Artículo
Comprando el artículo el PDF del mismo podrá ser descargado
Precio 19,34 €
Comprar ahora
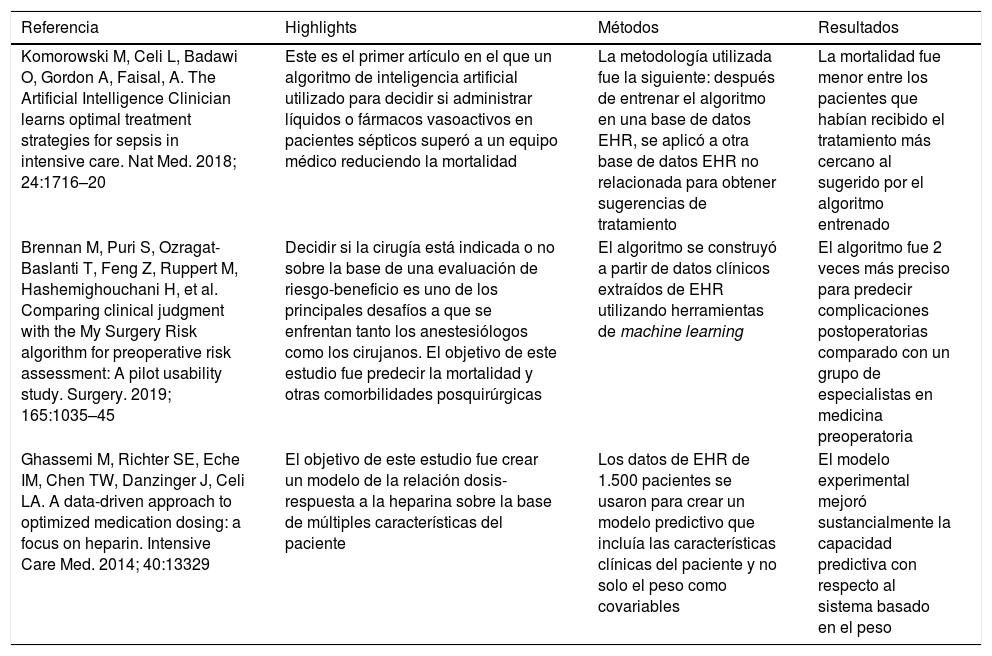
array:24 [ "pii" => "S0034935619301458" "issn" => "00349356" "doi" => "10.1016/j.redar.2019.07.004" "estado" => "S300" "fechaPublicacion" => "2019-12-01" "aid" => "1054" "copyright" => "Sociedad Española de Anestesiología, Reanimación y Terapéutica del Dolor" "copyrightAnyo" => "2019" "documento" => "simple-article" "crossmark" => 1 "subdocumento" => "cor" "cita" => "Rev Esp Anestesiol Reanim. 2019;66:555-8" "abierto" => array:3 [ "ES" => false "ES2" => false "LATM" => false ] "gratuito" => false "lecturas" => array:2 [ "total" => 76 "formatos" => array:2 [ "HTML" => 31 "PDF" => 45 ] ] "Traduccion" => array:1 [ "en" => array:19 [ "pii" => "S2341192919301714" "issn" => "23411929" "doi" => "10.1016/j.redare.2019.07.010" "estado" => "S300" "fechaPublicacion" => "2019-12-01" "aid" => "1054" "copyright" => "Sociedad Española de Anestesiología, Reanimación y Terapéutica del Dolor" "documento" => "simple-article" "crossmark" => 1 "subdocumento" => "cor" "cita" => "Revista Española de Anestesiología y Reanimación (English Version). 2019;66:555-8" "abierto" => array:3 [ "ES" => false "ES2" => false "LATM" => false ] "gratuito" => false "lecturas" => array:1 [ "total" => 0 ] "en" => array:10 [ "idiomaDefecto" => true "cabecera" => "<span class="elsevierStyleTextfn">Letter to the Director</span>" "titulo" => "<span class="elsevierStyleItalic">Big data</span> techniques for the secondary use of clinical data in the generation of medical knowledge. The MIMIC solution" "tienePdf" => "en" "tieneTextoCompleto" => "en" "paginas" => array:1 [ 0 => array:2 [ "paginaInicial" => "555" "paginaFinal" => "558" ] ] "titulosAlternativos" => array:1 [ "es" => array:1 [ "titulo" => "Técnicas Big data para el uso secundario de datos clínicos para la creación de conocimiento médico. La solución MIMIC" ] ] "contieneTextoCompleto" => array:1 [ "en" => true ] "contienePdf" => array:1 [ "en" => true ] "autores" => array:1 [ 0 => array:2 [ "autoresLista" => "X. Borrat, L.A. Celi, C. Ferrando" "autores" => array:3 [ 0 => array:2 [ "nombre" => "X." "apellidos" => "Borrat" ] 1 => array:2 [ "nombre" => "L.A." "apellidos" => "Celi" ] 2 => array:2 [ "nombre" => "C." "apellidos" => "Ferrando" ] ] ] ] ] "idiomaDefecto" => "en" "Traduccion" => array:1 [ "es" => array:9 [ "pii" => "S0034935619301458" "doi" => "10.1016/j.redar.2019.07.004" "estado" => "S300" "subdocumento" => "" "abierto" => array:3 [ "ES" => false "ES2" => false "LATM" => false ] "gratuito" => false "lecturas" => array:1 [ "total" => 0 ] "idiomaDefecto" => "es" "EPUB" => "https://multimedia.elsevier.es/PublicationsMultimediaV1/item/epub/S0034935619301458?idApp=UINPBA00004N" ] ] "EPUB" => "https://multimedia.elsevier.es/PublicationsMultimediaV1/item/epub/S2341192919301714?idApp=UINPBA00004N" "url" => "/23411929/0000006600000010/v1_201912172242/S2341192919301714/v1_201912172242/en/main.assets" ] ] "itemSiguiente" => array:19 [ "pii" => "S003493561930132X" "issn" => "00349356" "doi" => "10.1016/j.redar.2019.06.008" "estado" => "S300" "fechaPublicacion" => "2019-12-01" "aid" => "1053" "copyright" => "Sociedad Española de Anestesiología, Reanimación y Terapéutica del Dolor" "documento" => "simple-article" "crossmark" => 1 "subdocumento" => "cor" "cita" => "Rev Esp Anestesiol Reanim. 2019;66:558-61" "abierto" => array:3 [ "ES" => false "ES2" => false "LATM" => false ] "gratuito" => false "lecturas" => array:2 [ "total" => 110 "formatos" => array:2 [ "HTML" => 56 "PDF" => 54 ] ] "es" => array:11 [ "idiomaDefecto" => true "cabecera" => "<span class="elsevierStyleTextfn">CARTA AL DIRECTOR</span>" "titulo" => "Percepción del médico sobre las ayudas cognitivas para el manejo de la hipoxemia en anestesia y cuidados críticos" "tienePdf" => "es" "tieneTextoCompleto" => "es" "paginas" => array:1 [ 0 => array:2 [ "paginaInicial" => "558" "paginaFinal" => "561" ] ] "titulosAlternativos" => array:1 [ "en" => array:1 [ "titulo" => "Physician's perception of cognitive aids for hypoxemia management in anesthesia and critical care" ] ] "contieneTextoCompleto" => array:1 [ "es" => true ] "contienePdf" => array:1 [ "es" => true ] "resumenGrafico" => array:2 [ "original" => 0 "multimedia" => array:6 [ "identificador" => "fig0015" "etiqueta" => "Figura 3" "tipo" => "MULTIMEDIAFIGURA" "mostrarFloat" => true "mostrarDisplay" => false "figura" => array:1 [ 0 => array:4 [ "imagen" => "gr3.jpeg" "Alto" => 2345 "Ancho" => 2458 "Tamanyo" => 349642 ] ] ] ] "autores" => array:1 [ 0 => array:2 [ "autoresLista" => "J.M. Nieves Alonso, R.M. Méndez Hernández, F. Ramasco Rueda, A. Planas Roca" "autores" => array:4 [ 0 => array:2 [ "nombre" => "J.M." "apellidos" => "Nieves Alonso" ] 1 => array:2 [ "nombre" => "R.M." "apellidos" => "Méndez Hernández" ] 2 => array:2 [ "nombre" => "F." "apellidos" => "Ramasco Rueda" ] 3 => array:2 [ "nombre" => "A." "apellidos" => "Planas Roca" ] ] ] ] ] "idiomaDefecto" => "es" "Traduccion" => array:1 [ "en" => array:9 [ "pii" => "S2341192919301702" "doi" => "10.1016/j.redare.2019.06.009" "estado" => "S300" "subdocumento" => "" "abierto" => array:3 [ "ES" => false "ES2" => false "LATM" => false ] "gratuito" => false "lecturas" => array:1 [ "total" => 0 ] "idiomaDefecto" => "en" "EPUB" => "https://multimedia.elsevier.es/PublicationsMultimediaV1/item/epub/S2341192919301702?idApp=UINPBA00004N" ] ] "EPUB" => "https://multimedia.elsevier.es/PublicationsMultimediaV1/item/epub/S003493561930132X?idApp=UINPBA00004N" "url" => "/00349356/0000006600000010/v1_201912020617/S003493561930132X/v1_201912020617/es/main.assets" ] "itemAnterior" => array:19 [ "pii" => "S0034935619301574" "issn" => "00349356" "doi" => "10.1016/j.redar.2019.09.001" "estado" => "S300" "fechaPublicacion" => "2019-12-01" "aid" => "1062" "copyright" => "Sociedad Española de Anestesiología, Reanimación y Terapéutica del Dolor" "documento" => "simple-article" "crossmark" => 1 "subdocumento" => "cor" "cita" => "Rev Esp Anestesiol Reanim. 2019;66:554-5" "abierto" => array:3 [ "ES" => false "ES2" => false "LATM" => false ] "gratuito" => false "lecturas" => array:2 [ "total" => 97 "formatos" => array:2 [ "HTML" => 53 "PDF" => 44 ] ] "es" => array:10 [ "idiomaDefecto" => true "cabecera" => "<span class="elsevierStyleTextfn">Carta al Director</span>" "titulo" => "Comentarios al artículo «<span class="elsevierStyleItalic">Damage Control Resuscitation</span> en el paciente traumático»" "tienePdf" => "es" "tieneTextoCompleto" => "es" "paginas" => array:1 [ 0 => array:2 [ "paginaInicial" => "554" "paginaFinal" => "555" ] ] "titulosAlternativos" => array:1 [ "en" => array:1 [ "titulo" => "Article Comments «Damage Control Resuscitation in the Traumatic Patient»" ] ] "contieneTextoCompleto" => array:1 [ "es" => true ] "contienePdf" => array:1 [ "es" => true ] "autores" => array:1 [ 0 => array:2 [ "autoresLista" => "R. Navarro Suay, R. García Cañas, R. Tamburri Barain, E. López Soberón" "autores" => array:4 [ 0 => array:2 [ "nombre" => "R." "apellidos" => "Navarro Suay" ] 1 => array:2 [ "nombre" => "R." "apellidos" => "García Cañas" ] 2 => array:2 [ "nombre" => "R." "apellidos" => "Tamburri Barain" ] 3 => array:2 [ "nombre" => "E." "apellidos" => "López Soberón" ] ] ] ] ] "idiomaDefecto" => "es" "Traduccion" => array:1 [ "en" => array:9 [ "pii" => "S2341192919301684" "doi" => "10.1016/j.redare.2019.09.001" "estado" => "S300" "subdocumento" => "" "abierto" => array:3 [ "ES" => false "ES2" => false "LATM" => false ] "gratuito" => false "lecturas" => array:1 [ "total" => 0 ] "idiomaDefecto" => "en" "EPUB" => "https://multimedia.elsevier.es/PublicationsMultimediaV1/item/epub/S2341192919301684?idApp=UINPBA00004N" ] ] "EPUB" => "https://multimedia.elsevier.es/PublicationsMultimediaV1/item/epub/S0034935619301574?idApp=UINPBA00004N" "url" => "/00349356/0000006600000010/v1_201912020617/S0034935619301574/v1_201912020617/es/main.assets" ] "es" => array:14 [ "idiomaDefecto" => true "cabecera" => "<span class="elsevierStyleTextfn">Carta al Director</span>" "titulo" => "Big data techniques for the secondary use of clinical data in the generation of medical knowledge. The MIMIC solution" "tieneTextoCompleto" => true "saludo" => "<span class="elsevierStyleItalic">Estimado editor:</span>" "paginas" => array:1 [ 0 => array:2 [ "paginaInicial" => "555" "paginaFinal" => "558" ] ] "autores" => array:1 [ 0 => array:4 [ "autoresLista" => "X. Borrat, L.A. Celi, C. Ferrando" "autores" => array:3 [ 0 => array:4 [ "nombre" => "X." "apellidos" => "Borrat" "email" => array:1 [ 0 => "xborrat@clinic.cat" ] "referencia" => array:2 [ 0 => array:2 [ "etiqueta" => "<span class="elsevierStyleSup">a</span>" "identificador" => "aff0005" ] 1 => array:2 [ "etiqueta" => "<span class="elsevierStyleSup">*</span>" "identificador" => "cor0005" ] ] ] 1 => array:3 [ "nombre" => "L.A." "apellidos" => "Celi" "referencia" => array:2 [ 0 => array:2 [ "etiqueta" => "<span class="elsevierStyleSup">b</span>" "identificador" => "aff0010" ] 1 => array:2 [ "etiqueta" => "<span class="elsevierStyleSup">c</span>" "identificador" => "aff0015" ] ] ] 2 => array:3 [ "nombre" => "C." "apellidos" => "Ferrando" "referencia" => array:2 [ 0 => array:2 [ "etiqueta" => "<span class="elsevierStyleSup">d</span>" "identificador" => "aff0020" ] 1 => array:2 [ "etiqueta" => "<span class="elsevierStyleSup">e</span>" "identificador" => "aff0025" ] ] ] ] "afiliaciones" => array:5 [ 0 => array:3 [ "entidad" => "Servicio de Anestesiología y Reanimación, Hospital Clínic de Barcelona, Barcelona, España" "etiqueta" => "a" "identificador" => "aff0005" ] 1 => array:3 [ "entidad" => "Laboratory for Computational Physiology, Institute for Medical Engineering and Science, Massachusetts Institute of Technology, Cambridge, Estados Unidos" "etiqueta" => "b" "identificador" => "aff0010" ] 2 => array:3 [ "entidad" => "Division of Pulmonary, Critical Care and Sleep Medicine, Beth Israel Deaconess Medical Center, Boston, Estados Unidos" "etiqueta" => "c" "identificador" => "aff0015" ] 3 => array:3 [ "entidad" => "Servicio de Anestesiología y Reanimación, Hospital Clínic, Institut d’Investigació August Pi i Sunyer, Barcelona, España" "etiqueta" => "d" "identificador" => "aff0020" ] 4 => array:3 [ "entidad" => "CIBER de Enfermedades Respiratorias, Instituto de Salud Carlos III, Madrid, España" "etiqueta" => "e" "identificador" => "aff0025" ] ] "correspondencia" => array:1 [ 0 => array:3 [ "identificador" => "cor0005" "etiqueta" => "⁎" "correspondencia" => "Autor para correspondencia." ] ] ] ] "titulosAlternativos" => array:1 [ "en" => array:1 [ "titulo" => "Técnicas <span class="elsevierStyleItalic">Big data</span> para el uso secundario de datos clínicos para la creación de conocimiento medico. La solución MIMIC" ] ] "textoCompleto" => "<span class="elsevierStyleSections"><p id="par0005" class="elsevierStylePara elsevierViewall">Muchos aspectos de la vida moderna están cambiando debido al <span class="elsevierStyleItalic">big data</span> y el aprendizaje automático o <span class="elsevierStyleItalic">machine learning</span>. Netflix sabe qué películas les gusta ver a la gente, y Google sabe qué anuncios mostrar a los navegadores en función de su historial de búsqueda. Grandes cantidades de información personal de redes sociales, historiales de búsqueda y otros tipos de publicación se digitalizan y cargan en la nube, dejando un rastro de datos que ha llevado al desarrollo de un nuevo campo, el <span class="elsevierStyleItalic">Data Science</span> o ciencia de datos. La ciencia de datos es el resultado de la unificación de herramientas estadísticas, bases de datos y programación para interpretar datos con el fin de mejorar la predicción de eventos y la toma de decisiones. <span class="elsevierStyleItalic">Big data</span> es el sustrato y dio origen al campo de la ciencia de datos, y el término se usa cuando la cantidad de datos es tal que su tamaño ya no se mide en términos de volumen, sino en la velocidad de creación. Mientras tanto, el campo de la medicina ha experimentado una revolución digital en los últimos años, con la creación de extensas bases de datos basadas en registros electrónicos de salud (EHR) y otras aplicaciones utilizadas en servicios transversales, como laboratorios, servicios de diagnóstico por imágenes y farmacia hospitalaria. Estas otras aplicaciones para servicios transversales se conocen como aplicaciones departamentales. En los últimos 10 años, se han implementado EHR y aplicaciones departamentales en casi todos los hospitales grandes de España, y toda la información ahora se almacena en formato digital en lugar de en papel. Todas estas cantidades masivas de datos, más allá de su uso rutinario en gestión o a nivel asistencial, podrían ser utilizadas para ampliar el conocimiento médico.</p><span id="sec0005" class="elsevierStyleSection elsevierViewall"><span class="elsevierStyleSectionTitle" id="sect0005">Técnicas de <span class="elsevierStyleItalic">Big Data</span> (Preparación de un <span class="elsevierStyleItalic">DataMart</span> -Extracción-Análisis) para la generación de conocimiento a partir de EHR</span><p id="par0010" class="elsevierStylePara elsevierViewall">Los datos de EHR pueden tener un uso primario de salud (uso asistencial) o un uso secundario de investigación. Los datos médicos recopilados de la práctica clínica deben pasar por varias etapas antes de que puedan usarse para la investigación. Primero, se deben crear unas bases de datos con contenido clínicamente significativo, llamadas <span class="elsevierStyleItalic">DataMart</span> (ver solución MIMIC)<a class="elsevierStyleCrossRef" href="#bib0005"><span class="elsevierStyleSup">1</span></a> a partir de las bases de datos EHR. Brevemente, un <span class="elsevierStyleItalic">DataMart</span> consiste en una selección y reorganización de datos provenientes de la base de datos EHR sin procesar para incluir datos relevantes con un propósito específico. Por ejemplo, si el foco de la actividad de investigación son los resultados clínicos del paciente relacionados con el entorno clínico general del paciente, debemos incluir signos vitales, diagnósticos principales, datos de dispositivos de soporte, medicamentos, pruebas de laboratorio y también los resultados clínicos del paciente, entre otros. Pero, al mismo tiempo, deben excluirse muchos otros campos, como la información operativa u otros que no están relacionados con el enfoque de investigación. Una de las principales ventajas de los <span class="elsevierStyleItalic">DataMarts</span> es la accesibilidad de los datos con consultas simples para obtener un conjunto de datos <span class="elsevierStyleItalic">(data-sets)</span>.</p><p id="par0015" class="elsevierStylePara elsevierViewall">En segundo lugar, el <span class="elsevierStyleItalic">data-set</span> (un subconjunto de datos preparado para el análisis) debe crearse extrayendo los datos seleccionados de los casos incluidos en el <span class="elsevierStyleItalic">DataMart</span> (covariables de contexto). Por ejemplo, si queremos predecir, antes de la admisión, la duración de la estancia en el hospital en cirugía electiva, debemos incluir en el conjunto de datos todas las variables que pueden estar relacionadas con la duración de la estancia, como las comorbilidades, el tipo de cirugía, medicación regular y duración real de la hospitalización. Tener un <span class="elsevierStyleItalic">DataMart</span> disponible permite que esta fase se pueda hacer fácilmente en minutos usando un lenguaje de base de datos común como SQL. Será posible cambiar las variables de forma ágil, así como filtrar casos y, por lo tanto, ajustar el conjunto de datos a la pregunta clínica deseada<a class="elsevierStyleCrossRef" href="#bib0010"><span class="elsevierStyleSup">2</span></a>.</p><p id="par0020" class="elsevierStylePara elsevierViewall">Finalmente, el conjunto de datos debe ser analizado. El mejor método para analizar grandes conjuntos de datos es el <span class="elsevierStyleItalic">machine learning</span>, aunque cualquier tipo de prueba estadística, como la visualización de datos, los análisis descriptivos y las pruebas de hipótesis, se pueden aplicar al <span class="elsevierStyleItalic">data-set</span>. El <span class="elsevierStyleItalic">machine learning</span> es un término utilizado para describir un programa que aprende tareas como la predicción, clasificación y optimización en función de los datos proporcionados<a class="elsevierStyleCrossRef" href="#bib0015"><span class="elsevierStyleSup">3</span></a>. Esta es una definición muy amplia e incluye un espectro de algoritmos, que van desde aquellos que requieren una intervención humana considerable para elegir el modelo y las variables utilizadas (por ejemplo, regresión múltiple), hasta los modelos más sofisticados en los que se minimiza el requisito de intervención humana como las redes de confrontación generativas (GAN), donde el algoritmo deriva su representación de la verdad fundamental a partir de los datos proporcionados, con poco aporte humano. El aprendizaje automático es interesante porque, dado un gran número de casos y una población altamente heterogénea, puede crear modelos de predicción y clasificación aplicables tanto a poblaciones enteras como a individuos.</p></span><span id="sec0010" class="elsevierStyleSection elsevierViewall"><span class="elsevierStyleSectionTitle" id="sect0010">Medicina de precisión como alternativa a los ensayos controlados aleatorizados (ECA) para la toma de decisiones</span><p id="par0025" class="elsevierStylePara elsevierViewall">Los médicos buscan la evidencia en ensayos clínicos y estudios observacionales, pero muchas veces no existe la evidencia necesaria de manera específica. La idea de utilizar los datos de EHR generados a partir de la práctica clínica para fines de investigación es muy atractiva. A pesar de ser observacionales y retrospectivos, estos estudios tienen muchas ventajas sobre los ensayos controlados aleatorizados (ECA). Su naturaleza intrínseca facilita la generación de soluciones altamente individualizadas para un problema clínico dado. Los estudios basados en datos de EHR permiten a los investigadores investigar problemas clínicos globales y locales; en otras palabras, para resolver las preguntas clínicas del día a día rápidamente, sin riesgo para los pacientes, mientras se minimizan el coste. Además, gracias a la disponibilidad de una gran cantidad de datos y a los modelos predictivos basados en el <span class="elsevierStyleItalic">machine learning</span>, el uso secundario de los datos de EHR puede predecir la respuesta al tratamiento en todo tipo de pacientes, sin excluir grupos de pacientes, ya sea intencionalmente o no, por ejemplo, pacientes embarazadas, niños, etc. La inclusión de todos los tipos de pacientes con su enfermedad detallada «biométrica» en una gran muestra de población permite a los investigadores crear modelos que se ajustan a múltiples capas de confusión. Esto difiere del paradigma ECA de estandarización, en la medida en que nos acerca a la medicina de precisión individualizada. Además, la verdad fundamental cambia constantemente en medicina haciendo que incluso estudios perfectamente realizados y aplicables de hace unos años ya no puedan aplicarse hoy en día, ya sea por haberse incorporado nuevas pruebas diagnósticas, nuevas terapias o simplemente por los cambios demográficos poblacionales. El proceso de usar continuamente datos clínicos recopilados de forma rutinaria para actualizar el conocimiento médico y guiar la práctica clínica, vinculando íntimamente la generación de conocimiento y la prestación de atención, representa un nuevo paradigma que promete acercarnos a una verdadera atención basada en la evidencia<a class="elsevierStyleCrossRef" href="#bib0020"><span class="elsevierStyleSup">4</span></a>. Algunos ejemplos de uso de esta metodología los mostramos en la <a class="elsevierStyleCrossRef" href="#tbl0005">tabla 1</a>.</p><elsevierMultimedia ident="tbl0005"></elsevierMultimedia></span><span id="sec0015" class="elsevierStyleSection elsevierViewall"><span class="elsevierStyleSectionTitle" id="sect0015">Dificultades para extraer información de EHR</span><p id="par0030" class="elsevierStylePara elsevierViewall">El programa informático con el que los médicos acceden a las bases de datos de EHR está diseñado para la documentación individual con fines asistenciales y administrativos, incluida la facturación. Más importante aún, los datos generalmente están bloqueados en algún formato patentado y, por lo tanto, se necesita un software adicional para extraer y reunir información de grupos de pacientes con fines de investigación. Además, el esquema de datos de las bases de datos de EHR existentes difiere de una institución a otra, lo que dificulta la integración y la armonización.</p><p id="par0035" class="elsevierStylePara elsevierViewall">Esto crea una serie de problemas para los investigadores clínicos, como son:</p><p id="par0040" class="elsevierStylePara elsevierViewall">1.-Incapacidad para extraer datos de grandes grupos de pacientes. Cuando se necesitan datos de cientos o miles de pacientes, no es posible crear un conjunto de datos mediante la revisión de registros de pacientes uno a uno.</p><p id="par0045" class="elsevierStylePara elsevierViewall">2.- Falta de priorización por parte de los departamentos de Tecnología de la Información (TI), generando retrasos significativos en el desarrollo del proyecto.</p><p id="par0050" class="elsevierStylePara elsevierViewall">3.- Los datos no se pueden compartir. La naturaleza propietaria de los sistemas de información y las diferencias en la estructura de la base de datos entre hospitales hacen que sea difícil generar grandes repositorios a partir de datos compartidos. El intercambio de datos es un requisito previo para crear modelos generalizables e identificar las mejores prácticas en la aplicación del <span class="elsevierStyleItalic">machine learning</span> a los datos generados de la atención médica.</p></span><span id="sec0020" class="elsevierStyleSection elsevierViewall"><span class="elsevierStyleSectionTitle" id="sect0020">La solución MIMIC. ¿Podría ser replicada en España?</span><p id="par0055" class="elsevierStylePara elsevierViewall">Uno de los ámbitos de la atención médica con mayor capacidad para generar datos son las Unidades de Cuidados Intensivos (UCI). Los datos específicos del paciente relacionados con los signos vitales y los sistemas de monitorización de soporte vital, terapia farmacológica, hallazgos de laboratorio y pruebas de imágenes, etc., generan más de 10 veces la tasa de datos que pacientes ingresados en una sala de hospitalización convencional. Debido a esto, las UCI tienen el mayor potencial para generar y validar nuevos conocimientos en el campo de la atención médica.</p><p id="par0060" class="elsevierStylePara elsevierViewall">MIMIC (<span class="elsevierStyleItalic">Medical Information Mart for Intensive Care</span>) es un proyecto creado y mantenido por el Laboratorio de Fisiología Computacional (LCP) del MIT (Instituto de Tecnología de Massachusetts)<a class="elsevierStyleCrossRef" href="#bib0005"><span class="elsevierStyleSup">1</span></a>. A día de hoy, contiene datos de salud anonimizados de más de 60.000 pacientes de cuidados críticos. La base de datos tipo <span class="elsevierStyleItalic">DataMart</span> disponible públicamente consiste en tablas relacionales dentro de una estructura que es clínicamente intuitiva, brindando a los investigadores que están familiarizados con la UCI la autonomía y la agilidad que necesitan para construir <span class="elsevierStyleItalic">data-sets</span> dinámicos en cuestión de segundos<a class="elsevierStyleCrossRef" href="#bib0025"><span class="elsevierStyleSup">5</span></a>. La base de datos contiene datos demográficos del paciente, signos vitales, resultados de laboratorio, procedimientos, diagnósticos, tratamiento farmacológico, notas clínicas e información de supervivencia extraída tanto de EHR como de diferentes aplicaciones departamentales. El código fuente abierto y la estructura de acceso abierto de la base de datos es el resultado de un gran proyecto de investigación colaborativa que involucra a académicos y socios de la industria con fondos del gobierno. Los usuarios pueden aportar definiciones de conceptos y códigos para extraerlos, y también pueden usar el código existente creado por los más de 12.000 usuarios autorizados de MIMIC. El LCP también promueve la reproducibilidad al exigir a los usuarios de MIMIC que compartan sus métodos con suficiente detalle, además de los códigos y consultas empleados durante su análisis una vez que se publican sus documentos. La falta de reproducibilidad de los estudios está causando una creciente preocupación en la comunidad científica<a class="elsevierStyleCrossRef" href="#bib0030"><span class="elsevierStyleSup">6</span></a>, y este nuevo enfoque no solo facilita la replicación de los estudios, sino que permite a los investigadores aprovechar la investigación de los demás. De hecho, hasta la fecha, la población de la UCI MIMIC es la cohorte más ampliamente estudiada en pacientes críticos. El mismo grupo MIT promueve eventos (llamados <span class="elsevierStyleItalic">datathons</span>) en los que los médicos y los científicos de datos trabajan juntos usando la base de datos MIMIC para resolver problemas clínicos. A partir de estos eventos, y de otros grupos internacionales que trabajan con datos MIMIC, se generan múltiples publicaciones científicas basadas en el análisis de datos<a class="elsevierStyleCrossRef" href="#bib0020"><span class="elsevierStyleSup">4</span></a>.</p><p id="par0065" class="elsevierStylePara elsevierViewall">Hay otras otras iniciativas en la estandarización de datos de salud que vale la pena mencionar. Una de ellas es el Observational Health Data Sciences and Informatics (OHDSI), un consorcio internacional que está construyendo un esquema de base de datos de salud llamado OMOP-CDM (Modelo de datos comunes de asociación de resultados médicos observacionales)<a class="elsevierStyleCrossRef" href="#bib0035"><span class="elsevierStyleSup">7</span></a>. Este proyecto tiene como objetivo transformar los datos contenidos dentro de EHR en un formato común y una representación común (terminologías, vocabularios, esquemas de codificación). El consorcio también reúne y proporciona una biblioteca de herramientas analíticas que se han desarrollado en base a un formato común. Finalmente, el HL7® Fast Healthcare Interoperability Resources (FHIR®) que es un marco estándar que describe formatos y elementos de datos, así como una interfaz de programación de aplicaciones (API) para sistemas de información, incluidos teléfonos móviles y aplicaciones basadas en la web<a class="elsevierStyleCrossRef" href="#bib0040"><span class="elsevierStyleSup">8</span></a>.</p><p id="par0070" class="elsevierStylePara elsevierViewall">En resumen, ahora tenemos la oportunidad de complementar y llenar los vacíos de información en los sistemas de conocimiento médico que no son abordados por los ECA y la investigación científica básica. La comprensión de la ciencia de datos, combinada con su experiencia específica en el campo, permitirá a los médicos explotar los datos generados en su entorno de trabajo. Los proyectos como MIMIC deben replicarse en España para comprender mejor la salud y la enfermedad entre la población española.</p></span></span>" "textoCompletoSecciones" => array:1 [ "secciones" => array:5 [ 0 => array:2 [ "identificador" => "sec0005" "titulo" => "Técnicas de Big Data (Preparación de un DataMart -Extracción-Análisis) para la generación de conocimiento a partir de EHR" ] 1 => array:2 [ "identificador" => "sec0010" "titulo" => "Medicina de precisión como alternativa a los ensayos controlados aleatorizados (ECA) para la toma de decisiones" ] 2 => array:2 [ "identificador" => "sec0015" "titulo" => "Dificultades para extraer información de EHR" ] 3 => array:2 [ "identificador" => "sec0020" "titulo" => "La solución MIMIC. ¿Podría ser replicada en España?" ] 4 => array:1 [ "titulo" => "Bibliografía" ] ] ] "pdfFichero" => "main.pdf" "tienePdf" => true "multimedia" => array:1 [ 0 => array:8 [ "identificador" => "tbl0005" "etiqueta" => "Tabla 1" "tipo" => "MULTIMEDIATABLA" "mostrarFloat" => true "mostrarDisplay" => false "detalles" => array:1 [ 0 => array:3 [ "identificador" => "at1" "detalle" => "Tabla " "rol" => "short" ] ] "tabla" => array:1 [ "tablatextoimagen" => array:1 [ 0 => array:2 [ "tabla" => array:1 [ 0 => """ <table border="0" frame="\n \t\t\t\t\tvoid\n \t\t\t\t" class=""><thead title="thead"><tr title="table-row"><th class="td" title="\n \t\t\t\t\ttable-head\n \t\t\t\t " align="left" valign="\n \t\t\t\t\ttop\n \t\t\t\t" scope="col" style="border-bottom: 2px solid black">Referencia \t\t\t\t\t\t\n \t\t\t\t\t\t</th><th class="td" title="\n \t\t\t\t\ttable-head\n \t\t\t\t " align="left" valign="\n \t\t\t\t\ttop\n \t\t\t\t" scope="col" style="border-bottom: 2px solid black">Highlights \t\t\t\t\t\t\n \t\t\t\t\t\t</th><th class="td" title="\n \t\t\t\t\ttable-head\n \t\t\t\t " align="left" valign="\n \t\t\t\t\ttop\n \t\t\t\t" scope="col" style="border-bottom: 2px solid black">Métodos \t\t\t\t\t\t\n \t\t\t\t\t\t</th><th class="td" title="\n \t\t\t\t\ttable-head\n \t\t\t\t " align="left" valign="\n \t\t\t\t\ttop\n \t\t\t\t" scope="col" style="border-bottom: 2px solid black">Resultados \t\t\t\t\t\t\n \t\t\t\t\t\t</th></tr></thead><tbody title="tbody"><tr title="table-row"><td class="td-with-role" title="\n \t\t\t\t\ttable-entry\n \t\t\t\t ; entry_with_role_rowhead " align="left" valign="\n \t\t\t\t\ttop\n \t\t\t\t">Komorowski M, Celi L, Badawi O, Gordon A, Faisal, A. The Artificial Intelligence Clinician learns optimal treatment strategies for sepsis in intensive care. Nat Med. 2018; 24:1716–20 \t\t\t\t\t\t\n \t\t\t\t</td><td class="td" title="\n \t\t\t\t\ttable-entry\n \t\t\t\t " align="left" valign="\n \t\t\t\t\ttop\n \t\t\t\t">Este es el primer artículo en el que un algoritmo de inteligencia artificial utilizado para decidir si administrar líquidos o fármacos vasoactivos en pacientes sépticos superó a un equipo médico reduciendo la mortalidad \t\t\t\t\t\t\n \t\t\t\t</td><td class="td" title="\n \t\t\t\t\ttable-entry\n \t\t\t\t " align="left" valign="\n \t\t\t\t\ttop\n \t\t\t\t">La metodología utilizada fue la siguiente: después de entrenar el algoritmo en una base de datos EHR, se aplicó a otra base de datos EHR no relacionada para obtener sugerencias de tratamiento \t\t\t\t\t\t\n \t\t\t\t</td><td class="td" title="\n \t\t\t\t\ttable-entry\n \t\t\t\t " align="left" valign="\n \t\t\t\t\ttop\n \t\t\t\t">La mortalidad fue menor entre los pacientes que habían recibido el tratamiento más cercano al sugerido por el algoritmo entrenado \t\t\t\t\t\t\n \t\t\t\t</td></tr><tr title="table-row"><td class="td-with-role" title="\n \t\t\t\t\ttable-entry\n \t\t\t\t ; entry_with_role_rowhead " align="left" valign="\n \t\t\t\t\ttop\n \t\t\t\t">Brennan M, Puri S, Ozragat-Baslanti T, Feng Z, Ruppert M, Hashemighouchani H, et al. Comparing clinical judgment with the My Surgery Risk algorithm for preoperative risk assessment: A pilot usability study. Surgery. 2019; 165:1035–45 \t\t\t\t\t\t\n \t\t\t\t</td><td class="td" title="\n \t\t\t\t\ttable-entry\n \t\t\t\t " align="left" valign="\n \t\t\t\t\ttop\n \t\t\t\t">Decidir si la cirugía está indicada o no sobre la base de una evaluación de riesgo-beneficio es uno de los principales desafíos a que se enfrentan tanto los anestesiólogos como los cirujanos. El objetivo de este estudio fue predecir la mortalidad y otras comorbilidades posquirúrgicas \t\t\t\t\t\t\n \t\t\t\t</td><td class="td" title="\n \t\t\t\t\ttable-entry\n \t\t\t\t " align="left" valign="\n \t\t\t\t\ttop\n \t\t\t\t">El algoritmo se construyó a partir de datos clínicos extraídos de EHR utilizando herramientas de <span class="elsevierStyleItalic">machine learning</span> \t\t\t\t\t\t\n \t\t\t\t</td><td class="td" title="\n \t\t\t\t\ttable-entry\n \t\t\t\t " align="left" valign="\n \t\t\t\t\ttop\n \t\t\t\t">El algoritmo fue 2 veces más preciso para predecir complicaciones postoperatorias comparado con un grupo de especialistas en medicina preoperatoria \t\t\t\t\t\t\n \t\t\t\t</td></tr><tr title="table-row"><td class="td-with-role" title="\n \t\t\t\t\ttable-entry\n \t\t\t\t ; entry_with_role_rowhead " align="left" valign="\n \t\t\t\t\ttop\n \t\t\t\t">Ghassemi M, Richter SE, Eche IM, Chen TW, Danzinger J, Celi LA. A data-driven approach to optimized medication dosing: a focus on heparin. Intensive Care Med. 2014; 40:13329 \t\t\t\t\t\t\n \t\t\t\t</td><td class="td" title="\n \t\t\t\t\ttable-entry\n \t\t\t\t " align="left" valign="\n \t\t\t\t\ttop\n \t\t\t\t">El objetivo de este estudio fue crear un modelo de la relación dosis-respuesta a la heparina sobre la base de múltiples características del paciente \t\t\t\t\t\t\n \t\t\t\t</td><td class="td" title="\n \t\t\t\t\ttable-entry\n \t\t\t\t " align="left" valign="\n \t\t\t\t\ttop\n \t\t\t\t">Los datos de EHR de 1.500 pacientes se usaron para crear un modelo predictivo que incluía las características clínicas del paciente y no solo el peso como covariables \t\t\t\t\t\t\n \t\t\t\t</td><td class="td" title="\n \t\t\t\t\ttable-entry\n \t\t\t\t " align="left" valign="\n \t\t\t\t\ttop\n \t\t\t\t">El modelo experimental mejoró sustancialmente la capacidad predictiva con respecto al sistema basado en el peso \t\t\t\t\t\t\n \t\t\t\t</td></tr></tbody></table> """ ] "imagenFichero" => array:1 [ 0 => "xTab2173663.png" ] ] ] ] "descripcion" => array:1 [ "es" => "<p id="spar0005" class="elsevierStyleSimplePara elsevierViewall">Ejemplos del uso secundario de datos clínicos</p>" ] ] ] "bibliografia" => array:2 [ "titulo" => "Bibliografía" "seccion" => array:1 [ 0 => array:2 [ "identificador" => "bibs0015" "bibliografiaReferencia" => array:8 [ 0 => array:3 [ "identificador" => "bib0005" "etiqueta" => "1" "referencia" => array:1 [ 0 => array:1 [ "referenciaCompleta" => "MIMIC III project [consultado 1 May 2019]. Disponible en: <a target="_blank" href="https://mimic.physionet.org/">https://mimic.physionet.org</a>" ] ] ] 1 => array:3 [ "identificador" => "bib0010" "etiqueta" => "2" "referencia" => array:1 [ 0 => array:2 [ "contribucion" => array:1 [ 0 => array:2 [ "titulo" => "Big data analysis and machine learning in intensive care units" "autores" => array:1 [ 0 => array:2 [ "etal" => false "autores" => array:3 [ 0 => "A. Nuñez Reiz" 1 => "M.A. Armengol de la Hoz" 2 => "M. Sanchez Garcia" ] ] ] ] ] "host" => array:1 [ 0 => array:2 [ "doi" => "10.1016/j.medin.2018.10.007" "Revista" => array:6 [ "tituloSerie" => "Med Intensiva." "fecha" => "2019" "volumen" => "43" "paginaInicial" => "416" "paginaFinal" => "426" "link" => array:1 [ 0 => array:2 [ "url" => "https://www.ncbi.nlm.nih.gov/pubmed/30591356" "web" => "Medline" ] ] ] ] ] ] ] ] 2 => array:3 [ "identificador" => "bib0015" "etiqueta" => "3" "referencia" => array:1 [ 0 => array:2 [ "contribucion" => array:1 [ 0 => array:2 [ "titulo" => "Big data and machine learning in health care" "autores" => array:1 [ 0 => array:2 [ "etal" => false "autores" => array:2 [ 0 => "A.L. Beam" 1 => "I.S. Kohane" ] ] ] ] ] "host" => array:1 [ 0 => array:2 [ "doi" => "10.1001/jama.2017.18391" "Revista" => array:7 [ "tituloSerie" => "JAMA." "fecha" => "2018" "volumen" => "319" "paginaInicial" => "1317" "paginaFinal" => "1318" "link" => array:1 [ 0 => array:2 [ "url" => "https://www.ncbi.nlm.nih.gov/pubmed/29532063" "web" => "Medline" ] ] "itemHostRev" => array:3 [ "pii" => "S0049017211001454" "estado" => "S300" "issn" => "00490172" ] ] ] ] ] ] ] 3 => array:3 [ "identificador" => "bib0020" "etiqueta" => "4" "referencia" => array:1 [ 0 => array:2 [ "contribucion" => array:1 [ 0 => array:2 [ "titulo" => "Critical care, critical data" "autores" => array:1 [ 0 => array:2 [ "etal" => false "autores" => array:3 [ 0 => "C.V. Cosgriff" 1 => "L.A. Celi" 2 => "D.J. Stone" ] ] ] ] ] "host" => array:1 [ 0 => array:1 [ "Revista" => array:3 [ "tituloSerie" => "Biomed Eng Comput Biol." "fecha" => "2019" "volumen" => "10" ] ] ] ] ] ] 4 => array:3 [ "identificador" => "bib0025" "etiqueta" => "5" "referencia" => array:1 [ 0 => array:2 [ "contribucion" => array:1 [ 0 => array:2 [ "titulo" => "The MIMIC Code Repository: enabling reproducibility in critical care research" "autores" => array:1 [ 0 => array:2 [ "etal" => false "autores" => array:4 [ 0 => "A.E. Johnson" 1 => "D.J. Stone" 2 => "L.A. Celi" 3 => "T.J. Pollard" ] ] ] ] ] "host" => array:1 [ 0 => array:2 [ "doi" => "10.1093/jamia/ocx084" "Revista" => array:6 [ "tituloSerie" => "J Am Med Inform Assoc." "fecha" => "2018" "volumen" => "25" "paginaInicial" => "32" "paginaFinal" => "39" "link" => array:1 [ 0 => array:2 [ "url" => "https://www.ncbi.nlm.nih.gov/pubmed/29036464" "web" => "Medline" ] ] ] ] ] ] ] ] 5 => array:3 [ "identificador" => "bib0030" "etiqueta" => "6" "referencia" => array:1 [ 0 => array:1 [ "referenciaCompleta" => "Challenges in irreproducible research [consultado 1 May 2019]. Disponible en: <a target="_blank" href="http://www.nature.com/collections/prbfkwmwvz%0A">www.nature.com/collections/prbfkwmwvz%0A</a>" ] ] ] 6 => array:3 [ "identificador" => "bib0035" "etiqueta" => "7" "referencia" => array:1 [ 0 => array:1 [ "referenciaCompleta" => "OMOP Common Data Model [consultado 4 Jul 2019]. Disponible en: <a target="_blank" href="https://www.ohdsi.org/data-standardization/the-common-data-model">https://www.ohdsi.org/data-standardization/the-common-data-model</a>" ] ] ] 7 => array:3 [ "identificador" => "bib0040" "etiqueta" => "8" "referencia" => array:1 [ 0 => array:1 [ "referenciaCompleta" => "FHIR Infrastructure. Background [consultado 4 Jul 2019]. Disponible en: <a target="_blank" href="http://www.hl7.org/Special/committees/fiwg/overview.cfm">http://www.hl7.org/Special/committees/fiwg/overview.cfm</a>" ] ] ] ] ] ] ] ] "idiomaDefecto" => "es" "url" => "/00349356/0000006600000010/v1_201912020617/S0034935619301458/v1_201912020617/es/main.assets" "Apartado" => array:4 [ "identificador" => "78500" "tipo" => "SECCION" "es" => array:2 [ "titulo" => "Cartas al Editor" "idiomaDefecto" => true ] "idiomaDefecto" => "es" ] "PDF" => "https://static.elsevier.es/multimedia/00349356/0000006600000010/v1_201912020617/S0034935619301458/v1_201912020617/es/main.pdf?idApp=UINPBA00004N&text.app=https://www.elsevier.es/" "EPUB" => "https://multimedia.elsevier.es/PublicationsMultimediaV1/item/epub/S0034935619301458?idApp=UINPBA00004N" ]
Consulte los artículos y contenidos publicados en este medio, además de los e-sumarios de las revistas científicas en el mismo momento de publicación
Esté informado en todo momento gracias a las alertas y novedades
Acceda a promociones exclusivas en suscripciones, lanzamientos y cursos acreditados
La Revista Española de Anestesiología y Reanimación (REDAR) es el órgano científico de la Sociedad Española de Anestesiología, Reanimación y Terapéutica del Dolor. Esta es una revista mensual (10 números) que publica artículos científicos de todas las áreas que abarca la especialidad: anestesia clínica, reanimación-medicina intensiva y cuidados críticos, diagnóstico y tratamiento del dolor agudo y crónico, urgencias y emergencias, así como trabajos de ciencias básicas y relacionadas. La REDAR acepta trabajos tanto en español como en inglés.

Web of Science, Emerging Sources Citation Index (WoS, Clarivate), PubMed/Medline, Scopus, EMBASE, IBECS
Ver másEl factor de impacto mide la media del número de citaciones recibidas en un año por trabajos publicados en la publicación durante los dos años anteriores.
© Clarivate Analytics, Journal Citation Reports 2022
SJR es una prestigiosa métrica basada en la idea de que todas las citaciones no son iguales. SJR usa un algoritmo similar al page rank de Google; es una medida cuantitativa y cualitativa al impacto de una publicación.
Ver másSNIP permite comparar el impacto de revistas de diferentes campos temáticos, corrigiendo las diferencias en la probabilidad de ser citado que existe entre revistas de distintas materias.
Ver más
¿Es usted profesional sanitario apto para prescribir o dispensar medicamentos?
Are you a health professional able to prescribe or dispense drugs?
Você é um profissional de saúde habilitado a prescrever ou dispensar medicamentos