Introducción
La microbiología clínica en nuestro país, con gran tradición, se ha ido asentando en los últimos 40 años. En la actualidad es muy útil, tiene prestigio y gran producción científica. De vez en cuando se siente la necesidad de realizar una revisión científico-filosófica de su situación presente y futuro inmediato. En ese contexto creemos que se planteó el diseño de esta monografía, y así lo hemos considerado al escribir este artículo. Los temas relacionados con la identificación bacteriana ocupan muchos libros, manuales y miles de artículos, tanto en su relación con los patógenos humanos como en los asociados a otras disciplinas, por lo que sólo haremos un repaso sintético del tema, entremezclando algunas opiniones, probablemente compartidas por la mayoría de nosotros, derivadas de nuestro quehacer diario.
Taxonomía bacteriana
El asignar nombres a las bacterias se basa en la taxonomía, que es la ciencia de clasificación biológica que agrupa organismos según sus afinidades o similitudes. En ella se incluye tanto la identificación como la nomenclatura. Los elementos básicos de los caracteres taxonómicos son diversos: morfología, fisiología, bioquímica, ecología, biología molecular y genética. Con todos ellos se compila una serie de datos que llevan a la caracterización, clasificación y nomenclatura. Dos son los principales esquemas de clasificación: uno basado en las características observables de un organismo dado (fenotípica), y la que tiene en cuenta las relaciones de evolución ancestral de los seres vivos, en nuestro caso las bacterias (filogenética).
Las especies bacterianas son grupos de cepas (clones) con muchas características en común, y notables diferencias con otras especies; las taxoespecies presentan una gran similitud fenotípica; las especies genómicas un alto porcentaje común en las bases de su ADN, y las genotípicas gran homología en las secuencias de sus genes.
Los caracteres fenotípicos se extraen de la morfología bacteriana (tamaño, aspecto, inclusiones, esporas, cápsulas, flagelos, tipo de tinción), aspecto en los medios de cultivo (morfología, márgenes, elevación, superficie, textura, opacidad, pigmentación), propiedades fisiológicas (margen de crecimiento a distintas temperaturas, pH y cloruro sódico, y atmósfera de O2 libre), propiedades bioquímicas (metabolismo de nitrógeno, utilización de hidratos de carbono, degradación de macromoléculas, tipo de enzimas respiratorias), necesidades nutricionales, características quimiotaxonómicas (tipo de pared celular, membrana celular, proteínas, lipopolisacáridos), inhibición por diversas sustancias (colorantes, antibióticos, fagos, bacteriocinas), pruebas serológicas, y genómica (porcentaje de moles de G + C, hibridación con sondas de ADN).
La taxonomía clásica y tradicional se ha basado en las características morfológicas, comportamiento en los medios de cultivo, propiedades bioquímicas, fisiológicas y nutricionales. En la actualidad es preferible por su exactitud la taxonomía molecular, basada en la ubicuidad del ARN ribosómico (ARNr) en todos los organismos vivos. El 16 ARNr de las bacterias tiene una longitud de 1.650 nucleótidos y sus secuencias se han dividido en distintas áreas de acuerdo con su grado de variabilidad entre diferentes grupos taxonómicos. Las regiones altamente conservadas agrupan a las bacterias en altos órdenes taxonómicos, mientras que las regiones variables (mayor frecuencia de mutación) permiten una clasificación a niveles inferiores como género o especie. De esta manera pueden construirse los árboles filogenéticos que relacionan las bacterias.
La nomenclatura de las bacterias está regida por reglas internacionales desarrolladas y aplicadas por científicos dedicados a la taxonomía, de modo que una bacteria con un nombre dado es reconocida internacionalmente de modo inequívoco como el mismo. Estas reglas son publicadas en el Código Internacional de Nomenclatura de las Bacterias (Internacional Code of Nomenclature of Bacteria), con algunos principios: a) existe un solo nombre correcto para una bacteria; b) los nombres que causan error o confusión deben ser rechazados, y c) todos los nombres están en latín o latinizados, de palabras de este idioma o derivadas del griego1-7.
Lógicamente, la taxonomía es una ciencia dinámica, sujeta a cambios basados en los datos conocidos, la nueva información requiere con frecuencia cambios en la taxonomía; a su vez, estos cambios determinan otros cambios en la clasificación existente, en la nomenclatura, en los criterios de identificación y en el reconocimiento de nuevas especies.
Los microbiólogos clínicos deben mantenerse actualizados con respecto a los cambios taxonómicos, a fin de saber qué especies recientemente descritas presentan problemas clínicos potenciales, cuáles son patógenos humanos, y la manera de identificarlos.
Diagnóstico microbiológico de las enfermedades infecciosas
El diagnóstico microbiológico de las enfermedades infecciosas y su control epidemiológico tiene actualmente unas exigencias derivadas de mucho volumen de trabajo, necesidad de diagnósticos objetivos, rápidos, fiables, seguros y reproducibles, y todo ello ajustando el binomio coste-eficiencia. Las pruebas que se demandan cada vez son mayores, en cantidad, calidad y diferenciación, lo que hace que los laboratorios se encarezcan en el montante económico total.
La economía y la práctica dictan el uso de un número mínimo de pruebas diagnósticas, así, por necesidad, la identificación en el laboratorio clínico representará siempre un compromiso entre la exactitud y precisión por una parte, y la velocidad y economía por otra. Cómo se logra este compromiso es lo que determina la calidad de un laboratorio. La limitación práctica del número de propiedades que pueden usarse para la identificación de los microorganismos hace que la selección de las pruebas diagnósticas sea muy importante. Las mismas propiedades que se usan en la clasificación taxonómica se emplean en la identificación de los aislamientos clínicos.
La identificación de las especies bacterianas presentes en las muestras clínicas es una disciplina aplicada de la microbiología de gran significación práctica. La identificación de una bacteria sospechosa de ser la causa de una infección es uno de los instrumentos principales del diagnóstico de enfermedades infecciosas, ya que el conocimiento de éste nos lleva a saber con lo que se enfrenta el paciente, y saber de qué armas se dispone para ayudar a combatir la enfermedad. Diferenciar si estamos ante un microorganismo patógeno, uno potencialmente patógeno o un contaminante y deducir los posibles mecanismos de resistencia del microorganismo, conduce a tratar la enfermedad infecciosa con la mayor eficiencia posible, con antimicrobianos u otras terapias antiinfecciosas, y todo procurando el menor coste posible. La identificación correcta también permite detectar y seguir las infecciones hospitalarias y brotes infecciosos por microorganismos nosocomiales o de la comunidad social.
Proceso del diagnóstico microbiológico
El proceso del diagnóstico microbiológico en general y el bacteriológico en particular es secuencial, comienza por un problema clínico o de control epidemiológico, que hace necesaria la demanda de una o varias pruebas, toma y transporte adecuados de las muestras que se van a investigar, con los datos demográficos y clínicos pertinentes inscritos en un volante de petición, recepción en el laboratorio, procesamiento, análisis e interpretación de los resultados, y un informe final que una vez recibido en el punto de origen ayuda a tomar decisiones clinicoepidemiológicas y adecuación de tratamientos antimicrobianos8-10.
El informe del laboratorio, se espera que sea rápido, claro, objetivo, fiable, que ayude y no confunda, con pensamiento clínico y eficaz. Cuanto más rápido es el informe, aunque sea provisional, mayor es el interés del médico por el mismo; este es máximo en las primeras horas, descendiendo a mínimos a partir de 24-48 h, para volver a aumentar a partir de las 72 h, cuando los tratamientos iniciales empíricos han fallado o hay necesidad de completar, por varias causas, datos objetivos en el historial de enfermo. En la medicina de cada día, un diagnóstico microbiológico muy perfeccionista, a veces necesario, no va necesariamente acompañado de rentabilidad clínica, ya que puede confundir al médico, demorar tratamientos o provocar otros innecesarios.
La identificación de bacterias tiene sus etapas, procedimientos y grado. Una vez llegada la muestra al laboratorio se examina directamente, en fresco o por medio de tinciones generales o específicas, luego, las bacterias aisladas en los distintos cultivos se identifican por diversos métodos para conocer género, especie y biotipos, y luego, si procede, se hacen con ellas otras pruebas, entre ellas la de sensibilidad antibiótica11,12.
Examen en fresco
La observación directa al microscopio de muestras no teñidas, puede realizarse directamente, o con montaje en solución salina. Permite, entre otras cosas, evaluar la calidad de la muestra y es útil en la observación de la presencia de bacterias, disposición y su movilidad, así como la existencia de estructuras no bacterianas como levaduras, micelios de hongos filamentosos, otros parásitos y células. La incorporación de anticuerpos específicos permite la identificación de la cápsula en el caso de Streptococcus pneumoniae y Haemophilus influenzae. El examen en un microscopio con contraste de fases, o campo oscuro, es útil para la observación de espiroquetas.
Tinciones
Las tinciones son el primer paso, y a veces el único, para la identificación bacteriana. La mayoría de los colorantes utilizados en bacteriología se derivan de la anilina, que tienen como estructura básica anillos de benceno unidos que se comportan como cromóforos, y fuerte afinidad por el hidrógeno, unos son ácidos y otros básicos13-16. Las coloraciones más usadas e imprescindibles son la de azul de metileno y la de Gram; esta última, especialmente utilizada para ver las propiedades de tinción de todo tipo de bacterias, diferenciándolas en dos grandes grupos, grampositivas (color violeta) y gramnegativas (color rojo) a la vez que permite distinguir su morfología y forma de agruparse. El Gram es de utilidad para orientarnos con la mayoría de las bacterias, pero permite hacer una presunción, apoyados en el tipo de muestra y diagnóstico presuntivo, de algunas bacterias como S. pneumoniae, Neisseria, Bacillus, Clostridium, Corynebacterium, Listeria, Vibrio, Campylobacter, Enterobacteriaceae, Haemophilus y otras.
La coloración de Ziehl-Neelsen, basada en la capacidad de resistir a la decoloración de algunas especies, por su pared rica en ácidos micólicos, a una mezcla de alcohol y ácido, se mantiene en la actualidad como imprescindible para detectar la presencia de Mycobacterium en las muestras patológicas y en la identificación de sus colonias. También es útil, con decoloración más suave, "Ziehl dulce", para la sospecha de Nocardia y Rodococcus.
Las coloraciones por medio de colorantes panóptico como Giemsa y Wright, y con fluorocromos como naranja de acridina y auramina, dan un buen servicio para la observación de bacterias que tiene dificultad en teñirse con los colorantes de anilina, como los utilizados en la técnica de Gram o azul de metileno, o que tienen poco contraste con el material de fondo, entre las cuales tenemos los bacilos ácido-alcohol resistentes y algunos patógenos intracelulares como Legionella pneumophila y Chlamydia pneumoniae. Las tinciones fluorescentes, como el isocianato de fluoresceína, a pesar de lo complejo de la técnica, son útiles y mejoran de manera considerable si se asocian a anticuerpos monoclonales.
Otro tipo de tinciones habituales de gran utilidad para bacterias muy finas como Treponema pallidum y Borrelia burgdorferi son las de plata, como la de Fontana-Tribondeau, que permite recubrir a estos microorganismos con su precipitado y aumentar el tamaño, facilitando su visión microscópica.
Tanto la información derivada de los exámenes en fresco como el de las bacterias teñidas puede comunicarse inmediatamente a los médicos, dependiendo de su relevancia y utilidad.
En muchas ocasiones, los resultados de estos exámenes son suficientes para establecer el diagnóstico, al menos presuntivo, con un excelente rendimiento en rapidez, seguridad y coste-eficiencia. Las mayores aplicaciones, además de las generales de presencia o no de bacterias, su coloración, morfología y forma de agruparse, son en el diagnóstico de meningitis examinando el líquido cefalorraquídeo (LCR) (N. meningitidis, S. pneumoniae, H. influenzae, L. monocytogenes, bacilos gramnegativos); infecciones urinarias por la presencia de bacterias habituales y Leptospira; enfermedades de transmisión sexual por T. pallidum, C. trachomatis, N. gonorrhoeae y Gardnerella vaginalis; fiebre recurrente por Borrelia; conjuntivitis purulenta bacteriana y especialmente por C. trachomatis; infecciones intestinales por Vibrio y C. difficile; faringoamigdalitis por Corynebacterium difteriae y úlcera de Vincent; infecciones pulmonares u otra localización por Nocardia, M. tuberculosis, M. leprae y otras micobacterias, etc.
Identificación tras aislamiento por cultivo
La clave para identificar las bacterias como agentes patógenos depende de su aislamiento en cultivo puro; en 1840, Henle ya señaló la necesidad de dar este paso previo a la identificación final. Una colonia de cultivo puro está compuesta por un solo tipo de microorganismos y procede de una única célula. Las pruebas de identificación se deben hacer siempre en colonias únicas o cultivos puros. Es posible llevar a cabo una identificación preliminar de muchas bacterias con importancia médica, sobre la base de unas pocas características simples de las células.
En general, una vez que se han aislado las bacterias por medio de su cultivo correspondiente, común o selectivo, el paso siguiente para su identificación pasa por el conocimiento de las características ambientales de su crecimiento óptimo (aerobias, anaerobias, microaerófilas, tiempo de cultivo, grado de humedad, temperatura de crecimiento), estudio visual del aspecto de las colonias, hecho fundamental en el diagnóstico bacteriano, como morfología, elevación, bordes, tamaño, color, olor, tipo de superficie, densidad, consistencia, incrustación el medio de cultivo, crecimiento en sábana y otras características; su comportamiento frente a los distintos sustratos incorporados al medio de cultivo (hemólisis, fermentación de azúcares, utilización de sales; el comportamiento frente a los colorantes de los individuos bacterianos que forman las colonias, junto con su morfología, manera de agruparse y presencia de esporas o no; posteriormente, el siguiente paso es el estudio del comportamiento metabólico mediante pruebas bioquímicas, como la utilización de diversas fuentes orgánicas e inorgánicas con la producción de sustancias detectables por reacciones directas e indirectas; y también la detección de sus diversos antígenos con anticuerpos específicos17-22.
Las pruebas bioquímicas pueden ser rápidas y sencillas o más lentas y complejas; un ejemplo de las primeras son la detección de producción de catalasa por medio de H2O2, solubilidad en la bilis (desoxicolato de sodio), plasmocoagulasa en "porta", actividad de citocromo oxidasa, presencia de indol con el reactivo de Kovacs, etc.; el caso de las segundas suelen utilizarse más número de pruebas y requieren un período más largo de tiempo, pueden llevarse a cabo con técnicas manuales o con automatización del proceso. En el caso de las bacterias gramnegativas es común buscar la actividad de la citocromooxidasa, utilización de hidratos de carbono como fermentación de la glucosa, lactosa y otros azúcares, producción de betagalactosidasa, reducción de nitratos, actividad descarboxilasa o deshidrolasa sobre la ornitina, lisina y arginina, producción de citrato, de indol, de acetil-metil-carbinol, ureasa, etc., batería de pruebas que pueden hacerse con máquinas automáticas23,24.
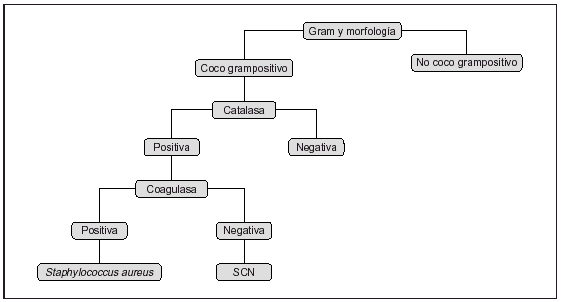
Una vez obtenidos los resultados de las pruebas bioquímicas y otras, la identificación final puede efectuarse por medio de los sistemas de esquemas clásicos como el de matriz de cuadrículas y los diagramas de flujo ramificados. En la matriz de cuadrículas se expresan las pruebas bioquímicas positivas frente a diferentes sustratos, y el porcentaje de positividad para distintas especies bacterianas. La precisión es bastante alta, pero la interpretación es tediosa hasta llegar a un patrón determinado para identificar la especie y el biotipo. Los diagramas de flujo reducen la complejidad de la lectura de toda la matriz, utilizando algoritmos, pero un fallo en uno de los puntos puede originar interpretaciones erróneas finales. Un esquema de algoritmo simple puede verse en la figura 1. En la actualidad, esto puede simplificarse con la ayuda de programas informáticos controlados por computadoras. Otro paso para facilitar la identificación bacteriana es la utilización de códigos numéricos aplicados a los resultados de las pruebas, tanto por medios manuales como asistidos por ordenadores, que trasladan las características de identificación a una secuencia de números binarios correspondientes a los resultados de pruebas positivas o negativas de los diferentes géneros y especies, transformados posteriormente en sus equivalentes octales hasta llegar a sistemas matemáticos más simples y comprensibles de códigos numéricos. Posteriormente, para la identificación definitiva habrá que apoyarse en el cálculo de probabilidades, previa comprobación y resolución de las posibles discrepancias. Por ejemplo, sistemas manuales basados en este sistema son el API (bioMérieux), tubo Oxi/Ferm (Becton-Dickinson), el BD-Crystal (Becton-Dickinson), Micro-ID (Remel), RapID (Innovative Diagnostics), Enterotube (Becton-Dickinson), Minitek (Becton-Dickinson), Biolog (Biolog Inc.), Sensititre (AccuMed Internacional), Sceptor (Becton-Dickinson), entre otros, alguno de ellos en desuso ante la irrupción de los sistemas automáticos o semiautomáticos.
Figura 1. Ejemplo simple de identificación por algoritmo en cascada. SCN: Staphylococcus coagulasa negativo.
El sistema de galería API es el más utilizado en la actualidad. En él se va obteniendo una puntuación según que cada prueba sea positiva o negativa, la identificación numérica de un perfil observado se apoya en el cálculo de su proximidad relativa a los distintos taxones de la base de datos y su proximidad al perfil más típico en cada taxón; a continuación, la clasificación de los taxones permite proponer un resultado de identificación. Básicamente, la base de datos establecida está constituida por porcentajes de reacción positiva para cada taxón/prueba, las frecuencias de aparición de las reacciones observadas derivan de los porcentajes de reacciones positivas y negativas y contemplan los posibles errores de lectura de cada prueba, al mismo tiempo se calculan las frecuencias de aparición de las reacciones del perfil más típico con probabilidad positiva cuando la probabilidad es igual o mayor a 50 y con probabilidad negativa cuando es menor a 50. La frecuencia de aparición del perfil observado en cada taxón es igual al producto del conjunto de frecuencias de aparición en el taxón de las reacciones observadas. Al mismo tiempo, se calcula para cada taxón la frecuencia de aparición del perfil más típico, igual al producto de las frecuencias de aparición de las reacciones del perfil más típico. A continuación, estas frecuencias se normalizan dividiendo cada una por su suma total; multiplicando el resultado por 100 se obtiene el porcentaje de identificación; al mismo tiempo se calcula para cada taxón una frecuencia modal a partir de las reacciones más típicas; luego se clasifican los taxones en orden decreciente de sus porcentajes de identificación y se calcula un índice T (tipificación) a partir de cada frecuencia modal. Si se obtiene un único taxón y su porcentaje de identificación es mayor o igual a 80, se propone para la identificación con un comentario según los valores de su porcentaje de identificación y de su índice T. Si se obtienen varios taxones, cuya suma es mayor o igual a 80, se proponen para la identificación con un comentario seleccionado según el valor de la suma de los porcentajes de identificación según el promedio de los índices T. Durante mucho tiempo, el API ha representado la identificación de referencia en el laboratorio, pero debido a que su realización es laboriosa, al igual que su interpretación, prefieren utilizarse otros métodos, como los automáticos.
Pruebas para identificar algunas bacterias
Para los bacilos gramnegativos aerobios del tipo enterobacterias se realiza una batería de pruebas que lleve a identificar lo mejor posible las más frecuentemente aisladas en nuestro medio. Una batería ejemplo como la expuesta en la tabla 1. Esta batería puede ayudar a su identificación final, a conocer el fenotipo de sensibilidad de dicha bacteria. Este tipo de batería de pruebas puede variar de un laboratorio a otro, ya que cada microbiólogo conoce hasta dónde quiere llegar en la identificación, y qué pruebas le resultan más fáciles y familiares para el reconocimiento de las bacterias.
Para cocos grampositivos, si son catalasa-positivos puede realizarse una prueba de manitol con 6,5% de cloruro sódico y una ADNasa, con lo que se diferenciará S. aureus del resto de estafilococos, que llamamos genéricamente estafilococos coagulasa-negativos. Si queremos llegar mas lejos en la identificación deberán realizarse más pruebas. En cocos grampositivos catalasa-negativos tenemos un gran número de géneros, por lo que la identificación preliminar va a desempeñar un papel esencial. Se puede realizar un esquema en cascada con unas pocas pruebas convencionales que permitan identificar el género y, en algún caso, llegar hasta especie: patrón hemolítico (si hay betahemólisis puede realizarse una prueba con antisueros que identifica los grupos antigénicos de Lancefield), crecimiento en satélite con una cepa de S. aureus hemolítica, piracinamidasa, hidrólisis del hipurato, prueba de CAMP, solubilidad en bilis y optoquina. Si interesa llegar a una identificación de especie deberán realizarse una gran cantidad de pruebas convencionales o ir a un sistema de identificación con más pruebas como el API rapid ID 32 STREP, o un sistema automático como el Vitek2 (bioMérieux Vitek), MicroScan WalkAway (Dade), BD Phoenix (Becton-Dickinson) o Wider (Soria Melguizo).
Para bacilos grampositivos, la diferenciación preliminar se realizará por la morfología de la colonia, color, hemólisis, características al microscopio y presencia de esporas. Pueden diferenciarse los tipos corineformes del género Bacillus, y con unas pocas pruebas convencionales (catalasa, motilidad, reducción de nitratos, hidrólisis de esculina y gelatina, fermentación de azúcares, urea y piracinamidasa) y conociendo la sensibilidad a los antimicrobianos, se consigue identificar la especie en un porcentaje importante de los bacilos grampositivos de mayor relevancia clínica. Para una mayor seguridad en la identificación se utilizará un sistema de galería para bacilos corineformes (API Coryne), o el API 50 CHB para el género Bacillus. También hay métodos automáticos como el BD Phoenix que identifica bien bacilos grampositivos.
Para la identificación de bacterias anaerobias, es importante la identificación preliminar que llevará en muchos casos hasta el género, basándose en la morfología, Gram, disposición bacteriana y hemólisis. Para una identificación definitiva puede realizarse una galería de pruebas como el API rapad ID 32 A, que incluye pruebas enzimáticos estandarizadas y miniaturizadas y una base específica, la incubación se realiza en aerobiosis y la lectura en solo 4 h.
Sistemas automáticos
Hemos comentado anteriormente que hay sistemas semiautomáticos y automáticos para la identificación de bacterias. Existen en el mercado varios sistemas de identificación, fáciles de aplicar, y rápidos en dar una identificación definitiva en el ámbito de especie, además de poder realizar las pruebas de sensibilidad al mismo tiempo; su principal aplicación son las identificaciones y pruebas de sensibilidad, en el quehacer diario del laboratorio, en asociación con los métodos convencionales. Los más usados en la actualidad son el Vitek (bioMérieux), Microscan Walkaway (Dade), BD Phoenix (Becton-Dickinson), Wilder (Soria Melguizo) y Sensititre (Accu Med). Básicamente, los sistemas consisten en unos paneles con una serie de pocillos con diversos sustratos. En cada uno de ellos se determina una prueba y dan el resultado en porcentaje de identificación o en porcentaje de tipificación. En algunos casos será necesario alguna prueba externa para diferenciar entre dos o más especies, como en los casos en que hay hemólisis, pigmentación, movilidad, reacción citocromo-oxidasa, etc. Estos sistemas incorporan un software denominado "experto" que aplica al resultado final una serie de las reglas programadas, normalmente siguiendo las recomendaciones del National Committee of Clinical Laboratory Standards (NCCLS) u otros organismos, con el fin de facilitar el estudio de las cepas identificadas, dando consejos taxonómicos y advirtiendo de resultados poco probables o imposibles, lo cual indica además otros caminos a seguir.
Todos los sistemas de automáticos actuales son parecidos en cuanto a identificación, con diferentes sus matices en la forma de preparar los paneles, unos son más laboriosos que otros. La diferencia fundamental la encontramos en las pruebas de sensibilidad y el sistema "experto" que utilizan, unos con reglas que se aplican a los resultados, y otros, como el Vitek2, utilizan el Sistema Experto Avanzado (AES) que está basado en el reconocimiento fenotípico a partir de la concentración inhibitoria mínima (CIM), aplicando una base de datos que incorpora los resultados de CIM publicadas en la bibliografía mundial, y que se actualiza periódicamente.
Como ejemplo de estos sistemas tenemos el Vitek2, que es un sistema totalmente automático de identificación basado en tecnología de fluorescencia para la detección de metabolismo bacteriano y prueba de susceptibilidad de microorganismos basado en el principio de curvas de crecimiento cinético según el patrón fenotípico de resistencia, además de la detección rápida de resistencia de alto nivel y emergentes. Está diseñado para identificar hasta el 90% de los aislados de rutina clínica. Estos resultados pueden obtenerse dentro de las 2 h para los cocos grampositivos y en 3 h para los bacilos gramnegativos. Esto identifica hasta 51 especies de levaduras y organismos afines en 15 h. El soporte físico para la realización de la prueba de identificación son unas tarjetas con 64 pocillos, en cada una de ellas se realiza una prueba, no todos los pocillos están en uso, el llenado de las tarjetas que usa es un proceso automático que se realiza por vacío dentro del propio módulo del sistema. Está programado para identificar 101 especies de enterobacterias y bacilos no fermentadores de relevancia clínica, y 53 especies para estafilococos y estreptococos.
El BD Phoenix identifica también bacterias de significado clínico y realiza pruebas de sensibilidad antimicrobiana. Tanto las pruebas de identificación como el antibiograma se procesan de manera simultánea en el mismo panel. Usa bandejas de poliestireno con 136 pocillos, 51 reservados para las pruebas bioquímicas y enzimáticas de identificación, y una segunda zona con 85 pocillos en la que se realizan las pruebas de sensibilidad a los antimicrobianos por la técnica de diluciones dobles seriadas de cada antibiótico, al menos con tres concentraciones para cada uno de ellos. La lectura se hace por medio de colorimetría, colorantes-redox y flurimetría. Los resultados, simultáneos, se obtienen a las 5-7 h para las bacterias grampositivas y a las 6-12 h para las gramnegativas.
El Microscan WalkAway, de diseño más antiguo, utiliza microplacas de 96 pocillos, independientes para la identificación y las pruebas de sensibilidad, haciendo la lectura tanto por turbidimetría, colorimetría y flurimetría. Emplea 32 sustratos reactivos y varias diluciones de antibióticos. Requiere más reactivos adicionales que los dos anteriores, aunque pueden incorporarse automáticamente, y la lectura se hace a las 2-18 h, dependiendo de las bacterias y el sistema de paneles.
El Wider es un sistema semiautomático, que transmite en 5 s la lectura de los pocillos donde se realizan las distintas pruebas por medio de una telecámara de alta resolución a un sistema computarizado donde se procesan los resultados. La visón de las imágenes de las distintas pruebas por el observador hace que sea un sistema que gusta a muchos microbiólogos.
El sistema de identificación Biolog, que utiliza también microplacas de 96 pocillos, tiene una base de datos mucho más amplia que los anteriores. Esta basado en la utilización, oxidación y producción de nicotinamida adenindinucleótido reducido (NADH), de una gran variedad sustratos de fuente carbono, 95 en concreto, cuya utilización se mide por medio de la reducción del colorante redox violeta de tetrazolio, de amplio uso en biología. Está diseñado para la identificación de bacterias ambientales, alimentos y otras procedencias menos relacionadas con los problemas médicos, pero de gran utilidad también en microbiología clínica. Esta misma firma dispone un sistema para la identificación por métodos génicos.
Otro sistema que se ha automatizado es el Sensititre, pero sólo para bacterias gramnegativas.
Lo principal de todos estos métodos automáticos es su fácil realización, capacidad al menos de identificar hasta grado de especie, y tener en su base de datos la gran mayoría de las bacterias de importancia en la práctica clínica. En general se identifican bien bacilos gramnegativos del tipo de enterobacterias y bacilos gramnegativos no fermentadores, estreptococos betahemolíticos, enterococos, algunos estreptococos alfahemolíticos y estafilococos. Algunos sistemas tienen paneles especiales para S. pneumoniae, levaduras y bacilos grampositivos del tipo de Listeria, Corynebacterium y Bacillus. De cualquier manera, la automatización considerada desde un punto de vista general, muy útil y extendida en los laboratorios generales de análisis clínicos y bioquímica, es más difícil en microbiología, porque las muestras son variables e inestables, las peticiones son heterogéneas y con frecuencia inconcretas, hay mucho apoyo, como se ha comentado, en los exámenes directos, es diversa y selectiva la elección de los medios de cultivo (dependiendo de la cantidad y calidad de la muestra y de los resultados de los exámenes directos) y la interpretación de muchos de los resultados hay que hacerla en el contexto de los datos del enfermo y su proceso nosológico. Las áreas donde es posible la automatización son, además de la identificación y antibiograma ya expuestas, los cultivos de muestreo de orinas (sólo el 60% de los casos), técnicas de enzimoinmunoanálisis (65% de la serología), crecimiento de micobacterias, extracción y amplificación de ácidos nucleicos, y parte de la secuenciación de genes, siempre valorando su ventaja sobre las técnicas convencionales. En la decisión de automatizar pruebas, y concretamente en la identificación de bacterias, hay que valorar si representa un ahorro en costes y en personal, lo que no siempre sucede. Por ejemplo, la identificación automatizada de bacterias, sólo en gasto de material fungible, es de »10-12 €, mientras que la manual no pasa de »0,6-2,5 €. Una ventaja de la automatización es la mayor objetividad, la mejor homologación de lo resultados interlaboratorio y la realización de mayor número de pruebas con menos personas.
Identificación génica de bacterias
Como se ha comentado al principio de este escrito, la tendencia para el futuro, y en algunos casos realidad en la actualidad, es utilizar la identificación bacteriana por medio de la taxonomía molecular. Las herramientas más utilizadas son la reacción en cadena de la polimerasa (PCR), usando primers para la diana 16S ARNr de la bacteria, o la ARNtm para las que tengan el ARNr muy conservado, que permite la amplificación génica y su posterior detección por hibridación u otros métodos, la identificación de genes codificadores de factores de virulencia, la detección microorganismos, únicos o múltiples, de difícil cultivo o no cultivables y la aplicación a situaciones nuevas emergentes desconocidas. Además, la disponibilidad de PCR en "tiempo real" acorta de manera considerable los tiempos de diagnóstico. Otros métodos, basados en la misma tecnología, son la aplicación de las micromatrices, la identificación multiespectral por emisión de fluorescencia, la secuenciación y posterior comparación con bases de datos de genes bacterianos y, lógicamente, la aplicación de algoritmos a la misma tecnología25-31. Es evidente que esto supondrá cambios en los conocimientos y habilidades del personal del laboratorio, sin abandonar los clásicos, aumento de la tecnología, adaptación a la misma e incremento de los costes, pero, probablemente, con un binomio coste-eficiencia favorable. De todo esto, de los métodos moleculares, se tratará en otro capítulo de esta monografía.