Muchos de los hongos degradadores de madera están implicados en la síntesis de metabolitos bioactivos de naturaleza antimicrobiana y terapéutica, así como de compuestos de importancia biotecnológica, incluyendo derivados indólicos, entre otros. Estos hongos brindan ciertos beneficios ecológicos a las plantas, entre los que se destacan la protección contra fitopatógenos y la promoción del crecimiento radicular. Xylaria sp. es un hongo degradador de celulosa (lignocelulolítico) con potencial biotecnológico. El ácido indol-3-acético (AIA) desempeña un papel sumamente importante en las interacciones planta-microorganismo, ya que es esencial para la fisiología y el correcto desarrollo morfológico vegetal. Se sabe que las enzimas nitrilo-hidrolíticas (nitrilasas) están involucradas en la síntesis de compuestos indólicos en las plantas, no obstante, se dispone de poca información acerca de la naturaleza de estas enzimas en el reino de los hongos. A través de una aproximación bioquímica y de genética molecular, se demuestra por primera vez que Xylaria sp. posee actividad enzimática nitrilasa utilizando compuestos ricos en nitrógeno y carbono como sustrato. La cepa estudiada aumentó sus niveles de expresión génica relativa y mostró crecimiento micelial, ambos en presencia de compuestos químicos como cianobenceno y KCN. Los resultados de este trabajo sugieren que el microorganismo es capaz de degradar moléculas nitrogenadas complejas. Por otra parte, mediante biofertilización con extractos fúngicos, se observó que Xylaria sp. promueve el desarrollo del sistema radicular de plántulas de Arabidopsis thaliana, además de sintetizar AIA.
Endophytic fungi inhabit plant tissues internally and asymptomatically, and many of them are involved in the synthesis of bioactive metabolites of antifungal and therapeutic nature, as well as other compounds of biotechnological importance including indole derivatives, among many others. Ecologically, they provide some benefits to plants including protection against phytopathogens and promotion of root growth. In this sense, Xylaria sp. is a cellulose-decomposing fungus with biotechnological potential. It is worth mentioning that indole-3-acetic acid (IAA) also plays an extremely important role in plant–micro-organism interactions, as it is essential for physiology and proper plant morphological development. It is known that nitrile-hydrolytic enzymes (nitrilases) are involved in the synthesis of plant indole compounds; however, relatively little information is available concerning the nature of these enzymes in the fungal kingdom. In view of the above, through a biochemical and molecular-genetic approach, it has been demonstrated for the first time that Xylaria sp. carries out nitrile-hydrolytic enzyme activity using nitrogen and carbon-rich compounds as substrate. The studied strain increased its relative gene expression levels and showed mycelial growth, both in the presence of chemical compounds such as cyanobenzene and KCN. Thus, the results of this work suggest that the micro-organism is capable of degrading complex nitrogenous molecules. On the other hand, through fungal biofertilization, it was observed that Xylaria sp. promotes the development of the root system of Arabidopsis thaliana seedlings, in addition to synthesizing IAA.
En los últimos años, se ha demostrado que los hongos producen una gran diversidad de metabolitos secundarios bioactivos, incluyendo biomoléculas con aplicaciones medicinales y farmacológicas. Lo anterior se extiende al grupo de los microorganismos que interactúan con las plantas sin inducir afección alguna. Esta coevolución significa que dichos individuos producen los mismos compuestos, o, al menos, biomoléculas similares a las que se originan en los vegetales38.
En este sentido, la mayoría de las especies fúngicas del orden Xylariales (clase: Sordariomycetes) se han relacionado con aplicaciones biotecnológicas y de control biológico28. Por otra parte, las propiedades antagónicas-microbiológicas del género Xylaria también se vinculan con la producción de ciertos compuestos con actividad antibiótica y de competencia por nutrientes37. Igualmente, se sabe que la síntesis de enzimas hidrolíticas (por ejemplo, nitrilasas) es un fenómeno relacionado con la promoción del crecimiento vegetal a través del metabolismo de compuestos indólicos42.
Las enzimas de la superfamilia de las nitrilasas, también denominadas CN-hidrolasas22, hidrolizan y condensan enlaces carbono-nitrógeno no peptídicos al correspondiente ácido carboxílico (R-COOH) y amoníaco, utilizando una tríada catalítica Glu-Lys-Cys que se repite en las regiones conservadas de los genes que transcriben estas enzimas5.
Los nitrilos son abundantes en los entornos vegetales, y se ha sugerido que las nitrilasas forman parte de un mecanismo que facilita la colonización microbiana de las plantas, con posibles funciones en la síntesis de hormonas vegetales, así como en la utilización de nitrógeno o la detoxificación ambiental de nitrilos y cianuro15,20,24.
El indol-3-acetonitrilo (IAN) se hidroliza por la actividad de las nitrilasas, lo cual lleva a la síntesis de ácido indol-3-acético (AIA) y ácido fenilacético. La vía del IAN es al menos una de varias que se han identificado para la biosíntesis de AIA en plantas14,15. Se sabe que algunas especies de Xylaria colonizan parte de la raíz de las plantas y sus efectos son evidentes en la promoción de su desarrollo7, probablemente debido a la intervención de este tipo de enzimas.
Si bien es conocido el papel biológico de las nitrilasas vegetales, la importancia de estas enzimas en los hongos ha sido poco estudiada. Existen reportes que sugieren que el L-triptófano (Trp) es un precursor del AIA en cultivos de Trichoderma, lo cual se encuentra vinculado a la promoción del desarrollo de raíces laterales en Arabidopsis thaliana12. Las nitrilasas también tienen una amplia gama de aplicaciones industriales y biotecnológicas, incluida la síntesis de ácidos carboxílicos de importancia industrial y la biorremediación de cianuro y compuestos nitrogenados heterocíclicos15,35.
En el presente estudio se proporciona la primera evidencia de actividad enzimática nitrilasa por parte de un hongo del género Xylaria y, de manera complementaria, se dilucida su relación con la síntesis de AIA. Además, se propone una reconstrucción filogenética de una secuencia homóloga putativa de nucleótidos que codifica este grupo de enzimas y se evalúan sus niveles de expresión relativa. Por otra parte, se demuestra que el hongo produce cantidades significativas de AIA al utilizar Trp como precursor, lo que se relacionaría con la promoción del crecimiento de raíces de A. thaliana. Asimismo, se determinó la capacidad de crecimiento micelial en presencia de cianobenceno.
Materiales y métodosAislamiento y caracterización de la cepa fúngica nativaSe logró aislar una cepa de Xylaria que fructificaba sobre un fruto de caoba (Swietenia macrophylla) en proceso de descomposición, en el suelo. Este material se recolectó en una región geográfica con características de bosque tropical, a 495 m.s.n.m., situada en el noroeste del estado de Veracruz, México (18°25′00″N 95°07′00″O), en la vertiente del golfo. La muestra biológica fue transportada al laboratorio, en donde se realizó una caracterización morfológica preliminar empleando claves taxonómicas25. De manera complementaria, se caracterizó molecularmente a la cepa, para lo cual se hicieron cortes de fragmentos del estroma previamente desinfectados en su superficie con NaClO. Los trozos se colocaron en el centro de placas de Petri con agar suplementado con harina de maíz (Corn Meal Agar, CMA, Becton Dickinson) para inducir la proliferación micelial.
A partir de cultivos de una semana se realizaron extracciones de ADN genómico utilizando metanol-cloroformo como disolvente, siguiendo el protocolo descrito por Lin et al.26. Así, se secuenciaron (método Illumina) transcritos previamente generados por PCR de regiones genómicas conservadas (ITS1-ITS2 y 5.8S rRNA) y factores de elongación alfa (EF-1a). De cada amplicón se calculó el promedio y la posición de las bases nitrogenadas empleando el modelo de probabilidad máxima compuesta40.
Se comparó la homología con la colección de nucleótidos (nr/nt) del Centro Nacional para la Información Biotecnológica (NCBI) empleando la herramienta de búsqueda de alineación básica local (BLAST), y se seleccionaron las accesiones que mostraron el mayor porcentaje de identidad. El conjunto de datos se analizó mediante comparación de secuencias múltiples por expectativa logarítmica (MUSCLE)29 empleando como elemento de referencia una única secuencia concatenada cuyas regiones consenso (≈ 90%) estaban conformadas por fragmentos de genes ITS como unidad flanqueada y, complementando el resto, con una secuencia corta de factor de elongación.
Determinación de la expresión génica relativa de Xylaria sp.La expresión génica relativa de una secuencia homóloga de Xylaria sp. que codifica enzimas nitrilasas putativas se analizó mediante qRT-PCR empleando ADNc independiente como plantilla. Este se obtuvo a partir del ARN total, que se extrajo de manera directa del micelio con TRIZOL® (Invitrogen Life Technologies, Carlsbad, CA), siguiendo las instrucciones del fabricante. Previamente se diseñaron los siguientes cebadores específicos y se comprobó que no se formaban dímeros: xyln-D (5́-CGGAAAGCCTGAAAACCGTG-3́) y xyln-R (5́-TGCAAATCGCGGTAAAGCAC-3́). Se empleó el gen de la β-tubulina como control endógeno constitutivo: Tub-D (5́-GATCGTCTAAGTCGTAATCTGAGA-3́) y Tub-R (5́- AGCTATTGCCTTAGCTGCATCAAG-3́), el cual sirvió como base para calcular los ciclos de umbral (CT) utilizando el software 7500 Fast DX PCR-System (Thermofisher). Los datos se expresaron como log10 de 2-ΔΔCT27. Las condiciones de la qRT-PCR fueron un ciclo de 10min a 95°C y 40 ciclos de dos pasos de 15 s a 95°C y 1min a 60°C.
Estudio de la evolución molecular taxonómica y caracterización de secuencias nucleotídicas homólogasLa reconstrucción filogenética de secuencias nucleotídicas homólogas que codifican enzimas nitrilasas (incluyendo la secuencia de la cepa fúngica nativa amplificada mediante qRT-PCR) se realizó empleando el método estadístico de unión de secuencias vecinas (NJ: Neighbor-Joining)36, computando 500 repeticiones17, y calculando las distancias evolutivas con el método de probabilidad máxima compuesta41. Cada análisis evolutivo se realizó con el software MEGA7 versión 7.023 y se generaron reconstrucciones óptimas seguidas de análisis BLASTn contra las secuencias nucleotídicas de referencia. Se complementó con un conjunto de datos que contenía más de 70 accesiones de genes putativos y caracterizados, que transcriben enzimas nitrilasas de especies de distintos reinos.
De manera adicional, se llevó a cabo la traducción bioinformática a aminoácidos del transcrito amplificado, se buscaron los marcos de lectura abierta (ORF) mediante el software EXPASy Translate Tool (https://web.expasy.org/translate/) y se identificaron las orientaciones 5́-3́ y 3́-5́ así como los sitios activos y de enlace, al igual que tríadas y residuos catalíticos. Los algoritmos MAKER-GMOD (http://www.yandell-lab.org/software/maker.html) y BUSCO v3 (https://busco-archive.ezlab.org/) permitieron analizar la secuencia respecto de su predicción funcional y evolución molecular a partir de copias únicas de genes ortólogos.
Elaboración de cultivos líquidos para la inducción de expresión génicaLa obtención de micelio de Xylaria para la extracción del ARN total y el posterior estudio de la expresión génica relativa se llevó a cabo siguiendo una estrategia de cultivos líquidos en dos etapas. Primero, el hongo se cultivó en matraces de 200ml con medio mínimo (MM)34 que contenía glucosa al 2% a 28°C durante 48h. Posteriormente, se recogieron los micelios, se filtraron y transfirieron a 200ml de MM con glucosa (2%) o sin dicha fuente de carbono, suplementando con distintas fuentes de nitrógeno a diferentes concentraciones, de modo que se obtuvieron los siguientes tratamientos: 1) ausencia de glucosa y de sulfato de amonio (testigo); 2) 2% de glucosa; 3) 2% de sulfato de amonio; 4) 2% de glucosa y 2% de sulfato de amonio; 5) 2% de glucosa y 0,25% de 4-cianopiridina (Sigma-Aldrich,St. Louis, MO, EE. UU.); 6) 2% de glucosa y 0,1% de cianuro potásico; 7) 2% de glucosa y 0,5% de cianobenceno; 8) 2% de glucosa y 0,1mM de Trp; 9) 0,25% de 4-cianopiridina; 10) 0,1% de cianuro potásico; 11) 0,5% de cianobenceno, y 12) 0,1mM de Trp. Los cultivos se mantuvieron a 28°C en un agitador rotatorio a 150rpm durante 4, 8 y 12h por separado. La biomasa se recuperó por filtración, se lavó dos veces, se congeló con nitrógeno líquido y se almacenó a −80°C hasta su uso.
Caracterización de la actividad enzimática nitrilasa de Xylaria sp.La actividad enzimática se midió utilizando 4-cianopiridina como sustrato en un caldo de patata y dextrosa (PDB), en cultivos de 7 días. Se añadió caldo de cultivo previamente filtrado con membrana de 0,22μm (0,1ml) a 0,7ml de solución de sustrato que contenía 40μl de 4-cianopiridina (50mM) en un tampón de acetato de sodio (100mM, pH 7,5). La mezcla se incubó a 37°C durante 20min y luego se añadieron 100μl de HCl 1M para detener la reacción. Se incubó un volumen de 50μl de la mezcla de reacción durante 5min con 500μl de reactivo de Nessler (Sigma-Aldrich, St. Louis, MO, EE. UU.). Tras la centrifugación (12.000×g, 3min), se tomaron muestras del sobrenadante y se midió la absorbancia a 435nm en un espectrofotómetro Bio-Rad SmartSpec 3000.
Se preparó una curva estándar de absorbancia a 435nm frente a un gradiente ascendiente de alícuotas de cloruro de amonio (10-1000mM) y se cuantificó el amonio residual4. Una unidad de actividad nitrilasa se definió como la cantidad de enzima que formaba un μmol de amonio por min bajo las condiciones anteriores. La actividad específica de la enzima se expresó como la actividad nitrilasa por mg de proteína. La cantidad de proteína total se determinó según Bradford6 utilizando albúmina de suero bovino como estándar. El efecto del pH y la temperatura sobre la actividad de la nitrilasa y la estabilidad enzimática se midieron en el rango de 4,0 a 9,0 y de 30 a 50°C, respectivamente, utilizando 200μl de tampón y 200μl de la muestra, con un tiempo de reacción de 1h. La estabilidad térmica y de pH de la enzima se analizó incubando una alícuota de tampón de muestra a temperaturas de 30 a 50°C (1h) y a pH 4,0 a 9,0 (24h), y se calculó el porcentaje de actividad residual. Todos los ensayos se realizaron por triplicado.
Ensayos de resistencia a cianobenceno (crecimiento micelial)Se evaluó la capacidad de crecimiento micelial en medio líquido en presencia de diferentes concentraciones de cianobenceno, siguiendo el método propuesto por Gómez-Reyes et al., 201718 con algunas modificaciones. Para tal fin, se utilizaron placas de microtitulación. Cada pocillo contenía 200μl de PDB y se suplementó con el compuesto contaminante (0,1; 0,25; 0,5 y 1% v/v, respectivamente). La solución testigo solo contenía PDB. La inoculación se efectuó con 100 conidios de Xylaria. El crecimiento micelial se determinó midiendo la absorbancia a 590nm luego de 48h a 28°C. Los datos se expresaron como porcentaje de crecimiento fúngico, donde el valor de absorbancia más alto corresponde a un crecimiento del 100%. Los ensayos se realizaron por triplicado y la significancia estadística se evaluó empleando pruebas no paramétricas de Mann-Whitney (p≤0,05).
Ensayos de biofertilización fúngicaPara realizar los ensayos de biofertilización, se utilizaron semillas de A. thaliana. Estas se desinfectaron de manera superficial con una solución de etanol al 70% (v/v) y Tritón X-100 al 1% (v/v), posteriormente se hicieron tres lavados con agua destilada estéril. Luego de una estratificación durante 24h a 4°C, las semillas se sembraron en placas de Petri que contenían 0,8% de agar mezclado con sales basales Murashige y Skoog (MS) (Sigma-Aldrich,St. Louis, MO, EE. UU.) y sacarosa al 2% (p/v), además de un volumen de cultivo de Xylaria de 72h obtenido en medio líquido (MM) suplementado con 100mg/l de Trp. El pH final se ajustó a 5,7 y las condiciones de germinación fueron las siguientes: temperatura de 22°C con fotoperíodos de 16h de luz y 8h oscuridad (100 a 120 ∝E m-2 s-1) en una cámara de crecimiento con el 35% de humedad. El proceso de germinación se extendió por 10 días; como testigo se adicionó el mismo volumen de MM mezclado con agar, sales basales, sacarosa y Trp, pero sin inoculación fúngica.
Para apreciar mejor el efecto de los tratamientos sobre la estructura radicular de las plántulas, estas se retiraron de las placas y se realizaron fotografías con una cámara digital acoplada a un sensor infrarrojo. Se obtuvieron los datos promedio de las raíces principales y laterales de cuatro placas, con ocho plántulas por placa, para cada condición. La significancia estadística del conjunto de datos obtenidos se evaluó empleando pruebas no paramétricas de Mann-Whitney (p≤0,05).
Detección de AIA mediante HPLC-MSSe usaron 100 conidios de Xylaria para inocular matraces de 1l que contenían 400ml de MM sin glucosa, suplementado con sacarosa al 2%, 7ng de AIA deuterado (IAA-d2) y 100mg/l de Trp. Se incubó a 28°C y 180rpm durante 72h. Los sobrenadantes se concentraron a 10ml mediante vacío y se ajustaron a pH 2,8 con HCl 1 N. Para confirmar la presencia de AIA, se separaron y analizaron los componentes por HPLC seguida de espectrometría de masas. El análisis se realizó mediante la inyección de alícuotas de 10μl en una columna SunFire® C18 (2,1×150mm i.d.; 5mm) conectada a una bomba de HPLC (Waters), controlando la absorbancia a 260nm.
La fase móvil incluyó un programa isocrático que constó de una mezcla de agua: acetonitrilo: ácido acético (40: 60: 1). Bajo estas condiciones, el AIA deuterado y el nativo eluyen a 3,55 y 3,6min, respectivamente, y son detectados por la formación de iones negativos. La velocidad de flujo fue de 1ml/min con 3,800psi de presión y el tiempo de retención de los picos se comparó con el del AIA deuterado añadido al MM. El sistema de HPLC se sincronizó con un espectrómetro de masas QTRAP® 4500 SCIEX con trampa de iones lineal (LIT), de modo que se obtuvo un cromatograma iónico total (TIC) durante 30min. Los espectros electromagnéticos se generaron a partir del TIC.
Secuencias de aminoácidos y ácidos nucleicosLa secuencia genómica de identidad de la cepa aislada fue depositada en el repositorio del NCBI bajo el número de acceso ON843621.1.
ResultadosAislamiento y caracterización de la cepa fúngica nativaEmpleando claves morfológicas25, se llevó a cabo la identificación taxonómica preliminar de una cepa fúngica que fue aislada de un fruto de caoba en proceso de descomposición. Se observaron peritecios semiclaros, con algunas áreas punteadas ennegrecidas que contenían ascosporas. Los peritecios mostraron una forma ovoide prominente, de color oscuro, de 0,8-1,3mm de diámetro. Las ascosporas mostraron formas elipsoides-inequilaterales de color café claro. El estroma mostró una estructura teleomórfica cilíndrica (≈10-145×1-3mm); el ectostroma era de textura escamosa clara brillante y superficie estriada.
Ya que se consideró que los resultados anteriores no fueron en su totalidad concluyentes para determinar la correcta clasificación taxonómica del hongo, se complementó la identificación con el análisis de una secuencia nucleotídica concatenada (698pb) conformada por transcritos de genes ITS1-ITS2 y ARNr 5.8S, aunado al de una secuencia corta del factor de elongación EF-1a, como se describe en el apartado de materiales y métodos. Así, la secuencia de ADN consenso se relacionaba molecularmente con el genoma del género Xylaria.
Al realizar un análisis BLAST para identificar posibles organismos que coincidieran con la muestra, se filtraron las accesiones del NCBI correspondientes a aquellas especies que mostraban un mayor porcentaje de similitud en el análisis. Las accesiones respectivas fueron las siguientes: 1) NR_153200.1: Xylaria bambusicola (98,13%); 2) AB495009.1 Xylaria sp. (97,61%); 3) MN846317.1 Xylaria necrophora (95,67%); 4) DQ322153.1 Xylaria maitlandii (98,60%), y 5) GU339495.1 Xylaria arbuscula var. plenofissura (93,67%). Finalmente, de las cinco accesiones anteriores, Xylaria sp. fue el microorganismo que sugirió un mayor grado de alineación de algunas de sus regiones conservadas de genes ITS y del factor de elongación con la secuencia nucleotídica concatenada obtenida a partir de la PCR empleando los marcadores moleculares respectivos. Este resultado fue corroborado por un análisis comparativo por expectativa logarítmica (MUSCLE)29.
En la figura 1 se muestra la alineación de las secuencias nucleotídicas pertenecientes a los seis hongos (incluida la cepa nativa). Al realizar una reconstrucción filogenética empleando el método NJ (500 réplicas), se generó una distribución de un clado y una reagrupación parafilética conformados por cuatro y dos taxones, respectivamente: 1) cepa nativa, X. maitlandii, X. bambusicola y Xylaria sp., y 2) X. arbuscula y Xylaria necrophora. El primero de ellos mostró un arreglo típico escalonado en donde Xylaria sp. enraíza la arquitectura de las ramas. La segunda reagrupación se origina a partir de una distribución parafilética. La reconstrucción sugiere que Xylaria sp. es el antecesor común de este grupo de hongos.
Análisis comparativo por expectativa logarítmica de seis regiones de secuencias genómicas altamente conservadas en especies del género Xylaria. El estudio incluye la secuencia óptima concatenada (nativa) de la cepa fúngica aislada en campo. Los asteriscos representan los residuos de nucleótidos idénticos. La reconstrucción filogenética se realizó empleando el método estadístico de Neighboor-Joining (500 réplicas) complementado por un modelo de probabilidad máxima compuesta.
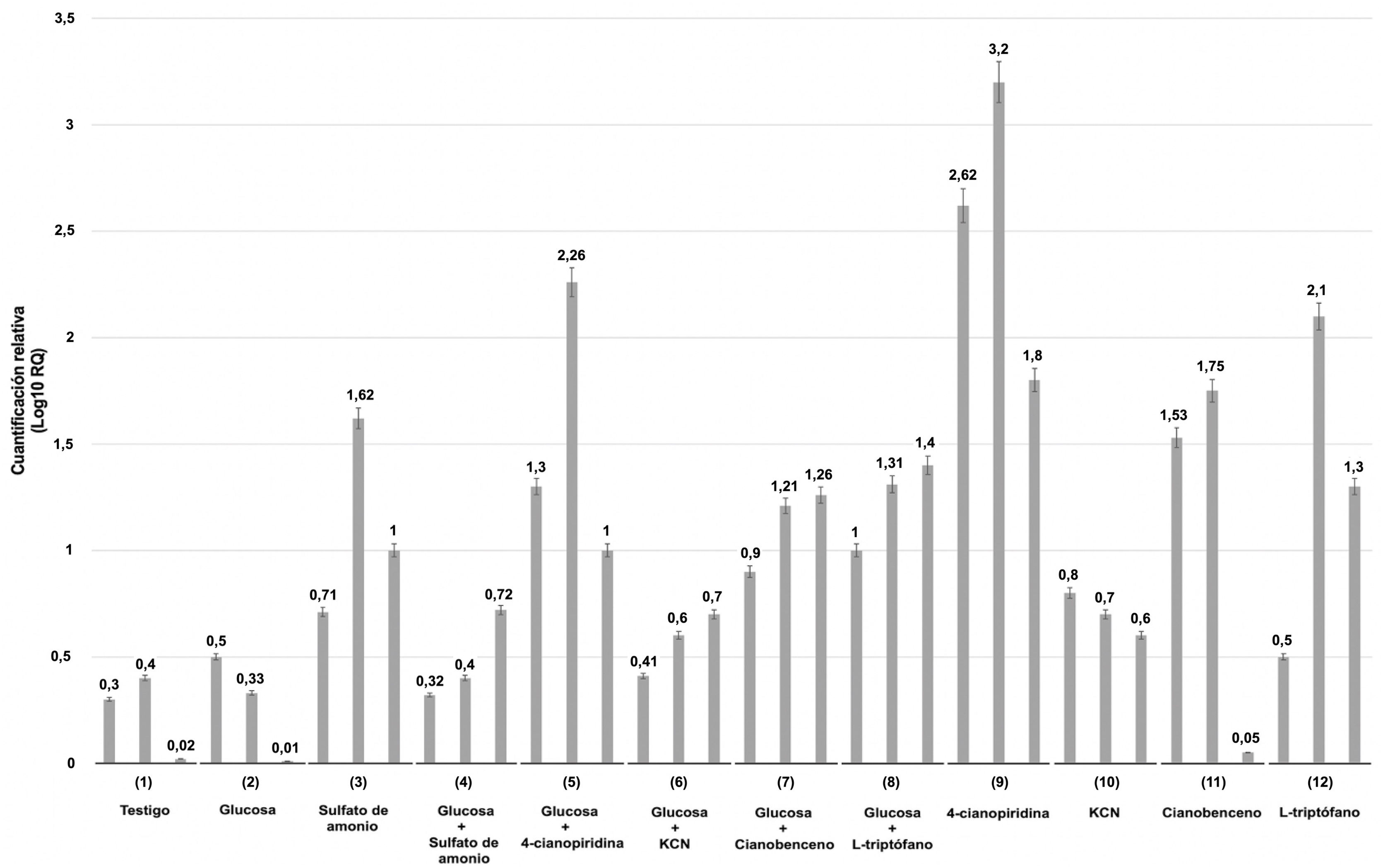
Se evaluó mediante qRT-PCR la expresión relativa de un gen de Xylaria sp. que codifica una enzima nitrilasa putativa bajo condiciones de cultivo tras 4, 8 y 12h de fermentación, en presencia o ausencia de glucosa como única fuente de carbono. De manera complementaria, los cultivos se suplementaron con las siguientes fuentes de nitrógeno inductoras: sulfato de amonio, 4-cianopiridina, cianuro potásico, cianobenceno y Trp.
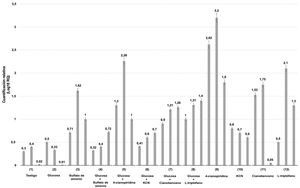
Los niveles de transcripción se compararon con aquellos detectados en las condiciones de cultivo testigo (ausencia de glucosa y de fuentes de nitrógeno). La figura 2 muestra que la expresión génica relativa se reprimió tanto en condiciones basales (ausencia de glucosa y nitrógeno) como en presencia de glucosa sola y cianobenceno solo a las 12h de cultivo. Los menores niveles de expresión se detectaron a las 4, 8 y 12h de fermentación en presencia de glucosa, glucosa más sulfato de amonio, glucosa más cianuro potásico, cianuro potásico y Trp.
Niveles de expresión génica relativa de una nitrilasa putativa de Xylaria sp. Los valores se expresan como log10 de 2-ΔΔCT. Se utilizó el gen de la β-tubulina como referencia endógena. Las líneas superiores de cada barra representan las desviaciones estándar de los valores de la media de las réplicas biológicas. Los niveles de expresión génica de cada ensayo (1-12) se muestran de manera individual y se componen del resultado de 4, 8 y 12h de fermentación.
Por otra parte, la presencia de sulfato de amonio, glucosa más cianobenceno, glucosa más Trp y cianobenceno en los medios de cultivo indujeron niveles medios de transcritos. Así, se sugiere que la mayor expresión génica es inducida por la presencia de glucosa suplementada con 4-cianopiridina, cianobenceno y, sobre todo, por la 4-cianopiridina sola. Es importante mencionar que la cinética enzimática sugiere un comportamiento exponencial, en donde, en promedio, el valor más alto de expresión se observa a las 8h de fermentación para la mayoría de los casos.
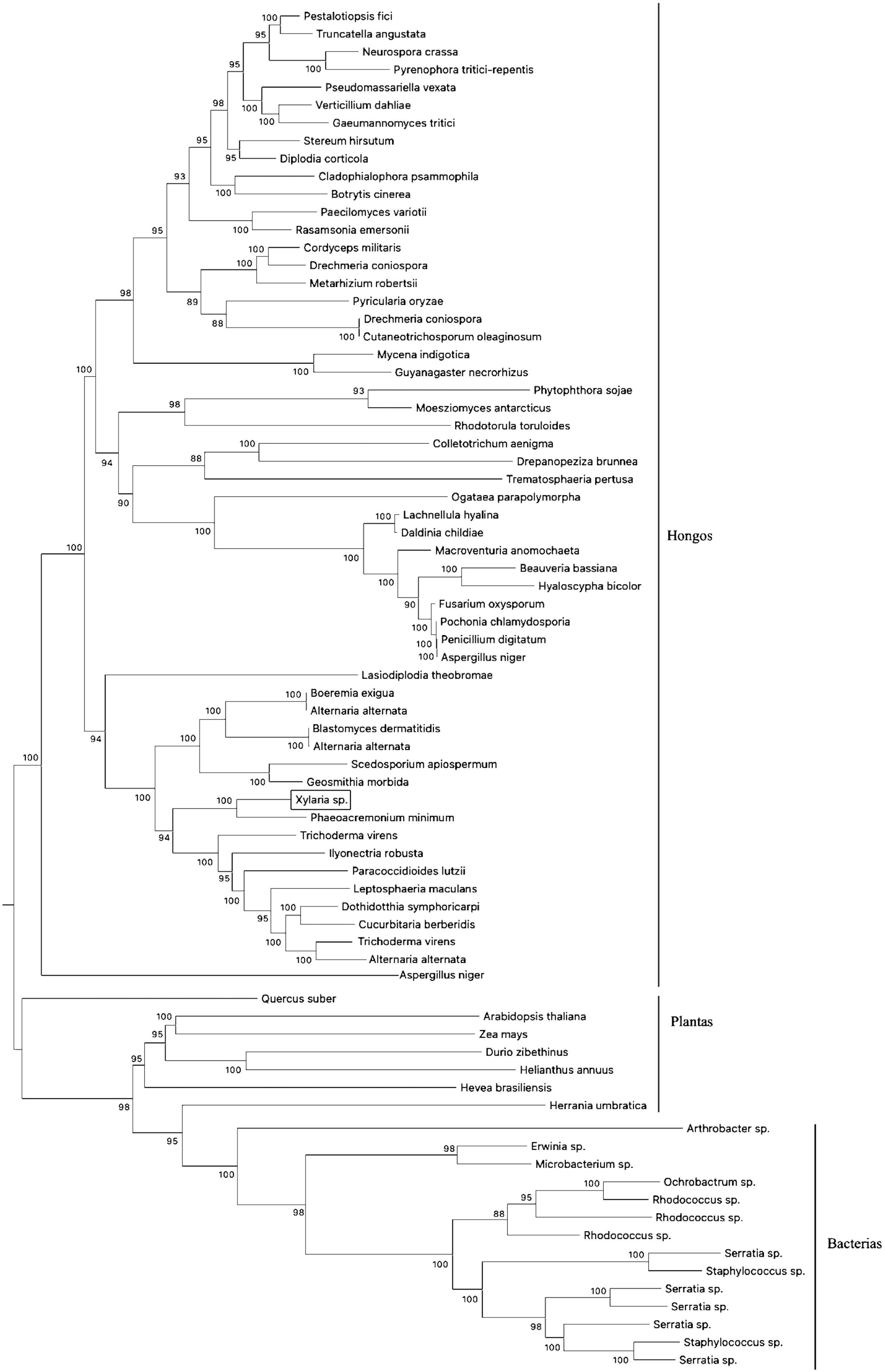
Evolución molecular taxonómica y caracterización de secuencias nucleotídicas homólogas de Xylaria sp.El resultado de la reconstrucción filogenética (análisis evolutivo molecular óptimo) de las secuencias de nucleótidos homólogas de genes caracterizados e hipotéticos que transcriben enzimas nitrilasas de diversas especies se muestra en la figura 3, en la que se incluye el transcrito amplificado de la cepa fúngica nativa. La suma total de la longitud de las ramas eliminando todas las posiciones que contenían espacios faltantes fue de 0,01778648 sustituciones por sitio; incluyendo las distancias evolutivas para cada una de estas tomando en cuenta la probabilidad máxima compuesta bajo el modelo de Tamura-Nei. Asimismo, se observó un total de 1.924.236 posiciones en el conjunto de datos finales y se calculó el resultado de divergencia evolutiva con estimadores mínimos.
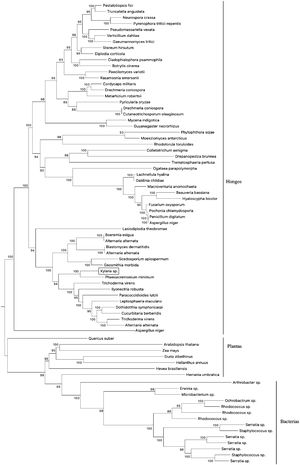
Reconstrucción filogenética de secuencias nucleotídicas caracterizadas e hipotéticas que transcriben enzimas nitrilo-hidrolíticas (incluyendo genes homólogos) en especies de distintos reinos. El cladograma se realizó empleando el método estadístico Neighbor-Joining (500 réplicas) complementado por un modelo de probabilidad máxima compuesta. La base de datos se construyó a partir de accesiones pertenecientes al NCBI. La secuencia homóloga de Xylaria sp. se encuentra resaltada en un recuadro.
La arquitectura de la divergencia evolutiva sugiere la presencia de 3 clados concomitantes que se reagrupan de acuerdo con su naturaleza (es decir, bacterias, plantas y hongos). El primer clado (bacterias) se compone de 14 taxones reagrupados a través de ramificaciones monofiléticas, con uno y 2 taxones terminales. Para todos los casos, los nodos mostraron una tasa de repetición de reconstrucción de ≈98% en el remuestreo replicado (bootstrapping). El segundo clado (plantas) se compone de siete taxones. Cuatro de estos provienen de ramificaciones monofiléticas y los restantes se generan a partir de nodos individuales. Este segundo clado se emparenta directamente con el primero a través de un taxón basal (Arthrobacter sp.).
El tercer clado (hongos) es el más representativo de todos y está compuesto por 55 taxones divididos en dos grupos. El primer grupo posee 18 taxones y en este se encuentra Xylaria sp., directamente emparentada con Phaeoacremonium minimum a través de un nodo parafilético. El segundo grupo posee ramas más grandes y se compone de 37 taxones separados por dos ramas monofiléticas. En este caso, la mayoría de los taxones poseen relaciones bilaterales a partir de nodos individuales.
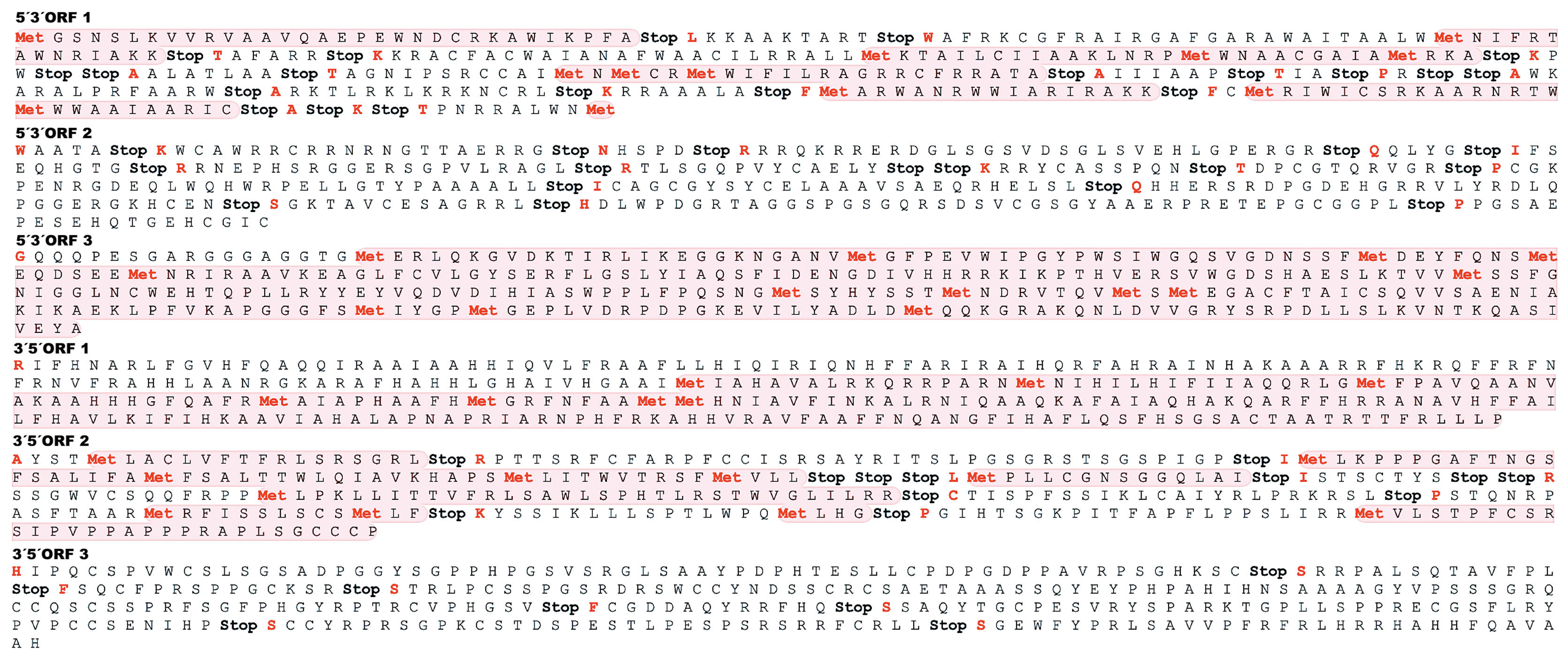
En la secuencia nucleotídica de Xylaria sp. se encontraron al menos seis ORF (3 para cada orientación), incluyendo tríadas catalíticas y codones de parada (fig. 4). Por otra parte, el análisis de dominios conservados a través del NCBI sugiere la presencia de al menos nueve sitios activos putativos, 3 tríadas catalíticas y varias interfaces de dimerización. La clasificación proteica dominante correspondió a la familia de las carbono-nitrógeno hidrolasas (ID 10166026) (superfamilia nitrilasa; cd07546).
Representación gráfica de seis orientaciones distintas de conjuntos de marcos de lectura abierta (ORF) de la secuencia homóloga de Xylaria sp. que transcribe enzimas nitrilasas (traducida a aminoácidos). Los fragmentos de secuencias de codones resaltadas en color claro representan los ORF para cada orientación. La palabra Stop representa un codón de parada. Los residuos marcados en rojo refieren sitios activos, tríadas catalíticas e interfaces de dimerización.
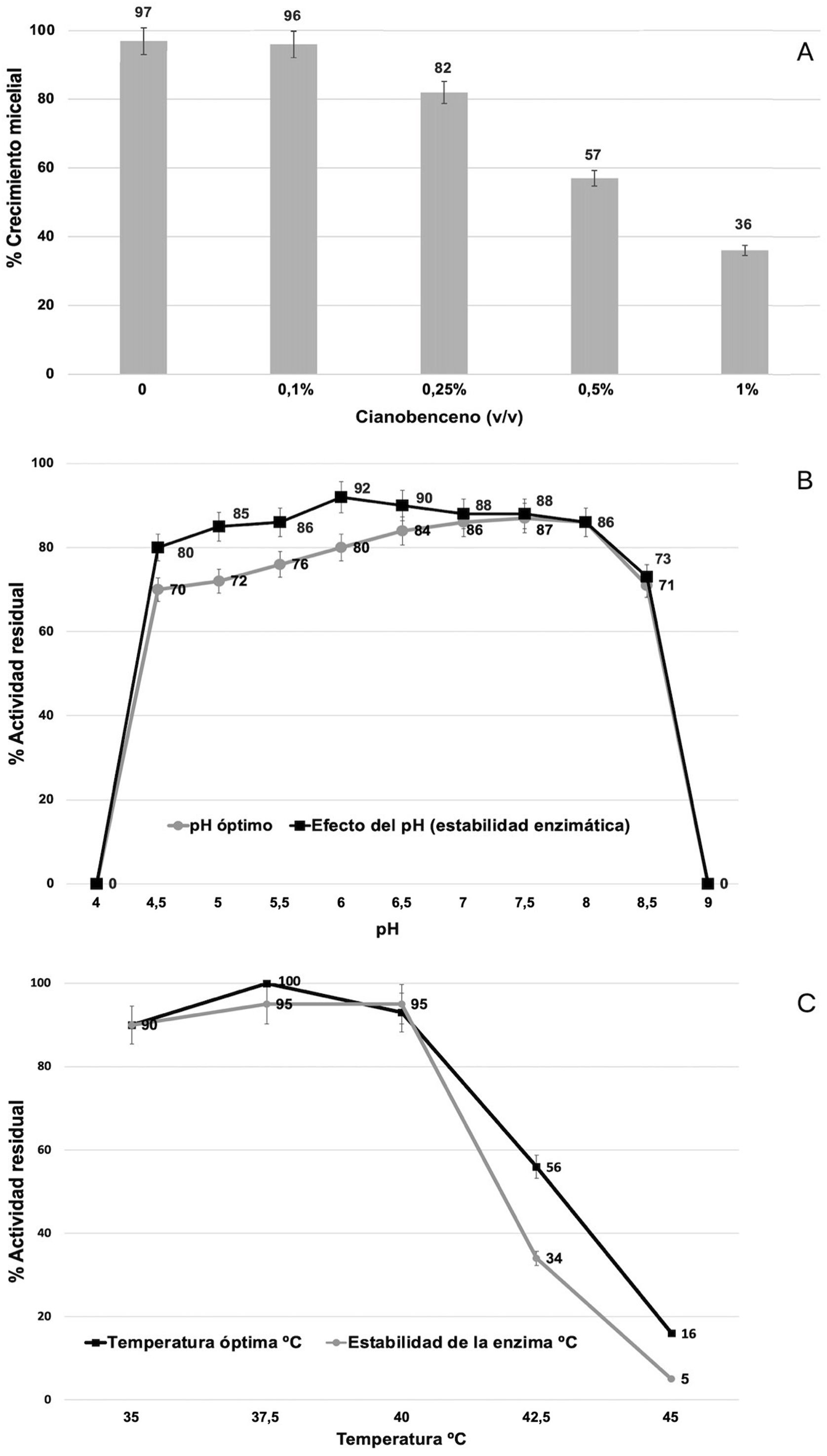
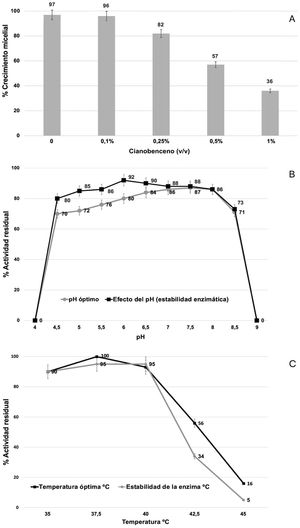
Dado que existe una correlación entre el modelo de expresión génica y la actividad enzimática nitrilasa de la cepa fúngica, se sugirió que Xylaria sp. podría llevar a cabo procesos de degradación de compuestos nitrogenados complejos. En virtud de ello, se ejecutaron ensayos de evaluación de crecimiento micelial en presencia de distintas concentraciones (0,1; 0,25; 0,5 y 1% v/v) de cianobenceno para analizar la tolerancia del hongo a dicho compuesto, cabe destacar que en ausencia de este y hasta una concentración del 0,25%, no se observaron diferencias estadísticamente significativas. Si bien el 100% del crecimiento se alcanzó en 48h, a concentraciones del 0,5 y 1%, la proliferación micelial disminuyó a más del 70%. Para ambos casos, las diferencias fueron estadísticamente significativas (fig. 5A).
Porcentajes de crecimiento micelial de la cepa de Xylaria sp. y caracterización de parámetros termodinámicos de actividad enzimática nitrilasa. A) El porcentaje de crecimiento micelial se evaluó en presencia de diferentes concentraciones (v/v) de cianobenceno a 28°C durante 48h. B) Efecto del pH en la actividad enzimática nitrilasa durante 1h de incubación. C) Efecto de la temperatura en la estabilidad de la enzima nitrilasa durante 24h de incubación. Para todos los casos se representan las desviaciones estándar de las medias de las tres réplicas.
Por otra parte, se evaluó la actividad enzimática nitrilasa de Xylaria sp. después de cultivar la cepa durante 7 días en medio PDB suplementado o no con 4-cianopiridina (50mM) como sustrato. Los valores de actividad nitrilo-hidrolítica registrados fueron los siguientes: cultivos en medio PDB: 0,023±0,006U/mg proteína; cultivos en medio PDB+4-cianopiridina: 0,645±0,021U/mg proteína. La diferencia entre estos valores fue estadísticamente significativa. Respecto del estudio de los parámetros termodinámicos de la enzima, se observó que esta funciona a un pH óptimo de entre 6,0 y 7,0, con una estabilidad máxima a pH 7,5 (fig. 5B), y que su temperatura óptima es de alrededor de 37°C, con una estabilidad máxima a 40°C (fig. 5C).
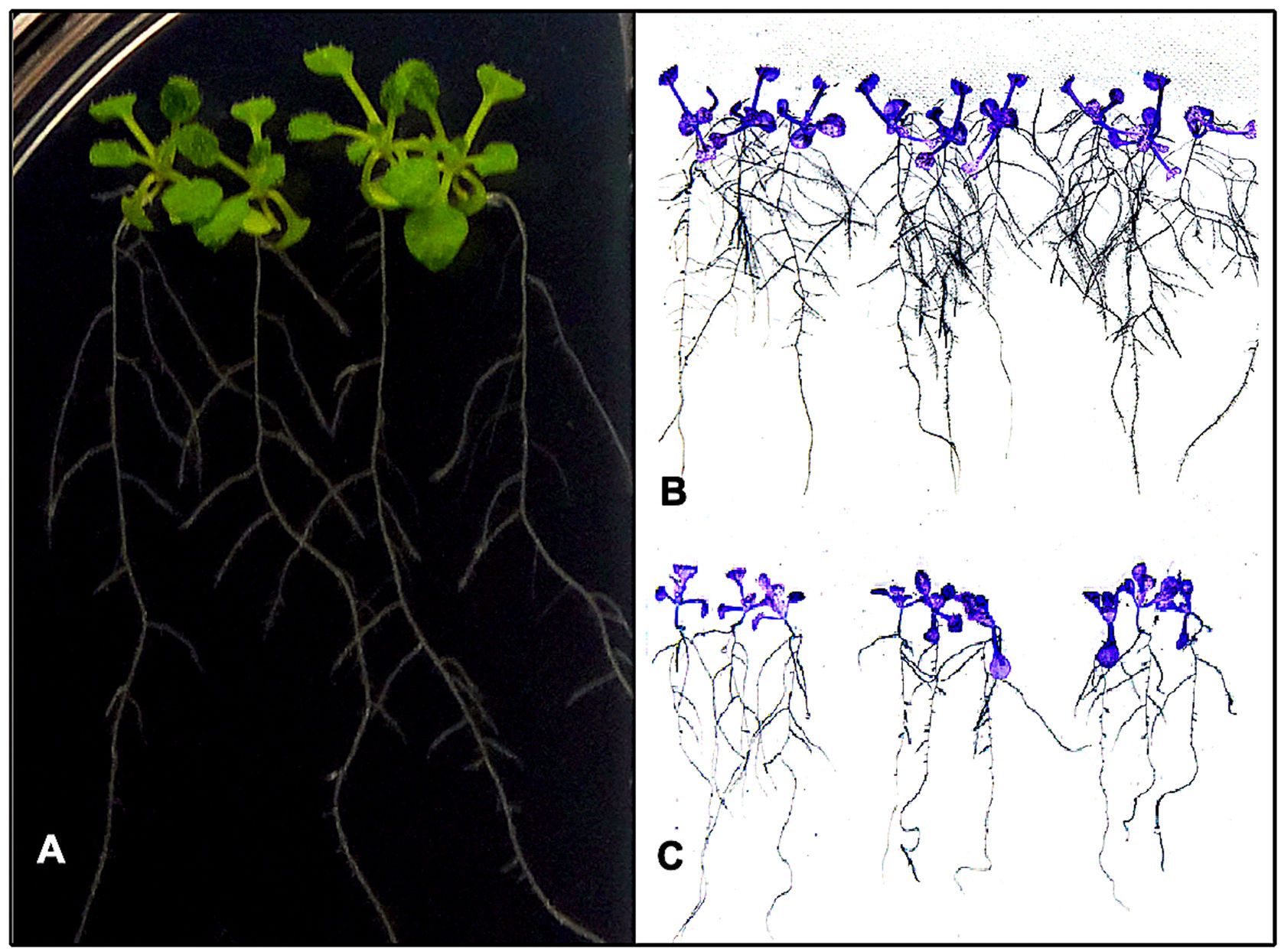
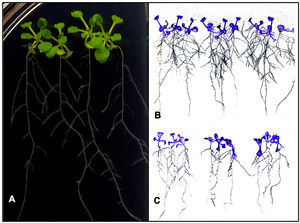
Efecto de la biofertilización fúngica en la arquitectura radicular de A. thalianaEs bien sabido que algunas cepas de Xylaria actúan como simbiontes de plantas16 y que las enzimas nitrilasas están involucradas en la biosíntesis de AIA9. Para estudiar los posibles efectos que ejerce Xylaria sp. en el crecimiento vegetal, se biofertilizaron plántulas de A. thalianain vitro, que se mantuvieron por hasta 10 días (fig. 6A). Transcurrido este tiempo, se observó un aumento considerable en la longitud de la raíz primaria en los individuos que fueron tratados con extractos de Xylaria (4,16±0,03cm), al igual que en la longitud (0,94±0,09cm) y el número (19±2,4) de sus raíces laterales (fig. 6B). En este sentido, cuando se inoculó únicamente con MM, la longitud de la raíz primaria fue menor (3,09±0,04cm), al igual que el número de raíces laterales (10,21±1,93) (fig. 6C). Estas diferencias fueron estadísticamente significativas.
Efecto de la biofertilización fúngica in vitro sobre el sistema radicular de plántulas de A. thaliana. A) Plántulas de cinco días de germinación cultivadas sobre agar (MS) previamente inoculado con un volumen de MM fermentado durante 72h con la cepa nativa de Xylaria. B) Plántulas biofertilizadas recuperadas de placas después de 10 días de germinación fotografiadas con sensor infrarrojo. C) Plántulas sin biofertilizar recuperadas de placas después de 10 días de germinación fotografiadas con sensor infrarrojo.
Dado que las nitrilasas están involucradas en la vía biosintética del AIA dependiente de Trp a través del IAN en plantas8,9, se analizó la capacidad de Xylaria sp. de biosintetizar este compuesto indólico. Las incubaciones se realizaron aplicando agitación en MM con sacarosa al 2%, suplementando o no con 100mg/l de L-Trp. Los caldos se filtraron, liofilizaron y extrajeron como se indica en el apartado de materiales y métodos.
Posteriormente, se llevó a cabo un análisis de HPLC/MS, que reveló la presencia de AIA; las cantidades más altas se encontraban en los extractos fúngicos suplementados con Trp. Así, el hongo produjo 170mg/ml de AIA en cultivos de 72h de fermentación. En ausencia del aminoácido en el medio, la cepa produjo cantidades inferiores a 35mg/ml de dicho compuesto.
DiscusiónEl presente estudio contribuye a la comprensión de la naturaleza de las enzimas nitrilasas incluyendo su relación con los hongos ascomicetes, a los que se les atribuyen propiedades benéficas para las plantas, entre otras. En esta investigación, se caracteriza y analiza por primera vez la actividad enzimática nitrilasa y la biosíntesis de ácido indol-3-acético por parte de Xylaria sp. En los hongos filamentosos, todas las nitrilasas conocidas son inducibles13,31, sin embargo, el mecanismo molecular de tal inducción aún no se ha dilucidado completamente.
Varios estudios han correlacionado la actividad de las nitrilasas con la conversión del IAN en AIA, el cual es considerado un factor de crecimiento vegetal imprescindible3,15,21,44. Desde la década iniciada en 1980, las bacterias han sido explotadas como fuente de enzimas hidrolíticas, lo que se ha aprovechado para diversas síntesis bioquímicas y, principalmente, para la remediación ambiental1. Las nitrilasas fúngicas han sido menos exploradas que las nitrilasas bacterianas, aun cuando varios informes sugieren que la presencia de péptidos aromáticos en hongos filamentosos es común24,31.
La superfamilia de las nitrilasas se divide en 13 ramas, según la identidad de la secuencia nucleotídica y la actividad catalítica22. Los péptidos que hidrolizan los nitrilos (R-CN) al correspondiente ácido carboxílico (R-COOH) y amoníaco son, quizás, los que han sido mejor caracterizados dentro de todos los miembros, con numerosos ejemplos en los diversos reinos19,24,40.
Mientras que la mayoría de las nitrilasas son específicas para los nitrilos aromáticos, otras sólo tienen preferencia por los arilacetonitrilos, los nitrilos alifáticos, el bromoxinil o el cianuro33. Una comparación de la capacidad de diferentes nitrilos para inducir actividades nitrilasas en hongos filamentosos mostró que la 2-cianopiridina, que es un sustrato pobre para las nitrilasas fúngicas, actúa como un potente inductor de nitrilasas en cepas pertenecientes a los géneros Fusarium, Aspergillus y Penicillium31. Nuestros resultados muestran que cuando se utilizaron compuestos nitrílicos como el cianobenceno o la 4-cianopiridina como única fuente de carbono y nitrógeno, los niveles de transcripción aumentaron.
Como se muestra en la figura 2, la expresión génica de Xylaria se redujo principalmente en medios de cultivo que contenían glucosa y aumentó en presencia de L-Trp y 4-cianopiridina a las 4, 8 y 12h de fermentación. Sin embargo, cuando el hongo creció en medios que contenían 0,1mM de Trp en condiciones de inanición de glucosa y sulfato amónico, se detectó un aumento progresivo de la expresión, principalmente a las 8h. Este resultado podría deberse a que el Trp es una fuente de nitrógeno relativamente accesible para el hongo.
Se sabe que las nitrilasas son inducidas por tres compuestos nitrílicos diferentes que se clasifican según su estructura. Estos compuestos son aromáticos o heterocíclicos (benzonitrilo), alifáticos (acetonitrilo) y arilacetonitrilos (fenilpropionitrilo). Muchas de las enzimas tienen una amplia especificidad de sustrato, mientras que otras son menos específicas20,32,39,40.
En estudios anteriores, se ha observado una elevada actividad específica de las nitrilasas fúngicas hacia sus sustratos preferentes, como el cianobenceno y sus análogos31. Sin embargo, se han encontrado diferencias notables entre las nitrilasas de Fusarium oxysporum f. sp. melonis y las de Fusarium solani o Aspergillus niger respecto de sus actividades relativas hacia los sustratos benzonitrilo y 4-cianopiridina. La rama de las nitrilasas se distingue de otros miembros de la superfamilia por la presencia de un motivo Cys-Trp-Glu conservado, posicionado en el residuo Cys de la tríada catalítica, además de que tienen un grupo sulfhidrilo que es esencial para la actividad catalítica20,22. En la figura 4 se observan algunos ORF en los que se sitúan sitios catalíticos activos de la secuencia nucleotídica homóloga de Xylaria sp. que codifica enzimas nitrilasas.
Así, los resultados de expresión génica sugieren que un probable gen putativo de la cepa posee actividad nitrilasa aromática y arilacetonitrilasa, similar a la observada en Brassica rapa20. En cualquier caso, los valores más altos de actividad nitrilasa se observaron en presencia de 4-cianopiridina, un sustrato arilacetonitrilasa. Las nitrilasas con la misma especificidad de sustrato a menudo muestran similitud de secuencia de aminoácidos y pueden reagruparse dentro del mismo clado en los análisis filogenéticos15. Como se muestra en la figura 3, el clado principal en el que se agrupó Xylaria sp. incluye taxones cuyas enzimas nitrilasas prefieren sustratos arilacetonitrilasa.
Por otro lado, las nitrilasas fúngicas conocidas presentan una mayor estabilidad que las nitrilasas bacterianas31, por lo que en este trabajo se utilizó micelio de cultivos de Xylaria de 7 días de edad para estudiar la actividad hidrolítica. No obstante, en trabajos anteriores con cepas de Fusarium, Aspergillus o Penicillium se utilizaron micelios de 2-3 días de edad, y se sugirió una vida media de 12 días a 35°C para la nitrilasa de F. solani43. De manera conjunta, los valores de la actividad hidrolítica indican que la presencia de 4-cianopiridina en el medio de cultivo aumentó los niveles de expresión de Xylaria, y también se observó una preferencia hacia el cianobenceno y el L-Trp.
La capacidad de las nitrilasas para degradar contaminantes ambientales es importante en la biorremediación2,20,22 y, dado que Xylaria pertenece a una clase de hongos de vida libre que son comunes en la rizosfera, analizamos el potencial de detoxificación de la cepa en presencia de cianobenceno, y se observó que el hongo es capaz de desarrollarse a concentraciones moderadas de este contaminante. Entre muchos compuestos químicos, el cianobenceno es una de las moléculas inorgánicas con mayor impacto ambiental, ya que es el componente activo de numerosos herbicidas30. Por lo anterior, se infiere que Xylaria sp. posee resistencia a estos tipos de compuestos nitrogenados.
Se ha documentado que la biosíntesis de AIA a partir de Trp procede a través de ácido indol-3-pirúvico (IPyA) e indol-3-acetaldehido (IAAld) en Ustilago esculenta11. En nuestro estudio, hemos detectado la presencia de AIA en cultivos líquidos de Xylaria cuando se añadieron al menos 100mg/l de Trp al medio nutritivo. Durante muchos años se asumió que el Trp era el único precursor del AIA. Sin embargo, un trabajo con mutantes auxotróficos de Trp sugirió que la biosíntesis de AIA puede ocurrir por una ruta independiente de este aminoácido14, aunque en presencia de Trp, los microorganismos liberan mayores cantidades de AIA y algunos otros compuestos indólicos relacionados3,12.
El IAN es un intermediario para la producción de AIA y la síntesis de esta biomolécula no se limita únicamente a las plantas superiores, ya que diversas vías intermediarias de compuestos indólicos operan en el metabolismo de miles de bacterias que habitan la rizosfera de las plantas: estas incluyen el IPyA, la indol-3-acetamida (IAM), la triptamina (TAM) y el IAN14. Asimismo, se han identificado AIA, triptofol, IAAld, IAM, IPyA y ácido indol-láctico a partir de cultivos de Colletotrichum acutatum suplementados con Trp, lo que demuestra que este hongo patógeno también puede sintetizar AIA10.
Como se mencionó anteriormente, se ha sugerido que Xylaria tiene la capacidad de colonizar el sistema radicular y promover el crecimiento de las plantas7. En este sentido, se observó que Xylaria sp. estimula el sistema radicular de plántulas de Arabidopsis. En otros trabajos se ha demostrado que Trichoderma virens promueve el crecimiento de las raíces laterales en Arabidopsis a través de un mecanismo dependiente de AIA o IAAld por igual12.
Nuestros ensayos de medición comparativa de raíces principales y laterales de plántulas de Arabidopsis germinadas en presencia de extractos de Xylaria sugieren que los efectos de estimulación del desarrollo vegetal por la inoculación fúngica probablemente se producen por la producción de AIA8,14,15. Por lo anterior y tomando en consideración nuestros resultados, se concluye que Xylaria es capaz de producir AIA, pero, sobre todo, en presencia de fuentes de nitrógeno, lo cual desencadenaría la estimulación y el desarrollo del sistema radicular en ciertas especies vegetales.
ConclusionesEl presente trabajo documenta la actividad nitrilo-hidrolítica llevada a cabo por una cepa del género Xylaria aislada en el campo. Si bien este microorganismo posee características que lo distinguen de diversos grupos de ascomicetes, como la biosíntesis de compuestos antibióticos y algunos promotores del crecimiento vegetal, los mecanismos de acción de tales biomoléculas sólo han sido dilucidados de manera parcial. En dicho sentido, el metabolismo y la actividad biológica de las enzimas nitrilasas forman parte del conjunto de rutas metabólicas imprescindibles para que se lleven a cabo estos fenómenos a través de interacciones planta-microorganismo. En virtud de las consideraciones anteriores, sin duda es importante que futuras investigaciones en el campo de la micología dirijan sus esfuerzos a comprender la naturaleza de las enzimas nitrilasas y, sobre todo, a profundizar en el estudio de su potencial biotecnológico.
FinanciaciónLa presente investigación no ha recibido ayudas específicas provenientes de agencias del sector público, sector comercial o entidades sin ánimo de lucro.
Conflicto de interesesLos autores declaran no tener conflicto de intereses.
A los revisores y lectores de este documento que contribuyeron sustancialmente a la mejora de su contenido, así como a la UNIDA del ITVER y la UV.