La utilización del antígeno prostático específico como herramienta diagnóstica en el cribado del cáncer de próstata se ve reflejada en un incremento en la incidencia, un incremento en el diagnóstico de cánceres más precoces y un aumento en los tratamientos con intención curativa, aun a costa de un sobretratamiento. Sabemos, por datos recogidos en la literatura, que no todo paciente con antígeno prostático específico elevado necesita biopsia, y que no todo paciente con diagnóstico de cáncer de próstata necesita tratamiento. Con los nuevos marcadores prostáticos emergentes vamos a tratar de mejorar la especificidad del antígeno prostático específico en la zona gris (4-10 ng/ml) evitando biopsias innecesarias, de mejorar la sensibilidad en la detección de cáncer de próstata significante con antígeno prostático específico bajo y a intentar reducir el riesgo de sobretratamiento. Por otro lado, los biomarcadores pronósticos con test genómicos nos van a ayudar a elegir la mejor opción terapéutica para el paciente.
The use of prostate-specific antigen as a diagnostic tool in the screening of prostate cancer is reflected in an increase in the incidence, an increase in diagnosis at initial stages, and an increase in radical therapies, even at the expense of over-treatment in some cases. It is known from the data collected in the literature that not every patient with high prostate-specific antigen needs a biopsy, and that not every patient diagnosed with prostate cancer needs treatment. With the new emerging prostate markers, we will try to improve the specificity of prostate-specific antigen in the grey area (4-10 ng/ml) should be improved. This should avoid unnecessary biopsies. The sensitivity in the detection of significant prostate cancer with low prostate-specific antigen should also be improved in an attempt to reduce the risk of over-treatment. On the other hand, prognostic biomarkers with genomic tests will help to choose the best therapeutic option for the patient.
El antígeno prostático específico (PSA) se ha venido utilizando ampliamente en la práctica clínica para el cribado, diagnóstico y seguimiento del cáncer de próstata (CaP). Un tacto sospechoso en pacientes con PSA < 4 ng/ml tiene un valor predictivo positivo de presentar cáncer del 5-30%1, el 60% de los cuales son no organoconfinados2. Como variable independiente, el PSA es mejor predictor de cáncer que el tacto rectal o la ecografía transrectal3. No existe un valor de PSA por debajo del cual no exista riesgo de padecer CaP, ya que con valores de PSA entre 0 y 0,5 ng/ml existe una probabilidad de cáncer del 6,6%4.
La amplia utilización del PSA para el cribado de CaP ha generado un aumento importante en el número de biopsias, con incremento en la incidencia de CaP5 y en los tratamientos con intención curativa6. La biopsia guiada por ecografía transrectal puede no detectar el 20-30% de los cánceres clínicamente significativos7, muchos de ellos indolentes8, pero otros pueden amenazar la vida del paciente.
Esta utilización masiva de PSA en programas de cribado conlleva un coste en sobrediagnóstico y sobretratamiento de enfermedad indolente. Existen, por tanto, una serie de motivos que harían necesaria la búsqueda de nuevos marcadores que representen herramientas útiles en el diagnóstico y manejo clínico del CaP:
- -
Mejorar la sensibilidad del PSA en la detección de CaP significativos con valores iniciales de PSA bajos (17% de riesgo de CaP entre 1,1 y 2,0 ng/ml).
- -
Mejorar la especificidad del PSA en la «zona gris» (4-10 ng/ml) y, de este modo, evitar biopsias innecesarias.
- -
Reducir el riesgo de sobretratamiento (5-46%) en CaP indolentes.
- -
Aportar información pronóstica.
Describimos a continuación aquellos biomarcadores (tabla 1) que, a nuestro juicio, presentan más interés desde el punto de vista práctico y que el médico de Atención Primaria debería conocer.
Nuevos biomarcadores. Orientación
| Nuevos biomarcadores |
|---|
| ¿A quién biopsiar? |
| • ProPSA |
| • Índice de salud prostática |
| • 4Kscore PCA3 |
| ¿Cuándo repetir biopsia? |
| - PCA3 |
| - Prostate Cancer Mitomic Test (PCMT) |
| - Confirm MDx Test |
| - Genes de fusión (TMPRSS2-ERG) |
| ¿A quién tratar? |
| - OncotypeDX Test |
| - Prolaris Test |
| - Decipher Test |
| - Prostavysion Test |
| ¿A qué fármaco responde? |
| - Caris Molecular Intelligence Profile |
| ¿Quién orienta al patólogo? |
| - Quadvysion Test |
| - Ourview Prognostic Panel |
Las alteraciones del ADN provocan cambios de la expresión génica, bien en la metilación del ADN bien en la acetilación de histonas, y están a menudo involucradas en el desarrollo, progresión y recidiva tumoral9.
Uno de los productos de un gen hipermetilado es la glutatión-S-transferasa P1 (GSTP1). La GSTP1 pertenece a una familia de enzimas que catalizan la desintoxicación celular y modulan las vías de señalización implicadas en la proliferación y diferenciación celular y en la apoptosis. Se consideran enzimas protectoras. Cerca de un 70-80% de los CaP están metilados, mientras que las hiperplasias prostáticas benignas están hipometiladas10. La metilación de GSTP1 conduce a una pérdida de su expresión, que es uno de los principales eventos en la transformación de un fenotipo preneoplásico en fenotipo maligno11.
Utilizando muestras de orina después de un masaje prostático, se demostró la presencia de este marcador en el 68% de los hombres con enfermedad organoconfinada, que está presente solo en el 2% de los hombres con hiperplasias prostáticas benignas12.
La detección de metilación GSTP1 en muestras de orina de pacientes con sospecha de CaP puede ser un complemento útil a las pruebas de detección en suero (como el PSA) y al tacto rectal, para identificar pacientes con más riesgo de presentar CaP después de una biopsia negativa13.
Del tejido obtenido de piezas de prostatectomía radical se estudió el gen GSTM2, perteneciente también a la familia de la glutatión-S-transferasa, y el gen PENK, una proencefalina expresada en el estroma prostático que constituye una vía de señalización14. La pérdida de función de GSTM2 y PENK por hipermetilación se demostró que estaba implicada en el desarrollo de CaP.
El mismo trabajo anterior ha estudiado el gen CFTR, perteneciente a una superfamilia de proteínas transportadoras transmembrana, y el gen HTR1B. Se demostró que la hipermetilación simultánea de ambos genes se asociaba con CaP de alto riesgo y peor pronóstico (grado de Gleason ≥ 8, y niveles altos de ki-67).
A modo de resumen:
- -
La utilización de reacción en cadena de la polimerasa para determinar la hipermetilación de GSTP1 en líquidos orgánicos representa una herramienta emergente factible para la detección de CaP.
- -
El silenciamiento epigenético de GSTM2 y PENK son importantes marcadores moleculares para el diagnóstico de CaP. La hipermetilación simultánea de HTR1B y CFTR podría discriminar entre tumores de próstata indolentes y agresivos.
Los miARN son pequeñas cadenas de nucleótidos de ARN no codificante que juegan un papel importante en la tumorogénesis. Estos se excretan en el espacio extracelular y pueden encontrarse en diversos fluidos corporales (sangre, orina o eyaculado).
La aglutinación y centrifugación de lectina ha demostrado una sobreexpresión significativa de miR-574-3p, miR-141-5p y miR-21-5p en orina de pacientes con CaP, y a un bajo coste15.
Otro estudio16 comparó la expresión de miARN 21 y 221 en sangre de pacientes con y sin CaP. Se observó una sobreexpresión estadísticamente significativa de miARN 21 y miARN 221 en el 90 y el 80% de los pacientes con CaP, respectivamente, frente a solo el 10 y el 20% de los pacientes sin CaP, respectivamente.
A modo de resumen:
- -
Su alto valor predictivo negativo (90% para miARN 21 y 80% para miARN 221) y los altos valores de sensibilidad y especificidad (90 y 80% para ambos, respectivamente) los convierten en prometedores marcadores no invasivos para evitar biopsias innecesarias.
En el año 2005 se descubrió la fusión de genes entre el gen TMPRSS2 y distintos miembros de la familia ETS en el CaP, que se mostró como precursor de oncogénesis17. Esta fusión no se observaba en enfermedad benigna (lo que lo hace marcador específico), pero solo está presente en el 50% de los CaP (esto condiciona su sensibilidad como herramienta diagnóstica).
La presencia del gen de fusión en la orina se asocia con indicadores de cáncer clínicamente significativos tanto en la biopsia como en la pieza de prostatectomía18, incluyendo el tamaño tumoral y un grado de Gleason alto en la prostatectomía.
A modo de resumen:
- -
La presencia del gen de fusión en orina y la no detección de CaP en la biopsia empieza a plantearse como una indicación clara de rebiopsia temprana.
El objetivo de estos paneles multigénicos es el de optimizar su rendimiento, mejorar su capacidad diagnóstica y aumentar la sensibilidad y especificidad.
Existen diversos paneles multigénicos que, asociados al grado de PCA3, aumentan la sensibilidad de este usado de forma aislada. En el año 2008 se presentó el primer panel multigénico en orina, que combinaba 7 biomarcadores, incluyendo PCA3 y TMPRSS2-ERG19. Este modelo superaba al PSA sérico y al PCA3 solo en la detección de CaP.
El primer kit comercializado ha sido el Prostarix20. Se basa en un panel de marcadores bioquímicos excretados en orina que analizan su concentración y genera un valor. Este, combinado con el PSA y el volumen prostático, da lugar al Prostarix-Plus Risk Score, que estratifica el riesgo del paciente con biopsia previa negativa para cáncer oculto. No está comercializado en España.
A modo de resumen:
- -
Los modelos que incorporan diferentes biomarcadores optimizan el rendimiento y mejoran su capacidad diagnóstica.
Se han comenzado a realizar comparaciones genómicas de alteraciones genéticas asociadas con el CaP, utilizando matrices que permiten analizar polimorfismos de un solo nucleótido (SNP). Analizando 3SNP del gen 8q24 y un SNP de cada uno de los genes 17q12 y 17q24.3, se vio que aquellos hombres que presentaban alguna combinación de estas 5variantes de riesgo presentaban mayor OR (IC 95%) de presentar CaP21. Si además se incluía la historia familiar, el efecto acumulativo era mayor.
Por otro lado, se ha estudiado el receptor androgénico (AR) en pacientes con CaP resistente a la castración (CPRC), para determinar si puede haber variantes genéticas predictivas de la capacidad del tumor para continuar creciendo en ausencia de andrógenos22. La variante AR-V1 y AR-V7 se expresan 20 veces más en pacientes con CPRC que pacientes con CaP hormononaivë, lo que puede relacionarse con resistencia a tratamientos hormonales.
A modo de resumen:
- -
El receptor androgénico se relaciona con resistencias a tratamientos hormonales y predice recidiva bioquímica después del tratamiento quirúrgico.
El diagnóstico de CaP se basa en el estudio histológico de las muestras recogidas de las biopsias de tejido prostático. La ausencia de células basales es un criterio importante para la confirmación histológica de adenocarcinoma de próstata23.
Los marcadores de células basales más comúnmente utilizados son las citoqueratinas (CK) de alto peso molecular (como 34βE12, CK5, CK5/6 y CK14) y p63. Existen también moléculas que se comportan como marcadores inmunohistoquímicos de CaP, el más ampliamente estudiado de los cuales es el α-metilacil-coA racemasa (AMACR). Este se expresa en el 80-90% de los CaP24, aunque también en neoplasia intraepitelial prostática, adenosis, hiperplasias prostáticas benignas o atrofias.
La combinación de un marcador de células basales (34βE12 o p63) y la tinción para AMACR25,26 ha demostrado ser una herramienta inmunohistoquímica útil en la valoración de biopsias de próstata difíciles, con una alta sensibilidad y especificidad en la detección de pequeños CaP focales, lo que incrementa la precisión diagnóstica25.
A modo de resumen:
- -
La combinación de un marcador de células basales y la tinción para AMACR es una herramienta útil en la valoración de biopsias de próstata difíciles.
El PSMA es una glicoproteína de membrana de tipo II que se expresa fuertemente en el epitelio acinar prostático benigno, en neoplasia intraepitelial prostática y en el adenocarcinoma prostático. Su expresión es mayor en cánceres de alto grado y hormonoinsensibles, y es independiente de la expresión de PSA27.
El ProstaScint fue aprobado en 1996 por la FDA como un agente diagnóstico por imagen en pacientes con CaP28. Se compone de un anticuerpo monoclonal murino (7E11) que, conjugado con un radiomarcador (indium 111), se une claramente al PSMA. En combinación con TC o RM, ayuda a determinar la localización y extensión de la enfermedad metastásica a ganglios linfáticos. Presenta actividad limitada en el diagnóstico de tumores dentro de la próstata y las vesículas seminales29. Puede utilizarse para detectar recidiva en el contexto de una recidiva bioquímica temprana30,31, como predictor independiente de respuesta a radioterapia y para ayudar en la selección de pacientes para ablación crioquirúrgica de rescate o braquiterapia32.
A modo de resumen:
- -
El uso de ProstaScint estaría indicado en pacientes diagnosticados de CaP con alto riesgo de metástasis ganglionar.
- -
No está indicado para el cribado de CaP ni para valorar la respuesta a la terapia.
En la circulación sistémica el PSA puede presentar 2 formas moleculares. Un 15% está representado por el PSA libre (fPSA), compuesto por al menos 3diferentes tipos de PSA inactivo: PSA benigno, PSA inactivo intacto y pro-PSA. El 85% restante lo constituye el PSA complejo, unido a α1-antiquimotripsina (AQT) o a la α2-macroglobulina. La célula prostática maligna puede sintetizar AQT y liberar hacia la sangre el complejo PSA-AQT, con lo que la forma fPSA en los CaP estaría disminuida33.
El pro-PSA es un precursor inactivo del fPSA que se libera de las células epiteliales prostáticas con una secuencia guía de 7 aminoácidos (aa) conocida como [-7]pro-PSA. La calicreína humana (hk) 2 y, en menor medida, la hk4 escinden esta secuencia guía de 7 aa para activar el PSA. Este PSA activo sufre proteólisis y genera PSA inactivo. También puede sufrir degradación interna y formar PSA benigno. La escisión parcial de la secuencia guía de 7 aa produce formas inactivas de pro-PSA ([-2]pro-PSA o [-4] pro-PSA). Estas isoformas de pro-PSA son formas de PSA potencialmente más cáncer-específicas que pueden ayudar a diferenciar de manera más fiable entre CaP y enfermedad prostática benigna.
El pro-PSA se expresa casi exclusivamente en la zona periférica, donde se desarrollan la mayoría de los CaP34. Se demostró que [-2]pro-PSA mejoraba la discriminación entre CaP y enfermedad benigna en hombres con PSA entre 2,5 y 10 ng/ml y tacto rectal normal35.
El [-2]pro-PSA y %pro-PSA se elevaban con el incremento del grado de Gleason y en cánceres más agresivos36.
La capacidad predictiva de [-2]pro-PSA en la detección del CaP dentro de la «zona gris» de diagnóstico (PSA entre 2 y 10 ng/ml) parece ser óptima cuando se evalúa como parte de un modelo matemático. El modelo matemático que incorpora [-2]pro-PSA, tPSA y fPSA (([-2]pro-PSA/free PSA) × √PSA) es conocido como el índice PHI. Está desarrollado para su aplicación en pacientes con niveles de PSA de 2-10 ng/ml sin sospecha de CaP en el tacto rectal. Esta prueba se realiza en sangre y da como resultado una puntuación o «PHI score» que mejora significativamente la especificidad clínica del PSA, lo que permite disminuir el número de biopsias innecesarias y recomendar la biopsia con un PHI ≥ 55 (riesgo 52,1% de padecer cáncer). Los valores elevados de PHI se asocian con mayor probabilidad de encontrar CaP con Gleason ≥ 737.
En aquellos pacientes estudiados mediante biopsia de próstata por niveles de PSA entre 2 y 10 ng/ml, el PHI y %2pro-PSA eran los mayores predictores de biopsia positiva38 que podían predecir enfermedades más agresivas en la pieza de prostatectomía radical: resultaba útil en el asesoramiento previo a la cirugía en pacientes con CaP localizado39.
La FDA ha aprobado el índice PHI en la decisión de primera biopsia en hombres >50 años, con tacto rectal no sospechoso y valores de tPSA entre 4 y 10 ng/ml. Tanto %2pro-PSA como PHI pueden optimizar la indicación de una 2.ª biopsia. En programas de vigilancia activa los niveles séricos de PHI como %2pro-PSA fueron significativamente más altos en hombres que al final desarrollaron biopsia desfavorable durante el seguimiento40,41.
A modo de resumen:
- -
[-2]pro-PSA es la isoforma de pro-PSA más cáncer-específica de todas, con mejor capacidad diagnóstica y mejor correlación con tumores más agresivos.
- -
Tanto el índice PHI como %2pro-PSA pueden predecir la agresividad del CaP tanto en la biopsia como en la pieza de prostatectomía radical.
- -
Comparado con otros marcadores, solo el índice PHI se correlaciona con el grado de Gleason, no así el PCA3 ni el TMPRSS2:ERG.
El gen DD3PCA3 es uno de los primeros biomarcadores que pueden utilizarse ya en la práctica clínica habitual. El gen fue identificado en 1999 mediante la comparación de patrones de expresión de ARNm entre el tejido tumoral y el adyacente no tumoral. Se localiza en el cromosoma 9q21-22 y se transcribe de forma exclusiva en tejido prostático, sobreexpresándose intensamente en casi todos los tumores prostáticos y en lesiones metastásicas de próstata en comparación con el benigno cercano42.
Se comercializa como un score definido como la ratio del ARNm-PCA3/ARNm-PSA×1.000. Su expresión se detecta en 30ml de orina recogida tras masaje prostático, con el que se obtiene un score de PCA3 (s-PCA3)43. Se considera positivo un s-PCA3 igual o superior a 35, que se interpreta como de probabilidad elevada de detectar CaP en una biopsia posterior43. La sobreexpresión de este gen es específica de CaP e independiente del volumen prostático, del número de biopsias previas, de la presencia de hiperplasias prostáticas benignas y del valor de PSA sérico44. El empleo de los inhibidores de la 5α-reductasa (ARI) no parece influir en el score45.
Existe una relación directamente proporcional entre el s-PCA3 y el porcentaje de biopsias positivas posteriores46. El PCA3 ahorra la mitad de las biopsias (49%), basado sobre todo en su alto valor predictivo negativo47,48.
El s-PCA3 se comporta, así mismo, como indicador de agresividad tumoral: el 76,7% de los casos con s-PCA3 superior a 50 presentaron CaP de alto grado (Gleason ≥ 7)49.
Dentro de un programa de cribado oportunista en pacientes con PSA ≥ 3 ng/ml y tacto rectal normal, se pueden ahorrar un 65,7% de las biopsias empleando el PCA3 como segundo biomarcador con un punto de corte de 3550.
Varios estudios han identificado una relación entre el s-PCA3 y el grado de Gleason, el volumen tumoral y la invasión extracapsular47,49,51.
El PCA3 mejora el diagnóstico de biopsias de repetición. Aquellos con s-PCA3 < 25 presentaron una probabilidad 4,56 veces mayor de tener una segunda biopsia negativa que los que tuvieron un s-PCA3 ≥ 2552.
El Pregensa PCA3 assay fue autorizado por la FDA en 2012 para varones de 50 años o más con una o más biopsias prostáticas negativas, si bien algún estudio considera su utilización en pacientes sin biopsia previa y con un s-PCA3 > 6053.
En cuanto al valor pronóstico de PCA3, este puede mejorar los modelos predictivos de bajo volumen tumoral y de CaP insignificante cuando se añade el PCA3 a factores de riesgo clínico estándar54.
A modo de resumen:
- -
Su principal indicación es determinar si es necesario repetir la biopsia tras una primera o varias biopsias negativas.
- -
Se puede considerar su utilización en pacientes sin biopsia previa y con un s-PCA3 > 60.
- -
Un s-PCA3 > 35 presenta una alta probabilidad de CaP en la biopsia, con valor pronóstico cuantitativo (Gleason).
- -
Su valor no depende del volumen prostático, del nivel de PSA, de una biopsia previa, de inflamación ni del empleo de 5ARI.
Vickers et al.55 desarrollaron una prueba sérica comercializada con el nombre de 4Kscore test o test de las 4calicreínas. El 4Kscore combina 4biomarcadores específicos de la próstata (tPSA, fPSA, PSA intacto y hk 2), junto con la edad y el tacto rectal, y calcula un riesgo.
Este panel se estudió inicialmente en una cohorte de pacientes derivados del estudio de screening ERSCP a quienes se realizó biopsia de próstata por presentar PSA ≥3 ng/ml55. Aplicando el test a 1.000 hombres con un riesgo ≥20% en el test de las calicreínas, se reducirían 513 biopsias (51,3% de biopsias innecesarias), se perderían 54 de 177 cánceres de bajo grado y 12 de 100 cánceres de alto grado.
El panel de las 4 calicreínas también fue analizado como predictor de CaP insignificante en piezas de prostatectomía radical56.
Se analizó el comportamiento del panel de las 4 calicreínas en el suero de pacientes a quienes se les habían realizado biopsias de próstata (10 cilindros) por presentar PSA ≥ 3 ng/ml57. El panel mejoró la detección de CaP en comparación con la edad y el PSA.
A modo de resumen:
- -
Predice el riesgo de CaP y su posible agresividad.
- -
Se puede utilizar antes de la biopsia, o después de una biopsia negativa, y puede predecir la probabilidad de que el cáncer se propague a otras partes del cuerpo en los siguientes 20 años.
La capacidad para detectar y aislar células tumorales circulantes (CTC) en pacientes con CPRC ya es una realidad clínica58. La detección de células epiteliales en sangre podría indicar la presencia de células tumorales que han abandonado la próstata y la probable enfermedad metastásica, lo que presenta peor pronóstico frente a los que son indetectables59,60.
Se exploró61 la relación entre el número de CTC (> 5 cél/7,5ml de sangre) y los patrones de diseminación metastásica. Se observaron niveles más altos de CTC en pacientes con metástasis óseas, en aquellos que previamente había recibido quimioterapia citotóxica y en aquellos con supervivencia global más corta62.
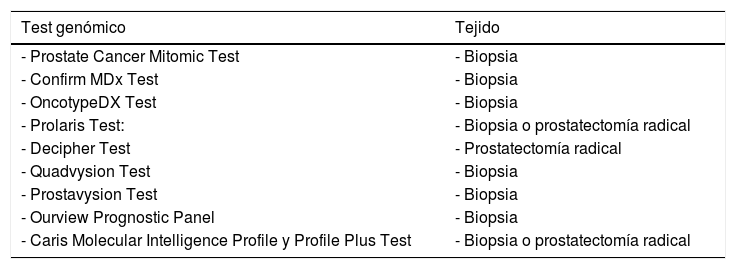
Revisamos a continuación varios biomarcadores con test genómicos realizados en tejido prostático (de la biopsia o de la pieza de prostatectomía radical). A partir del estudio de este tejido prostático (tabla 2), se puede hacer un análisis del ADN genético (alteraciones estructurales o mutaciones), del ADN epigenético (metilación ADN) o del ARN. Estos nuevos test permiten un mejor conocimiento de la enfermedad y ayudan al médico y al paciente en la toma de decisiones:
Test genómicos
| Test genómico | Tejido |
|---|---|
| - Prostate Cancer Mitomic Test | - Biopsia |
| - Confirm MDx Test | - Biopsia |
| - OncotypeDX Test | - Biopsia |
| - Prolaris Test: | - Biopsia o prostatectomía radical |
| - Decipher Test | - Prostatectomía radical |
| - Quadvysion Test | - Biopsia |
| - Prostavysion Test | - Biopsia |
| - Ourview Prognostic Panel | - Biopsia |
| - Caris Molecular Intelligence Profile y Profile Plus Test | - Biopsia o prostatectomía radical |
El Prostate Cancer Mitomic Test (PCMT)63 predice con exactitud cuándo una biopsia es verdadera positiva mediante la detección de deleciones de ADN mitocondrial que habitualmente se encuentran en el tumor o próximo a este.
Tiene como ventaja el ser muy sensible en detectar CaP. Así, tanto si la biopsia como si el PCMT son negativos, no hay necesidad de repetir la biopsia, con un 91% de precisión.
No determina agresividad tumoral ni predice el riesgo.
Confirm MDX TestEs un test que detecta hipermetilación de ADN. Determina el estatus epigenético (metilación de ADN) de genes asociados a CaP: GSTP1, APC y RASSF1. Está destinado a definir pacientes que no van a necesitar rebiopsia64.
Tampoco predice agresividad ni riesgo.
OncotypeDX Test (genomic prostate score)Marca el riesgo biológico del tumor al estudiar 17 genes que determinan la agresividad del tumor en cuanto a recidiva local o metastásica65,66. El resultado de la prueba se expresa mediante un número (genomic prostate score), cuyos rangos varían de 0 (bajo) a 100 (alto).
En pacientes diagnosticados de CaP de muy bajo o bajo riesgo, o con el grado de Gleason 7 con bajo volumen, identifica a los de bajo riesgo según el test, para ayudar en la toma de decisiones en cuanto al tratamiento o la vigilancia activa.
Predice los tumores de alto grado o enfermedad no organoconfinada en la prostatectomía radical y proporciona información del riesgo individual de recidiva bioquímica a los 5 años.
Prolaris test (score)El test de Prolaris67,68 o test de progresión del ciclo celular mide el promedio de expresión de ARN de 31 genes que reflejan la frecuencia de división celular. Cuanto más agresivo es el cáncer, mayor es la división y proliferación celulares, y mayor es el valor de Prolaris. Evita el tratamiento a pacientes con pocas posibilidades de beneficiarse de él. Predice la mortalidad cáncer-específica después de una biopsia. En pacientes posprostatectomía radical estima el riesgo de recidiva bioquímica, ayudando en la decisión de a quién realizar tratamientos más agresivos, adyuvantes a la cirugía.
Decipher test (genomic prostate cancer classifier)Este test evalúa 22 biomarcadores genómicos, lo que puede mejorar significativamente la predicción de recidiva bioquímica y metástasis después de una prostatectomía radical. Puede identificar a hombres de alto riesgo que precisaran de radioterapia o de privación androgénica69.
QuadVysion y ProstaVysion testsQuadVysion ayuda al patólogo a mejorar la precisión del diagnóstico mediante la tinción inmunohistoquímica utilizando 4 anticuerpos (AMACR, C-myc, HMW P63 y CK).
ProstaVysion70,71 proporciona una puntuación en el material de biopsia que ayuda a determinar la agresividad del cáncer y el pronóstico a largo plazo mediante la tinción inmunohistoquímica de 3biomarcadores (HOXD3, ERG y PTEN). Un triple resultado positivo indica mal pronóstico, mientras que un triple negativo indica un pronóstico favorable, lo que puede hacer que nos decantemos por terapias más conservadoras.
OurView Prognostic PanelEste panel72 incorpora factores clínicos, grado de Gleason, PTEN, ki-67 y ploidía del ADN, como segunda opinión patológica de la biopsia. Mejora la precisión de la biopsia y ayuda en la toma de decisiones.
ConclusionesLa determinación de PSA sérico es el test más utilizado en la actualidad para el diagnóstico y seguimiento del CaP. Este marcador es a menudo solicitado por el paciente desde las consultas de Atención Primaria y, de este modo, hace realidad la detección oportunista. El cribado conlleva un sobrediagnóstico, de ahí la necesidad de optimizarlo para evitar biopsias innecesarias y su morbilidad asociada. Con el resultado de la biopsia se nos plantean a su vez nuevas dudas. Si el resultado es negativo, necesito saber si el paciente tiene alta probabilidad de tener CaP para plantearle o no nuevas biopsias. Si el resultado es positivo, debería saber si ese cáncer es indolente y planearle vigilancia activa o, por el contrario, si es un tumor de alto riesgo y debería ofrecerle tratamiento activo desde ese momento.
Los nuevos biomarcadores nos brindan nuevos caminos en cuanto a detección precoz, pronóstico y respuesta al tratamiento. En parte, como hemos visto, nos van a ayudar en la toma de muchas decisiones, aun a sabiendas de que en la actualidad no disponemos del marcador ideal.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.