: Los trastornos psiquiátricos forman parte de las enfermedades más frecuentes en la población chilena y, a pesar de que la mayoría de estos cuadros son tratables farmacológicamente de manera eficaz, hay un porcentaje de pacientes que no responden adecuadamente, o presentan severos efectos adversos que obligan a la discontinuidad de su tratamiento. En la actualidad, no existe ningún método fiable para predecir qué tratamiento farmacológico será el mejor para un determinado paciente
El metabolismo de los psicofármacos está mediado, principalmente, por el complejo enzimático Citocromo P450 (CYP450), que incluye las isoformas 1A2, 2C9, 2C19, 2D6, 3A4 y 3A5. Los genes que codifican para estas enzimas, presentan variantes polimórficas que influyen en la actividad enzimática. La identificación de estos polimorfismos del CYP450, nos permite clasificar a los pacientes como metabolizadores lentos, intermedios, normales o ultrarrápidos, facilitando el diseño de una estrategia farmacológica personalizada predeciblemente eficaz para el paciente
Objetivo: Genotipificar las variantes alélicas más relevantes del CYP450 1A2, 2C9, 2C19, 2D6, 3A4 y 3A5 en pacientes chilenos que consultan por trastornos psiquiátricos
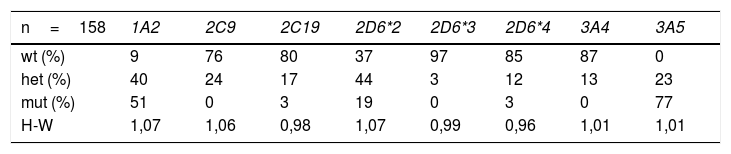
Resultados: En este estudio observacional, descriptivo y exploratorio se evaluaron las variantes genéticas de 6 enzimas del CYP450 mediante RFLP y secuenciación en 158 pacientes chilenos, consultantes por trastornos psiquiátricos, cuyo rango etario es de 6-74 años (mediana de 35 años), siendo 66 hombres y 92 mujeres. Las frecuencias alélicas obtenidas mostradas como alelo silvestre/alelo polimórfico fueron las siguientes: 1A2 (1A/1F 0,290/0,710); 2C9 (1A/2A 0,880/0,120); 2C19 (1A/2A 0,885/0,115); 2D6*2 (1/2 0,590/0,410); 2D6*3 (1/30,985/0,0015); 2D6*4 (1/4 0,910/0,090); 3A4 (1A/1B 0,935/0,065); y 3A5 (1A/3A 0,115/0,885). En nuestro estudio, las frecuencias genotípicas para todos los polimorfismos estudiados en la población estaban en equilibrio de Hardy-Weinberg
Conclusiones: Las frecuencias alélicas obtenidas para el grupo de estudio son similares a las descritas por otros autores tanto para población chilena como de otros países del mundo y por tanto permite ajustar las dosis de los medicamentos de acuerdo a las pautas internacionales en concordancia con el uso de una medicina personalizada. En psiquiatría, contar con un examen que nos permita implementar, desde el comienzo, una terapia predeciblemente eficaz para el paciente, puede hacer una gran diferencia en la práctica clínica
: Psychiatric disorders are one of the most frequent illnesses in the Chilean population and, even though most of these conditions are effectively pharmacologically treatable, there is a percentage of patients who do not respond adequately or have severe adverse effects that require patients to discontinue their treatment. At present, there is no reliable method to predict which drug treatment will be the best for a given patient.
The metabolism of psychotropic drugs is mainly mediated by the enzyme complex Cytochrome P450 (CYP450), which includes the isoforms 1A2, 2C9, 2C19, 2D6, 3A4, and 3A5. The genes that code for these enzymes have polymorphic variants that influence enzyme activity. Identification of these CYP450 polymorphisms allows us to classify patients as poor, intermediate, normal, or ultra-rapid metabolizers, facilitating the design of a personalized drug strategy that is predictably effective for the patient.
Objective: Genotyping the most relevant allelic variants of CYP450 1A2, 2C9, 2C19, 2D6, 3A4, and 3A5 in Chilean patients consulting for psychiatric disorders.
Results: In this observational, descriptive and exploratory study, the genetic variants of 6 CYP450 enzymes were evaluated by RFLP and sequencing in 158 Chilean patients, consulting for psychiatric disorders whose age range was 6-74 years (median of 35 years), being 66 men and 92 women. The allele frequencies obtained shown as wild allele/polymorphic allele were the following: 1A2 (1A/1F 0.290/0.710); 2C9 (1A/2A 0.880/0.120); 2C19 (1A/2A 0.885/0.115); 2D6 * 2 (1/2 0.590/0.410); 2D6 * 3 (1/3 0.985/0.0015); 2D6 * 4 (1/4 0.910/0.090); 3A4 (1A/1B 0.935/0.065); and 3A5 (1A/3A 0.115/0.885). In our study, the genotype frequencies for all the polymorphisms studied in the population were in Hardy-Weinberg equilibrium.
Conclusions: The allelic frequencies obtained for the study group are similar to those described by other authors for both the Chilean population and those of other countries in the world and therefore allow adjusting the drugs doses according to international guidelines on personalized medicine. In psychiatry, having a test that allows us to implement a predictably effective therapy for the patient right from the start can make a big difference in clinical practice.
Los trastornos psiquiátricos ocupan el tercer lugar entre las patologías más prevalentes en la población chilena, de acuerdo con los datos publicados en 2016 por Vicente et al. presentando una frecuencia de alrededor del 17%1. A nivel mundial, los trastornos mentales también tienen una alta prevalencia, y se pueden medir en lo que se denomina la carga de enfermedad mediante dos indicadores básicos, los años perdidos por discapacidad (APD) y los años de vida ajustados en función de la discapacidad (AVAD), siendo APD un 32% y AVAD un 13%2. La enfermedad mental en Chile es frecuente y causa mucha discapacidad considerando la enfermedad misma, los efectos a nivel educacional (escolar y universitario), laboral y familiar, pero además hay que considerar la muerte prematura asociada a la enfermedad, no solo por suicidio, sino que también por complicaciones asociadas al uso de medicamentos, terapias crónicas extensas y también por un menor acceso a la atención de salud. Chile, dentro de la región de las Américas, ocupa el segundo lugar con un 36% de APD y el tercer lugar con un 21% de AVAD3. Dentro de los trastornos mentales, la depresión ocupa el primer lugar y es dos veces más frecuente en mujeres que en hombres. A nivel regional, es la primera causa de discapacidad y en Chile la depresión representa un 8,1% de la discapacidad total medida en APD y afecta a población principalmente joven de 15 a 50 años, siendo la enfermedad que presenta el primer lugar con el mayor porcentaje de AVAD del país3–5.
Muchos de los cuadros de la enfermedad mental son tratables farmacológicamente y tienen una expectativa de recuperación ad integrum. Sin embargo, causan un importante deterioro en la calidad de vida de quienes los padecen, cuando el paciente posterga la búsqueda de ayuda, muchas veces por temor a los efectos adversos de los psicofármacos o porque el tratamiento apropiado tarda en ser implementado, ya que no se obtienen los resultados esperados, obligando al psiquiatra a intentar diversos esquemas terapéuticos6.
Hoy en día los fármacos para el tratamiento de la enfermedad mental se prescriben sobre la base de las características del cuadro clínico y la probabilidad de obtener resultados que puedan ser clínicamente reproducibles. Pese a su probada eficacia, una proporción importante de pacientes no responde adecuadamente o presenta severos efectos adversos, que obligan a su discontinuidad. De este modo, la respuesta diferencial al tratamiento farmacológico constituye una fuente principal de morbilidad y mortalidad de los pacientes. De hecho, más de dos millones de casos de reacciones adversas ocurren anualmente en Estados Unidos, incluyendo 100.000 muertes7. Cerca del 5 al 13% de los pacientes hospitalizados o ambulatorios presentan efectos adversos a la droga y efectos subterapéuticos de la terapia con fármacos8,9. Además, los efectos secundarios a los medicamentos determinan cerca de un 7% de hospitalizaciones10 y un 20% de todas las readmisiones hospitalarias11.
Los pacientes responden a los medicamentos de manera variable e impredecible. Esta respuesta variable se debe, entre otros, a factores genéticos y epigenéticos, que incluyen la edad, la polimedicación, enfermedades concomitantes, y la dieta del paciente que afectan a las enzimas que metabolizan la droga, a los receptores o a ambos. Cerca del 20 al 30% de la variabilidad interindividual de la respuesta a la droga se puede explicar en base a los polimorfismos genéticos12,13 La diferencia en el metabolismo de las drogas, inter e intraindividual, contribuye a la variabilidad en la respuesta a los tratamientos, ya que finalmente éste es el determinante principal de la concentración plasmática14. En suma, actualmente no existe un método fiable, para predecir cuál tratamiento farmacológico será el mejor para un determinado paciente, y esta situación se ve agravada, además, por la latencia en la aparición de respuesta, que en la mayoría de los casos es de 2 a 3 semanas.
La medicina personalizada, esto es, la adaptación de las terapias basada en el perfil genético y molecular del paciente, es uno de los aspectos más promisorios de la medicina moderna y ha sido una de las primeras aplicaciones de la farmacogenómica15,16. La identificación de la relación entre genotipo y respuesta a drogas, incluyendo tanto el efecto terapéutico como el perfil de efectos adversos, afectará profundamente la práctica médica porque se mejora la respuesta del paciente por disminución de efectos adversos y la eficacia de la medicación17. Así, la farmacogenética se establece cada día más como una disciplina relevante para la elección del tratamiento farmacológico porque se personaliza la medicina usando las características genéticas del paciente, las cuales predecirán la respuesta a la droga, guiando al médico en la selección del fármaco y en el cálculo de dosis óptimas 18,19. Esto elimina la prescripción basada en la prueba y error, permitiendo un tratamiento más efectivo, seguro, y costo-efectivo17,20. Las variaciones genéticas particulares del paciente pueden expresarse a distintos niveles, ya sea a nivel farmacocinético (absorción, distribución, metabolismo, eliminación) como farmacodinámico (modificación del blanco de la droga o la via biológica que altere la sensibilidad del efecto farmacológico), lo que finalmente deriva en una respuesta distinta y particular a un tratamiento específico. En la actualidad, existen fármacos concretos en cuya ficha técnica la FDA recomienda realizar una monitorización farmacogenética para mejorar su prescripción y/o dosificación. En algunos casos, esta información se ha incorporado a la ficha técnica mucho tiempo después de la autorización del fármaco por las agencias reguladoras21,22. Así la información farmacogenética está contenida en la etiqueta de cerca de un 10% de fármacos aprobados por la FDA, la mayoría de los cuales se refiere a los genes CYP2C9, CYP2C19 y CYP2D623. Los centros médicos más importantes del mundo, como el Instituto Pasteur, el Hospital Clínico de Londres y la Clínica Mayo, están incluyendo la caracterización farmacogenética de sus pacientes en todas las especialidades. Por ejemplo, la Clínica Mayo el año 2011 genotipificó al 35% de todos los pacientes que ingresaban para recibir tratamiento farmacológico. Todos los datos de genotipo están incorporados en la ficha electrónica del paciente, por lo que cuando un clínico prescribe una droga, si hay alguna que no sea compatible aparece una alerta. Las decisiones clínicas se toman en base a datos farmacogenéticos y farmacocinéticos de acuerdo a las propias guías que han desarrollado24.
El metabolismo o biotransformación de alrededor del 80% de los psicofármacos, está mediado, principalmente, por el complejo enzimático Citocromo P450 (CYP450), que corresponde a una superfamilia de monooxigenasas, responsables del metabolismo oxidativo de un gran número de compuestos endógenos y xenobióticos y constituye, por lo tanto, uno de los principales moduladores del metabolismo de las drogas25,26. Los genes CYP450 son altamente polimórficos, es decir, presentan una gran variabilidad entre distintos individuos y a través de las poblaciones en el mundo, lo que determina importantes implicancias en la bioactivación o desintoxicación de los fármacos27. El complejo CYP450 incluye las isoformas 1A2, 2C9, 2C19, 2D6, 3A4 y 3A5, entre otras. Polimorfismos genéticos de estas enzimas del CYP, pueden producir cambios profundos en la actividad enzimática, determinando así la respuesta individual a un fármaco en particular, lo que permite clasificar fenotípicamente a los pacientes como metabolizadores pobres, intermedios, extensos o ultrarrápidos28,30.
El objetivo principal de este estudio fue determinar las frecuencias alélicas de diversas isoformas del CYP450 en un grupo de la población de Chile, consultante por cuadros ansiosos y/o depresivos, con el fin de describir los polimorfismos genéticos más frecuentes. Esto puede ser útil para el diseño de una estrategia terapéutica personalizada, que redunde en una mejor respuesta a la farmacoterapia. En el ámbito de la psiquiatría, donde los psicofármacos tienen una efectividad promedio de alrededor del 60%, contar con un examen que permita implementar una terapia predeciblemente eficaz para el paciente, puede hacer una gran diferencia en la práctica clínica6,31.
2Materiales y método2.1Tipo de estudio, población y muestrasEl presente estudio es un estudio observacional, descriptivo y transversal realizado en Clínica Las Condes de Santiago, Chile. Los pacientes institucionales y provenientes de consultas privadas, fueron enrolados consecutiva y prospectivamente desde abril del 2012 hasta agosto del 2021. Se incluyeron a todos los pacientes adultos y pediátricos de ambos sexos que consultaron por trastornos psiquiátricos y a los cuales se les realizó la genotipificación del CYP450. Se obtuvo muestras de sangre de 158 pacientes cuyo rango etario es de 6-74 años (mediana de 35 años), siendo 66 hombres y 92 mujeres. Esta investigación fue autorizada por el Comité de Ética de Clínica Las Condes.
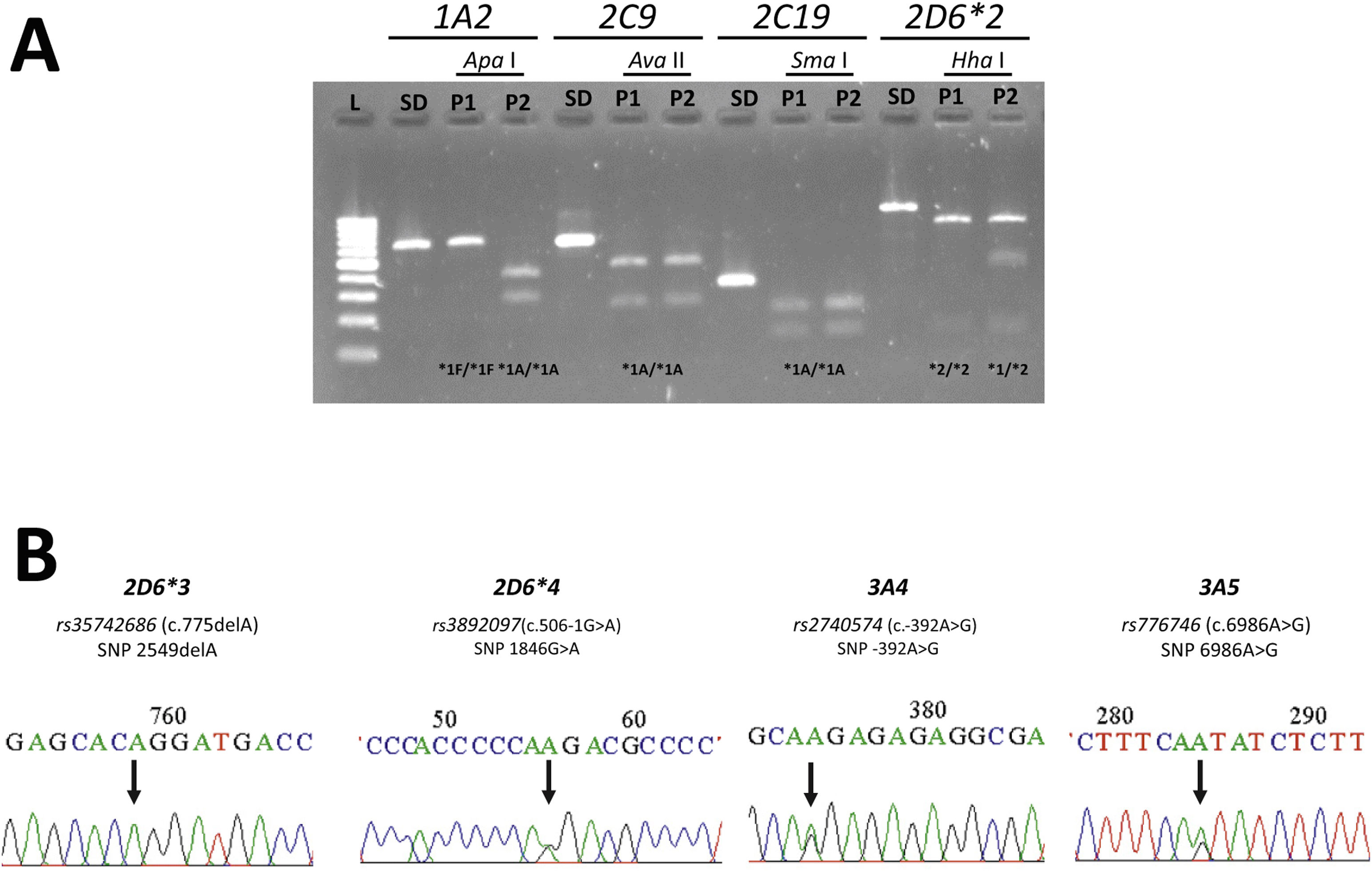
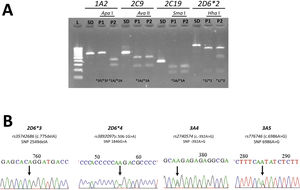
2.2Extracción de DNA genómico y genotipificaciónEl DNA genómico se extrajo desde 5mL de sangre periférica mediante el método de Lahiri32. Posteriormente, a partir de 100ng de DNA total se llevó a cabo la amplificación por PCR usando partidores específicos para las isoformas CYP1A2, CYP2C9, CYP2C19, CYP2D6, CYP3A4 y CYP3A5, diseñados para este estudio o tomados desde publicaciones previas33,34. La detección de polimorfismos CYP1A2*1F, CYP2C9*2, CYP2C19*2 y CYP2D6*2 se realizó por la técnica de RFLP (Restriction Fragment Length Polymorphism), con enzimas de restricción específicas (Apa I, Ava II, Sma I y Hha I, respectivamente). El análisis se realizó a través de electroforesis en gel de agarosa al 2% de acuerdo con métodos reportados previamente (Fig. 1A)35–37. Para la detección de los polimorfismos CYP3A4*1B, CYP3A5*3, CYP2D6*2, CYP2D6*3 y CYP2D6*4, los productos de amplificación fueron secuenciados por el método de Sanger en ambas hebras del DNA y se comparó la secuencia obtenida con las secuencias publicadas definidas como tipo silvestre (Fig. 1B). Para el caso específico de CYP2D6 además, se realizó la detección de la duplicación de la variante CYP2D6*2 en 59 pacientes, dado que esta alteración produce una enzima considerada de actividad aumentada. Para ello, se utilizó el método descrito por Steijns y Van der Weide que puede discriminar mediante la utilización de dos parejas de partidores y Taq polimerasa de alta resolución, la presencia de duplicación génica38.
Métodos utilizados para la evaluación de los distintos polimorfismos. A) Ejemplo de RFLP (Restriction Fragment Length Polymorphism) para las 4 variantes estudiadas CYP1A2, CYP2C9, CYP2C19 y CYP2D6*2. Se muestran los resultados para dos pacientes distintos (P1 y P2). El primer carril L corresponde a un patrón de tamaños de DNA de 100 pb (New England Biolabs). SD: producto de PCR sin digerir. Las enzimas de restricción usadas corresponden a Apa I para 1A2, Ava II para 2C9, Sma I para 2C19 y Hha I para 2D6*2. Se observa para el caso de 1A2 y 2D6*2 la diferencia en el patrón de digestión entre ambos pacientes. En el caso de 1A2 el P1 es tipo mutado (*1F/*1F), y el P2 es tipo silvestre (*1A/*1A); mientras que en 2D6*2 el P1 es tipo mutado (*2/*2) y el P2 es tipo heterocigoto (*1/*2). Para los genes 2C9 y 2C19 tanto el P1 como el P2 son tipo silvestre (*1A/*1A). B) Se muestran los cromatogramas de la secuenciación Sanger para variantes de los genes CYP2D6, CYP3A4 y CYP3A5. En el caso de CYP2D6 se muestra el perfil tipo silvestre para la variante *3 (rs 35742686), y el tipo heterocigoto para la variante *4 (rs 3892097) indicado por la flecha observándose la doble señal en dicha posición (G/A). Para las variantes CYP3A4 *1B (rs 2740574) y CYP3A5 *3A (rs 776746) el patrón heterocigoto indicado por la flecha.
Dada la naturaleza descriptiva de este estudio, los datos categóricos obtenidos que indican la presencia de los distintos polimor-fismos para cada isoforma de CYP450 estudiada, se resumieron en frecuencias y porcentajes. El equilibrio de Hardy-Weinberg para cada alelo estudiado fue calculado usando el método de Weir39.
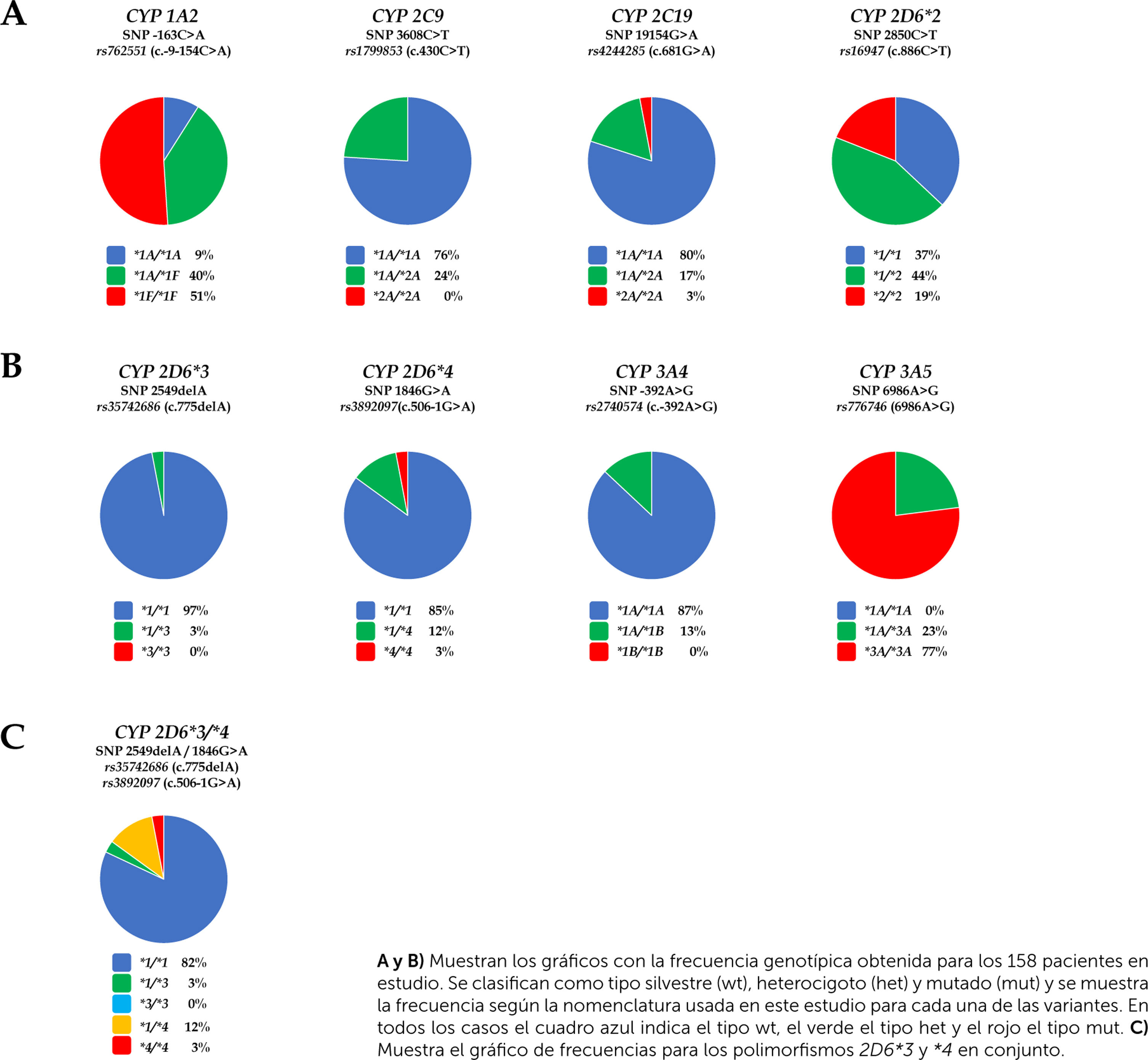
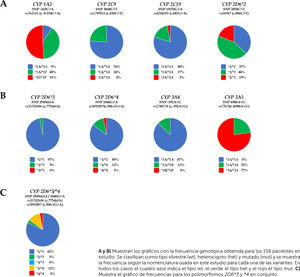
3ResultadosPara el caso de la isoforma CYP1A2 se obtuvo que el 9% (14/158) de los pacientes estudiados presentó la forma tipo silvestre (wt, wild type) *1A/*1A, mientras que el 40% (63/158) el genotipo heterocigoto (het) *1A/*1F y el 51% (81/158) de nuestra población el genotipo homocigoto (mut) *1F/*1F cuya actividad enzimática es inducible por varios factores (tabaco, insulina, modafinilo, nafcilina [análogo de la penicilina], omeprazol, vegetales crucíferas [brócoli, repollo, repollos de Bruselas, berros], café y carnes grilladas) (ver Fig. 2, Tablas 1 y 2). Para diferenciar el genotipo homocigoto silvestre del que presenta el polimorfismo, utilizaremos en este trabajo la abreviatura mut (mutado) aun cuando este término se utiliza para cambios que se encuentran en menos del 1% de la población y no representa una mutación propiamente tal40. Cuando analizamos la isoforma CYP2C9, el 76% (120/158) de los pacientes presentó el genotipo silvestre (wt) *1A/*1A y el 24% restante (38/158) el genotipo heterocigoto (het) *1A/*2A. Ningún paciente presentó el genotipo homocigoto (mut) *2A/*2A (ver Fig. 2, Tablas 1 y 2). CYP2C19 presentó una frecuencia del 80% (127/158) para el tipo silvestre *1A/*1A, 17% (27/158) para el genotipo heterocigoto *1A/*2A, y 3% (4/158) para el genotipo homocigoto *2A/*2A que determina la ausencia completa de actividad enzimática (ver Fig. 2, Tablas 1 y 2). Para CYP2D6, el 82% de los pacientes (129/158) presentan la forma silvestre *1/*1, 3% (4/158) el genotipo heterocigoto *1/*3, 12% (19/158) el genotipo heterocigoto *1/*4, 3% (5/158) el genotipo homocigoto *4/*4, que tiene total ausencia de actividad enzimática, y 0% (0/158) el genotipo homocigoto *3/*3 (ver Fig. 2c). Para el CYP2D6*2 observamos un 37% (58/158) de pacientes que presentan la forma silvestre*1/*1, mientras que un 44% (70/158) el genotipo heterocigoto *1/*2 y un 19% (30/158) el genotipo homocigoto *2/*2. Ninguno de los 59 pacientes analizados en nuestra población mostró la duplicación génica para la variante CYP2D6*2, cuya presencia indica un paciente metabolizador ultrarrápido.
Distribución de las variantes genotípicas para CYP 1A2, 2C9, 2C19, 2D6, 3A4 y 3A5. A y B) Muestran los gráficos con la frecuencia genotípica obtenida para los 158 pacientes en estudio. Se clasifican como tipo silvestre (wt), heterocigoto (het) y mutado (mut) y se muestra la frecuencia según la nomenclatura usada en este estudio para cada una de las variantes. En todos los casos el cuadro azul indica el tipo wt, el verde el tipo het y el rojo el tipo mut. C) Muestra el gráfico de frecuencias para los polimorfismos 2D6*3 y *4 en conjunto.
Variantes de CYP450 analizadas
| Polimorfismo estudiado | rs | Genotipo Silvestre | Genotipo Heterocigoto | Genotipo Homocigoto | Actividad enzimática | Referencias |
|---|---|---|---|---|---|---|
| CYP1A2*1Fc.-163C>A | 762551 | *1A/*1A C/C | *1A/*1F C/A | *1F/*1F A/A | Inducible | 38,45,46,47 |
| CYP2C9*2Ac.3608C>T | 1799853 | *1A/*1A C/C | *1A/*2A C/T | *2A/*2A T/T | Disminuida | 66,67 |
| CYP2C19*2Ac.19154G>A | 4244285 | *1A/*1A G/G | *1A/*2A G/A | *2A/*2A A/A | Ausencia | 55 |
| CYP2D6*2c.2850C>T | 16947 | *1/*1C/C | *1/*2C/T | *2/*2T/T | Aumentada o Normal | 42,49,56-59 |
| CYP2D6*3c.2549delA | 35742686 | *1/*1 | *1/*3 | *3/*3 deleción | Ausencia | 50-52 |
| CYP2D6*4c.1846G>A | 3892097 | *1/*1G/G | *1/*4G/A | *4/*4A/A | Ausencia | 50-53 |
| CYP3A4*1Bc.-392A>G | 2740574 | *1A/*1A A/A | *1A/*1B A/G | *1B/*1B G/G | Disminuida | 65 |
| CYP3A5*3Ac.6986A>G | 776746 | *1A/*1A A/A | *1A/*3A A/G | *3A/*3A G/G | Severamente disminuida | 34,55 |
rs: reference Single Nucleotide Polymorphism (SNP) number.
Porcentaje de frecuencias genotípicas observadas y equilibrio Hardy-Weinberg
| n=158 | 1A2 | 2C9 | 2C19 | 2D6*2 | 2D6*3 | 2D6*4 | 3A4 | 3A5 |
|---|---|---|---|---|---|---|---|---|
| wt (%) | 9 | 76 | 80 | 37 | 97 | 85 | 87 | 0 |
| het (%) | 40 | 24 | 17 | 44 | 3 | 12 | 13 | 23 |
| mut (%) | 51 | 0 | 3 | 19 | 0 | 3 | 0 | 77 |
| H-W | 1,07 | 1,06 | 0,98 | 1,07 | 0,99 | 0,96 | 1,01 | 1,01 |
wt: genotipo silvestre; het: genotipo heterocigoto; mut: genotipo homocigoto mutado; H-W: equilibrio Hardy Weinberg.
Para la isoforma CYP3A4 el 87% (137/158) presentó el tipo silvestre *1A/*1A, un 13% (20/158) el genotipo heterocigoto *1A/*1B, y ningún paciente el genotipo homocigoto *1B/*1B, la presencia de la variante alélica 1*B, le confiere una actividad reducida a esta enzima (ver Fig. 2 y Tablas 1 y 2). Por último, en el caso de CYP3A5, sorpresivamente, ningún paciente mostró la versión silvestre *1A/*1A descrita ampliamente en la literatura (0/158), mientras que un 22% (35/158) de los pacientes presentó el genotipo heterocigoto *1A/*3A y un 78% (122/158) presentó el genotipo homocigoto *3A/*3A, cuya actividad enzimática está severamente disminuida (ver Fig. 2, Tablas 1 y 2). En nuestro estudio, las frecuencias genotípicas para todos los polimorfismos estudiados en la población estaban en equilibrio de Hardy-Weinberg (Tabla 2).
4DiscusiónExisten pocos trabajos en población chilena en donde se estudia la distribución de las distintas variantes polimórficas de los distintos genes de la familia del CYP450. Un artículo publicado por Roco et al. en el año 2012 analiza algunas de las variantes alélicas estudiadas en nuestro trabajo, entre otras 23 en el contexto de respuesta a terapia oncológica en una población de 253 sujetos41 y un segundo, que analiza la presencia de polimorfismos en CYP2D6 en 321 individuos de población general42. Al comparar los resultados, podemos observar que las frecuencias alélicas para CYP1A2 *1F, CYP2C19 *2A, CYP2D6 *2/*3 y CYP3A4 *1B (ver Tabla 3) son iguales a las descritas por Roco y Varela341,42, mientras que para las variantes CYP2C9*2A encontramos una frecuencia alélica mayor siendo 0,12 versus 0,06, y para CYP3A5*3A también es mayor y corresponde a 0,88 versus 0,7641. A pesar del tamaño de muestra de nuestro estudio, es posible hacer un análisis de los resultados e inferir interesantes conclusiones respecto de la presencia de polimorfismos en pacientes chilenos que consultan por trastornos ansiosos y depresivos. Por ejemplo, al analizar la distribución del polimofismo *1F que se observa para la isoforma CYP450 1A2, vemos que un 51% de nuestros pacientes presenta el genotipo*1F/*1F. Se ha descrito en población europea que el 33%43,44 presenta la forma inducible *1F, sin embargo, nosotros hemos encontrado un porcentaje de 51% para el alelo *1F y sumando la variante heterocigota más la homocigota representan un 91% de nuestra población, teniendo una frecuencia total el alelo *1F de 0,71 muy parecido a los valores descritos para una muestra de población chilena de 253 sujetos, en que el alelo *1F tiene una frecuencia de 0,7741. Este hecho es relevante porque esta forma enzimática es inducible en presencia de sustancias como tabaco y café, entre otros, y tiene una alta actividad45–47, lo que determina, por ejemplo, que pacientes psiquiátricos fumadores con este genotipo, sometidos a terapia con olanzapina, aumenten la actividad enzimática, reduciendo los niveles plasmáticos de la droga a concentraciones subterapéuticas, alejando al paciente del beneficio terapéutico de la droga. Alternativamente, es posible apoyar al paciente para dejar de fumar mientras dure el tratamiento, o ajustar la dosis del fármaco, compensando la mayor actividad enzimática con dosis mayores para alcanzar el efecto terapéutico deseado.
Frecuencias alélicas para las variantes de CYP450 estudiadas
| 1A2 | 2C9 | 2C19 | 2D6*2 | 2D6*3 | 2D6*4 | 3A4 | 3A5 |
|---|---|---|---|---|---|---|---|
| 1A 0,290 | 1A 0,880 | 1A 0,885 | 1 0,590 | 1 0,985 | 1 0,910 | 1A 0,935 | 1A 0,115 |
| 1F 0,710 | 2A 0,120 | 2A 0,115 | 2 0,410 | 3 0,015 | 4 0,090 | 1B 0,065 | 3A 0,885 |
La fila superior contiene el alelo tipo silvestre (1A) y la inferior el alelo polimórfico según sea el gen en estudio (1B, 1F, 2A, 3A).
En el caso de CYP450 2D6*2, existe controversia con respecto a si es una isoforma con actividad normal o aumentada. Esta discrepancia estaría explicada por la presencia de la variante -1584C> G (rs1080985 o CYP2D6*2A) en la región promotora del gen la cual no fue evaluada en nuestro estudio42,48,49. Por otra parte, para CYP450 2D6*4, identificamos 5 pacientes portadores de la variante homocigota *4/*4, determinando que esta enzima tenga nula actividad en dichos pacientes50–52. La frecuencia de este genotipo en población caucásica es del 19%, y en nuestra población de estudio, considerando las variantes homocigota y heterocigota, alcanzamos un porcentaje del 15%, y que se traduce en una frecuencia alélica correspondiente al 9% que comparados con otros resultados publicados para población chilena son muy parecidos y correspondiente al 11,8%42. Además, la presencia de la variante *4 de forma heterocigota va a significar una reduccion de la actividad de la enzima, con lo que la concentración plasmática de los fármacos utilizados será también mayor comparado con los metabolizadores normales52,53. Cabe destacar que CYP2D6 es una enzima hepática capaz de metabolizar cerca del 25% de todos los fármacos actualmente utilizados en clínica. Otro resultado interesante es con la isoforma CYP4503A5, donde ningún paciente presentó la forma silvestre *1A/*1A44.54, y el 78% de la población analizada presentó el genotipo*3A/*3A, cuya actividad enzimática se encuentra severamente disminuida3455. Esta mayor frecuencia genotípica también es descrita por Roco et al. alcanzando un valor de 59,3% en su muestra41. Cabe preguntarse si en la población chilena en general, la forma prevalente corresponde a la versión *1A/*1A o a la *3A/*3A. En general, para todas las isoformas estudiadas la mayor parte de la población presenta las variantes silvestres, excepto, 3A5. Sin embargo, la combinación de las distintas isoformas en los pacientes, permite que cada uno de ellos tenga una distribución única de ellas y, por lo tanto, su respuesta a la terapia va a ser muy distinta al resto de los pacientes.
En el caso de la isoforma CYP2D6*2 donde existen publicaciones con resultados discrepantes sobre la actividad enzimática asociada a dicha variante; la correlación del genotipo al fenotipo se lograría midiendo por ejemplo la metabolización de debrisoquina en pacientes que presentan esta isoforma y determinar si su actividad es normal o aumentada42,49,56–59.
Con respecto a la detección de la duplicación de la variante CYP2D6*2, al no ser identificada en los 59 pacientes analizados, no se continuó con su estudio. Uno de los pocos estudios publicados que describe la duplicación *2 demostró que en población caucásica (589 pacientes alemanes) la frecuencia para esta variante es del 1,3%60. Recientemente se han publicado dos reportes, uno en población china de Hong Kong donde el 1,9% corresponde a la duplicación de los alelos CYP2D6*1 y *2, y que no muestran el valor por separado61, mientras que el segundo es una revisión sistemática de la literatura realizada en población europea. En dicho reporte también se presenta la duplicación de las variantes *1 y *2 y se encontró que, en un total de 82.791 individuos analizados, la duplicación está disminuida en pacientes del sudeste al noroeste de Europa. De este modo, la duplicación en Turquía y Grecia es cercana al 6% mientras que en España, Portugal e Italia la frecuencia corresponde al 3% y en países como Suecia, Dinamarca, Austria y Alemania va desde el 0,5% al 1,6%62. Considerando estos datos y el número de pacientes analizados en nuestro estudio (N=59 para la duplicación), esperaríamos obtener de 0 a 2 pacientes que presenten la duplicación considerando la frecuencia mínima europea y como máximo la observada en la península ibérica (0,5-3%), teniendo en cuenta la dotación genética de nuestra población que contempla genes amerindios y europeo-españoles, siendo una mezcla del 37% amerindio-caucásica según la distribución de los grupos sanguíneos ABO63,64.
Una de las debilidades de nuestro estudio, fue la falta de medición de mejoría de los pacientes con encuestas validadas por parte del médico tratante que permitan tener un registro de la efectividad del estudio farmacogenético en el tratamiento de patologías psiquiátricas. Sin embargo, de acuerdo a comunicaciones personales, sí ha habido mejoría en la respuesta clínica de los pacientes. A pesar de esta falencia, este estudio es el primer reporte publicado en nuestro país analizando específicamente pacientes con trastornos psiquiátricos. Nuestro estudio incluyó un grupo heterogéneo de pacientes con cuadros ansiosos o depresivos, principalmente, derivados por sus médicos tratantes para la realización del estudio genético, buscando poder explicar la falta de respuesta y/o la mala tolerancia a la farmacoterapia. Es importante destacar que tras el análisis farmacogenético realizado en nuestro estudio a los pacientes se les modificó empíricamente la terapia en uso para obtener una respuesta adecuada o mejorada a la misma, con lo cual se demuestra que efectivamente este tipo de análisis es trascendente para llevar a cabo una medicina personalizada. Es más, desde la aprobación de los primeros test farmacogenéticos comerciales por la FDA los datos de referencia no han tenido siempre resultados óptimos en pacientes de América Latina debido tal vez a diferencias étnicas. En nuestra población por ejemplo la frecuencia alélica del CYP2D6*4 (rs 3892097) que genera un fenotipo de pacientes metabolizadores lentos es de un 12% y su frecuencia genotípica es del 3% a diferenciadeloobservadoenpoblacióneuropea, dondeseobserva una frecuencia alélica del 20% o en el Este asiático del 57%. Por lo cual estudiar este polimorfismo en nuestra población tendrá un impacto menor si se estima el número de pacientes probables que estarán afectados, pero de gran relevancia al momento de tratar a estos pacientes. Existen reportes de la literatura donde, por ejemplo, en el caso del genotipo de metabolizadores pobres de CYP2D6, bajo terapia con antidepresivos tricíclicos (amitriptilina, clomipramina, imipramina entre otros), está asociado con un riesgo incrementado de cambiar a otro antidepresivo durante las 6 primeras semanas del tratamiento presentado un OR de 5,77 (IC95% 1,59-21,03 p=0,01). Se demostró además que las dosis de mantención para estos metabolizadores pobres eran mucho menores cuando se comparan con los pacientes metabolizadores normales53. Considerando los últimos reportes sobre carga global de enfermedades se ha establecido que en el caso específico de desórdenes depresivos ha habido un aumento desde 1990 a 2019 de un 61,1% de AVAD68 y además, considerando la contingencia actual de la pandemia por COVID-19, otro estudio sobre la prevalencia global de desórdenes de ansiedad o depresivos en 204 países, que no ha considerado datos de estudios en pacientes chilenos, ha demostrado un aumento en desorden depresivo mayor y desórdenes de ansiedad del 27,6% y 25,6%, respectivamente69. Estos datos pueden ser extrapolables a población general chilena y consolida, por lo tanto, el contar en Psiquiatría con un examen que nos permita implementar desde el comienzo una terapia predeciblemente eficaz para el paciente, y que puede hacer una gran diferencia en la práctica clínica. Este tipo de análisis en pacientes con trastornos psiquiátricos podría ser extendido a otras áreas de la clínica en nuestro país, para las que ya se han descrito distintos genes y sus polimorfismos como agentes determinantes en el curso de diferentes enfermedades o respuesta a fármacos.
4.1AgradecimientosLos autores agradecen a Dirección Académica de Clínica Las Condes por financiar la implementación de los métodos de genotipificación a través del proyecto de investigación PI2012-DA012 otorgado a la Dra. Lina Ortiz y a los pacientes que aceptaron y consintieron ser parte del estudio.
Declaración de conflicto de interésLos autores declaran no tener conflictos de interés.