La caracterización molecular de cepas silvestres de Pleurotus es importante para la conservación del germoplasma y su posterior uso en la mejora genética. No se han realizado estudios moleculares con los monocariontes utilizados para la producción de cepas híbridas, ni de las cepas reconstituidas obtenidas al aparear tales monocariontes. Por consiguiente, la caracterización molecular de cepas dicarióticas parentales, híbridas y reconstituidas, así como de cepas monocarióticas, es de suma importancia.
ObjetivosCaracterizar molecularmente cepas dicarióticas silvestres e híbridas, así como cepas monocarióticas de Pleurotus djamor.
MétodosSe recolectaron cinco cepas silvestres de P. djamor en diferentes estados de México y se identificaron molecularmente mediante la secuenciación de la región ITS1-5.8-ITS2 con los oligonucleótidos universales ITS1 e ITS4. Se seleccionaron dos cepas silvestres y se generaron cuatro cepas híbridas por apareamiento de neohaplontes compatibles. Para la caracterización molecular de las cepas monocarióticas y dicarióticas seleccionadas y producidas se utilizaron seis marcadores ISSR.
ResultadosCon los marcadores ITS se obtuvo un producto de amplificación de 700 pb en las cinco cepas silvestres con una similitud del 99-100% con la especie P. djamor. Con los marcadores ISSR se obtuvieron un total de 95 fragmentos con un 99% de polimorfismo.
ConclusionesLas cepas silvestres se identificaron como P. djamor. Los marcadores ISSR generaron bandas polimórficas en las cepas monocarióticas y dicarióticas, y separaron ambos tipos de cepas. El alto grado de polimorfismo indica la diversidad genética de P. djamor, una ventaja para la producción de hongos y para la mejora de la especie.
Molecular characterisation of wild type Pleurotus species is important for germplasm conservation and its further use for genetic improvement. No molecular studies have been performed with monokaryons used for producing hybrid strains, either with the reconstituted strains obtained by pairing those monokaryons. The molecular characterisation of parental dikaryons, hybrid, and reconstituted strains as well as monokaryotic strains, is therefore of utmost importance.
AimsTo carry out the molecular identification of Pleurotus djamor strains, i.e. dikaryotic wild type strains, hybrid strains, and the monokaryotic strains used for the hybrid formation.
MethodsFive wild type strains of P. djamor from different states in Mexico were collected and molecularly identified by sequencing the ITS1-5.8-ITS2 region using ITS1 and ITS4 universal oligonucleotides. Four hybrid strains were obtained by pairing neohaplonts of two wild type strains selected. Six ISSR markers were used for the molecular characterisation of monokaryotic and dikaryotic strains.
ResultsUsing the ITS markers, an amplified product of 700bp was obtained in five wild type strains, with a 99-100% similarity with P. djamor. A total of 95 fragments were obtained using the ISSR markers, with 99% of polymorphism.
ConclusionsWild type strains were identified as P. djamor, and were clearly grouped with Mexican strains from other states of Mexico. ISSR markers allowed the generation of polymorphic bands in monokaryotic and dikaryotic strains, splitting both types of strains. The high degree of polymorphism indicates the genetic diversity of P. djamor, an advantage in mushroom production and in the improving of the species.
Las cepas silvestres de hongos comestibles del género Pleurotus constituyen un material genético importante tanto para la subsistencia de los ecosistemas como para su posterior uso en la mejora genética de cultivos comerciales; por ello es necesaria la conservación y caracterización del germoplasma de estas especies. Una estrategia de mejora genética de cepas de hongos del género Pleurotus es la formación de cepas híbridas a partir de sus monocariontes, que se realiza a través de diferentes procesos: 1) germinación de las esporas de los cuerpos fructíferos13,14,26,27; 2) fusión de los protoplastos obtenidos al incubar el micelio dicariótico en soluciones enzimáticas6,15,19 y 3) desdicariotización química, que implica la homogenización de los micelios dicarióticos y el posterior crecimiento en una solución peptona-glucosa para la recuperación de los monocariontes o neohaplontes16,23,24,30,31. Con el proceso de desdicariotización química ha sido posible la producción de cepas híbridas intraespecie, interespecie e incluso intergénero6,9,23,24,30,31 y con ello se ha incrementado la probabilidad de la mejora genética de las especies implicadas.
Para la caracterización taxonómica molecular, el estudio de secuencias del ADN ha proporcionado información valiosa para varios grupos de basidiomicetos; en particular, la región ITS ha permitido el análisis de especies estrechamente relacionadas en muchos géneros, incluyendo especies de hongos cultivados5,18,25. Otra herramienta importante la constituye el uso de marcadores moleculares inter simple secuence repeat (ISSR), que tienen la ventaja de amplificar gran número de bandas polimórficas7 y han sido eficientes en la detección de polimorfismos en cepas de Pleurotus8,19,33. Nuestro estudio tuvo como objetivo la caracterización molecular de cinco cepas silvestres de Pleurotus así como la de las cepas híbridas y cepas monocarióticas (obtenidas por desdicariotización) que fueron utilizadas para la producción de los híbridos.
Materiales y métodosMaterial biológicoLas cepas silvestres de Pleurotus se recogieron en los años 2014 y 2015 en los estados de Hidalgo (CC053), Oaxaca (CC050 y CC051), Puebla (CC052) y San Luis Potosí (CC054). Las cepas de colección procedían del Instituto de Ecología A.C. (IE201, clave CC056), Colegio Posgraduados, Puebla (UAP9, clave CC059) y Facultad de Ciencias-UNAM (PCM, clave CC061). La cepa comercial se compró a un productor de México D. F. (CC055). Los cultivos axénicos de todas las cepas se encuentran depositados en el Laboratorio de Cultivos Celulares de la Unidad Profesional Interdisciplinaria de Biotecnología del Instituto Politécnico Nacional en México.
Obtención de cepas híbridas y reconstituidasLas cepas híbridas se formaron a partir de los neohaplontes obtenidos de la cepa blanca CC050 y la cepa rosa CC051, ambas recogidas en Oaxaca. Las cepas híbridas y reconstituidas se obtuvieron a partir de neohaplontes generados por desdicariotización química16 denominados CC0517 (R7), CC0515 (R5), CC0513 (R3), CC0511 (R1), CC0506 (B6), CC0504 (B4). Siguiendo el método descrito por Valencia del Toro et al.31, se obtuvieron las cepas híbridas que se denominaron CC0517xCC0504 (R7B4), CC0515xCC0504 (R5B4), CC0513xCC0504 (R3B4), CC0511xCC0504 (R1B4) y las reconstituidas CC0517xCC0511 (R7R1) y CC0506xCC0504 (B6B4).
Extracción de ADNPara la extracción del ADN genómico se siguió el protocolo Charge Switch gDNA Plant Kit (Invitrogen, EE. UU.). Se utilizaron 0,5g de micelio recuperado por filtración y congelado, procedente de un cultivo líquido (EM 28°C) de tres semanas de cada una de las cepas fúngicas.
Amplificación y secuenciación de ADNPara amplificar la región ITS-5.8-ITS2 del ADN de las cepas mediante la reacción en cadena de la polimerasa (PCR) se utilizaron los oligonucleótidos universales ITS1 (TCCGTAGGTGAACCTGCGG) e ITS4 (TCCTCCGCTTATTGATATGC)1,2,11,26,34. La PCR se llevó a cabo en un volumen de 50μl, que contenía 43μl de la mezcla de PCR SuperMix (Invitrogen, EE. UU.) y 7μl con la siguiente composición: 10ng de ADN total de cada cepa y, 0,1μM de cada cebador ITS1 e ITS4. Para la amplificación de las cuatro cepas híbridas y reconstituidas se utilizó el equipo Sure Cycler 88000 Thermal Cycler (Agilent Technologies, EE. UU.) y la PCR se realizó con las siguientes condiciones: un ciclo de desnaturalización inicial a 95°C durante 5min, seguido por 55 ciclos de desnaturalización a 95°C durante 30 s, alineamiento a 40-60°C durante 30 s, en función de cada marcador, elongación a 72°C durante unminuto y una extensión final a 72°C durante 10min. Se tomaron 5μl de los productos de amplificación y se analizaron por electroforesis en geles de agarosa al 1% (1,5h, 60V, cámara de electroforesis Thermo EC Midicell® Primo™). Las muestras se examinaron en el fotodocumentador BioRad ChemiDocTMMP Imaging System y el número de pares de bases de cada banda se determinó por comparación con el marcador de 100 pb (Sigma) y 1 Kb plus (Invitrogen, EE. UU.). Los productos amplificados fueron purificados con PureLink PCR Purification (Invitrogen, EE. UU.). La secuenciación de los productos amplificados fue realizada en el Instituto de Biotecnología de la UNAM y cada muestra se secuenció en ambas direcciones.
Análisis ISSRDe las cepas silvestres recogidas se seleccionaron los neohaplontes de la cepa blanca (CC050) y de la rosa (CC5051), y se efectuó la hibridación formando cuatro cepas híbridas y dos reconstituidas. Se utilizaron 6 iniciadores (ISSR) con las siguientes secuencias nucleotídicas: UBC807 (AGAGAGAGAGAGAGAGT), UBC811 (GAGAGAGAGAGAGAGAC), ISR02 (CAGCAGCAGCAGCAG), ISR11 (CACCACCACGC), ISR12 (GAGGAGGAGGC), ISR15 (GCAGCAGCACT). La mezcla para la PCR incluyó 30 ng de ADN de cada cepa, 2μM del oligonucleótido, 2mM de MgCl2 y 23μl de PCR SuperMix (Invitrogen, EE. UU.), con un volumen final de 25μl. Las condiciones de reacción fueron las siguientes: desnaturalización inicial a 94°C durante 5min y 40 ciclos de desnaturalización a 94°C durante unminuto, alineamiento durante un minuto a la temperatura de cada marcador, extensión a 72°C durante 3min y, finalmente, una extensión a 72°C durante 10min. La presencia de los productos de PCR fue verificada por electroforesis en gel de agarosa al 1,5% en las mismas condiciones que las utilizadas para los marcadores ITS. Las muestras se examinaron en el fotodocumentador BioRad ChemiDocTMMP Imaging System y el número de pares de bases de cada banda se determinó por comparación con el marcador 100 pb (Sigma) y 1 Kb plus.
Análisis de datosLas secuencias obtenidas con los cebadores ITS fueron comparadas con las secuencias de Pleurotus publicadas en la base de datos de ADN (GenBank) mediante el programa Basic Local Alignment Search Tool (BLAST). Posteriormente las secuencias fueron alineadas con BioEdit Sequence Alignment Editor y el programa Mega 6.6 y se llevó a cabo un análisis taxonómico con el algoritmo Neighbour-Joining. Finalmente las secuencias quedaron depositadas en el GenBank.
Para el análisis de los productos obtenidos por ISSR las bandas se consideraron caracteres binarios y se evaluaron como presencia (1) o ausencia (0). La matriz de similitud se analizó a través de las distancias genéticas usando el coeficiente de Jaccard. El dendrograma se generó igualmente con el algoritmo Neighbour-Joining. Los análisis se realizaron con el programa NTSYSpc 2.1 y SPSS versión 22. El análisis de la eficacia de los cebadores, los perfiles genéticos de las cepas y el rendimiento de los cebadores se efectuó mediante el cálculo del contenido de información polimórfica, índice de marcador y el poder de resolución. También se calcularon los parámetros relación múltiple efectiva, loci polimórficos, loci no polimórficos y el poder de resolución de cada oligonucleótido22,32.
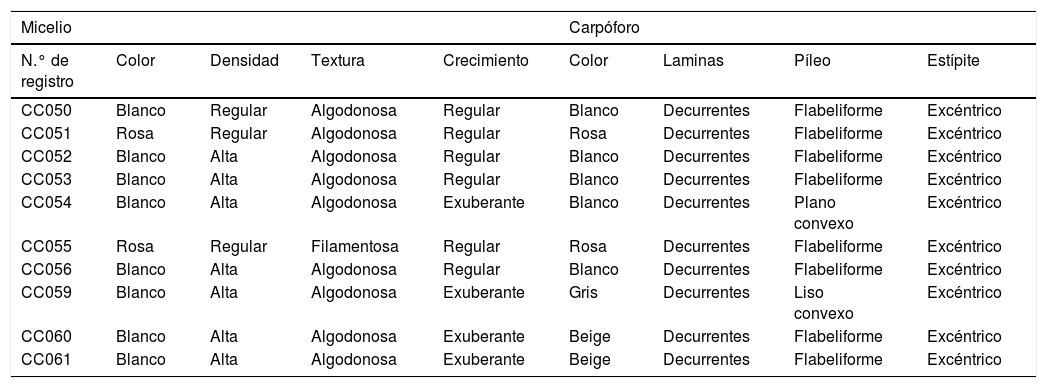
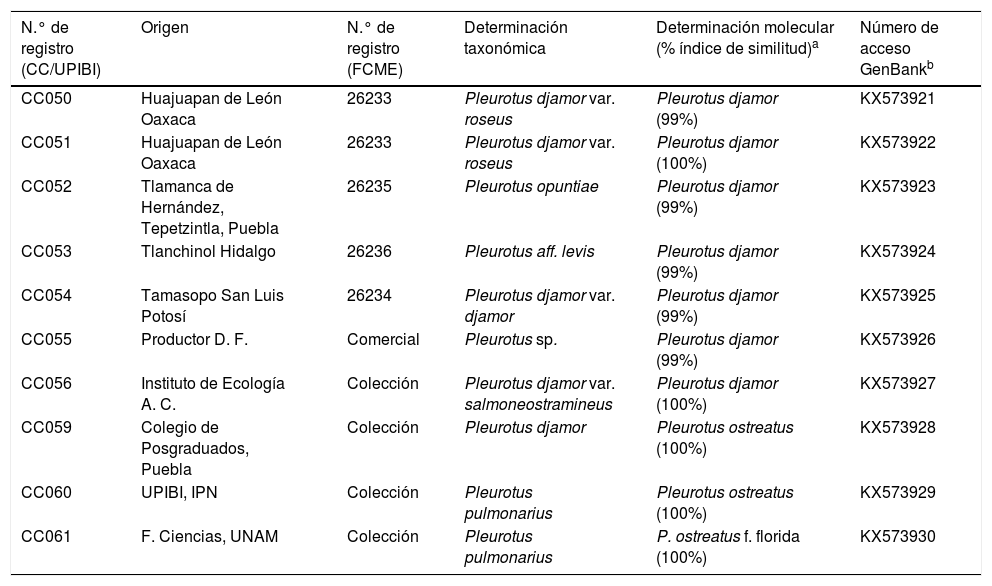
ResultadosDeterminación taxonómica de las cepas silvestresCon base en su morfología (ver tabla 1), las cepas de Pleurotus nativas, comerciales y de colección incluidas en este estudio fueron identificadas taxonómicamente por el personal de la Colección de Macromicetos del herbario de la Facultad de Ciencias de la UNAM (FCME). En la tabla 2 se muestran los números de registro y el nombre científico asignado a cada cepa.
Morfología del micelio y cuerpos fructíferos de las cepas de Pleurotus comerciales, de colección y silvestres
| Micelio | Carpóforo | |||||||
|---|---|---|---|---|---|---|---|---|
| N.° de registro | Color | Densidad | Textura | Crecimiento | Color | Laminas | Píleo | Estípite |
| CC050 | Blanco | Regular | Algodonosa | Regular | Blanco | Decurrentes | Flabeliforme | Excéntrico |
| CC051 | Rosa | Regular | Algodonosa | Regular | Rosa | Decurrentes | Flabeliforme | Excéntrico |
| CC052 | Blanco | Alta | Algodonosa | Regular | Blanco | Decurrentes | Flabeliforme | Excéntrico |
| CC053 | Blanco | Alta | Algodonosa | Regular | Blanco | Decurrentes | Flabeliforme | Excéntrico |
| CC054 | Blanco | Alta | Algodonosa | Exuberante | Blanco | Decurrentes | Plano convexo | Excéntrico |
| CC055 | Rosa | Regular | Filamentosa | Regular | Rosa | Decurrentes | Flabeliforme | Excéntrico |
| CC056 | Blanco | Alta | Algodonosa | Regular | Blanco | Decurrentes | Flabeliforme | Excéntrico |
| CC059 | Blanco | Alta | Algodonosa | Exuberante | Gris | Decurrentes | Liso convexo | Excéntrico |
| CC060 | Blanco | Alta | Algodonosa | Exuberante | Beige | Decurrentes | Flabeliforme | Excéntrico |
| CC061 | Blanco | Alta | Algodonosa | Exuberante | Beige | Decurrentes | Flabeliforme | Excéntrico |
Las cepas CC050 a CC061 están depositadas en el Laboratorio de Cultivos Celulares de la UPIBI (CC/UPIBI), del Instituto Politécnico Nacional de México.
Número de acceso de las cepas de Pleurotus comerciales, de colección y silvestres utilizadas para el estudio
| N.° de registro (CC/UPIBI) | Origen | N.° de registro (FCME) | Determinación taxonómica | Determinación molecular (% índice de similitud)a | Número de acceso GenBankb |
|---|---|---|---|---|---|
| CC050 | Huajuapan de León Oaxaca | 26233 | Pleurotus djamor var. roseus | Pleurotus djamor (99%) | KX573921 |
| CC051 | Huajuapan de León Oaxaca | 26233 | Pleurotus djamor var. roseus | Pleurotus djamor (100%) | KX573922 |
| CC052 | Tlamanca de Hernández, Tepetzintla, Puebla | 26235 | Pleurotus opuntiae | Pleurotus djamor (99%) | KX573923 |
| CC053 | Tlanchinol Hidalgo | 26236 | Pleurotus aff. levis | Pleurotus djamor (99%) | KX573924 |
| CC054 | Tamasopo San Luis Potosí | 26234 | Pleurotus djamor var. djamor | Pleurotus djamor (99%) | KX573925 |
| CC055 | Productor D. F. | Comercial | Pleurotus sp. | Pleurotus djamor (99%) | KX573926 |
| CC056 | Instituto de Ecología A. C. | Colección | Pleurotus djamor var. salmoneostramineus | Pleurotus djamor (100%) | KX573927 |
| CC059 | Colegio de Posgraduados, Puebla | Colección | Pleurotus djamor | Pleurotus ostreatus (100%) | KX573928 |
| CC060 | UPIBI, IPN | Colección | Pleurotus pulmonarius | Pleurotus ostreatus (100%) | KX573929 |
| CC061 | F. Ciencias, UNAM | Colección | Pleurotus pulmonarius | P. ostreatus f. florida (100%) | KX573930 |
Las cepas CC055 a CC061 son cepas depositadas en el Laboratorio de Cultivos Celulares de la UPIBI (CC/UPIBI), del Instituto Politécnico Nacional de México.
FCME: Colección de Macromicetos del Herbario de la Facultad de Ciencias de la UNAM, México.
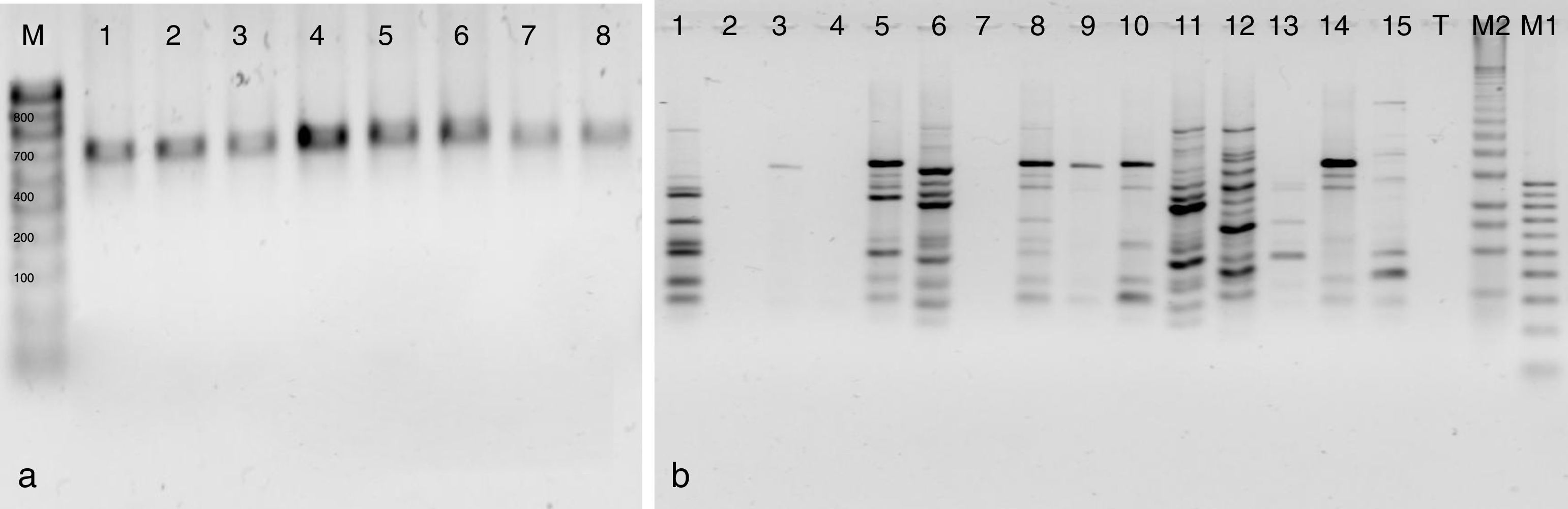
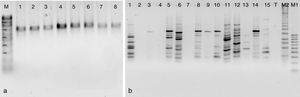
Con respecto al análisis molecular, en la región ITS de las cepas silvestres y de colección se obtuvo un solo producto de amplificación con un tamaño de 700-800 pb (fig. 1a). Las cepas silvestres y de colección se identificaron como Pleurotus djamor y Pleurotus ostreatus con una similitud del 99-100% (tabla 2).
a) Productos amplificados del ADN de las cepas de Pleurotus mediante cebadores ITS. Calle M: marcador molecular, calle 1: CC052, calle 2: CC051, calle 3: CC056, calle 4: CC054, calle 5: CC053, calle 6: CC055, calle 7: CC050, calle 8: CC059. b) Productos amplificados mediante el oligonucleótido UBC811 para las cepas de Pleurotus parentales, híbridas, reconstituidas y neohaplontes. Marcadores moleculares M1 (100 pb) y M2 (1 Kb plus), calle 1: CC050, calle 2: CC051, calle 3: R7B4, calle 4: R5B4, calle 5: R3B4, calle 6: R1B4, calle 7: R7R1, calle 8: B6B4, calle 9: R7, calle 10: R5, calle 11: R3, calle 12: R1, calle 13: B6, calle 14: B4, calle 15: R1, calle 16: Testigo (T).
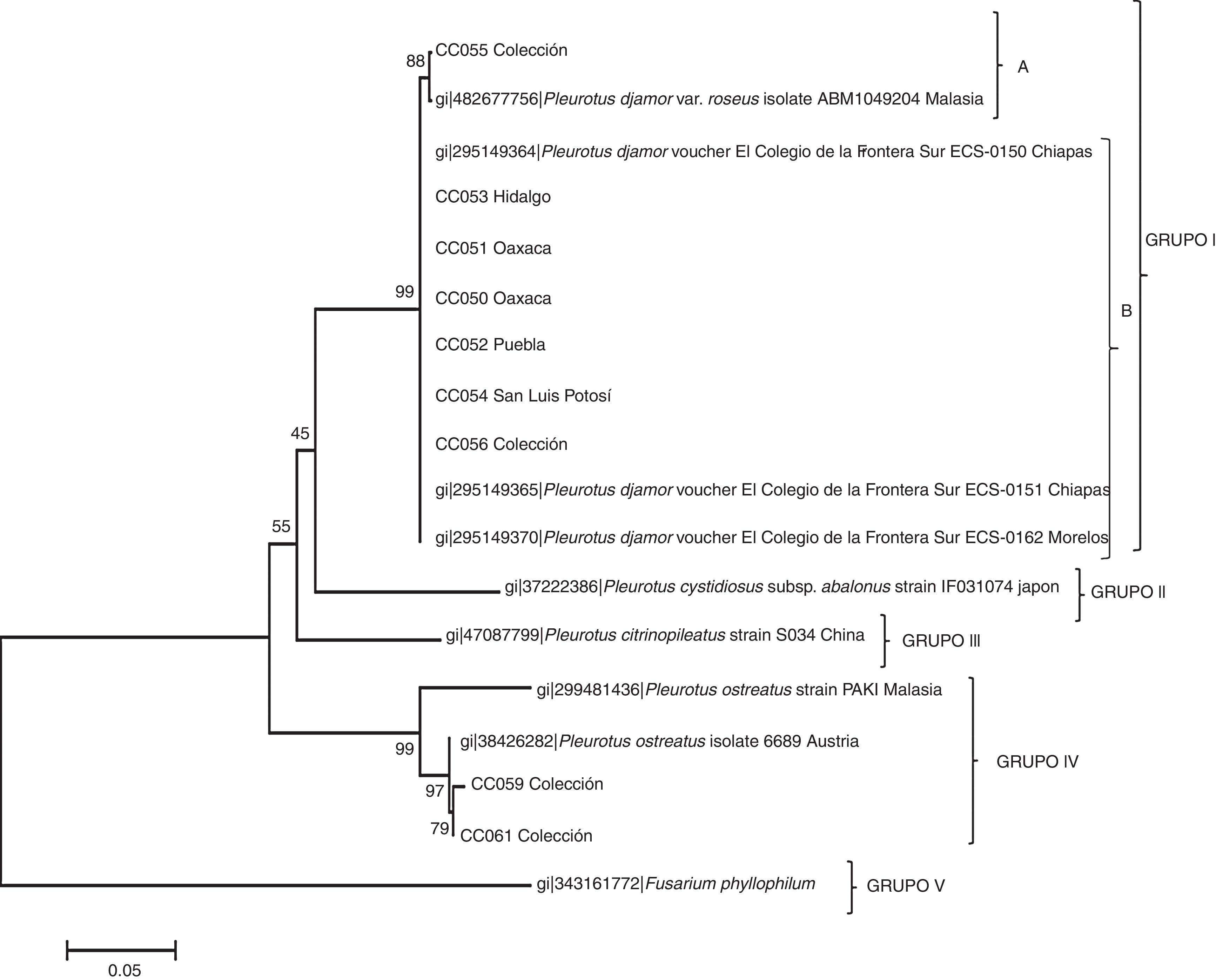
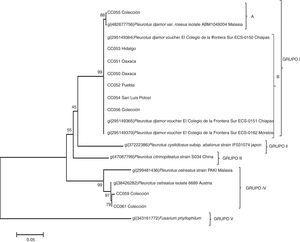
Uno de los hallazgos de este estudio fue que las cinco cepas silvestres recogidas en Huajuapan de León, Oaxaca (CC050, CC051), Tlamanca de Hernández en Tepetzintla, Puebla (CC052), Tlanchinol, Hidalgo (CC053), Tamasopo y San Luis Potosí (CC054) fueron identificadas por los métodos moleculares como P. djamor. También una de las cepas de colección (CC056) y una comercial (CC055) correspondieron a la especie P. djamor. Otras dos cepas de colección (CC059 y CC061) fueron identificadas como P. ostreatus (tabla 2). Con las secuencias de ITS se elaboró el árbol filogenético (fig. 2) que permitió separar las especies de Pleurotus en grupos en función de las homologías de sus secuencias con aquellas del GeneBank. El grupo I, con P. djamor, estaba constituido por dos subgrupos, el A con un 88% de homología e integrado por la cepa comercial CC055 y la cepa P. djamor var. roseus (aislamiento ABM1049204 de la región de Malasia) y el subgrupo B, con una homología del 99%, que englobaba las cepas silvestres CC053, CC056, CC054, CC052, CC051 y CC050, en conjunto con la cepa ECS0162 del estado de Morelos y las cepas ECS0151 y ECS0150 provenientes de Chiapas. El grupo II estaba formado únicamente por Pleurotus cystidiosus subespecie abalonus (cepa IFO31074 de Japón). El grupo III quedó conformado por Pleurotus citrinopileatus (cepa S034 de China), y en el grupo IV, con una homología del 97%, se situó P. ostreatus con las cepas de colección CC059 y CC061 junto con la cepa 6689 de Austria y la cepa PAK1 de Malasia. Finalmente, el grupo V contuvo la especie Fusarium phyllophilum como grupo externo.
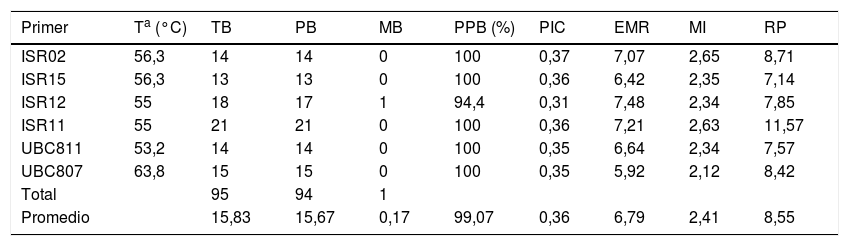
Análisis de ISSRLos seis marcadores ISSR utilizados mostraron una alta capacidad para generar bandas de ADN polimórfico amplificado (ver fig. 1b y tabla 3). En total se visualizaron 95 productos de amplificación; los seis oligonucleótidos ISSR amplificaron fragmentos en las 14 cepas estudiadas, con un número de fragmentos amplificados que van desde 13 (ISR15) hasta 21 (ISR11). El oligonucleótido ISR11 presentó el mayor poder de resolución (11,57). El promedio de polimorfismo obtenido con los seis marcadores utilizados fue del 99% y los tamaños de las bandas se situaron entre 100 y 4.000 pb. El valor más alto en el contenido de información polimórfica fue de 0,37 para el oligonucleótido ISR02, mientras que el menor valor (de 0,31) fue para el oligonucleótido ISR12; no obstante, este marcador presentó el valor más alto (7,48) en la relación múltiple efectiva. El valor promedio del índice de marcador fue de 2,41; el valor más alto (2,65) fue para el oligonucleótido ISR02 y el más bajo el del oligonucleótido UBC807 (2,1).
Parámetros de eficiencia genética de los marcadores ISSR utilizados
| Primer | Ta (°C) | TB | PB | MB | PPB (%) | PIC | EMR | MI | RP |
|---|---|---|---|---|---|---|---|---|---|
| ISR02 | 56,3 | 14 | 14 | 0 | 100 | 0,37 | 7,07 | 2,65 | 8,71 |
| ISR15 | 56,3 | 13 | 13 | 0 | 100 | 0,36 | 6,42 | 2,35 | 7,14 |
| ISR12 | 55 | 18 | 17 | 1 | 94,4 | 0,31 | 7,48 | 2,34 | 7,85 |
| ISR11 | 55 | 21 | 21 | 0 | 100 | 0,36 | 7,21 | 2,63 | 11,57 |
| UBC811 | 53,2 | 14 | 14 | 0 | 100 | 0,35 | 6,64 | 2,34 | 7,57 |
| UBC807 | 63,8 | 15 | 15 | 0 | 100 | 0,35 | 5,92 | 2,12 | 8,42 |
| Total | 95 | 94 | 1 | ||||||
| Promedio | 15,83 | 15,67 | 0,17 | 99,07 | 0,36 | 6,79 | 2,41 | 8,55 |
EMR: relación múltiple efectiva; MB: número de loci monomórficos; MI: índice de marcador; PB: número de loci polimórficos; PIC: contenido de información polimórfica; PPB: porcentaje de bandas polimórficas; RP: poder de resolución; TB: bandas totales.
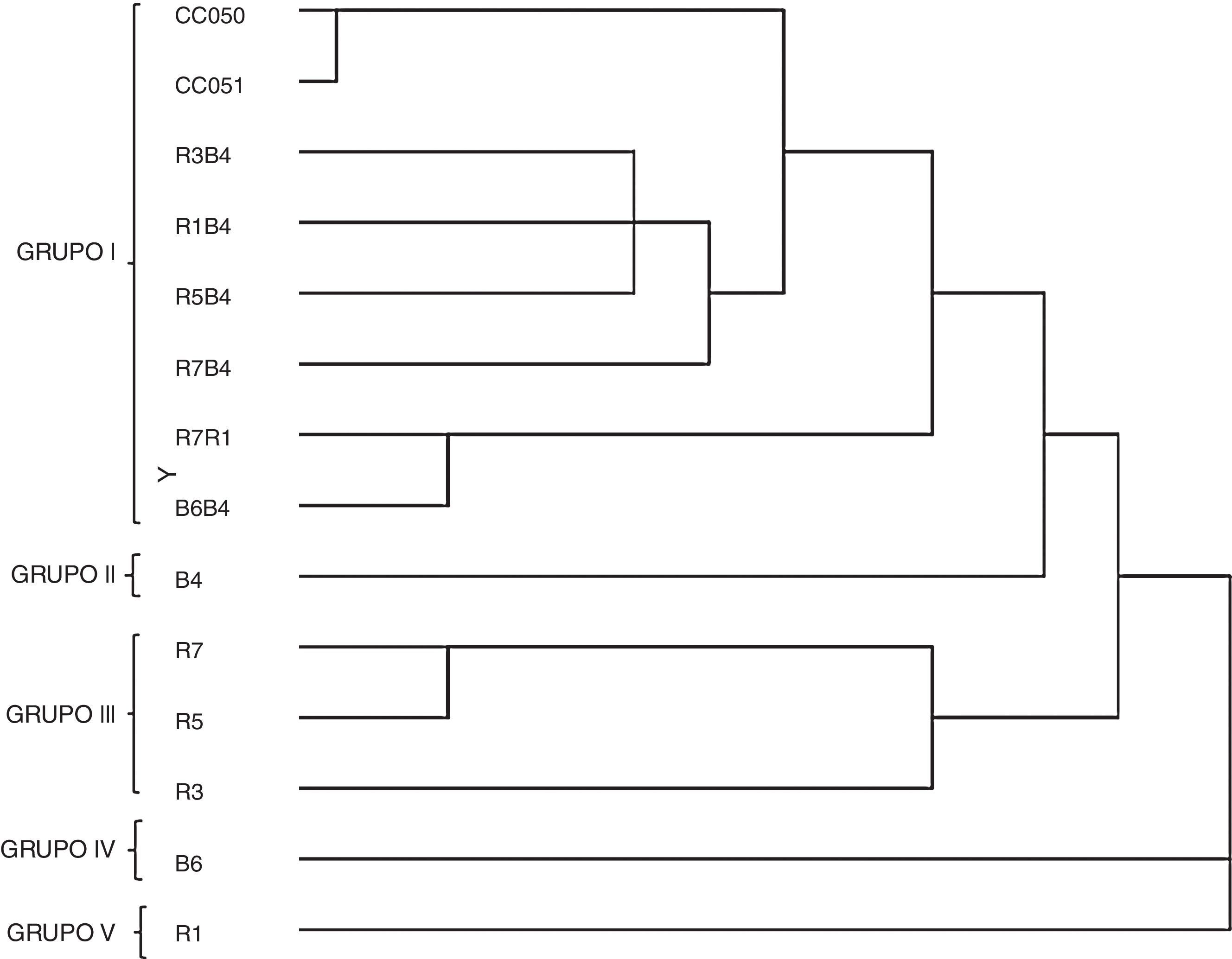
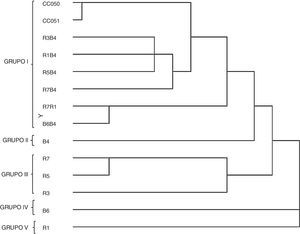
La figura 3 muestra el dendrograma basado en los datos obtenidos con los seis marcadores ISSR. Las 14 cepas se dividieron en cinco grupos. El primer grupo estuvo conformado por las ocho cepas dicarióticas: las cepas parentales silvestres CC050 (cepa blanca) y CC051 (cepa rosa), las cepas híbridas R3B4, R1B4, R7B4 y R5B4, y las cepas reconstituidas R7R1 y B6B4. El segundo grupo lo formó la cepa monocariótica B6 (proveniente de la cepa parental blanca CC050). El tercer grupo lo integraron tres neohaplontes (R7, R5 y R3) provenientes de la cepa parental rosa (CC051). El cuarto grupo lo constituyó el neohaplonte B6 (proveniente de la cepa parental blanca CC050); el neohaplonte R1 (proveniente de la misma cepa parental rosa) conformó el grupo cinco.
Dendrograma elaborado mediante el algoritmo de agrupamiento del vecino más cercano (Neighbour-Joining) de las cepas de Pleurotus parentales (CC051 y CC052), reconstituidas (R7R1 y B6B4), híbridas (R3B4, RIB4, R5B4 y R7B4), neohaplontes (B4 y B6) de la cepa blanca y neohaplontes (R1, R3, R5 y R7) de la cepa rosa.
La amplificación de la región ITS del ADN de las cinco cepas silvestres, tres cepas de colección y una cepa comercial con los cebadores ITS1 e ITS2 permitió obtener un fragmento único de 700 pb. Estos datos son consistentes con los obtenidos por Zervakis et al.36 para las cepas de Pleurotus abalonus, P. cystidiosus, P. cystidiosus var. formosensis, Pleurotus fuscoquamulosus, Pleurotus smithii y Pleurotus australis. Asimismo, Imtiaj et al.12 obtuvieron fragmentos de 600-800 pb en cepas de P. djamor, Pleurotus cornucopiae, P. cystidiosus, Pleurotus eryngii, Pleurotus ferulae, Pleurotus nebrodensis, P. ostreatus y Pleurotus pulmonarius. Es evidente que el número de pares de bases obtenido con las cepas analizadas en el presente estudio se encuentra en el intervalo reportado para especies del género Pleurotus. Por otro lado, es importante señalar que, al secuenciar estos productos de amplificación, las cepas de Pleurotus estudiadas quedaron identificadas en dos especies diferentes, P. djamor y P. ostreatus, y que estos resultados difieren de los obtenidos sobre la base de los caracteres morfológicos. El análisis molecular de las cepas silvestres CC052 y CC053 indicó que ambas corresponden a P. djamor y morfológicamente fueron identificadas como Pleurotus opuntiae y Pleurotus levis, respectivamente (ver tabla 2). Una situación similar tuvo lugar con las cepas de colección CC055 y CC056, que morfológicamente se identificaron como P. pulmonarius, mientras que el estudio molecular las identificó como P. ostreatus.
La utilización de marcadores ISSR permitió detectar una amplia diversidad genética de las cepas de P. djamor utilizadas, con polimorfismos del 99% y 15,8 bandas de promedio por cebador. Otros autores han documentado también una alta diversidad genética en otras especies del género Pleurotus; así, Ying et al.35 obtuvieron valores promedio de polimorfismo del 74,6%, con 10,6 bandas por cebador para diferentes cepas de P. pulmonarius, mientras que Wang et al.33 obtuvieron un porcentaje promedio de polimorfismo del 84,3%, con 16,2 bandas por cebador en cepas de P. eryngii. Zhang et al.37, al utilizar once marcadores ISSR en cepas de Pleurotus citrinopilatus, encontraron un polimorfismo del 82,9%, con 10,5 bandas por cebador. También para cepas de Pleurotus florida, Lentinula edodes y los híbridos formados entre ellos se han encontrado polimorfismos del 98,4%, con 11 bandas promedio por cebador19.
Los marcadores ISSR fueron una herramienta útil al unirse a diferentes sitios del ADN genómico de las cepas analizadas y generar un alto grado de bandas polimórficas. Los oligonucleótidos ISR02 e ISR11 presentaron los valores más altos de contenidos de información polimórfica (0,37 y 0,36, respectivamente) y de poder de resolución (8,71 y 11,57, respectivamente), y tienen un alto potencial para estudios adicionales que permitan investigar más ejemplares o sitios de muestreo con un número reducido de cebadores22,38.
Además de realizar la determinación molecular de las cepas silvestres, otro propósito de este trabajo fue utilizar los marcadores ISSR con la finalidad de poder caracterizar molecularmente las cepas dicarióticas híbridas, parentales y reconstituidas de P. djamor. Se esperaba que todas las cepas dicarióticas presentaran los mismos patrones de bandas y se ubicaran en un mismo grupo en el dendrograma elaborado, lo que efectivamente observamos al situarse en el primer grupo (fig. 3) las cepas dicarióticas, con el subgrupo de las cepas híbridas (R3B4, R1B4, R5B4 y R7B4) como el más cercano a la cepas parentales CC050 y CC051; el subgrupo de las cepas reconstituidas (R7R1 y B6B4) se encontró más distante. Una situación similar se presentó en el trabajo de Avin et al.4, con un dendrograma en el que las cepas dicarióticas parentales e híbridas de P. pulmonarius se situaron en el mismo grupo. Es importante precisar que las dos cepas silvestres de P. djamor se ubicaron en el mismo subgrupo, a pesar de que el color de los cuerpos fructíferos de la cepa CC050 es blanco y los de la cepa CC051, rosa (ver tabla 1). Las cepas monocarióticas o neohaplontes obtenidas por desdicariotización química se situaron en un grupo independiente, lo que llevó a la separación de las cepas monocarióticas (neohaplontes) con respecto a las dicarióticas (híbridas, parentales y reconstituidas), hecho que coincide con lo observado por otros investigadores en cepas de Agaricus bisporus: con los oligonucleótidos ISSR se generaron dendrogramas en los que claramente se separan las cepas monocarióticas de las dicarióticas20,29. En cepas de Pleurotus se han utilizado diferentes marcadores para detectar cepas monocarióticas; en P. sajor-caju se utilizaron marcadores RAPD10; para P. ostreatus var. florida, P. djamor var. roseus y los híbridos formados a partir de estas cepas se utilizaron marcadores RFLP28, y en P. pulmonarius se han utilizado marcadores ISSR para secuencias del espaciador intergénico 2 (IGS2)3. En este estudio fue posible agrupar las cepas monocarióticas fuera del grupo de las cepas dicarióticas. Por otro lado, es importante resaltar que, a su vez, las cepas monocarióticas se separaron en diferentes grupos, por lo que se requieren estudios con mayor número y diferente tipo de cepas monocarióticas (neohaplontes y monospóricos) para así determinar la posible pérdida de bandas en estos componentes, como se ha reportado con anterioridad20,29.
También es importante indicar que con los oligonucleótidos utilizados fue posible la amplificación de bandas polimórficas para cepas monocarióticas de Pleurotus. En general, este estudio demostró la utilidad de los marcadores ISSR para el análisis de homocariones y heterocariones en cepas de P. djamor.
Aunque los estudios previos registran la existencia de P. djamor en los estados de Chiapas, Yucatán y Tabasco (zona sur), Puebla, Morelos y Michoacán (zona centro), Veracruz (este), Jalisco (oeste)15 y la región sur del estado de Oaxaca17, los trabajos realizados demuestran la distribución de la especie por todo el país. También es valioso indicar que las cepas CC053 y CC054 de Hidalgo y San Luis Potosí, respectivamente, son registros nuevos de la especie P. djamor. Respecto a las cepas CC050 y CC051 de la región Mixteca de Oaxaca, el registro anterior fue de la Sierra Sur de Oaxaca17; como no estaba documentada la presencia de P. djamor para Huajuapan de León, este también es el primer registro en esta zona. La cepa CC052 de la Sierra Norte de Puebla es registrada por primera vez en la zona de Tepetzintla y fue documentada con anterioridad en el altiplano del municipio de Atlixco11. Teniendo en cuenta el origen de las cepas evaluadas, puede afirmarse que P. djamor es una especie ampliamente distribuida en las regiones tropicales y subtropicales de México11,21.
La alta diversidad genética obtenida en la cepas dicarióticas y monocarióticas de P. djamor puede ser explicada por el hecho de que se trabajó con cepas silvestres, que presentan genotipos adaptados al medio ambiente natural37. Por otro lado, la gran variabilidad genética de las cepas utilizadas es importante para los programas de mejora genética.
ConclusionesEl uso de los marcadores ITS permitió la identificación molecular de las cepas silvestres de Pleurotus, que forman un grupo inter-estéril P. djamor. Con la obtención del árbol filogenético se observó la relación entre las cepas silvestres y las cepas de P. djamor previamente recogidas en diferentes regiones de México, lo que puso en evidencia la presencia de esta especie en varias regiones de nuestro país.
Los oligonucleótidos ISSR utilizados llevaron a la amplificación del ADN de P. djamor tanto en las cepas dicarióticas como en las monocarióticas, lo que permitió su separación. El alto polimorfismo de estas cepas puso en evidencia la diversidad genética de P. djamor, que puede utilizarse en la producción comercial y en programas de mejoramiento genético.
Conflicto de interesesLos autores declaran que no existe conflicto de intereses en relación con el contenido de este manuscrito.
Este trabajo ha sido apoyado por la Secretaría de Investigación y Posgrado del Instituto Politécnico Nacional (SIP-IPN: 20160240).
Se agradece el apoyo brindado por los investigadores Dr. Joaquín Cifuentes Blanco y Biól. Lilia Pérez Ramírez, de la Colección de Macromicetos del herbario de la Facultad de Ciencias de la UNAM (FCME), por la caracterización taxonómica de las cepas de Pleurotus.