La susceptibilidad a la tuberculosis pulmonar (TB) es multifactorial, por lo que factores genéticos como las moléculas del complejo mayor de histocompatibilidad (CMH) y los receptores tipo inmunoglobulinas presentes en células NK (KIR) podrían predisponer al desarrollo de la misma.
ObjetivoEvaluar si algún alelo de HLA clasei en combinación con determinados KIR podría estar relacionado con el desarrollo de TB en la comunidad amerindia Wichi en el noreste argentino.
MétodosEn un estudio de cohorte se incluyeron 18 familias, 35 individuos afectados con TB, 84 convivientes familiares y 63 controles sanos del mismo grupo étnico.
Los loci A y B de HLA clasei se tipificaron mediante amplificación genérica seguida de hibridación reversa (Dynal), el locusC por PCR-SSOP. Los receptores KIR fueron amplificados con primers de secuencia específica SSP-PCR.
ResultadosSe observó una asociación altamente significativa con el alelo B*35:19/47 en TB vs. contactos familiares [Pc=0,0051] y vs. controles [Pc=0,0033] y con el alelo HLA-C*03 en TB vs. contactos [Pc=0,014] y vs. controles [Pc=0,0033]. Cuando se analizaron los receptores KIR, se observó aumento de la frecuencia KIR2DL3/KIR2DL3 en combinación con el grupo C1 de HLA-C (p=0,018). C*03 pertenece al grupoC1, por lo que podemos pensar que esta combinación ejerza una fuerte acción inhibitoria sobre la célula infectada con Mycobacterium.
ConclusiónHLA-B35:19/47-HLA-C*3 podrían ser un factor de susceptibilidad a TB y la combinación KIR2DL3-HLA-C1, por su efecto inhibitorio sobre las células NK, podría contribuir al curso clínico de la infección por M.tuberculosis.
The susceptibility to pulmonary tuberculosis (TB) is multifactorial, thus genetic factors such as HLA and immunoglobulins-like killer receptors (KIR) could be predisposed to the development of the disease.
Aim To evaluate whether any HLA classi allele and its combination with KIR could be related to the development of TB in the Wichi Amerindian community in north-eastern Argentina.
MethodsA cohort study was conducted that included 18 families, 35 individuals affected with TB, 84 cohabiting families, and 63 controls of the same ethnic group.
A and B loci of HLA classi were typed by generic PCR followed by reverse hybridization (Dynal), locus C by PCR-SSOP. KIR receptors were studied using sequence specific PCR.
ResultsThere was a highly significant association with allele B*35:19/47 in TB vs. household contacts [Pc=0.0051] and vs. controls [Pc=0.0033], and with allele HLA-C*03 in TB vs. household contacts [Pc=0.014] and vs. controls [Pc=0.0033]. KIR receptors had shown increased KIR2DL3/KIR2DL3 frequency in combination with the C1 group of HLA-C (P=.018). HLA-C*03 belongs to C1 group, and this combination could have a strong inhibitory action on the infected cell.
ConclusionHLA-B35:19/47-C*03 haplotype could be a susceptibility factor to TB and KIR2DL3-HLA-C1 combination have an inhibitory capacity on NK cells, and might contribute to the course of the infection by Mycobacterium tuberculosis.
La tuberculosis pulmonar (TB) es una de las principales enfermedades infecciosas con alta mortalidad, sobre todo en países en desarrollo. La infección por Mycobacterium tuberculosis se debe fundamentalmente al tropismo que tiene este bacilo por el tejido pulmonar y el desarrollo de la enfermedad depende fundamentalmente de la respuesta inmune del huésped, donde la inmunidad innata juega un importante rol1.
La fagocitosis del M.tuberculosis por los macrófagos alveolares es el primer evento en la relación huésped-patógeno, en la que intervienen los receptores toll-like (TLR) involucrados en el reconocimiento celular de la micobacteria a través de los patrones moleculares de este bacilo, desencadenando una compleja serie de eventos que conducen a una mayor expresión de genes pro-inflamatorios. Las mutaciones en los TLR pueden contribuir a una mayor susceptibilidad a las infecciones2–4.
Por otro lado, las células NK están involucradas en el reconocimiento del patógeno a través de sus receptores KIR que interactúan con las moléculas HLA clasei que se expresan en la superficie de las células infectadas. Un balance entre las señales de inhibición y activación vía receptores KIR de las células NK controla la actividad efectora de este linaje celular5–7.
La especificidad de la interacción KIR-HLA puede verse influenciada por la presencia de ciertos residuos de aminoácidos en posiciones específicas. Las moléculas KIR2DL1 interactúan con alotipos HLA-C del grupoC2 (-Cw2, -Cw4, -Cw5, -Cw6, -Cw15, -Cw1602, -Cw17 y -Cw18) con Lys (K) en posición 80, mientras que moléculas KIR2DL2, KIR2DL3 interactúan con alotipos del grupo C1 (-Cw1, Cw3, -Cw7, -Cw8, -Cw12, -Cw14) con asparagina (N) en posición 807–9. Con respecto a los ligandos de los receptores KIR de activación, son controvertidos y aún no están definidos, aunque hay algunas evidencias de que el receptor KIR3DS1 tendría como ligando al epitope de HLA-Bw4 y el residuo 138 del receptor KIR3DS1 tendrían relevancia en la unión a la molécula HLA-Bw410. KIR3DS1/3DL1 segregan en un mismo locus y definen haplotiposA y B de los receptores KIR y se han demostrados distintos genotipos KIR en diferentes grupos étnicos11,12.
En la región sanitaria 6 de la provincia del Chaco se encuentra el mayor número de tribus amerindias (Mataco, Wichi, Toba) las pautas culturales-religiosas de los diferentes grupos étnicos llevan al retraso en la consulta y por lo tanto dan lugar a las formas graves de presentación. Con el fin de establecer si el elevado número de individuos con tuberculosis en población aborigen podría deberse a factores genéticos, se estudiaron los alelos de HLA clasei y su combinación con receptores KIR de activación y/o inhibición con el objetivo de establecer si pueden contribuir al desarrollo de tuberculosis pulmonar en la comunidad Wichi.
Material y métodoSe estudiaron 182 individuos de origen Wichi que viven en Misión Nueva Pompeya Chaco, en el noreste de Argentina (localizado en latitud 24¿ 56′ y longitud 61¿ 31′), la misma se refiere a población rural que vive agrupada en distintos parajes, la gran mayoría desarrolla tareas rurales. Cuenta con una población aproximada de 2.496 habitantes con una tasa de prevalencia de tuberculosis en el año 2006 de 247,09 por 100.000 habitantes. Los recién nacidos son vacunados con BCG de acuerdo con el Programa Nacional, con buena cobertura en la zona. Las metas básicas del Programa de Control son detectar el 70% de los casos, y que el 85% de estos complete el tratamiento bajo supervisión.
El protocolo de investigación fue aprobado por el Comité de Ética del Departamento de Docencia e Investigación del Hospital y por el programa de Control de la Tuberculosis del Ministerio de Salud Pública de la Provincia del Chaco y respetando la declaración de Helsinki de 1975.
El estudio se llevó a cabo sobre 18 familias que tenían al menos un integrante afectado con TB, confirmado en forma clínica, radiológica y por la presencia del bacilo-ácido alcohol resistente en el análisis de esputo, realizado al menos en 2 ocasiones separadas, diagnosticados durante el período 2002-2006. Estas familias estaban integradas por hijos, padres, abuelos y tíos, con un total de 119 individuos, 35 de ellos afectados con TB y 84 individuos convivientes no afectados. El grupo control estuvo constituido por 63 individuos no relacionados con las familias estudiadas, pero pertenecientes al mismo grupo étnico. Tanto los familiares convivientes como el grupo control no tenían signos síntomas clínicos ni de laboratorio de TB. La población fue altamente homogénea y afectada a las mismas condiciones ambientales.
Genotipificación HLASe realizó la extracción de ADN de células mononucleares y tipificación de los locusA y B del HLA clasei por medio de amplificación genérica por PCR y revelados por hibridación reversa (Dynal RELI-SSO) y analizadas con el software suministrado por el fabricante de acuerdo a la nomenclatura del comité de WHO y la base de datos del IMGT/HLA (www.tissue-typing.com).
La genotipificación de HLA-Cw fue realizada por PCR-SSOP según Cereb et al.13.
Genotipicación KIRLos receptores KIR de inhibición y activación fueron estudiados usando PCR con primers de secuencia específica según metodología establecida por Uhrberg et al.14. Se utilizaron cebadores para los siguientes receptores: KIR3DS1, KIR3DL1, KIR2DL2, KIR2DL3.
Los productos de PCR fueron visualizados por electroforesis en gel al 2% de agarosa.
Análisis estadísticoLa frecuencia génica es expresada en porcentaje y definida por el número de alelos testeados (N) dividido por el número de individuos multiplicado por 2. El estudio de asociación (casos y controles) se llevó a cabo con la frecuencia alélica utilizando el Test de contingencia 2 por 2 x2 test (doscolas) o el test de Mantel-Haenszel. El OR fue calculado usando el método de Wolf's. Se aplicó la corrección de Bonferroni para múltiples comparaciones a los alelos HLA (la Pc se obtuvo al multiplicar el valor de p×30 para el locusB, y ×15 para el locusC). En la frecuencia de los haplotipos Bw4, Bw6, KIR y gruposC1 y C2 se aplicó la corrección de Yates sobre el valor de p según Mantel-Haenszel.
ResultadosNosotros evaluamos en primer lugar la frecuencia de los diferentes alelos HLA clasei (locusA, B y C) y en segundo término a los receptores KIR3DS1, KIR2DL1, KIR2DL2 y KIR2DL3 y su combinación con los epitopes de Bw4, Bw6 y HLA-C, grupo C1 y C2. Se comparó la frecuencia de los genes en los pacientes con TB (n=35) respecto a sus contactos familiares (n=84) y al grupo control (n=63). El total de las familias con individuos afectados fueron 18, de las cuales 8 tuvieron un solo afectado, 6 con 2 afectados, 3 con 3 afectados y una con 6 individuos afectados. Todos ellos pertenecientes al mismo grupo étnico y con las mismas condiciones socioeconómicas y ambientales.
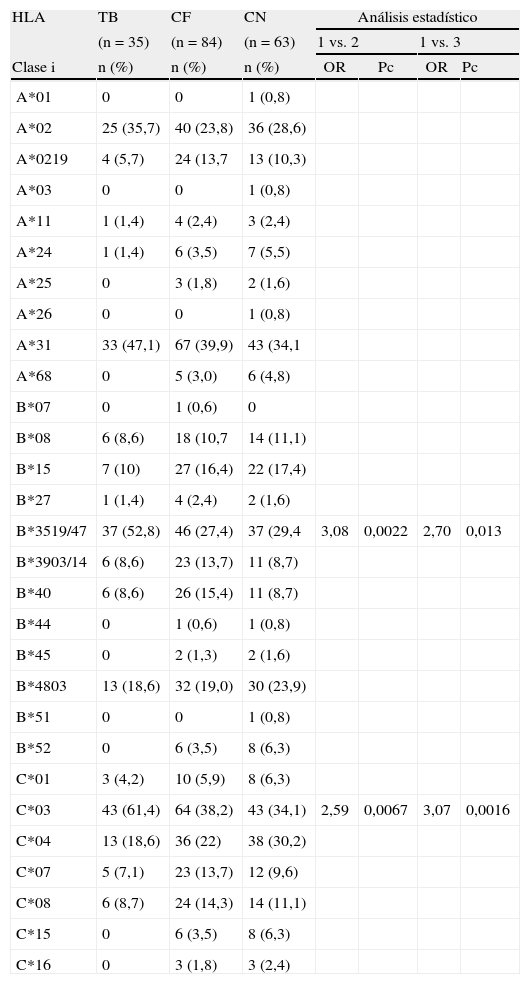
Cuando analizamos HLA clasei no observamos diferencias en la distribución de los alelos del locusA entre los distintos grupos estudiados. En el locusB, una asociación altamente significativa fue observada para el alelo B*35:19/47 en pacientes con TB vs. contactos familiares [OR=2,97; p=0,00017; Pc=0,0051] y vs. controles (OR=2,70; p=0,0011; Pc: 0,033). Respecto al locusC, el alelo HLA-C*03 se encontró significativamente aumentado en TB vs. contactos familiares [OR=2,59; p=0,0009; Pc=0,014] y vs. controles [OR=3,07; p=0,00022; Pc=0,0033] (tabla 1).
Frecuencia de alelos de HLA clasei en tuberculosis pulmonar (TB), contactos familiares (CF) y controles normales (CN)
| HLA | TB | CF | CN | Análisis estadístico | |||
| (n=35) | (n=84) | (n=63) | 1 vs. 2 | 1 vs. 3 | |||
| Clase i | n (%) | n (%) | n (%) | OR | Pc | OR | Pc |
| A*01 | 0 | 0 | 1 (0,8) | ||||
| A*02 | 25 (35,7) | 40 (23,8) | 36 (28,6) | ||||
| A*0219 | 4 (5,7) | 24 (13,7 | 13 (10,3) | ||||
| A*03 | 0 | 0 | 1 (0,8) | ||||
| A*11 | 1 (1,4) | 4 (2,4) | 3 (2,4) | ||||
| A*24 | 1 (1,4) | 6 (3,5) | 7 (5,5) | ||||
| A*25 | 0 | 3 (1,8) | 2 (1,6) | ||||
| A*26 | 0 | 0 | 1 (0,8) | ||||
| A*31 | 33 (47,1) | 67 (39,9) | 43 (34,1 | ||||
| A*68 | 0 | 5 (3,0) | 6 (4,8) | ||||
| B*07 | 0 | 1 (0,6) | 0 | ||||
| B*08 | 6 (8,6) | 18 (10,7 | 14 (11,1) | ||||
| B*15 | 7 (10) | 27 (16,4) | 22 (17,4) | ||||
| B*27 | 1 (1,4) | 4 (2,4) | 2 (1,6) | ||||
| B*3519/47 | 37 (52,8) | 46 (27,4) | 37 (29,4 | 3,08 | 0,0022 | 2,70 | 0,013 |
| B*3903/14 | 6 (8,6) | 23 (13,7) | 11 (8,7) | ||||
| B*40 | 6 (8,6) | 26 (15,4) | 11 (8,7) | ||||
| B*44 | 0 | 1 (0,6) | 1 (0,8) | ||||
| B*45 | 0 | 2 (1,3) | 2 (1,6) | ||||
| B*4803 | 13 (18,6) | 32 (19,0) | 30 (23,9) | ||||
| B*51 | 0 | 0 | 1 (0,8) | ||||
| B*52 | 0 | 6 (3,5) | 8 (6,3) | ||||
| C*01 | 3 (4,2) | 10 (5,9) | 8 (6,3) | ||||
| C*03 | 43 (61,4) | 64 (38,2) | 43 (34,1) | 2,59 | 0,0067 | 3,07 | 0,0016 |
| C*04 | 13 (18,6) | 36 (22) | 38 (30,2) | ||||
| C*07 | 5 (7,1) | 23 (13,7) | 12 (9,6) | ||||
| C*08 | 6 (8,7) | 24 (14,3) | 14 (11,1) | ||||
| C*15 | 0 | 6 (3,5) | 8 (6,3) | ||||
| C*16 | 0 | 3 (1,8) | 3 (2,4) | ||||
La frecuencia génica es expresada en porcentaje y definida por el número de alelos testeados (n) dividido por el número de individuos ×2. Los valores estadísticos se expresaron en OR con intervalos de confianza de 95%; el valor de p fue corregido según Bonferroni.
HLA-B*35:19/47 en TB vs. Contactos familiares: OR=2,90 (1,60<OR<5,53), p=0,00017.
HLA-B*35:19/47 en TB vs. Controles: OR=2,70 (1,41<OR<5,18), p=0,00115.
HLA-C*03 en TB vs. Contactos familiares: OR=2,59 (1,40<OR<4,79), p=0,00097.
HLA-C*03 en TB vs. Controles: OR=3,07 (1,61<OR<5,91), p=0,00022.
HLA clasei muestra un fuerte desequilibrio de ligamento entre los alelos HLA B*35:19/47 y HLA-C*03.
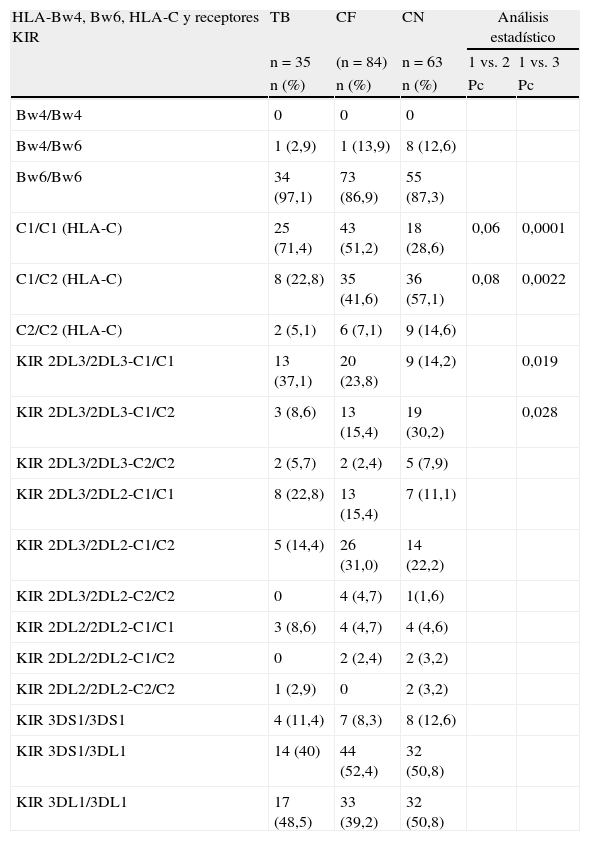
En la tabla 2 se muestra la distribución de los epitopes HLA-Bw4 y Bw6, los genes de los gruposC1 y C2 del locusC, la frecuencia de los haplotipos KIR (genotipoA, B o AB) y la combinación de estos receptores KIR2DL3, KIR2DL2 con HLA-C1 y C2, así como también la combinación de Bw4/Bw6 con los receptores KIR3DS1 y KIR3DL1.
Frecuencia de los genotipos HLA-Bw4, Bw6, HLA-C1, -C2, receptores KIR y sus ligandos HLA en tuberculosis pulmonar (TB), contactos familiares (CF) y controles normales (CN)
| HLA-Bw4, Bw6, HLA-C y receptores KIR | TB | CF | CN | Análisis estadístico | |
| n=35 | (n=84) | n=63 | 1 vs. 2 | 1 vs. 3 | |
| n (%) | n (%) | n (%) | Pc | Pc | |
| Bw4/Bw4 | 0 | 0 | 0 | ||
| Bw4/Bw6 | 1 (2,9) | 1 (13,9) | 8 (12,6) | ||
| Bw6/Bw6 | 34 (97,1) | 73 (86,9) | 55 (87,3) | ||
| C1/C1 (HLA-C) | 25 (71,4) | 43 (51,2) | 18 (28,6) | 0,06 | 0,0001 |
| C1/C2 (HLA-C) | 8 (22,8) | 35 (41,6) | 36 (57,1) | 0,08 | 0,0022 |
| C2/C2 (HLA-C) | 2 (5,1) | 6 (7,1) | 9 (14,6) | ||
| KIR 2DL3/2DL3-C1/C1 | 13 (37,1) | 20 (23,8) | 9 (14,2) | 0,019 | |
| KIR 2DL3/2DL3-C1/C2 | 3 (8,6) | 13 (15,4) | 19 (30,2) | 0,028 | |
| KIR 2DL3/2DL3-C2/C2 | 2 (5,7) | 2 (2,4) | 5 (7,9) | ||
| KIR 2DL3/2DL2-C1/C1 | 8 (22,8) | 13 (15,4) | 7 (11,1) | ||
| KIR 2DL3/2DL2-C1/C2 | 5 (14,4) | 26 (31,0) | 14 (22,2) | ||
| KIR 2DL3/2DL2-C2/C2 | 0 | 4 (4,7) | 1(1,6) | ||
| KIR 2DL2/2DL2-C1/C1 | 3 (8,6) | 4 (4,7) | 4 (4,6) | ||
| KIR 2DL2/2DL2-C1/C2 | 0 | 2 (2,4) | 2 (3,2) | ||
| KIR 2DL2/2DL2-C2/C2 | 1 (2,9) | 0 | 2 (3,2) | ||
| KIR 3DS1/3DS1 | 4 (11,4) | 7 (8,3) | 8 (12,6) | ||
| KIR 3DS1/3DL1 | 14 (40) | 44 (52,4) | 32 (50,8) | ||
| KIR 3DL1/3DL1 | 17 (48,5) | 33 (39,2) | 32 (50,8) | ||
Los valores estadísticos se expresaron en OR, con intervalos de confianza del 95%; el valor de Pc fue calculado según corrección de Yates.
Homocigoto para C1 en TB vs. Contactos familiares: OR=2,38 (0,95<OR<6,10), p=0,042.
Homocigoto para C1 en TB vs. Controles: OR=6,25 (2,29<OR<17,43), p=0,00041.
KIR2DL3/2DL3-C1/C1 en TB vs. Controles: OR=3,55 (1,20<OR<10,62), p=0,009.
KIR2DL3/2DL3-C1/C2 en TB vs. Controles: OR=0,22 (0,05<OR<0,87), p=0,014.
Se observó ausencia del epitope homocigoto Bw4/Bw4, una baja frecuencia de la forma heterocigota Bw4/Bw6 y elevada frecuencia de Bw6/Bw6 en las 3 poblaciones estudiadas. Un estudio sobre 200 individuos de población criolla normal realizado en nuestro laboratorio mostró la siguiente distribución: Bw4/Bw4 12% (n=23), Bw4/Bw6 39% (n=78) y Bw6/Bw6 49% (n=99), lo que difiere con la población aborigen, donde el epitope homocigoto Bw6 se encuentra en 87,3%. Los haplotipos A y AB de los genes KIR estuvieron presentes con mayor frecuencia, sin diferencias entre los grupos estudiados.
Solo se observó un aumento del grupo C1 de HLA-C en TB respecto a sus familiares y respecto del grupo control, como así también un aumento de la frecuencia en la combinación del receptor homocigota para KIR2DL3 con el grupo C1 (Pc=0,018). El alelo HLA-C*03 que se encontró aumentado en esta población pertenece al grupo C1.
DiscusiónEn este estudio nosotros examinamos los alelos HLA clasei en población amerindia Wichi y comparamos los pacientes que desarrollaron TB con sus contactos familiares y con controles sanos, todos pertenecientes al mismo grupo étnico.
El análisis detallado de HLA clasei se muestra en tabla 1. En el locusB, solo el alelo B*35:19/47 se encontró incrementado en forma altamente significativa en pacientes con TB vs. contactos familiares y vs. controles sanos, mientras que en el locusC solo C*03 estuvo asociado con TB comparado con contactos familiares y controles.
Aunque los linfocitos T CD4+ son esenciales en la inmunidad protectiva para M.tuberculosis, numerosos reportes parecen indicar que las células T CD8+ en el contexto de las moléculas HLA clasei pueden jugar un rol crítico en el control de la enfermedad. Rajalingam et al.15 encuentran un aumento de HLA-A2 en pacientes con análisis de esputo negativos, lo que sugiere un posible papel en la mediación de la actividad supresora de las células TCD8+contra el M.tuberculosis, lo que resulta en el desarrollo de una enfermedad limitada en estos pacientes. Además, encuentra una reducción de HLA-B*18 en los pacientes con TB, en comparación con los controles. Ninguno de los antígenos de clasei se asoció con la dinámica de la gravedad de la quimioterapia o la enfermedad según la evaluación del grado de compromiso pulmonar en la radiografía de tórax. También Balamurugan et al.16 estudiaron la asociación de HLA de clasei con tuberculosis, basándose en un motif de secuencia compartida en el sitio de unión al péptido de las moléculas HLA. En los pacientes con tuberculosis pulmonar y tuberculosis miliar observaron un aumento significativo de las frecuencias de supertipos de A3 y disminución de la frecuencia de supertipos A1, resultando para ambos grupos de alelos una asociación positiva y negativa, respectivamente. Además sugieren que la presencia del motivo de unión al péptido, tipoA3 y el incremento de la expresión de ligandos para KIR2DL2 pueden influir en el curso de la tuberculosis
En el estudio de Lakshmi et al.17 se encontró una asociación positiva de la tuberculosis con el alelo HLA-B*51. Dubaniewicz et al.18 reportaron una frecuencia aumentada de HLA-B62(15) y Cw5 en tuberculosis respecto a controles. En la población estudiada se observó ausencia de Cw5 y baja frecuencia de B*51.
Una de las conclusiones de este estudio fue el aumento de la frecuencia del haplotipo HLA-B*35:19/47-C*03 asociado a TB en esta población, marcando la importancia de algunos alelos como fuertes candidatos para la susceptibilidad a esta enfermedad. Es de señalar que el HLA-B35 también se reportó asociado a TB en otras poblaciones, como en caucásicos europeos19 y en población china20.
En la tabla 2 nosotros comparamos la frecuencia de los epitopes HLA-Bw4, Bw6, HLA-C1 y HLA-C2, haplotipos KIR (A, B y AB), receptores KIR y su interacción con HLA-C. Se observó un aumento del haplotipo homocigoto para HLA-C1 en TB vs. contactos familiares, perdiendo significación cuando se aplica la corrección de Yates, mientras que en TB vs. controles sanos la diferencia fue altamente significativa (Pc=0,0001). Es de señalar que el alelo HLA-C*03 pertenece al grupoC1 y estuvo significativamente aumentado en este estudio.
Como habíamos indicado, HLA-C1 tiene como ligando los receptores KIR2DL3 y 2DL2, mientras que HLA-C2 tiene como ligando a KIR2DL1 presente en los 2 haplotipos KIR (A y B).
No fue posible analizar la interacción KIR3DL1-Bw4 o KIR3DS1-Bw4 debido a la baja frecuencia de Bw4 en esta población.
Varios estudios genéticos señalan un modelo de inhibición de las células NK por determinadas combinaciones KIR-HLA. En los modelos de inhibición, la interacción KIR2DL1-HLA-C2 es muy fuerte, seguido de KIR2DL2-HLA-C1 y finalmente KIR2DL3-HLA-C18,21,22.
Recientes estudios23 demuestran que las variaciones en los receptores KIR —y por lo tanto del repertorio de las células NK— pueden afectar la producción de citoquinas en respuesta a las micobacterias, lo que podría explicar los diferentes niveles de susceptibilidad a M.tuberculosis.
En este estudio encontramos una asociación significativa del haplotipo KIR2DL3-HLA-C1 en TB vs. controles. También Hirayasu et al.24 encuentran una fuerte asociación de este haplotipo con malaria cerebral. KIR2DL3 fue encontrado asociado significativamente a TB en población libanesa25.
Cabe señalar que el alelo HLA-B*35:19/45 pertenece al grupo Bw6, y no se ha demostrado hasta el momento un receptor KIR que interaccione con este epitope. Colantonio et al.26 señalan que la ausencia de receptores KIR que reconozcan el epitope Bw6 parecería reflejar la pérdida de receptores de esta especificidad durante la evolución humana. Por otro lado, HLA-C*03 estuvo también presente en forma significativa en TB vs. contactos y controles. Este alelo une fuertemente a un receptor KIR de inhibición (2DL3), por lo que podemos pensar que esta combinación KIR2DL3-HLA-C1 tenga una fuerte acción inhibitoria sobre la célula infectada con Mycobaterium.
En conclusión, HLA-B35:19/47-HLA-C*03 podría ser un factor de susceptibilidad a TB, mientras que la combinación KIR2DL3-HLA-C1, por su capacidad inhibitoria de la acción de estas células NK, podría contribuir al curso clínico de la infección por M.tuberculosis. Probablemente otros factores puedan influir en el curso de la TB, entre los que figuran las pautas culturales-religiosas del grupo étnico estudiado, a pesar de la fuerte campaña instaurada para el control de TB, donde se realiza una búsqueda activa de los pacientes y seguimiento del tratamiento.
FinanciaciónDonations received by Dr. Laurent Caignault Mannager DYNAL BIOTECH 2002, Distributor and Innogenetics-be through the sales manager: Ingrid Casteleyn. 2002.
Esta investigación recibió la colaboración del Hospital Julio C. Perrando y del Ministerio de Salud Pública de la Provincia del Chaco durante el desarrollo de la investigación en 2000-2005.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Agradecemos a los técnicos Irma Sabadini y Héctor Fernández por su extensa colaboración; ambos forman parte del equipo del Servicio de Histocompatibilidad y Genética Molecular del Hospital Julio C. Perrando.