Staphylococcus aureus resistente a la meticilina (SARM) y enterococo multirresistente continúan siendo un problema en la infección nosocomial y han surgido nuevos retos en su control. SARM ha aumentado su perfil multirresistente; se han descrito aislados resistentes a vancomicina y linezolid, y con menor sensibilidad a daptomicina. Asimismo, han surgido nuevos clones (ST398), inicialmente asociados a explotaciones ganaderas de cerdos, y variantes mec (mecC) con origen en animales de granja que escapan a la detección en sistemas moleculares basados en el gen mecA. En enterococo, se han descrito aislados con resistencia al linezolid y menor sensibilidad a daptomicina, y se ha comunicado en Europa el aislamiento de Enterococcus faecium resistente a ampicilina por producción de β-lactamasa. El control de SARM y enterococo multirresistente debe combinar una mejor utilización de los antimicrobianos y medidas epidemiológicas, incluyendo la detección de portadores, que reduzca la presión de colonización y, por tanto, su transmisión.
Methicillin -resistant Staphylocccus aureus (MRSA) and multirresistant entorococci are still problematic in nosocomial infections and new challenges have emerged for their containment. MRSA has increased the multiresistant profile; it has been described vancomycin and linezolid resistant isolates and isolates with decreased daptomycin susceptibility. Moreover, new clones (ST398) have emerged, initially associated with piggeries, and new mec variants (mecC) with livestock origin that escape to the detection with current molecular methods based on mecA gene have been detected. In enterococci, linzeolid resistant isolates and isolates with deceased susceptibility to daptomycin have been described. Moreover, ampicillin resistant Enterococcus faecium due to β-lactamase production has been recently found in Europe. Control of MRSA isolates and multiresistant enteroccocci should combined antibiotic stewardship strategies and epidemiological measures, including detection of colonized patients in order to reduce colonization pressure and their transmission.
A pesar de la mayor proporción de infecciones nosocomiales causadas por microorganismos gramnegativos (aproximadamente un 55% en España) cuando se comparan con las producidas por los microorganismos grampositivos (35%), estas últimas continúan siendo un problema importante en el manejo de los pacientes hospitalizados. Según el estudio de prevalencia publicado recientemente por el European Center for Diseases Control and Prevention (ECDC)1, Escherichia coli (E. coli) (15,9%) es el patógeno más importante en la infección nosocomial, siendo el segundo y tercero Staphylococcus aureus (S. aureus) (15,9%) y Enterococcus spp. (9,6%), respectivamente. Estos últimos destacan también por su perfil multirresistente; globalmente en toda Europa, el 41,2% de los aislados de S. aureus fueron resistentes a la meticilina (SARM) y el 10,2% de los enterococos fueron resistentes a la vancomicina (ERV). Aunque España está por debajo de estas cifras, ambos microorganismos tienen gran relevancia en nuestro país. El presente artículo revisa y actualiza diferentes aspectos relacionados con las infecciones nosocomiales producidas por estos microorganismos.
Epidemiología de Staphylococcus aureus resistente a la meticilinaS. aureus es paradigma de la adquisición de mecanismos de resistencia y el desarrollo de antimicrobianos ha sido parejo a su descripción. La resistencia a la meticilina fue comunicada en 1961 pero no fue hasta los años ochenta en los que las cifras de SARM alcanzaron cantidades alarmantes, siendo en la actualidad un patógeno relevante en la infección nosocomial. No obstante, existen variaciones importantes entre distintas áreas geográficas y en algunos países, gracias a la implantación de medidas epidemiológicas de control y mejora de pruebas de detección, se ha producido un importante descenso.
En Asia, la prevalencia de SARM en los hospitales es en general muy elevada, con cifras que superan el 60% de los aislados en países como Corea del Sur, Vietnam o Taiwán2. En Europa, el estudio EARS-Net, que recoge aislados de S. aureus invasivos, ilustra las grandes variaciones entre distintos países, incluso geográficamente cercanos. Mientras que las cifras son muy bajas (< 1%) en el norte de Europa (Noruega, Suecia, Dinamarca y Holanda), en otros se supera el 45% de los aislados (Malta, Portugal o Rumanía)3. En algunos países, su incidencia ha disminuido de forma importante, resaltando el descenso en Irlanda, España, Francia, Bélgica, Alemania y Reino Unido. En estos países las cifras alcanzaron en 2011 el 23,7, el 22,5, el 17,4, el 16,1 y el 13,6%, respectivamente.
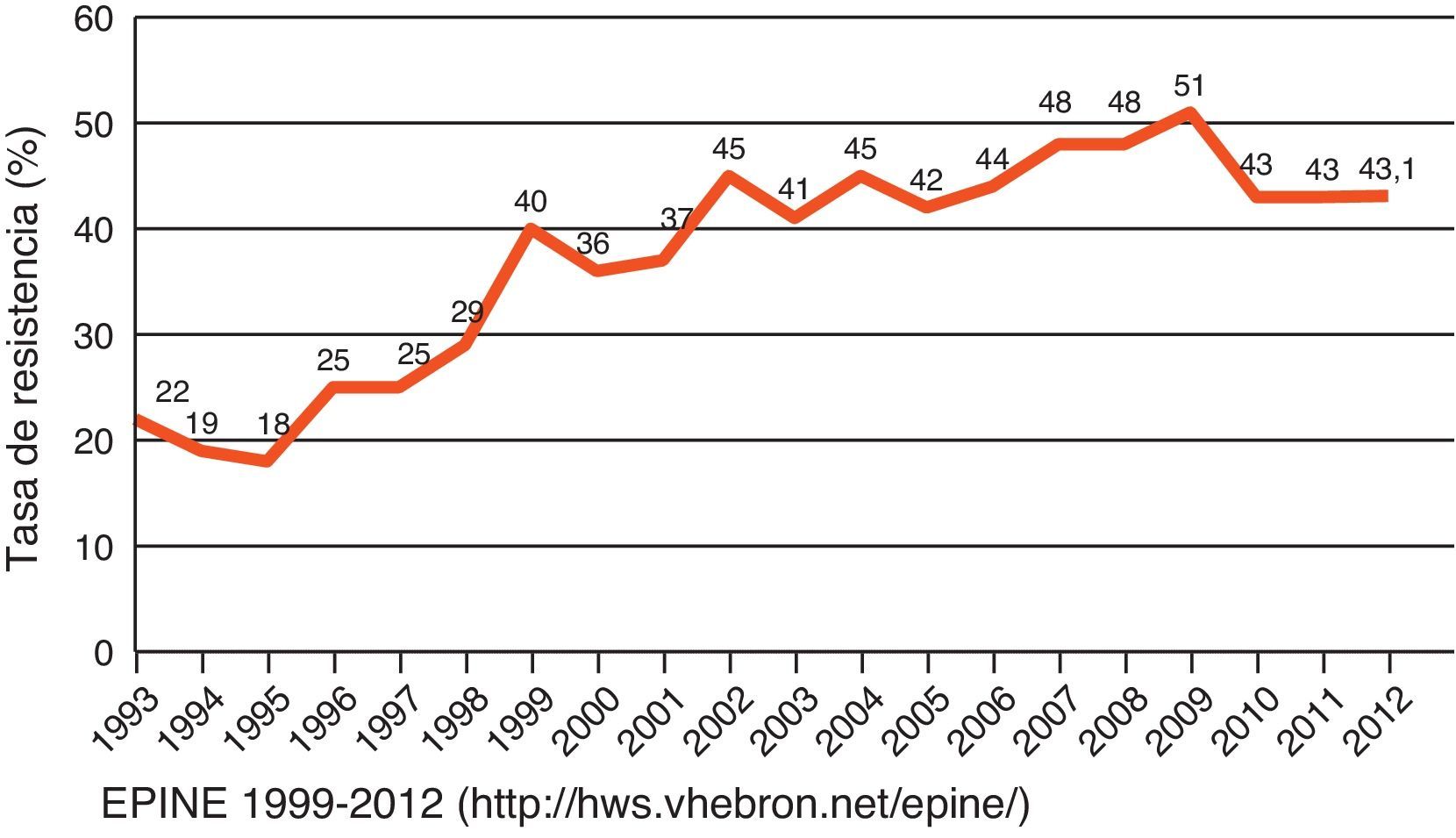
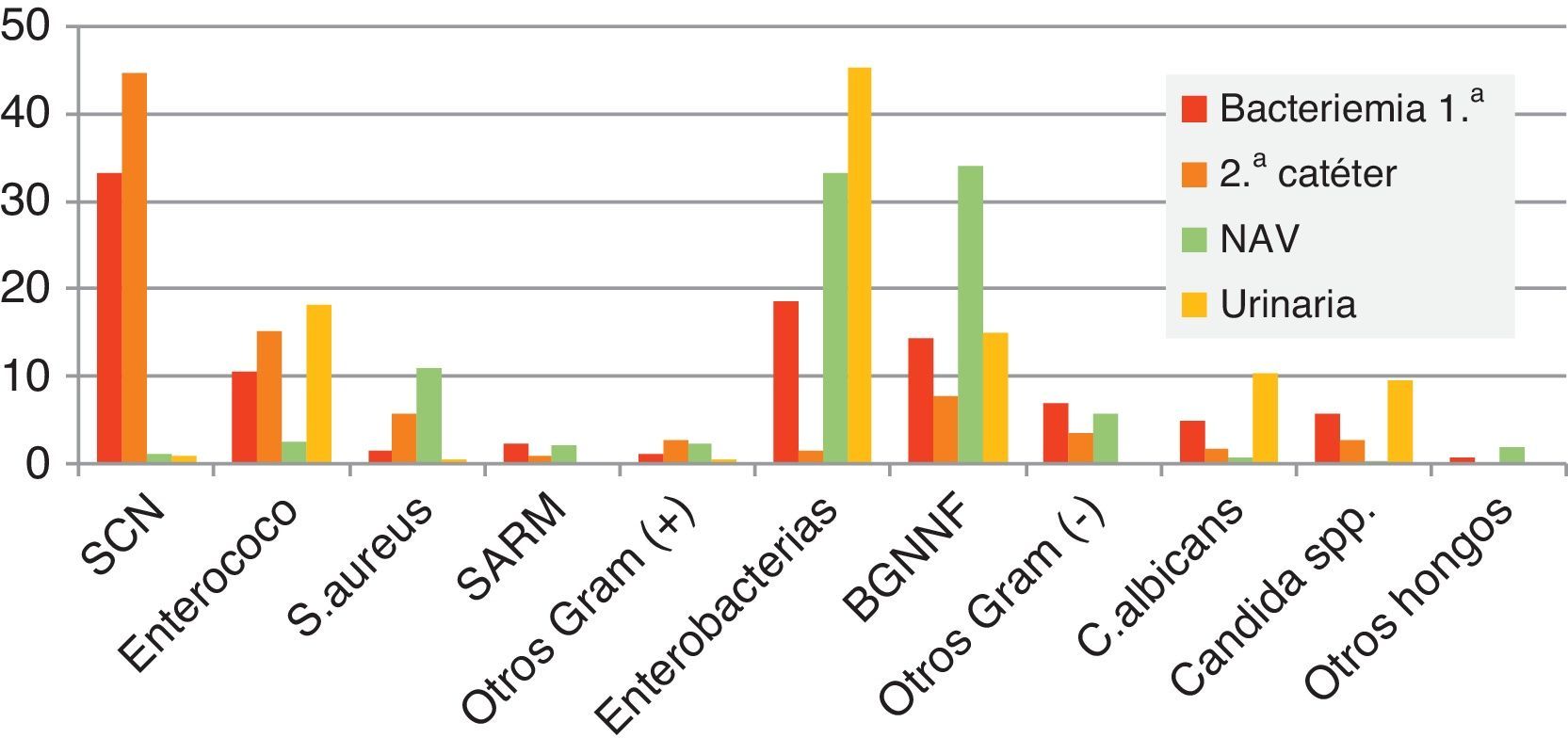
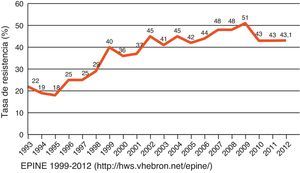
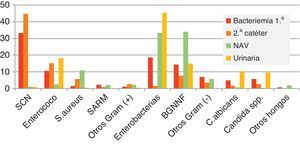
El Estudio de Prevalencia de la Infección Nosocomial en España (EPINE) del año 2012 confirma la importancia de SARM en nuestro país4. S. aureus fue el segundo patógeno en frecuencia (7,6%) por detrás de E. coli (28,1%). Un 43,1% de los aislados de S. aureus fueron resistentes a la meticilina, observándose un ligero descenso en relación con el porcentaje en años previos (fig. 1). Estos datos contrastan con los obtenidos en el Estudio Nacional de Vigilancia de Infección Nosocomial en los Servicios de Medicina Intensiva (ENVIN)5, que recoge los datos de incidencia de la infección nosocomial en las unidades de cuidados intensivos (UCI) en España y que tendría valores más cercanos al estudio EARS-Net; tan solo la cuarta parte de los aislados de S. aureus se corresponde con cepas resistentes a la meticilina. En la figura 2 se indica comparativamente con otros microorganismos la frecuencia de S. aureus y el porcentaje de aislados resistentes a la meticilina en los diferentes tipos de infección nosocomial obtenidos en el estudio ENVIN. SARM es más frecuente en la bacteriemia primaria (2,3%) y en la neumonía asociada a ventilación mecánica (2,2%) que en la bacteriemia asociada a catéter (0,9%) o en la infección urinaria (< 0,1%). Es de resaltar que estas cifras son más bajas que las recogidas en años anteriores en el estudio ENVIN. Este hecho sería debido a la implantación en España de los protocolos de «paquetes» de medidas (bundles) y programas de control de la infección nosocomial como Neumonía Zero o Bacteriemia Zero (http://hws.vhebron.net/formacion-Nzero/, http://hws.vhebron.net/formacion-BZero/).
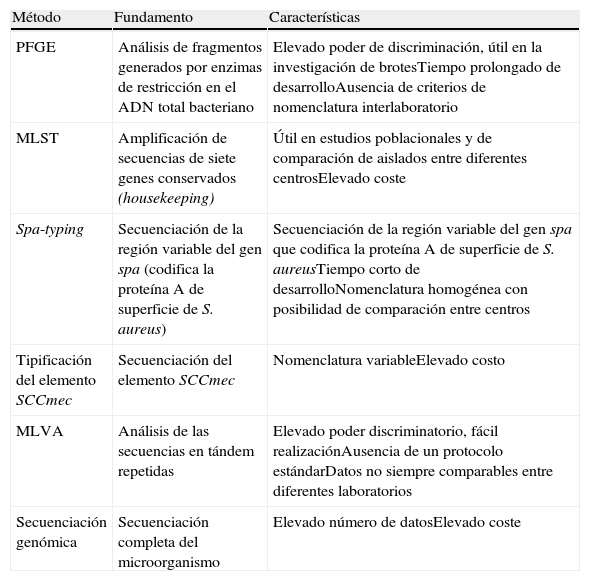
Un aspecto relevante en el análisis del SARM es el estudio de su estructura poblacional, ya que permite documentar las epidemias locales, la emergencia y la dispersión de los diferentes clones más allá de un único centro o compartimento (p. ej., hospitalario, extrahospitalario o veterinario) y explicar los cambios epidemiológicos observados. En la tabla 1 se recogen los métodos más comúnmente utilizados en la tipificación de SARM y se indican sus características esenciales y utilidad. Estos métodos han permitido confirmar la introducción del gen mecA (responsable de la producción de la PBP2a) en diferentes clones de S. aureus sensibles a la meticilina en unas pocas ocasiones y su posterior dispersión mundial. También han facilitado conocer las diferencias entre los clones eminentemente hospitalarios (hospital-associated-MRSA [HA-MRSA]), los comunitarios (community acquired/associated-MRSA [CA-MRSA]) y los relacionados con animales (livestock-associated-MRSA [LA-MRSA]).
Características de los métodos más comúnmente utilizados en la tipificación de Staphylococcus aureus resistente a la meticilina
| Método | Fundamento | Características |
| PFGE | Análisis de fragmentos generados por enzimas de restricción en el ADN total bacteriano | Elevado poder de discriminación, útil en la investigación de brotesTiempo prolongado de desarrolloAusencia de criterios de nomenclatura interlaboratorio |
| MLST | Amplificación de secuencias de siete genes conservados (housekeeping) | Útil en estudios poblacionales y de comparación de aislados entre diferentes centrosElevado coste |
| Spa-typing | Secuenciación de la región variable del gen spa (codifica la proteína A de superficie de S. aureus) | Secuenciación de la región variable del gen spa que codifica la proteína A de superficie de S. aureusTiempo corto de desarrolloNomenclatura homogénea con posibilidad de comparación entre centros |
| Tipificación del elemento SCCmec | Secuenciación del elemento SCCmec | Nomenclatura variableElevado costo |
| MLVA | Análisis de las secuencias en tándem repetidas | Elevado poder discriminatorio, fácil realizaciónAusencia de un protocolo estándarDatos no siempre comparables entre diferentes laboratorios |
| Secuenciación genómica | Secuenciación completa del microorganismo | Elevado número de datosElevado coste |
La electroforesis en campo pulsante (PFGE) continúa siendo el método de referencia a nivel local. No obstante, se están imponiendo otras técnicas con el objetivo de detectar nuevos clones, comparar aislados de más de un centro y realizar análisis filogenéticos, sobre todo en los centros de referencia6. Una de las técnicas de mayor éxito es el spa-typing; se fundamenta en la secuenciación de la región variable del gen spa que codifica la proteína A de superficie de S. aureus. Al igual que el multilocus sequence typing (MLST), basado en la secuenciación de regiones de ADN definidas de 7 genes conservados (arc, aro, glp, gmk, pta, tpi e yqi), el spa-typing utiliza una nomenclatura estandarizada a través de una base de datos específica que controla los datos generados (www.SeqNet.org). El MLST determina secuencias tipo (sequence type [ST]), que se agrupan en complejos clonales (clonal complex [CC]) y en el spa-typing, en función de la secuencia de la región variable del gen spa, se genera un spa-type. El poder discriminatorio de ambas técnicas es elevado y están recomendadas por el ECDC.
También, aunque con menos entusiasmo por su mayor complejidad, se recomienda la secuenciación del elemento SCCmec, estructura que integra el gen mec. Ofrece información precisa en cuanto a tipos y subtipos de los aislados SARM pero carece, por el momento, de una nomenclatura unificada. Hasta el momento se han establecido 11 tipos de SCCmec en función del alotipo de los complejos genéticos ccr y mec que lo integran. Los subtipos se establecen en función de los polimorfismos o las variaciones en las regiones J (joining-region, J1, J2 y J3) del SSCmec7. A pesar de su complejidad, se ha asociado el tipo del SSCmec con su origen; los tipos i, ii y iii tendrían un origen nosocomial, el iv tanto comunitario como nosocomial, el v se asocia al ganado porcino y el xi al ganado vacuno. En igual situación se encontraría la técnica multilocus variable-number tandem repeat analysis (MLVA) que, a pesar de su rapidez, elevado poder discriminatorio y fácil realización, no tiene un protocolo estándar y los datos pueden no ser comparables entre diferentes laboratorios.
En la definición de los clones suele añadirse la posible producción de marcadores de virulencia, como es la leucocidina de Panton-Valentine (PVL), presente inicialmente en la mayoría de las cepas de origen comunitario (CA-MRSA)8.
Recientemente, con el abaratamiento de los costes de los métodos moleculares, se ha propuesto la secuenciación masiva como una técnica útil en la investigación y control de brotes por SARM, aunque su implantación rutinaria es, por el momento, escasa y precisa de herramientas de análisis bioinformático que ayuden a interpretar la ingente cantidad de datos obtenidos9.
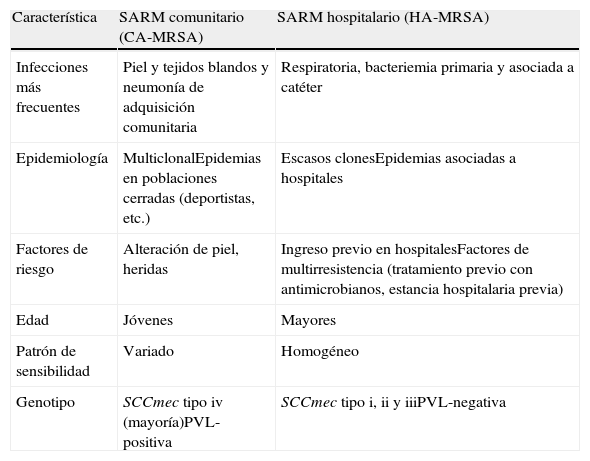
Clones hospitalarios, comunitarios y asociados a animalesLos clones hospitalarios (HA-MRSA) fueron los primeros que surgieron y los responsables del gran aumento de SARM en los hospitales durante los años ochenta y noventa. Los CC más prevalentes desde 1961 hasta nuestros días han sido CC5, CC8, CC22, CC30 y CC456. Cada CC incluye diferentes ST, con una epidemiología y localización geográfica variable. Los más prevalentes serían los integrados en los CC5 y CC8. Este último alberga al denominado clon ibérico (UK-EMRSA-5) o ST247-HA-MRSA-I, responsable durante los años noventa del fuerte incremento en España de aislados de SARM resistente a todos los aminoglucósidos y las fluoroquinolonas. Con posterioridad, este clon ha sido desplazado por otros clones sensibles a la gentamicina pero resistentes a la amikacina10. El CC30 integra al ST36 (o UK-EMRSA-16), inicialmente descrito en Inglaterra y que dio lugar, superpuesto con el ST22 (UK-EMRSA-15), al incremento de SARM en el Reino Unido en los años ochenta y noventa. En algunos países asiáticos, como Corea del Sur, Taiwán o Vietnam, se ha observado la dispersión de clones HA-MRSA en la comunidad, comportándose como los catalogados como CA-MRSA.
Durante la década de los 2000 se observó en EE. UU. un rápido aumento de cepas de SARM en la comunidad, demostrándose que la práctica totalidad de los aislados pertenecían al clon USA300 SCCmec de tipo iv productor de PVL y sensible a rifampicina, cotrimoxazol, clindamicina y tetraciclina. Se asociaba esencialmente a infecciones de piel y tejidos blandos pero también causaba neumonías necrosantes severas con mal pronóstico. En Europa, este clon ha sido infrecuente y las cepas de SARM productoras de PVL encontradas en la comunidad tienen una estructura policlonal, siendo el ST80 el mayoritario6. En estudios recientes se ha demostrado la invasión de clones con SSCmec de tipo iv, típico de los CA-MRSA, en el compartimento hospitalario, desdibujando la separación tradicional entre clones comunitarios y hospitalarios (tabla 2). Este hecho podría estar en relación con la mayor competencia o fitness de los aislados comunitarios con respecto a los hospitalarios11.
Características tradicionalmente asociadas a los clones hospitalarios y comunitarios
| Característica | SARM comunitario (CA-MRSA) | SARM hospitalario (HA-MRSA) |
| Infecciones más frecuentes | Piel y tejidos blandos y neumonía de adquisición comunitaria | Respiratoria, bacteriemia primaria y asociada a catéter |
| Epidemiología | MulticlonalEpidemias en poblaciones cerradas (deportistas, etc.) | Escasos clonesEpidemias asociadas a hospitales |
| Factores de riesgo | Alteración de piel, heridas | Ingreso previo en hospitalesFactores de multirresistencia (tratamiento previo con antimicrobianos, estancia hospitalaria previa) |
| Edad | Jóvenes | Mayores |
| Patrón de sensibilidad | Variado | Homogéneo |
| Genotipo | SCCmec tipo iv (mayoría)PVL-positiva | SCCmec tipo i, ii y iiiPVL-negativa |
PVL: leucocidina de Panton-Valentine.
Más recientemente, en 2003, se describieron aislados de SARM que pertenecían al ST398 en cuidadores de granjas de cerdos en Holanda, comprobándose su origen animal. Estos aislados representan los denominados LA-MRSA. La mayoría se han encontrado en países con baja incidencia de SARM (Holanda, Bélgica, Dinamarca y Austria), demostrándose, a pesar de su rápida y amplia difusión, una baja incidencia en hospitales12. No obstante, estudios posteriores, incluyendo los realizados en España, han mostrado la penetración y la circulación de clones LA-MRSA tanto en el ambiente hospitalario como extrahospitalario, y que podrían cambiar la epidemiología actual13.
Emergencia de nuevas variantes mecEn 2011 se describió por primera vez un análogo de mecA, inicialmente denominado mecALGA251 y en la actualidad mecC, asociado a una nueva estructura de SCCmec (tipo xi) en aislados procedentes de humanos y de ganado bovino en Dinamarca y Reino Unido14,15. Su caracterización indicó transmisión entre el hombre y el ganado bovino, aunque también se han aislado en animales domésticos y salvajes. La secuenciación genómica demostró su relación, evidenciándose un origen común. En Europa, se ha descrito en Dinamarca, Irlanda, Francia, Noruega, Alemania, Suiza, Holanda, Suecia, Bélgica y España. A pesar de su aparente dispersión, su epidemiología no es bien conocida, ya que las pruebas de laboratorio basadas en la amplificación del gen mecA o en la aglutinación relacionada con la PBP2a dan resultados negativos, por lo que es necesario implementar nuevas técnicas para su detección16.
Perfil de multirresistencia de Staphylococcus aureus resistente a la meticilinaSARM suele ser resistente a los macrólidos y las fluoroquinolonas, con un patrón de sensibilidad variable a los aminoglucósidos. Este perfil se adquirió a partir de la década de los años setenta, siendo más reciente la descripción de aislados con resistencia al linezolid y los glucopéptidos (vancomicina y teicoplanina). También se han descrito aislados con sensibilidad disminuida a la daptomicina e incluso a la ceftarolina, cefalosporina con alta afinidad por la PBP2a.
La resistencia al linezolid en el SARM se describió primeramente ligada mutaciones ribosomales y más recientemente a la adquisición del gen cfr17. Este gen, responsable de la producción de una metilasa que interactúa con el 23sRNA, se encontró inicialmente en estafilococos coagulasa negativa de origen animal. En España se describió por primera vez a nivel mundial una epidemia por SARM resistente al linezolid, asociándose su aparición al incremento del uso del linezolid18. El gen cfr suele estar localizado en plásmidos transferibles y confiere un perfil de resistencia que también afecta a las pleuromutilinas, las lincosamidas, las estreptogramina A y el cloranfenicol. Aunque los valores de CMI al linezolid en las cepas que presentan el gen cfr suelen estar entre 16 y 32mg/L, su detección con técnicas de difusión (discos o tiras en gradiente) no es sencilla, ya que se producen halos dobles que impiden una lectura adecuada de las pruebas de sensibilidad. En la actualidad, se han descrito también aislados que presentan simultáneamente mutaciones ribosomales y el gen cfr.
La resistencia o pérdida de sensibilidad a los glucopéptidos ha originado grandes debates acerca de su importancia epidemiológica y clínica. Los comités EUCAST y CLSI establecen como punto de corte de sensibilidad para vancomicina ≤ 2mg/L. El CLSI clasifica las cepas con valores de CMI para vancomicina de 4 y 8mg/L como intermedias (cepas VISA o GISA) y ≥ 16mg/L como resistentes. EUCAST no reconoce desde un punto de vista clínico la categoría intermedia y considera clínicamente resistentes cuando se obtienen valores de CMI > 2mg/L para vancomicina. Los aislados GISA o VISA se asocian con fracaso terapéutico y deben esencialmente estos valores a un engrosamiento de la pared que impide la actuación adecuada de la vancomicina. Esta misma característica la tendrían algunas de las cepas que presentan valores de CMI a la vancomicina de 2mg/L pero que expresarían de forma heterogénea esta particularidad (fenómeno denominado como heterorresistencia). También se han asociado con mayor fracaso terapéutico y con una pérdida de la actividad bactericida (tolerancia) de los glucopéptidos19. Las cepas altamente resistentes a la vancomicina (CMI ≥ 32mg/L), denominadas SARV, deben estar particularidad a la adquisición del gen vanA de enterococo a través de plásmidos que portan el transposón Tn154620. Hasta hace poco, su descripción se ha limitado a EE. UU. y algunos países de la península arábiga, pero se también se han descrito en Sudamérica y Europa (Portugal)21.
La pérdida de sensibilidad a la daptomicina (CMI ≥ 1mg/L) es infrecuente y los aislados que la presentan tiene mutaciones en genes relacionados con la membrana celular (mprF, yycFG, …) y engrosamiento de la pared celular, siendo más frecuente en aislados VISA (o GISA) con menor sensibilidad a los glucopéptidos. También se afecta la daptomicina en aislados con alteraciones en procesos de polarización de membrana22.
Un tema que preocupa en el control de la infección por SARM es la resistencia a la mupirocina, antimicrobiano empleado en la decolonización del estado de portador nasal de SARM. Su frecuencia varía de unos centros a otros y se relaciona con el uso de este antimicrobiano. La resistencia de alto nivel, asociada al fracaso terapéutico de la decolonización nasal, está mediada por el gen mupA ligado a plásmidos conjugativos que también presentan genes de resistencia a otros antimicrobianos. La resistencia de bajo nivel se asocia a mutaciones en el gen ileS, siendo incierta su repercusión clínica23.
Impacto clínico de la infección nosocomial por Staphylococcus aureus resistente a la meticilina y diagnóstico de laboratorioSARM se aísla en una gran variedad de infecciones nosocomiales, entre ellas las de localización quirúrgica, la respiratoria, la bacteriemia y, de manera testimonial, la infección urinaria (fig. 2). Los factores de riesgo para desarrollar infecciones por SARM varían según el tipo de aislado, HA-MRSA o CA-MRSA, aunque coinciden en el tratamiento previo con antimicrobianos, la colonización por SARM y el contacto sociosanitario o con individuos colonizados o infectados por este microorganismo. Por ello, se han establecido programas para detectar a los individuos colonizados tratando de reducir el riesgo de adquisición de una infección por SARM. En algunos países se han introducido programas de cribado universal de los pacientes que ingresan en los hospitales con el fin de establecer medidas de barrera que impidan su transmisión. Esta actuación ha permitido una disminución drástica de la infección por SARM.
Los métodos de detección en el laboratorio a partir de muestras clínicas incluyen la utilización de medios cromogénicos en el cultivo y métodos moleculares basados en la amplificación del gen mecA por técnicas de reacción en cadena de la polimerasa en tiempo real24. Estos últimos incluyen sistemas cada vez más rápidos y sencillos de utilizar. Su sensibilidad y especificidad son elevadas y hacen innecesario recurrir a cultivos con enriquecimiento previo. No obstante, la aparición de nuevas variantes mec obligará a desarrollar nuevas estrategias de detección. El diagnóstico de laboratorio debe completarse con el análisis epidemiológico de los aislados, que permite definir situaciones de brote.
La importancia de SARM se ha hecho evidente no solo por las implicaciones en el pronóstico, sino también por la mayor demanda de recursos sanitarios. Los pacientes infectados por SARM requieren mayor número de pruebas diagnósticas y antimicrobianos, y se asocian a estancias hospitalarias prolongadas. Se han publicado varias revisiones y metaanálisis que relacionan SARM con una mayor mortalidad25,26.
Tratamiento de las infecciones producidas por Staphylococcus aureus resistente a la meticilina y control epidemiológicoEl tratamiento de las infecciones producidas por SARM tiene grandes limitaciones por el perfil multirresistente que suele presentar. Los glucopéptidos, esencialmente vancomicina, han sido considerados tradicionalmente tratamiento de elección. No obstante, en los últimos años, se ha abierto un gran debate al comprobar cifras elevadas de fracasos terapéuticos en situaciones en las que los aislados de SARM presentaban valores de CMI ≥ 1,5mg/L, sobre todo cuando se comparan con los que se producen con aislados con valores de CMI de vancomicina ≤ 1mg/L27. En el primer caso, aun ajustando el tratamiento con vancomicina a valores valle de 15 a 20mg/L, se produce un porcentaje elevado de fracasos tras el tratamiento con vancomicina28.
En España, se han publicado diferentes guías y documentos de consenso que recogen la experiencia publicada y las opiniones de experto. La elección del tratamiento está estratificada según el tipo de infección, los valores de CMI de la vancomicina y, en el caso del tratamiento empírico, en la epidemiología local de los aislados con CMI ≥ 1,5mg/L29-31. En una de estas guías se recomienda no utilizar vancomicina de forma empírica cuando la prevalencia de este tipo de aislados supere el 10%29. El linezolid sería de elección en la infección respiratoria, mientras que la daptomicina y el linezolid en la infección de piel y tejidos blandos, la infección quirúrgica y la bacteriemia, incluyendo la asociada a catéter. La tigeciclina también se ha utilizado como alternativa en infecciones de piel y tejidos blandos e intraabdominales, sobre todo en las que tengan un componente polimicrobiano, Recientemente, se ha aprobado el uso de ceftarolina, cefalosporina con actividad frente a SARM, en la neumonía adquirida en la comunidad y en la infección de piel y tejidos blandos. Presenta una buena actividad frente a SARM debido su gran afinidad por la PBP2a.
Medias de prevención de la infección nosocomial por Staphylococcus aureus resistente a la meticilinaSARM coloniza con facilidad la mucosa nasal y la epidermis, esencialmente en los pliegues, así como las úlceras y heridas quirúrgicas. Por esta razón, los programas de cribado de pacientes colonizados incluyen generalmente estas localizaciones, ajustando la estrategia a las peculiaridades de cada centro32. La transmisión hospitalaria de SARM se suele producir con facilidad a través de las manos contaminadas del personal sanitario, de superficies o el instrumental médico contaminado o del contacto entre los pacientes, por lo que es importante la instauración de programas de cribado y aplicación de medidas de aislamiento. El ambiente hospitalario es un nicho proclive a la persistencia de SARM debido a la gran cantidad de antimicrobianos que se utilizan, el perfil multirresistente de SARM y los procesos de coselección que se produce. Al igual que para otros microorganismos multirresistentes, el riesgo de que un paciente se colonice por SARM es mayor al aumentar el número de pacientes colonizados en una determinada área hospitalaria (presión de colonización) y el tiempo de ingreso hospitalario. Este último favorece el contacto con el resto de los pacientes y los trabajadores sanitarios.
El laboratorio de microbiología desempeña un papel importante en la prevención de la infección por SARM con la detección de los nuevos casos, incluyendo la de los pacientes no hospitalizados, la implantación de técnicas de detección de este tipo de aislados y del análisis de la clonalidad para definir situaciones de brote o epidemia.
El aislamiento de los pacientes colonizados por SARM sigue siendo objeto de debate, aunque está generalizada su realización y se recoge en la totalidad de las guías de actuación en el control de la infección-colonización por SARM32. El aislamiento suele prolongarse todo el ingreso o hasta que al menos 3 muestras de vigilancia consecutivas sean negativas. No existe un consenso del tiempo que debe transcurrir entre la recogida de las muestras. Con mayor acuerdo está la utilización de guantes, mascarillas y batas desechables en el manejo de los pacientes y siempre el lavado de manos del personal sanitario tras la manipulación de los pacientes. En la tabla 3 se esquematizan las medidas habitualmente empleadas en el control de SARM. Todas estas medidas deben adaptarse a las particularidades y posibilidades de cada centro, y a su situación epidemiológica.
Medidas habitualmente empleadas en el control de SARM y ERV
| Medidas generales | Medidas específicas |
| Detección-identificación de casos | Implantación de indicadores de infecciónEstablecimiento de sistemas de alerta a equipos de infecciónImplementación de métodos de detección de pacientes colonizados (cultivos de vigilancia con medios cromogénicos y técnicas moleculares)Implementación de métodos de tipificación en la caracterización de brotesDetección del personal sanitario colonizado |
| Medidas de prevención y control de la transmisión | Lavado de manos con soluciones alcohólicasUso racional de guantes y mascarillasAislamiento de pacientes colonizados o infectadosHigiene de pacientes (baño/ducha o uso de esponjas desechables con clorhexidina)Desinfección de aparatos de exploración de pacientes |
| Descolonización de pacientes | Decolonización nasal con mupirocina con o sin la utilización de higiene con clorhexidina (en particular para SARM) |
| Limpieza/desinfección medioambiental | Limpieza de superficies, equipos y habitaciones |
| Control del uso de antimicrobianos | Restricción del uso de antimicrobianos (cefalosporinas y fluoroquinolonas) |
| Formación e implicación de los directivos | Formación del personal sanitarioCreación de equipos de seguimiento, incluyendo directivos en la institución |
ERV: enterococo resistente a la vancomicina; SARM: Staphylococcus aureus resistente a la meticilina.
La concienciación de que los centros sociosanitarios y atención extrahospitalaria desempeñan un papel esencial en el mantenimiento y la transmisión de SARM hace también necesario establecer estrategias de control en estas áreas. El número de estudios realizados es menor y las medias deben partir de la experiencia obtenida en los hospitales. En el caso de los aislados relacionados con animales (LA-MRSA), el control debe centrarse en las granjas o instalaciones de producción y en sus trabajadores. No obstante, no existe un consenso de cuáles deben ser las medidas establecidas.
Infección nosocomial por Enterococcus spp.Los enterococos forman parte de la microbiota de humanos y animales, siendo Enterococcus faecalis (E. faecalis) y Enterococcus faecium (E. faecium) las especies más abundantes en el tracto intestinal de los humanos. Aunque desde el siglo xix se conoce el papel de los enterococos como agentes causales de endocarditis, tradicionalmente se les ha considerado microorganismos con bajo potencial patógeno. No obstante, en la epidemiología de la infección enterocócica se han producido importantes cambios y desde la década de los ochenta se consideran importantes patógenos nosocomiales33. Estos cambios son consecuencia de factores relacionados con la propia biología de la bacteria y con el huésped. Los enterococos son bacterias intrínsecamente resistentes a diferentes antibióticos como las cefalosporinas o los aminoglucósidos, pero también poseen una especial capacidad para desarrollar resistencias a otros antibióticos, bien por adquisición de genes de resistencia localizados en plásmidos o en transposones (p. ej., resistencia a los glucopéptidos) o por mutaciones espontáneas que aumentan el nivel de resistencia a algunos antibióticos (p. ej., resistencia a las fluoroquinolonas). Además, los enterococos son capaces de sobrevivir en el ambiente en condiciones adversas, facilitando su diseminación y transmisión entre pacientes. Por otra parte, como consecuencia de los avances en la medicina, y al igual que lo acontecido con S. aureus, se ha producido un aumento en el número de pacientes con factores de riesgo para desarrollar una infección nosocomial por enterococo (ingreso en UCI, inmunosupresión, uso previo de antibióticos, uso de catéteres, etc.).
Epidemiología de la infección nosocomial por enterococo. Cambio epidemiológico de diferentes especiesLos enterococos son responsables del casi el 10% de infecciones relacionadas con la asistencia sanitaria según los datos recogidos en un estudio de prevalencia realizado por el ECDC en hospitales de agudos en Europa (2011-2012)1. En función del tipo de infección, los enterococos principalmente se relacionaron con infecciones de localización quirúrgica (14,5%), infecciones del tracto urinario (12,5%) y bacteriemias (8,2%). En España, el estudio EPINE mostró que los enterococos también causaron alrededor del 10% de las infecciones de origen nosocomial en el año 20124. Atendiendo al tipo de infección, los enterococos se aislaron mayoritariamente en infecciones de localización quirúrgica, urinaria y bacteriemia en un 14, un 13 y un 8,2% de los episodios, respectivamente, cifras muy similares a las descritas en otros países europeos. El estudio ENVIN también reflejó unos porcentajes infección enterocócica de más del 10% en pacientes críticos5 (fig. 2). En función de la localización de la infección, de nuevo se asociaron principalmente a infección del tracto urinario relacionada con el uso de sonda uretral (18%) y bacteriemias (13%).
De todas las especies de enterococo, E. faecalis es la especie más frecuente (80-90% de las muestras clínicas) seguida de E. faecium (5-10%)33. Antes de la década de los noventa, la ratio de infecciones causadas por E. faecalis y E. faecium era de 10:1, pero a finales de los años noventa comenzó a describirse un importante aumento en el número de infecciones originadas por E. faecium, alcanzando una ratio de 3:134,35. En nuestro país, el 6,6 y el 3,7% de las infecciones de adquisición nosocomial en el año 2012 estuvieron causadas por E. faecalis y E. faecium, respectivamente4, representando ya una ratio de 2:1. No obstante, esta ratio puede estar sujeta a variaciones geográficas, lugar de adquisición y tipo de infección5,34. Así, por ejemplo, en pacientes críticos en la infección urinaria asociada al uso de sonda urinaria, E. faecalis predomina sobre E. faecium (14% vs. 4%), mientras que en bacteriemias secundarias a infección de otros focos, el aislamiento de E. faecium es ligeramente superior al de E. faecalis (6,5% vs. 5,8%)5.
Otras especies de enterococo, como Enterococcus durans, Enterococcus avium, Enterococcus raffinosus, Enterococcus gallinarum y Enterococcus casselifalvus, solo representan un pequeño porcentaje de los aislados clínicos33. Las bacteriemias por estas especies de enterococos se han asociado a pacientes inmunodeprimidos y estancias hospitalarias prolongadas36.
Nuevos retos de resistencia: cepas productoras de β-lactamasa, resistencia al linezolid y nuevas variantes resistentes a la vancomicinaE. faecium presenta mayores problemas de multirresistencia que E. faecalis, por lo que su aumento como agente causal de infecciones relacionadas con la asistencia sanitaria presenta importantes implicaciones clínicas. En E. faecium, se ha producido un importante incremento de las tasas de resistencia de alto nivel a la ampicilina. Este tipo de aislados se describió en EE. UU. a finales de los años ochenta, mientras que Europa este aumento se comenzó a observar una década después33. En la actualidad, el porcentaje de E. faecium resistente a la ampicilina supera el 80% en muchos países europeos, entre ellos España3. La resistencia de alto nivel a las penicilinas en E. faecium es debida principalmente a la hiperproducción de la PBP5, que tiene una baja afinidad natural por las penicilinas, o también a mutaciones en el gen pbp5, que confieren incluso una menor afinidad por las penicilinas37. Por el contrario, en E. faecalis, la resistencia a la ampicilina es poco común, habiéndose descrito casos esporádicos de cepas productoras de una β-lactamasa idéntica a la de S. aureus en EE. UU., Canadá, Argentina, Líbano e India. Igualmente, y de forma excepcional, se ha descrito la presencia de cepas de E. faecium productoras de β-lactamasa en EE. UU. y más recientemente en Italia37,38.
En la década de los ochenta, se describieron en Francia y en el Reino Unido las primeras cepas de E. faecium resistentes a la vancomicina. En la prevalencia de resistencia a glucopéptidos en enterococo existen variaciones temporales y geográficas importantes. En los EE. UU., en la década de los noventa se originó un importante aumento de la resistencia a vancomicina, llegando a alcanzar desde entonces niveles endémicos en muchos hospitales33. En Europa, por el contrario, los ERV se asociaron a animales de granja y voluntarios sanos de la comunidad como consecuencia del uso de la avoparcina como promotor del crecimiento en animales. Aunque el consumo de avoparcina en animales fue prohibido en 1996, disminuyendo así la prevalencia de ERV en la comunidad, paralelamente en los hospitales europeos se ha producido un importante incremento de estas cepas33. En Europa existen grandes variaciones en la prevalencia de ERV en los distintos países. Según datos del EARS-Net de 2011, hay países como Grecia, Irlanda o Portugal que presentan una prevalencia de ERV superior al 20%, mientras que otros países, entre los que se encuentra España, la prevalencia es inferior al 5%3. En América del Sur y Asia, la prevalencia de ERV es baja, aunque se han descrito brotes nosocomiales en diferentes países33. La resistencia a los glucopéptidos se asocia mayoritariamente a cepas de E. faecium resistentes a ampicilina. En E. faecalis esta resistencia es menos frecuente, no superando una prevalencia del 5% incluso en países altamente endémicos.
La resistencia adquirida a glucopéptidos en enterococo esta mediada por 8 operones diferentes denominados van (vanA, vanB, vanD, vanE, vanG, vanL, vanM y van), siendo vanA y vanB los genes detectados con más frecuencia37. El gen vanA confiere resistencia inducible de alto nivel a vancomicina y teicoplanina, y se adquiere generalmente a través del transposón Tn1546 o de transposones relacionados de la familia Tn3. Por el contrario, el gen vanB confiere resistencia inducible de alto o moderado nivel a vancomicina y permanece sensible a teicoplanina, y con frecuencia se encuentra presente en el transposón Tn5382 junto con la resistencia a ampicilina.
En la actualidad, preocupa la emergencia de resistencias a antibióticos que pueden ser empleados como alternativas terapéuticas a la vancomicina, como son el linezolid y la daptomicina. Así, se han descrito casos aislados y brotes hospitalarios causados por enterococo resistente al linezolid39. El mecanismo de resistencia más frecuente es la mutación G2576T en la subunidad 23S del ARN ribosómico. Esta resistencia por mutaciones ribosomales se ha descrito sobre todo en pacientes que habían recibido tratamiento previo con linezolid. Más recientemente, se ha descrito en enterococo la resistencia transferible a linezolid mediado por el gen cfr que codifica una metilasa ribosómica y descrito previamente en estafilococo37.
La daptomicina es un antibiótico bactericida que también presenta actividad frente a ERV. No obstante, se han descrito cepas de E. faecalis y E. faecium con sensibilidad disminuida a la daptomicina, alcanzando valores de CMI de hasta 16mg/L40. En estas cepas existen alteraciones estructurales en la membrana celular como consecuencia de mutaciones en los genes que codifican las proteínas LiaF y GdpD implicadas en el metabolismo de los fosfolípidos y que afectarían al mecanismo de actuación de la daptomicina41.
Análisis de clonalidad y dispersión de clones resistentes y multirresistentes de enterococoAl igual que en SARM, la PFGE continúa siendo la técnica de referencia en el análisis de los brotes originados por enterococos multirresistentes y son otras técnicas las empleadas en los estudios a mayor escala y de análisis filogenético. Los cambios observados en la epidemiología de las resistencias en enterococo están íntimamente relacionados con las dinámicas poblacionales de estas bacterias. Los estudios de estructura poblacional, tanto en E. faecalis como E. faecium, han puesto de manifiesto la existencia en ambas especies de los denominados clones de alto riesgo (CAR) asociados, entre otras características, con perfiles de multirresistencia antibiótica y presencia de diferentes genes de virulencia42.
El análisis de la estructura poblacional de E. faecium mediante MLST con los genes gdh, gyd, pstS, gki, aroE, xpt e yiq identificó una subpoblación policlonal designada como complejo clonal-17 (CC17) en la que se agrupaban la mayor parte de las cepas de origen hospitalario42. Estas cepas se caracterizan por presentar resistencia a la vancomicina, ampicilina y quinolonas, aunque en la mayor parte de los países europeos E. faecium-CC17 es todavía sensible a la vancomicina. Asimismo, la secuenciación de los genomas de cepas pertenecientes a esta subpoblación hospitalaria ha identificado una gran variedad de genes que codifican factores de virulencia, secuencias de inserción e islas de patogenicidad que codifican nuevas rutas metabólicas que no están presentes en cepas que colonizan a individuos sanos de la comunidad y animales42. Todo este material genético presente exclusivamente en CC17 podría haber facilitado la adaptación de esta población al medio hospitalario. Más recientemente, el análisis mediante Bayesian-based population genetic modelling (BAPS) ha reclasificado a CC17 en 2 subgrupos denominados BAPS 2-1 y BAPS 3-3, confirmando la existencia de 3 linajes genéticos (ST17, ST18 y ST78) que podrían haber evolucionado de forma independiente43.
En E. faecalis el análisis de la estructura poblacional mediante MLST con los genes adk, atpA, ddl, gyd, gdh, purK y pstS también demostró la existencia de CAR representados por diferentes CC (CC2, CC9, CC28 y CC40)44. Estos CC agrupan la mayor parte de E. faecalis resistentes a vancomicina y causantes de brotes hospitalarios42,44.
Impacto clínico de la infección nosocomial por enterococo multirresistente y opciones terapéuticasLos enterococos pueden originar una gran variedad de infecciones nosocomiales, entre ellas las urinarias, de localización quirúrgica, bacteriemias, infecciones intraabdominales y endocarditis. Los pacientes con mayores factores de riesgo para desarrollar infecciones por enterococos multirresistentes son los pacientes neutropénicos, especialmente aquellos con mucositis, trasplantados de médula ósea o precursores hematopoyéticos, así como trasplantados de órgano sólido, pacientes en hemodiálisis, con cirugías previas, con catéteres urinarios o vasculares o pacientes con estancia hospitalaria prolongada y que han recibido antibioterapia previa39. En estos pacientes, la colonización por enterococos multirresistentes es un paso previo al desarrollo de la infección. La colonización de sondas urinarias o catéteres vasculares puede originar infecciones urinarias o bacteriemias, mientras que en pacientes neutropénicos con mucositis puede originar bacteriemias por translocación desde el tracto gastrointestinal. Ahora bien, no todos los pacientes colonizados presentan el mismo riesgo de desarrollar infección, ya que depende de la población estudiada. Así, por ejemplo, más de un 11% de los pacientes con un trasplante de órgano sólido colonizados por ERV desarrollan una infección, mientras que esta cifra asciende al 24% si se trata de pacientes oncohematológicos39. Por el contrario, en los individuos sanos colonizados por ERV el riesgo de desarrollar una infección es mínimo. No obstante, es importante recordar que la significación clínica de los enterococos en muchas ocasiones es incierta, ya que en muchas ocasiones se aíslan en el contexto de infecciones polimicrobianas como las intraabdominales o en heridas o úlceras donde su papel como microorganismo patógeno todavía no ha sido claramente establecido.
Las infecciones invasivas por enterococos multirresistentes se han asociado a una peor respuesta clínica. En pacientes con bacteriemia por ERV se ha descrito una mortalidad cruda que puede alcanzar hasta el 60%, aunque estas tasas dependen en gran medida del grupo de población estudiada39. Estas elevadas tasas de mortalidad pueden ser debidas a que estas infecciones se desarrollan en pacientes con serios problemas de inmunidad y múltiples comorbilidades.
Uno de los aspectos más evaluados de la infección enterocócica nosocomial ha sido el impacto de la resistencia a vancomicina en la respuesta clínica de las bacteriemias. En un metaanálisis en el que se incluyeron 1.614 episodios de bacteriemia enterocócica (638 por ERV y 931 por enterococos sensibles a vancomicina [ESV]), los pacientes con bacteriemia por ERV presentaron una mayor mortalidad en comparación con los pacientes con bacteriemias por ESV (OR = 2,52; IC del 95%, 1,9-3,4)45. La peor respuesta del grupo de pacientes con bacteriemia por ERV podría ser debida al retraso en el tratamiento antibiótico adecuado, una peor actividad de los antibióticos empleados para el tratamiento de ERV o bien diferencias en la virulencia de las poblaciones ERV y ESV. No obstante, la mayor mortalidad atribuida a las bacteriemias por ERV es un tema aún controvertido, ya que otros estudios no han confirmado estos hallazgos39. Por el contrario, lo que sí se ha demostrado es que las infecciones por ERV se asocian a una mayor estancia hospitalaria y un mayor consumo de recursos sanitarios en comparación con las producidas por ESV.
La mayor trascendencia clínica de las infecciones producidas por ERV son las limitadas opciones terapéuticas que existen frente a estas infecciones. El linezolid y la daptomicina son los antibióticos que principalmente se han usado como alternativa46. Linezolid ha demostrado ser efectivo en el tratamiento de las infecciones por ERV, aunque en infecciones como la endocarditis su uso es controvertido por su acción bacteriostática y aún existe poca experiencia clínica. Por su parte, la experiencia de la utilización de la daptomicina en las infecciones por ERV tampoco es muy extensa y los datos actuales parecen indicar que deberían emplearse altas dosis47. La emergencia de resistencias o sensibilidad disminuida a estos antibióticos en cepas de ERV es un tema preocupante, ya que compromete seriamente el tratamiento de estas infecciones al no existir alternativas adecuadas comercializadas.
Medias de prevención de la infección nosocomial por enterococoEl tracto gastrointestinal de los pacientes ingresados constituye el principal reservorio de ERV en el hospital. Su transmisión se produce a través de las manos contaminadas del personal sanitario, a través de superficies o instrumental médico contaminado. En los pacientes portadores de ERV, también es frecuente la colonización de la zona perianal, inguinal y otras partes del cuerpo, hecho que facilita la contaminación del ambiente y de las manos del personal sanitario39. Además, los enterococos pueden persistir en el ambiente durante largos periodos debido a su capacidad de sobrevivir en condiciones adversas, aumentando la probabilidad de diseminación.
El riesgo de que un paciente se colonice por ERV es mayor al aumentar el número de pacientes colonizados en una determinada área hospitalaria (presión de colonización), por la proximidad de otros pacientes colonizados, estancias hospitalarias prolongadas, el número de contactos con los trabajadores sanitarios y el grado de contaminación ambiental48. Los pacientes hospitalizados están sometidos a múltiples factores (uso de dispositivos médicos, tratamientos antibióticos, antiácidos, etc.) que inducen alteraciones en su microbiota intestinal, perdiendo así el efecto protector que esta ejerce para evitar la colonización por bacterias patógenas (resistencia de colonización)39. Se ha visto que los antibióticos con potente efecto anaerobicida promueven la colonización por ERV49. Una vez que se ha establecido la colonización, los tratamientos antibióticos también favorecen el sobrecrecimiento intestinal de ERV, lo que incrementa la probabilidad de contaminación de la piel y ambiente próximo al paciente, incrementando a su vez el riesgo de transmisión. Así, por ejemplo, se ha demostrado que la piperacilina-tazobactam o el metronidazol son antibióticos que promueven la persistencia de un alto nivel de colonización por ERV49.
La mayor parte de los estudios sobre la dinámica de transmisión hospitalaria de enterococo se han centrado en ERV, aunque podría hacerse extensible a otros enterococos multirresistentes. Existen pocos datos sobre la colonización y la dinámica de transmisión de E. faecium resistente a ampicilina. Se ha descrito que, al igual que sucede con ERV, en los pacientes portadores de E. faecium resistente a ampicilina se produce un importante sobrecrecimiento intestinal de estos microorganismos y hay altas tasas de contaminación ambiental50. En un estudio realizado en nuestro hospital con baja prevalencia de ERV, más del 30% de los pacientes hospitalizados están colonizados por E. faecium resistente a ampicilina (Pedrosa et al., datos no publicados). Este dato indica que los hospitales son un importante reservorio de E. faecium resistente a ampicilina que, junto con un potencial elevado grado de contaminación ambiental, podrían explicar el aumento de las infecciones causadas por estos microorganismos.
Las medidas de control de infección de los enterococos multirresistentes deben ir encaminadas a reducir las tasas de transmisión. Todas estas medidas se han focalizado en el control de ERV y, al igual que en el SARM, se recomienda la aplicación de paquetes de medidas (bundles) encaminadas a controlar la dispersión de ERV. Entre estas medidas, se encuentran la correcta higiene de manos del personal sanitario, aplicación de precauciones de contacto, el uso prudente de los antibióticos, establecer un programa de vigilancia activa y la correcta limpieza ambiental (tabla 3). Además, un punto esencial es la educación del personal sanitario para garantizar la adherencia a estas medidas. De todas las medidas propuestas, se ha demostrado que algunas tienen un elevado impacto en la disminución de las tasas de transmisión, como es la higiene de manos del personal sanitario y el aislamiento de pacientes48. Otras medidas, como la implantación de una vigilancia activa universal o la restricción del uso de vancomicina, han sido más debatidas. Es indiscutible que la realización de cultivos de vigilancia es pieza clave para identificar pacientes portadores de ERV. Las muestras más habituales para el cultivo son los frotis rectales o perianal y las muestras de heces, y en situaciones especiales, como por ejemplo un brote hospitalario, se puede realizar un estudio ambiental. Ahora bien, la aplicación de sistemas de vigilancia activa ha sido un tema controvertido, ya que su instauración de forma universal no es una medida coste-efectiva. No obstante, se ha demostrado que pueden ser de gran utilidad en pacientes de riesgo, como aquellos con ingresos hospitalarios previos, procedentes de centros de crónicos o con larga estancia, pacientes que han recibido antibióticos en los 30 días previos al ingreso o pacientes en hemodiálisis51. Por otra parte, los estudios que tratan de evaluar el impacto de la restricción de vancomicina en la reducción de las tasas de infección/colonización por ERV muestran resultados muy contradictorios52. Por el contrario, las restricciones en el uso de cefalosporinas de amplio espectro han demostrado tener un mayor impacto en la reducción de de las tasas de infección/colonización por ERV49.
En resumen, y al igual que con SARM, para garantizar el éxito de las medidas de control de los enterococos multirresistentes es necesario un enfoque multidisciplinar, la aplicación de paquetes de medidas y la educación del personal sanitario para garantizar la adherencia a las mismas.
FinanciaciónRafael Cantón y Patricia Ruiz Garbajosa son integrantes de la Red Española de Patología Infecciosa (REIPI) (financiado por el Plan Nacional de I+D+i 2008-2011 y el Instituto de Salud Carlos III, Subdirección General de Redes y Centros de Investigación Cooperativa, Ministerio de Economía y Competitividad, Red Española de Investigación en Patología Infecciosa [REIPI RD12/0015], cofinanciada por el Fondo Europeo de Desarrollo Regional «Una manera de hacer Europa» FEDER).
Conflicto de interesesLos autores declaran que no existe ningún conflicto de intereses.
Nota: sección acreditada por el Consell Català de Formació Continuada de les Professions Sanitàries. Consultar preguntas de cada artículo en: http://www.eslevier.es/eimc/formacion