En los últimos años, la incidencia de Enterococcus faecium resistente a glucopéptidos (ERG) como patógeno nosocomial está aumentando en España, debido a su alta capacidad de colonizar pacientes y personal durante largos periodos, así como de persistir en el medio ambiente hospitalario donde puede sobrevivir mucho tiempo. La principal presentación epidemiológica de ERG es en forma de brotes esporádicos1, como el ocurrido en la Unidad de Cuidados Intensivos Neonatal (UCIN) de nuestro hospital durante 2015 y que presentamos a continuación.
Desde el 21 de abril al 23 de junio de 2015 se aisló ERG en los frotis rectales de vigilancia (FRV) de diez neonatos ingresados en la UCIN de nuestro hospital (universitario de tercer nivel). La Unidad Neonatal de nivel IIIA (que no incluye cirugía), consta de seis puestos de cuidados intensivos (CIV) y 11 de cuidados intermedios (CIM) en salas independientes. El personal a cargo de la misma incluye 5 neonatólogos en plantilla más 2 en guardias, un turno matinal de 4 enfermeros y 3 técnicos de cuidados auxiliares de enfermería (TCAE), y turnos de tarde y noche de 3 enfermeros y 2 TCAE. Durante el año 2015 tuvo 282 ingresos con una estancia media de 9,10 días y registró un consumo total de vancomicina de tan solo 6,5g en 2015 (2,0g en 2014) sin consumo de ceftazidima en ambos años. A los recién nacidos se les realizan cultivos de vigilancia en el momento de ingreso (frotis nasal, faríngeo y ótico cultivados en agar sangre durante 48h a 35°C y en atmósfera con 5% de CO2) y semanalmente se realizan cultivos de vigilancia de enterobacterias productoras de β-lactamasas de espectro extendido (BLEE) mediante cultivo de FRV, en placas chromID-ESBL® (BioMérieux). Tras el aislamiento de ERG, se añadió una placa cromogénica selectiva para su cribado: chromID-VRE® (BioMérieux). Ambas placas se incuban 48h a 37°C en aerobiosis2.
Los primeros aislados de ERG se detectaron en los FRV del día 21 de abril en dos pacientes (tasa de colonización [TC]: 33%, 2 pacientes de 6 ingresados). Ambos ERG eran resistentes a ampicilina, fluoroquinolonas, presentaban resistencia de alto nivel a gentamicina y estreptomicina, y eran resistentes a vancomicina y a teicoplanina con una CMI >16mg/l, sin embargo mantenían la sensibilidad a linezolid. Comunicados estos resultados el viernes 24 de abril (23 festivo), se establecen precauciones de aislamiento de contacto y limpieza dos veces al día con SANIT P20® (cloruro de didecildimetilamonio al 4,5% y monoetanolamina al 8%), tomándose ese mismo día nuevamente FRV, que mostraron la colonización de un tercer neonato (TC 30%, 3 de 10). En la vigilancia semanal del 28 de abril, otros 4 neonatos presentaron FRV con crecimiento de ERG con el mismo fenotipo de sensibilidad, permaneciendo ingresados en ese momento un total de 6 neonatos colonizados (uno de los 3 previos había recibido el alta) (TC 55%, 6 de 11). Ante esta situación y la sospecha clínica de sepsis en un colonizado, el día 29 de abril en comisión multidisciplinar, se decidió no admitir nuevos ingresos en toda la Unidad, realizar el aislamiento en cohorte situando a todos los colonizados en la sala CIM con personal exclusivo (no compartido con el de la sala CIV), reforzar la plantilla de limpiadoras y cumplimiento de protocolos de limpieza, reforzar las medidas de aislamiento de contacto (educación del personal y padres), restringir las visitas a los padres, dotándoles de taquillas personalizadas para objetos personales y uso de lavabos diferentes.
El 4 de mayo se detectó un nuevo neonato colonizado (TC 43%, 3 de 7). El 14 de mayo tras el alta del último niño colonizado, se procedió a una doble limpieza terminal siguiendo los protocolos de limpieza de áreas críticas de nuestro hospital: retirada del material de enfermería y limpieza exhaustiva (SANIT P20®) incluyendo paredes y techos, pies de gotero, cama en su totalidad y colchón, cubos de basura y mesas auxiliares, por dos veces. Tras la finalización de la limpieza se tomaron muestras ambientales incluyendo diferentes superficies, equipos médicos, grifos, incubadoras y cunas, carros de cura, básculas, teclados de ordenador etc. utilizándose para las tomas hisopos previamente humedecidos en caldo cerebro-corazón (BHI-T® BioMérieux), que se introdujeron después de la toma en este medio y tras agitación se inocularon 0,5ml en una placa con medio selectivo chromID-VRE. A las 18-24h de incubación del caldo (37°C, en aerobiosis) se resembraron 0,5ml en el medio chromID-VRE3. En una columna móvil (equipos de apoyo) se aisló ERG.
Después de la limpieza, antes de los resultados de los cultivos, se reabre la Unidad por presión asistencial y en los FRV del día 19 de mayo se aisló ERG en una nueva paciente (TC 20%, 1 de 3) que es dada de alta el 20 de mayo junto a otro ingresado, quedando vacía la sala CIM (solo quedó un niño ingresado en CIV). Tras limpieza terminal de CIM, se toman muestras ambientales aislándose en su cuna el mismo ERG. Se procede a una nueva limpieza terminal, reabriéndose la CIM el 25 de mayo. Al cabo de un mes sin detectar ningún caso, el 23 de junio se volvió a detectar un nuevo portador (TC 20%, 1 de 5). Desde ese momento y hasta la fecha no se han vuelto a documentar nuevos aislamientos.
En resumen el brote afectó a 5 niños y 5 niñas, con edades gestacionales comprendidas entre 29 y 40 semanas, con pesos adecuados para las mismas y estancia media de 18,3 días (rango 5-40 días). La fecha de ingreso fue la fecha de nacimiento salvo en tres neonatos que ingresaron a las 48h, 7 y 9 días de vida. Ningún paciente había sido tratado con vancomicina. En cuanto a la clínica, ningún niño presentó diarrea y solo un paciente presentó un cuadro de sepsis clínica con hemocultivos negativos y sin aislamiento de ERG en ninguna otra muestra clínica que fue tratado eficazmente con linezolid durante 7 días. Seis neonatos tenían cultivos de frotis rectal previos negativos, uno tenía frotis rectal previo positivo con Enterobacter cloacae productor de una carbapenemasa de tipo VIM y los otros tres no tenían FRV previos, si bien ingresaron en la UCIN en la fecha de nacimiento y presentaron los frotis faríngeo, nasal y ótico de recién nacido sin crecimiento.
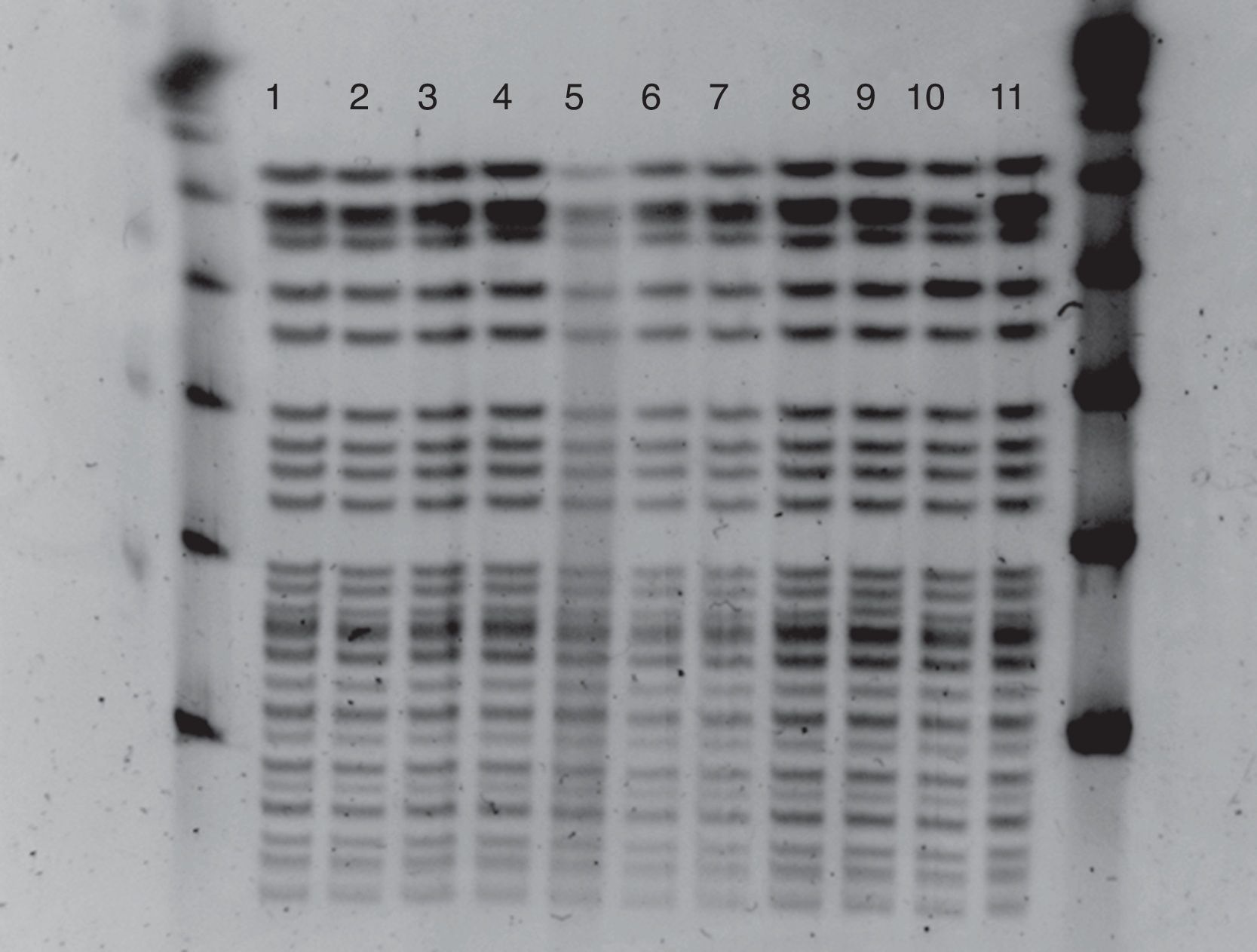
La identificación y estudio de sensibilidad se efectuó mediante VITEK2® (BioMérieux) confirmándose la resistencia a glucopéptidos mediante E-test (BioMérieux). Las cepas se enviaron al Programa de Vigilancia de Resistencia a Antibióticos del Centro Nacional de Microbiología para estudio del mecanismo de resistencia y epidemiología molecular. En todos ellos de detectó el gen vanA. Mediante electroforesis en campo pulsado (PFGE) tras digestión del ADN total con la enzima de restricción SmaI se observó que los aislamientos (incluidos los 2 ambientales) presentaban un mismo perfil (fig. 1) perteneciente al secuenciotipo 17 (ST17) por multilocus sequence typing (MLST).
El ST17da nombre al complejo clonal 17 (CC17) ampliamente distribuido en el ambiente hospitalario a nivel mundial4. Además de la resistencia clásica a ampicilina y a fluoroquinolonas del CC17, en los últimos años han aumentado los brotes producidos por aislados de este complejo clonal resistentes a glucopéptidos4,5.
En nuestro caso, los estudios rutinarios de vigilancia permitieron la rápida detección del brote en el que resultaron 10 pacientes colonizados6,7. Los cultivos ambientales demostraron la elevada capacidad de diseminación de ERG, así como su persistencia en superficies inanimadas8.
Las primeras medidas de aislamiento de contacto tomadas tras los dos primeros casos, no fueron suficientes para evitar la rápida colonización de otros 5 niños, quizás influido por el retraso en un día en su implantación, unido al fin de semana (jueves festivo en nuestra área geográfica). Queremos resaltar la dificultad que supuso para el éxito de las medidas implementadas el no disponer de habitaciones individuales y encontrarse muy próximos unos puestos de otros. Además por presión asistencial, se reabrió la Unidad inmediatamente tras la limpieza terminal, sin tener todavía el resultado de los primeros cultivos ambientales, en los que se aisló ERG, por lo que era posible la nueva aparición de colonizados.
En este estudio también es de destacar que las placas chromID-ESBL® (no selectivas para ERG) permitieron la detección de ERG, por lo que, la placa chromID-VRE, si bien facilitó la detección de ERG durante el brote, podría dejarse de utilizar, en ausencia de brotes, para reducir costes. Somos conscientes del consumo de tiempo y falta de coste-efectividad en muchos casos de los cultivos de vigilancia, pero en pacientes críticos como los neonatos, con predisposición a sufrir infecciones, se han mostrado eficaces en la presunción de los agentes causales de infección9, pudiendo permitir el inicio oportuno de una terapia antimicrobiana adecuada, habida cuenta del reto terapéutico que suponen10. Además la detección precoz de pacientes colonizados puede ayudar a mejorar las medidas de control de la infección, que limiten una rápida y amplia propagación, con el fin de evitar el incremento potencial de infecciones entre los pacientes (ratio colonización-infección, 10:1)1,6.
En conclusión, describimos un brote por ERG-ST17 en el que la realización de estudios de vigilancia de portadores de bacterias multirresistentes nos permitió la detección temprana del mismo y la aplicación precoz de medidas para su control, que incluyeron aislamiento de contacto, cohortes de pacientes, refuerzo de protocolos de limpieza, y cierre temporal de la unidad a nuevos ingresos.
A Elena Infante López (médico especialista de la Unidad de Cuidados Intensivos Neonatal), y Aurora Sacristán Salgado (médico especialista del Servicio de Medicina Preventiva) del Hospital Universitario Río Hortega (HURH), por su colaboración durante el brote y apoyo en la elaboración de este texto y a Soledad Sañudo García (médico de Admisión y Documentación Clínica) y Tomás Caro-Patón Carmona (farmacéutico especialista del Servicio de Farmacia Hospitalaria) también del HURH, por los datos aportados.