En el complejo Mycobacterium tuberculosis se engloban las especies M. tuberculosis, Mycobacterium africanum, Mycobacterium bovis, Mycobacterium bovis-Calmette y Guérin, Mycobacterium microti, Mycobacterium caprae, Mycobacterium pinnipedii y Mycobacterium canettii. Estas especies son las causantes de la tuberculosis en humanos y animales. Tradicionalmente la identificación de estas especies se ha basado en el estudio de métodos fenotípicos. No obstante, en los últimos años se han desarrollado numerosas técnicas moleculares. El objetivo de este trabajo es la evaluación de cada una de éstas para crear un esquema de identificación rápido y sencillo.
Material y métodosMediante el esquema propuesto se analizaron 251 cepas escogidas al azar entre las estudiadas en el año 2004 y se analizaron 797 cepas recibidas en el Laboratorio de Referencia de Micobacterias entre los años 2005 y 2007. La caracterización fenotípica de 4.183 cepas aisladas en este período se realizó mediante el estudio morfológico de la colonia, el aspecto del cultivo, la reducción de nitratos, la producción de niacina y el crecimiento en presencia de 10μg/ml de isoniacida del ácido 2-tiofencarboxílico y 50μg/ml de pirazinamida. El esquema de identificación diseñado consiste en lo siguiente: 1) PCR (polymerase chain reaction ‘reacción en cadena de la polimerasa’)-RFLP (restriction fragment length polymorphism ‘polimorfismo del ácido desoxirribonucleico’) del gen gyrB y digestión con las enzimas RsaI, TaqI o Sac II y PCR-RFLP del gen hsp65 y digestión con la enzima HhaI, y 2) PCR multicebador para detectar la ausencia o la presencia de las regiones RD9 y RD1.
ResultadosLos resultados obtenidos muestran una concordancia del 100% entre el esquema fenotípico y el esquema molecular.
ConclusiónEste esquema de identificación es un método rápido y sencillo basado en técnicas moleculares que puede implantarse en la mayoría de los laboratorios a un bajo coste y con un 100% de sensibilidad y de especificidad.
The Mycobacterium tuberculosis complex includes the following species: Mycobacterium tuberculosis, Mycobacterium africanum, Mycobacterium bovis, Mycobacterium bovis-BCG, Mycobacterium microti, Mycobacterium caprae, Mycobacterium pinnipedii, and Mycobacterium canettii. These species cause tuberculosis in humans and animals. Identification of mycobacterial strains has classically been performed by phenotype study. Over the last years, laboratories have developed several molecular techniques to differentiate between these species. The aim of this study is to evaluate these methods and develop a simple, fast, identification scheme.
Material and methodsWe analyzed 251 strains randomly obtained from the strains studied in 2004, and 797 strains received by the Reference Laboratory between 2005 and 2007. Phenotype characterization of 4183 strains isolated during that period was done by studying the colony morphology, characteristics in culture, nitrate reduction, niacin accumulation, and growth in the presence of thiophen-2-carboxylic acid hydrazide 10μg/mL and pyrazinamide 50μg/mL. The molecular identification scheme designed was as follows: 1) gyrB PCR-RFLP with RsaI, TaqI or SacII and hsp65 RFLP/PCR with HhaI., and 2) multiplex-PCR to determine the presence/absence of the RD9 and RD1 regions.
ResultsThe results showed 100% agreement between phenotype study and the molecular scheme.
DiscussionThis molecular identification scheme is a simple and fast method, with 100% sensitivity and specificity, that can be implemented in most clinical laboratories at a low cost.
Hoy en día la tuberculosis es una enfermedad infecciosa emergente con 8 millones de casos nuevos y que causa alrededor de 2 millones de muertes al año en todo el mundo ( http://www.who.int/tb/publications/global_report/2007 )1. Las micobacterias que causan tuberculosis en hombres y animales se agrupan dentro del denominado complejo Mycobacterium tuberculosis (CMTB). Actualmente, el CMTB se compone de las especies M. tuberculosis, Mycobacterium africanum (subtipo I similar a Mycobacterium bovis [más frecuente en la zona oeste del continente africano] y subtipo II similar a M. tuberculosis [más frecuente en la zona este de África])2,3, M. bovis, M. bovis-Calmette y Guérin (BCG), Mycobacterium microti, Mycobacterium caprae, Mycobacterium pinnipedii y Mycobacterium canettii (que se considera como una subespecie de M. tuberculosis)4. La M. tuberculosis es la principal causa de tuberculosis humana en casi todo el mundo e infecta a más de un tercio de la población mundial ( http://www.who.int/tb/publications/global_report/2007 )1, excepto en algunas regiones de África en las que el agente causal es M. africanum2 en el 60% de los casos de tuberculosis diagnosticados. Principalmente, M. bovis produce tuberculosis en animales salvajes y domésticos; el ganado bovino es el mayor reservorio de esta especie, lo que también produce, con relativa frecuencia, enfermedad en el hombre5. M. caprae6 y M. microti7 infectan al ganado caprino y a pequeños roedores, respectivamente, y ambas especies son causantes de casos de zoonosis, aunque en menor medida que M. bovis. M. pinnipedii8 es la especie que causa tuberculosis en pinnípedos, se aísla mayoritariamente en focas de varios continentes y por el momento no se han descrito casos de enfermedad en el hombre. M. bovis-BCG es una variante avirulenta obtenida en el laboratorio a partir de una cepa de M. bovis que por sucesivos pases perdió su poder patógeno y mantuvo su capacidad antigénica; actualmente se la utiliza como vacuna para la prevención de la tuberculosis, como vehículo recombinante para otras vacunas y como tratamiento inmunógeno9 que puede causar enfermedad en individuos inmunodeprimidos10 al tratarse de una cepa viva.
Un diagnóstico diferencial en la especie de las cepas que forman este complejo es importante, pues implica la administración de una adecuada pauta de tratamiento y porque conocer la especie productora de tuberculosis es una herramienta básica en la vigilancia de la salud pública.
Tradicionalmente, la identificación de las especies de este complejo se ha basado en sus características morfológicas, pruebas bioquímicas y tolerancia a pirazinamida (50μg/ml) y a isoniacida del ácido 2-tiofencarboxílico (TCH) (10μg/ml). Debido al lento crecimiento de estos microorganismos, la interpretación de los resultados puede ser subjetiva, especialmente la que se refiere a las características morfológicas que pueden variar debido a la pérdida de virulencia o debido a mutaciones asociadas a resistencia a fármacos11, lo que puede producir errores en la identificación. Clásicamente, la tolerancia a TCH se ha asignado a M. tuberculosis. Sin embargo, Yates et al12 mostraron que una gran mayoría de las cepas de M. tuberculosis aisladas de pacientes asiáticos mostraba sensibilidad al TCH, por lo que subdividió M. tuberculosis en «variedad clásica» y «variedad asiática». Además, las cepas de M. bovis resistentes a isoniacida también son resistentes al TCH, del mismo modo que hay cepas de M. tuberculosis monorresistentes a pirazinamida (característica que clásicamente se ha atribuido a M. bovis y a M. bovis-BCG). La M. africanum tiene un fenotipo intermedio entre M. tuberculosis y M. bovis y es susceptible a pirazinamida, pero en el caso de cepas monorresistentes a este fármaco, la identificación puede dificultarse13. Por esto, es necesario complementar la identificación mediante la utilización de técnicas moleculares.
Hay numerosas técnicas moleculares encaminadas a la identificación genotípica de estas especies que han desarrollado diversos grupos de trabajo. Todas éstas contribuyen a la identificación de alguna de las especies del CMTB pero ninguna recoge un esquema que permita la identificación molecular de todas las especies13,14,15,16,17,18,19,20. El Laboratorio de Referencia de Micobacterias ha evaluado cada una de estas técnicas para elaborar un esquema de identificación rápido y sencillo que pueda implementarse en la mayoría de los laboratorios hospitalarios con un bajo coste. El desarrollo y la validación de este esquema es el objetivo básico de este trabajo.
Material y métodosCepasCepas controlSe utilizaron como cepas control para todas las técnicas empleadas cepas de la Colección Americana de Cepas Tipo (ATCC) del Instituto Pasteur y del Instituto Nacional y Salud Pública y Medio Ambiente Holandés: M. bovis ATCC 35.726, M. bovis-BCG ATCC 35.733, M. africanum ATCC 25.420, M. tuberculosis H37Rv ATCC 25.618, M. microti ATCC 35.781, M. caprae CIP 105.776T y M. canettii RIVM 17.728.
Cepas estudiadas fenotípicamenteDurante los últimos 3 años (2005–2007) se estudiaron fenotípicamente 4.183 cepas del CMTB, 4.175 cepas de origen humano (sólo se incluyen en este estudio los resultados del primer aislamiento de cada paciente), 7 cepas de origen animal y una cepa aislada de un gastroscopio. Diversos hospitales de la Red Sanitaria Pública y un centro de Sanidad Animal remitieron estas cepas.
Cepas estudiadas molecularmenteSe estudió a un total de 251 cepas de las que se recibieron en el Laboratorio de Referencia de Micobacterias en el año 2004 identificadas mediante métodos fenotípicos. Se eligieron 207 cepas de origen humano y 44 cepas de origen bovino: 150 cepas de M. tuberculosis, de las que 20 se identificaron como «variedad asiática», 73 cepas de M. bovis, 20 cepas de M. bovis-BCG y 8 cepas de M. africanum.
Durante los últimos 3 años (2005–2007), se aplicó el esquema molecular a todas las cepas que se aislaron de pacientes de origen africano (545) con el fin de diferenciar M. tuberculosis y M. Africanum, a todas las cepas que se aislaron de pacientes procedentes de Asia (35) para diferenciar M. tuberculosis de «variedad asiática» de M. africanum, a todas las muestras de orina (122) para identificar M. bovis-BCG y a todas las cepas (95) que no presentaban las características fenotípicas de M. tuberculosis.
La caracterización fenotípica de estas cepas se realizó mediante la aplicación de métodos clásicos: morfología de la colonia, aspecto del cultivo, reducción de nitratos (NO3), producción de niacina y crecimiento en presencia de 10μg/ml de TCH y en presencia de 50μg/ml de pirazinamida21.
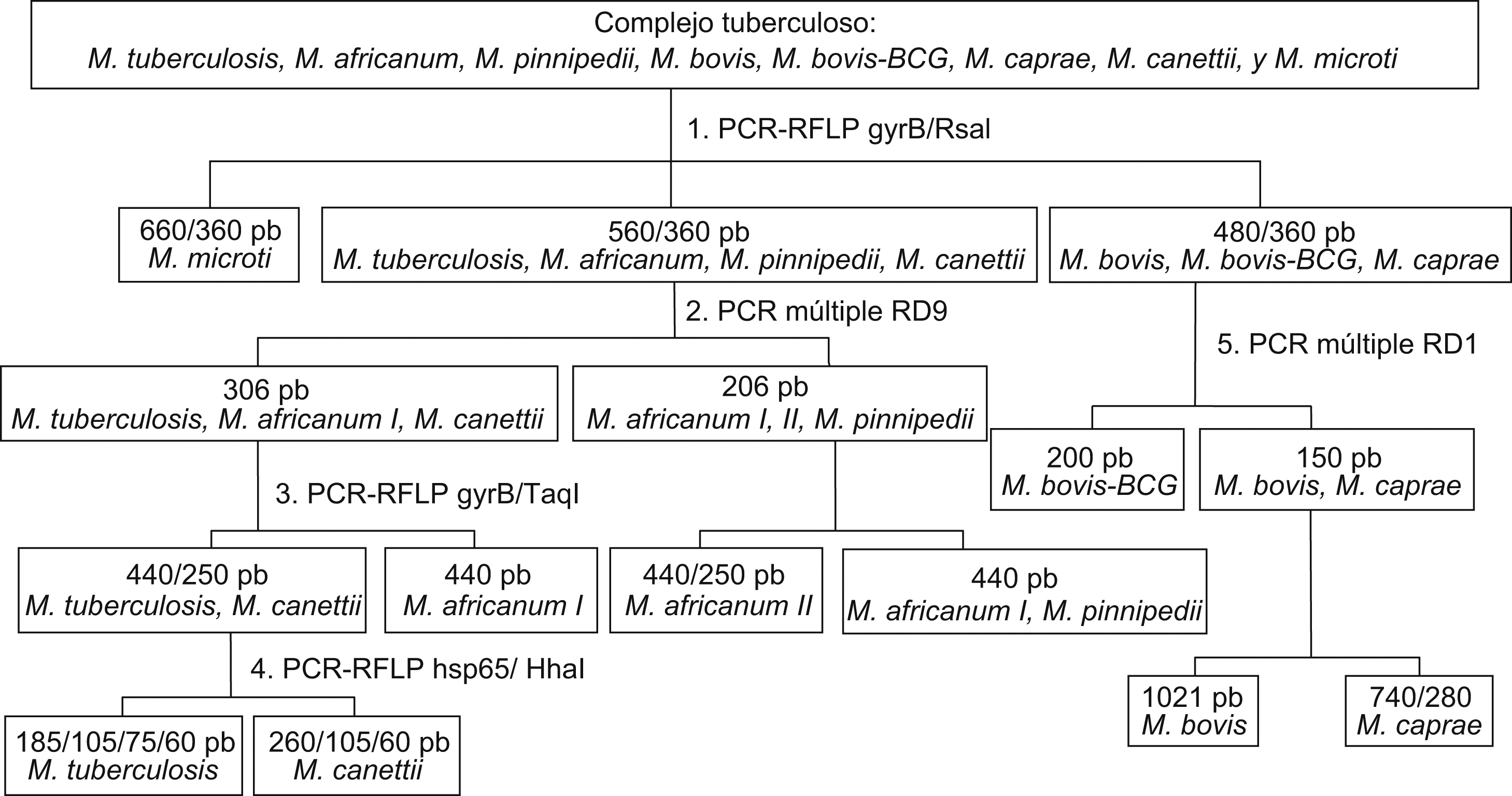
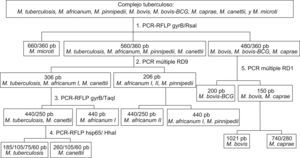
Caracterización genotípicaLa identificación individual de las especies incluidas en el grupo CMTB se realizó mediante estudios moleculares según el esquema que se muestra en la figura 1.
Figura 1. Esquema para la identificación de las especies del complejo Mycobacterium tuberculosis.
Todas las PCR (polymerase chain reaction 'reacción en cadena de la polimerasa’) se realizaron con el equipo comercial «PureTaq Ready-To-Go PCR Beads» (GE Healthcare Life Science, Piscataway NJ, EE. UU.) con 5μl de extracto crudo. Las reacciones de amplificación se llevaron a cabo en un termociclador PTC-100 Peltier (M.J. Research, Inc). La secuencia de los cebadores y de las condiciones de cada PCR se resume en la tabla 1. El tamaño de los productos amplificados se comprobó mediante electroforesis en 0,8% de agarosa mediante la utilización de marcadores de tamaño molecular (Marcador X de Roche y marcador de tamaño de 50pb y 100pb de GE Healthcare Life Sciences). El análisis de los patrones de restricción se realizó mediante electroforesis en 2% de agarosa, incluidos los mismos marcadores de tamaño. Los geles se observaron con luz ultravioleta tras tinción con bromuro de etidio.
Tabla 1. Secuencia de los cebadores utilizados en las reacciones en cadena de la polimerasa y las condiciones de amplificación
| Diana de amplificación | Secuencia de los cebadores (5′→3′) | Condiciones de amplificación | Tamaño esperado del amplificado (pares de bases) |
| Gen gyrB | MTUBF: TCGGACGCGTATGCGATATC | Un ciclo (5min a 94°C), 35 ciclos (1min a 95°C, 1min a 60°C y 90 s a 72°C) | 1.020 |
| MTUBR: ACATACAGTTCGGACTTGCG | |||
| RD9 | RD9FF: GTGTAGGTAAGCCCCATCC | Un ciclo (5min a 95°C), 40 ciclos (30 s a 94°C, 1min a 65°C y 10min a 72°C) | 306 o 206 |
| RD9FR: GCTACCCTCGACCAAGTGTT | |||
| RD9INT: CAATGTTTGTTGCGCTGC | |||
| RD1 | ET1: AAGCGGTTGCCGCCGACCGACC | Un ciclo (5min a 94°C), 35 ciclos (1min a 95°C, 1min a 60°C y 90 s a 72°C) | 200 o 150 |
| ET2: CTGGCTATATTCCTGGGCCCGG | |||
| ET3: GACGCGATCTGGCGGTTTGGGG | |||
| Gen hsp65 | Tb11: ACCAACGATGGTGTGTCCAT | Un ciclo (5min a 95°C), 35 ciclos (1min a 95°C, 1min a 60°C y 90 s a 72°C) | 440 |
| Tb12: CTTGTCGAACCGCATACCCT | |||
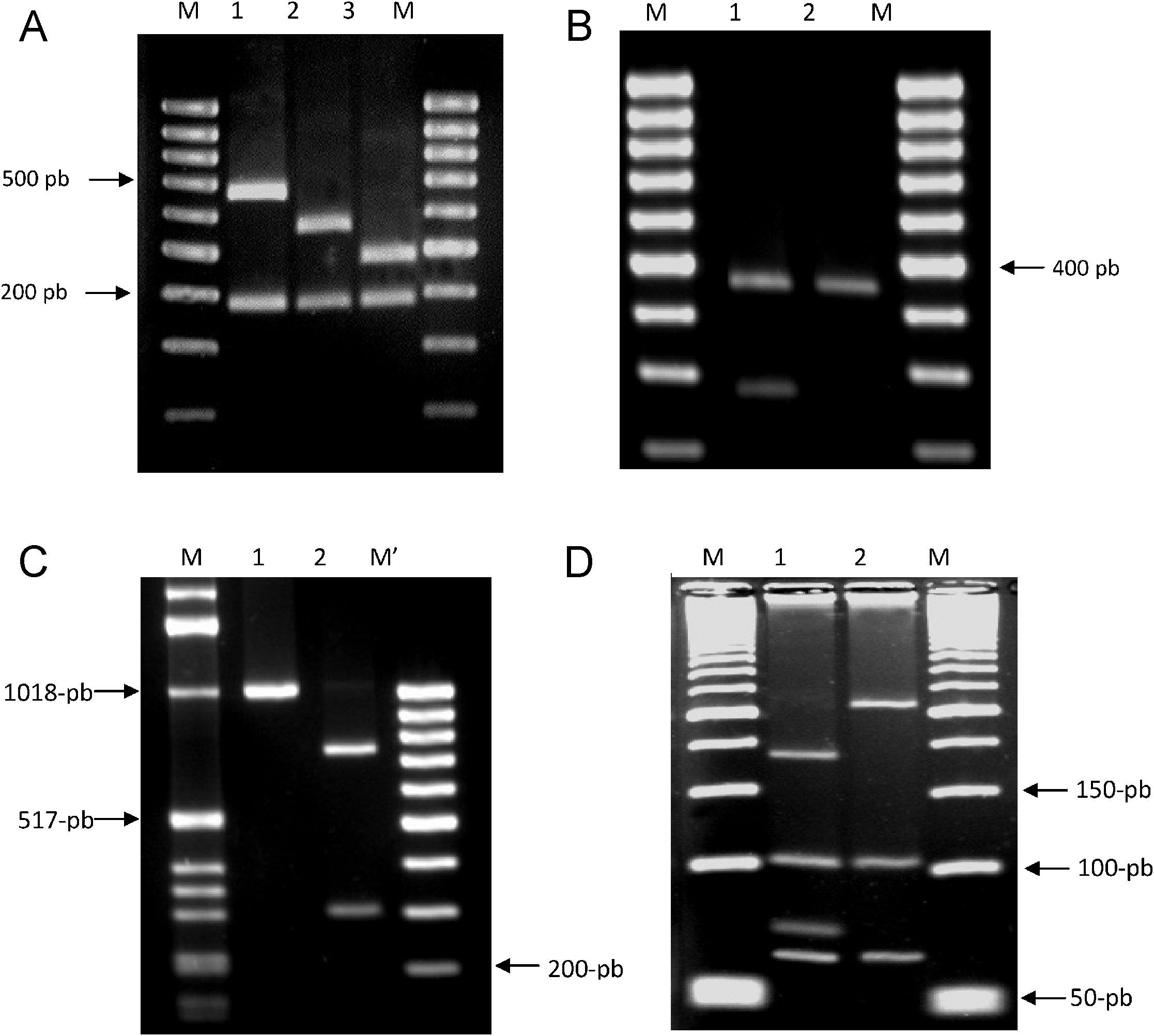
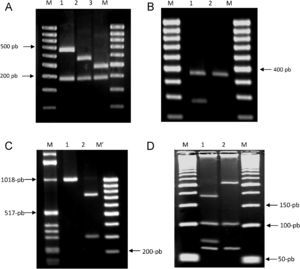
PCR-RFLP (restriction fragment lenght polymorfism ‘polimorfismo del ácido desoxirribonucleico’). La amplificación de un fragmento de 1.020pb del gen gyrB se llevó a cabo mediante PCR mediante la utilización de los cebadores que describieron Kasai et al22. Se digirieron 10μl del producto de amplificación con las enzimas RsaI, TaqI o Sac II (MBI Fermentas, España) según el protocolo que describieron Niemann et al18. El tamaño de los fragmentos obtenidos tras digestión con las enzimas RsaI, TaqI y Sac II se muestra en las figura 2A, 2B y 2C, respectivamente.
Figura 2. Patrones obtenidos al digerir el gen gyrB con las enzimas de restricción Rsa, TaqI y Sac II y el gen hsp65 con la enzima HhaI. 2A) Patrones obtenidos al digerir el gen gyrB con RsaI. Carriles: M: marcador 100pb; 1: Mycobacterium microti; 2: Mycobacterium tuberculosis; 3: Mycobacterium bovis. 2B) Patrones obtenidos al digerir el gen gyrB con la enzima TaqI. Carriles: M: marcador 100pb; 1: Mycobacterium africanum tipo ii ; 2: M. africanum tipo i . 2C) Patrones obtenidos al digerir un fragmento del gen gyrB con SacI. M: marcador 100pb; 1: M. bovis; 2: M. caprae. 2D) Patrones obtenidos al digerir un fragmento del gen hsp65 con HhaI. M: marcador 50pb; 1: M. tuberculosis; 2: M. canettii.
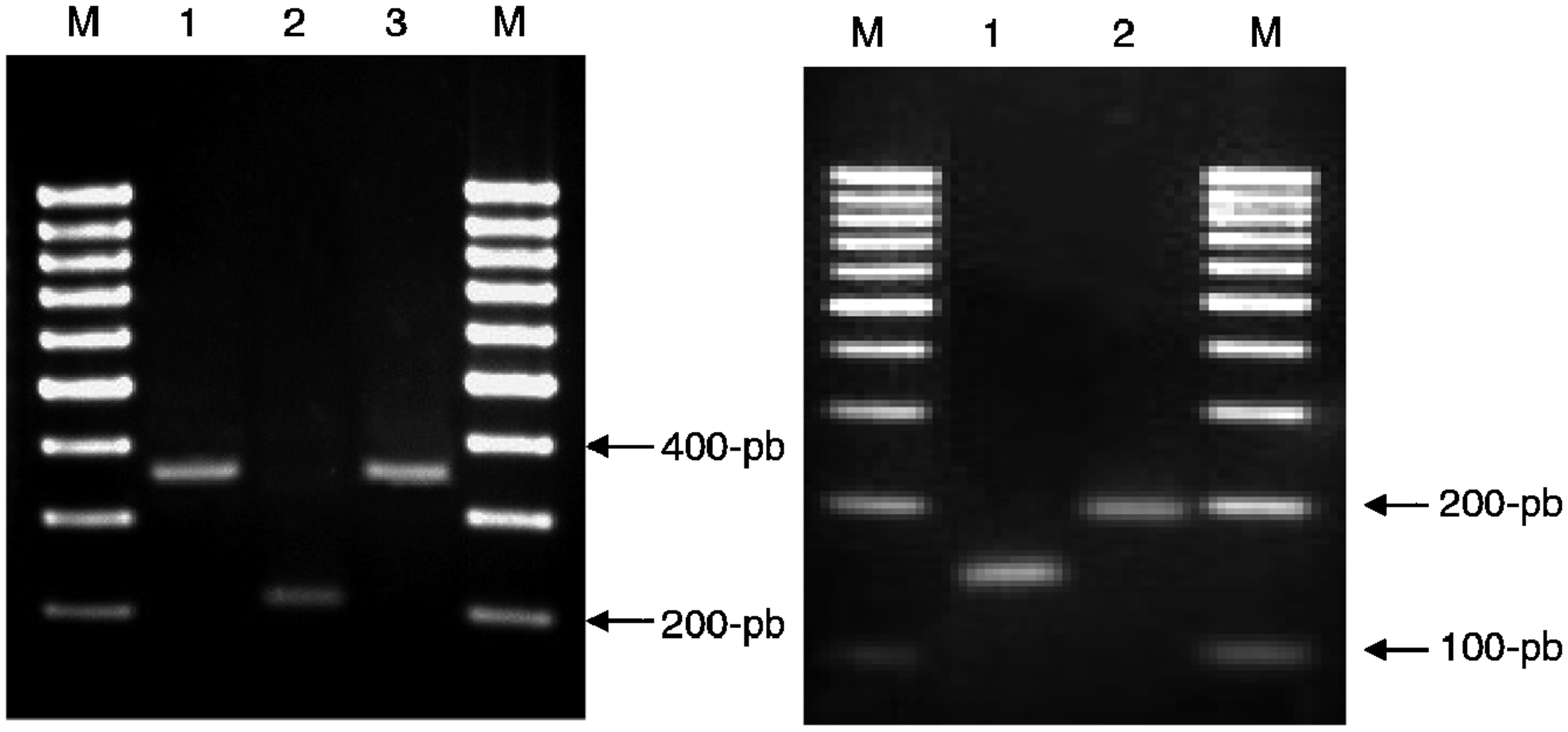
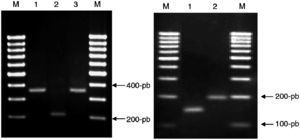
PCR multicebador (RD9). Para detectar la presencia o la ausencia de la región RD9 se siguió el protocolo que describieron Parsons et al13. El tamaño de los fragmentos obtenidos se muestra en la figura 3A. Si la región RD9 está presente, el cebador RD9INT se une en su extremo 3′ y amplifica una región de 306pb. La región que acota la pareja de cebadores RD9FF y RD9FR tendría un tamaño de 2.236pb, por lo que no se produce su amplificación debido a las condiciones de las PCR empleadas. Sin embargo, si la región RD9 se encuentra ausente, la región que acota la pareja de cebadores RD9FF y RD9FR es de 206pb y el cebador RD9INT no se uniría al no estar presente su secuencia homóloga.
Figura 3. Productos de amplificación obtenidos en las PCR múltiples. A) RD9 y B) RD1. 3A) PCR múltiple RD9. Carriles: M: marcador 100pb; 1: Mycobacterium tuberculosis; 2: Mycobacterium africanum tipo II; 3: M. africanum tipo I. 3B) PCR múltiple RD1. Carriles: M: marcador 100pb; 1: Mycobacterium bovis; 2: Mycobacterium bovis-Calmette y Guérin. PCR: Polymerase chain reaction ‘reacción en cadena de la polimerasa’.
PCR multicebador (RD1). Para detectar la posible deleción de la región genómica RD1 se realizó el protocolo que describieron Talbot et al17. El tamaño de los fragmentos se muestra en la figura 3B. De forma similar a RD9, si la región RD1 está presente, el cebador ET2 se une a su extremo 3′ y produce la amplificación de un fragmento de 150pb, ya que la región que acota la pareja de cebadores ET1 y ET2 es de 9.455pb (tamaño no amplificado bajo las condiciones de PCR empleadas). Si la región RD1 está ausente, los cebadores ET1 y ET2 amplifican un fragmento de 200pb.
PCR-RFLP. La amplificación de un fragmento de 441pb entre las posiciones 398 y 836 de la secuencia del gen hsp65 de M. tuberculosis se llevó a cabo mediante PCR con los cebadores que describieron Telenti et al23. Se digirieron 10μl del producto de amplificación con la enzima HhaI (MBI Fermentas, España) según el protocolo que previamente describieron Goh et al19. Los patrones que corresponden a cada especie se muestran en la figura 2D.
Al analizar los resultados obtenidos en la caracterización fenotípica, en 3.966 (94,81%) de las 4.183 cepas recibidas en el laboratorio las colonias fueron rugosas y eugónicas, redujeron los NO3, produjeron niacina y fueron resistentes al TCH, por lo que se identificaron fenotípicamente como M. tuberculosis. Los resultados de las características morfológicas y bioquímicas de las cepas de este estudio se muestran en la tabla 3.
Caracterización molecularEn las 251 cepas elegidas entre las identificadas fenotípicamente en el laboratorio durante el año 2004 se identificaron 130 cepas (51,79%) de M. tuberculosis «variedad clásica», 73 cepas (29,08%) de M. bovis, 20 cepas (7,97%) de M. tuberculosis «variedad asiática», 20 cepas (7,97%) de M. bovis-BCG y 8 cepas (3,19%) de M. africanum. En todos los casos los resultados fenotípicos y genotípicos coincidieron. Los patrones que se obtuvieron según las especies se muestran en las figura 2A, figura 2B, figura 2C, figura 2D, figura 3A y figura 3B.
En las 545 cepas aisladas de pacientes procedentes de África se identificaron 460 cepas (84,4%) de M. tuberculosis, 69 cepas (12,7%) de M. africanum, 14 cepas (2,6%) de M. bovis y 2 cepas (0,4%) de M. caprae.
En las 35 cepas aisladas de pacientes procedentes de Asia se identificaron 25 cepas (71,4%) de M. tuberculosis «variedad clásica» y 10 cepas (28,6%) de «variedad asiática».
En relación con las 122 cepas aisladas de orina, se identificaron 93 cepas (76,2%) de M. tuberculosis, 23 cepas (18,8%) de M. bovis-BCG, 4 cepas (3,3%) de M. bovis y 2 cepas (1,6%) de M. africanum.
La distribución de estas cepas por especies junto con la distribución de aquellas que no presentaban las características propias de M. tuberculosis «variedad clásica» se muestran en la tabla 2.
Tabla 2. Distribución por especies de las cepas del grupo del complejo Mycobacterium tuberculosis al aplicar estudios moleculares
| Especie | n de cepas |
| Pacientes africanos | |
| Mycobacterium tuberculosis | 460 |
| Mycobacterium africanum tipo I | 57 |
| M. africanum tipo II | 12 |
| Mycobacterium bovis | 14 |
| Mycobacterium caprae | 2 |
| Pacientes asiáticos | |
| M. tuberculosis | 25 |
| M. tuberculosis «variedad asiática» | 10 |
| Cepas aisladas de orinas | |
| M. tuberculosis | 93 |
| Mycobacterium bovis- Calmette y Guérin | 23 |
| M. bovis | 4 |
| M. africanum tipo I | 2 |
| Cepas sin M. tuberculosis | |
| M. bovis | 60 |
| M. caprae | 20 |
| M. tuberculosis Nitratos negativos | 6 |
| M. bovis-Calmette y Guérin | 4 |
| M. africanum tipo I | 3 |
| M. tuberculosis «variedad asiática» | 2 |
No se identificó ninguna cepa como M. microti, M. pinnipedii o M. canettii.
Mayoritariamente las cepas procedían de muestras clínicas de pacientes, excepto 7 cepas de origen animal y una cepa obtenida de un control de esterilidad de un gastroscopio, que se identificó como M. tuberculosis. De las 7 cepas de origen animal, 3 se identificaron como M. bovis (todas éstas procedían de ganado bovino) y 4 cepas se identificaron como M. caprae (3 cepas aisladas de animales salvajes y una cepa aislada de cabra).
DiscusiónLa identificación de las especies que forman parte del CMTB ha estado clásicamente basada en el análisis de un número muy limitado de métodos fenotípicos. Estos métodos tienen una serie de inconvenientes: son lentos, al ser necesario el crecimiento de la bacteria (que para estas especies es de al menos 15 días), son de lectura subjetiva y pueden llevar a errores en la identificación11,13 basándose en las características de estas bacterias. La M. tuberculosis posee una nitrato reductasa muy activa, pero en este estudio un número muy pequeño de cepas (18) carecía de esta enzima. Doce de estas 18 cepas se identificaron como M. tuberculosis «variedad asiática» al ser sensibles al TCH12. También se ha demostrado la resistencia cruzada entre el TCH y la isoniacida, por lo que las cepas de M. africanum tipo I y las cepas de M. bovis resistentes a isoniacida lo son también a TCH24. En este trabajo se pone de manifiesto la variabilidad fenotípica intraespecífica en las cepas de CMTB (tabla 3), por lo que se considera que el estudio exclusivo de estas características no es un buen método para la identificación de la especie y se debe complementar con la aplicación de técnicas moleculares basadas en el estudio de aquellos genes o de aquellas regiones del genoma en los que hayan diferencias interespecíficas.
Tabla 3. Características fenotípicas de las cepas estudiadas
| Especie | cepas, n | Morfología colonias | Aspecto cultivo | Reducción nitratos | Producción niacina | Crecimiento 10μg/ml TCH | Crecimiento 50μg/ml PZ |
| Mycobacterium tuberculosis | 3.966 | Rugosas | Eugónico | + | + | + | Variable |
| M. tuberculosis | 6 | Rugosas | Eugónico | − | + | + | − |
| M. tuberculosisa | 12 | Rugosas | Disgónico | − | + | − | − |
| Mycobacterium africanum tipo I | 59 | Rugosas | Disgónico | − | − | − | − |
| M. africanum tipo I b | 3 | Rugosas | Disgónico | − | − | + | − |
| M. africanum tipo II | 8 | Rugosas | Disgónico | + | + | + | − |
| M. africanum tipo II | 4 | Rugosas | Disgónico | + | − | − | − |
| Mycobacterium bovis | 76 | Lisas | Disgónico | − | − | − | + |
| M. bovisb | 2 | Lisas | Disgónico | − | − | + | + |
| Mycobacterium bovis-Calmette y Guérin | 20 | Rugosas | Eugónico | + | − | − | + |
| M. bovis-Calmette y Guérin | 7 | Rugosas | Eugónico | − | − | − | + |
| Mycobacterium caprae | 20 | Lisas | Disgónico | − | − | − | − |
PZ: pirazinamida; TCH: ácido 2-tiofencarboxílico.
a Diez pacientes procedían de Asia.
b Estas cepas fueron resistentes a isoniacida.
Las especies que forman parte de este complejo están estrechamente relacionadas genéticamente. Poseen una homología superior al 95% en la hibridación ADN-ADN, tienen idénticas secuencias en los genes rpoB, 16S ácido ribonucleico ribosómico y en el espacio intergénico de los genes 16S y 23S ácido ribonucleico ribosómico y sólo la subespecie M. canettii difiere en el gen hsp65. Tienen el mismo perfil lipídico, excepto M. bovis-BCG, que presenta pequeñas diferencias con el resto de las especies. En base a estos datos, Van Soolingen et al indican que las especies reconocidas actualmente dentro del CMTB deberían considerarse taxonómicamente como subespecies de una sola especie. Las diferencias básicas entre éstas radican en el huésped, su origen geográfico y su patogenidad4.
El esquema de identificación que se propone reúne las técnicas que aportan otros trabajos previamente publicados13,17,18,19, pero interrelaciona unas con otras para llegar a la identificación individual de todas las especies del CMTB. En el esquema que propusieron Parsons et al13, se llega a la identificación de M. bovis, M. bovis-BCG, M. africanum y M. tuberculosis, pero no se diferencia entre subtipo I y II de M. africanum ni se diferencia al resto de las especies del complejo, mientras que en el esquema de Niemann et al18 se llega a la diferenciación específica de M. africanum tipo i , M. bovis, M. bovis-BCG y M. microti, pero no diferencia M. africanum tipo II de M. tuberculosis ni identifica M. canettii. Por su parte, Goh et al19 y Talbot et al17 desarrollaron un método específico para la diferenciación de M. canettii y M. bovis-BCG, respectivamente. Con el esquema que se propone en este trabajo se llega a la identificación de todas las especies que componen el CMTB, excepto M. pinnipedii que, según los resultados de las pruebas moleculares estudiadas, sería indistinguible de M. africanum de tipo i 14,25. Actualmente, M. pinnipedii no ha producido tuberculosis en humanos, sólo se ha aislado de diferentes especies animales (principalmente de pinnípedos)26, por lo que la imposibilidad de identificar esta especie no sería un problema diagnóstico. Además, ambas especies pueden diferenciarse fenotípicamente, ya que M. pinnipedii, a diferencia de M. africanum, tiene preferencia por el piruvato14.
Un diagnóstico diferencial de la especie es importante, ya que implica la administración de una adecuada pauta de tratamiento. Todas las cepas de M. bovis son resistentes de forma intrínseca a pirazinamida debido a un cambio del nucleótido guanina por citosina en la posición 169 que origina la sustitución del aminoácido histidina por ácido aspártico, lo que produce la inactivación de la pirazinamidasa (enzima necesaria para convertir la pirazinamida en el principio activo eficaz sobre el bacilo tuberculoso)27. De los 4.183 casos de tuberculosis estudiados, se identificaron como M. bovis 3 cepas de origen bovino y 75 cepas de origen humano que fueron la causa del 1,8% de los casos de tuberculosis en los que fue necesario un cambio en el tratamiento para evitar la aparición de nuevas resistencias por un procedimiento inadecuado. No se apreciaron diferencias estadísticamente significativas en el porcentaje de casos de tuberculosis que causó esta especie durante los 3 años analizados, 23 casos en 2005, 29 casos en 2006 y 26 casos en 2007. Se identificaron 20 cepas como M. caprae, 3 cepas se aislaron a partir de muestras de animales salvajes, una cepa a partir de muestra de cabra y el resto de las cepas a partir de muestras de pacientes de diversas comunidades autónomas, mayoritariamente con profesiones relacionadas con el tratamiento de este tipo de animales.
M. bovis-BCG se utiliza como complemento para potenciar la actividad inmunógena en el tratamiento de algunas enfermedades por su alto poder antigénico. En estos casos, un diagnóstico de la especie es fundamental para conocer si el aislamiento es M. bovis-BCG o si se trata de una infección que causa otra especie del grupo CMTB; en este caso hay que administrar un tratamiento antituberculoso urgente. De las 27 cepas identificadas como M. bovis-BCG, 23 fueron aislamientos obtenidos a partir de orina tras la aplicación de un tratamiento inmunógeno sin valoración clínica. La aplicación del esquema molecular permitió diferenciar esta especie de otras especies causantes de una tuberculosis renal (93 cepas de M. tuberculosis, 2 cepas de M. africanum y 4 cepas de M. bovis). Se identificó M. bovis-BCG en 3 cepas de lesiones cutáneas producidas en el lugar de la vacunación con afectación ganglionar, en las que fue necesario aplicar tratamiento; este dato confirma las complicaciones posvacunales ya descritas en la literatura médica28. También se aisló M. bovis-BCG en una muestra respiratoria de un paciente que recibió tratamiento inmunógeno vesical, en el que se había producido una diseminación de la micobacteria por su estado de inmunodepresión (caso poco habitual pero no único)29.
La diferenciación de las especies de este complejo también es una herramienta útil para un mejor conocimiento de la epidemiología de la enfermedad. Según el Sistema de Información Microbiológica, M. tuberculosis fue el agente causal de la mayoría de los casos de tuberculosis diagnosticados en España entre los años 2005 y 2006 (99,6%) (URL: http://www.isciii.es/htdocs/centros/epidemiologia/informacion_microbiologica/sim2006.pdf )30. De las 12 cepas identificadas como M. tuberculosis «variedad asiática», 10 se aislaron de pacientes de ese origen geográfico y sólo en 2 casos los pacientes fueron españoles. Asimismo, se identificaron 74 cepas como M. africanum; todas las cepas, excepto 3, se aislaron de pacientes inmigrantes procedentes de África, mayoritariamente África subsahariana. Se ha observado un aumento paulatino en el número de aislamientos durante estos 3 años, lo que coincide con el aumento de la inmigración procedente de países en los que M. africanum es causante de un número muy elevado de casos de tuberculosis. La nacionalidad de los enfermos con tuberculosis que causa M. africanum y M. tuberculosis «variedad asiática» confirma la relación entre la especie y la procedencia geográfica, pero cada vez es más frecuente aislar estas especies de pacientes autóctonos que nunca han visitado esos continentes debido a los movimientos migratorios y a la integración de estas personas en la sociedad.
Este esquema basado en el estudio genotípico de las cepas del CMTB facilita y a su vez complementa el estudio fenotípico. Los resultados han demostrado que una combinación de ambos métodos proporciona una sensibilidad y una especificidad del 100% en la identificación de la especie de todas las cepas incluidas en este complejo, por lo que se propone como método idóneo para adscribir una cepa productora de tuberculosis, tanto en humanos como en animales, a una especie o a una subespecie concreta de este complejo.
Recibido 14 Mayo 2008
Aceptado 7 Enero 2009
Autor para correspondencia. lherrera@isciii.es