A diferencia de otras áreas del laboratorio, la automatización del laboratorio de microbiología no ha sido fácil, sin embargo es una necesidad clínica para mejorar el manejo de pacientes críticos. Para lograr este objetivo, es necesario optimizar la productividad y flujos de trabajo, partiendo desde el pre-analítico, pasando por los diferentes test de identificación y susceptibilidad, hasta el post-analítico, que en gran medida se ha automatizado a través de la informatización de los resultados. Se muestra en este documento, cuáles son algunos avances en las diferentes áreas, respecto de las metodologías tradicionales, proponiendo esquemas de trabajo para identificación precoz mediante metodología MALDI-TOF, así como elementos de juicio para realizar un correcto informe de antibiograma. La implementación de metodologías rápidas y automatizadas en el laboratorio de microbiología implica una adaptación del personal técnico y profesional, interpretando correctamente los resultados obtenidos, tanto en identificación como en estudios de susceptibilidad. También constituye una exigencia para los administradores, que deben hacer las inversiones necesarias y modificaciones en la planta física y humana, para mantener el servicio en una base de 24/7, aprovechando al máximo las ventajas ofrecidas por la automatización de los procesos.
Unlike other areas of the laboratory, microbiology laboratory automation has not been easy, but it is a clinical need to improve the management of critically ill patients. To achieve this goal, it is necessary to optimize productivity and workflow, starting from the pre-analytical, through different identification and susceptibility tests, to the post-analytical, which has been largely automated through computerized results. It is shown in this paper, some of the advances in the different steps, for traditional methodologies, proposing schemes of work for early identification by MALDI-TOF methodology as well as criteria for correct susceptibility report. The implementation of rapid methodologies and automated microbiology laboratory equipment in turn leads to the need for an adaptation of technical and professional staff, correctly interpreting the results obtained, both for identification and susceptibility studies. It is also a requirement for managers who must make the necessary investments and changes, to maintain service on a 24/7 basis, maximizing the advantages offered by automation.
La microbiología ha sido tradicionalmente una disciplina manual. A diferencia de otras áreas del laboratorio, la automatización de ésta no ha sido fácil, principalmente debido a la gran variabilidad en el tipo de muestras, diversidad y número de microorganismos a identificar y un volumen relativamente menor de exámenes (en comparación con el número de exámenes químicos y hematológicos), que hace menos rentable la incorporación de nuevas tecnologías1,2.
Sin embargo y a pesar de estas consideraciones, hay evidencia científica creciente que demuestra que el pronóstico de un paciente crítico infectado, depende del inicio precoz con el antimicrobiano adecuado, para lo cual es fundamental que el laboratorio sea capaz de proveer identificación microbiana confiable y oportuna, así como reportes de susceptibilidad estandarizados y reproducibles3,4.
Es así que durante muchos años, la identificación de microorganismos ha dependido de la producción local de medios de cultivos para siembra y realización de pruebas bioquímicas, con la consiguiente menor estandarización de estas pruebas. Asimismo, el antibiograma ha sido realizado clásicamente por técnicas de difusión manual, que si bien cada vez se han estandarizado mejor y están en la actualidad adecuadamente validados, es una técnica laboriosa y que requiere de más tiempo para obtener resultados que las técnicas automatizadas disponibles actualmente.
Para lograr el objetivo de la automatización en microbiología, es necesario optimizar la productividad y flujos de trabajo, partiendo desde el pre-analítico, pasando por los diferentes test de identificación y susceptibilidad, hasta el pos analítico, que en gran medida se ha automatizado a través de la informatización de los resultados1,5.
En el pre analítico se han desarrollado equipos que de manera robotizada, logran sembrar muestras en formato líquido, sobre diversas placas para luego ser incubadas y analizadas6.
En cuanto a la identificación bacteriana y los estudios de susceptibilidad, se ha desarrollado un gran número de técnicas rápidas, semiautomatizadas o automatizadas. Estos equipos aportan estandarización y velocidad, pero no han logrado resolver toda la problemática que el estudio microbiológico plantea, por lo que aún es necesario complementar su uso con pruebas manuales. En relación a la identificación, la incorporación del análisis proteómico de las especies bacterianas o fúngicas aisladas, ha cambiado el paradigma microbiológico en los últimos años5,7–9.
Si bien el laboratorio de microbiología involucra además técnicas de diagnóstico inmunológico (tanto para conocer el estado inmune de un paciente, como determinar si hubo contacto reciente o antiguo con algún patógeno específico), técnicas rápidas para detectar presencia de antígenos virales o bacterianos en diversas secreciones o muestras, y técnicas de biología molecular, que permiten detectar secuencias de ácidos nucleicos propias de cada patógeno, así como factores de virulencia específicos, en el presente documento no se abordará estas metodologías, que sin duda también han sido objeto de grandes evoluciones hacia la automatización, circunscribiendo la discusión solamente al ámbito de identificación y estudio de susceptibilidad de microorganismos bacterianos y fúngicos por métodos fenotípicos.
AUTOMATIZACIÓN DEL PRE-ANALÍTICOLa inoculación automatizada de las diferentes muestras clínicas en la superficie de medios de cultivo que permitan el desarrollo de microorganismos aeróbicos, facultativos, fastidiosos y anaeróbicos, ha sido un punto largamente anhelado con el fin de lograr optimizar los flujos de trabajo.
Existe hoy en el mercado diferentes equipos que abordan esta necesidad. Todos ellos intentan resolver problemas de calidad y estandarización de la estría o inóculo, contaminación cruzada, tiempo de procesamiento y costos. Al lograr una siembra con colonias bien aisladas, se reduce la necesidad de hacer traspasos y subcultivos, con el consiguiente ahorro en tiempo y reactivos. En términos generales, todos estos equipos utilizan muestras líquidas o en medio de transporte líquido. En Tabla 1, se muestra una comparación somera de los principales equipos inoculadores disponibles en el mercado y sus características1,10.
AUTOMATIZACIÓN DEL ANALÍTICO: IDENTIFICACIÓN Y ESTUDIOS DE SUSCEPTIBILIDAD A ANTIMICROBIANOSEl ideal actual consiste en la “automatización total” del laboratorio de microbiología, lo que comprende equipos en línea para siembra, incubación, análisis remoto de colonias desarrolladas mediante digitalización de imágenes y posterior identificación mediante espectometría de masas (MALDI-TOF; Matrix Assisted Laser Desorption Ionization -Time of Flight) con realización de antibiograma automatizado a las colonias así identificadas. Todos estos equipos en línea, estarían interconectados mediante un software que además comunica con equipos de hemocultivo automatizado, tinción de Gram y la realización de técnicas serológicas.
Si bien el ideal de automatización involucra el proceso completo del laboratorio de microbiología, la automatización total es un concepto aún lejano en la realidad de la mayor parte de los laboratorios. Lo que sí es posible en la actualidad, es contar con metodologías de identificación más rápida que la convencional.
IDENTIFICACIÓNLos procedimientos convencionales de identificación de microorganismos consisten en observación de características físicas y tintoriales (morfología de colonias, tinción de Gram) y de reacciones bioquímicas. Inicialmente, estas pruebas bioquímicas se realizaban en medios en tubos, los que con el tiempo se miniaturizaron en formatos que permitieron aumentar el número de pruebas y eventualmente automatizar la lectura de estos resultados. Dependiendo de las reacciones de cada microorganismo frente a diferentes sustratos, se obtiene un perfil bioquímico, que al ser comparados con perfiles conocidos, permite la identificación5,8,9.
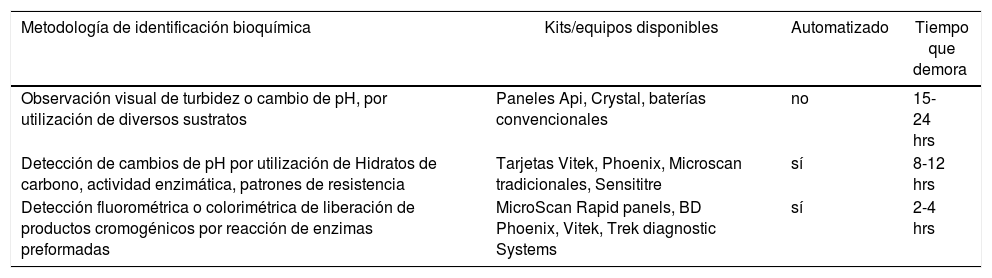
El tiempo de la identificación convencional era de 24-48hrs para permitir la expresión de la reacción, ya que debía lograrse un número crítico de bacterias, que dependen de su velocidad de replicación. Con la automatización se logró reducir este tiempo a 8-10 horas, ya sea porque se detecta reacción con sustratos preformados, o se logra visualizar la reacción bioquímica con un menor número de replicaciones, dada la miniaturización de la reacción. Esta reducción en los tiempos exige la necesidad a contar con personal capacitado durante las 24hrs en el laboratorio, para optimizar y hacer más eficientes los procesos, aprovechando así las ventajas que ofrece esta tecnología más rápida. En Tabla 2 se presentan los diferentes tipos de equipos y kits disponibles para identificación bioquímica11.
Sistemas para identificación mediante pruebas bioquímicas
| Metodología de identificación bioquímica | Kits/equipos disponibles | Automatizado | Tiempo que demora |
|---|---|---|---|
| Observación visual de turbidez o cambio de pH, por utilización de diversos sustratos | Paneles Api, Crystal, baterías convencionales | no | 15-24 hrs |
| Detección de cambios de pH por utilización de Hidratos de carbono, actividad enzimática, patrones de resistencia | Tarjetas Vitek, Phoenix, Microscan tradicionales, Sensititre | sí | 8-12 hrs |
| Detección fluorométrica o colorimétrica de liberación de productos cromogénicos por reacción de enzimas preformadas | MicroScan Rapid panels, BD Phoenix, Vitek, Trek diagnostic Systems | sí | 2-4 hrs |
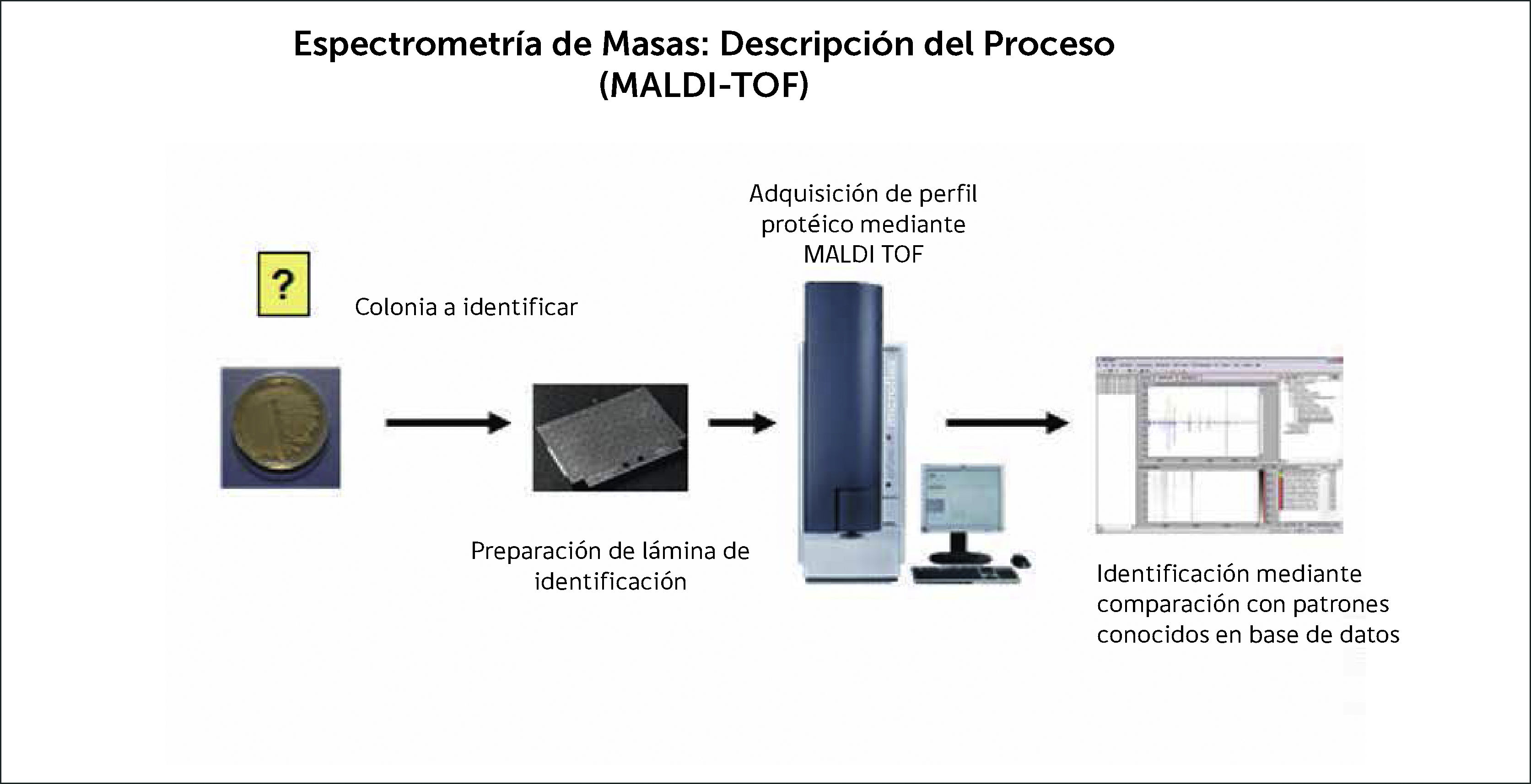
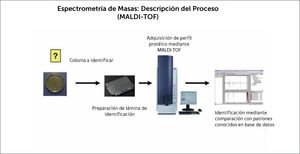
Durante los últimos años, cada vez más laboratorios están implementando la tecnología de espectometría de masas MALDI-TOF, que mediante el análisis proteómico de cepas bacterianas y fúngicas, permite su identificación en minutos. La Figura 1 muestra un esquema de esta tecnología y sus principales etapas. Existen diversos equipos disponibles en el mercado, siendo los más representativos: MALDI-Biotyper de Bruker y Vitek MS, de BioMerieux, ambos presentan fortalezas y debilidades.
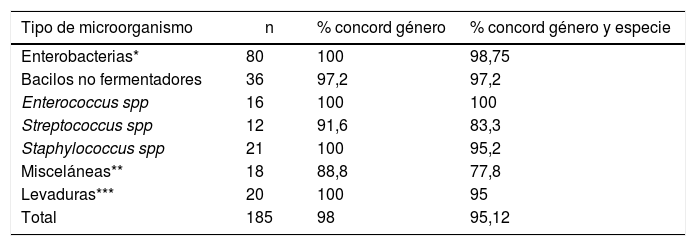
En nuestro centro, realizamos una verificación de la tecnología MALDI-TOF, con el objetivo de conocer la concordancia en identificación de género y de especie, entre metodología convencional y espectometría de masas, comparando los resultados obtenidos por Vitek 2 Compact y Vitek MS en 205 cepas, obteniendo una concordancia global de 95% para género y especie y 98% para género solamente. Cabe destacar que la identificación de enteropatógenos, no es satisfactoria por esta metodología, ya que no permite diferenciar cepas de E. coli de Shigella, ni tipificar las cepas de Salmonella spp. En Tabla N° 3 se muestra los resultados por grupo de microorganismo. La probabilidad de identificación de una cepa bacteriana o fúngica por espectometría de masas, depende de que el perfil proteómico de esa cepa esté incluido en la biblioteca de perfiles del equipo, es así que las especies “misceláneas” de la tabla, tienen un menor grado de identificación, posiblemente por ser cepas aisladas con menor frecuencia y por ende, menos representadas en esta biblioteca o base de datos12.
Rendimiento identificación por metodología MALDI-TOF vs técnicas convencionales
| Tipo de microorganismo | n | % concord género | % concord género y especie |
|---|---|---|---|
| Enterobacterias* | 80 | 100 | 98,75 |
| Bacilos no fermentadores | 36 | 97,2 | 97,2 |
| Enterococcus spp | 16 | 100 | 100 |
| Streptococcus spp | 12 | 91,6 | 83,3 |
| Staphylococcus spp | 21 | 100 | 95,2 |
| Misceláneas** | 18 | 88,8 | 77,8 |
| Levaduras*** | 20 | 100 | 95 |
| Total | 185 | 98 | 95,12 |
(Comparado Vitek MS vs Vitek 2).
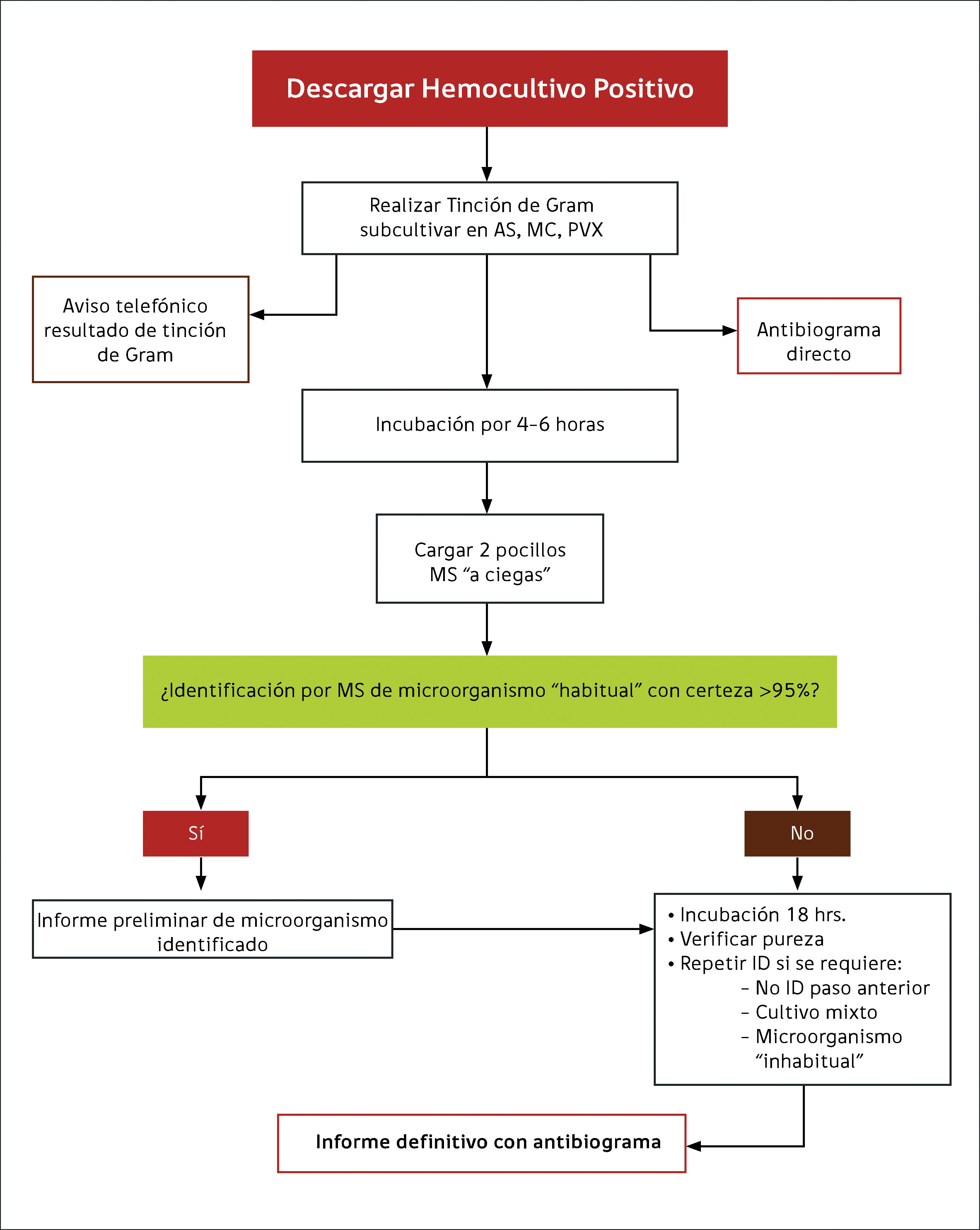
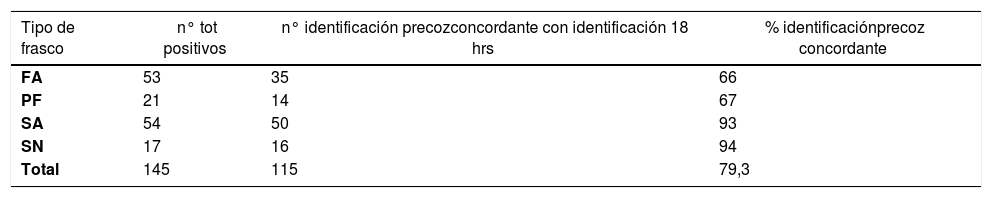
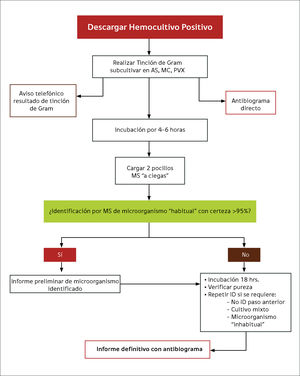
El análisis proteómico de microorganismos por espectrometría de masas (MS) es una metodología confiable, económica y rápida, que muestra resultados promisorios al compararlo con métodos convencionales (MC). Se han publicado reportes que indican que MS puede ser utilizado con buenos resultados en la identificación a partir de muestra directa, con protocolos publicados para urocultivo y hemocultivo, destacando el aporte que esto significa en el manejo clínico. Sin embargo, la aplicación de los protocolos publicados para hemocultivo, resulta laboriosa e interrumpe el flujo normal de trabajo de un laboratorio, ya que en el momento en que un hemocultivo da alarma de positivo, debe haber un profesional disponible para realizar la secuencia completa de concentración-centrifugación y resuspensión de pellet que indica el procedimiento, lo que no siempre existe en la práctica diaria. En nuestro centro aplicamos este protocolo el año 2012 en 39 muestras de hemocultivo, logrando un 74% de diagnóstico directo, que se veía fuertemente reducido si el frasco de hemocultivo tenía resina/carbón activado. Dado que este resultado no fue satisfactorio, se mantuvo el método convencional de identificación (incubación por 18 horas en placas y luego identificación de colonias). Durante el primer semestre de 2014, establecimos un algoritmo acortado del método tradicional, realizando una identificación a ciegas a partir de placas incubadas por 4 a 6 horas (sin colonias visibles), para conocer la correlación del diagnóstico adelantado a ciegas por MS en hemocultivos positivos inoculados en placas de agar sangre, comparado con método convencional (MC), según tipo de frasco y tipo de microorganismo. Se incluyó en el estudio 145 muestras de hemocultivo positivo detectados por equipo Bact /Alert. Los resultados de ambos procesos fueron comparados en relación a resultado de identificación. Se toma en cuenta el tipo de frasco y tipo de microorganismo detectado. Los resultados se muestran en Tablas 4 y 5, evidenciando que el estudio de hemocultivos positivos en forma directa por MS, sembrando a ciegas a las 4 a 6 horas de inoculación en placas, permite identificar en forma segura el 81% de los casos, adelantando el dg en 12-16 horas respecto del método tradicional. Este resultado mejora cuando se utilizan frascos sin resina o carbón activado (dg correcto en 93% de los casos), y se observa mayor precocidad para diagnosticar bacilos Gram negativo que cocáceas Gram positivo. En base a estos resultados, se estableció un algoritmo que se muestra en Figura 2, que permite adelantar la ID sin aumentar los costos ni la carga de trabajo del laboratorio de microbiología13,14.
Rendimiento de Identificación precoz, según tipo de frasco hemocultivo positivo, por Vitek MS
| Tipo de frasco | n° tot positivos | n° identificación precozconcordante con identificación 18 hrs | % identificaciónprecoz concordante |
|---|---|---|---|
| FA | 53 | 35 | 66 |
| PF | 21 | 14 | 67 |
| SA | 54 | 50 | 93 |
| SN | 17 | 16 | 94 |
| Total | 145 | 115 | 79,3 |
FA y PF: frascos con carbón activado. SA, SN: frascos sin carbón activado.
Rendimiento identificación precoz según tipo de microorganismo, hemocultivo positivo por Vitek MS
| Tipo de Microorganismo | n° totalidentificados | n° concordantes por identificación precoz vs identificación 18 hrs | % identificaciónprecoz concordante |
|---|---|---|---|
| BNEG | 72 | 62 | 86 |
| CPOS | 62 | 49 | 79 |
| LEVAD | 7 | 4 | 57 |
| BPOS | 2 | 0 | 0 |
| mixto | 2 | 0 | 0 |
| total | 145 | 115 | 79,3 |
BNEG: bacilo gram negativo; CPOS:cocácea gram positivo; LEVAD:levadura;BPOS:bacilo gram positivo. Mixto: desarrollo de mas de un microorganismo.
En cuanto a estudios de susceptibilidad a antimicrobianos, existen diferentes metodologías, que se pueden sistematizar en: convencionales o fenotípicos, y automatizados ya sea fenotípicos o genotípicos. Cada vez más, se hace necesario detectar de manera oportuna y precisa, la existencia de mecanismos de resistencia con implicancia epidemiológica, como son: Enterococcus resistentes a vancomicina, S. aureus con sensibilidad reducida a vancomicina, enterobacterias productoras de Betalactamasas de Espectro Extendido (BLEE) o productoras de carbapenemasas. Estos mecanismos de resistencia pueden ser detectados por técnicas rápidas, de screening, o confirmatorias, ya sea fenotípicas o genotípicas, que deben estar disponibles para realizar frente a una alerta en la interpretación del antibiograma (generalmente estas alertas están dadas por la identificación de alguna especie en particular, o la detección de concentraciones inhibitorias mínimas (CIM) sugerentes).15–18.
Los métodos convencionales o fenotípicos, a su vez pueden ser:
- a)
Cuantitativos: permiten conocer la CIM de un antimicrobiano frente a un determinado microorganismo (en ug/ml), lo que es de gran utilidad al momento de definir las dosificaciones, especialmente en pacientes críticos. Existen métodos cuantitativos de referencia o Gold standard (ya sea micro o macro dilución en caldo y/o dilución en agar) y aquellos que no son de referencia (E-test, Just-One), pero que facilitan de manera importante la realización de antibiograma por CIM en la práctica diaria a la vez que da flexibilidad respecto de a qué antimicrobianos estudiar la CIM y a cuáles no.
- b)
Cualitativos (difusión en disco o Kirby Bauer), que a través de la medición de un halo en mm permite correlacionar con la CIM y entregar una categoría de Susceptible, Intermedio o Resistente frente a cada antimicrobiano.
- c)
De screening y punto de corte, permiten separar cepas con algún mecanismo de resistencia específico, de aquellas que no lo poseen. Para esto, existen diversos agares cromógenos o de screening, que contienen una concentración de antimicrobiano que constituye el punto de corte para identificar las cepas resistentes.
Los métodos automatizados disponibles son:
- a)
Microdilución rápida (métodos comerciales fenotípicos: Vitek, Phoenix, Microscan). Estos métodos, al igual que la identificación automatizada, consisten en paneles con pocillos miniaturizados que contienen diferentes concentraciones de antimicrobianos, según los puntos de corte que permiten diferenciar cepas resistentes de cepas intermedias o sensibles. Estos paneles o tarjetas contienen antimicrobianos definidos para los distintos grupos de microorganismos (bacilos, cocáceas, levaduras, gram positivo, gram negativo), se ingresan al equipo respectivo, el cual mediante turbidimetría establece las CIM de cada cepa. Actualmente estos equipos cuentan con softwares de interpretación o reglas de experto, para informar resultados coherentes de susceptibilidad. Estos equipos permiten realizar la identificación (ya sea bioquímica o por MALDI) y estudio de susceptibilidad en forma integrada19.
- b)
PCR (métodos comerciales genotípicos), que detectan presencia de genes de resistencia conocidos, o bien,
- c)
La detección de perfiles proteómicos mediante MALDI-TOF post exposición a un determinado antimicrobiano.
Las últimas dos mencionadas, son técnicas en desarrollo o ya comercializadas, que escapan el ámbito de esta revisión20,21.
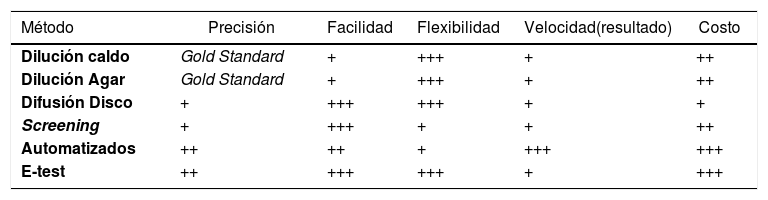
En Tabla N°6 se muestra una comparación de las principales características de las diferentes metodologías fenotípicas mencionadas.
Cuadro comparativo principales características de los métodos de estudio fenotípicos de susceptibilidad a antimicrobianos
| Método | Precisión | Facilidad | Flexibilidad | Velocidad(resultado) | Costo |
|---|---|---|---|---|---|
| Dilución caldo | Gold Standard | + | +++ | + | ++ |
| Dilución Agar | Gold Standard | + | +++ | + | ++ |
| Difusión Disco | + | +++ | +++ | + | + |
| Screening | + | +++ | + | + | ++ |
| Automatizados | ++ | ++ | + | +++ | +++ |
| E-test | ++ | +++ | +++ | + | +++ |
Actualmente persiste un desfase entre la velocidad de identificación que se logra por espectometría de masas, y la menor velocidad en obtener un resultado de susceptibilidad a antimicrobianos, ya que a pesar de su automatización, esta última sigue basada en la velocidad de replicación del microorganismo.
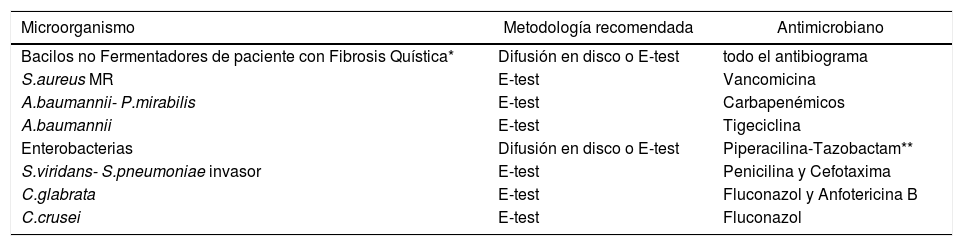
Es necesario considerar también que, pese a los grandes avances logrados en relación a automatización de estudios de susceptibilidad, en este momento aún no es posible prescindir del todo de los métodos manuales, ya que hay algunos microorganismos, o combinaciones de microorganismo/antimicrobiano (Tabla 7) que presentan falsas sensibilidades o resistencias22.
Recomendaciones específicas para realizar estudio de Susceptibilidad por metodología manual
| Microorganismo | Metodología recomendada | Antimicrobiano |
|---|---|---|
| Bacilos no Fermentadores de paciente con Fibrosis Quística* | Difusión en disco o E-test | todo el antibiograma |
| S.aureus MR | E-test | Vancomicina |
| A.baumannii- P.mirabilis | E-test | Carbapenémicos |
| A.baumannii | E-test | Tigeciclina |
| Enterobacterias | Difusión en disco o E-test | Piperacilina-Tazobactam** |
| S.viridans- S.pneumoniae invasor | E-test | Penicilina y Cefotaxima |
| C.glabrata | E-test | Fluconazol y Anfotericina B |
| C.crusei | E-test | Fluconazol |
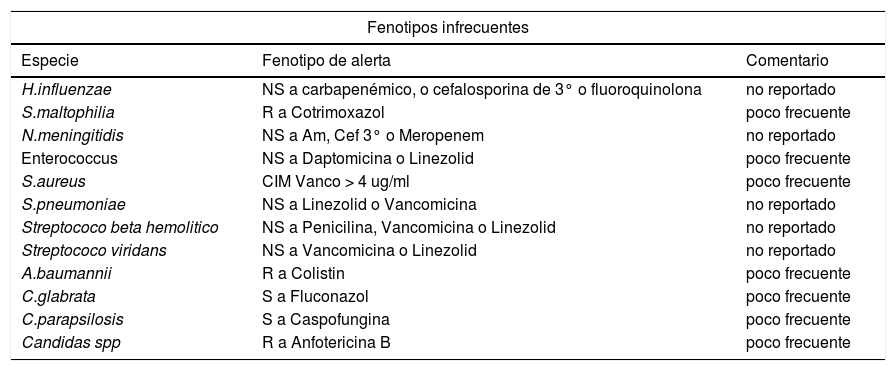
Es fundamental, para una correcta interpretación de los resultados de susceptibilidad, contar con una identificación precisa y confiable, ya que existen microorganismos con resistencias intrínsecas o naturales, que no siempre se evidencian in vitro, por lo que el laboratorio de microbiología debe emitir un informe de susceptibilidad interpretado de acuerdo a la identificación del microorganismo, y de los mecanismos de resistencia subyacentes. También existen fenotipos de resistencia inusuales, que deben hacer sospechar al microbiólogo y repetir el estudio, o intentar confirmarlo por algún otro método. En Tabla 8 se presentan los microorganismos con fenotipos de resistencia intrínseca más frecuentemente encontrados en la práctica clínica y en Tabla 9 aquellos fenotipos de resistencia poco habituales, que deben llevar a realizar más pruebas23.
Resistencia intrínseca a tener en cuenta al momento de informar un antibiograma
| Resistencia Intrínseca | |
|---|---|
| C.krusei | R a Fluconazol |
| Citrobacter freundii | R a ampi, sulbactam-ampicilina, amoxi-clavulánico y cefalosporinas de 1° y 2° |
| Enterobacter aerogenes y cloacae | R a ampi, sulbactam-ampicilina, amoxi-clavulánico y cefalosporinas de 1° y 2° |
| Hafnia alvei | R a ampi, sulbactam-ampicilina, amoxi-clavulánico y cefalosporinas de 1° |
| K.pneumoniae | R a ampicilina |
| Morganella morgannii | R a ampi, amoxi-clavulánico y cefalosporinas de 1° y 2°, tetraciclinas, Nitrofurantoina, Colistin |
| Proteus mirabilis | Tetraciclinas, Nitrofurantoina, Colistin |
| Proteus vulgaris | R a ampi, cefalosporinas de 1° y 2°, tetraciclinas, Nitrofurantoina, Colistin |
| Proteus Providence y Morganella | Pueden presentar CIM elevado a Imipenem por mecanismo de impermeabilidad. |
R: Resistencia.
Table CLSI MIDO S24.
Fenotipos de alerta, implican confirmación o repetición
| Fenotipos infrecuentes | ||
|---|---|---|
| Especie | Fenotipo de alerta | Comentario |
| H.influenzae | NS a carbapenémico, o cefalosporina de 3° o fluoroquinolona | no reportado |
| S.maltophilia | R a Cotrimoxazol | poco frecuente |
| N.meningitidis | NS a Am, Cef 3° o Meropenem | no reportado |
| Enterococcus | NS a Daptomicina o Linezolid | poco frecuente |
| S.aureus | CIM Vanco > 4 ug/ml | poco frecuente |
| S.pneumoniae | NS a Linezolid o Vancomicina | no reportado |
| Streptococo beta hemolitico | NS a Penicilina, Vancomicina o Linezolid | no reportado |
| Streptococo viridans | NS a Vancomicina o Linezolid | no reportado |
| A.baumannii | R a Colistin | poco frecuente |
| C.glabrata | S a Fluconazol | poco frecuente |
| C.parapsilosis | S a Caspofungina | poco frecuente |
| Candidas spp | R a Anfotericina B | poco frecuente |
NS: No sensible.
Asimismo, el laboratorio de microbiología debe proveer a sus clínicos de tablas de susceptibilidad de la microbiota local actualizadas en forma periódica, de manera de orientar adecuadamente los tratamientos empíricos que se inician frente a una sospecha clínica determinada24.
CONCLUSIÓNLa microbiología clínica está cambiando rápidamente en el último tiempo, como respuesta a la evidencia científica de contar con información de manera precoz, mejora el pronóstico de los pacientes, así como la necesidad de las instituciones de hacer sus procesos más eficientes. Esto implica a su vez, la exigencia de una adaptación del personal técnico y profesional a las nuevas tecnologías, interpretando correctamente los resultados obtenidos, tanto en identificación como en estudios de susceptibilidad. También constituye una exigencia para los administradores, que deben hacer las inversiones necesarias y modificaciones en la planta física y de personal, para mantener el servicio en una base de 24/7, aprovechando al máximo las ventajas ofrecidas por la automatización de los procesos.
La autora declara no tener conflictos de interés, en relación a este artículo.