La candidemia es una complicación infecciosa que afecta fundamentalmente a pacientes ingresados en unidades de cuidados intensivos así como en otros servicios hospitalarios, y cuya mortalidad puede alcanzar el 40%. La candidemia es una infección de adquisición típicamente hospitalaria, por lo que la transmisión horizontal de Candida spp. puede traducirse en la aparición de brotes nosocomiales. La caracterización genotípica de los aislamientos de Candida causantes de candidemia puede ayudar a esclarecer el origen de la infección, detectar los servicios hospitalarios con transmisión activa y, consecuentemente, mejorar su prevención. Diversas técnicas de tipificación molecular han sido empleadas para el estudio genotípico de aislamientos de Candida. Las técnicas basadas en microsatélites son reproducibles y presentan un alto poder de discriminación, lo que las convierte en opciones atractivas para el estudio de brotes de candidemia. La mayor parte de los brotes han sido descritos en pacientes ingresados en unidades de cuidados intensivos, fundamentalmente en neonatos. La presente revisión pretende discutir el papel de la caracterización genotípica de aislamientos de Candida causantes de candidemia para el estudio de brotes hospitalarios, así como describir las poblaciones más frecuentemente afectadas por las cepas epidémicas.
Candidemia is an infectious complication mainly affecting hospitalized patients, particularly those admitted to intensive care units. Patient mortality can reach up to 40%. Candidemia is typically nosocomially-acquired, and horizontal transmission of Candida spp. can lead to the presence of outbreaks of candidemia. Genotyping of isolates of Candida causing candidemia can help us to understand the source of the infection, detect the hospital wards with active Candida spp. transmission and, consequently, improve the prevention of the infection. Several genotyping tools have been used for the molecular characterization of Candida isolates involved in outbreaks of candidemia. Genotyping procedures based on microsatellites are reproducible and show a high discriminatory power. Microsatellites are recommended for the study of outbreaks of candidemia. In most hospital outbreaks of candidemia, patients admitted to intensive care units are involved, mostly neonatal patients. The role of genotyping Candida isolates causing candidemia for the study of nosocomial outbreaks of candidemia is reviewed, as well as the patients more commonly affected by epidemic strains.
Candida agrupa a varias especies de levaduras capaces de causar un amplio espectro de infecciones, entre las que destaca la candidiasis invasiva que afecta a pacientes hospitalizados. La candidemia es la forma de candidiasis invasiva más común. Candida ocupa el cuarto lugar en la incidencia de infecciones del torrente circulatorio y las cifras de candidemia oscilan entre 1,7 y 10 episodios por cada 100.000 habitantes66. Aunque se han descrito casos de candidemia causados hasta por 17 especies de Candida, más del 90% de los casos se atribuyen a Candida albicans, Candida parapsilosis, Candida glabrata, Candida tropicalis y Candida krusei65.
C. albicans, microorganismo habitual de la piel y mucosas del ser humano, es la principal causa de candidemia y responsable del 50% de los casos. Gracias a su capacidad para formar biopelículas puede adherirse a catéteres y otros dispositivos médicos implantables; también se ha encontrado en soluciones de nutrición parenteral y en el medio hospitalario66. Sin embargo, se ha constatado un aumento en el número de casos de candidemia causados por otras especies de Candida, aunque con marcadas diferencias geográficas. C. parapsilosis, la segunda especie más frecuente en España y Latinoamérica2,6,43,61,63, puede encontrarse en piel y mucosas. Su alta capacidad de formar biopelículas hace que sea posible encontrarla en catéteres y otros dispositivos implantables, así como persistir en el medio hospitalario; esto la convierte en un patógeno de transmisión hospitalaria por parte del personal sanitario. Su menor capacidad patogénica explica la menor tasa de mortalidad de los pacientes infectados83. Por el contrario, en Estados Unidos y norte de Europa, C. glabrata es la segunda especie en términos de frecuencia, siendo la tercera especie encontrada en España; su incidencia parece estar aumentando83.
C. tropicalis es la segunda especie más frecuentemente aislada en algunas regiones como Brasil o la región Asia-Pacífico. Es causa conocida de infecciones sistémicas en pacientes con cáncer, especialmente aquellos con leucemia, y presenta una mortalidad que oscila entre el 33 y el 90%83. Por último, C. krusei es una especie menos frecuente, que se asocia a pacientes con neutropenia con tumores hematológicos o sólidos. Raramente se asocia la infección con el catéter. Su resistencia intrínseca al fluconazol y el uso amplio de este antifúngico como profilaxis puede explicar el aumento en las infecciones por C. krusei66.
La mayor parte de los casos de candidemia aparecen durante la estancia hospitalaria del paciente67. El carácter nosocomial de esta infección se manifiesta de forma más evidente cuando aparecen brotes hospitalarios, en los que se supone que la transmisión horizontal de Candida spp. juega un papel importante. En este contexto, la caracterización de los aislamientos de Candida responsables de candidemia puede ser útil para el estudio de brotes hospitalarios.
Rutas de adquisición de la candidemiaInfección por cepas endógenasLa presencia de Candida en el tubo gastrointestinal es un factor de riesgo importante para el desarrollo de candidemia en algunos pacientes56. El sobrecrecimiento de Candida en el tubo digestivo tras la exposición prolongada a antibióticos de amplio espectro que reducen la microbiota bacteriana se ha asociado al desarrollo de candidemia23,56. Otro factor de riesgo importante es la traslocación de cepas de Candida desde el intestino al torrente circulatorio, hecho que ocurre en pacientes tras cirugía abdominal o trasplante de hígado, en aquellos con pancreatitis, o en inmunodeprimidos con mucositis58. En todas estas situaciones, la candidemia se desarrolla cuando las cepas endógenas propias del paciente acceden al torrente circulatorio por diversas razones. Por tanto, en este escenario, aunque la candidemia se diagnostique durante el ingreso hospitalario, el papel de la transmisión horizontal tiene un bajo impacto.
Infección por cepas exógenasGracias a la capacidad de Candida para formar biopelículas, esta levadura puede encontrarse en diferentes localizaciones del medio hospitalario como superficies, dispositivos médicos, soluciones de nutrición parenteral, o sustancias de naturaleza plástica como los catéteres intravasculares66. La presencia ubicua de este microorganismo en el hospital lo predispone a acceder al torrente circulatorio de pacientes con roturas de la barrera cutánea, fundamentalmente en portadores de catéteres intravasculares. El catéter es, por tanto, un origen frecuente de candidemia15,58. Hay estudios que demuestran que las manos del personal sanitario se colonizan por Candida; así, la manipulación del catéter por parte de estos trabajadores facilita su colonización y el posterior desarrollo de candidemia42,89,92.
Es conocida la alta incidencia de candidemia por C. parapsilosis en neonatos, sobre todo en recién nacidos prematuros con bajo peso53. Estudios en los que se ha estudiado el papel de la transmisión vertical durante el parto evidencian que tanto la colonización previa del canal vaginal de la madre como la transmisión horizontal de cepas por parte del personal sanitario juegan un papel relevante en estos pacientes3,12,53,84. Por otra parte, la estancia prolongada de pacientes en unidades de cuidados intensivos es un factor de riesgo para el desarrollo de candidemia, ya que la mayoría de estos pacientes presentan factores de riesgo adicionales (tales como ser portadores de catéteres intravasculares) y además son susceptibles de infectarse por cepas exógenas, fundamentalmente de C. parapsilosis88.
Resulta lógico que los brotes hospitalarios de candidemia los causen cepas exógenas adquiridas por los pacientes durante el ingreso hospitalario. La identificación de las fuentes de infección y las posibles rutas de transmisión es importante para el desarrollo de estrategias de prevención y control de esta infección58.
Los brotes de candidemia como problema hospitalarioAunque la candidemia es una enfermedad con elevada mortalidad, su incidencia es baja cuando se compara con otras infecciones como la neumonía o la bacteriemia18,30. Por ello, un incremento en el número de casos de candidemia en relación con los habitualmente observados en una misma unidad, en un determinado periodo de tiempo, no debería pasar desapercibido.
La presencia de brotes nosocomiales de candidemia ha sido ilustrada en diversos estudios7,34,48. Los pacientes involucrados en estos brotes generalmente se encuentran ingresados en unidades de cuidados intensivos, unidades postoperatorias, o unidades de neonatología, documentándose una mayor incidencia en pacientes con edades extremas (>65 años o<1 año)66. Las unidades de cuidados intensivos neonatales son áreas de máximo riesgo para la aparición de brotes de candidemia, causados fundamentalmente por C. albicans o C. parapsilosis.
También se han descrito brotes en las unidades de oncohematología, donde se encuentran pacientes con alto riesgo que son frecuentemente portadores de catéteres intravasculares9,27,70.
En situaciones en las que aparece un brote, sería deseable estudiar tanto los aislamientos infectantes como aquellos aislamientos ambientales recogidos en el entorno de los pacientes, incluso los provenientes de las manos del personal sanitario. Si aquellos aislamientos estudiados perteneciesen a la misma especie de Candida, las técnicas genotípicas serían útiles a la hora de esclarecer la fuente del brote y su posterior erradicación. La tipificación molecular es una herramienta que permite detectar cepas epidémicas, determinar los orígenes de la infección, determinar las rutas de adquisición y transmisión de dichas cepas, monitorizar la emergencia de cepas resistentes de origen clonal, así como conocer la diversidad clonal de la población de cepas estudiada29.
Sin un estudio genotípico de los aislamientos de Candida causantes de candidemia no es posible cuantificar la proporción de casos causada por genotipos epidémicos. En estas circunstancias, la definición de «brote de candidemia» descansaría exclusivamente en la constatación de un aumento en el número de casos en una unidad hospitalaria concreta en un momento dado. Por tanto, las rutas de la infección no pueden ser trazadas.
Técnicas de caracterización genotípica de aislamientos de Candida spp.Las técnicas de tipificación molecular permiten estudiar la relación genética existente entre 2 o más aislamientos de la misma especie de Candida. Por tanto, el paso previo al estudio genotípico es obtener una identificación precisa de la especie.
Se ha demostrado que las técnicas de identificación bioquímicas como el API ID 32C son insuficientes para discriminar entre especies relacionadas como es el caso de C. parapsilosis, Candida orthopsilosis o Candida metapsilosis. Es deseable que se realice previamente una identificación a nivel molecular de las cepas en cuestión. Las regiones internal transcribed spacers (ITS) son utilizadas universalmente para la identificación de hongos. Son regiones no codificantes que separan a las regiones que codifican las subunidades 18S, 5,8S y 26S del ARN ribosómico. Se trata de un conjunto de genes multicopia dispuestos en tándem, lo que facilita su amplificación por medio de parejas de cebadores universales (ITS1 e ITS4) que resulta en un fragmento de 400 a 800 pares de bases. La secuenciación de este fragmento y su posterior comparación con secuencias depositadas en bases como GenBank permiten identificar la especie aislada95.
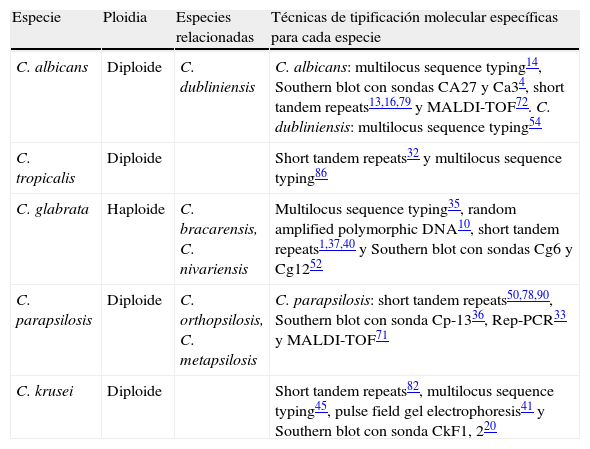
La elección de la técnica genotípica a aplicar dependerá del objetivo del estudio, de la infraestructura del laboratorio y de la experiencia del personal, así como de la especie de Candida a estudiar. Se han desarrollado métodos de caracterización molecular para las diferentes especies de Candida más frecuentemente causantes de candidemia. Las características genéticas de dichas especies se resumen en la tabla 1.
Características genéticas de las principales especies de Candida y técnicas de tipificación descritas
| Especie | Ploidia | Especies relacionadas | Técnicas de tipificación molecular específicas para cada especie |
| C. albicans | Diploide | C. dubliniensis | C. albicans: multilocus sequence typing14, Southern blot con sondas CA27 y Ca34, short tandem repeats13,16,79 y MALDI-TOF72. C. dubliniensis: multilocus sequence typing54 |
| C. tropicalis | Diploide | Short tandem repeats32 y multilocus sequence typing86 | |
| C. glabrata | Haploide | C. bracarensis, C. nivariensis | Multilocus sequence typing35, random amplified polymorphic DNA10, short tandem repeats1,37,40 y Southern blot con sondas Cg6 y Cg1252 |
| C. parapsilosis | Diploide | C. orthopsilosis, C. metapsilosis | C. parapsilosis: short tandem repeats50,78,90, Southern blot con sonda Cp-1336, Rep-PCR33 y MALDI-TOF71 |
| C. krusei | Diploide | Short tandem repeats82, multilocus sequence typing45, pulse field gel electrophoresis41 y Southern blot con sonda CkF1, 220 |
Idealmente, la técnica genotípica debe mostrar un alto porcentaje de tipificabilidad (definido como la capacidad para analizar todas las cepas estudiadas), debe ser estable y reproducible, en la medida de lo posible debe ser económica y poco laboriosa, debe permitir un análisis objetivo y sencillo de los resultados, y, por último, debe mostrar un alto poder discriminatorio29,50.
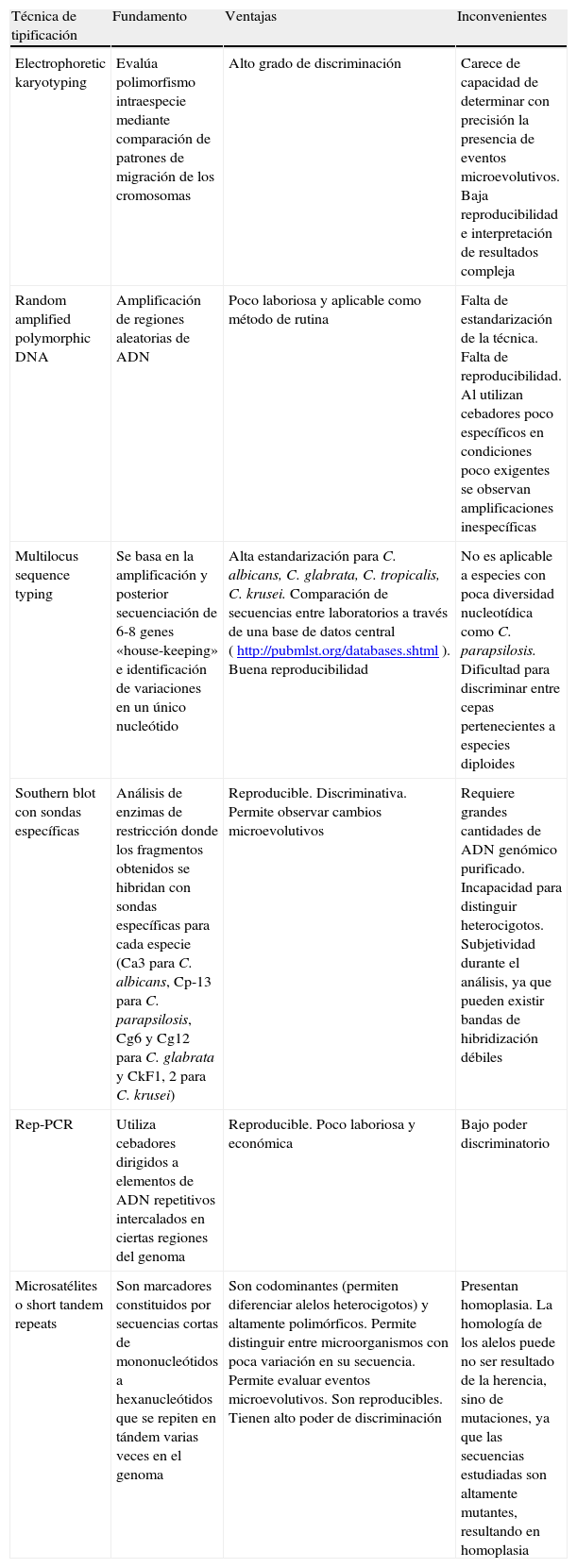
Las técnicas de tipificación molecular pueden agruparse en base a los elementos que evalúen en 2 grandes grupos. Uno de ellos engloba las técnicas basadas en procedimientos de análisis del ADN, y el otro evalúa indirectamente la expresión genética mediante el análisis de enzimas o patrones proteicos. Las principales técnicas genotípicas descritas para la caracterización de cepas de Candida spp. se recogen en las tablas 1 y 2.
Fundamento, ventajas e inconvenientes de las principales técnicas empleadas para el estudio genotípico de cepas de Candida spp
| Técnica de tipificación | Fundamento | Ventajas | Inconvenientes |
| Electrophoretic karyotyping | Evalúa polimorfismo intraespecie mediante comparación de patrones de migración de los cromosomas | Alto grado de discriminación | Carece de capacidad de determinar con precisión la presencia de eventos microevolutivos. Baja reproducibilidad e interpretación de resultados compleja |
| Random amplified polymorphic DNA | Amplificación de regiones aleatorias de ADN | Poco laboriosa y aplicable como método de rutina | Falta de estandarización de la técnica. Falta de reproducibilidad. Al utilizan cebadores poco específicos en condiciones poco exigentes se observan amplificaciones inespecíficas |
| Multilocus sequence typing | Se basa en la amplificación y posterior secuenciación de 6-8 genes «house-keeping» e identificación de variaciones en un único nucleótido | Alta estandarización para C. albicans, C. glabrata, C. tropicalis, C. krusei. Comparación de secuencias entre laboratorios a través de una base de datos central (http://pubmlst.org/databases.shtml). Buena reproducibilidad | No es aplicable a especies con poca diversidad nucleotídica como C. parapsilosis. Dificultad para discriminar entre cepas pertenecientes a especies diploides |
| Southern blot con sondas específicas | Análisis de enzimas de restricción donde los fragmentos obtenidos se hibridan con sondas específicas para cada especie (Ca3 para C. albicans, Cp-13 para C. parapsilosis, Cg6 y Cg12 para C. glabrata y CkF1, 2 para C. krusei) | Reproducible. Discriminativa. Permite observar cambios microevolutivos | Requiere grandes cantidades de ADN genómico purificado. Incapacidad para distinguir heterocigotos. Subjetividad durante el análisis, ya que pueden existir bandas de hibridización débiles |
| Rep-PCR | Utiliza cebadores dirigidos a elementos de ADN repetitivos intercalados en ciertas regiones del genoma | Reproducible. Poco laboriosa y económica | Bajo poder discriminatorio |
| Microsatélites o short tandem repeats | Son marcadores constituidos por secuencias cortas de mononucleótidos a hexanucleótidos que se repiten en tándem varias veces en el genoma | Son codominantes (permiten diferenciar alelos heterocigotos) y altamente polimórficos. Permite distinguir entre microorganismos con poca variación en su secuencia. Permite evaluar eventos microevolutivos. Son reproducibles. Tienen alto poder de discriminación | Presentan homoplasia. La homología de los alelos puede no ser resultado de la herencia, sino de mutaciones, ya que las secuencias estudiadas son altamente mutantes, resultando en homoplasia |
Se ha empleado para caracterizar cepas de las principales especies de Candida21,76. Se evalúa la variación de la movilidad electroforética de un conjunto de enzimas tras variaciones en su composición de aminoácidos, que implica cambios en la secuencia de ADN. Permite evaluar variaciones en diferentes loci, así como diferenciar entre homocigotos y heterocigotos en organismos diploides, ya que evalúan marcadores codominantes, y es altamente reproducible.
Espectrofotometría de masas de desorción/ionización láser (MALDI-TOF)En esta técnica, las células (levaduras) se mezclan con una matriz orgánica compuesta por ácido α-ciano-4-hidroxicinámico que se ioniza; posteriormente los iones son acelerados en un campo eléctrico y dirigidos a un detector, obteniéndose un patrón característico y específico para cada especie. La espectrofotometría de masas de desorción/ionización láser también puede tener aplicabilidad en la caracterización a nivel de subespecie tras un análisis de proyección principal de espectros, consistente en la comparación de «la huella peptídica» de cada aislamiento analizado y la evaluación de su relación construyendo un árbol filogenético8. Los resultados obtenidos hasta la fecha con C. albicans72 y C. parapsilosis son comparables a la tipificación molecular por microsatélites71.
Técnicas basadas en el análisis de ácido desoxirribonucleico mediante electroforesisElectrophoretic karyotypingPermite analizar los cromosomas mediante el análisis de bandas tras una electroforesis de campo pulsado. Las variaciones en los tamaños y patrones de migración se relacionan con variaciones polimórficas de los cromosomas y permiten distinguir entre cepas relacionadas. Es una técnica laboriosa, con poco poder de discriminación, por lo que se recomienda asociarla a otras técnicas de tipificación molecular24.
Restriction fragment length polymorphismEn esta técnica el ADN genómico se digiere con ciertas enzimas de restricción obteniéndose fragmentos cortos. Tras la electroforesis, se comparan los patrones de bandas obtenidos. Es una técnica rápida y poco laboriosa, aunque el análisis de los patrones de bandas puede ser subjetivo y, además, no permite la evaluación de eventos microevolutivos. El análisis por restriction fragment length polymorphism o electrophoretic karyotyping puede aplicarse a cualquier especie de Candida69.
Southern blot con sondas de hibridización específicasSe trata de un análisis de enzimas de restricción donde los fragmentos obtenidos se hibridan con sondas específicas para cada especie. Las sondas contienen secuencias repetitivas y se forman bandas de hibridación con las secuencias homólogas. Las sondas que se emplean para el análisis de C. albicans son CA27 y Ca325, Cp3-13 para C. parapsilosis36, Cg6 y Cg12 para C. glabrata52, y CkF1,2 y para C. krusei20.
Técnicas basadas en el análisis de ácido desoxirribonucleico mediante reacción en cadena de la polimerasaRandom amplified polymorphic deoxyribonucleid acidEsta técnica se ha desarrollado para C. albicans, C. parapsilosis y C. glabrata. Consiste en la amplificación aleatoria de fragmentos de ADN con cebadores arbitrarios, lo que genera productos de amplificación de diferente tamaño que se corresponden con un patrón específico para cada cepa. Está limitada por su baja reproducibilidad y la interpretación subjetiva de los resultados10.
Amplificación por reacción en cadena de la polimerasa de elementos palindrómicos extragénicos repetitivosSe trata de una amplificación de secuencias repetitivas de regiones no codificantes del genoma, que tras electroforesis arroja un patrón de bandas usado para comparar las cepas analizadas. Tiene un poder discriminatorio moderado, es reproducible y barato, y permite caracterizar cepas de C. albicans y C. parapsilosis24,33.
Multilocus sequence typingConsiste en la secuenciación de un conjunto de genes constitutivos o «house-keeping»62. Permite estudiar cepas de C. albicans, C. glabrata, C. krusei, C. tropicalis y Candida dubliniensis. No se puede aplicar a cepas de C. parapsilosis por su baja variabilidad nucleotídica, además de ser una especie aneuploide, lo que dificulta la estandarización de esta técnica62. Las secuencias obtenidas se pueden comparar con bases de datos que contienen secuencias disponibles on-line, lo que permite establecer comparaciones con cepas estudiadas con anterioridad14,62.
Se trata de una técnica laboriosa (6 a 8 reacciones de PCR por cepa, dependiendo de cada especie). Además, la detección de heterocigotos puede ser errónea, ya que requiere un análisis exhaustivo del cromatograma en busca de dobles picos que diferencien mutaciones puntuales del ruido de fondo.
Microsatélites o short tandem repeatsSe trata de regiones cuya secuencia está formada por motivos de uno o más nucleótidos que se repiten en tándem a lo largo del genoma y cuyas regiones flanqueantes están conservadas, lo que permite el diseño de cebadores81. Los microsatélites más informativos aparecerán, preferiblemente, en regiones no codificantes por su mayor polimorfismo. Los cebadores usados para amplificar estas regiones se marcan con diferentes fluorocromos y pueden combinarse en la misma PCR, lo que permite detectar la combinación de los productos de amplificación tras una electroforesis capilar. Cada fragmento tendrá una motilidad dependiente de su peso molecular, que a su vez se corresponde con el número de repeticiones del marcador.
Los microsatélites presentan muchas ventajas, entre las cuales están su elevado polimorfismo, codominancia, reproducibilidad, facilidad de automatización y análisis objetivo de los resultados mediante el empleo de programas informáticos. Al ser marcadores codominantes permiten la distinción entre microorganismos con poca variación en su secuencia81. Sin embargo, aunque se ha descrito que los microsatélites pueden intercambiarse entre laboratorios, el uso de diferentes equipos y condiciones de electroforesis puede alterar de manera mínima la longitud de los fragmentos de PCR. Para solventar este inconveniente, se puede realizar una calibración mediante escaleras alélicas asegurando así la reproducibilidad de resultados entre laboratorios31,38.
Dos cepas se definen como idénticas cuando se observa la misma composición alélica para todos los marcadores estudiados, diferentes cuando se presentan diferencias en varios loci, o como clonalmente relacionadas cuando se constatan eventos microevolutivos. Sin embargo, actualmente no existe un consenso para la definición de cepas clonalmente relacionadas. Algunos autores definen como complejos clonales aquellos genotipos que presentan cambios en un único locus33.
Se han desarrollado diversos microsatélites para las principales especies de Candida, entre las que destacan C. albicans13,16,79, C. parapsilosis50,78,90, C. glabrata1,37,40, C. tropicalis32 y C. krusei82.
Aplicación de la tipificación molecular como herramienta para la caracterización de brotes nosocomiales causados por Candida spp.Hay diversos estudios que demuestran la aplicación de las técnicas de tipificación molecular para esclarecer el origen de los brotes hospitalarios de candidemia. Los brotes descritos hasta la fecha afectan a pacientes ingresados en unidades de cuidados intensivos34,46,68,85, unidades quirúrgicas incluyendo aquellas en las que se desarrolla cirugía cardiotorácica33,64,93 o neurocirugía49, unidades de hematología17,51,60, o unidades de trasplante de progenitores hematopoyéticos91. También se han descrito brotes de candidemia en servicios pediátricos como oncología9,59,70, o en las unidades de cuidados intensivos neonatales5,11,26,28,44,47,55,73–75,87,89,90.
Las especies que más comúnmente causan brotes nosocomiales de candidemia son C. albicans11,44,49,64,73,85,87,93, C. parapsilosis9,17,28,33,34,51,55,68,70,75,89,90, C. glabrata59,91, C. tropicalis26,46,74 y C. krusei60. Recientemente se han descrito brotes por especies menos frecuentes, como Candida pelliculosa5 y otras especies de Candida47.
La transmisión horizontal entre pacientes ha demostrado ser la principal vía de transmisión de Candida en el medio hospitalario, jugando el personal sanitario un papel relevante en la transmisión de clones. Aunque se desconoce el reservorio de cepas de Candida dentro del hospital, estas pueden colonizar piel, manos y uñas del personal sanitario, que a su vez actúan como vehículo de transmisión de las cepas a los pacientes9,11,22,44,49,73,89,90. Sin embargo, se han propuesto otros reservorios de cepas, como las bolsas de desechos de orina en pacientes sondados46, o las soluciones de nutrición parenteral de naturaleza lipídica que se contaminan durante su preparación o manipulación antes de ser administradas a los pacientes57,68,74.
Los recién nacidos con bajo peso, al estar hospitalizados en unidades de cuidados intensivos neonatales y requerir nutrición parenteral, son un grupo de especial riesgo para el desarrollo de candidemia. Además, su manipulación frecuente por parte del personal sanitario les predispone a infectarse por cepas propias del personal de la unidad de ingreso73,80,89. Las cepas de Candida que colonizan la piel de estos pacientes también colonizan los catéteres intravasculares y las preparaciones de nutrición parenteral que reciben, lo que les convierte en la puerta de acceso al torrente sanguíneo28,55. Sin embargo, también se han descrito brotes en pacientes sin catéteres intravasculares, o en los que la infección se originó tras el uso de productos contaminados, como ungüentos tópicos19, preparaciones de medicamentos94 o botellas de leche59.
El catéter es, con frecuencia, el foco de la infección en muchos pacientes con candidemia34,39,51,55,70,77. En los casos en los que se ha podido estudiar el aislamiento obtenido de la sangre del paciente, junto con los aislamientos aislados del catéter y del personal sanitario, se ha demostrado que el genotipo encontrado tanto en la sangre como en el catéter se encuentra también en las manos del personal sanitario, lo que sugiere una contaminación del catéter tras su manipulación51,70. No obstante, el catéter no es el único origen de la candidemia causada por cepas exógenas, puesto que se han descrito casos en los que la puerta de acceso al torrente circulatorio son las mucosas o las heridas64.
Papel del laboratorio de microbiología en la caracterización de brotes hospitalarios de candidemiaLa detección de un brote hospitalario y el esclarecimiento de su origen es esencial para evitar que aparezcan nuevos casos, y el laboratorio de microbiología tiene un papel fundamental en este sentido. La sospecha de un brote suele ser consecuencia de la observación de un número de casos de candidemia anormalmente altos en una unidad concreta. La caracterización genotípica de los aislamientos será de gran utilidad para determinar si las cepas de los pacientes involucrados pertenecen al mismo clon y, por tanto, sugieren un origen común. Detectar el origen del brote es el comienzo del final del mismo, tras la implantación de medidas correctoras. La recogida minuciosa y apropiada de los aislamientos es, quizás, el paso más importante para la caracterización genotípica. Se debe contar con los aislamientos invasivos provenientes de cada paciente involucrado. Para trazar el origen es necesario muestrear el ambiente de los pacientes (manos del personal sanitario, superficies de las habitaciones y del mobiliario, medicación sospechosa de estar infectada, etc.). Cualquier elemento susceptible de estar contaminado con los aislamientos causantes del brote debe ser examinado y muestreado. Adicionalmente, se deben incluir aislamientos de pacientes localizados en diferentes unidades para ser usados como controles. La elección de la técnica genotípica dependerá de las posibilidades del laboratorio, así como de las especies involucradas en el brote, tal y como se comentó anteriormente.
ConclusionesLos brotes de candidemia pueden ser un problema en las unidades de ingreso de pacientes críticos, particularmente en aquellas con neonatos. A la hora de investigar un brote, es primordial conocer la relación genética entre los aislamientos involucrados. Las técnicas moleculares son útiles para estudiar el origen de los brotes; entre estas destacan los microsatélites, que permiten conocer de forma rápida, reproducible y discriminativa la relación entre cepas. En base a los estudios de tipificación realizados, se ha podido demostrar que existe transmisión cruzada de Candida en diversas áreas del hospital y que la vía de infección más común es la contaminación del catéter intravascular por parte del personal sanitario.
FinanciaciónEste estudio ha sido parcialmente financiado por el proyecto del Fondo de Investigación Sanitaria (FIS) con número de referencia PI11/00167.
Laura Judith Marcos-Zambrano disfruta de una beca predoctoral del FIS (FI12/00265). Pilar Escribano disfruta de un contrato posdoctoral Sara Borell del FIS (CD09/00230). Jesús Guinea disfruta de un contrato para investigadores del Sistema Nacional de Salud, programa Miguel Servet (MS09/00055).
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.