Los hongos fitopatógenos Alternaria alternata y Colletotrichum gloeosporioides pueden provocar enfermedades en los tejidos vegetales y pérdidas significativas en la poscosecha. El uso de fungicidas químicos para su control tiene efectos negativos sobre el ambiente y la salud. Los metabolitos secundarios de bacterias halófilas son una alternativa prometedora para obtener nuevos compuestos antifúngicos. En el presente estudio, se aislaron y caracterizaron bacterias halófilas provenientes de dos sitios con suelos salinos, denominados blanquizales, en Campeche, México. En total, se aislaron 64 bacterias; entre los géneros identificados por métodos moleculares, se encontraron Agrobacterium, Bacillus, Inquilinus, Gracilibacillus, Metabacillus, Neobacillus, Paenibacillus, Priestia, Staphylococcus, Streptomyces y Virgibacillus. Se investigó el potencial antifúngico del sobrenadante de cultivo (SC) de 39 bacterias halófilas contra C. gloeosporioides y A. alternata. Las bacterias que ejercieron una mayor inhibición del crecimiento micelial (ICM) fueron Bacillus subtilis CPO 4.292, Metabacillus sp. CPO 4.266, Bacillus sp. CPO 4.295 y Bacillus sp. CPO 4.279. Asimismo, el SC de Bacillus sp. CPO 4.279 fue el que presentó la mayor actividad y su extracto en acetato de etilo (AcOEt) el que más inhibió la germinación de conidios de C. gloeosporioides, con valores de inhibición del crecimiento media.
(IC50) de 8.630μg/ml e IC90 de 10.720μg/ml. La partición orgánica del extracto en AcOEt permitió la obtención de tres fracciones, la de acetonitrilo (FAcB9) presentó la mayor actividad antifúngica, con un porcentaje de inhibición de la germinación de condios superior al 66%. Se concluye que las bacterias halófilas de suelos blanquizales del género Bacillus sp. producen metabolitos con propiedades antifúngicas para inhibir al hongo fitopatógeno C. gloeosporioides.
Phytopathogenic fungi Alternaria alternata and Colletotrichum gloeosporioides cause diseases in plant tissues as well as significant postharvest losses. The use of chemical fungicides for their control has negative effects on health and the environment. Secondary metabolites from halophilic bacteria are a promising alternative for new antifungal compounds. In the present study, halophilic bacteria were isolated and characterized from two sites with saline soils called branquizales in Campeche, Mexico. A total of 64 bacteria were isolated. Agrobacterium, Bacillus, Inquilinus, Gracilibacillus, Metabacillus, Neobacillus, Paenibacillus, Priestia, Staphylococcus, Streptomyces and Virgibacillus were among the identified genera. The antifungal potential of the culture supernatant (CS) of 39 halophilic bacteria was investigated against C. gloeosporioides and A. alternata. The bacteria showing the greatest inhibition of mycelial growth corresponded to Bacillus subtilis CPO 4292, Metabacillus sp. CPO 4266, Bacillus sp. CPO 4295 and Bacillus sp. CPO 4279. The CS of Bacillus sp. CPO 4279 exhibited the highest activity and its ethyl acetate extract (AcOEt) inhibited the germination of C. gloeosporioides, with IC50 values of 8,630μg/ml and IC90 of 10,720μg/ml. The organic partition of the AcOEt extract led to three fractions, with acetonitrile (FAcB9) showing the highest antifungal activity, with values exceeding 66%. Halophilic bacteria from ‘blanquizales’ soils of the genus Bacillus sp. produce metabolites with antifungal properties that inhibit the phytopathogenic fungus C. gloeosporioides.
La agricultura tropical a nivel mundial se ve afectada por la presencia de enfermedades de origen fúngico, que ocasionan pérdidas durante los ciclos de producción y en la poscosecha14. Los hongos fitopatógenos Alternariaalternata y Colletotrichumgloeosporioides producen daños en la poscosecha de varios cultivos, que pueden reducir hasta en un 40% la producción13,46. La principal estrategia de control de estos hongos es mediante el uso de fungicidas de origen químico; no obstante, el empleo excesivo de estos productos ha provocado contaminación ambiental y resistencia de los hongos a los compuestos específicos5,29. Por lo tanto, es importante encontrar nuevas fuentes de compuestos con actividad antifúngica amigables con el ambiente y efectivos contra hongos fitopatógenos resistentes a los productos actuales42.
En este sentido, las bacterias halófilas constituyen un grupo diverso de microorganismos caracterizado por habitar en ambientes con altas concentraciones de sales30,47. Se considera que estas bacterias han desarrollado múltiples mecanismos de adaptación para sobrevivir, entre ellos, la producción de metabolitos que funcionan como defensa frente a organismos competidores, lo que supone una fuente importante de nuevos compuestos antifúngicos6,10,38,47. En este contexto, Sadfi-Zouaoui et al.37 reportaron que las bacterias halófilas Bacillus, Halobacillus, Halomonas, Marinococcus, Planococcus y Salinicoccus, aisladas de lagos salados en Túnez, inhibieron el crecimiento del fitopatógeno Botrytiscinerea en un 50 a 91%. A su vez, Elyasi et al.10 aislaron del desierto de sal de la provincia de Semnan, en el norte de Irán, las bacterias halófilas Bacillus subtilis Dar y D6A, con capacidad para inhibir el crecimiento de los hongos fitopatógenos Fusariumoxysporum y B. cinerea. También, se ha reportado la actividad antifúngica de extractos orgánicos provenientes del sobrenadante de cultivo (SC) de bacterias halófilas. Por ejemplo, Nas et al.27 encontraron que los extractos crudos de acetato de etilo (AcOEt) y cloroformo del SC de una cepa halófila de Bacillus sp. fueron capaces de inhibir el crecimiento de Aspergillusflavus. Entre los principales compuestos identificados en los extractos con actividad antifúngica se encontraron el 2,4-di-terc-butilfenol, el 1-nonilcicloheptano y el 1,3-di-terc-butilbenceno. De igual forma, Tian et al.44 identificaron en el SC del actinomiceto Nocardiopsisgilva YIM 90087 p-terfenil, novobiocina y ciclodipéptidos con actividad antifúngica contra los hongos fitopatógenos Fusariumavenaceum, Fusariumgraminearum, Fusariumculmorum y Pyriculariaoryzae. Esto indica que los compuestos producidos por bacterias halófilas tienen un alto potencial para ser utilizados como antifúngicos.
México posee diversos hábitats hipersalinos, de los cuales solo algunos han sido investigados con respecto a la composición de su comunidad microbiana7,12. En la Reserva de la Biósfera de los Petenes (RBLP), en Campeche, México, existen ciertos ambientes conocidos como blanquizales, que son suelos hipersalinos y calcáreos, en los que se registran altos valores de salinidad y con condiciones mesohalinas en la temporada de lluvias (27,1± 8,0g/l) e hipersalinas en la temporada seca (47,0±7,0g/l)4. Estos ambientes son considerados únicos a nivel internacional, ya que se encuentran ubicados dentro de una planicie costera con sedimentación palustre, adyacente al mar, en la que existen tres distintas geoformas de origen: litoral, palustre y kárstico22. A pesar de ello, no hay reportes de la diversidad microbiana existente y el potencial de sus bacterias halófilas para producir metabolitos secundarios con actividad antifúngica contra A. alternata y C. gloeosporioides. El presente estudio tiene por objetivo aislar e identificar molecularmente bacterias halófilas de suelos de la RBLP y determinar su potencial para liberar compuestos antifúngicos.
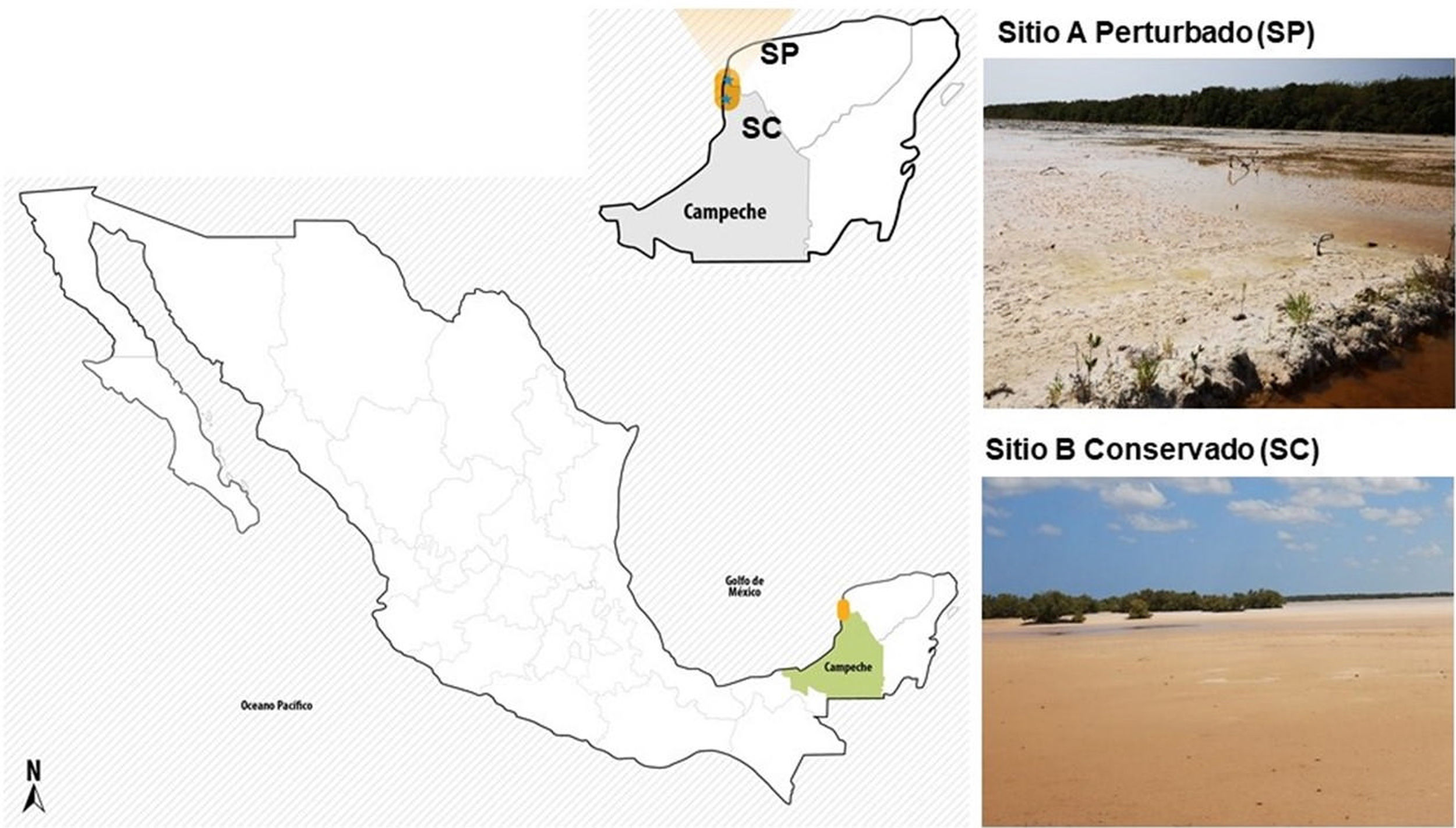
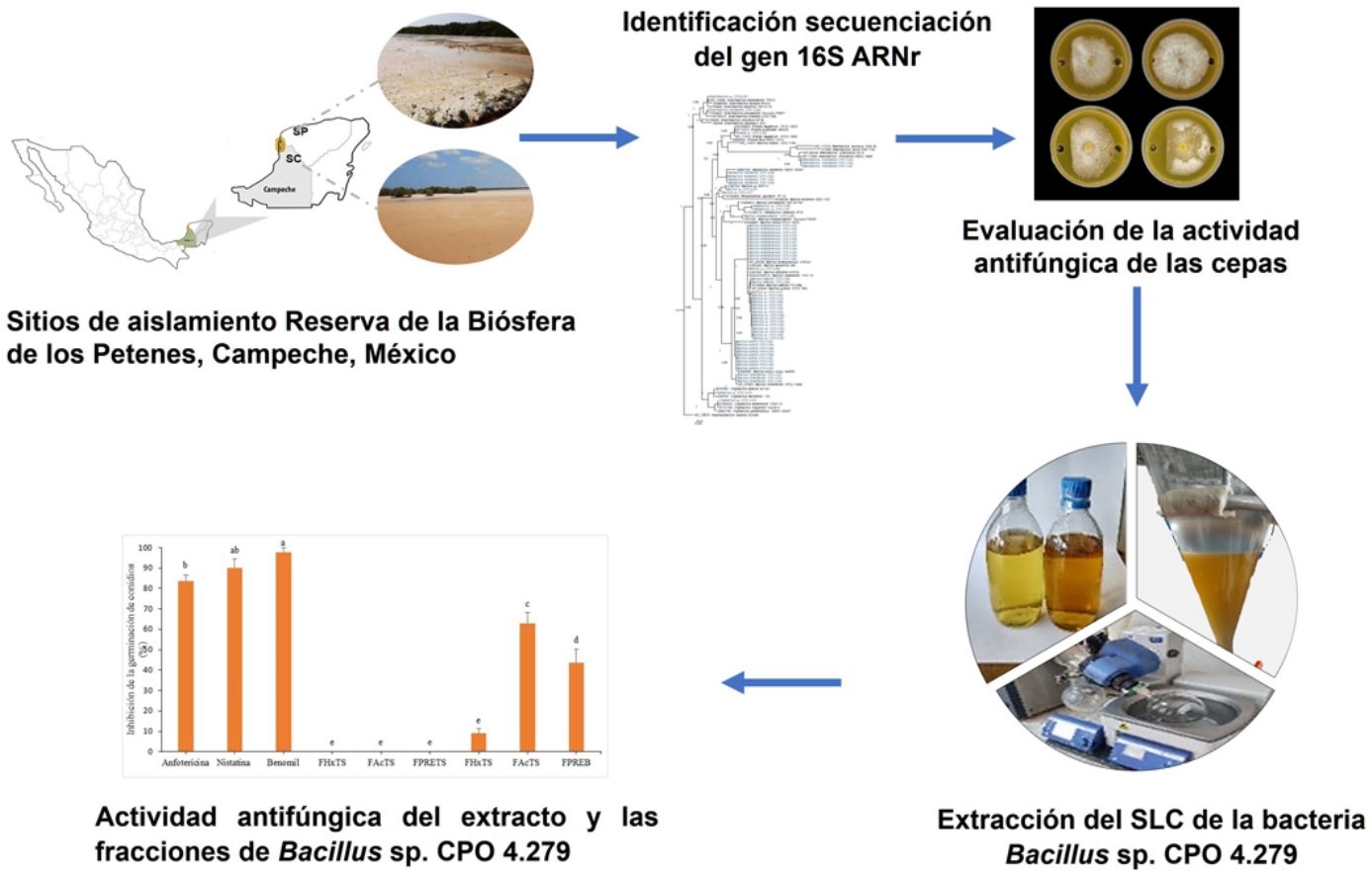
Materiales y métodosSitios de muestreo de los suelos salinosEn mayo de 2019, se colectaron muestras de suelo blanquizal en dos sitios de la RBLP con diferente grado de conservación: el ejido de Tankuché (20° 15′ 40,7″ N, 90° 28′ 31,0″ W), que se consideró suelo de blanquizal perturbado, y el ejido de Nunkiní (20° 32′ 46,9″ N, 90° 25′ 45,6″ W), que se consideró suelo de blanquizal conservado (Fig. 1S). En cada sitio se tomaron cinco submuestras de suelo a una profundidad de 30cm, las cuales se homogenizaron para formar una muestra compuesta representativa de cada sitio (1kg).
Aislamiento de las bacterias halófilasEl aislamiento de las bacterias halófilas se realizó por diluciones decimales. Para ello, se diluyeron 10g de suelo salino seco en 90ml de solución salina estéril al 25% m/v7. A partir de esta suspensión, se prepararon diluciones desde 10-1 hasta 10-5. Alícuotas de cada dilución (0,1ml) se esparcieron con una espátula Drigalski de vidrio estéril en placas de Petri con los medios de aislamiento agar tripticasa de soya (TSA)40, medio de cultivo agar R2A3 y agar extracto de malta (EM)40, suplementados con 0,5M de cloruro de sodio (NaCl) para favorecer el crecimiento de un mayor número de bacterias halófilas. Los cultivos se incubaron a 28±3°C. Las colonias con diferentes morfologías se aislaron y purificaron por estría por agotamiento.
La capacidad de las cepas bacterianas de tolerar la salinidad se evaluó haciendo crecer cada una de las cepas en medio TSA con diferentes concentraciones de NaCl (0,5; 2 y 3M)40 por un tiempo de incubación máximo de 48horas. Aquellas bacterias que presentaron crecimiento óptimo en 24horas a 0,5M se categorizaron como halotolerantes, las que crecieron a 2M como halófilas moderadas y las que desarrollaron a 3M como halófilas extremas, según el esquema de clasificación propuesto por Kushner23.
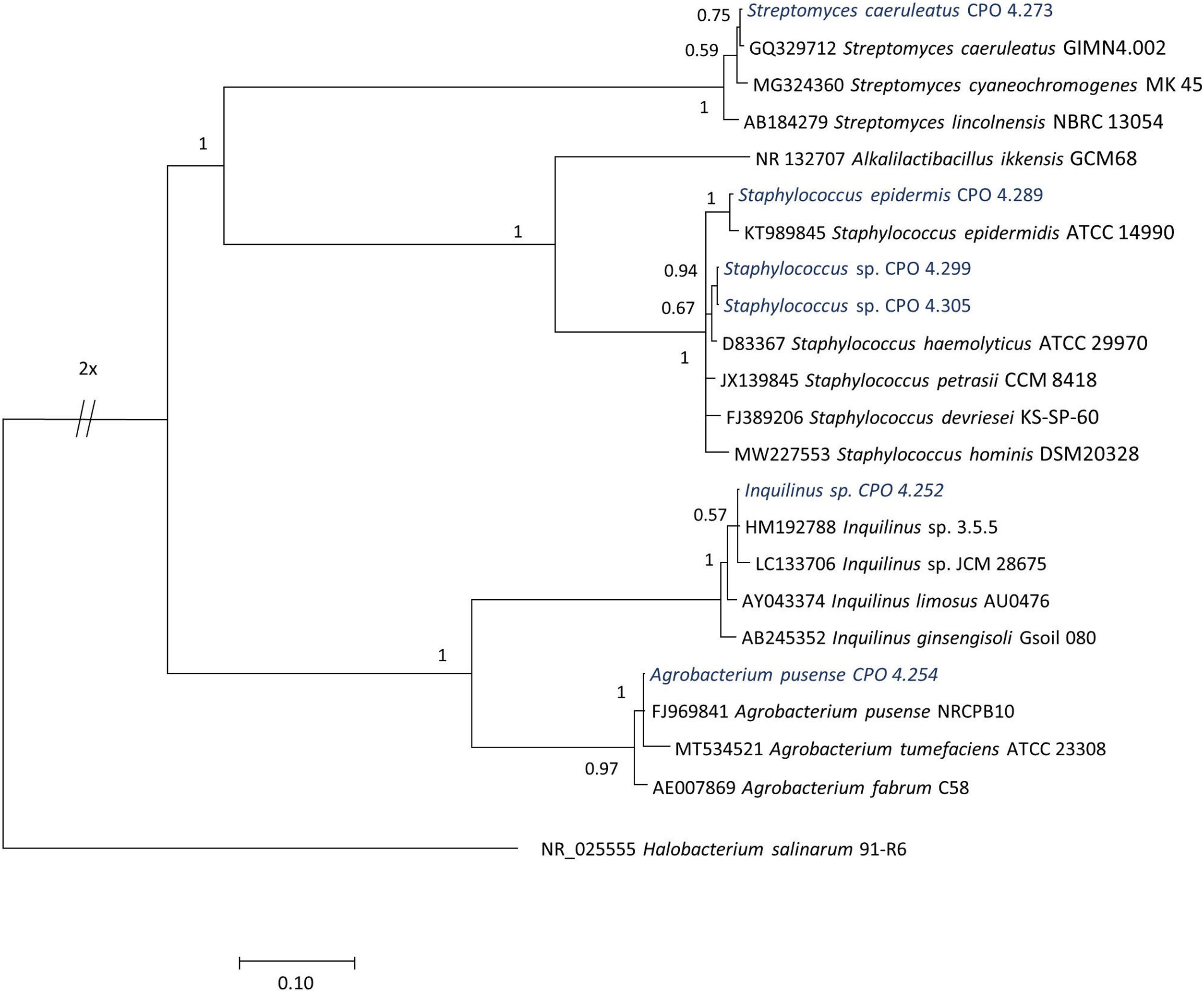
Identificación de las bacterias por secuenciación del gen 16S ARNrLas cepas bacterianas se identificaron con base en la secuenciación parcial del gen 16S ARNr. El ADN se extrajo con bromuro de hexadeciltrimetilamonio (CTAB) al 2% m/v, de acuerdo con lo propuesto por Doyle et al.8 La integridad del ADN se determinó mediante electroforesis en gel de agarosa al 1,5% suplementado con GelRed® (Biotium, EE. UU.). La amplificación del fragmento de 1.500 pb se llevó a cabo con los iniciadores universales 8F (5′-AGAGTTTGATCCTGGCTCAG-3′) y 1492 R (5′-GTTACCTTGTTACGACTT-3′)9, y con fD1 (5’-AGAGTTTGATCCTGGCTCAG-3’) y rD1 (5’-AAGGAGGTGATCCAGCC-3’)49. La mezcla de reacción se preparó en un volumen final de 15μl y el programa de amplificación que se aplicó es el descrito por Eden et al.9. Los productos de reacción en cadena de la polimerasa (PCR), de aproximadamente 1.500 pb, se purificaron con EXO-SAP® (Affymetrix, EE. UU.), siguiendo las instrucciones del fabricante. Los amplicones purificados se secuenciaron con los iniciadores 518F (5’-CCAGCAGCCGCGGTAATACG-3′) y 800R (5’-TACCAGGGTATCTAATCC-3′) en la compañía Macrogen, mediante secuenciación capilar. Las secuencias de ambas hebras de cada cepa se ensamblaron y editaron usando el programa BioEdit versión v7.2.516 (Thomoas A. Hall, 2013, California, EE. UU.) y se analizaron utilizando el algoritmo BLASTN del software BLAST/NCBI1 (National Center for Biotechnology Information, 2011, Maryland, EE. UU.) y el Ribosomal Database Project release 1150. Se realizó el alineamiento múltiple de secuencias utilizando el programa MAFFT v.7 (Kazutaka Katoh, 2002, Osaka, Japón; http://mafft.cbrc.jp/aligment/server/)21. La reconstrucción filogenética de todas las secuencias se realizó con inferencia bayesiana usando el programa Mr. Bayes v3.235 (Ronquist Fredrik et al., 2012, Uppsala, Suecia) y el modelo de sustitución nucleotídica fue (GTR+G) (por sus siglas en inglés General Time Reversible [tiempo reversible general]) con 3.000.000 de generaciones. Para la construcción de los árboles filogenéticos, se incluyeron las cepas Alkalilactibacillus ikkensis GCM68 (NR_132707) y Halobacterium salinarum 91-R6 (NR_025555) como fuera de grupo. Los árboles filogenéticos se visualizaron y editaron en FigTree V1.4.4 (Rambautet Andrew, 2016, Edimburgo, Escocia; https://tree.bio.ed.ac.uk/software/figtree/). Todas las secuencias se depositaron en la base de datos del National Center for Biotechnology Information (NCBI)-GenBank (N.° de acceso MW429711-MW429774).
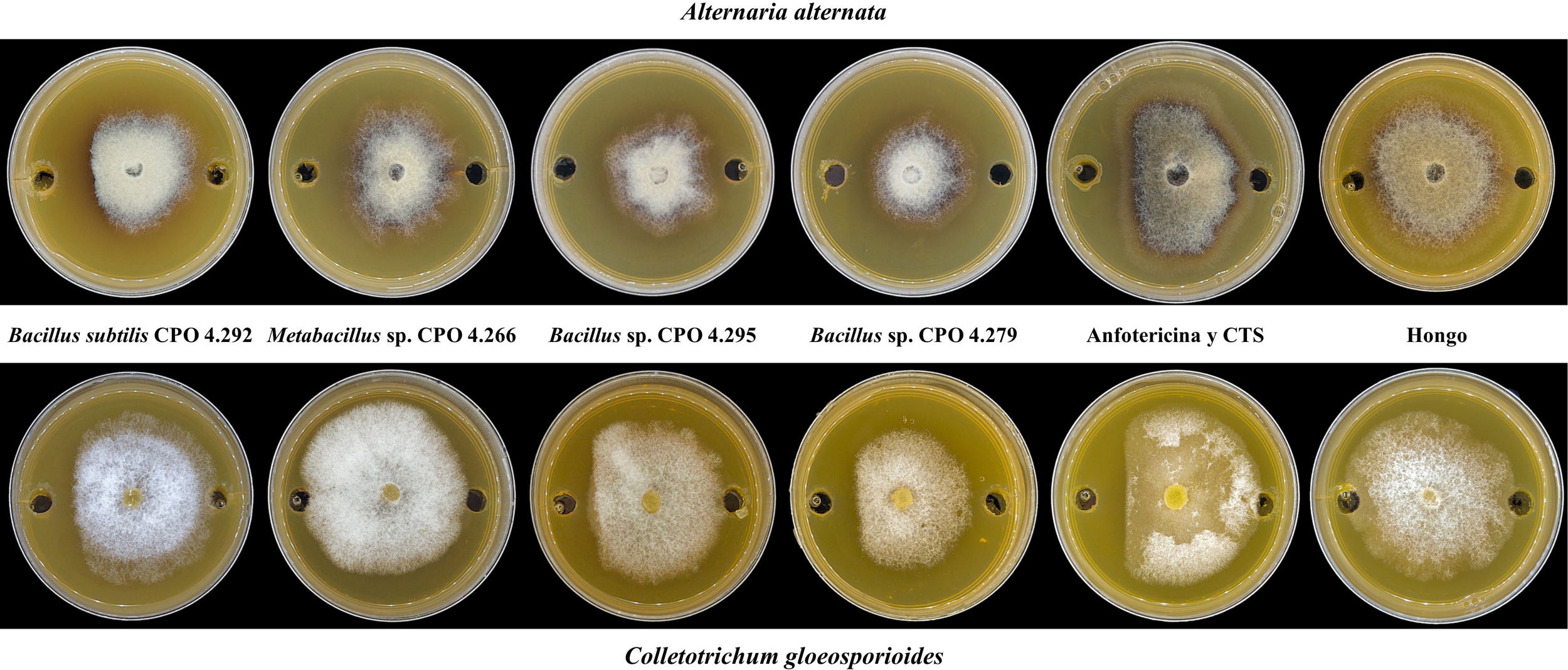
Evaluación de la actividad antifúngica de las cepas bacterianas halófilasDe las 64 cepas bacterianas aisladas, 25 perdieron su viabilidad. Por lo tanto, solo 39 se reactivaron y cultivaron en 1ml de caldo de tripticasa de soya (CTS) con 0,5M de NaCl40, a 28°C 33±3°C por 15 días33 a 180rpm34 en agitación orbital. Para obtener el SC, estos se centrifugaron a 6.000rpm durante 20min y se filtraron a través de una membrana estéril (0,22μm). Para evaluar la actividad antifúngica de las bacterias halófilas, se utilizó el ensayo de difusión en pozo, de acuerdo con la metodología descrita por Elyasi et al.10. Para ello, en placas de Petri con medio de cultivo papa dextrosa agar (PDA), se realizaron dos perforaciones de 6mm de diámetro en los extremos de la placa y en ellas se depositó el SC (70μl) de cada cepa bacteriana. Finalmente, en el centro de cada placa se colocó un disco invertido de micelio de A. alternata JG14 o de C. gloeosporioides CGP6. Se incluyó anfotericina B como control positivo, a una concentración de 8μg/ml para C. gloeosporioides y de 4μg/ml para A. alternata. Como control negativo, se utilizó CTS con 0,5M de NaCl en el pozo y el hongo fitopatógeno. Las placas se incubaron a 28±3°C hasta que el micelio de las placas del control negativo alcanzó el borde de los pozos. La actividad antifúngica se expresó mediante el porcentaje de inhibición del crecimiento micelial (ICM) empleando la fórmula de Shi et al.39:
donde R1 (mm) es el crecimiento del hongo del control negativo y R2 (mm) es el crecimiento del hongo en presencia del SC de cada cepa bacteriana.
Selección de la cepa bacteriana halófila con mayor actividad antifúngica contra C.gloeosporioidesLas cepas bacterianas halófilas más activas se cultivaron nuevamente en 1ml de CTS con 0,5M de NaCl por 48h para obtener un preinóculo20, que luego se transfirió a tubos con 2.500ml de CTS con 0,5M de NaCl, ajustado a una concentración de 1,5×108 UFC/ml20. Las condiciones de incubación fueron 28°C33±3°C por 15 días33, con una agitación orbital a 180rpm34. El SC se obtuvo como se describió anteriormente. La cepa fitopatógena A. alternata no fue capaz de producir una cantidad elevada de conidios, los cuales son necesarios para los ensayos de inhibición de la germinación de conidios (IGC) y la inhibición del crecimiento (IC), por lo que se decidió excluirla en el proceso de selección de la cepa más activa. Por lo tanto, los SC masivos de las cepas con mayor actividad se evaluaron únicamente contra C. gloeosporioides mediante los ensayos de IGC, IC e ICM. El porcentaje de ICM se determinó como se describió anteriormente, mediante el método de difusión en pozo.
La IGC se determinó de acuerdo con la descripción de Suwan et al.43, con ligeras modificaciones. El SC de las bacterias se concentró a 2×por liofilización. La suspensión de conidios del hongo fitopatógeno C. gloeosporioides se preparó con solución salina estéril y se ajustó a una concentración de 2×105 conidios/ml con un hemocitómetro. Posteriormente, se depositaron 20μl del SC concentrado en un tubo de microcentrífuga y se mezclaron con 20μl de una suspensión de conidios de C. gloeosporioides. El tubo fue incubado a 28°C±3°C por 12horas. Se utilizó como control positivo anfotericina B (8μg/ml), nistatina (6,25μg/ml) y benomil (1000μg/ml), y como control negativo CTS concentrado a 2×. Después de 12horas, se tomó una alícuota de 10μl de la mezcla y se determinó el IGC contando aleatoriamente 100 conidios por tratamiento, empleando un microscopio óptico (Prisma 209, Optika Microscopes, Italia) con aumento de 10×. Se registró el número de conidios germinados; se consideró conidio germinado aquel cuyo tubo germinativo tenía el doble de longitud que el conidio. El porcentaje de IGC se estimó con la fórmula siguiente:
IGC (%)=[(Eg1 – Eg2) / (Eg2)] x 100
donde Eg1 es el número de conidios germinados en el control negativo y Eg2 es el número de conidios germinados en presencia del SC de cada bacteria.
La IC se determinó mediante el método de microdilución, como describen Escalante-Rendiz et al. 11, con ligeras modificaciones. El SC de las bacterias se concentró a un cuarto de su volumen (4×) por liofilización. Luego, se depositó el SC concentrado (50μl) y el medio sintético RPMI-1640 (50μl) en una microplaca de 96 pozos. Finalmente, se transfirieron 100μl de una suspensión de conidios de C. gloeosporioides (1×105 conidios/ml). Se utilizó el medio CTS concentrado 4×como control negativo y como control positivo se emplearon los mismos compuestos que los usados en la determinación de la IGC. Las pruebas se realizaron por triplicado y las placas se incubaron a 28°C±3°C por 96horas. La IC de C. gloeosporioides por el SC se evaluó visualmente usando la escala numérica establecida por el Comité Nacional de Normas de Laboratorio Clínico28.
Extracción del SC de la bacteria Bacillus sp. CPO 4.279La bacteria Bacillus sp. CPO 4.279 fue cultivada en CTS con 0,5M de NaCl a 28°C33±3°C durante 15 días33, en agitación orbital a 180rpm34, para obtener el SC. La extracción orgánica del SC se realizó mediante una partición líquido-líquido con AcOEt dos veces (2:1, 1.1, v/v). El disolvente se eliminó a presión reducida en un evaporador rotatorio (Ika RV 10 Control, Optika Microscopes, Italia; IKA, Alemania) para obtener el extracto crudo EcB9 (594,80mg). También se incluyó un blanco de extracción del medio caldo tripticasa libre de la bacteria (EcTS; 102,50mg).
Fraccionamiento del extracto de acetato de etilo del SC de Bacillus sp. CPO 4.279Los extractos de AcOEt EcB9 (100mg) y EcTS (100mg) se resuspendieron en acetonitrilo (AcN) (10ml) y se fraccionaron con hexano (Hx) tres veces (2:1, 1:1, 1:1, v/v). El disolvente se eliminó a presión reducida para obtener, a partir del extracto crudo de EcB9, las fracciones de baja polaridad (FHxB9; 4,80mg) y mediana polaridad (FAcB9; 116,90mg), y un precipitado (FPREB9; 45,80mg). Las fracciones obtenidas del extracto EcTS fueron FHxTS (0,60mg), FAcTS (2,80mg) y FPRETS (32,80mg), respectivamente36.
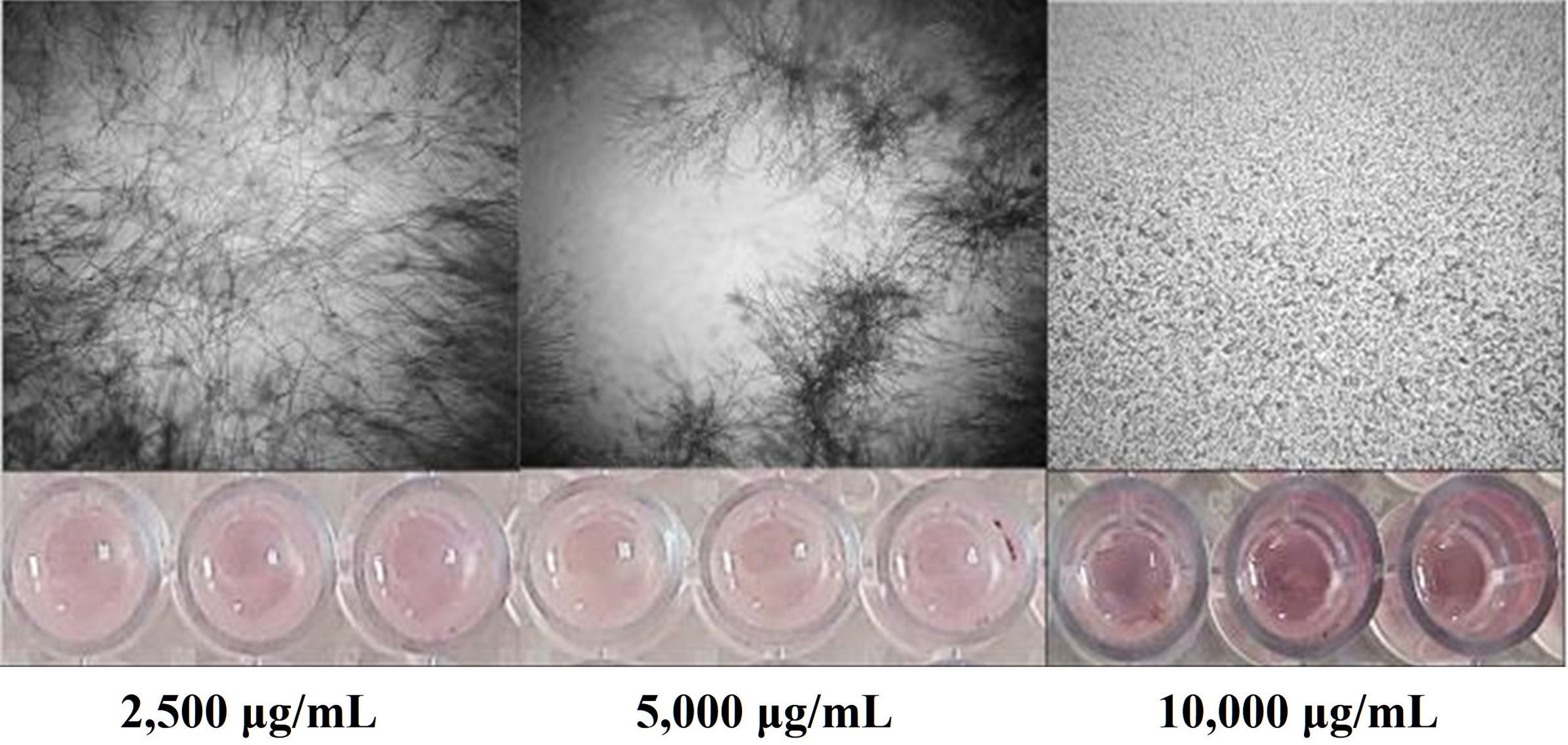
Actividad antifúngica del extracto y de las fracciones de Bacillus sp. CPO 4.279La concentración inhibitoria mínima (CIM) de los extractos y de las fracciones se determinó mediante el ensayo de microdilución en caldo, siguiendo la metodología de Medina-Baizabal et al.26, con modificaciones. Los extractos EcB9 y EcTS se disolvieron en dimetil sulfóxido (DMSO) a una concentración de 40mg/ml. Posteriormente, se mezclaron con el medio sintético RPMI-1640 y se depositaron en una microplaca de 96 pozos. Se realizaron diluciones seriadas hasta obtener concentraciones finales del extracto de 10.000, 5.000 y 2.500μg/ml. Finalmente, se depositaron 100μl de la suspensión de conidios de C. gloeosporioides a una concentración final de 2,5×104 conidios/ml. Se utilizaron como controles positivos anfotericina B (8μg/ml), nistatina (6,25μg/ml) y benomil (1mg/ml), y como controles negativos DMSO al 5% con el hongo fitopatógeno y el blanco del extracto EcTS (10.000μg/ml) más el hongo. Se efectuaron pruebas por triplicado y se incubaron las placas a 28°C±3°C durante 96horas. La CIM se definió como la concentración más baja en la que no se observó crecimiento visible del hongo después de la incubación.
También se calculó la concentración inhibitoria media y noventa (CI50 y CI90) del extracto crudo EcB9 mediante el método de IGC por un análisis Probit con diferentes concentraciones del extracto (8.000, 10.000 y 12.000μg/ml); los resultados se analizaron utilizando el sistema de análisis estadístico (SAS, Institute Inc., 2004, Carolina del Norte, EE. UU.), versión 9 para Windows36.
La actividad antifúngica de las fracciones obtenidas del extracto EcB9 se determinó mediante el método IGC, utilizando una concentración final de 8.000μg/ml. Se emplearon tres réplicas por tratamiento y como controles se usaron los descritos anteriormente.
Análisis estadísticoLas medias se analizaron mediante un análisis de varianza (ANOVA) seguido de la prueba Tukey de comparación múltiple (p=0,05), con el objetivo de encontrar diferencias significativas entre tratamientos. Se empleó el software SAS versión 9 para Windows.
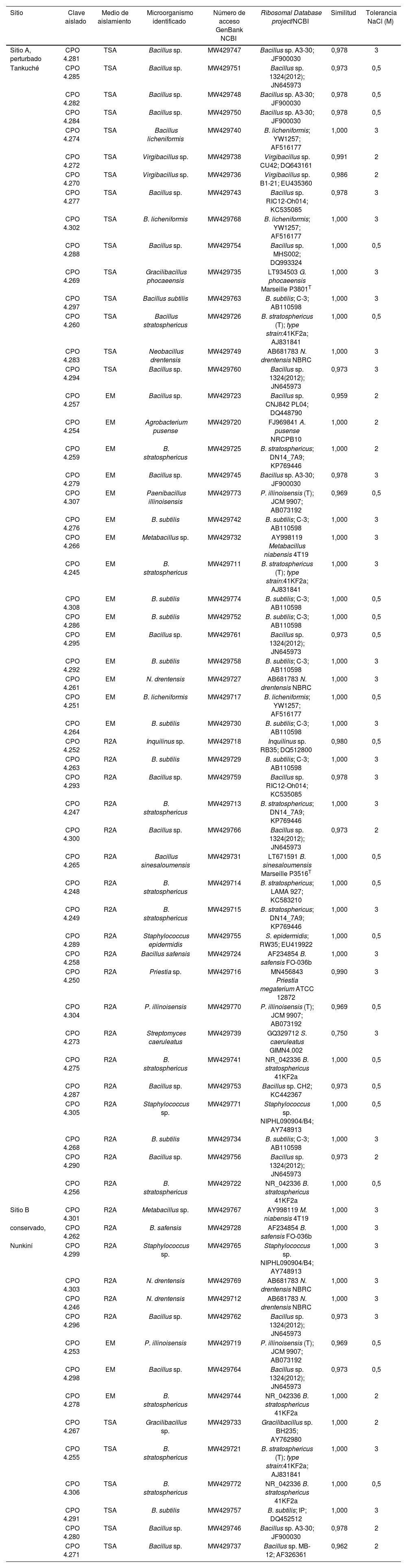
ResultadosAislamiento e identificación de las bacterias halófilasSe aislaron en total 64 cepas bacterianas halófilas a partir de suelos salinos de los blanquizales de la RBLP; se obtuvo un mayor número de bacterias (n=49) a partir del sitio perturbado en comparación con el sitio conservado (n=15) (tabla 1). Asimismo, del total de cepas bacterianas aisladas, un gran porcentaje mostró capacidad de crecer a 3 y 0,5M de NaCl; por lo tanto, se categorizaron como halófilas extremas (31 cepas, 48%) y halotolerantes (22 cepas, 34%), respectivamente. Solo 11 cepas (17%) se clasificaron como halófilas moderadas al crecer a una concentración 2M de NaCl (tabla 1).
Identificación molecular de cepas halófilas aisladas de dos sitios de suelos salinos blanquizales de la Reserva de la Biósfera de los Petenes, México
| Sitio | Clave aislado | Medio de aislamiento | Microorganismo identificado | Número de acceso GenBank NCBI | Ribosomal Database project/NCBI | Similitud | Tolerancia NaCl (M) |
|---|---|---|---|---|---|---|---|
| Sitio A, perturbado | CPO 4.281 | TSA | Bacillus sp. | MW429747 | Bacillus sp. A3-30; JF900030 | 0,978 | 3 |
| Tankuché | CPO 4.285 | TSA | Bacillus sp. | MW429751 | Bacillus sp. 1324(2012); JN645973 | 0,973 | 0,5 |
| CPO 4.282 | TSA | Bacillus sp. | MW429748 | Bacillus sp. A3-30; JF900030 | 0,978 | 0,5 | |
| CPO 4.284 | TSA | Bacillus sp. | MW429750 | Bacillus sp. A3-30; JF900030 | 0,978 | 0,5 | |
| CPO 4.274 | TSA | Bacillus licheniformis | MW429740 | B. licheniformis; YW1257; AF516177 | 1,000 | 3 | |
| CPO 4.272 | TSA | Virgibacillus sp. | MW429738 | Virgibacillus sp. CU42; DQ643161 | 0,991 | 2 | |
| CPO 4.270 | TSA | Virgibacillus sp. | MW429736 | Virgibacillus sp. B1-21; EU435360 | 0,986 | 2 | |
| CPO 4.277 | TSA | Bacillus sp. | MW429743 | Bacillus sp. RIC12-Oh014; KC535085 | 0,978 | 3 | |
| CPO 4.302 | TSA | B. licheniformis | MW429768 | B. licheniformis; YW1257; AF516177 | 1,000 | 3 | |
| CPO 4.288 | TSA | Bacillus sp. | MW429754 | Bacillus sp. MHS002; DQ993324 | 1,000 | 0,5 | |
| CPO 4.269 | TSA | Gracilibacillus phocaeensis | MW429735 | LT934503 G. phocaeensis Marseille P3801T | 1,000 | 3 | |
| CPO 4.297 | TSA | Bacillus subtilis | MW429763 | B. subtilis; C-3; AB110598 | 1,000 | 3 | |
| CPO 4.260 | TSA | Bacillus stratosphericus | MW429726 | B. stratosphericus (T); type strain:41KF2a; AJ831841 | 1,000 | 0,5 | |
| CPO 4.283 | TSA | Neobacillus drentensis | MW429749 | AB681783 N. drentensis NBRC | 1,000 | 3 | |
| CPO 4.294 | TSA | Bacillus sp. | MW429760 | Bacillus sp. 1324(2012); JN645973 | 0,973 | 3 | |
| CPO 4.257 | EM | Bacillus sp. | MW429723 | Bacillus sp. CNJ842 PL04; DQ448790 | 0,959 | 2 | |
| CPO 4.254 | EM | Agrobacterium pusense | MW429720 | FJ969841 A. pusense NRCPB10 | 1,000 | 2 | |
| CPO 4.259 | EM | B. stratosphericus | MW429725 | B. stratosphericus; DN14_7A9; KP769446 | 1,000 | 2 | |
| CPO 4.279 | EM | Bacillus sp. | MW429745 | Bacillus sp. A3-30; JF900030 | 0,978 | 3 | |
| CPO 4.307 | EM | Paenibacillus illinoisensis | MW429773 | P. illinoisensis (T); JCM 9907; AB073192 | 0,969 | 0,5 | |
| CPO 4.276 | EM | B. subtilis | MW429742 | B. subtilis; C-3; AB110598 | 1,000 | 3 | |
| CPO 4.266 | EM | Metabacillus sp. | MW429732 | AY998119 Metabacillus niabensis 4T19 | 1,000 | 3 | |
| CPO 4.245 | EM | B. stratosphericus | MW429711 | B. stratosphericus (T); type strain:41KF2a; AJ831841 | 1,000 | 3 | |
| CPO 4.308 | EM | B. subtilis | MW429774 | B. subtilis; C-3; AB110598 | 1,000 | 0,5 | |
| CPO 4.286 | EM | B. subtilis | MW429752 | B. subtilis; C-3; AB110598 | 1,000 | 0,5 | |
| CPO 4.295 | EM | Bacillus sp. | MW429761 | Bacillus sp. 1324(2012); JN645973 | 0,973 | 0,5 | |
| CPO 4.292 | EM | B. subtilis | MW429758 | B. subtilis; C-3; AB110598 | 1,000 | 3 | |
| CPO 4.261 | EM | N. drentensis | MW429727 | AB681783 N. drentensis NBRC | 1,000 | 3 | |
| CPO 4.251 | EM | B. licheniformis | MW429717 | B. licheniformis; YW1257; AF516177 | 1,000 | 0,5 | |
| CPO 4.264 | EM | B. subtilis | MW429730 | B. subtilis; C-3; AB110598 | 1,000 | 3 | |
| CPO 4.252 | R2A | Inquilinus sp. | MW429718 | Inquilinus sp. RB35; DQ512800 | 0,980 | 0,5 | |
| CPO 4.263 | R2A | B. subtilis | MW429729 | B. subtilis; C-3; AB110598 | 1,000 | 3 | |
| CPO 4.293 | R2A | Bacillus sp. | MW429759 | Bacillus sp. RIC12-Oh014; KC535085 | 0,978 | 3 | |
| CPO 4.247 | R2A | B. stratosphericus | MW429713 | B. stratosphericus; DN14_7A9; KP769446 | 1,000 | 3 | |
| CPO 4.300 | R2A | Bacillus sp. | MW429766 | Bacillus sp. 1324(2012); JN645973 | 0,973 | 2 | |
| CPO 4.265 | R2A | Bacillus sinesaloumensis | MW429731 | LT671591 B. sinesaloumensis Marseille P3516T | 1,000 | 0,5 | |
| CPO 4.248 | R2A | B. stratosphericus | MW429714 | B. stratosphericus; LAMA 927; KC583210 | 1,000 | 0,5 | |
| CPO 4.249 | R2A | B. stratosphericus | MW429715 | B. stratosphericus; DN14_7A9; KP769446 | 1,000 | 3 | |
| CPO 4.289 | R2A | Staphylococcus epidermidis | MW429755 | S. epidermidis; RW35; EU419922 | 1,000 | 0,5 | |
| CPO 4.258 | R2A | Bacillus safensis | MW429724 | AF234854 B. safensis FO-036b | 1,000 | 3 | |
| CPO 4.250 | R2A | Priestia sp. | MW429716 | MN456843 Priestia megaterium ATCC 12872 | 0,990 | 3 | |
| CPO 4.304 | R2A | P. illinoisensis | MW429770 | P. illinoisensis (T); JCM 9907; AB073192 | 0,969 | 0,5 | |
| CPO 4.273 | R2A | Streptomyces caeruleatus | MW429739 | GQ329712 S. caeruleatus GIMN4.002 | 0,750 | 3 | |
| CPO 4.275 | R2A | B. stratosphericus | MW429741 | NR_042336 B. stratosphericus 41KF2a | 1,000 | 0,5 | |
| CPO 4.287 | R2A | Bacillus sp. | MW429753 | Bacillus sp. CH2; KC442367 | 0,973 | 0,5 | |
| CPO 4.305 | R2A | Staphylococcus sp. | MW429771 | Staphylococcus sp. NIPHL090904/B4; AY748913 | 1,000 | 0,5 | |
| CPO 4.268 | R2A | B. subtilis | MW429734 | B. subtilis; C-3; AB110598 | 1,000 | 3 | |
| CPO 4.290 | R2A | Bacillus sp. | MW429756 | Bacillus sp. 1324(2012); JN645973 | 0,973 | 2 | |
| CPO 4.256 | R2A | B. stratosphericus | MW429722 | NR_042336 B. stratosphericus 41KF2a | 1,000 | 0,5 | |
| Sitio B | CPO 4.301 | R2A | Metabacillus sp. | MW429767 | AY998119 M. niabensis 4T19 | 1,000 | 3 |
| conservado, | CPO 4.262 | R2A | B. safensis | MW429728 | AF234854 B. safensis FO-036b | 1,000 | 3 |
| Nunkiní | CPO 4.299 | R2A | Staphylococcus sp. | MW429765 | Staphylococcus sp. NIPHL090904/B4; AY748913 | 1,000 | 3 |
| CPO 4.303 | R2A | N. drentensis | MW429769 | AB681783 N. drentensis NBRC | 1,000 | 3 | |
| CPO 4.246 | R2A | N. drentensis | MW429712 | AB681783 N. drentensis NBRC | 1,000 | 3 | |
| CPO 4.296 | R2A | Bacillus sp. | MW429762 | Bacillus sp. 1324(2012); JN645973 | 0,973 | 3 | |
| CPO 4.253 | EM | P. illinoisensis | MW429719 | P. illinoisensis (T); JCM 9907; AB073192 | 0,969 | 0,5 | |
| CPO 4.298 | EM | Bacillus sp. | MW429764 | Bacillus sp. 1324(2012); JN645973 | 0,973 | 0,5 | |
| CPO 4.278 | EM | B. stratosphericus | MW429744 | NR_042336 B. stratosphericus 41KF2a | 1,000 | 2 | |
| CPO 4.267 | TSA | Gracilibacillus sp. | MW429733 | Gracilibacillus sp. BH235; AY762980 | 1,000 | 2 | |
| CPO 4.255 | TSA | B. stratosphericus | MW429721 | B. stratosphericus (T); type strain:41KF2a; AJ831841 | 1,000 | 3 | |
| CPO 4.306 | TSA | B. stratosphericus | MW429772 | NR_042336 B. stratosphericus 41KF2a | 1,000 | 0,5 | |
| CPO 4.291 | TSA | B. subtilis | MW429757 | B. subtilis; IP; DQ452512 | 1,000 | 3 | |
| CPO 4.280 | TSA | Bacillus sp. | MW429746 | Bacillus sp. A3-30; JF900030 | 0,978 | 2 | |
| CPO 4.271 | TSA | Bacillus sp. | MW429737 | Bacillus sp. MB-12; AF326361 | 0,962 | 2 |
TSA: agar tripeteína de soja; R2A: medio agar R2A; EM: agar extracto de malta, suplementados con 0,5M de NaCl;
A. pusense: Agrobacterium pusense; B. licheniformis: Bacillus licheniformis; B.stratosphericus: Bacillus stratosphericus; B. subtilis: Bacillus subtilis; G. phocaeensis: Gracilibacillus phocaeensis; M. niabensis: Metabacillus niabensis; NaCl: cloruro de sodio; NCBI: National Center for Biotechnology Information; N. drentensis: Neobacillusdrentensis; P. illinoisensis: Paenibacillusillinoisensis; B. sinesaloumensis: Bacillus sinesaloumensis; S. epidermidis: Staphylococcusepidermidis; B. safensis: Bacillus safensis; S. caeruleatus: Streptomyces caeruleatus.
El análisis de secuenciación permitió detectar que las 64 cepas aisladas presentaban una máxima similitud (0,959-1,000) con secuencias previamente reportadas. El género predominante correspondió a Bacillus y las especies más frecuentemente encontradas correspondieron a especies aún no descritas, que se nombraron como Bacillus sp. (CPO 4.257, CPO 4.277, CPO 4.279, CPO 4.281, CPO 4.282, CPO 4.284, CPO 4.285, CPO 4.287, CPO 4.288, CPO 4.290, CPO 4.293, CPO 4.294, CPO 4.295, CPO 4.300, CPO 4.271, CPO 4.280, CPO 4.296 y CPO 4.298). Dentro de especies ya descritas, predominaron aquellas asignables a Bacillus stratosphericus (CPO 4.245, CPO 4.247, CPO 4.248, CPO 4.249, CPO 4.256, CPO 4.259, CPO 4.260, CPO 4.275, CPO 4.255, CPO 4.278 y CPO 4.306) y B. subtilis (CPO 4.263, CPO 4.264, CPO 4.268, CPO 4. 276, CPO 4.286, CPO 4.291, CPO 4.292, CPO 4.297 y CPO 4.308). Entre las especies menos frecuentes se encontraron Bacillus licheniformis (CPO 4.251, CPO 4.274 y CPO 4.302), Paenibacillus illinoisensis (CPO 4.253, CPO 4.304 y CPO 4.307), Bacillus safensis (CPO 4.258 y CPO 4.262), Virgibacillus sp. (CPO 4.270 y CPO 4.272), Bacillus sinesaloumensis (CPO 4.265), Gracilibacillus phocaeensis (CPO 4.269) y Gracilibacillus sp. (CPO 4.267). Otras especies identificadas por secuenciación fueron Neobacillus drentensis (CPO 4.246, CPO 4. 283, CPO 4.261 y CPO 4.303), Metabacillus sp. (CPO 4.266 y CPO 4.301) y Priestia sp. (CPO 4. 250), que fueron recientemente reclasificadas como nuevos géneros15,32 (fig. 1). De igual forma, se encontraron otros géneros que se agruparon con bacterias gram positivas, entre las que se cuentan Streptomyces caeruleatus (CPO 4.273), Staphylococcus epidermis (CPO 4.289) y Staphylococcus sp. (CPO 4.299 y CPO 4.305); entre las gram negativas, se detectaron Inquilinus sp. (CPO 4.252) y Agrobacterium pusense (CPO 4.254) (tabla 1, fig. 2).
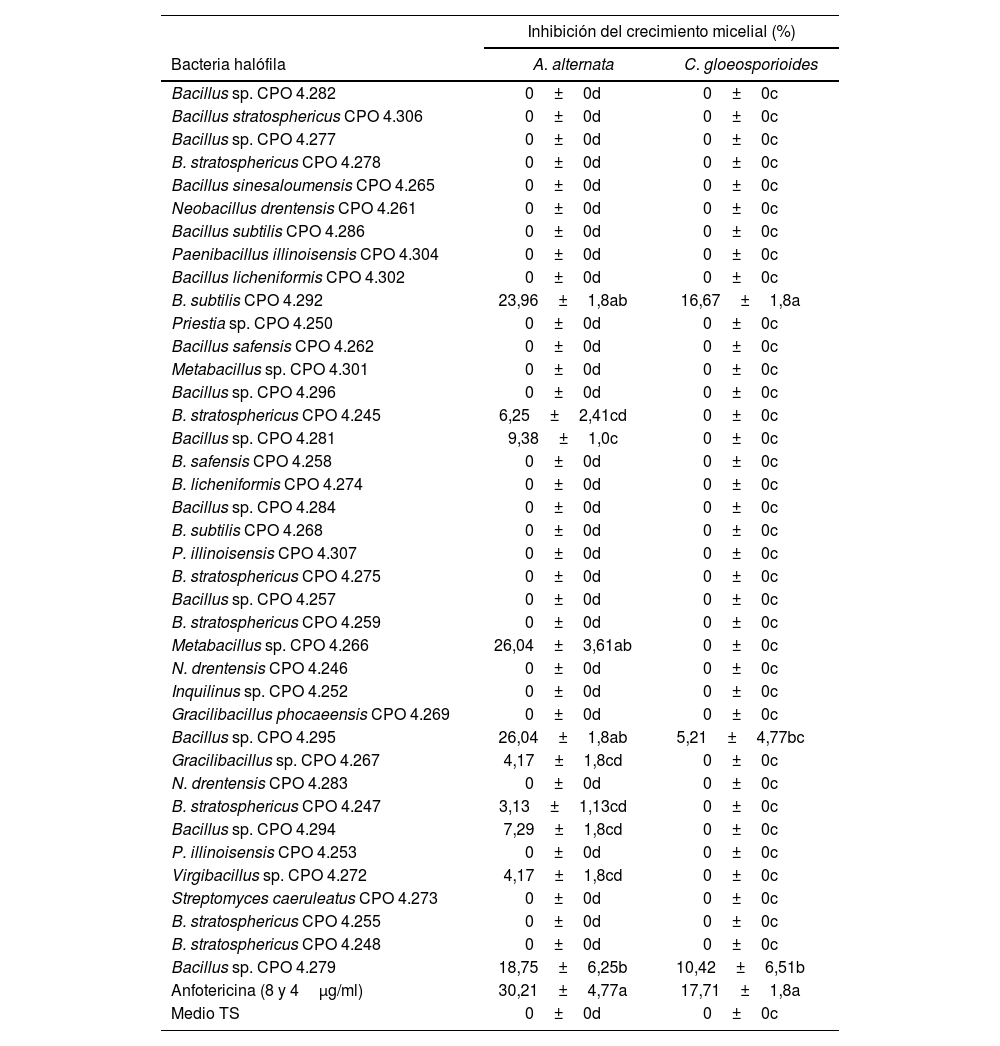
Del total de cepas bacterianas halófilas evaluadas (39), el 28% presentó actividad antifúngica contra alguno de los dos hongos fitopatógenos evaluados. El análisis estadístico de la ICM contra A. alternata indicó que las cepas con mayor acción antifúngica correspondieron a Metabacillus sp. CPO 4.266, Bacillus sp. CPO 4.295 y B. subtilis CPO 4.292, las que tuvieron un desempeño equivalente al control positivo de anfotericina B (4μg/ml) (p ≤ 0,05). Contra C. gloeosporioides, fue B. subtilis CPO 4.292 el aislamiento que exhibió la mayor actividad antifúngica, con una acción igual a la de la anfotericina B (8μg/ml). Asimismo, la cepa Bacillus sp. CPO 4.279 mostró una actividad antifúngica media contra ambos fitopatógenos (tabla 2).
Inhibición del crecimiento micelial de Alternaria alternata y Colletotrichum gloesporioides por el SC de las bacterias halófilas
| Inhibición del crecimiento micelial (%) | ||
|---|---|---|
| Bacteria halófila | A. alternata | C. gloeosporioides |
| Bacillus sp. CPO 4.282 | 0±0d | 0±0c |
| Bacillus stratosphericus CPO 4.306 | 0±0d | 0±0c |
| Bacillus sp. CPO 4.277 | 0±0d | 0±0c |
| B. stratosphericus CPO 4.278 | 0±0d | 0±0c |
| Bacillus sinesaloumensis CPO 4.265 | 0±0d | 0±0c |
| Neobacillus drentensis CPO 4.261 | 0±0d | 0±0c |
| Bacillus subtilis CPO 4.286 | 0±0d | 0±0c |
| Paenibacillus illinoisensis CPO 4.304 | 0±0d | 0±0c |
| Bacillus licheniformis CPO 4.302 | 0±0d | 0±0c |
| B. subtilis CPO 4.292 | 23,96±1,8ab | 16,67±1,8a |
| Priestia sp. CPO 4.250 | 0±0d | 0±0c |
| Bacillus safensis CPO 4.262 | 0±0d | 0±0c |
| Metabacillus sp. CPO 4.301 | 0±0d | 0±0c |
| Bacillus sp. CPO 4.296 | 0±0d | 0±0c |
| B. stratosphericus CPO 4.245 | 6,25±2,41cd | 0±0c |
| Bacillus sp. CPO 4.281 | 9,38±1,0c | 0±0c |
| B. safensis CPO 4.258 | 0±0d | 0±0c |
| B. licheniformis CPO 4.274 | 0±0d | 0±0c |
| Bacillus sp. CPO 4.284 | 0±0d | 0±0c |
| B. subtilis CPO 4.268 | 0±0d | 0±0c |
| P. illinoisensis CPO 4.307 | 0±0d | 0±0c |
| B. stratosphericus CPO 4.275 | 0±0d | 0±0c |
| Bacillus sp. CPO 4.257 | 0±0d | 0±0c |
| B. stratosphericus CPO 4.259 | 0±0d | 0±0c |
| Metabacillus sp. CPO 4.266 | 26,04±3,61ab | 0±0c |
| N. drentensis CPO 4.246 | 0±0d | 0±0c |
| Inquilinus sp. CPO 4.252 | 0±0d | 0±0c |
| Gracilibacillus phocaeensis CPO 4.269 | 0±0d | 0±0c |
| Bacillus sp. CPO 4.295 | 26,04±1,8ab | 5,21±4,77bc |
| Gracilibacillus sp. CPO 4.267 | 4,17±1,8cd | 0±0c |
| N. drentensis CPO 4.283 | 0±0d | 0±0c |
| B. stratosphericus CPO 4.247 | 3,13±1,13cd | 0±0c |
| Bacillus sp. CPO 4.294 | 7,29±1,8cd | 0±0c |
| P. illinoisensis CPO 4.253 | 0±0d | 0±0c |
| Virgibacillus sp. CPO 4.272 | 4,17±1,8cd | 0±0c |
| Streptomyces caeruleatus CPO 4.273 | 0±0d | 0±0c |
| B. stratosphericus CPO 4.255 | 0±0d | 0±0c |
| B. stratosphericus CPO 4.248 | 0±0d | 0±0c |
| Bacillus sp. CPO 4.279 | 18,75±6,25b | 10,42±6,51b |
| Anfotericina (8 y 4μg/ml) | 30,21±4,77a | 17,71±1,8a |
| Medio TS | 0±0d | 0±0c |
Los valores representan el promedio de tres repeticiones±error estándar. Las diferentes letras dentro de la misma columna indican diferencias significativas (Tukey, p<0,05).
A. alternata: Alternaria alternata; C. gloeosporioides: Colletotrichum gloeosporioides; B. stratosphericus: Bacillus stratosphericus; B. subtilis: Bacillus subtilis; N. drentensis: Neobacillus drentensis; P. illinoisensis: Paenibacillus illinoisensis; B. safensis: Bacillussafensis; SC: sobrenadante de cultivo; TS: tripeteína de soja.
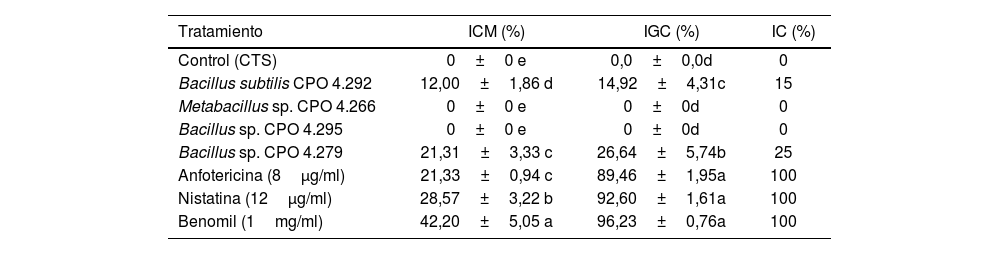
Las cuatro cepas más activas se cultivaron de nuevo para obtener los SC masivos y se evaluaron nuevamente contra C. gloeosporioides, para seleccionar la cepa más activa para el proceso de fraccionamiento. Los resultados indicaron que el SC de Bacillus sp. CPO 4.279 presentó la mayor ICM en el ensayo de difusión en pozo, la mayor IGC y la IC en el ensayo en microdilución (tabla 3, Anexo, fig. S2).
Selección de la bacteria con mayor actividad antifúngica contra Colletotrichum gloeosporioides
| Tratamiento | ICM (%) | IGC (%) | IC (%) |
|---|---|---|---|
| Control (CTS) | 0±0 e | 0,0±0,0d | 0 |
| Bacillus subtilis CPO 4.292 | 12,00±1,86 d | 14,92±4,31c | 15 |
| Metabacillus sp. CPO 4.266 | 0±0 e | 0±0d | 0 |
| Bacillus sp. CPO 4.295 | 0±0 e | 0±0d | 0 |
| Bacillus sp. CPO 4.279 | 21,31±3,33 c | 26,64±5,74b | 25 |
| Anfotericina (8μg/ml) | 21,33±0,94 c | 89,46±1,95a | 100 |
| Nistatina (12μg/ml) | 28,57±3,22 b | 92,60±1,61a | 100 |
| Benomil (1mg/ml) | 42,20±5,05 a | 96,23±0,76a | 100 |
Los valores representan el promedio de tres repeticiones±error estándar. Las diferentes letras dentro de la misma columna indican diferencias significativas (Tukey, p <0,05).
CTS: caldo de tripticasa soya; ICM: inhibición del crecimiento micelial; IGC: inhibición de la germinación de conidios; IC: inhibición del crecimiento por el método de microdilución.
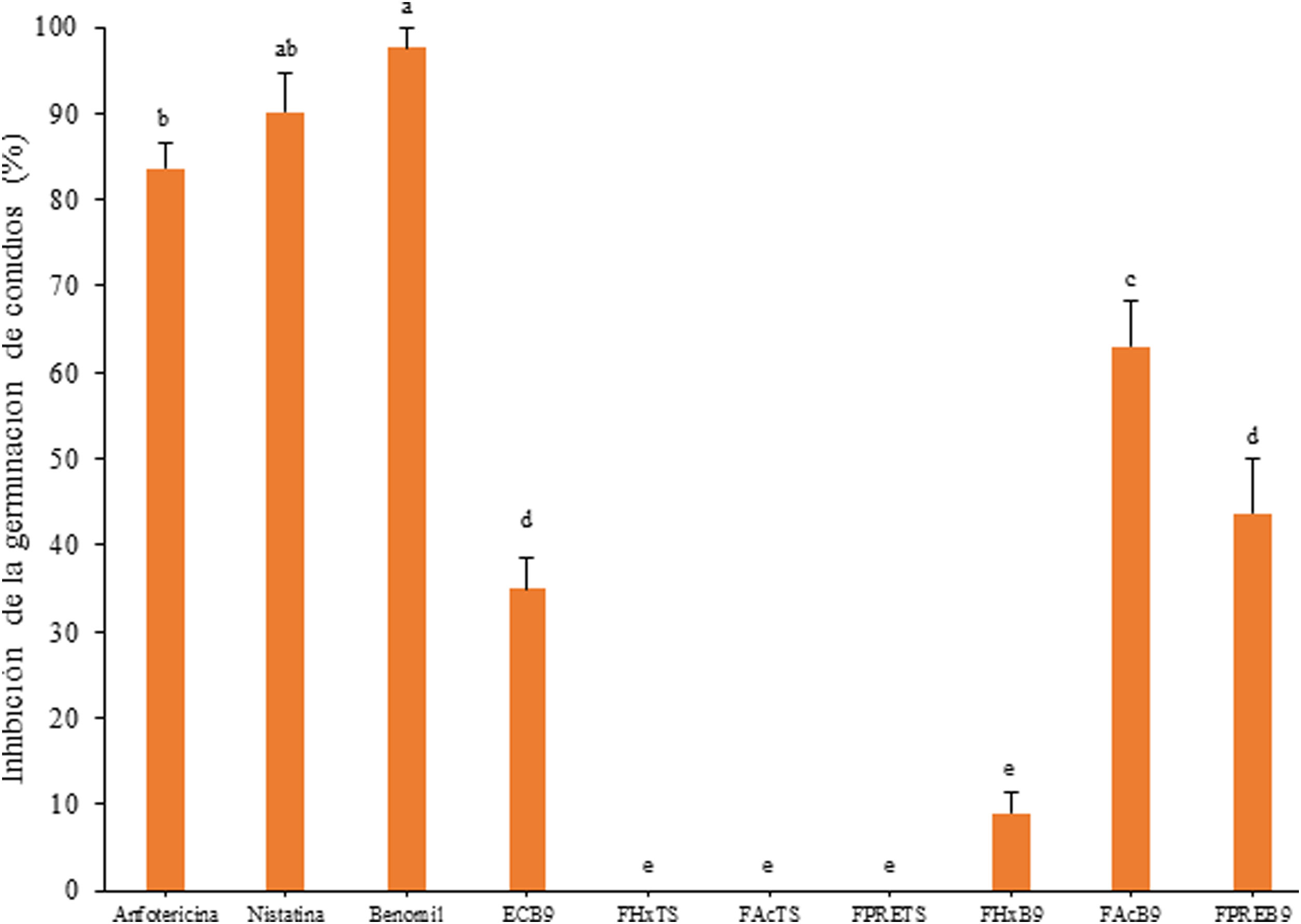
El extracto crudo de AcOEt (EcB9) obtenido del SC de Bacillus sp. CPO 4.279 presentó una CIM de 10μg/ml para inhibir el crecimiento de C. gloeosporioides por el método de microdilución (Anexo, fig. 3S). De igual forma, presentó una CI50 y CI90 de 8,63μg/ml y 10,72μg/ml, respectivamente, para inhibir la germinación de conidios de C. gloeosporioides. La fracción del extracto EcB9 con mayor IGC correspondió a la de mediana polaridad (FAcB9), con 62,99±5,36% de inhibición, un valor significativamente diferente del registrado con el resto de las fracciones (p ≤ 0,05). El precipitado (FPREB9) y el extracto (EcB9) presentaron una actividad moderada, con 43,65±6,41% y 35,00±3,61% de inhibición, respectivamente, valores significativamente superiores al de la fracción hexánica (FHxB9), cuya IGC fue del 8,98±2,48%. Las fracciones del blanco no mostraron actividad antifúngica (fig. 3).
Inhibición de la germinación de conidios de C. gloeosporioides por el extracto crudo de Bacillus sp. CPO 4.279 (EcB9) a una concentración de 8,0μg/ml y de sus distintas fracciones orgánicas: FHxB9: baja polaridad; FAcB9: mediana polaridad; FPREB9: precipitado. Control negativo: mismas fracciones orgánicas del CTS. Control positivo: anfotericina B (8μg/ml), nistatina (12μg/ml) y benomil (1,0μg/ml). Las letras indican las diferencias significativas (Tukey, p <0,05). CTS: caldo de tripticasa soya; C. gloeosporioides: Colletotrichum gloeosporioides.
La presente investigación corresponde a la primera prospección de bacterias en suelos halófilos del trópico mexicano enfocada en la búsqueda de nuevos agentes antifúngicos. En los suelos salinos de la RBLP, las cepas bacterianas halófilas aisladas se agruparon dentro de tres principales filos, Bacillota (= Firmicutes), que incluyó los géneros Bacillus, Gracilibacillus, Metabacillus, Neobacillus, Paenibacillus, Priesta, Staphylococcus y Virgibacillus; Pseudomonadata (= Proteobacteria), representada por una sola cepa de los géneros Agrobacterium e Inquilinus, y, finalmente, una cepa bacteriana del filo Actinomycetota (= Actinobacteria), identificada como del género Streptomyces.
En los sitios muestreados, Bacillus aparece como el género dominante; sin embargo, algunas cepas no se lograron identificar a nivel especie, ya que sus secuencias no han sido descritas aún en la literatura. En otros suelos salinos se han encontrado como dominantes especies de Bacillus2,19,48, lo que se ha atribuido a su reconocida plasticidad metabólica para sobrevivir bajo diversas condiciones ambientales6,18. En el presente estudio, las cepas con mayor actividad también correspondieron a Bacillus; sin embargo, se observó una variación en la actividad antifúngica del SC. Esto indica que la producción de los metabolitos secundarios en estas cepas es inestable cuando se producen en una escala masiva, por lo que el proceso de producción del SC aún debe estandarizarse. A pesar de esto, el SC masivo de la cepa Bacillus sp. CPO 4.279 presentó una mayor actividad antifúngica contra C. gloeosporioides, lo que sugiere que esta cepa posee una mayor estabilidad en la producción de metabolitos secundarios. En este sentido, los halos de ICM observados en el ensayo de difusión en pozo por la cepa Bacillus sp. CPO 4.279 sugieren la secreción constitutiva de metabolitos extracelulares antifúngicos. De forma similar, Irshad et al.19 encontraron que las bacterias halófilas Streptomyces sampsonii M1C-15 y Marinobacter flavimaris M1A-1 mostraron capacidad antifúngica contra C. gloeosporioides. Torres et al.45 reportaron que la cepa halotolerante Bacillus velezensis inhibió el crecimiento micelial de A. alternata. Esto indica que las bacterias halófilas son capaces de producir y secretar metabolitos con actividad antifúngica.
Asimismo, el extracto (EcB9) del SC de la cepa Bacillus sp. CPO 4.279 presentó actividad antifúngica contra C. gloesporioides. Sin embargo, se observó una CIM más alta que la reportada previamente para dos cepas halófilas de Streptomyces2,17. Esto daría cuenta de la presencia de metabolitos secundarios con actividad antifúngica en el extracto EcB9, aunque en muy bajas concentraciones, por lo que se realizó el fraccionamiento del extracto para potenciar la actividad antifúngica. El fraccionamiento permitió aumentar la actividad antifúngica, lo que se evidenció por la IGC de C. gloeosporioides (fig. 3). Además, se observó que los compuestos de mediana polaridad provenientes de la fracción de AcN (FAcB9) son los principales responsables de la actividad antifúngica.
Actualmente, existen pocos estudios sobre la actividad antifúngica de fracciones orgánicas de cepas halófilas de Bacillus spp. contra C. gloeosporioides. Los resultados del presente trabajo son concordantes con los de estudios previos que han mostrado que las fracciones orgánicas de cepas mesófilas de Bacillus spp. tienen actividad antifúngica contra este hongo. De igual manera, Park et al.31 observaron una disminución notable en el crecimiento micelial de C. gloeosporioides por la fracción de mediana polaridad obtenida de AcN de la cepa B.velezensis GH1-13, lo que revela que las principales sustancias antifúngicas contenidas en esta fracción son lipopéptidos cíclicos de tipo surfactina. Por otro lado, en el presente estudio, la fracción de baja polaridad (FHxB9) de Bacillus sp. CPO 4.279 presentó baja actividad antifúngica, contrariamente a lo informado por Matloub et al.25, quienes documentaron que la fracción Hx del SC de B.subtilis NCIB 3610 presentó los mayores halos de inhibición (10mm) contra Aspergillusniger mediante el ensayo de difusión en pozo. Estas diferencias pueden deberse a la naturaleza de los compuestos producidos por las diferentes cepas de Bacillus y el blanco evaluado. En bacterias halófilas del género Bacillus con actividad antifúngica, se han detectado compuestos bioactivos como 2,4-di-terc-butilfenol, 1-nonilcicloheptano, 1,3-di-terc-butilbenceno, iturina F1, iturina F2, iturina A9, iturina A8, subtilisina A, glucoproteínas y proteínas24,27,41. Estos reportes nos sugieren que la cepa Bacillus sp. CPO 4.279 también podría ser productora de estos tipos de compuestos antifúngicos.
ConclusionesEn el presente estudio se efectuó la caracterización molecular de 64 bacterias halófilas aisladas de suelos salinos en ambientes tropicales en Campeche, México. Las cepas Bacillus sp. CPO 4.279, Bacillus sp. CPO 4.295, B. subtilis CPO 4.292 y Metabacillus sp. CPO 4.266 fueron capaces de inhibir el crecimiento de A. alternata y C. gloeosporioides. El análisis biodirigido del extracto crudo de AcOEt de Bacillus sp. CPO 4.279 indicó la presencia de una fracción orgánica de mediana polaridad con capacidad de inhibir la germinación de C. gloeosporioides. En suma, Bacillus sp. CPO 4.279 presenta potencial para la producción de compuestos con actividad antifúngica contra C. gloeosporioides, por lo que es conveniente avanzar en la caracterización de los metabolitos implicados. Las bacterias halófilas del trópico mexicano poseen alto potencial para desarrollar productos alternativos en el control de C. gloeosporioides, agente causal de antracnosis en cultivos agrícolas de importancia.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Agradecemos al Consejo Nacional de Ciencia y Tecnología por la beca de maestría concedida al primer (1099478) y segundo autor (723269); así como al proyecto Cátedra CONACYT 364 «Reconversión productiva sustentable para el desarrollo de los productores rurales de Campeche» del cual es integrante el autor de correspondencia de este manuscrito. Además, agradecemos al Consejo Nacional de Humanidades, Ciencias y Tecnologías (CONAHCYT) por la beca otorgada para realizar los estudios de posgrado.