Las infecciones hospitalarias causadas por bacilos gram negativos resistentes a carbapenems (BGNCR) están asociadas al aumento de morbimortalidad y gasto sanitario. La identificación mediante cultivos de vigilancia y las medidas de control de infecciones permiten reducir su diseminación. El objetivo del estudio fue evaluar el impacto de un programa de vigilancia integrado a protocolos de control de infecciones sobre la incidencia de BGNCR y conocer su epidemiología molecular en una unidad de cuidados intensivos. Se realizaron auditorías seguidas de un programa de cultivo de vigilancia activa y caracterización molecular de BGNCR, antes y después de la implementación de programas de prevención y control de infecciones. El screening microbiológico se realizó en medios cromogénicos; la caracterización molecular de β-lactamasas (blaKPC, blaOXA-48-like, blaVIM, blaIMP, blaNDM, blaSHV y blaCTX-M) por PCR y la tipificación molecular por PFGE y MLST para Klebsiella pneumoniae. El protocolo desarrollado permitió reducir la colonización global de 16,92% al 9,67%. La diseminación de K. pneumoniae fue a expensas de diversos clones portadores de KPC-2 asociada a BLEE SHV-2 y CTX-M-15, y distribuidos en varios secuenciotipos (ST17, ST13, ST2256, ST353); no se observó persistencia de un clon particular y ningún aislamiento presentó factores de virulencia asociados a hipervirulencia. Los aislamientos de Acinetobacter baumannii fueron mayoritariamente productores de IMP-1. El análisis PFGE individualizó 3 clusters, asumiendo que la diseminación fue clonal. El protocolo permitió evaluar el impacto del programa sobre la incidencia de colonización por BGNCR. Demostramos que los cultivos de vigilancia activa en unidad de cuidados intensivos reducen significativamente la transmisión nosocomial de estos microorganismos.
Hospital-acquired infections caused by carbapenem-resistant Gram-negative bacteria (CRGNB) have been increasingly reported worldwide and are associated with high rates of mortality especially in intensive care units(ICUs). Early identification through rectal surveillance cultures and implementation of infection control measures(ICM) including contact precautions, staff education on cleaning and hand hygiene may reduce the spread of these microorganisms. The aim of this work was to assess the impact of enhanced ICM on CRGNB colonization and to describe the molecular epidemiology of these bacteria in a polyvalent ICU in a tertiary level hospital.
A prospective study including audits and active surveillance culture program, with molecular characterization, was conducted before and after the implementation of prevention programs and infection control measures. Microbiological screening was performed in chromogenic media; PCR targeting β-lactamases genes (blaKPC, blaNDM, blaVIM and blaOXA-48, blaSHV and blaCTX-M), molecular typing by PFGE; and MLST in K. pneumoniae were performed. CRGNB colonization was reduced from 16.92% to 9.67% upon implementing the infection control measures. In K. pneumoniae the most frequent carbapenemase type was KPC-2 associated with SHV-2 and CTX-M-15, and was disseminated in various STs (ST17, ST13, ST2256, ST353); there was no persistence of particular clones and virulence factors showed no association with hypervirulence. IMP-1 carbapenemase predominated in A. baumannii and the PFGE analysis individualized 3 clusters, assuming that the dissemination in the ICU was clonal. The early detection of patients colonized with CRBGN by using epidemiological surveillance cultures and the implementation of prophylactic measures are key to reducing the incidence of these microorganisms.
Las infecciones hospitalarias causadas por bacilos gram negativos resistentes a carbapenems (BGNCR) son notificadas cada vez más en todo el mundo11,21 y están asociadas a un aumento de la morbilidad, la mortalidad y el gasto sanitario12. La emergencia y propagación de Klebsiella pneumoniae, Pseudomonas aeruginosa y Acinetobacter baumannii resistentes a carbapenems en entornos hospitalarios, especialmente en unidades de cuidados intensivos (UCI), plantea un reto permanente y ha sido reconocida como un problema sanitario importante8,10,14,35.
La identificación temprana de BGNCR mediante cultivos de vigilancia rectal y la aplicación de medidas básicas de control de infecciones (MCI), incluidas las precauciones de contacto, el uso de desinfectantes apropiados y la educación del personal sobre la limpieza e higiene de las manos, pueden reducir la diseminación de estos microorganismos8,17,33. Varios estudios han investigado el impacto de las mejoras en las MCI, complementadas con auditorías y retroalimentación mediante reuniones multidisciplinarias sobre la eliminación/disminución de la propagación de BGNCR entre pacientes críticamente enfermos8,17,33. Sin embargo, un número limitado de estudios han examinado el efecto de la implementación de estas medidas en las tasas de incidencia de BGNCR en pacientes de UCI8. Algunos estudios realizados en nuestra región describen múltiples clones de K. pneumoniae resistentes a carbapenems productores de carbapenemasas que contribuyen a explicar la gran diversidad genética, su alto poder de diseminación y la endemicidad de estas enzimas15,32.
El objetivo principal de este estudio fue evaluar el efecto de un programa de vigilancia activa integrado a los protocolos de control de infecciones sobre la incidencia de BGNCR y conocer la epidemiología molecular de las colonizaciones por estos microorganismos en una UCI polivalente de un hospital de tercer nivel.
Materiales y métodosDiseño experimentalEste estudio se realizó en la Unidad de Cuidados Intensivos 1 (UCI1) del Hospital Ángel Cruz Padilla, Tucumán, Argentina; la sala cuenta con 18 camas para la recepción y cuidado de pacientes críticos. Se definió como paciente colonizado a todo paciente con hisopado rectal positivo para BGNCR y sin manifestaciones sistémicas atribuibles a infección por el microorganismo. El protocolo ejecutado fue aprobado por el Comité de Docencia y el Comité de Ética del hospital; las patologías por las cuales los pacientes tuvieron ingreso al servicio fueron registradas al momento de tomar la muestra en la ficha clínica. El estudio fue aprobado por el Comité de Ética y formó parte de las actividades del Comité de Control de Infecciones, por lo cual no fue necesario el consentimiento informado de los pacientes.
Se realizaron auditorías seguidas de un programa de cultivo de vigilancia activa que consistieron en la detección rectal y caracterización molecular de BGNCR antes y después de implementar un programa de prevención y control de infecciones (IPC).
Recolección de muestrasSe tomaron muestras durante 9 semanas consecutivas en 2 períodos diferentes: período 1, octubre-diciembre de 2018; período 2, abril-junio de 2019. La primera semana muestral perteneciente a cada período fue considerada como de «auditoría o sala de situación», lo que implicó recolectar hisopados rectales a todos los pacientes internados en ese momento en la sala, una sola vez y en el mismo día. Luego se procedió a implementar el protocolo de cultivo de vigilancia activa durante 8 semanas consecutivas. Los hisopados rectales se tomaron en 3 momentos de la hospitalización del paciente: al ingresar a la UCI1, a las 48h de internación y, en caso de mantenerse negativos, semanalmente mientras permanecían en la UCI1.
Screening microbiológicoLas muestras recolectadas se enriquecieron en caldo cerebro corazón (BHI) (Britania®) con 2μg/ml de ertapenem por 6h; posteriormente, se sembraron en medios de cultivo cromogénicos (CHROMagarTM mSuperCARBATM base, Medica-Tec, Argentina) y se incubaron en aerobiosis a 37°C por 24-48h; a partir de las colonias desarrolladas en el medio cromogénico, se confirmó la identificación bacteriana por espectrometría de masas acoplada al tiempo de vuelo de proteínas (MALDI-TOF) (Bruker®); se aceptó su identificación con scores superiores o iguales a 2.000, tanto para enterobacterias y P. aeruginosa como para A. baumannii9.
Sensibilidad a los antimicrobianosLos ensayos se realizaron solo frente a los aislamientos de K. pneumoniae y A. baumannii por el método automatizado Vitek 2® System (BioMerieux, Merci l’Etoile, Francia) incluyendo los siguientes antimicrobianos: ampicilina, ampicilina/sulbactam, piperacilina/tazobactam, cefalotina, cefotaxima (CTX), ceftazidima, cefepime, meropenem, imipenem (IMP), gentamicina, amikacina, trimetoprima/sulfametoxazol y ciprofloxacina. Los puntos de corte se interpretaron siguiendo el documento M100-S30 del CLSI (Clinical and Laboratory Standards Institute)7. La susceptibilidad a colistina se determinó por microdilución en caldo, según las recomendaciones del EUCAST (https://www.eucast.org/).
Caracterización molecular de β-lactamasasEl ADN bacteriano total se extrajo por boiling de colonias desarrolladas en el medio cromogénico. Los genes blaKPC, blaOXA-48-like, blaVIM, blaIMP y blaNDM fueron detectados siguiendo los protocolos desarrollados por la Administración Nacional de Laboratorios e Institutos de Salud «Dr. Carlos G. Malbrán» (http://antimicrobianos.com.ar/category/algoritmos-manuales-protocolos); blaSHV y blaCTX-M se detectaron por PCR múltiple24. Los productos de amplificación se visualizaron en un gel de agarosa al 1,5% coloreado con GelRed en un transiluminador UV, posteriormente fueron secuenciados con ABI3130CL (Applied Biosystems, EE. UU.) y las secuencias fueron analizadas en el National Center for Biotechnology Information (NCBI).
Estudio de genes de virulencia en K. pneumoniaePara caracterizar los factores de virulencia se realizaron 4 reacciones de PCR múltiple. Se estudiaron genes asociados a las siguientes características: resistencia a la fagocitosis (uge, ycfM, Wca6), fimbrias (fimH, mrkD, kpn), elementos capsulares (magA, k2A, rmpA), sideróforos (entB, iroN, iutA), estructuras de pared (WabG), metabolismo de alantoína (allS), proteínas líticas de glóbulos rojos (hly) y citotoxina (cnf-1). Los productos de PCR se analizaron en un gel con 1,8% de agarosa en TAE 1X, corrido durante 2h a 150V3.
Tipificación molecularEn K. pneumoniae se realizó por electroforesis de campo pulsado (PFGE) y MLST. El ADN genómico fue digerido con la endonucleasa de restricción XbaI (Promega) y los fragmentos separados por electroforesis en un gel de agarosa al 1% SeaKem Gold (BioRad, EE. UU.) en buffer TBE 0,5X (45mM Tris, 45mM ácido bórico, 1,9mM EDTA; pH 8.0) a 6V/cm y 14°, alternando pulsos en un ángulo de 120° cada 2,2-54,2seg por 20h. Se seleccionó un representante de cada pulsotipo para el estudio de las variables alélicas que permitieron definir el secuenciotipo (ST) utilizando la metodología descripta por el Instituto Pasteur (http://bigsdb.pasteur.fr/) y el banco de datos provisto por MLST.net.
En A. baumannii, el ADN genómico fue digerido con la endonucleasa de restricción ApaI (Promega) y los fragmentos separados por electroforesis en un gel de agarosa preparado como ya fue descripto para K. pneumoniae, pero con los pulsos cada 5-35seg por 24h.
Salmonella enterica serovariedad Braenderup H9812 fue utilizada como marcador de corte. El análisis de los fragmentos y la determinación de la relación entre los aislamientos se realizó con el software BioNumerics versión 8.0 (AppliedMaths, Kortrijk, Bélgica). Los patrones de ADN fueron interpretados según Tenover et al.29; se consideró que las cepas eran del mismo cluster si su identidad genética era superior al 75% o si presentaban menos de 3 bandas diferentes en los perfiles de PFGE.
Implementación del programa de prevención y control de infecciones (PCI)El equipo multidisciplinario involucrado estuvo conformado por infectólogos, microbiólogos, enfermeros, kinesiólogos y personal de mantenimiento. Una vez finalizada la primera auditoría (2018), durante la implementación del protocolo de vigilancia activa y en los 3 meses posteriores a su finalización (enero-abril 2019), bajo la coordinación del Comité de Control de Infecciones y el Servicio de Infectología del nosocomio, se realizaron actividades para optimizar las MCI complementadas con auditorías y reuniones multidisciplinarias.
Se trabajó sobre los siguientes aspectos: a) higiene de manos (monitorización y evaluación de la adherencia al programa de lavado de manos, se suministró jabón antibacteriano con triclosán al 0,4% p/v y solución antiséptica con gluconato de clorhexidina al 0,5% p/v, y alcohol isopropílico al 60% p/v delante de camas de UCI; b) precauciones de contacto para pacientes colonizados por BGNCR, incluyendo cohorte paciente y personal (designación de personal de enfermería para atender únicamente a los pacientes BGNCR positivos durante el mismo turno); c) limpieza ambiental, incluyó la desinfección de las camas y su entorno (solución de cloro para el suelo y paredes adyacentes o compuestos de amonio cuaternario para monitores, ventiladores, etc.), durante la hospitalización del paciente y después del alta; d) administración antimicrobiana (formulario restringido para carbapenems, piperacilina/tazobactam, cefalosporinas, fluoroquinolonas, colistina y tigeciclina); y e) educación del personal, se realizaron reuniones periódicas y multidisciplinarias para impartir educación y capacitar al personal de la UCI (especial referencia a la diseminación BGNCR y a la importancia de aplicar la MCI para reducirla al mínimo)16.
ResultadosSe incluyeron en este estudio 169 pacientes, 37 registrados durante las auditorías y 132 participantes del programa de vigilancia activa (períodos 1 y 2).
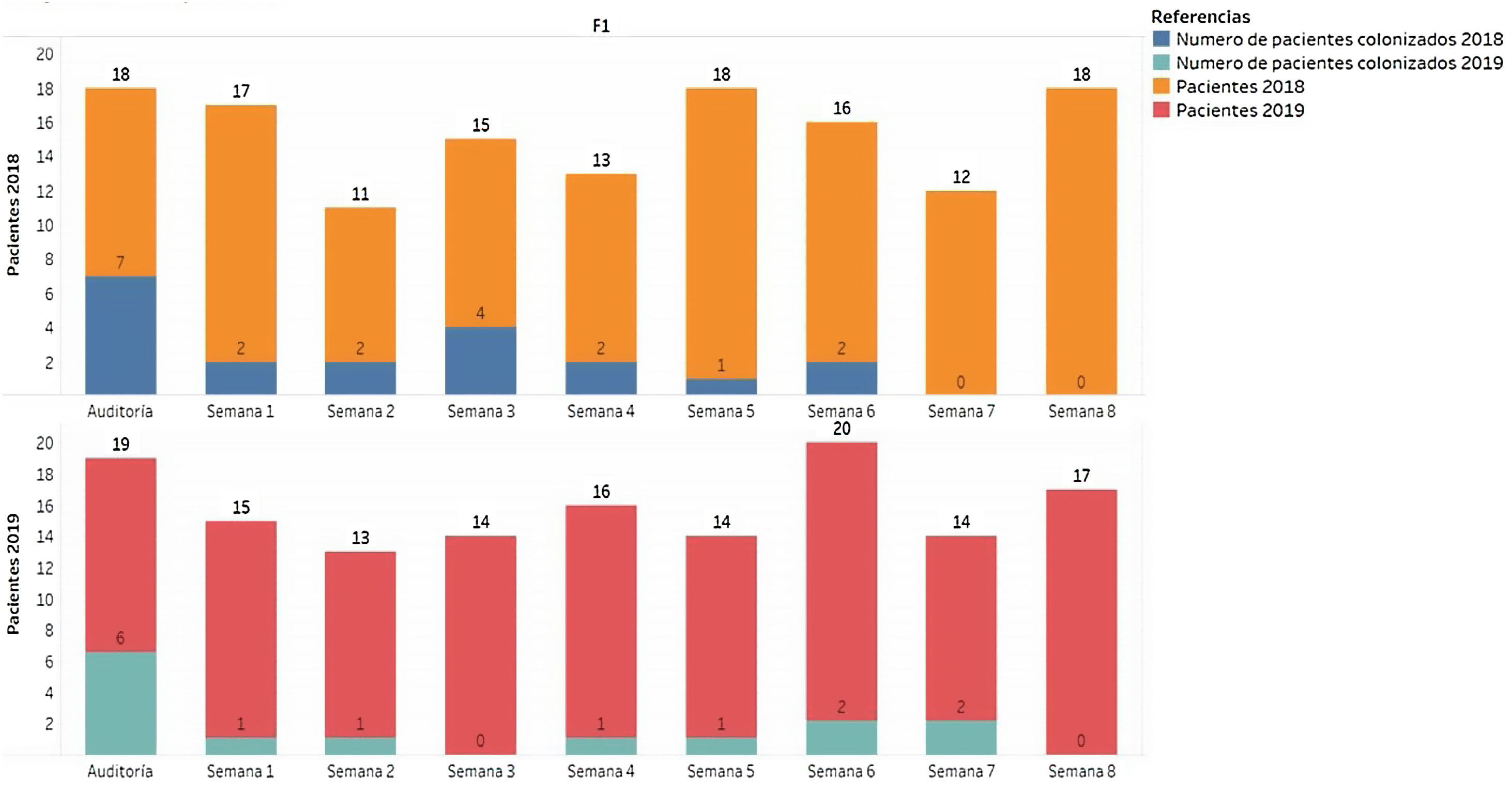
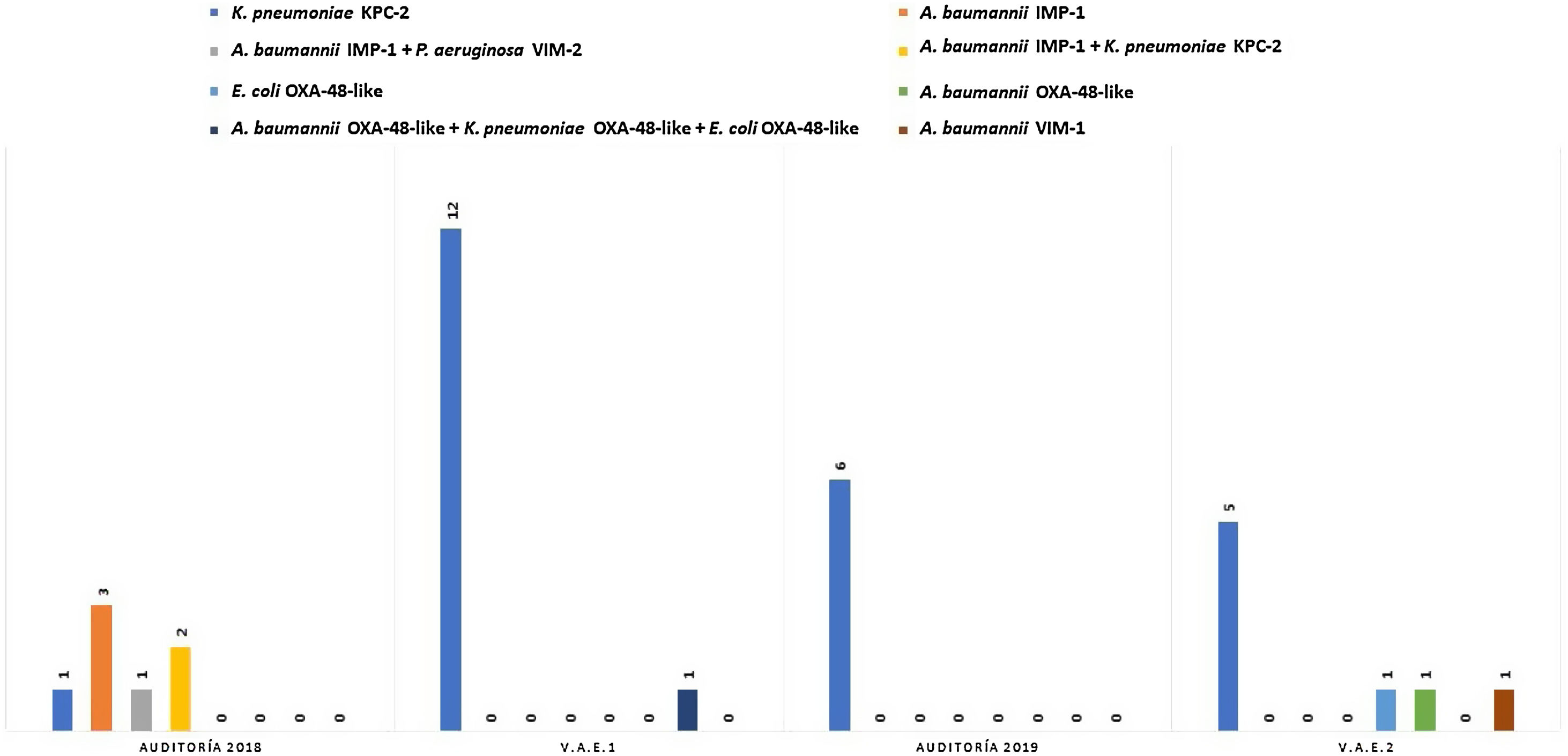
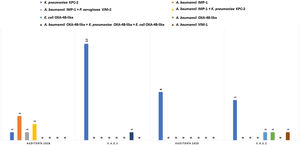
Evaluamos la distribución semanal de pacientes colonizados vs. total de pacientes muestreados a lo largo del estudio (fig. 1), además de los BGNCR aislados y las carbapenemasas asociadas a estos (fig. 2).
En la primera auditoría, 7/18 pacientes (38,9%) presentaron colonización rectal por BGNCR: A. baumannii productor de IMP-1 (n=3), K. pneumoniae KPC-2 (n=1), asociación de diferentes especies (n=3), A. baumannii IMP-1+K. pneumoniae KPC-2 (n=2) y A. baumannii IMP-1+P. aeruginosa VIM-2 (n=1). En la segunda auditoría, 6/19 pacientes resultaron colonizados (31,6%), con predominio de K. pneumoniae productor de KPC-2 (n=6). Ningún paciente presentó asociación de BGNCR en esta auditoría (fig. 2).
En el programa de vigilancia activa, 13 de los 68 pacientes (19%) que participaron en el período 1 resultaron colonizados con BGNCR, 12 (92%) con K. pneumoniae KPC-2 y un paciente (8%) simultáneamente con K. pneumoniae, Escherichia coli y A. baumannii productores de OXA-48-like. Posteriormente, en el segundo período de este programa participaron 64 pacientes, de los cuales 8 demostraron estar colonizados con BGNCR (12%), 5 con K. pneumoniae KPC-2, 2 (25%) con A. baumannii OXA-48-like y VIM-1 y un paciente (12%) con E. coli OXA-48-like (fig. 2). Los perfiles de sensibilidad a los antimicrobianos ensayados se describen en las tablas 1A y 1B del material suplementario.
Entre ambas etapas, auditorías/vigilancia activa, se implementaron mejoras y capacitaciones en el servicio, como internación por cohorte de los pacientes colonizados en un sector de la terapia; adhesión al procedimiento de lavado de manos y su correcta realización; compra de material apropiado para realizar la limpieza y división de tareas entre el personal (limpieza-alimentación-recolección de residuos), restricción del ingreso de pacientes y alumnos al servicio; limitación del flujo del personal por la unidad de terapia intensiva; disminución de la permanencia de pacientes colonizados; uso racional de antibióticos; jornadas de capacitación para el personal perteneciente a la UCI y de otros servicios asociados y reuniones multidisciplinarias para informar al equipo los resultados obtenidos; de esta manera, se pusieron en relieve las deficiencias y los problemas y se logró reforzar la aplicación de una gestión integrada.
El análisis estadístico comparando ambas etapas del programa indicó diferencias significativas y demostró una reducción de la colonización global dentro de la UCI1 del 16,92% al 9,67% (X2=0,014).
Para evaluar la efectividad global del protocolo desarrollado, no fueron considerados los aislamientos recuperados con anterioridad a las 72 horas de permanencia en el servicio ni los pacientes positivos durante las auditorías. Teniendo en cuenta esta consideración, la incidencia global de bacilos gram negativos resistentes a carbapenems en la etapa previa a implementar mejoras y capacitaciones fue del 16,92% vs. 9,67%. El coeficiente arrojado fue X2=0,014; lo que implicó diferencia significativa (X2<p).
El análisis de PFGE en A. baumannii individualizó 6 pulsotipos diferentes designados del i al vi y agrupados en 3 clusters; ningún aislamiento fue productor de las enzimas BLEE estudiadas. En la figura 3 se detallan las características clínicas y moleculares asociadas a estos aislamientos.
Características clínicas y estructura poblacional de A. baumannii resistentes a carbapenems recuperados de pacientes internados en UCI1 durante el desarrollo del protocolo de vigilancia. PLT: politraumatismo; TEC: traumatismo encéfalo-craneal; TRM: traumatismo raquimedular; V.A.E.1: vigilancia activa etapa 1; V.A.E. 2: vigilancia activa etapa 2.
A: coexistencia con P. aeruginosa VIM-2.
B: coexistencia con K. pneumoniae KPC-2
C: coexistencia con K. pneumoniae OXA-48-like y E. coli OXA-48-like.
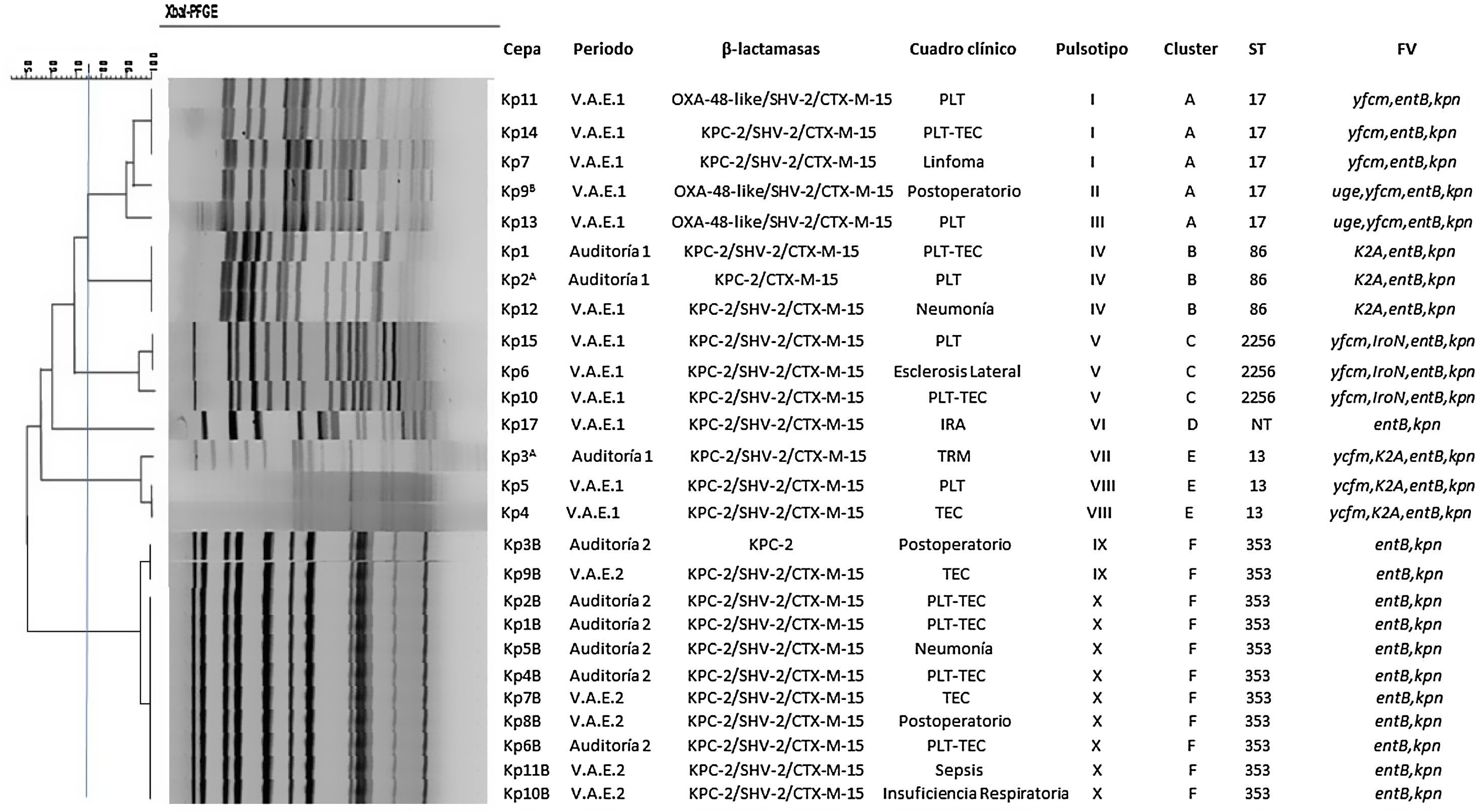
En K. pneumoniae, el análisis de PFGE individualizó 10 pulsotipos designados del i al x y agrupados en 6 clusters correspondientes a 6 secuenciotipos: ST17 (n=5), ST 86 (n=3), ST2256 (n=3), ST13 (n=3), ST353 (n=11) y un secuenciotipo no determinado NT (n=1). En la figura 4 se detallan las características clínicas y moleculares asociadas a estos.
Características clínicas y estructura poblacional de K. pneumoniae resistentes a carbapenems recuperados de pacientes internados en UCI1 durante el desarrollo del protocolo de vigilancia. V.A.E.1: vigilancia activa etapa 1; V.A.E. 2: vigilancia activa etapa 2; NT: no tipificable; IRA: insuficiencia renal aguda; PLT: politraumatismo; TEC: traumatismo encéfalo-craneal; TRM: traumatismo raquimedular; FV: factores de virulencia; uge y yfcm (resistencia a la fagocitosis), iroN y entB (sideróforos), K2A (serogrupo capsular K2), kpn (adhesina no fimbrial).
A: coexistencia con A. baumannii IMP-1; B: coexistencia con A. baumannii OXA-48-like y E. coli OXA-48-like.
Los resultados de este estudio permitieron determinar la magnitud del problema respecto de la incidencia de colonización por BGNCR en pacientes hospitalizados en la UCI y las carencias en la ejecución de un programa de control de infecciones adecuado en el nosocomio. A partir de los resultados de los cultivos de vigilancia activa y la epidemiologia molecular, se implementaron medidas de contingencia para controlar la diseminación de estos microorganismos y, finalmente, se demostró una disminución en la incidencia de colonización.
La emergencia y diseminación de BGN productores de carbapenemasas, como paradigma actual de la resistencia extrema y de la panresistencia a los antibióticos en nuestro ámbito sanitario, es una grave amenaza para la salud de los pacientes y para la salud pública.
El máximo impacto de esta problemática se debe a la dispersión de cepas de K. pneumoniae productoras de KPC-2 y de otros BGN, como A. baumannii y P. aeruginosa, productores de OXA-48-like y VIM-1. La capacidad de estas cepas para extenderse de forma rápida y eficaz hace necesaria la existencia de sistemas de alerta precoz interconectados, que nos permitan detectarlas inmediatamente y conocer su epidemiología23,34. Numerosos autores consideran que la aplicación de medidas de control de la infección es clave4,5 y requiere de un abordaje multidisciplinar, que incluya aspectos relacionados con la vigilancia (detección precoz del caso índice y detección activa de la colonización en pacientes), la implementación de precauciones estándar y de contacto, incluso de medidas de cohorting, si fuera necesario, además de la limpieza y desinfección ambiental4,5. Estos procedimientos coinciden con los implementados en este estudio, en el cual utilizamos un sistema de alerta precoz para detectar inmediatamente los pacientes colonizados, ejecutar un aislamiento de cohorte en la UCI y aplicar los protocolos establecidos en el PCI.
En un estudio reciente realizado en Nueva York en el que se comparaban las prácticas de control de infecciones entre 9 hospitales vecinos, se evidenció que los hospitales que llevaban a cabo cultivos de vigilancia activa tuvieron más éxito en la disminución de la tasa de adquisición de bacilos gram negativos productores de KPC18. Por otro lado, Thatrimontrichai et al.30. y Lledo et al.20. aseguran que la detección de portadores de BGNCR es beneficiosa en el control y la reducción de casos de infecciones asociadas, tanto en brotes como en situaciones no epidémicas, impulsando de este modo a la vigilancia activa de pacientes críticos cuando sea posible. La implementación de programas de optimización del uso de antimicrobianos es otra de las medidas que puede ayudar a limitar la selección de cepas productoras de carbapenemasas26. Con nuestro protocolo, los resultados obtenidos demostraron que las medidas adoptadas durante el estudio, en un período acotado, en conjunto con la ejecución del Programa de Uso Racional de Antimicrobianos (PROA) y la vigilancia microbiológica sistemática de los pacientes permitieron reducir significativamente la colonización global dentro de la UCI1, del 16,92% al 9,67% (X2=0,014).
Tras implementar un programa multidisciplinario de vigilancia epidemiológica, Hayden et al.13 demostraron una reducción significativa de los casos de colonización por enterobacterias productoras de KPC (50%) y de la frecuencia de infecciones asociadas a estos microorganismos (30%) en pacientes pertenecientes a un hospital de estancia prolongada; ese estudio no incorporó un programa de uso racional de los antimicrobianos o, al menos, no se hace referencia a esto.
Algunos trabajos realizados en grupos poblacionales de riesgo, como pacientes internados y neonatos, señalan que el tiempo de hospitalización es una variable asociada al riesgo de colonización por BGNCR26,28. En este sentido, Lin et al.19 aseguran que la colonización en centros sanitarios donde la internación es prolongada es cercana al 30%, contra un 3% en los centros con internaciones breves. Si bien nuestro estudio se desarrolló solo en una UCI y el número de pacientes estudiados es una limitante, el protocolo implementado en cada etapa durante 8 semanas consecutivas nos permitió observar diferentes tiempos de colonización en los pacientes admitidos. Santolin et al.27. consideran que la omisión del cultivo de vigilancia realizado a las 48-72h de la admisión en la unidad incrementaría el número de pacientes colonizados por la rápida diseminación horizontal de los BGNCR; en nuestro trabajo, solo 2 pacientes (uno correspondiente a cada etapa muestral) positivizaron sus cultivos a las 48h de permanencia en el servicio.
El estudio de epidemiología molecular muestra diferentes patrones de diseminación de BGNCR en la UCI. Por una parte, la emergencia y diseminación de K. pneumoniae se produce a expensas de diversos clones, mayoritariamente portadores de KPC-2 asociada con BLEE del tipo SHV-2 y CTX-M-15, lo que agrava el problema, por presentar múltiples resistencias a otros grupos de antimicrobianos. En nuestro hospital, esta epidemiologia fue descripta anteriormente por Jure et al.15 y Vargas et al.32., por lo cual nuestros resultados confirman que, si bien no existe la persistencia de un clon particular, el clon ST17 nuevamente se comporta como uno de los prevalentes en la región. En cuanto a la distribución geográfica, ST17 también fue descripto por Argente et al.2 en España, asociado a la producción de OXA-48, y por Andrade et al.1 en Brasil; dicho clon fue asociado a KPC-2 y no fue registrado en estudios epidemiológicos recientes realizados en Argentina por Cejas et al.6. En la base de datos del Instituto Pasteur (bigsdb.pasteur.fr) se encuentran registrados 103 aislamientos de K. pneumoniae ST17 procedentes de muestras de sangre, materia fecal, esputo, orina, abscesos y respiratorias, de pacientes colonizados e infectados. En cuanto al tipo capsular, en este trabajo no se detectaron los tipos capsulares ensayados (K1 y K2), previamente descriptos en nuestra región por Vargas et al.32, quienes informaron un aislamiento ST17 asociado al tipo capsular K2; estos resultados difieren de los de Martin et al.22, quienes reportan ST17/ K23, y de los de Andrade et al.1, quienes describen ST17/ K112.
Es importante recalcar que en el período 2, la población policlonal de K. pneumoniae descripta previamente en este hospital se ve sustituida por la presencia de un único clon, ST353. Analizando el perfil de sensibilidad, el corto período en el que fueron obtenidas estas cepas, los perfiles de restricción y secuenciotipos idénticos, asumimos que nos encontrábamos epidemiológicamente en una situación de brote por un secuenciotipo no descripto antes en nuestra región y asociado a KPC2/SHV-2 y CTX-M-15. Este secuenciotipo también fue reportado por Thomas et al.31, quienes describen un brote por K. pneumoniae ST 353, coproductora de enzimas tipo OXA-48 y SHV, en pacientes colonizados.
En cuanto a la virulencia de las cepas de K. pneumoniae colonizantes, demostramos la ausencia de genes asociados a hipervirulencia (rmpA, iutA) y a elementos fimbriales tipo 3 (genes mrk) y la presencia de genes entB y kpn, ambos ampliamente distribuidos en el género Klebsiella. Solo los aislamientos pertenecientes a los secuenciotipos ST13 y ST86 estuvieron asociados al serogrupo capsular K2.
Los aislamientos de A. baumannii fueron mayoritariamente productores de IMP-1. El análisis de PFGE individualizó 6 pulsotipos agrupados en 3 clusters, con lo que podríamos asumir que la diseminación en la UCI fue clonal, en concordancia con otros estudios que demuestran la clonalidad de estas cepas25.
En conclusión, implementamos un protocolo multidisciplinario de vigilancia adaptado a una UCI, evaluamos su impacto en la incidencia de colonización por BGNCR y demostramos que el cultivo de vigilancia activa en unidades de alto riesgo reduce significativamente la colonización por estos microorganismos; por lo tanto, el cumplimiento riguroso de medidas de control de infecciones y un protocolo de uso racional de antimicrobianos, monitorizado, son necesarios en entornos de alto riesgo para prevenir brotes o infecciones invasivas por BGNCR.
FinanciaciónEl presente trabajo ha sido financiado por un proyecto otorgado por SCAIT (Secretaría de Ciencia, Arte e Innovación Tecnológica, Tucumán, Argentina) (proyecto D640-1).
Conflicto de interesesNinguno para declarar.
Los autores expresamos nuestra gratitud al Equipo de Control de Infecciones del Hospital Ángel C. Padilla y al personal médico, de enfermería y mantenimiento de UCI1, presentes durante el desarrollo del estudio por la colaboración y predisposición demostrada.