A medida que el acceso a tratamiento antirretroviral avanza globalmente, son necesarios esfuerzos encaminados a simplificar la toma y el envío de muestras a laboratorios de referencia para realizar técnicas de diagnóstico serológico y genético, análisis de carga viral y resistencia a fármacos antirretrovirales en países con recursos limitados. Por tanto, es necesario implementar métodos más baratos y prácticos de obtención, almacenaje y transporte de muestras biológicas y/o sanguíneas. La recogida de muestras en papeles de filtro es una alternativa económica y práctica al plasma para la monitorización del tratamiento antirretroviral, principalmente en laboratorios con equipamientos mínimos y sin acceso a cadenas de frío y/o refrigeración. En esta revisión abordaremos algunas de las aplicaciones clínicas del uso de sangre total seca recogida en papeles de filtro (DBS, dried blood spots). Nos centraremos en su aplicabilidad en la monitorización de la infección por VIH para su posible aplicación por los programas nacionales de salud de los países en vías de desarrollo. Haremos un repaso a los distintos estudios que han empleado DBS en la cuantificación de la carga viral, en la monitorización de mutaciones de resistencias a antirretrovirales, en el diagnóstico perinatal temprano, en el diagnóstico serológico en adultos, en la detección de antígeno p24 viral e incluso en la identificación de variantes virales para estudios de epidemiología molecular del VIH. Hablaremos de las variables que afectan a las determinaciones hechas con DBS en el campo del VIH y explicaremos cómo se pueden optimizar los procedimientos usando DBS para aumentar su sensibilidad. El objetivo final será dar a conocer las aplicaciones y ventajas del uso de los DBS en el campo del VIH, sobre todo en países de recursos limitados.
As access to antiretroviral treatment increases in the developing countries, efforts towards making it easier and less costly to collect, store, and deliver the biological samples to reference laboratories, where the serological and genetic diagnosis techniques are performed, have become a high priority. Blood sampling on filter papers is an inexpensive and practical alternative to plasma for antiretroviral treatment monitoring in countries with limited resources and no access to cold chains or refrigeration. The main clinical applications and uses of blood-sampling onto filter papers (dried blood spots [DBS]) are reviewed, focusing on how these can be applied in monitoring HIV infection, particularly for use in National Health Programs in developing countries, or in resource-limited settings. A review is presented of studies that have used the DBS technique for quantifying viral load, analysis of antiretroviral drug-resistance mutations, early infant diagnosis, adult serological diagnosis, detection of viral p24 antigen, and molecular epidemiology of HIV-1, in different geographical locations. Those variables that could affect the use of DBS, particularly in the HIV field, as well as explaining how these procedures can be optimised to increase their sensitivity are also reviewed. The aim of this study was to review the advantages of implementing the DBS technique in the HIV field, especially in resource-constrained regions.
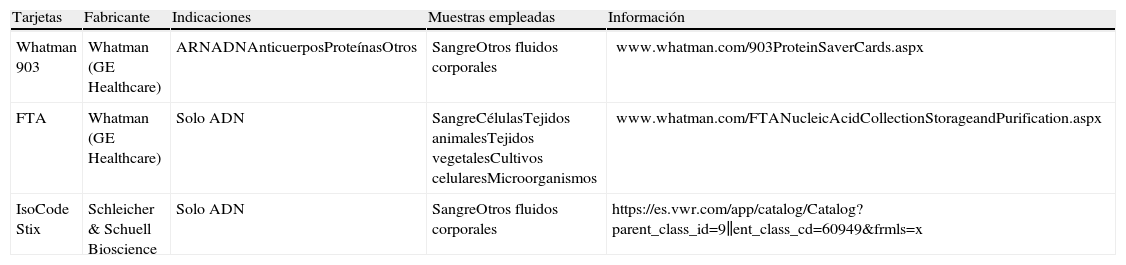
De los más de 34 millones de infectados por el virus de la inmunodeficiencia humana tipo 1 (VIH-1) en el mundo, el 93% viven en países de bajos recursos1. A medida que el acceso al tratamiento antirretroviral (TAR) avanza globalmente, también son necesarios esfuerzos encaminados a monitorizar correctamente la terapia reduciendo costes y simplificando procedimientos. La recogida de muestras en papeles de filtro está considerada una alternativa barata y práctica al plasma para la monitorización del TAR, principalmente en laboratorios con equipamientos mínimos y sin acceso a cadenas de frío y/o refrigeración2. Los tipos de tarjetas existentes para la recogida de muestras biológicas se detallan en la tabla 1. Las muestras secas, ya sean de sangre total (DBS), plasma (DPS), suero (DSS) o incluso leche materna3 de pacientes infectados y recogidas en papeles de filtro Whatman 9034 son un método accesible de toma, almacenaje y envío de muestras a laboratorios de referencia. Pueden emplearse para la cuantificación de la viremia o carga viral (CV)5–19, monitorización de mutaciones de resistencias a antirretrovirales18–29, diagnóstico perinatal7,11,30–32, diagnóstico serológico en adultos33, detección de p24 viral34 e identificación de variantes del VIH18,22–25,29. La experiencia de cribado en neonatos empleando DBS ha mostrado ser eficaz y sencilla35. Los DBS también han sido empleados para el diagnóstico de diversas enfermedades genéticas, metabólicas e infecciosas35,36. Actualmente no está aprobado el uso de los DBS para cuantificar linfocitos CD4+ por inmunoensayo, por la poca correlación con los valores obtenidos mediante citometría de flujo. Algunas aplicaciones de los DBS se resumen en la tabla 2.
Tipos de tarjetas para la recogida de muestras biológicas
| Tarjetas | Fabricante | Indicaciones | Muestras empleadas | Información |
| Whatman 903 | Whatman (GE Healthcare) | ARNADNAnticuerposProteínasOtros | SangreOtros fluidos corporales | www.whatman.com/903ProteinSaverCards.aspx |
| FTA | Whatman (GE Healthcare) | Solo ADN | SangreCélulasTejidos animalesTejidos vegetalesCultivos celularesMicroorganismos | www.whatman.com/FTANucleicAcidCollectionStorageandPurification.aspx |
| IsoCode Stix | Schleicher & Schuell Bioscience | Solo ADN | SangreOtros fluidos corporales | https://es.vwr.com/app/catalog/Catalog?parent_class_id=9∥ent_class_cd=60949&frmls=x |
Los tres dispositivos consisten en una pequeña «tarjeta» portadora que presenta un recorte de papel de algodón o nitrocelulosa adherido a un formulario para recoger la información identificativa del paciente. Las tarjetas 903 están especialmente diseñadas para la recogida y el almacenamiento de sangre para los programas de cribado neonatal. Las proteínas, ARN y otras biomoléculas se pueden archivar en papel 903 y almacenar entre 4°C y –80°C. Las tarjetas FTA están tratadas con formulaciones químicas que desnaturalizan proteínas, protegen de la degradación de ácidos nucleicos por las nucleasas y facilitan la unión de ADN. Los FTA se emplean para archivar ADN a temperatura ambiente de manera indefinida. Las tarjetas Isocode son tarjetas 903 modificadas que permiten la elución y amplificación del ADN por la reacción en cadena de la polimerasa (PCR), al tener impregnados inactivadores de muchos de los inhibidores de la PCR presentes en la sangre.
Algunas aplicaciones prácticas del uso de los DBS
| Aplicaciones de los DBS | Ejemplos |
| Diagnóstico de infecciones (serológico/genético) | VIHVHCCMVHVS-2Treponema pallidum (sífilis)Tripanosoma cruzi (Chagas)Plasmodium falciparum (malaria)Toxoplasma gondiiEsquistosomiasisetc. |
| Monitorización de la infección por HIV | Cuantificación de la carga viralIdentificación de mutaciones de resistenciaIdentificación de subtipos y recombinantes del VIH-1Estudios farmacocinéticos de antirretroviralesCuantificación de antígeno p24 viral |
| Estudios epidemiológicos de enfermedades infecciosas | |
| Estudio de marcadores de alteraciones genéticas y metabólicasa | Anemia falciforme, fenilcetonuria, fibrosis quística, hipotiroidismo neonatal, hiperfenilalalinemia, talasemia, hidrocefalia congénita, leucemia, galactosemia, diabetes tipo 1, etc. |
| Estudios de haplotipo | |
| Estudios de marcadores de enfermedad | |
| Detección y cuantificación de compuestos y drogas por HPLC |
VIH: virus de la inmunodeficiencia humana; VHC: virus de la hepatitis C; CMV: citomegalovirus; HVS-2: virus herpes simplex tipo 2; HPLC: cromatografía líquida de alta eficacia.
El uso de los DBS en lugar de sangre tiene la ventaja de emplear un menor volumen de sangre total (50-100μl) recogido mediante un pequeño pinchazo en el talón o en el dedo, evitando manejar jeringuillas y reduciendo el gasto en infraestructuras y material de laboratorio. Además, las muestras sanguíneas recogidas en papel de filtro no son infecciosas. Por ello el empleo de los DBS podría simplificar la monitorización de la infección por VIH en los programas de salud pública de países en desarrollo. El objetivo de esta revisión es detallar las posibles aplicaciones de los DBS en el diagnóstico y la monitorización de enfermedades, profundizando en su uso para el diagnóstico y la monitorización de la infección por el VIH.
Extracciones de ácidos nucleicos a partir de dried blood spots para la amplificación viralMétodos de extracción de ácidos nucleicos (ARN, ADN) a partir de dried blood spotsLa mayoría de las técnicas de cuantificación de CV, detección de resistencias, diagnóstico genético temprano perinatal y detección de variantes empleando DBS se basan en la amplificación de ácidos nucleicos, principalmente por la reacción en cadena de la polimerasa (PCR). El ARN viral (de virus circulantes) y/o el ADN proviral (de virus integrados en células sanguíneas infectadas) extraído del DBS también se puede usar para otros estudios que requieran secuenciación viral o clonaje molecular del VIH, incluso ensayos de resistencia fenotípica o detección molecular de otros virus del paciente. Sin embargo, todos ellos requieren un paso previo de extracción de material genético del DBS.
No todas las técnicas de extracción (comerciales y no comerciales) disponibles tienen la misma eficacia37. De hecho, los fallos en la extracción del ARN viral son la causa más frecuente de obtener valores de CV infraestimados, viremias negativas o fallos de amplificación en la detección de resistencias en muestras de pacientes donde se esperaban CV altas. La mayoría de estudios de determinación de la CV, resistencias y epidemiología molecular y diagnóstico genético empleando DBS extraen ácidos nucleicos por técnicas basadas en el uso de sílice38, principalmente mediante el empleo del NucliSens isolation Kit (BioMérieux).
Otros métodos de extracción (comerciales y no comerciales) de ácidos nucleicos en DBS alternativos al uso de la sílice también se han usado para cuantificar la viremia del VIH6,7,10,15,16,37 y para detectar resistencias22. La extracción por NucliSens resultó ser más precisa y sensible que otras durante la cuantificación de la viremia5 y la detección de resistencias37.
Integridad del material genético en los dried blood spotsAunque los DBS se pueden almacenar a temperatura ambiente, hay factores que hay que tener en cuenta para preservar la integridad del material genético a extraer del fluido seco en el DBS (tabla 3), y han sido extensamente estudiados6,7,13,14,26,32,37,39–42. Se ha comparado la integridad del material genético extraído del DBS considerando distintas temperaturas y grado de humedad durante la toma de la muestra, el transporte y el almacenamiento de los DBS. También se ha evaluado la eficacia de las distintas técnicas en DBS almacenados durante distintos tiempos hasta su uso, comparando las metodologías en DBS, DPS y plasma y empleando distintos números de círculos de sangre seca en la extracción. Aunque normalmente se usan dos círculos para CV y resistencias, la sensibilidad puede aumentar extrayendo ARN de un mayor número de círculos, al relacionarse con una mayor cantidad de muestra de sangre seca a partir de la cual se hará la extracción.
Variables que afectan a las determinaciones hechas con DBS en VIH
| Toma, manipulación y secado correcto del DBS |
| Volumen de sangre recogida en cada DBS |
| Temperatura, humedad y tiempo de almacenamiento hasta su uso |
| Método de extracción del material genético (ARN y/o ADN) del DBS |
| Número de círculos de sangre seca usados en la extracción |
| Preservación del material genético extraído hasta su uso |
| Uso del ADN proviral (más estable) vs. ARN viral (resistencias/diagnóstico/detección de variantes) |
| Técnica de cuantificación viral y de determinación de resistencias usada |
| Tamaño del fragmento viral amplificado del DBS |
| Variabilidad genética del VIH |
| Interferencia del ADN proviral en las determinaciones |
ADN proviral: integrado en las células infectadas de la sangre del DBS; ARN viral: extraído del plasma de la sangre del DBS.
Para una mejor recuperación del ARN viral para la detección de resistencias o de CV es muy importante señalar que los DBS deben ser almacenados lo antes posible en un congelador a –20°C, y preferiblemente a temperaturas menores (ultracongelador de –80°C). Ello contribuirá a mantener la integridad del ARN de la muestra hasta su procesamiento. Sin embargo, cuando la criopreservación no es posible, los DBS pueden ser almacenados o transportados a temperatura ambiente hasta 14 días después de su toma. Después de ese tiempo deben ser procesados o almacenados a –80°C (preferentemente) o a –20°C hasta su uso. Cabe destacar que, en muestras con baja CV, a mayor tiempo de almacenaje a temperatura ambiente, mayor es el riesgo de degradación del ARN viral del DBS.
Se sabe que el ADN es más estable que el ARN, y ello se aplica también a los DBS11. El ARN viral se degrada a mayor tiempo de almacenaje del DBS hasta su uso y, por tanto, la eficacia de cuantificación viral o de detección de resistencias puede verse afectada41. El ARN en los DBS y DPS puede ser estable al menos durante un año a –70°C18,40 o incluso más. Debemos recalcar que la estabilidad del ARN viral recuperado y el rendimiento de la cuantificación de CV y monitorización de resistencias a partir del DBS será mayor cuanto menores sean la temperatura, la humedad y el tiempo de almacenamiento del DBS hasta su procesado. La sensibilidad del ensayo aumentará cuanto mayor sea el volumen de sangre en cada círculo y el número de círculos usados para la extracción del ARN. Por otro lado, cuanto menor sea el tamaño de la región del VIH a amplificar, mayor será la eficacia de amplificación por PCR necesaria para la monitorización de resistencias y diagnóstico genético en niños. Si es posible, los DBS deberían ser almacenados una vez secos en nevera o preferentemente en congelador en el punto de recogida de muestras, almacenados en bolsas de plástico cerradas e individuales, y deberían contener una bolsa de material desecante para reducir la humedad hasta su uso.
Carga viral y dried blood spotsCarga viral del VIH: uso clínicoLa cuantificación de la CV del VIH en plasma sanguíneo es la herramienta de laboratorio recomendada para monitorizar la respuesta al TAR en pacientes infectados, detectar fracaso terapéutico y evaluar la necesidad de cambio de tratamiento. Sin embargo, en contextos con bajos recursos, esta técnica no se implementa y se siguen otros parámetros para este seguimiento. La cuantificación de la CV también ha sido empleada para el diagnóstico temprano en niños7,30,31,34. Valores de CV indetectables en plasma reflejan un adecuado control de la replicación viral en pacientes en TAR. Por el contrario, niveles detectables de viremia pueden indicar un mal control de la infección, fallos de adherencia o emergencia de virus con mutaciones de resistencia a fármacos en uso.
Ensayos de determinación de la carga viralExisten 2 metodologías disponibles para medir ARN del VIH-1 en las que se basan las técnicas de cuantificación viral comerciales en plasma. La primera metodología requiere una extracción previa de ácidos nucleicos de la muestra infectada para su amplificación posterior. La amplificación puede realizarse por transcripción inversa o RT-PCR (Amplicor HIV-1 Monitor) que puede emplear amplificación a tiempo real (Amplicor COBAS® TaqMan® HIV-1; Abbott Real Time™ m2000rt) y mediante la técnica NASBA (NucliSENS EasyQ® HIV-1). La segunda metodología se basa en métodos de amplificación de señal (VERSANT® HIV-1 RNA bDNA) y no requiere extracción previa de ácidos nucleicos.
La determinación de CV en plasma presenta altos costes y limitaciones técnicas para países con bajos recursos, y se necesitarían ensayos baratos, rápidos y fáciles de usar en países con instalaciones limitadas. Por ello se han desarrollado métodos no comerciales de menor coste22,32,43–47. Algunos están basados en la primera metodología: Generic HIV-1 viral load (Biocentric), Primagen Retina-Rainbow, RT-PCR manuales (caseras o in house) o RT-PCR a tiempo real32,44,47. Otras técnicas de cuantificación alternativas miden la actividad de la transcriptasa inversa de VIH-1 (Cavidi ExavirLoad v2 y v3)48. No obstante, la detección de falsos positivos, las necesidades de equipamiento caro y de personal cualificado siguen siendo las principales barreras para su puesta en práctica en laboratorios poco equipados o con poca experiencia en estas técnicas45. Las ventajas e inconvenientes de varios métodos de cuantificación de CV comerciales y no comerciales ya han sido publicadas43.
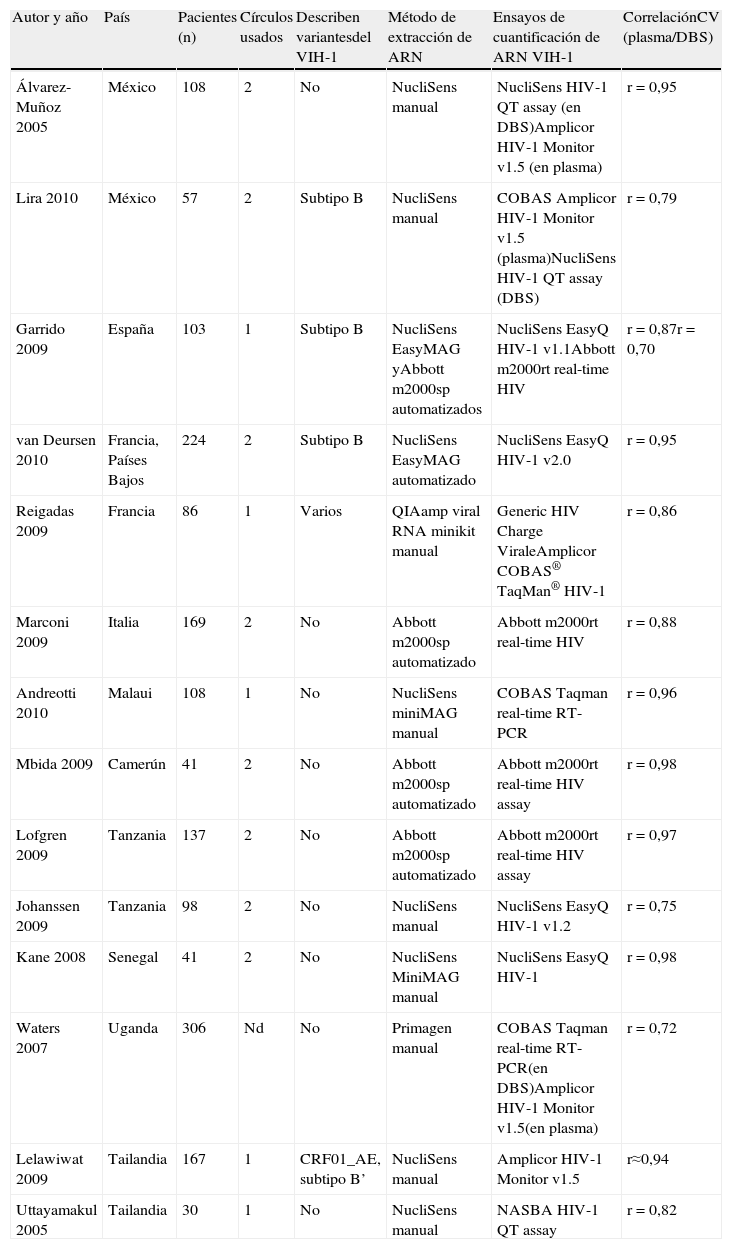
Variabilidad entre técnicas de carga viral en plasma y en dried blood spotsLas técnicas de cuantificación de CV del VIH-1 presentan diferencias en precisión y sensibilidad. Aunque la mayor parte de los artículos muestran buena correlación entre los valores de CV de plasmas y DBS5–18, algunos han observado falsos positivos10, atribuidos al efecto del ADN proviral en la cuantificación a partir de DBS en algunas muestras con CV plasmática baja5,8,13. El bajo volumen de sangre recogido en los DBS puede limitar la sensibilidad de la cuantificación de CV si se compara con la cuantificación convencional usando volúmenes de plasma mucho mayores. La sensibilidad de cualquier técnica puede disminuir si la muestra no ha sido bien conservada y principalmente en muestras con CV más bajas. No es fácil definir en qué grado las diferencias de sensibilidad empleando DBS vs. plasma observadas en estos estudios pueden ser atribuidas al método de extracción, al ensayo de CV específico, a la conservación de la muestra o incluso a la experiencia del laboratorio, al observarse también variabilidad de resultados entre laboratorios empleando las mismas muestras40. La tabla 4 resume algunos trabajos representativos que comparan la CV a partir de plasma y DBS empleando varias técnicas, principalmente comerciales. A pesar de todo, el rango de sensibilidad y la correlación de los valores de plasma deben ser determinados para cada ensayo comercial y versión. Independientemente del uso de plasma o sangre seca, es importante que no se cambie de técnica de cuantificación de viremia a la hora de monitorizar a un paciente durante su seguimiento clínico.
Comparación de distintas técnicas de cuantificación de la carga viral del VIH-1 comerciales usando DBS vs. plasma
| Autor y año | País | Pacientes (n) | Círculos usados | Describen variantesdel VIH-1 | Método de extracción de ARN | Ensayos de cuantificación de ARN VIH-1 | CorrelaciónCV (plasma/DBS) |
| Álvarez-Muñoz 2005 | México | 108 | 2 | No | NucliSens manual | NucliSens HIV-1 QT assay (en DBS)Amplicor HIV-1 Monitor v1.5 (en plasma) | r=0,95 |
| Lira 2010 | México | 57 | 2 | Subtipo B | NucliSens manual | COBAS Amplicor HIV-1 Monitor v1.5 (plasma)NucliSens HIV-1 QT assay (DBS) | r=0,79 |
| Garrido 2009 | España | 103 | 1 | Subtipo B | NucliSens EasyMAG yAbbott m2000sp automatizados | NucliSens EasyQ HIV-1 v1.1Abbott m2000rt real-time HIV | r=0,87r=0,70 |
| van Deursen 2010 | Francia, Países Bajos | 224 | 2 | Subtipo B | NucliSens EasyMAG automatizado | NucliSens EasyQ HIV-1 v2.0 | r=0,95 |
| Reigadas 2009 | Francia | 86 | 1 | Varios | QIAamp viral RNA minikit manual | Generic HIV Charge ViraleAmplicor COBAS® TaqMan® HIV-1 | r=0,86 |
| Marconi 2009 | Italia | 169 | 2 | No | Abbott m2000sp automatizado | Abbott m2000rt real-time HIV | r=0,88 |
| Andreotti 2010 | Malaui | 108 | 1 | No | NucliSens miniMAG manual | COBAS Taqman real-time RT-PCR | r=0,96 |
| Mbida 2009 | Camerún | 41 | 2 | No | Abbott m2000sp automatizado | Abbott m2000rt real-time HIV assay | r=0,98 |
| Lofgren 2009 | Tanzania | 137 | 2 | No | Abbott m2000sp automatizado | Abbott m2000rt real-time HIV assay | r=0,97 |
| Johanssen 2009 | Tanzania | 98 | 2 | No | NucliSens manual | NucliSens EasyQ HIV-1 v1.2 | r=0,75 |
| Kane 2008 | Senegal | 41 | 2 | No | NucliSens MiniMAG manual | NucliSens EasyQ HIV-1 | r=0,98 |
| Waters 2007 | Uganda | 306 | Nd | No | Primagen manual | COBAS Taqman real-time RT-PCR(en DBS)Amplicor HIV-1 Monitor v1.5(en plasma) | r=0,72 |
| Lelawiwat 2009 | Tailandia | 167 | 1 | CRF01_AE, subtipo B’ | NucliSens manual | Amplicor HIV-1 Monitor v1.5 | r≈0,94 |
| Uttayamakul 2005 | Tailandia | 30 | 1 | No | NucliSens manual | NASBA HIV-1 QT assay | r=0,82 |
Nd: no determinado; VERSANT® HIV-1 RNA bDNA (Siemens Medical Solution); Amplicor HIV-1 Monitor y COBAS Taqman real-time RT-PCR (Roche Diagnostics); Abbott RealTime m2000rt (Abbott Molecular Diagnostics); NASBA HIV-1 QT assay (Organon Técnica); NucliSens EasyQ® HIV-1 (Biomerieux); NASBA o NucliSens manual: empleando su buffer de lisis; Generic HIV Charge Virale (Biocentric); técnica manual, no automatizada; Abbott m2000sp: sistema automatizado de extracción de ácidos nucleicos en muestras (Abbott sample preparation system m2000sp). Todas las correlaciones de medidas de CV entre plasma y DBS fueron significativas (p<0,001).
La cuantificación de CV en DBS no puede alcanzar los mismos niveles de sensibilidad que empleando plasma. Mientras que en el DBS se emplean normalmente uno o 2 círculos de sangre seca, cada uno saturado por 50-100μl de sangre total, la nueva generación de ensayos de CV emplea 1ml de plasma equivalente a 2,5ml de sangre total. Por tanto, empleando plasma se pueden alcanzar límites de detección de 40-50 copias ARN-VIH/ml (1,3-1,7log) y en DBS se establece próximo a las 1.000 copias ARN-VIH/ml (3log)6,7,14, aunque algunos estudios detectaron hasta 200 copias ARN-VIH/ml (2,3log)15,32.
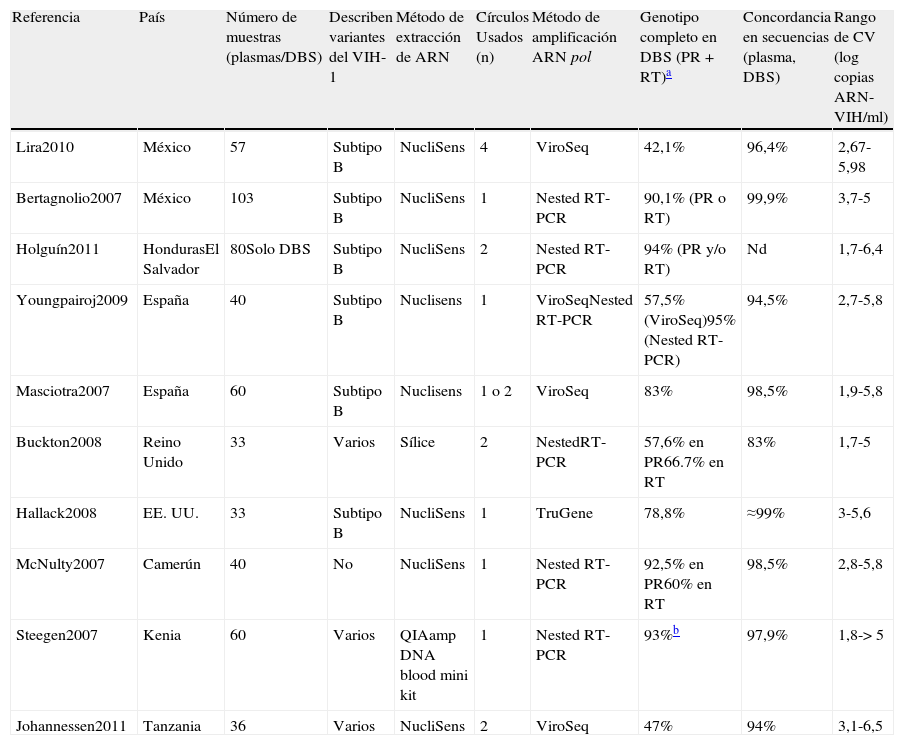
Resistencias y dried blood spotsEficacia de amplificación y ensayos de resistencia genotípicaLa Organización Mundial de la Salud (OMS) recomienda el empleo de DBS en instalaciones con recursos limitados para la vigilancia de resistencias transmitidas a fármacos del VIH-1 en pacientes naïve y para monitorizar resistencias en pacientes en TAR19. La tabla 5 resume algunos de los estudios que evalúan resistencias genotípicas a antirretrovirales empleando DBS18,20–29. La tasa de éxito de amplificación de material genético del VIH empleando DBS difiere según la CV de las muestras, y depende de las condiciones de almacenamiento de los DBS antes de su procesamiento, del método de extracción de ARN empleado y de la longitud de la región de pol del VIH amplificada. Como ocurre para la CV, la eficacia de amplificación para resistencias en DBS es mayor con CV superiores a 3log de copias ARN-VIH/ml, reduciéndose drásticamente en muestras con CV indetectables (<1,7log), como ocurre usando plasma.
Algunos estudios que monitorizan VIH-1 resistentes a fármacos antirretrovirales en DBS
| Referencia | País | Número de muestras (plasmas/DBS) | Describen variantes del VIH-1 | Método de extracción de ARN | Círculos Usados (n) | Método de amplificación ARN pol | Genotipo completo en DBS (PR+RT)a | Concordancia en secuencias (plasma, DBS) | Rango de CV (log copias ARN-VIH/ml) |
| Lira2010 | México | 57 | Subtipo B | NucliSens | 4 | ViroSeq | 42,1% | 96,4% | 2,67-5,98 |
| Bertagnolio2007 | México | 103 | Subtipo B | NucliSens | 1 | Nested RT-PCR | 90,1% (PR o RT) | 99,9% | 3,7-5 |
| Holguín2011 | HondurasEl Salvador | 80Solo DBS | Subtipo B | NucliSens | 2 | Nested RT-PCR | 94% (PR y/o RT) | Nd | 1,7-6,4 |
| Youngpairoj2009 | España | 40 | Subtipo B | Nuclisens | 1 | ViroSeqNested RT-PCR | 57,5% (ViroSeq)95% (Nested RT-PCR) | 94,5% | 2,7-5,8 |
| Masciotra2007 | España | 60 | Subtipo B | Nuclisens | 1 o 2 | ViroSeq | 83% | 98,5% | 1,9-5,8 |
| Buckton2008 | Reino Unido | 33 | Varios | Sílice | 2 | NestedRT-PCR | 57,6% en PR66.7% en RT | 83% | 1,7-5 |
| Hallack2008 | EE. UU. | 33 | Subtipo B | NucliSens | 1 | TruGene | 78,8% | ≈99% | 3-5,6 |
| McNulty2007 | Camerún | 40 | No | NucliSens | 1 | Nested RT-PCR | 92,5% en PR60% en RT | 98,5% | 2,8-5,8 |
| Steegen2007 | Kenia | 60 | Varios | QIAamp DNA blood mini kit | 1 | Nested RT-PCR | 93%b | 97,9% | 1,8->5 |
| Johannessen2011 | Tanzania | 36 | Varios | NucliSens | 2 | ViroSeq | 47% | 94% | 3,1-6,5 |
Nd: no descrito; PR: proteasa; RT: transcriptasa inversa; NucliSens: EasyMag y MiniMag (Biomerieux); QIAamp DNA blood mini kit (Qiagen); TruGene; TruGene HIV-1 genotyping assay (Siemens Healthcare Diagnostics; 1,3kb); ViroSeq HIV-1 genotyping system (Abbott Molecular; 1,8kb); Nested RT-PCR: técnica de amplificación de la PR y RT virales no comercial en uno o 2 amplicones.
Los ensayos de resistencia genotípica comerciales en pol (ViroSeq HIV-1 genotyping system; Abbott Molecular y TruGene HIV-1 genotyping assay; Siemens Healthcare Diagnostics) se han empleado para determinar resistencias en DBS18,25,26,28. Estos amplifican la proteasa y transcriptasa inversa virales en uno o 2 amplicones de 1,8kb (ViroSeq) y 1,3kb (TruGene), respectivamente. Sin embargo, son más numerosos los estudios que detectan resistencias en DBS empleando métodos no comerciales o caseros de amplificación basados en nested RT-PCR, de menor coste y que parecen ser tanto o más eficientes que ViroSeq o TruGene al tener mayores tasas de éxito de amplificación (tabla 5). Ello se debe a que en muchas ocasiones los PCR caseros amplifican la proteasa y transcriptasa inversa por separado, y la amplificación de regiones más cortas es más eficaz que las largas empleando DBS.
El éxito de amplificación del fragmento de 1,8kb por ViroSeq usando DBS fue variable en diversos estudios, dependiendo de las condiciones de almacenamiento del DBS a su uso y de su CV. En un estudio, ViroSeq consiguió amplificar el 79% de muestras con CV mayores de 5,1log copias RNA-VIH/ml, del 42,8% entre 4,2-5log, y ninguna amplificación con CV inferiores a 4,1log18. En otro estudio, ViroSeq amplificó las 46 muestras con viremias entre 2,7 y 5,8log en DBS almacenados a –20°C durante 6 meses, pero la amplificación falló en 26 de ellas cuando los DBS de los mismos pacientes se almacenaron a 4°C durante 12 meses hasta su uso26. En otro trabajo, ViroSeq amplificó el 75% de muestras con viremias mayores a 4log, pero solo el 13% con viremias inferiores28 en DBS almacenados a –20°C cerca de 4 meses, transportados a temperatura ambiente durante 20 días y guardados otro mes a –20°C hasta su procesamiento. Otros lograron genotipar por ViroSeq las 38 muestras en DBS con viremias mayores de 3,3log, pero solo el 54,5% con CV inferiores en DBS transportados y almacenados a –20°C durante una media de 5 meses, aunque consiguieron genotipar el 95% de las 46 muestras empleando una técnica no comercial de RT-PCR23. TruGene pudo amplificar el 90,5% de muestras en DBS con viremias mayores a 3,7log25.
Por todo ello, a pesar de que los kits comerciales son eficientes en muestras con altos niveles de viremia, como en pacientes en fracaso terapéutico o en la mayoría de los pacientes sin tratamiento, los ensayos de nested RT-PCR caseros que amplifican la proteasa y transcriptasa reversa por separado parecen dar más amplificaciones positivas en un porcentaje mayor de muestras con CV bajas. Además, el coste de las técnicas no comerciales normalmente es bastante inferior a las comerciales. En cualquier caso, cualquier técnica de detección de resistencias, tanto casera como comercial, requiere una buena integridad del ARN y, por tanto, las mismas condiciones favorables de conservación de los DBS.
Comparación de genotipos de resistencia obtenidos a partir de dried blood spots y plasmaAdemás de analizar el éxito de amplificación a partir de muestras de DBS, la mayoría de los estudios tienen como objetivo comparar las secuencias generadas a partir de DBS y plasma de las mismas muestras. Existe una alta concordancia (83-99,9%) entre mutaciones de resistencia a fármacos encontradas en plasma y DBS en los diversos estudios18–28 (tabla 5). Algunas mutaciones adicionales solo fueron detectadas en DBS principalmente como mezclas de aminoácidos incluyendo el residuo mutante y salvaje, pudiendo también deberse a la influencia del ADN proviral amplificado de las células infectadas junto al ARN viral en el DBS. Ello reflejaría mutaciones archivadas en el ADN proviral y que podrían volver a expresarse en las condiciones adecuadas de presión selectiva.
Interferencia del ADN proviral recuperado de los dried blood spots en la carga viral y determinación de resistenciasEl ADN proviral extraído de sangre seca contribuye significativamente al producto de la amplificación de ácidos nucleicos en ausencia de transcriptasa inversa21,23,41. Su contribución sería más relevante a mayor CV de la muestra21 y a mayor tiempo de almacenamiento de los DBS hasta la determinación de la resistencia41, confirmando la degradación más rápida del ARN respecto del ADN en los DBS. Aunque el papel FTA puede emplearse en la monitorización de resistencias, el papel 903 es más adecuado para recuperar ARN24.
Algunos autores han descrito interferencias del ADN proviral en la cuantificación basada en amplificación por RT-PCR y en los resultados genotípicos de resistencia, que puede conducir a problemas a la hora de valorar o interpretar un resultado positivo10. Ello ocurre principalmente cuando se emplean extracciones de ácidos nucleicos que recuperan tanto ADN como ARN al no incluir tratamiento con DNasas, como son los métodos basados en sílice, como el NucliSens. Ello se puede minimizar tratando los DBS previamente con DNasas para asegurar que solo se amplifica y cuantifica ARN viral41. Así se consigue que las CV usando una técnica de RT-PCR a tiempo real (Generic HIV Change Virale, Biocentric) y las eficiencias de amplificación por PCR de las mismas muestras tomadas en DBS y DPS almacenados 2 meses a 20 o 37°C sean comparables41. La influencia del ADN proviral en la cuantificación de la CV no se confirma en otros estudios en los que se extrae el material genético con el extractor m2000sp (Abbott) cuantificando con Abbott real-time HIV-16 o se extrae el ARN usando el QIAmpviral RNA mini Kit (Quiagen) cuantificando con Amplicor COBAS® TaqMan® HIV-116. En ambos estudios encontraron cuantificaciones similares en DBS y DPS sin amplificación de ADN proviral en los DBS y sin usar DNasa.
Por otro lado, cuantificando la CV por NucliSens se evita la amplificación del ADN proviral extraído del DBS durante la extracción previa por sílice, ya que esta técnica usa la ARN polimerasa de T7 para amplificar selectivamente el ARN de los ácidos nucleicos extraídos del DBS con una amplificación isoterma (4°C). En ausencia de desnaturalización por calor (esencial para cualquier amplificación de material genético por PCR), la doble cadena de ADN que pudiera existir en la extracción no puede participar en el proceso de amplificación, no sobrestimando los valores de ARN viral de la muestra en DBS.
El efecto de la interferencia de ADN proviral en la CV también está condicionado por la eficacia del tratamiento antirretroviral en el paciente22. Así, en pacientes en fallo virológico se tiende a encontrar más mutaciones de resistencia a fármacos en plasma que en células infectadas, especialmente cuando las CV son bajas. Por el contrario, en pacientes que han interrumpido el tratamiento, el ADN proviral obtenido a partir de células mononucleares de sangre periférica (CMSP) puede actuar como un archivo de mutaciones de resistencia de tratamientos pasados, y los DBS darían más información que el plasma en este tipo de pacientes.
Diagnóstico infantil empleando dried blood spotsSegún datos de la OMS, 390.000 niños menores de 15 años fueron infectados por VIH-1 en 20101. El diagnóstico temprano de la infección perinatal por VIH-1, principalmente en hijos de madres seropositivas, es esencial para instaurar el TAR, prevenir infecciones oportunistas y reducir la mortalidad infantil. Recientemente, en países en desarrollo se ha empezado a extender el uso de DBS en lugar de sangre para el diagnóstico perinatal. El diagnóstico en niños menores de 18 meses requiere detectar el material genético de VIH-1 (de ARN viral o ADN proviral) tras amplificarlo por PCR cualitativa preferiblemente en 3 regiones30,31 o por PCR cuantitativa7,11,32, aunque su sensibilidad puede variar con la edad31. Antes de 18 meses de edad las técnicas serológicas convencionales no deben emplearse, ya que los anticuerpos maternos pasan desde la madre al neonato y pueden persistir hasta entonces en el niño. Los DBS también se han usado para el diagnóstico serológico del VIH-1 por ELISA ultrasensible de detección de antígeno viral p24 en niños34 y para el diagnóstico serológico en adultos33.
Uso de dried blood spots en estudios epidemiológicosLas variantes no-B (subtipos no-B y recombinantes) del VIH-1 causan el 90% de los 34 millones de infecciones por VIH-1 y son prevalentes en gran parte de los países en desarrollo, en los que conviene implantar el uso de los DBS por sus ventajas a nivel metodológico. La alta variabilidad genética del VIH-1 puede afectar la eficacia de técnicas de amplificación por PCR del material genético viral extraído del DBS para cuantificar la CV, para detectar resistencias24 o para el diagnóstico perinatal. Las variantes no-B pueden ser responsables de falsos negativos o de fallos de sensibilidad de las técnicas diagnósticas, al estar principalmente diseñadas y validadas para el subtipo B, prevalente en países industrializados. Por ello, aunque pueden emplearse para otros subtipos, la sensibilidad puede variar. Así, se han descrito CV subestimadas en cierta muestra de plasma de subtipos no-B y recombinantes del VIH-1 empleando diferentes métodos de cuantificación comerciales49.
La toma de muestras en DBS constituye una técnica barata y práctica para diseñar grandes estudios de epidemiología molecular que analicen la distribución global y diseminación de variantes del VIH en el mundo. El análisis filogenético de las secuencias virales recuperadas de muestras infectadas es el método más adecuado para identificar variantes del VIH. Así, se puede subtipar a partir de la secuencia generada para identificar resistencias, y también de las secuencia de productos amplificados por PCR en cualquier región para el diagnóstico genético en niños. También existen páginas web gratuitas para el subtipaje rápido: se introduce la secuencia y se identifica la variante del VIH-1 en pocos segundos. Aunque estas son útiles para identificar subtipos B, presentan limitaciones importantes en la caracterización de ciertas variantes no-B del VIH-1, principalmente recombinantes complejos, cada vez más prevalentes en la pandemia50.
Aún hay pocos artículos que incluyan la información sobre el subtipo o recombinante del VIH-1 de las muestras de estudio cuando comparan eficacia de distintas técnicas empleando sangre seca infectada por VIH tomada en DBS (tablas 4 y 5). Ya que la mayoría de los pacientes con VIH están infectados por variantes no-B del VIH-1, es importante validar todos los métodos de monitorización del VIH-1 con paneles que incluyan un alto número de muestras de cada subtipo y recombinante circulante. Así se podrá determinar la sensibilidad y la especificidad de cada técnica a emplear para cada uno de ellos y se podrán diseñar bien los primers o sondas para una amplificación eficaz del genoma viral recuperado del DBS para todas las variantes del VIH-1.
ConclusiónA lo largo de esta revisión hemos expuesto cómo los DBS son útiles en la monitorización del VIH, sobre todo en países con recursos limitados. Su toma es sencilla, barata, requiere entrenamiento mínimo del personal. Evita venopunciones y uso de jeringuillas, requiere volúmenes mínimos de sangre —lo que favorece su uso en neonatos—, ocupan poco espacio durante su almacenaje y son fáciles de transportar y de manejar. Su validez respecto al plasma ha sido demostrada por distintos estudios a nivel internacional. Por ello, la OMS (http://www.who.int/hiv/topics/drugresistance/dbs_protocol.pdf) recomienda su uso en países de recursos limitados para la toma de muestras y monitorización de la infección del VIH cuando la recogida de sangre por venopunción, extracción de plasma, almacenamiento y transporte a laboratorios de referencia conlleva dificultades a nivel práctico. Además, más de 6,7 millones de personas están recibiendo tratamiento antirretroviral en estos países1. Así, se requiere implantar o reforzar los sistemas de monitorización del tratamiento empleando la detección de resistencias y favoreciendo el diagnóstico precoz de la infección. En ese contexto, el uso de los DBS es una gran alternativa. Sin embargo, en la mayoría de los países de bajos recursos donde la prevalencia de la infección por VIH es más elevada, su uso no está implantado. Por ello, es necesario unificar y validar procedimientos de laboratorio empleando los DBS. Se requiere invertir tiempo y recursos para optimizar los métodos de extracción, amplificación y cuantificación de ARN viral empleando DBS al menor coste posible. Se debe reforzar la capacitación técnica de los laboratorios en estos países para que se puedan implementar correctamente el uso de los DBS y obtener su máximo rendimiento. Es la única manera de garantizar unas buenas prácticas de toma, almacenamiento y empleo de los DBS para mantener la integridad del material genético y aumentar el éxito de los procesos. Por todo ello, invertir en extender y optimizar el uso de los DBS con las técnicas de monitorización del VIH aprobadas en cada país podría ser más rentable en costes para los planes nacionales de salud que invertir en logística tradicional de muestras.
Todo ello, unido al gran número de aplicaciones empleando DBS en el diagnóstico serológico y genético de otras infecciones, refleja el enorme potencial a nivel epidemiológico y clínico que su uso puede tener en países de recursos limitados.
FinanciaciónEste trabajo se realizó gracias a la financiación del Fondo de Investigaciones Sanitarias (FIS 09/00284). AH está financiada por la Agencia Laín Entralgo.
Conflicto de interesesLos autores declaran no tener conflicto de intereses.