Este texto incluye una actualización de las recomendaciones sobre el diagnóstico de la infección fúngica invasora de la Sociedad Española de Microbiología Clínica y Enfermedades Infecciosas (SEIMC) elaboradas en 2004 (Enferm Infecc Microbiol Clin. 2004, 22:32-9). En la actualización de 2010 se incluye una revisión exhaustiva de las novedades tecnológicas de los últimos años, así como los niveles de evidencia para recomendar cada una de las técnicas de diagnóstico. En primer lugar, se analizan los métodos convencionales, examen microscópico y cultivo, con sus limitaciones, lo que ha llevado a desarrollar métodos alternativos como la cuantificación de antígenos y de ADN fúngico. Las indicaciones de los métodos alternativos se analizan para las diferentes infecciones fúngicas, candidiasis, aspergilosis y micosis invasoras por otras especies. Por último, se incluye una breve descripción de los métodos de identificación molecular y se revisan las pruebas para realizar estudios de sensibilidad a los antifúngicos, los procedimientos de referencia, las técnicas comerciales y sus indicaciones.
These guidelines are an update of recommendations for the diagnosis of invasive fungal infections by the Spanish Society of Clinical Microbiology and Infectious Diseases (SEIMC) published in 2004 (Enferm Infecc Microbiol Clin. 2004, 22:32-9). In this updated version of the guidelines, a comprehensive review of the most recent diagnostic innovations and levels of evidence to recommend those diagnostic procedures are included. We first analyse conventional diagnostic methods, microscopic examination and culture, underlining their limitations which have led to the development of alternative methods, such as fungal antigen and DNA quantification. Those alternative methods of diagnosis are analysed by fungal infection. We also briefly review the methods for molecular identification of fungal species and recommendations for carrying out susceptibility tests for antifungal drugs, including reference procedures, commercial techniques and their indications.
En el año 2004, el Grupo de Estudio de Micología Médica de la Sociedad Española de Microbiología Clínica y Enfermedades Infecciosas (GEMICOMED-SEIMC) participó en la elaboración de unas recomendaciones sobre el diagnóstico de la infección fúngica invasora (IFI) (Enferm Infecc Microbiol Clin. 2004, 22:32-9). Desde entonces, se han producido diferentes avances y novedades tecnológicas que han modificado los procedimientos de diagnóstico microbiológico de la infección fúngica invasora.
Esta versión actualizada de las recomendaciones sobre el diagnóstico de la IFI analiza la utilidad de las técnicas de diagnóstico clásico. Actualmente, el examen microscópico y el cultivo continúan siendo los métodos de referencia para el diagnóstico de la IFI, sin embargo, su sensibilidad es limitada y no pueden considerarse técnicas de diagnóstico precoz. Por ello, aunque se ha reducido su mortalidad en los últimos años, la IFI presenta cifras elevadas de letalidad, ya que la infección se detecta cuando está muy avanzada, por lo que la respuesta al tratamiento suele ser pobre. Estas limitaciones han llevado a desarrollar técnicas alternativas al cultivo basadas en la cuantificación de antígenos, ácidos nucleicos y otros componentes fúngicos. Estos métodos se han diseñado con la intención de detectar la infección precozmente y, en algunos casos como la cuantificación de galactomanano, se han convertido en técnicas diagnósticas fiables en algunos grupos de enfermos.
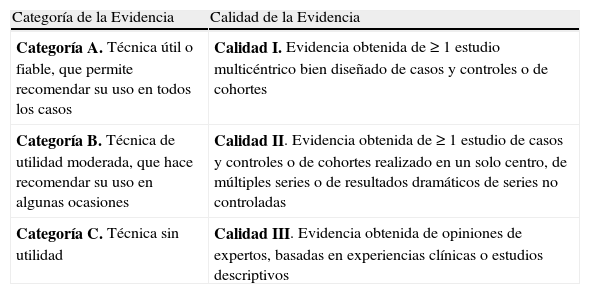
Estas recomendaciones actualizadas hacen una revisión de las evidencias disponibles sobre las diferentes técnicas de diagnóstico de la IFI, los métodos convencionales, los alternativos, las técnicas de sensibilidad y la identificación molecular. En el caso de las técnicas alternativas se realiza la revisión por separado de las evidencias disponibles para candidiasis, aspergilosis y otras infecciones fúngicas. Asimismo, se incluye una clasificación de las recomendaciones sobre la mayor parte de estas técnicas, con grado de evidencia según la calidad de los estudios publicados (tabla 1).
Grados de evidencia empleados para realizar las recomendaciones sobre las técnicas diagnósticas de la IFI
| Categoría de la Evidencia | Calidad de la Evidencia |
| Categoría A. Técnica útil o fiable, que permite recomendar su uso en todos los casos | Calidad I. Evidencia obtenida de ≥ 1 estudio multicéntrico bien diseñado de casos y controles o de cohortes |
| Categoría B. Técnica de utilidad moderada, que hace recomendar su uso en algunas ocasiones | Calidad II. Evidencia obtenida de ≥ 1 estudio de casos y controles o de cohortes realizado en un solo centro, de múltiples series o de resultados dramáticos de series no controladas |
| Categoría C. Técnica sin utilidad | Calidad III. Evidencia obtenida de opiniones de expertos, basadas en experiencias clínicas o estudios descriptivos |
El diagnóstico microbiológico convencional de las IFI se basa en el aislamiento e identificación de los hongos responsables, empleando los medios de cultivo idóneos y estudiar –si es pertinente- su sensibilidad a los antifúngicos. Para ello es fundamental la recogida de la muestra clínica adecuada, así como su correcta manipulación, conservación y transporte al laboratorio.
Recogida y transporte de las muestrasPara optimizar el adecuado procesamiento, las muestras1,2 deben ser representativas del foco de infección, estar correctamente identificadas, y ser tomadas en condiciones asépticas antes de instaurar el tratamiento antimicrobiano, evitando la contaminación accidental o con flora comensal, y en contenedores estériles.
Un importante aspecto a tener en cuenta en el diagnóstico micológico consiste en determinar, si es posible, el volumen necesario de muestra para asegurar la recuperación de ciertos hongos, ya que en determinadas circunstancias es preciso concentrar las muestras o sembrar un mayor volumen de las mismas. Para la toma de la muestra, es preferible evitar el empleo de hisopos y siempre aconsejable la aspiración con jeringa o si es posible la toma de biopsias.
El transporte de las muestras al laboratorio debe hacerse en contenedores estériles a prueba de vertidos, y debe ser lo más rápido posible, procurando evitar que se demore más de dos horas, ya que su viabilidad se ve reducida por la desecación, las temperaturas excesivamente elevadas (>37°C) o bajas (<4°C), la falta de nutrientes, el sobrecrecimiento bacteriano y/o la presencia de algunos enzimas leucocitarios u otros. Para evitar la desecación de la muestra puede añadirse una pequeña cantidad de solución salina estéril (0,85% ClNa). Debe evitarse el transporte en condiciones de anaerobiosis pues reduce la viabilidad de algunos hongos. En caso de que se prevea un transporte prolongado (> 2h), las muestras procedentes de localizaciones habitualmente estériles (sangre, líquido cefalorraquídeo [LCR],...) pueden conservarse a temperatura ambiente, pero el resto de las muestras deberían conservarse a 4°C para evitar el sobrecrecimiento bacteriano.
Como en cualquier otro proceso infeccioso, la mejor muestra clínica es aquella que procede directamente de una zona activa de infección. Aunque, en ocasiones, la agresividad de algunas técnicas de recogida de muestras o el precario estado de salud del paciente obligan a la obtención de muestras alternativas con menor rentabilidad diagnóstica.
El hemocultivo, aun a pesar de la escasa sensibilidad global (≈50%), sigue siendo la mejor técnica para el diagnóstico microbiológico de las fungemias3,4. El volumen de sangre extraída no debería ser inferior a 10ml (ni superior a 30ml para evitar la aparición de anemia nosocomial), siendo recomendable realizar dos o tres extracciones independientes por episodio séptico, procurando sembrar 20ml de sangre por extracción (o entre 1 y 5ml si se trata de niños pequeños o neonatos), ya que se ha demostrado que la diferencia entre utilizar 10ml o 20ml puede suponer incrementos del índice de recuperación de levaduras cercanos al 30%5.
El lavado broncoalveolar (LBA) es la técnica de elección para el diagnóstico de las micosis invasoras oportunistas por hongos filamentosos del tracto respiratorio inferior en los enfermos inmunodeprimidos, con un valor predictivo positivo ≥80%, según el tipo de inmunosupresión. Como muestras alternativas se recomienda el esputo obtenido por expectoración espontánea o inducido, o el aspirado traqueal en los pacientes intubados, aunque el valor de estos últimos está muy condicionado por la calidad de la muestra6. Estas muestras se consideran con valor diagnóstico en las infecciones causadas por hongos dimórficos patógenos primarios (Histoplasma capsulatum, Paracoccidioides brasiliensis, etc.) y en la neumonía por Pneumocystis jiroveci, pero en las neumonías causadas por hongos oportunistas (Aspergillus, etc.) sólo tienen valor presuntivo, pues no existen criterios que permitan diferenciar entre infección, colonización y contaminación.
La muestra de elección para el diagnóstico de las meningitis fúngicas es el LCR, y al igual que los otros líquidos estériles, como el pleural, el peritoneal, el pericárdico o el sinovial, debe obtenerse en condiciones de máxima asepsia y enviarse rápidamente al laboratorio.
En caso de sospechar la presencia de una micosis sistémica, especialmente si hay evidencia de infección de uno o varios órganos profundos, debe plantearse, siempre que sea posible, la realización una biopsia del tejido afectado. Las biopsias deben recogerse empleando técnicas quirúrgicas, con las máximas medidas de asepsia y deben enviarse al laboratorio con la máxima premura, procurando evitar su desecación. Antes de la siembra, y para facilitar el aislamiento de la mayoría de los hongos, debe procederse a la homogenización y trituración del tejido, pero es importante recordar que ésta deberá evitarse siembre que se sospeche de la presencia de zigomicetos. En estas muestras debe realizarse tanto el estudio microbiológico como el histológico. En muchas ocasiones, la histopatología es clave para demostrar la invasión tisular y para alcanzar un diagnostico de certeza de una micosis invasiva.
Examen microscópicoLa observación microscópica de las muestras puede, en ocasiones, detectar la presencia de elementos fúngicos suficientemente característicos como para diagnosticar la etiología de una infección fúngica. Uno de los principales inconvenientes del examen microscópico directo de las muestras clínicas es su baja sensibilidad; por lo que un examen microscópico negativo no excluye nunca la infección y no es recomendable su práctica cuando el material clínico del que se dispone es escaso. Por otro lado, también carece de la especificidad deseada, pues ciertas estructuras o artefactos pueden confundirse con elementos fúngicos. Sin embargo, siempre que se pueda es recomendable la realización del mismo, especialmente en aquellos casos en los que el paciente está recibiendo terapia antifúngica y en la muestra puede haber microorganismos no viables sólo detectados por microscopía7.
El examen directo en micología se basa en el empleo de colorantes o fluorocromos que se unen inespecíficamente a la pared fúngica7, como la tinción de Gram8,9, la tinción con blanco de calcoflúor, la tinción de la tinta china o las tinciones argénticas o del ácido periódico de Schiff, especialmente empleadas en histopatología10,11. Las levaduras son fáciles de observar, pudiéndose apreciar las gemaciones y las pseudohifas, cuando estas están presentes. Los hongos filamentosos se tiñen con más dificultad por los colorantes de la tinción de Gram, aunque si el número de hifas es elevado pueden reconocerse fácilmente, incluso a pequeños aumentos. Las características de las hifas, el grado de angulación de las ramificaciones y la presencia o ausencia de tabiques permiten distinguir entre hongos hialinos, feoides y zigomicetos. Las técnicas inmunológicas de examen directo, basadas en el empleo de anticuerpos marcados dirigidos contra antígenos fúngicos específicos pueden incrementar considerablemente la sensibilidad de la observación microscópica, pero desgraciadamente se dispone de un arsenal de anticuerpos muy limitado y sólo suelen emplearse en centros de referencia.
CultivoAl igual que en otros procesos infecciosos, el cultivo es un paso esencial y necesario en el diagnóstico de las infecciones fúngicas y además permite el aislamiento del agente causal, lo que es indispensable para una correcta identificación del mismo y, en caso de estar indicados, hace posible la práctica de estudios de sensibilidad o estudios epidemiológicos.
Los hongos no son especialmente exigentes y existen muchos medios útiles para su cultivo. Algunos están especialmente diseñados para el aislamiento primario de hongos potencialmente patógenos. El empleo de unos u otros depende en gran parte de la experiencia personal, de la disponibilidad y del coste. En la práctica clínica diaria, las necesidades suelen cubrirse con los medios de cultivo selectivos, generalmente derivados del agar glucosado de Sabouraud y, habitualmente, suplementados con antibióticos, como el cloranfenicol, la gentamicina o la amicacina, para impedir el crecimiento de bacterias. Es aconsejable sembrar más de un medio de cultivo y, en ciertas situaciones, puede estar indicada la utilización de medios específicos para el aislamiento de determinados hongos, como Malassezia12, Cryptococcus13 u hongos dimórficos patógenos primarios, así como medios de cultivo diferenciales que, merced a la adición de determinados componentes, permiten la identificación presuntiva de los hongos basándose en la morfología de sus colonias14.
Para el hemocultivo, los medios bifásicos3, que tradicionalmente habían sido considerados los mejores para el diagnóstico de las fungemias, han ido sustituyéndose por medios líquidos con formulaciones capaces de permitir una correcta recuperación de los hongos; o por técnicas de cultivo más complejas como la lisis-centrifugación o la lisis-filtración15. Aunque es complicado establecer el valor relativo de los diferentes medios y/o técnicas, ya que las diferencias metodológicas de los diversos estudios y las indefiniciones en cuanto a la población estudiada hacen difícil su comparación, estas últimas parecen ser las que presentan un mayor índice de recuperaciones en caso de hongos endémicos y de algunos hongos filamentosos.
Debe tenerse presente, que muchos de los hongos causantes de infecciones tienen un crecimiento lento, por ello los cultivos deben incubarse se incuban un mínimo de 4 o 6 semanas antes de considerarlos negativos, aunque en algunos casos, como cuando se sospecha una histoplasmosis, la incubación puede alargarse hasta 12 semanas. No debe desecharse ningún cultivo sin completar el periodo de incubación, ni aunque se haya aislado un posible patógeno, ya que podría tratarse de un contaminante o de una colonización, y que las colonias del verdadero agente causal todavía no fueran evidentes.
La interpretación del resultado de los cultivos siempre debe asociarse a la situación clínica del enfermo. El aislamiento de un hongo patógeno primario en cualquier tipo de muestra tiene siempre un valor diagnóstico, aunque estas infecciones son infrecuentes en nuestro país. Los cultivos de las biopsias de tejidos estériles tampoco suelen presentar problemas de interpretación, ya que el aislamiento de cualquier hongo debería considerarse significativo, pero la posibilidad de una contaminación accidental hace que sea muy recomendable disponer también de estudios anatomopatológicos que confirmen la invasión tisular por elementos fúngicos. También los aislamientos fúngicos en líquido cefalorraquídeo u otros líquidos estériles suelen ser significativos.
Por el contrario, el aislamiento en muestras de esputo, broncoaspirados o LBA siempre plantea una gran dificultad de interpretación, siendo difícil conocer su trascendencia clínica, si no se evalúan los datos clínicos del caso. Debe tenerse presente que la mayoría de infecciones fúngicas invasoras del tracto respiratorio inferior son producidas por Aspergillus fumigatus o Aspergillus flavus16. Además, no todos los hongos tienen capacidad para producir una micosis invasiva del tracto respiratorio inferior, como las especies del género Candida, que están presentes en las secreciones respiratorias del 20% de la población sana y en más del 50% de los pacientes que han recibido antibióticoterapia17. Candida spp. no causan infecciones respiratorias salvo en neumonías por aspiración o secundarias a diseminación hematógena18.
Los hemocultivos suelen presentar una baja rentabilidad en el diagnóstico de las infecciones invasivas por hongos filamentosos oportunistas19. Ello es debido a que las infecciones por ellos producidas no suelen cursar con una fase hematógena detectable por las técnicas de hemocultivos. Sin embargo existen notables excepciones, como por ejemplo Penicillium marneffei que se aísla en hemocultivos en el 75% de los pacientes infectados20; Fusarium spp. y Scedosporium en el 50-70%21, o Aspergillus terreus, que tiene la capacidad de producir en los tejidos formas pseudolevaduriformes que pueden ser detectadas en los hemocultivos22.
IdentificaciónLa identificación de los hongos permite su clasificación taxonómica y realizar estudios epidemiológicos y plantear estrategias terapéuticas. La identificación se basa esencialmente en la morfología macroscópica de las colonias, las características bioquímicas y la morfología microscópica23, aunque una vez aislado un hongo, los procedimientos de identificación dependerán de si es una levadura, un hongo filamentoso o un hongo dimórfico.
La identificación de los hongos levaduriformes se puede llevar a cabo atendiendo a criterios morfológicos, bioquímicos, inmunológicos o genéticos24. Los criterios morfológicos pueden ser, a su vez, macro o microscópicos. Los criterios macroscópicos tienen en cuenta el aspecto de las colonias al crecer en los diferentes medios de cultivo usados rutinariamente en el laboratorio de microbiología. Ciertas características microscópicas son muy útiles para la identificación de algunas especies de levaduras. Las más utilizadas en la práctica son las pruebas del tubo germinal o filamentación precoz, la formación de hifas, blastoconidias, clamidosporas y artrosporas.
Entre los criterios bioquímicos o enzimáticos podemos destacar los medios cromógenos, diseñados para el aislamiento e identificación presuntiva de algunas especies de Candida. El fundamento de los mismos se basa en la detección de determinadas actividades enzimáticas de las levaduras, mediante la hidrólisis de un sustrato cromógeno en presencia de un indicador de la enzima. Una de las principales ventajas de estos medios es que permiten detectar fácilmente los cultivos mixtos. Entre estos medios podemos destacar CHROMagar Candida® (CHROMagar Company)25, Cromogen Albicans® (Biomedics), chromID Candida® (bioMérieux-Vitek), CandiSelect® (Bio-Rad)26, Candida chromogenic agar® (Conda), Agar Fluoroplate Candida® (Merck) y agar SDCA-MUAG® (Biolife).
Además, existen varios sistemas enzimáticos comercializados para la identificación rápida de C. albicans a partir de colonias en cultivo puro. Consisten en la utilización de sustratos fluorogénicos o cromogénicos para la detección de una o dos enzimas (β- galactosidasa y L-prolina aminopeptidasa), sólo presentes en C. albicans27. Cryptococcus neoformans también puede ser identificado por métodos enzimáticos como la detección de ureasa, la de la nitrato-reductasa o la prueba de la fenol-oxidasa.
No obstante, para la identificación de la mayor parte de la especies de levaduras deben utilizase técnicas basadas en la asimilación de diferentes fuentes de carbono o nitrógeno (auxonograma), que se realiza sobre un medio sintético base. Para tal propósito existen diferentes métodos comercializados, como las galerías Auxacolor® (Bio-Rad) y Uni-Yeast Tek® (Remel). También existen sistemas semiautomáticos como API 20C AUX® (bioMérieux), API ID32C® (bioMérieux), Vitek® (bioMérieux), Micronaut-Candida System®28, y sistemas automáticos como Vitek 2® (bioMérieux), Biolog YT MicroPlate® (Biolog), y Rapid Yeast Identification Panel MicroScan® (Dade Behring). También se han comercializado técnicas de identificación rápida (4 horas) de levaduras como el RapID Yeast Plus System® (Innovative Diagnostic Systems) y Fungiscreen 4H® (Bio-Rad). La técnica GLABRATA RTT® (Foumouze Diagnostics) es un método sencillo que en sólo 20 minutos permite la identificación de C. glabrat29.
También se han desarrollado técnicas inmunológicas para la identificación de levaduras. Cabe citar los tests Bichro-latex albicans® (Fumouze)], Krusei-color® (Fumouze) y Bichro-Dubli® (Fumouze)30, que se basan en la aglutinación de partículas de látex utilizando un anticuerpo monoclonal específico de C. albicans, C. krusei o C. dubliniensis, respectivamente31.
La identificación de los hongos filamentosos se realiza en base a la morfología macroscópica de las colonias y, especialmente, al examen microscópico de sus estructuras de reproducción generalmente asexual. Un inconveniente importante radica en el hecho de que tales estructuras suelen ser de desarrollo lento no apareciendo hasta al cabo de varios días o semanas de incubación. Las características metabólicas suelen ser de escasa ayuda en la identificación de los hongos filamentosos con la excepción de las llamadas levaduras negras.
Para la identificación de los hongos endémicos es deseable demostrar tanto la fase levaduriforme como la filamentosa, aunque se han desarrollado técnicas alternativas para demostrar mediante anticuerpos específicos la producción de exoantígenos solubles32. Debe destacarse que para trabajar con estas especies se deben disponer de instalaciones de bioseguridad del tipo GR-3, ya que pueden causar infecciones en el personal del laboratorio al inhalar las conidias que desprende la fase filamentosa.
RecomendacionesEn la tabla 2 se incluyen algunas recomendaciones básicas para los laboratorios clínicos sobre las técnicas de diagnóstico clásico de la IFI. No se incluyen los grados de evidencia de cada una de estas recomendaciones, ya que no existen estudios comparativos controlados sobre las técnicas de diagnóstico convencional, pero debe insistirse en que siguen siendo los métodos de diagnóstico de referencia.
Recomendaciones sobre las técnicas convencionales de diagnóstico de la IFI
| Recomendaciones |
| Toma de muestras adecuada siguiendo procedimientos habituales |
| Remisión al laboratorio en < de dos horas. Si no es posible, conservar a 4°C |
| Realizar siempre un examen microscópico microbiológico e histopatológico |
| Cultivar en varios medios de cultivo, incluyendo medios selectivos |
| Los medios de cultivo diferenciales son útiles para algunas especies como Candida |
| Deben identificarse la especie de todas las cepas clínicas causantes de infección |
| Los criterios morfológicos y bioquímicos identifican la mayor parte de las cepas clínicas |
| La identificación molecular de las cepas clínicas debe utilizarse en casos seleccionados y realizarse en centros de referencia |
Debido a su elevada morbimortalidad, la candidiasis invasora (CI) se ha convertido en un problema importante en los hospitales terciarios de todo el mundo. Además, en algunos países Candida ocupa el cuarto lugar entre los microorganismos aislados en hemocultivo. El diagnóstico, de una infección sistémica por Candida es difícil debido a la ausencia de síntomas patognomónicos de la enfermedad y a la dificultad para aislar el hongo en los hemocultivos33, que sigue siendo la técnica de referencia, aunque muestra una baja sensibilidad y no detecta precozmente la infección, lo que induce al uso de terapia antifúngica empírica. Por ello, se han desarrollado técnicas alternativas basadas en métodos inmunológicos (detección de antígenos y anticuerpos) o en la detección de componentes fúngicos estructurales o metabólicos como el (1-3)-ß-D-glucano (BG), el D-arabinitol o el ADN. Sin embargo, todas las técnicas empleadas hasta ahora presentan importantes limitaciones y ninguna tiene la suficiente fiabilidad para su uso habitual en el laboratorio de microbiología clínica34.
Detección de manano y anti-mananoLa detección, mediante ELISA, de antígeno manano y de anticuerpos frente a este antígeno de Candida está disponible comercialmente desde hace años (Platelia Candida Ag® y Platelia Candida Ab/Ac/Ak®; Bio-Rad). Para evitar el escaso rendimiento de estas técnicas cuando se emplean por separado, se recomienda la realización conjunta de ambas pruebas en todo paciente con sospecha de CI. Recientemente se ha evaluado la utilidad de estas técnicas en sujetos neutropénicos, comprobándose que en estos pacientes un resultado negativo permitiría excluir una CI, debido a su elevado valor predictivo negativo (95%)35. Además, se ha constatado que la presencia de anticuerpos anti-manano se asocia a un mayor riesgo de CI en pacientes neutropénicos, y que se adelanta a la aparición de manifestaciones clínicas36. Sin embargo, debido a la alta prevalencia de anticuerpos anti-manano en la población sana o colonizada, se han investigado otros anticuerpos más específicos de CI. Entre ellos, destacan los dirigidos contra antígenos expresados en la fase micelial de C. albicans (anti-micelio) y contra los antígenos citoplasmáticos.
Detección de anticuerpos anti-micelioLa detección de anticuerpos anti-micelio es una técnica desarrollada en el Departamento de Inmunología, Microbiología y Parasitología de la Universidad del País Vasco, que se ha comercializado recientemente en forma de inmunofluorescencia indirecta, Candida albicans IFA IgG® (Laboratorios Vircell). Este método detecta anticuerpos contra antígenos expresados en la fase micelial de C. albicans pero que también es positiva en los pacientes con CI por C. tropicalis, C. parapsilosis, C. krusei, C. glabrata, C. guilliermondii o C. dubliniensis. La técnica presenta buenos resultados de sensibilidad (84,4%) y especificidad (94,7%) para el diagnóstico de CI, cuando se utiliza un título de anticuerpos antimicelio > 1:16037. Además de su utilidad para el diagnóstico, también permite el seguimiento evolutivo de la infección ya que en los pacientes que responden al tratamiento antifúngico se detectan títulos decrecientes de anticuerpos, que finalmente llegan a desaparecer38. En un estudio multicéntrico prospectivo, realizado para valorar la utilidad de esta técnica en pacientes críticos, se observó una tasa de positividad mayor de la esperada; además, la mortalidad fue significativamente menor en los pacientes con títulos progresivamente en aumento que fueron tratados con antifúngicos39.
Detección de otros anticuerposRecientemente ha sido evaluada una nueva técnica de ELISA comercializada en EE.UU., Candida Detect® (Micro Detect Inc), que detecta IgG, IgM e IgA frente antígenos citoplasmáticos de C. albicans, demostrando una excelente reproducibilidad y facilidad de uso40. Sin embargo, todavía no se conocen la sensibilidad, especificidad y valores predictivos de esta técnica.
Recientemente también se ha constatado la utilidad de un test diagnóstico, comercializado para la enfermedad de Crohn (IBDX®, Glycominds, Israel), que detecta anticuerpos anti-manano de Saccharomyces cerevisiae, anti-glucano y anti-quitina, como biomarcador de CI por C. albicans41. Esta prueba es capaz de discriminar entre colonización e infección por esta especie y sus resultados preliminares, aunque son alentadores, necesitan ser confirmados por nuevos estudios.
Detección de (1-3)-ß-D-glucanoEl BG es un polisacárido ubicuo en la naturaleza que se encuentra principalmente en las plantas y los hongos. En estos últimos, el BG es un componente abundante de la pared celular, donde tiene una función estructural muy importante. A principios de la década de los 1990 comenzó a estudiarse su utilidad como marcador de IFI, ya que se libera durante la infección por la mayoría de los hongos de importancia clínica y no se encuentra en los mamíferos, la mayoría de las bacterias o los virus.
La detección de BG se realiza mediante una técnica muy sensible basada en la activación de la cascada de coagulación del cangrejo herradura (Limulus polyphemus y otras especies). La interpretación de los resultados requiere personal con experiencia en la prueba, por lo que se recomienda que la detección de BG se realice en laboratorios de referencia. Actualmente existen cuatro pruebas comercializadas para su detección con fines diagnósticos: Fungitec G® (Seikagaku Kogyo Corporation), Wako® WB003 (Wako Pure Chemical Industries) y B-G Star® (Maruha Corporation) que se utilizan en Japón, y Fungitell® (Associates of Cape Cod Inc.), que se está utilizando en Estados Unidos y Europa. Aunque las cuatro pruebas detectan BG, presentan diferencias en su límite de detección y, por tanto, son necesarios puntos de corte diferentes en cada una de las pruebas comercializadas42.
El BG se libera de la pared fúngica y puede detectarse en los líquidos biológicos (principalmente suero) de pacientes con distintas micosis incluyendo candidiasis, aspergilosis, neumocistosis, pero no en las criptococosis ni en las mucormicosis. Los mucorales liberan in vitro al medio de cultivo cantidades de BG generalmente < 200pg/ml, mientras que Candida spp. y Aspergillus spp. liberan una media de 2.119pg/ml y 1.915pg/ml, respectivamente43. Se trata de un marcador panfúngico, por lo que un resultado positivo no permite identificar la especie causante de la infección.
La detección de BG presenta resultados falsos positivos y negativos que deben tenerse en cuenta para obtener el máximo rendimiento de la prueba. Debido a su ubicuidad, una de las causas de falsa positividad es la contaminación de los materiales de laboratorio con BG. También se han descrito resultados falsos positivos en pacientes en hemodiálisis con membranas de acetato de celulosa, en contacto con gasas y esponjas quirúrgicas o en tratamientos con inmunoglobulinas humanas intravenosas, polisacáridos antitumorales (lentinano y polisacárido K), albúmina, factores de coagulación, proteínas plasmáticas, quimioterapia antitumoral, amoxicilina-ácido clavulánico y piperacilina-tazobactam44. Los sueros hemolizados, algunas bacteriemias por grampositivos (Streptococcus spp.) y gramnegativos (Alcaligenes spp. y Pseudomonas aeruginosa) son otras causas conocidas de resultados falsos positivos. Los falsos negativos se asocian con la existencia de sueros hiperpigmentados (bilirrubina y triglicéridos elevados), el tratamiento empírico o profilaxis antifúngica y con especies que liberen poca cantidad de BG durante la infección44,45.
Detección de ácidos nucleicosLa detección de ADN de Candida en sangre de pacientes con candidiasis pretende paliar la relativa insensibilidad del cultivo y anticipar su diagnóstico. Aún no hay consenso sobre la fracción de sangre (sangre completa, suero o plasma) más adecuada para la extracción de ADN. Algunos estudios, aunque incluyendo pocas muestras, demuestran mayor sensibilidad tras la extracción a partir de suero o plasma46,47. La extracción de ADN es fundamental porque requiere la rotura de la pared fúngica. Se han evaluado diferentes sistemas comerciales para la extracción/purificación del ADN. Aunque la eficiencia es dependiente del sistema elegido, todavía no puede recomendarse alguno en concreto. Los métodos automatizados suponen un ahorro del tiempo necesario para el procesamiento. Para amplificar el ADN de Candida purificado se han evaluado diferentes métodos basados en la reacción en cadena de la polimerasa (PCR). Entre estos destaca la PCR en tiempo real, que supone un gran avance ya que muestra un bajo número de falsos positivos y ofrece la posibilidad de identificar el patógeno en 4-5h.
Destacan tres estudios prospectivos en los que los investigadores desarrollaron su propio sistema de PCR48–50. Lin et al estudiaron la presencia de ADN de Candida en muestras de sangre de 42 pacientes pediátricos neutropénicos. En 24 episodios de candidiasis, la PCR resultó positiva (sólo en 17 se acompañó de hemocultivos positivos) y supuso un adelanto en el diagnóstico de 1-8 días48. Klingspor et al desarrollaron una PCR multiplex a tiempo real para la detección genérica y específica de Candida en diversas muestras clínicas de pacientes con alta sospecha de micosis49. Este sistema demostró mayor sensibilidad que el hemocultivo, especialmente en pacientes con cáncer hematológico y con candidiasis hepatoesplénica y del sistema nervioso central. McMullan y colaboradores realizaron recientemente un estudio con 157 pacientes adultos críticos. La PCR demostró una sensibilidad y especificidad del 90,9% y 100%, respectivamente, y permitió alcanzar el diagnóstico de CI en menos de 6 horas50. El sistema comercial SeptiFast® (Roche Diagnostics) supone un primer paso hacia la estandarización de la PCR a tiempo real, sobre todo en pacientes con sospecha de sepsis. Westh y colaboradores describieron 25 casos de candidiasis invasora en los que el SeptiFast demostró mayor sensibilidad que el hemocultivo51. Dierkes y colaboradores estudiaron 77 pacientes con sospecha de sepsis siendo el SeptiFast complementario al hemocultivo en 6 casos de CI52. Por su parte, von Lilienfeld-Toal et al, observaron que el SeptiFast y el hemocultivo complementaban el diagnóstico en pacientes con neutropenia febril53. Son de destacar algunos trabajos en los que se detectó ADN de Candida en botellas de hemocultivo a través de PCR multiplex en tándem con posterior identificación de la levadura al nivel de especie en 2 horas54. Otros métodos basados en hibridación in situ con sondas fluorescentes detectan la presencia de C. albicans en botellas de hemocultivos y su aplicación ha demostrado tener impacto clínico en el ahorro de antifúngicos de amplio espectro55.
RecomendacionesLa detección conjunta de manano y de anticuerpos anti-manano se ha demostrado útil (B-II) en sujetos neutropénicos debido a su elevado valor predictivo negativo (95%)35. Por su parte, la detección de anticuerpos anti-micelio presenta una sensibilidad (84,4%) y una especificidad (94,7%) elevadas (B-II)37. Además, también permite el seguimiento evolutivo de la infección38,56 (tabla 3).
Recomendaciones y grados de evidencia de las técnicas alternativas al cultivo para el diagnóstico de la candidiasis invasora
| Técnica diagnóstica | Grado de recomendación |
| Ag manano y Ac anti-manano combinados | B-II |
| Ac anti-micelio | B-II |
| Detección de (1-3)-ß-D-glucano | A-I |
| Detección de ácidos nucleicos | B-II |
Ag: Antígeno; Ac: Anticuerpo.
La detección de BG en suero es un buen marcador de IFI, especialmente en pacientes con candidiasis o aspergilosis invasora34,42,57–60 y se considera un criterio microbiológico de infección probable, exceptuando la criptococosis y la mucormicosis, en los criterios conjuntos de la European Organization for Research and Treatment of Cancer (EORTC) y el Mycoses Study Group del NIAID (National Institute of Allergy and Infectious Diseases) de Estados Unidos (A-I) (tabla 1)61. Se recomienda que la realización de esta prueba se lleve a cabo en laboratorios de referencia, con una periodicidad de al menos una vez por semana durante el periodo de máximo riesgo de padecer una IFI12,26. El BG suele detectarse antes que otros marcadores diagnósticos como el cultivo, la histología, la fiebre o la radiología58,59. Sin embargo, al tratarse de un marcador panfúngico, la prueba debe complementarse con otras que permitan la identificación del hongo infectante44. La detección de BG en combinación con otro biomarcador disminuye los falsos positivos59. Los niños tienen concentraciones séricas de BG superiores a los de los adultos62. La detección de BG suele ser de ayuda en el establecimiento del tratamiento antifúngico anticipado de la IFI en pacientes trasplantados de hígado durante el año posterior al trasplante63.
Por su parte, la falta de estandarización del método de extracción, de los cebadores para la amplificación del ADN genómico y del sistema elegido para la detección del material amplificado ha hecho que EORTC siga sin incluir las técnicas moleculares como criterio diagnóstico de CI. La PCR a tiempo real parece ser el formato más conveniente. El sistema SeptiFast® (Roche Diagnostics) que ha sido comercializado en los últimos años es esperanzador ya que está basado en este formato y pueden realizarse ejercicios de estandarización. Sin embargo, su elevado coste y la escasa experiencia que existe con este test exigen prudencia en su recomendación. Es por ello que en la actualidad la PCR debe ser considerada una herramienta complementaria al cultivo de sangre y no un sustituto (B-II). Sin embargo, la detección de Candida mediante técnicas moleculares puede ser muy útil en el diagnóstico de casos de CI en los que se sabe a priori que el hemocultivo tiene poca rentabilidad, como pacientes con candidiasis hepatoesplénica o aquellos que están recibiendo antifúngicos en el momento de la extracción de sangre.
Diagnóstico de aspergilosis alternativo al cultivoEl diagnóstico de la aspergilosis invasora (AI) continúa siendo difícil por la poca especificidad de las manifestaciones clínicas y la inexistencia de hallazgos radiológicos patognomónicos, aunque se empleen técnicas tan sensibles como la tomografía axial computerizada de alta resolución (TAC), la resonancia magnética (RM) o la tomografía por emisión de positrones (PET). Además, los métodos convencionales de diagnóstico de laboratorio tienen una baja sensibilidad64. Con el objetivo de mejorar y adelantar el diagnóstico para instaurar un tratamiento antifúngico dirigido que permita disminuir la mortalidad asociada a la AI, se han desarrollado técnicas microbiológicas alternativas al cultivo basadas en la detección de antígenos –como el galactomanano- (GM), de componentes de la pared celular, como el BG o de ácidos nucleicos de Aspergillus. Algunos de estos biomarcadores, como el GM, han sido validados en estudios con pacientes inmunodeficientes con un alto riesgo de adquirir AI. Por otro lado, la metodología e instrumental necesarios para realizar la detección de GM están al alcance de la mayoría de los laboratorios clínicos. La detección de BG es más compleja de realizar y la de ácidos nucleicos se encuentra en el terreno de la investigación clínica aunque los resultados son muy prometedores. La detección de anticuerpos frente a Aspergillus o la de otros componentes antigénicos de este hongo no han demostrado utilidad diagnóstica.
Detección de galactomananoEl GM es un componente de la pared celular de Aspergillus que se libera durante la invasión de los tejidos y puede ser detectado en el suero, LBA, biopsias, orina o líquidos cefalorraquídeo, pericárdico y pleural de los enfermos con AI65. La técnica más empleada por su sencillez, rapidez y reproducibilidad es un ELISA que emplea el anticuerpo monoclonal EBA-2 (Platelia Aspergillus®, Bio-Rad). El límite de detección de GM mediante esta técnica es de 1 ng/ml de GM en suero y en el momento actual, aunque no existen recomendaciones clara del fabricante, la mayor parte de los expertos acepta el índice de GM en suero de 0,5 como punto de corte diagnóstico. El grado de validez diagnóstica de la detección de GM depende del tipo de pacientes. Los estudios publicados en pacientes onco-hematológicos y receptores de precursores hematopoyéticos han demostrado que la detección de GM tiene una especificidad alta (alrededor del 90%), mientras que su sensibilidad es variable (entre 30 y 100%). La variabilidad en la sensibilidad ha sido relacionada con las diferencias en los criterios utilizados para definir los casos de AI, con los puntos de corte diagnósticos de los índices de GM y con las distintas poblaciones de pacientes incluidos en cada estudio66–75.
Su mayor utilidad se ha demostrado en pacientes onco-hematológicos con alto riesgo de sufrir una AI. En los pacientes con neutropenia prolongada después de la quimioterapia o de la recepción de un trasplante alogénico de precursores hematopoyéticos67,70,71,73,76, la sensibilidad de la prueba supera el 85% y la especificidad el 95%. En esta población, la detección de GM antecede a los hallazgos clínicos y radiológicos, y podría permitir la instauración más temprana del tratamiento antifúngico. Además los datos existentes apuntan a que la detección de GM es útil para el seguimiento de la evolución de la AI y la respuesta al tratamiento porque los índices de GM se correlacionan con la carga fúngica en los tejidos65,68,70,77. Sin embargo, pueden observarse aumentos paradójicos de GM en la insuficiencia renal, en los pacientes sometidos a hemodiálisis, así como en pacientes tratados con caspofungina, a pesar de que la respuesta clínica sea favorable78,79.
La detección de GM en el LBA de pacientes onco-hematológicos con neutropenia tiene también un valor diagnóstico alto74,75 y se han observado valores aceptables en pacientes críticos inmunodeprimidos72 y en trasplantados de pulmón80. Por el contrario, los valores diagnósticos de la detección de GM en suero han sido peores en pacientes sin neutropenia, como los receptores de trasplante de hígado u otros órganos sólidos65,66.
Se ha observado un número importante de falsos positivos en la población pediátrica, sobre todo en neonatos con colonización intestinal por Bifidobacterium spp., en pacientes con enfermedad crónica de injerto contra huésped, y en personas que siguen dietas ricas en proteínas de soja y otros alimentos. También se ha descrito una reactividad cruzada con otros hongos (Penicillium spp., Alternaria spp., Paecilomyces spp. y Cryptococcus spp.) y fármacos derivados total o parcialmente de estos (piperacilina-tazobactam o amoxicilina-ácido clavulánico). Los falsos negativos son infrecuentes y se asocian a las aspergilosis muy localizadas como traqueobronquitis en receptores de trasplante de pulmón. También se ha descrito que A. fumigatus puede tener menos concentración de GM que otras especies. La profilaxis o el tratamiento empírico con antifúngicos activos frente a Aspergillus spp. también reduce la sensibilidad de la técnica60,65,71,73,80.
La detección de GM es un criterio micológico de AI probable incluido en las definiciones de la EORTC/MSG61. Se recomienda realizar determinaciones seriadas, ya que la detección de GM ≥ 0,5 en dos sueros consecutivos aumenta tanto la especificidad como el valor predictivo positivo de la prueba. Si se realiza una única determinación, un índice de GM ≥ 0,7 se considera positivo. La detección de GM en LBA con índices ≥ de 0,5-1, también se considera como un criterio diagnóstico de AI probable70,81.
Detección de (1-3)-ß-D-glucanoComo se ha mencionado previamente, el BG es un componente de la pared celular fúngica que se libera con el desarrollo de la infección, pudiendo detectarse en el suero de los pacientes y que se considera una técnica de detección panfúngica, también útil en la detección de la aspergilosis41,76,82. Los métodos de detección de BG han demostrado una sensibilidad superior al 60% y una especificidad entre el 85 y el 100% en enfermos con neutropenia y micosis invasoras.
Se han realizado varios estudios prospectivos en pacientes con alto riesgo de padecer AI34,41,60,70,76,82,83. En el caso concreto de Fungitell, que emplea una detección cinética de BG y un punto de corte de 60-120 pg/ml34,70, se han obtenido valores diagnósticos aceptables en el diagnóstico de la AI en pacientes onco-hematológicos con neutropenia, con una sensibilidad del 64 al 88% y una especificidad cercana al 90%, con valores predictivos positivos y negativos del 70 al 89% y del 73-96%, respectivamente. La detección de BG precedía en muchos pacientes a la aparición de síntomas y signos clínico-radiológicos y al tratamiento antifúngico empírico.
Como ya se ha expuesto anteriormente, se han descrito falsos positivos en la detección de BG. Si bien el tratamiento empírico con antifúngicos no interfiere en la detección del BG, el uso de azitromicina y pentamidina intravenosas puede provocar falsos negativos45,84.
Detección de ácidos nucleicosSe han desarrollado numerosas técnicas para la detección de ADN de Aspergillus mediante PCR. Estas técnicas son muy sensibles (detectan entre 1 y 10 fg de ADN fúngico) y se pueden adaptar tanto a la detección de secuencias de ADN específicas de Aspergillus (PCR específica) como de secuencias de ADN comunes entre hongos (PCR panfúngica), en la mayoría de las muestras clínicas. La falta de un método estandarizado es la causa de que se describan resultados divergentes en la literatura, siendo difícil establecer estudios comparativos entre los distintos trabajos publicados debido a la variabilidad de los métodos utilizados. Por otro lado debe tenerse en cuenta que el número de casos de AI probada o probable en los distintos estudios realizados no es alto, lo que influye en la rentabilidad diagnóstica de la prueba85.
Es por ello que los trabajos publicados presentan una gran variabilidad en cuanto a su valor diagnóstico (sensibilidad 36-100%, especificidad 28-100%), pero la sensibilidad de la detección de ADN en el LBA puede ser superior al de la sangre y suero sobre todo cuando se ha instaurado un tratamiento antifúngico. El diagnóstico de la AI por PCR puede ser una alternativa útil en un futuro próximo, tanto para el diagnóstico como para la detección de resistencias potenciales a los antifúngicos o la monitorización del tratamiento antifúngico, ya que en los pacientes con AI con una respuesta favorable al tratamiento, desciende el número de muestras positivas76,86–89. Se está realizando un gran esfuerzo en varios centros europeos para estandarizar la detección de ADN de Aspergillus por PCR90 y alcanzar un consenso sobre el significado de un resultado positivo, establecer el tipo de muestra clínica óptima y la periodicidad de obtención de las muestras clínicas. La comercialización de métodos, como el LightCycler® SeptiFast Test (Roche)91,53, que detectan ADN e identifican varios agentes patógenos, incluido A. fumigatus, puede facilitar la realización de estudios multicéntricos que permitan inferir la utilidad diagnóstica de la técnica bajo las mismas premisas controladas y estandarizadas.
Combinación de técnicasSe han realizado varios estudios que han combinado la detección de GM y BG70,82 o la de GM, BG y PCR76, con el objetivo de mejorar el diagnóstico de la AI. En estos estudios se observaba un descenso de los falsos positivos y una mejora de la especificidad del diagnóstico de AI. Sin embargo, la aplicabilidad de estas combinaciones para su uso en el laboratorio clínico requiere estudios adicionales.
RecomendacionesCon los datos actuales se puede recomendar la detección de GM en sangre (≥ 0,5 en dos sueros consecutivos o ≥ 0,7 en un solo suero) y LBA (0,5-1) como una técnica de ayuda diagnóstica de la AI y como una herramienta de cribado para detectar anticipadamente la AI, durante los periodos de mayor riesgo en los enfermos onco-hematológicos adultos y receptores de trasplante de precursores hematopoyéticos (A-I) (tabla 4). En estos pacientes, parece haber una correlación estrecha entre el GM sérico y el pronóstico67,70,71,73–75. Para otros grupos de pacientes la detección de GM puede ser de utilidad, pero hasta el momento no existen suficientes datos que lo demuestren (B-II). Está sin definir la utilidad de la detección de GM en poblaciones de pacientes sin neutropenia, como los receptores de trasplante de órgano sólido (con excepción de los trasplantes de pulmón), los que padecen enfermedad granulomatosa crónica, los pacientes críticos, los infectados por el VIH o sida, etc.66,69,72,73.
Recomendaciones y grado de evidencia de las técnicas diagnósticas alternativas al cultivo en la aspergilosis invasora
| Técnica diagnóstica | Grado de recomendación |
| Detección de galactomanano en suero o lavado broncoalveolar de enfermos oncohematológicosa | A-I |
| Detección de galactomanano en suero, LCR o lavado broncoalveolar de otros pacientes | B-II |
| Detección de (1-3)-ß-D-glucano | A-I |
| Detección de ácidos nucleicos | B-II |
LCR: líquido cefalorraquídeo; LBA: lavado broncoalveolar; GM: Galactomanano.
La prueba de detección de BG se considera una técnica útil para detectar AI en todos los grupos de pacientes (A-I), aunque es un método escasamente utilizado en los laboratorios clínicos, por lo que existe poca experiencia. Los métodos basados en la PCR pueden ser herramientas útiles para el cribado de la AI81 y de otras micosis invasoras en pacientes de alto riesgo (B-II) aunque aun no existen evidencias sólidas como para recomendarlos como métodos habituales en un laboratorio asistencial70,76,34,87,89.
Diagnóstico de otras micosis alternativo al cultivoComo ya se ha indicado con anterioridad, las técnicas convencionales son poco sensibles y detectan la infección cuando ésta se encuentra muy avanzada, por lo que no suele responder al tratamiento antifúngico. En las dos últimas décadas del siglo pasado, se desarrollaron métodos de diagnóstico precoz para detectar infecciones virales en enfermos inmunodeprimidos, y se creyó que esta estrategia también era aplicable en micología médica y, más concretamente, en el diagnóstico de las IFIs oportunistas92. En este contexto, se desarrollaron técnicas para la detección de diferentes componentes fúngicos y de ácidos nucleicos. En este apartado revisaremos las técnicas diagnósticas existentes para detectar algunos hongos oportunistas como Cryptococcus entre otros, así como las especies que causan micosis primarias endémicas.
CriptococosisLa detección del antígeno capsular de Cryptococcus es uno de los métodos alternativos al cultivo que ha demostrado una mayor utilidad diagnóstica en el campo de las enfermedades infecciosas. Existen varias técnicas diseñadas, muchas de ellas comercializadas, que se utilizan rutinariamente en los laboratorios clínicos de todo el mundo. Estas técnicas pueden emplear anticuerpos policlonales o monoclonales antiglucuroxilomanano93. La mayor parte de ellas están basadas en la aglutinación de partículas de látex o en el enzimoinmunoanálisis, y se emplean en muestras de LCR, hematológicas, secreciones respiratorias y orina. Los falsos positivos son infrecuentes, aunque se han descrito en presencia de factor reumatoide, infecciones por Trichosporon spp., algunas bacterias y neoplasias94.
Estas técnicas se vienen aplicando en el diagnóstico clínico desde los años 70 del siglo pasado. Varios estudios demostraron su gran fiabilidad para detectar infecciones diseminadas y criptococosis meníngeas, con un descenso en su sensibilidad en caso de infecciones localizadas como las cutáneas o las pulmonares95,96. La aparición del sida alteró la epidemiología de la criptococosis y, por tanto, la detección del antígeno capsular empezó a utilizarse de forma habitual. En enfermos VIH+ con criptococosis, la sensibilidad de estas técnicas es cercana al 100% en LCR y alrededor del 95% en sangre, cifras superiores a las del examen microscópico y el cultivo. Los títulos que se consideran positivos dependen de la técnica empleada, pero generalmente títulos de 1/8 indican enfermedad activa97–99. Asimismo, la técnica puede emplearse para evaluar la evolución de los enfermos, realizando determinaciones seriadas del antígeno. Títulos en LCR superiores a 1/1.024 son indicativos de mal pronóstico y de posible fallo terapéutico.
Tras la aparición de la terapia antirretroviral combinada, la criptococosis ha disminuido significativamente en enfermos con sida. Sin embargo, se ha empezado a detectar como una micosis emergente en otros grupos de enfermos. En enfermos inmunodeprimidos no VIH+, la criptococosis suele aparecer como una infección diseminada, con fiebre, pérdida de peso y alteraciones de conciencia, con tendencia a la focalización pulmonar. En estos enfermos la sensibilidad de la detección antigénica es sólo del 75% en LCR o sangre, e incluso menor si sólo presentan una infección pulmonar localizada100. Recientemente se ha publicado un estudio multicéntrico en trasplantados de órgano sólido con criptococosis, en el que se observó que un título antigénico de 1/64 en suero puede ser diagnóstico de esta infección101.
La cuantificación del BG, que se utiliza como una técnica de detección panfúngica, parece ser ineficaz para diagnosticar estas infecciones, ya que Cryptococcus spp. apenas tienen este glucano entre los componentes de su pared.
En lo que se refiere a la detección de esta especie en muestras clínicas mediante técnicas moleculares, hay pocos estudios publicados con un número significativo de muestras102. No obstante, sí que se utilizan técnicas moleculares para la identificación de Cryptococcus en cultivo, así como para su clasificación y tipificación103.
Diagnóstico alternativo de la zigomicosis y otras especies de hongos oportunistas emergentesEl aumento en la prevalencia de la zigomicosis ha hecho que ascienda el interés en desarrollar técnicas de detección precoz con mayor rentabilidad que las técnicas convencionales, cuya fiabilidad para diagnosticar esta micosis no es muy elevada104. No obstante, hasta la fecha, el diagnóstico de la zigomicosis sigue basado en los procedimientos del examen microscópico y cultivo, ya que, la detección de antígenos y las técnicas de biología molecular apenas se han evaluado como técnicas de diagnóstico clínico. Estas especies tampoco tienen BG en su pared, aunque en algunos estudios publicados recientemente se incluyen algunos casos de mucormicosis, en los que lo enfermos tenían cantidades elevadas de este antígeno en sangre. Debe señalarse, no obstante, que el número de pacientes incluidos en estos trabajos es reducido y no se puede recomendar la técnica como un método de diagnóstico alternativo en esta micosis42;45.
Otra de las estrategias que puede ser de utilidad es la identificación directa de las especies de zigomicetos en muestras tisulares mediante técnicas de biología molecular basadas en la PCR. Con estas técnicas se amplifica el ADN fúngico de las biopsias donde se observan hifas y ya se han publicado algunos estudios donde se ha demostrado una rentabilidad superior a la del cultivo104. Sin embargo, esta técnica tiene la limitación de que muchas veces el estado del enfermo o la localización de la lesión no permiten la obtención de biopsias, por lo que se están desarrollando técnicas de detección de ácidos nucleicos en otros tipos de muestras clínicas. En 2008 se publicó un estudio realizado con un modelo animal de zigomicosis pulmonar en conejos. Se analizó la utilidad de una técnica de PCR en tiempo real en LBA y tejidos, con capacidad para diferenciar cuatro especies de zigomicetos, pertenecientes a los géneros Rhizopus, Mucor, Rhizomucor y Cunninghamella. Los cultivos tuvieron una sensibilidad del 60-95%, mientras que la sensibilidad de la PCR fue del 100% en todas las muestras105.
En lo que se refiere a otras especies oportunistas emergentes de géneros tales como Fusarium, Scedosporium o Trichosporon, la información disponible es escasa por lo que no pueden hacerse recomendaciones basadas en estudios clínicos significativos. Muchas de estas especies pueden detectarse con las técnicas de detección de BG, pero no hay datos que apoyen su utilización. En muchas de estas especies se han desarrollado técnicas de identificación y de tipificación basadas en la PCR, para ser utilizadas en cultivo o en muestras clínicas106. Algunas de ellas se han validado en modelos animales o con muestras clínicas humanas, pero no hay información suficiente como para hacer recomendaciones107,108.
Diagnóstico alternativo de la neumocistosisExisten varias técnicas para el diagnóstico de las infecciones causadas por P. jiroveci, hongo que no crece en los cultivos convencionales y cuyo diagnóstico ha estado basado en la microscopía. En los últimos años se han desarrollado técnicas alternativas, generalmente basadas en la PCR, siendo de mayor aplicabilidad clínica las PCR cuantitativas en tiempo real realizadas en muestras de esputo inducido y en LBA, que han permitido distinguir la colonización de la infección mediante el establecimiento de un cut-off. En pacientes VIH+, la sensibilidad y especificidad de estas pruebas superan el 95%. En enfermos no VIH, la sensibilidad es algo inferior, con valores predictivos positivos del 50% y negativo del 98%109,110. Por tanto, en este último tipo de pacientes, una PCR negativa podría descartar la presencia de la neumocistosis.
Otra posibilidad es la utilización de la detección del BG, compuesto que también se encuentra en P. jiroveci. Obviamente su detección no sería específica de esta especie, pero en enfermos con clínica compatible, la detección de este glucano podría ayudar a diagnosticar la infección, como se ha demostrado en algunos estudios42,111.
Diagnóstico alternativo de las micosis endémicasHistoplasmosisLa detección de un antígeno polisacárido de Histoplasma capsulatum en suero y orina se ha convertido en una técnica alternativa al cultivo de gran utilidad diagnóstica. Su principal limitación es que no se encuentra disponible fuera de zonas endémicas y no se ha comercializado112,113. La sensibilidad es más elevada en orina (80-90’%) que en suero y LBA (50-80%), y ha sido validada para enfermos VIH+ con infección diseminada. Recientemente se ha observado que en pacientes no VIH, la antigenuria y la antigenemia son de menor cuantía, por lo que la sensibilidad de este test desciende al 60-75%. Por ello, en enfermos inmunocompetentes con histoplasmosis aguda pulmonar, se recomienda realizar las determinaciones de antígeno en LBA, donde ha mostrado una sensibilidad del 93%114,115. Se han detectado falsos positivos con muestras de pacientes que tenían otras micosis. Por último, esta prueba es útil para monitorizar la respuesta del paciente al tratamiento en enfermos VIH+ con histoplasmosis113.
En cuanto a otras técnicas alternativas al cultivo, la detección de anticuerpos tiene una utilidad limitada en el diagnóstico de esta micosis. Los anticuerpos aparecen entre la segunda y la sexta semana de infección y, por tanto, no suelen servir para detectar la infección aguda aunque pueden ayudar a diagnosticar las complicaciones crónicas de esta infección, así como la meningitis113. En enfermos VIH+ con histoplasmosis sólo son detectables en el 50% de los casos y la presencia de un título aislado sólo indica que el paciente ha tenido contacto con H. capsulatum.
En los últimos años se han desarrollado técnicas diagnósticas basadas en la PCR, sobre todo en la cuantitativa en tiempo real. Se han empleado sólo en unas pocas decenas de enfermos, pero parecen ser muy útiles en pacientes VIH+ con infección diseminada. Son métodos muy específicos y con una sensibilidad muy elevada en muestras respiratorias y en biopsias, aunque algo inferior en sangre y suero116,117.
BlastomicosisLa detección de anticuerpos no es una técnica muy útil en esta enfermedad, ya que tiene una sensibilidad baja, alrededor de un 10% en algunas publicaciones, y reacciones cruzadas con muchas especies fúngicas118. Por ello, se han desarrollado técnicas de detección antigénica, tanto en orina como en muestras respiratorias. Aunque no se ha evaluado un número representativo de enfermos, la sensibilidad de esta técnica es del 90% en orina y del 75% en LBA. Su principal limitación son las reacciones cruzadas con otras infecciones fúngicas tanto endémicas como oportunistas, principalmente aspergilosis y criptococosis119,120. Apenas hay datos publicados sobre técnicas de diagnóstico molecular aplicables a la detección de esta micosis121.
CoccidioidomicosisLa detección de anticuerpos mediante inmunodifusión, ELISA, aglutinación de partículas de látex o fijación de complemento son útiles para diagnosticar la infección en enfermos inmunocompetentes, presentando elevados valores de sensibilidad y especificidad. Los anticuerpos suelen detectarse durante la primera semana de infección y también ayudan a realizar el seguimiento de la respuesta al tratamiento. En enfermos inmunodeprimidos el diagnóstico es más complicado, por lo que se están desarrollando técnicas de detección antigénica y de ácidos nucleicos, aunque se han aplicado en pocos casos y no se conoce su utilidad clínica121–123.
ParacoccidioidomicosisPara el diagnóstico de la paracoccidioidomicosis existen pruebas de detección de anticuerpos, que muestran una gran sensibilidad en enfermos inmunocompetentes, sobre todo si se combinan dos de estas técnicas. Se han desarrollado métodos basados en la inmunodifusión, la contrainmunoelectroforesis, la fijación del complemento, la inmunofluorescencia indirecta y el ELISA. Los títulos son más elevados en aquellos pacientes que tienen la forma grave de la enfermedad, pudiéndose detectar durante dos años después de tratar la infección118. También se han desarrollado técnicas de detección antigénica para enfermos inmunodeprimidos y para realizar el seguimiento de la infección. El antígeno más utilizado es el gp43, que se ha evaluado en muestras sanguíneas de centenares de enfermos, demostrando una gran sensibilidad diagnóstica y utilidad para realizar el seguimiento de esta infección. También muestra una buena fiabilidad en LCR y muestras respiratorias. Asimismo se han publicado algunos estudios sobre la detección del antígeno gp70 para el diagnóstico de esta micosis124–126. Existen pruebas de diagnóstico molecular basadas en la PCR que podrían tener utilidad clínica en breve, pero deben ser evaluadas con mayor profundidad127.
Infecciones por Penicillium marneffeiA diferencia de las cuatro micosis anteriores, la infección por P. marneffei es una micosis oportunista endémica, que afecta a personas VIH+ del sudeste asiático. Se han descrito técnicas de detección de anticuerpos, de antígenos y moleculares basadas en la PCR convencional y cuantitativa. En estudios realizados con varias decenas de enfermos VIH+, la detección de antígenos ha mostrado una sensibilidad y una especificidad superiores al 90%. Las técnicas moleculares parecen ser muy rentables, pero no han sido evaluadas suficientemente hasta la fecha128–130.
RecomendacionesTras la revisión de la literatura disponible, en la tabla 5 se incluyen las recomendaciones para la utilización de las distintas técnicas alternativas al cultivo.
Recomendaciones y grado de evidencia de las técnicas diagnósticas alternativas al cultivo en otras especies fúngicas
| Infección fúngica/Técnica o método diagnóstico | Grado de recomendación |
| Criptococosis | |
| Detección de antígeno en LCR y en suero en enfermo VIH+ con infección diseminada o meníngea | A-II |
| Detección de antígeno en enfermos no VIH+ | B-II |
| Detección de antígeno sérico en enfermos con infección localizada (pulmonar o cutánea) | B-II |
| Detección de antígeno para seguimiento de la respuesta al tratamiento | B-II |
| Detección sérica de (1,3)-β-D-glucano | C-III |
| Detección de ácidos nucleicos | Sin resolver |
| Zigomicosis y otros hongos oportunistas | |
| Detección sérica de (1,3)-β-D-glucano para diagnosticar la zigomicosis | C-III |
| Detección de ácidos nucleicos para diagnosticar la zigomicosis | B-III |
| Utilización de técnicas alternativas al cultivo en especies emergentes como Fusarium, Scedosporium, Trichosporon y Saccharomyces | Sin resolver |
| Neumocistosis | |
| Detección sérica del antígeno (1,3)-β-D-glucano | B-II |
| Detección de ácidos nucleicos en enfermos VIH+ en muestras respiratorias | A-II |
| Detección de ácidos nucleicos en enfermos no VIH+ en muestras respiratorias | B-II |
| Histoplasmosis | |
| Detección de antígeno en orina de enfermos VIH+ con infección diseminada | A-II |
| Detección de antígeno en sangre o lavado broncoalveolar de enfermos VIH+ con infección diseminada | B-II |
| Detección de antígeno en orina o en sangre de enfermos no VIH | B-II |
| Detección de antígeno en lavado broncoalveolar de enfermos no VIH con infección pulmonar localizada | A-II |
| Detección de antígeno para seguimiento de la respuesta al tratamiento | B-II |
| Detección de anticuerpos en enfermos VIH+ | C-II |
| Detección de anticuerpos en enfermos inmunocompetentes con histoplasmosis en fase aguda | C-II |
| Detección de anticuerpos en enfermos con histoplasmosis crónica o meníngea | A-II |
| Detección de ácidos nucleicos | B-III |
| Blastomicosis | |
| Detección de anticuerpos | C-II |
| Detección de antígenos en sangre, orina o muestras respiratorias | B-III |
| Detección de ácidos nucleicos | B-III |
| Coccidioidomicosis | |
| Detección de anticuerpos para diagnosticar la infección | A-II |
| Detección de anticuerpos para realizar seguimiento de la respuesta al tratamiento | B-II |
| Detección de antígenos en sangre, orina o muestras respiratorias | B-III |
| Detección de ácidos nucleicos | B-III |
| Paracoccidioidomicosis | |
| Detección de anticuerpos para diagnosticar la infección, combinando dos técnicas serológicas diferentes | A-II |
| Detección de anticuerpos en enfermos inmunodeprimidos | C-II |
| Detección de anticuerpos para realizar seguimiento de la respuesta al tratamiento | C-II |
| Detección de antígenos en sangre de enfermos inmunodeprimidos | B-II |
| Detección de antígenos en sangre para realizar seguimiento de la respuesta al tratamiento | B-III |
| Detección de ácidos nucleicos | B-III |
| Infección por Penicillium marneffei | |
| Detección de anticuerpos | C-III |
| Detección de antígenos en sangre y otras muestras | B-II |
| Detección de ácidos nucleicos | B-III |
La identificación de las especies fúngicas mediante métodos de clasificación molecular se ha aproximado a la práctica clínica, desde la automatización de las técnicas de secuenciación de ácidos nucleicos. Estos métodos permiten conocer la secuencia de las cepas clínicas e identificarlas al introducirlas en bases de datos de secuencias conocidas. La revisión de estas técnicas no se encuentra entre los objetivos de estas recomendaciones, por lo que se incluyen varias referencias para el lector interesado131–134.
Debe indicarse que la identificación molecular está demostrando que la clasificación por técnicas convencionales es poco fiable para algunas especies, fundamentalmente las poco habituales. Además, se ha observado que las especies más frecuentes son, en ocasiones, conjuntos de especies y que existen numerosas especies crípticas. No obstante, la mayor parte de estos hallazgos no tienen aun trascendencia clínica, por lo que no se pueden hacer recomendaciones prácticas sobre la identificación molecular.
Para poder realizar estas recomendaciones hace falta estandarizar las técnicas de secuenciación y consensuar las dianas de ácidos nucleicos con poder de discriminación suficiente, como ya se ha hecho con algunas especies132. Desde el punto de vista de los laboratorios clínicos, las limitaciones de las técnicas de identificación convencional tienen importancia cuando una clasificación errónea de una especie puede llevar a instaurar un tratamiento antifúngico inadecuado. Estos casos son excepcionales, por lo que en la mayor parte de los casos, es preferible realizar un test de sensibilidad a los antifúngicos que realizar métodos de identificación molecular que actualmente sólo se encuentran disponibles en algunos centros de referencia.
Técnicas de sensibilidad in vitro a los antifúngicosDesde la estandarización de los métodos de referencia para hacer estudios de sensibilidad a los antifúngicos por parte de comités como el EUCAST (European Committee on Antimicrobial Susceptibility Testing) y el CLSI estadounidense (Clinical Laboratory Standard Institute)135–140, los laboratorios clínicos pueden realizar estas pruebas y conocer el perfil de sensibilidad de las cepas clínicas. Desde la aparición de nuevas moléculas antifúngicas, la detección de la resistencia es fundamental a la hora de elegir una alternativa terapéutica u otra141–144.
Asimismo, se han establecido puntos de corte para interpretar los resultados de los estudios de sensibilidad in vitro. Estos puntos de corte sólo son aplicables a las infecciones por Candida spp., y sólo para algunos de los antifúngicos disponibles. Para las infecciones por otras especies de levaduras y por hongos filamentosos, aun no se han establecido puntos de corte, aunque se recomienda no tratar con fármacos que sean inactivos in vitro frente a la especie causante de la micosis142,143,145.
Respecto a las técnicas que pueden ser utilizadas por los laboratorios clínicos debe resaltarse que se han desarrollado varios métodos comerciales y algunas técnicas de difusión en agar, que muestran una gran correlación con los procedimientos de referencia y que son más prácticos para los laboratorios clínicos. Por ello, se recomienda que los laboratorios asistenciales utilicen métodos de difusión con discos o técnicas comerciales que hayan demostrado su fiabilidad en diferentes estudios146–154.
RecomendacionesNo existen estudios que hayan aportado suficientes evidencias como para hacer recomendaciones estrictas sobre las indicaciones de los estudios de sensibilidad, por lo que éstas se basan en opiniones de expertos, conferencias de consenso y en algunos estudios descriptivos. Según estos datos, se han elaborado las recomendaciones que figuran en la tabla 6.
Recomendaciones y grado de evidencia de los estudios de sensibilidad in vitro
| Situación | Recomendación |
| Cepas procedentes de infecciones invasoras | B-III |
| Cepas procedentes de enfermos inmunodeprimidos | B-III |
| Cepas procedentes de enfermos con fracaso terapéutico | A-II |
| Fungemias de brecha | A-II |
| Cepas procedentes de enfermos que han recibido profilaxis | A-II |
| Cepas pertenecientes a especies poco frecuentes | A-II |
| Estudios epidemiológicos | A-I |
Algunos expertos recomiendan realizar estudios de sensibilidad de todas las cepas que proceden de infecciones invasoras o de enfermos con algún tipo de inmunosupresión, particularmente en el caso de infecciones por Candida. Esta recomendación se basa en el concepto de la vigilancia epidemiológica y en las evidencias que demuestran que los microorganismos resistentes a los antifúngicos se aíslan, más frecuentemente, en enfermos inmunodeprimidos que han sido tratados con antifúngicos en varias ocasiones.
Asimismo, hay varias situaciones en las que realizar un estudio de sensibilidad in vitro puede aportar información muy útil para el manejo del enfermo. Entre estas destacan, cepas que provengan de fracasos terapéuticos, fungemias de brecha, casos de enfermos que hayan recibido profilaxis antifúngica previa y en especies poco frecuentes, de las que se desconoce su espectro de sensibilidad in vitro.
Conviene hacer especial énfasis en la importancia de los estudios epidemiológicos. Varios trabajos han demostrado que la vigilancia epidemiológica de las infecciones fúngicas ayuda a conocer los reservorios, las vías de transmisión y los factores de riesgo de la infección, así como el perfil de sensibilidad de las distintas especies y su prevalencia149,155. De esta forma se pueden establecer cuáles son los tratamientos iniciales más adecuados o si se debe cambiar de tratamiento una vez que se ha identificado la especie.
Conflicto de interesesEn los últimos cinco años, E.M.M. ha recibido fondos de investigación del Fondo de Investigación Sanitaria (FIS). Ha recibido honorarios por ponencias de Astellas Pharma y Pfizer.
En los últimos cinco años, G.Q. ha recibido fondos de investigación de la Consejería de Educación, Universidades e Investigación y del Departamento de Industria, Comercio y Turismo del Gobierno Vasco-Eusko Jaurlaritza, del Fondo de Investigación Sanitaria (FIS), de la Universidad del País Vasco-Euskal Herriko Unibertsitatea, de Astellas Pharma y de Pfizer. Ha recibido honorarios por ponencias de Astellas Pharma y Pfizer.
En los últimos cinco años, F.S. ha recibido fondos de investigación de Astellas Pharma. Ha recibido ayudas de Gilead Sciences, Merck Sharp and Dohme, Pfizer, y Schering Plough.
En los últimos cinco años, J.G.R. ha recibido fondos de investigación de Gilead Sciences, Pfizer. Ha sido asesor/consultor de Gilead Sciencies. Ha recibido honorarios por ponencias de Pfizer, Merck Sharp and Dohme y Astellas.
En los últimos cinco años, J.G. ha recibido fondos de investigación de Basilea Pharmaceutica, bioMerieux, Fondo de Investigación Sanitaria (FIS), Fundación Mutua Madrileña, y la European Society for Clinical Microbiology and Infectious Diseases (ESCMID). Ha sido asesor/consultor de Gilead Sciences. Ha recibido honorarios por ponencias de Gilead Sciences, Pfizer y Astellas Pharma.
En los últimos cinco años, J.L.R.T. ha recibido fondos de investigación de Astellas Pharma, Gilead Sciences, Merck Sharp and Dohme, Pfizer, Schering Plough, Soria Melguizo SA, the European Union, the Spanish Agency for International Cooperation, the Spanish Ministry of Culture and Education, The Spanish Health Research Fund, The Instituto de Salud Carlos III, The Ramon Areces Foundation, The Mutua Madrileña Foundation. Ha sido consultor/asesor de the Panamerican Health Organization, Gilead Sciences, Merck Sharp and Dohme, Mycognostica, Pfizer, y Schering Plough. Ha recibido honorarios por ponencias de Gilead Sciences, Merck Sharp and Dohme, Pfizer, y Schering Plough.
En los últimos cinco años, M.C.E. ha recibido fondos de investigación de Astellas Pharma, bioMerieux, Gilead Sciences, Merck Sharp and Dohme, Pfizer, Schering Plough, Soria Melguizo SA, the European Union, the ALBAN program, the Spanish Agency for International Cooperation, the Spanish Ministry of Culture and Education, The Spanish Health Research Fund, The Instituto de Salud Carlos III, The Ramon Areces Foundation, The Mutua Madrileña Foundation. Ha sido asesor/consultor de the Panamerican Health Organization, Gilead Sciences, Merck Sharp and Dohme, Pfizer, and Schering Plough. Ha recibido honorarios por ponencias de Gilead Sciences, Merck Sharp and Dohme, Pfizer, Astellas Pharma y Schering Plough.
Otros autores: Sin conflictos
En memoria de José Pontón, coautor de estas Recomendaciones y amigo de todos nosotros.
Los miembros del Grupo de Estudio de Micología de la SEIMC (GEMICOMED) que han participado como comité de consenso de las recomendaciones son los siguientes: José M. Aguado (Hospital Doce de Octubre), Benito Almirante (Hospital de Vall d¿Hebron), Francisco Alvarez-Lerma (Hospital del Mar), Marcio Borges (Hospital Son Llátzer), Jordi Carratalá (Hospital de Bellvitge), Carlos Cervera (Hospital Clinic de Barcelona), Rafael de la Cámara (Hospital de la Zarzuela), Julián de la Torre Cisneros (Instituto Maimonides de Investigación Biomédica de Córdoba (IMIBIC)-Hospital Reina Sofía-Universidad de Córdoba), Jesús Fortún (Hospital Ramón y Cajal), José Garnacho (Hospital Virgen del Rocío), Joan Gavaldá (Hospital de Vall d¿Hebron), Alvaro Lassaletta (Hospital del Niño Jesús), Cristóbal León (Hospital de Valme), Manuel Lizasoain (Hospital Doce de Octubre), Luis Madero (Hospital del Niño Jesús), Pilar Martín-Dávila (Hospital Ramón y Cajal), José Mensa (Hospital Clinic de Barcelona), Asunción Moreno (Hospital Clinic de Barcelona), Patricia Muñoz (Hospital Gregorio Marañón), José T. Ramos Amador (Hospital de Getafe), Montserrat Rovira (Hospital Clinic de Barcelona), Isabel Ruiz (Hospital de Vall d¿Hebron), Jesús Ruiz-Contreras (Hospital Doce de Octubre), Miguel Salavert (Hospital La Fé), Miguel A. Sanz (Hospital La Fé) y Lourdes Vázquez (Hospital Clínico de Salamanca).
En el Anexo 1 se incluye el listado de los investigadores del grupo de Estudio de Micología Médica de la SEIMC (GEMICOMED).