Los perfiles de expresión génica (PEG) en las primeras semanas de tratamiento de pacientes con hepatitis C crónica pueden servir para evaluar la eficacia de la terapia basada en interferón. El objetivo de este trabajo fue estudiar los PEG de pacientes respondedores y no respondedores precoces antes y tras 12 semanas de tratamiento con peginterferón alfa y ribavirina.
MétodosEstudio observacional donde se analizaron los PEG de 12 pacientes con hepatitis C crónica candidatos a recibir tratamiento con peginterferón alfa y ribavirina.
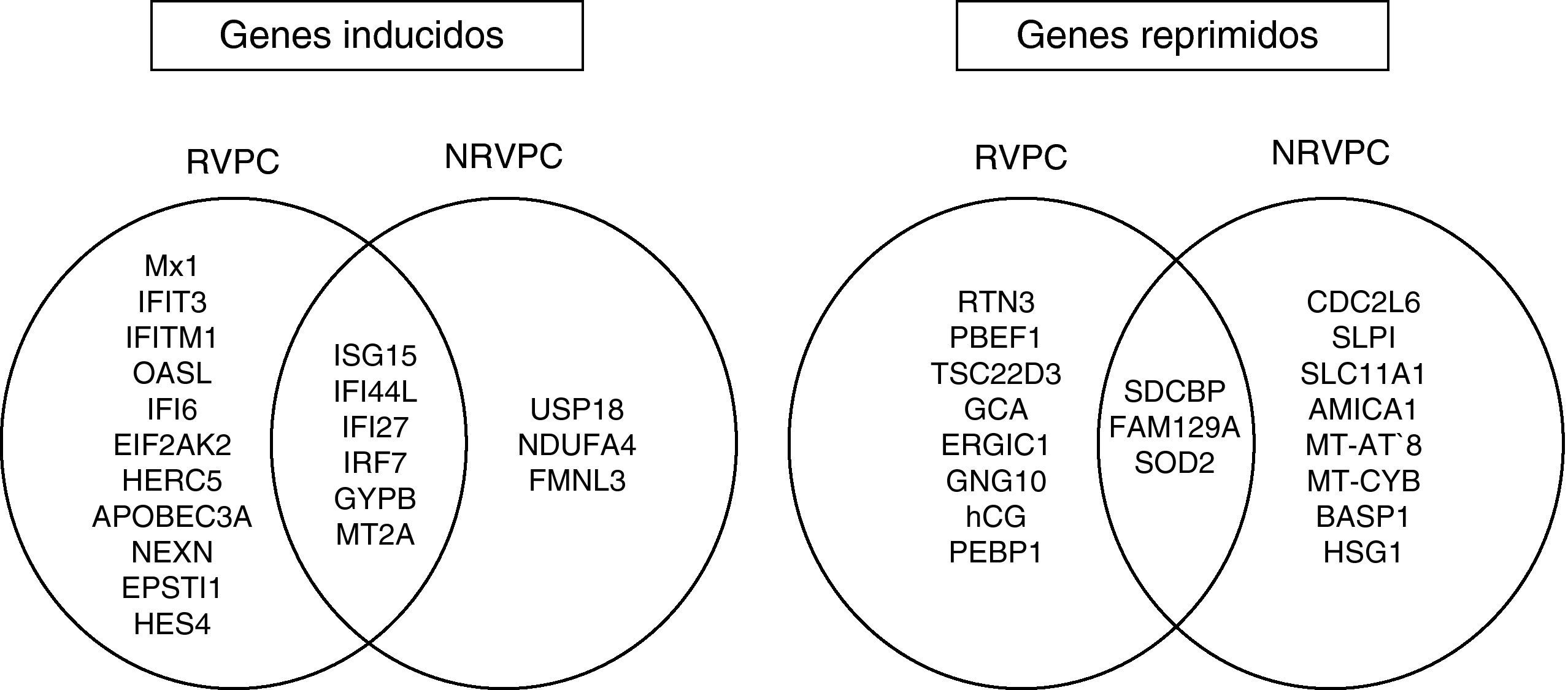
ResultadosDe los 12 pacientes estudiados, seis mostraron una respuesta virológica precoz completa, mientras que seis no lograron controlar la viremia. En respondedores precoces, el tratamiento con peginterferón y ribavirina indujo un aumento de la expresión de un mayor número de genes de respuesta al interferón (ISG15, IFI6, IFI44L, IFI27, MX1, OASL, IRF7, IFIT3, IFITM1, EIF2AK2, HERC5 y APOBEC3A) que en no respondedores (ISG15, IFI44L, IFI27, IRF7, USP18) (p<0,05). En ambos grupos, se observaron cambios en los niveles de algunos genes hasta ahora escasamente descritos en el tratamiento de la hepatitis C.
ConclusionesLos PEG descritos en este trabajo pueden ayudar a comprender mejor la patogénesis de la infección, así como las bases fisiológicas de la respuesta al tratamiento. Así, el mayor efecto del tratamiento sobre la expresión de genes de respuesta al interferón observado en respondedores podría explicar su mejor control de la carga viral.
Gene expression profiling in the first weeks of treatment of patients with chronic hepatitis C may contribute to better evaluate the response to interferon-based therapy. The objective of this study was to evaluate the gene expression profiles of early responders and non-responders before, and after 12 weeks of treatment with peginterferon alfa and ribavirin.
MethodsGene expression profiles were analysed in 12 patients with chronic hepatitis C, and scheduled for treatment with peginterferon alpha and ribavirin.
ResultsOf the 12 patients studied, six showed a complete early virological response, while six failed to control viremia. In early responders, treatment with peginterferon and ribavirin induced an increased expression of a larger number of interferon-induced genes (ISG15, IFI6, IFI44L, IFI27, MX1, OASL, IRF7, IFIT3, IFITM1, EIF2AK2, HERC5 and APOBEC3) than in non-responders (ISG15, IFI44L, IFI27, IRF7, USP18) in the first twelve weeks of treatment (P<0.05). In both groups, there were changes in the levels of certain genes poorly described in the treatment of hepatitis C so far.
ConclusionsThe gene expression profiles described in this study provide a new insight to understanding the pathogenesis of the disease and treatment effect. The more marked effect of the treatment on the expression of interferon-response genes observed in early responders could explain their better control of viral load.
El virus de la hepatitis C (VHC) constituye la causa más prevalente de hepatitis crónica en los países desarrollados. La historia natural de la infección por el VHC se caracteriza por una gran tendencia a la cronicidad, haciendo que esta enfermedad sea una causa importante de morbilidad y mortalidad. La infección por el VHC está implicada en el aumento de la incidencia de carcinoma hepatocelular (CHC) en muchos países desarrollados, donde la proporción atribuible al VHC oscila del 50 al 70%1–3. La terapia combinada de peginterferón alfa y ribavirina induce una respuesta virológica sostenida (RVS) en aproximadamente el 45% de los pacientes con genotipo 1 y más elevada, alrededor del 80%, en los infectados con los genotipos 2 y 34,5.
Además de los factores virales y ambientales, la diversidad genética del huésped contribuye al espectro de resultados clínicos de pacientes crónicamente infectados por el VHC. Los estudios de expresión génica abren la oportunidad de desarrollar perfiles que puedan distinguir, identificar y clasificar subconjuntos concretos de la enfermedad y predecir la evolución de la misma, así como la respuesta al tratamiento para distinguir respondedores de no respondedores6. El objetivo de este estudio fue evaluar los perfiles de expresión génica de pacientes respondedores y no respondedores precoces antes y tras 12 semanas de tratamiento con peginterferón alfa y ribavirina.
Material y métodosDiseño del estudio y pacientesSe llevó a cabo un estudio observacional prospectivo en el cual se incluyeron 14 pacientes ambulatorios pertenecientes a los Servicios de Medicina Digestiva del Hospital Clínico Universitario de Valladolid (HCUV) y del Hospital Universitario Río Hortega (HURH) desde mayo de 2008 hasta septiembre de 2009.
Criterios de inclusión: pacientes mayores de 18 años con infección crónica por el VHC candidatos a recibir tratamiento con peginterferón alfa 2a (180μg/semana) o 2b (1,5μg/kg/semana) y ribavirina (800mg/día para pacientes con genotipo no 1 y 1.000-1.200mg/día en función del peso para pacientes con genotipo 1). Todos los pacientes firmaron el consentimiento escrito de participación.
Criterios de exclusión: se excluyeron los pacientes que presentaron coinfecciones con el VIH u otras hepatitis, los que abandonaron el tratamiento y los que no firmaron la hoja de consentimiento informado.
Población control: se incluyeron controles sanos (n=5) que fueron trabajadores sanitarios de edad similar a pacientes y que no presentaron infección por el VHC ni antecedentes clínicos relevantes.
Se recogió una hoja de consentimiento informado. El protocolo del estudio, tanto en los aspectos científicos como éticos, fue aprobado por el Comité Científico de Investigación Clínica de los dos hospitales participantes.
MuestrasPara la cuantificación de la expresión génica, las muestras de sangre de los pacientes se recogieron en tubos PAXgene antes y tras doce semanas de iniciar el tratamiento. En controles sanos, también se obtuvo una muestra en el mismo tipo de tubo. Los tubos PAXgene se mantuvieron en posición vertical a temperatura ambiente un tiempo mínimo de dos horas antes de su procesado o de su congelación (−70°C) para asegurar la lisis completa de las células y la estabilización del ARN. Se recogió una segunda muestra de sangre en un tubo de EDTA para la determinación del polimorfismo rs1297860 de la IL28B, así como para la determinación de parámetros hematológicos. Se recogió además una tercera muestra de sangre en tubo seco para la determinación de parámetros bioquímicos, genotipo y carga viral.
Cuantificación de la expresión génicaPara la extracción del ARN total se empleó el Kit «PAXgene blood RNA kit» de PreAnalytiX siguiendo las instrucciones del fabricante. La calidad del ARN total obtenido se evaluó mediante el equipo «QIAxcel System» de Qiagen. El chip utilizado para estudiar los perfiles de expresión del genoma humano completo fue el «Human genome oligo array service V2.0» suministrado por CapitalBio. Para la determinación de la expresión génica, en primer lugar, se realizó una etapa de amplificación y marcaje del ARN con el kit «Amino Allyl MessageAmp II aRNA Amplification Kit» de Applied Biosystems siguiendo las instrucciones del fabricante (Cy5: fluoróforo para el etiquetado de las muestras de pacientes; Cy3: fluoróforo para el etiquetado del pool de muestras control) y se fragmentó el ARN etiquetado obtenido mediante el reactivo «RNA Fragmentation Reagents» de Applied Biosystems. Posteriormente, la hibridación de la mezcla (muestra+pool control) con los microarrays se llevó a cabo a 42°C durante toda la noche en un ambiente de humedad con el aparato BioMixerTM II. El escaneado se realizó con el aparato «LuxScan 10K» de CapitalBio.
Análisis del polimorfismo rs1297860 de la IL28BPara la detección del polimorfismo rs1297860 de la IL28B se empleó el kit Lightmix IL28B y el kit LightCycler FastStart DNA Master HybProbe de Roche® para realizar la mezcla de reacción siguiendo las instrucciones del fabricante, mediante el uso del temociclador LightCycler® 480II de Roche®.
Recogida de datos clínicos y de laboratorioEl ARN del VHC fue analizado en una muestra de suero usando el kit «COBAS® TaqMan® HCV Test, v2.0» en el instrumento COBAS® AmpliPrep de Roche® y el genotipo viral fue identificado con un ensayo de sondas en tira de nitrocelulosa (LiPA: Line Probe Assay) usando el kit «VERSANT HCV Amplification 2.0» y «VERSANT® HCV Genotype 2.0 Assay (LiPA)» de Siemens® con el analizador Auto-LIPA 48 (INNOGENETICS®).
La duración de la infección por el VHC, en pacientes con historia de uso de drogas por vía parenteral se estimó tomando como punto inicial el momento en el que se comenzó su uso. Para receptores de sangre, el punto inicial para estimar la duración de la infección fue la fecha de la primera transfusión que recibieron. Para aquellos pacientes con un origen de transmisión no identificado, el punto inicial fue el momento del diagnóstico.
En relación al consumo de alcohol se consideró el consumo de cantidades superiores a 50 gramos de alcohol por día durante más de doce meses como un consumo elevado de alcohol.
El grado de fibrosis se estimó mediante el índice APRI (aspartate aminotransferase to platelet count ratio index). Se calculó mediante la siguiente fórmula: valor (AST/ULN/Recuento de plaquetas*103/μl)*100 (el límite superior de normalidad [ULN] en nuestro laboratorio fue 39 UI/L). Un índice APRI ≤ 0,5 fue considerado como ausencia de fibrosis hepática, un índice APRI > 1,5 fue considerado como indicativo de fibrosis7 y entre 0,5 y 1,5 como valores indeterminados.
Estadística y análisis de datosPara la normalización, la estadística y el clustering de los datos se utilizó el paquete bioinformático de Biotools en lenguaje R8. El modelo lineal utilizado para el análisis estadístico se realizó con el paquete limma9 en R. Se calculó el logaritmo en base 2 del ratio (fluorescencia muestra/fluorescencia controles) para cada gen analizado en cada array para conocer la expresión génica de los pacientes respecto al grupo control. Posteriormente se realizó un ajuste de la estadística lineal para obtener el valor medio de los valores logarítmicos en cada grupo a comparar. De esta manera, para evaluar las variaciones en la expresión génica entre el inicio y la semana 12 de tratamiento en respondedores y no respondedores teniendo como basal el grupo control, se realizó la diferencia de los valores medios logarítmicos correspondientes a los dos grupos de comparación (logEG). Las comparaciones de la expresión génica entre el inicio y la semana 12 distribuyendo a los pacientes en función de su evolución clínica (RVS: respuesta virológica sostenida; NRVS: no respuesta virológica sostenida) se realizaron de manera similar.
Para el clustering se seleccionaron los genes que presentaron un logEG mayor de 1 o menor de –1 (p<0,05). La corrección para controlar la tasa de falsos positivos del análisis múltiple se realizó con el método Benjamini y Hochberg10.
ResultadosSe obtuvo ARN de calidad adecuada para el análisis de microarrays de 12 pacientes. De ellos, 6 mostraron una respuesta virológica precoz completa (RVPC), mientras que 6 no lograron controlar la viremia (NRVPC) mostrando carga viral detectable a las doce semanas después del comienzo del tratamiento.
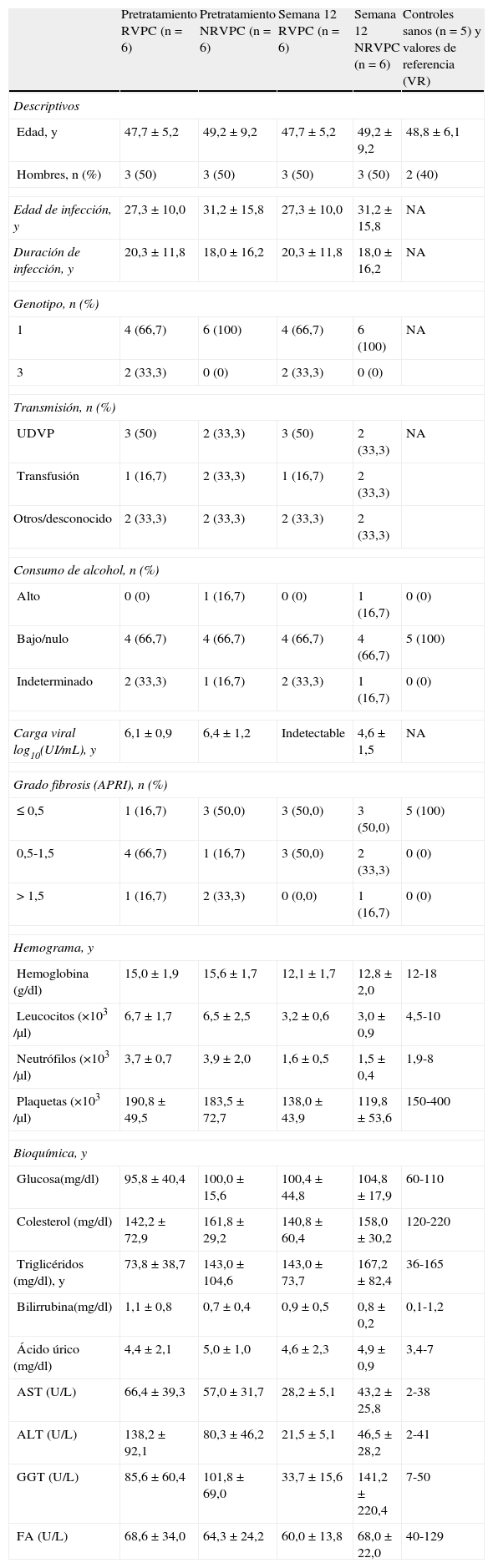
Características clínicas y analíticas de los pacientesEl 70% de los pacientes RVPC y el 100% de los pacientes NRVPC (tabla 1) presentaron genotipo viral 1. El grado de fibrosis estimado mediante el índice APRI fue similar en ambos grupos tras 12 semanas de tratamiento. El tratamiento provocó una disminución de las series roja, blanca y plaquetar, así como un aumento en el nivel de triglicéridos y una disminución en los niveles de transminasas en ambos grupos siendo más acusado el descenso de GOT y GPT en el grupo RVPC. La GGT descendió en RVPC, pero aumentó de forma llamativa en el grupo NRVPC.
Características clínicas de pacientes y grupo control
| Pretratamiento RVPC (n=6) | Pretratamiento NRVPC (n=6) | Semana 12 RVPC (n=6) | Semana 12 NRVPC (n=6) | Controles sanos (n=5) y valores de referencia (VR) | |
| Descriptivos | |||||
| Edad, y | 47,7±5,2 | 49,2±9,2 | 47,7±5,2 | 49,2±9,2 | 48,8±6,1 |
| Hombres, n (%) | 3 (50) | 3 (50) | 3 (50) | 3 (50) | 2 (40) |
| Edad de infección, y | 27,3±10,0 | 31,2±15,8 | 27,3±10,0 | 31,2±15,8 | NA |
| Duración de infección, y | 20,3±11,8 | 18,0±16,2 | 20,3±11,8 | 18,0±16,2 | NA |
| Genotipo, n (%) | |||||
| 1 | 4 (66,7) | 6 (100) | 4 (66,7) | 6 (100) | NA |
| 3 | 2 (33,3) | 0 (0) | 2 (33,3) | 0 (0) | |
| Transmisión, n (%) | |||||
| UDVP | 3 (50) | 2 (33,3) | 3 (50) | 2 (33,3) | NA |
| Transfusión | 1 (16,7) | 2 (33,3) | 1 (16,7) | 2 (33,3) | |
| Otros/desconocido | 2 (33,3) | 2 (33,3) | 2 (33,3) | 2 (33,3) | |
| Consumo de alcohol, n (%) | |||||
| Alto | 0 (0) | 1 (16,7) | 0 (0) | 1 (16,7) | 0 (0) |
| Bajo/nulo | 4 (66,7) | 4 (66,7) | 4 (66,7) | 4 (66,7) | 5 (100) |
| Indeterminado | 2 (33,3) | 1 (16,7) | 2 (33,3) | 1 (16,7) | 0 (0) |
| Carga viral log10(UI/mL), y | 6,1±0,9 | 6,4±1,2 | Indetectable | 4,6±1,5 | NA |
| Grado fibrosis (APRI), n (%) | |||||
| ≤ 0,5 | 1 (16,7) | 3 (50,0) | 3 (50,0) | 3 (50,0) | 5 (100) |
| 0,5-1,5 | 4 (66,7) | 1 (16,7) | 3 (50,0) | 2 (33,3) | 0 (0) |
| > 1,5 | 1 (16,7) | 2 (33,3) | 0 (0,0) | 1 (16,7) | 0 (0) |
| Hemograma, y | |||||
| Hemoglobina (g/dl) | 15,0±1,9 | 15,6±1,7 | 12,1±1,7 | 12,8±2,0 | 12-18 |
| Leucocitos (×103 /μl) | 6,7±1,7 | 6,5±2,5 | 3,2±0,6 | 3,0±0,9 | 4,5-10 |
| Neutrófilos (×103 /μl) | 3,7±0,7 | 3,9±2,0 | 1,6±0,5 | 1,5±0,4 | 1,9-8 |
| Plaquetas (×103 /μl) | 190,8±49,5 | 183,5±72,7 | 138,0±43,9 | 119,8±53,6 | 150-400 |
| Bioquímica, y | |||||
| Glucosa(mg/dl) | 95,8±40,4 | 100,0±15,6 | 100,4±44,8 | 104,8±17,9 | 60-110 |
| Colesterol (mg/dl) | 142,2±72,9 | 161,8±29,2 | 140,8±60,4 | 158,0±30,2 | 120-220 |
| Triglicéridos (mg/dl), y | 73,8±38,7 | 143,0±104,6 | 143,0±73,7 | 167,2±82,4 | 36-165 |
| Bilirrubina(mg/dl) | 1,1±0,8 | 0,7±0,4 | 0,9±0,5 | 0,8±0,2 | 0,1-1,2 |
| Ácido úrico (mg/dl) | 4,4±2,1 | 5,0±1,0 | 4,6±2,3 | 4,9±0,9 | 3,4-7 |
| AST (U/L) | 66,4±39,3 | 57,0±31,7 | 28,2±5,1 | 43,2±25,8 | 2-38 |
| ALT (U/L) | 138,2±92,1 | 80,3±46,2 | 21,5±5,1 | 46,5±28,2 | 2-41 |
| GGT (U/L) | 85,6±60,4 | 101,8±69,0 | 33,7±15,6 | 141,2±220,4 | 7-50 |
| FA (U/L) | 68,6±34,0 | 64,3±24,2 | 60,0±13,8 | 68,0±22,0 | 40-129 |
ALT: alanina aminotransferasa; APRI: índice ratio entre aspartato aminotransferasa y contaje de plaquetas; AST: aspartato aminotransferasa; FA: fosfatasa alcalina; GGT: gamma glutamil transferasa; NA: no aplicable; UDVP: usuarios de drogas por vía parenteral; y=media±desviación estándar.
En cuanto a la evolución clínica de los pacientes todos los RVPC alcanzaron una RVS y de los 6 NRVPC, 5 no consiguieron RVS mientras que un paciente obtuvo carga viral indetectable a los 6 meses postratamiento.
Expresión de genes estimulados por interferón (ISGs) antes del tratamientoSe determinó la expresión de ISGs basales en aquellos pacientes que consiguieron una RVS y en aquellos que no la consiguieron obteniendo menores niveles de la mayoría de ISGs basales en aquellos pacientes RVS. Los niveles de expresión obtenidos de ISGs antes del tratamiento expresados en escala logarítmica en base 2 fueron los siguientes (RVS vs NRVS): ISG15 (0,46 vs 0,60); IFI44L (0,82 vs 0,96); IFI27 (1,64 vs -0,14); MX1 (0,03 vs 1,34); IRF7 (0,17 vs 0,56); IFIT3 (0,08 vs 0,55); IFITM1 (-0,21 vs 0,042); OASL (-0,003 vs 0,54); IFI6 (0,49 vs 0,86); EIF2AK2 (0,56 vs 1,12); HERC5 (0,46 vs 0,98); APOBEC3A (0,49 vs 0,54); USP18 (0,36 vs 1,71).
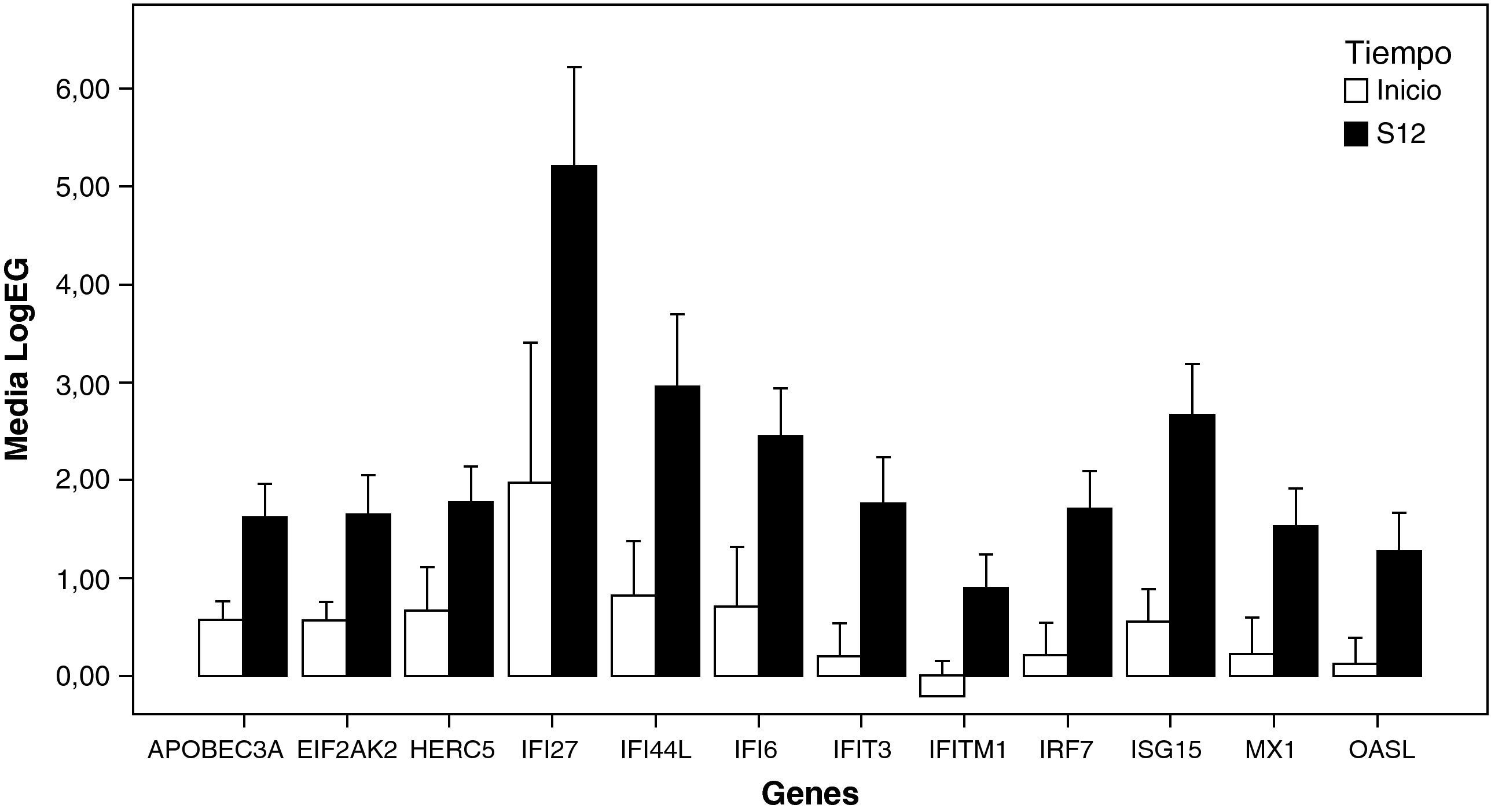
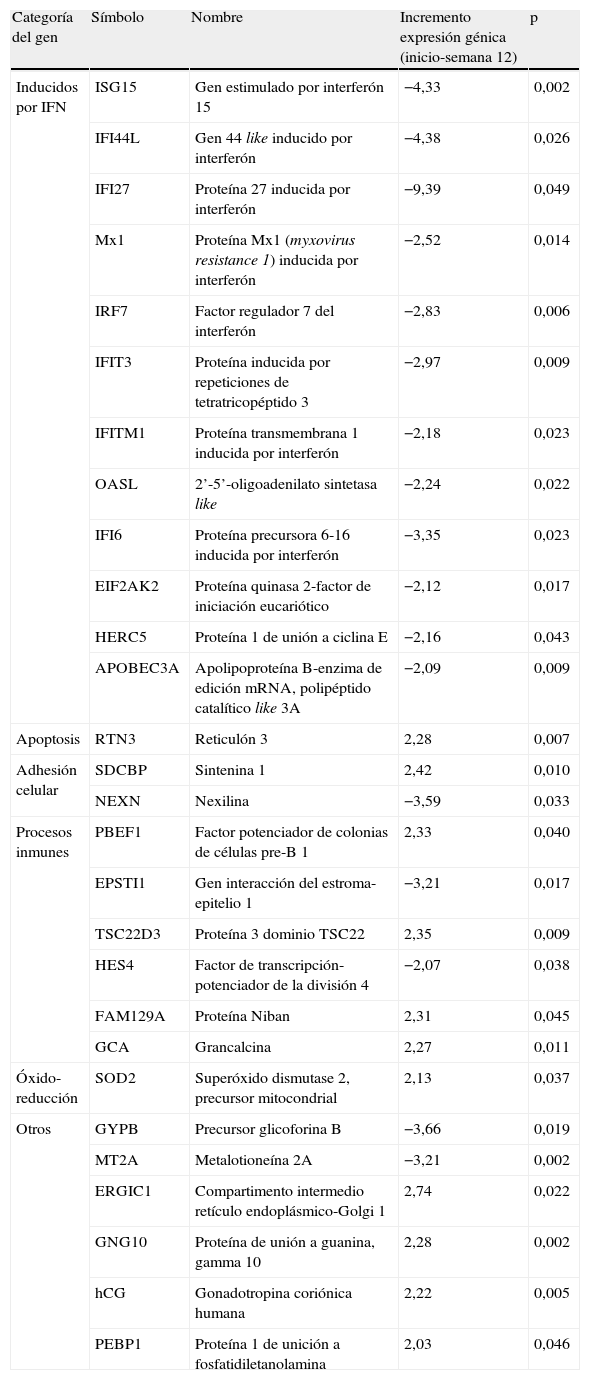
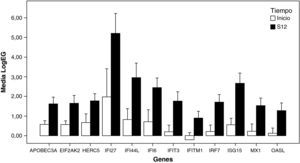
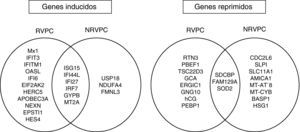
Efecto del tratamiento sobre los perfiles de expresión génica en RVPCEl análisis no supervisado de la expresión génica mediante clustering agrupó diferencialmente las muestras en función del tiempo (inicio y semana doce). El tratamiento con peginterferón alfa y ribavirina indujo un aumento de la expresión de un amplio número de genes que participan en la ruta del interferón como ISG15, IFI6, IFI44L, IFI27, MX1, OASL, IRF7, IFIT3, IFITM1, EIF2AK2, HERC5 y APOBEC3A en las primeras doce semanas de tratamiento (fig. 1). También se observó una sobreexpresión de otros genes involucrados en procesos inmunes (EPSTI1), en adhesión celular (NEXN), en la regulación de la transcripción (HES4) y en otras funciones (GYPB y MT2A) (tabla 2).
Representación de los niveles de expresión de genes inducidos por el interferón en RVPC: se seleccionaron aquellos genes con «veces de cambio inicio-semana 12 > 2» y p<0,05 (barras de error±error típico).
LogEG: logaritmo de la expresión de cada gen al inicio y a la semana 12, normalizada frente al control; S12: semana 12.
Por otra parte, los genes que presentaron mayor expresión génica antes del tratamiento que en la semana doce fueron genes involucrados en la proliferación celular (PBEF1), en la adhesión celular (SDCBP), en la apoptosis (RTN3), en otros procesos inmunes (TSC22D3, FAM129A, GCA) y en procesos de óxido-reducción (SOD2) entre otros (tabla 2).
Genes diferencialmente expresados en RVPC entre el inicio y la semana 12 de tratamiento
| Categoría del gen | Símbolo | Nombre | Incremento expresión génica (inicio-semana 12) | p |
| Inducidos por IFN | ISG15 | Gen estimulado por interferón 15 | −4,33 | 0,002 |
| IFI44L | Gen 44 like inducido por interferón | −4,38 | 0,026 | |
| IFI27 | Proteína 27 inducida por interferón | −9,39 | 0,049 | |
| Mx1 | Proteína Mx1 (myxovirus resistance 1) inducida por interferón | −2,52 | 0,014 | |
| IRF7 | Factor regulador 7 del interferón | −2,83 | 0,006 | |
| IFIT3 | Proteína inducida por repeticiones de tetratricopéptido 3 | −2,97 | 0,009 | |
| IFITM1 | Proteína transmembrana 1 inducida por interferón | −2,18 | 0,023 | |
| OASL | 2’-5’-oligoadenilato sintetasa like | −2,24 | 0,022 | |
| IFI6 | Proteína precursora 6-16 inducida por interferón | −3,35 | 0,023 | |
| EIF2AK2 | Proteína quinasa 2-factor de iniciación eucariótico | −2,12 | 0,017 | |
| HERC5 | Proteína 1 de unión a ciclina E | −2,16 | 0,043 | |
| APOBEC3A | Apolipoproteína B-enzima de edición mRNA, polipéptido catalítico like 3A | −2,09 | 0,009 | |
| Apoptosis | RTN3 | Reticulón 3 | 2,28 | 0,007 |
| Adhesión celular | SDCBP | Sintenina 1 | 2,42 | 0,010 |
| NEXN | Nexilina | −3,59 | 0,033 | |
| Procesos inmunes | PBEF1 | Factor potenciador de colonias de células pre-B 1 | 2,33 | 0,040 |
| EPSTI1 | Gen interacción del estroma-epitelio 1 | −3,21 | 0,017 | |
| TSC22D3 | Proteína 3 dominio TSC22 | 2,35 | 0,009 | |
| HES4 | Factor de transcripción-potenciador de la división 4 | −2,07 | 0,038 | |
| FAM129A | Proteína Niban | 2,31 | 0,045 | |
| GCA | Grancalcina | 2,27 | 0,011 | |
| Óxido-reducción | SOD2 | Superóxido dismutase 2, precursor mitocondrial | 2,13 | 0,037 |
| Otros | GYPB | Precursor glicoforina B | −3,66 | 0,019 |
| MT2A | Metalotioneína 2A | −3,21 | 0,002 | |
| ERGIC1 | Compartimento intermedio retículo endoplásmico-Golgi 1 | 2,74 | 0,022 | |
| GNG10 | Proteína de unión a guanina, gamma 10 | 2,28 | 0,002 | |
| hCG | Gonadotropina coriónica humana | 2,22 | 0,005 | |
| PEBP1 | Proteína 1 de unición a fosfatidiletanolamina | 2,03 | 0,046 |
Se seleccionaron aquellos genes con «veces de cambio pretratamiento-semana doce > 2», y p<0,05.
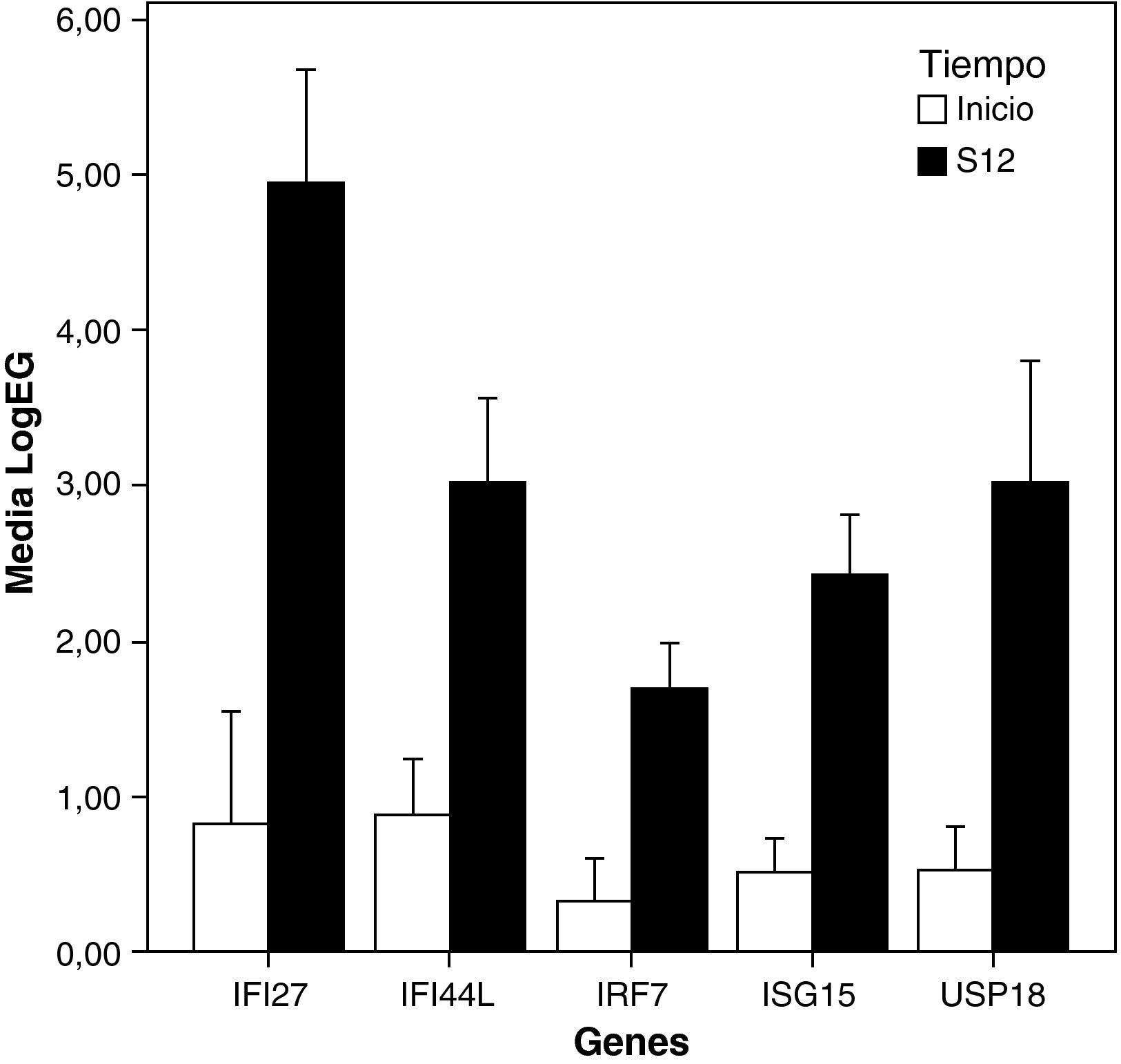
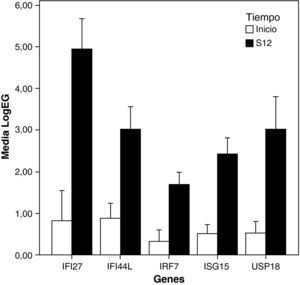
El estudio de los perfiles de expresión génica diferencial entre el inicio del tratamiento y la semana 12 mostró que el tratamiento indujo la sobreexpresión de un menor número de ISGs en pacientes NRVPC que en el caso de los pacientes RVPC (ISG15, IFI44L, IFI27, IRF7). Además, y a diferencia del caso de los RVPC, en los no respondedores se observó también un aumento de la expresión de un gen que presenta efecto inhibitorio en la ruta del interferón (USP18) (fig. 2 y fig. 3).
Representación de los niveles de expresión de genes inducidos por el interferón en NRVPC: se seleccionaron aquellos genes con «veces de cambio inicio-semana 12 > 2» y p<0.05 (barras de error ± error típico). LogEG: logaritmo de la expresión de cada gen al inicio y a la semana 12, normalizada frente al control. S12: Semana 12.
Por el contrario, el tratamiento indujo un descenso de la expresión de genes involucrados en adhesión celular (SDCBP), en el ciclo celular (CDC2L6), en procesos inmunes (FAM129A, SLPI, SLC11A1, AMICA1) y en procesos de óxido-reducción y defensa de estrés oxidativo (SOD2, MT-ATP8, MT-CYB) en las primeras doce semanas (tabla 3).
Genes diferencialmente expresados en NRVPC entre el inicio y la semana 12 de tratamiento
| Categoría del gen | Símbolo | Nombre | Incremento expresión génica (inicio-semana 12) | p |
| Inducidos por IFN | ISG15 | Gen estimulado por interferón 15 | −3,25 | 0,016 |
| IFI44L | Gen 44 like inducido por interferón | −4,35 | 0,047 | |
| IFI27 | Proteína 27 inducida por interferón | −28,65 | 0,000 | |
| IRF7 | Factor regulador 7 del interferón | −2,37 | 0,047 | |
| Inhibidores ruta IFN | USP18 | Peptidasa específica para ubiquitina 18 | −5,82 | 0,046 |
| Adhesión celular | SDCBP | Sintenina 1 | 3,01 | 0,029 |
| Ciclo celular | CDC2L6 | Proteína quinasa 6 del ciclo de división celular like 2 | 2,55 | 0,012 |
| Procesos inmunes | FAM129A | Proteína Niban | 2,55 | 0,012 |
| SLPI | Inhibidor de peptidasa de secreción leucocitaria | 2,13 | 0,004 | |
| SLC11A1 | Familia de transportadores de soluto 11, miembro 1 | 2,09 | 0,012 | |
| AMICA1 | Molécula de adhesión de interacción con CXADR antígeno 1 | 2,07 | 0,013 | |
| Óxido-reducción | SOD2 | Superóxido dismutasa 2, precursor mitocondrial | 2,54 | 0,026 |
| MT-ATP8 | ATP sintasa 8 codificada mitocondrialmente | 2,30 | 0,011 | |
| MT-CYB | Citocromo b codificado mitocondrialmente | 2,06 | 0,002 | |
| NDUFA4 | NADH deshidrogenasa (ubiquinona) 1 subcomplejo subunidad 4 | −2,95 | 0,029 | |
| Otros | BASP1 | Proteína soluble brain acid 1 | 2,04 | 0,012 |
| GYPB | Precursor glicoforina B | −2,95 | 0,008 | |
| MT2A | Metalotioneína 2A | −2,12 | 0,049 | |
| HBG1 | Hemoglobina gamma A | 5,21 | 0,040 | |
| FMNL3 | Formina-like 3 isoforma 2 | −2,19 | 0,027 |
Se seleccionaron aquellos genes con «veces de cambio pretratamiento-semana doce > 2», y p<0,05.
Se comparó la expresión génica en las primeras doce semanas de tratamiento distribuyendo a los pacientes en función de si alcanzaron RVS o no y se obtuvieron valores de incremento/disminución de la expresión génica con una tendencia similar a los obtenidos en las comparaciones anteriores (RVPC y NRVPC, tablas 2 y 3).
En el grupo de pacientes que consiguieron una RVS, el incremento/disminución de la expresión expresado en veces de cambio para los genes que resultaron significativos en la comparación anterior fueron los siguientes: ISG15: -4,79; IFI44L: -4,79; IFI27: -3,26; Mx1: -2,83, IRF7: -2,89; IFIT3: -3,10; IFITM1: -2,15; OASL: -2,38; IFI6: -4,01; EIF2AK2: -2,16; HERC5: -2,58; APOBEC3A: -2,26; RTN3: 2,16; SDCBP: 2,32; NEXN: -4,10; PBEF1: 2,42; EPSTI1: -3,53; TSC22D3: 2,33; HES4: -2,08; FAM129A: 2,43; GCA: 2,35; SOD2: 2,16; GYPB: -3,71; MT2A: -3,29; ERGIC1: 2,74; GNG10: 2,18; hCG: 2,01; PEBP1: 1,7.
En el grupo de pacientes que no consiguieron una RVS, las veces de cambio de la expresión génica en las primeras doce semanas de tratamiento (inicio-semana 12) fueron: ISG15: -2,68; IFI44L: -3,90; IFI27: -23,20; IRF7: -2,22; USP18: -6,65; SDCBP: 3,36; CDC2L6: 2,91; FAM129A: 2,42; SLPI: 2,00; SLC11A1: 2,35; AMICA1: 2,43; SOD2: 2,33; MT-ATP8: 2,32; MT-CYB: 1,94; NDUFA4: -3,18; BASP1: 1,94; GYPB: -2,72; MT2A: 1,87; HBG1: -4,36; FMNL3: -2,59.
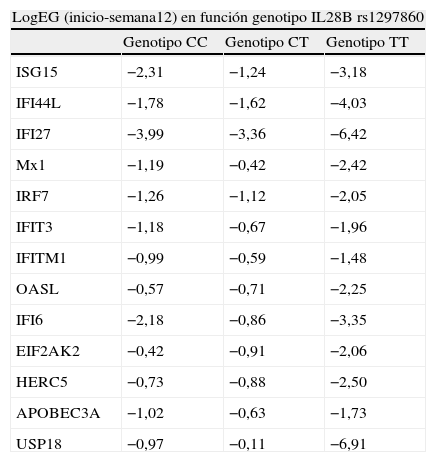
Expresión génica en las primeras doce semanas de tratamiento en función del genotipo de la IL28B rs1297860Los dos pacientes que presentaron genotipo CC consiguieron una RVS. En cuanto al resto de pacientes, de aquellos que presentaron el genotipo CT (n=7), dos consiguieron la RVS mientras que 5 no. Por otra parte, los tres pacientes con genotipo TT alcanzaron la RVS.
En la tabla 4 se representan las variaciones de la expresión de ISGs en las primeras doce semanas de tratamiento en función del genotipo de la IL28B rs 1297860 (log expresión inicio–log expresión semana 12). Se observa un mayor incremento de la expresión de ISGs en los pacientes que presentaron genotipo TT que en aquellos que presentaron genotipo CT o CC.
Expresión diferencial de ISGs en las primeras doce semanas de tratamiento en función del genotipo de la IL28B rs1297860
| LogEG (inicio-semana12) en función genotipo IL28B rs1297860 | |||
| Genotipo CC | Genotipo CT | Genotipo TT | |
| ISG15 | −2,31 | −1,24 | −3,18 |
| IFI44L | −1,78 | −1,62 | −4,03 |
| IFI27 | −3,99 | −3,36 | −6,42 |
| Mx1 | −1,19 | −0,42 | −2,42 |
| IRF7 | −1,26 | −1,12 | −2,05 |
| IFIT3 | −1,18 | −0,67 | −1,96 |
| IFITM1 | −0,99 | −0,59 | −1,48 |
| OASL | −0,57 | −0,71 | −2,25 |
| IFI6 | −2,18 | −0,86 | −3,35 |
| EIF2AK2 | −0,42 | −0,91 | −2,06 |
| HERC5 | −0,73 | −0,88 | −2,50 |
| APOBEC3A | −1,02 | −0,63 | −1,73 |
| USP18 | −0,97 | −0,11 | −6,91 |
Los datos son expresados en valores logarítmicos en base 2. Log EG: logaritmo de la expresión génica.
Teniendo en cuenta los efectos secundarios y el coste del tratamiento, la predicción de la respuesta al tratamiento es importante. Los perfiles de expresión génica de pacientes con hepatitis C crónica pueden ser biomarcadores útiles para la terapia basada en interferón11. El estudio de la expresión génica en las primeras semanas de tratamiento puede dilucidar rutas moleculares claves que influyan en la probabilidad de lograr una respuesta virológica12.
El tratamiento con peginterferón alfa y ribavirina en RVPC indujo un aumento de la expresión de un gran número de genes involucrados en la respuesta a virus, en la respuesta inmune y de defensa. La mayoría de los genes sobreexpresados en las primeras doce semanas fueron ISGs implicados en la respuesta a virus (ISG15, Mx1, EIF2AK2, APOBEC3A), en la respuesta inmune (IFI27, Mx1, OASL, IFIT3, IFITM1, EIF2AK2, ISG15, IFI6), en la regulación negativa de la proliferación celular (IFITM1, EIF2AK2), en el ciclo celular (IFITM1, EIF2AK2, HERC5) y en la señalización celular (ISG15). La activación de esta amplia cantidad de ISGs con el tratamiento se ha descrito con anterioridad en otros trabajos13,14 incluso en las primeras cuatro semanas de tratamiento en respondedores rápidos15. El aumento de la expresión de estos genes podría ser clave en el aclaramiento del virus producido en respondedores.
En nuestro conocimiento, esta es la primera ocasión en la que se describe una inducción de la sobreexpresión de genes como EPTSTI1, NEXN, HES4 y GYPB por el tratamiento utilizado en la hepatitis C crónica. La expresión del gen EPSTI1ha mostrado una fuerte correlación positiva con la expresión de ISG20 en otros trabajos16 sugiriendo que EPSTI1 podría tener un papel clave en eventos relacionados con el interferón. Otro de los genes sobreexpresados con la terapia basada en interferón fue HES4. En un trabajo reciente realizado in vitro, se describió un aumento de la expresión de HES4, MT2A y OASL durante la maduración de células dendríticas estimuladas con IFN gamma lo cual indicaría, según nuestros resultados, que la activación de estos genes también es inducida por interferón alfa. La expresión de MT2A aumentó con el tratamiento en ambos grupos (RVPC y NRVPC), aunque lo hizo en mayor grado en RVPC. Devitt et al observaron mayor sobreexpresión del gen MT2A en respondedores que en no respondedores incluso en las primeras 24h de tratamiento17.
Con peginterferón alfa y ribavirina se observó una represión de genes, implicados en procesos inmunes, que habían sido activados antes del tratamiento por el virus. Entre estos genes se encontraron ERGIC1 que podría estar implicado en el transporte entre el retículo endoplásmico y el aparato de Golgi siendo importante en la formación de membranas del virus18, SDCBP implicado en adhesión celular localizado en membrana así como en el retículo endoplásmico, RTN3 principalmente localizado en retículo endoplásmico e involucrado en inducción de apoptosis y FAM129A implicado en la respuesta al estrés del retículo endoplásmico19 y en la regulación de la traducción de ciertas proteínas. Por consiguiente, la activación de la expresión de estos genes antes del tratamiento podría deberse a una replicación activa del virus y su posterior represión en las primeras doce semanas de tratamiento deberse a un descenso en los niveles de virus circulantes por el tratamiento. Con el tratamiento también se observó la represión de otros genes como PBEF1 con efecto pro-inflamatorio y cuya concentración se encuentra incrementada significantemente en pacientes infectados por el VHC20, GCA involucrada en la adhesión de neutrófilos21 y PEBP1 que se ha descrito recientemente como un posible biomarcador de daño hepático22. La represión de estos genes podría ser importante en la prevención del daño hepático, la inflamación hepática y fibrosis causada por la respuesta inmune a la infección por el VHC.
En pacientes NRVPC, el tratamiento con peginterferón alfa y ribavirina indujo un incremento de la expresión de un menor número de ISGs comparado con el grupo RVPC. El estudio de los niveles de expresión en estos pacientes revela que el tratamiento no induce amplios cambios en la expresión de ISGs sobre los niveles de pretratamiento. Según se ha descrito previamente en otros trabajos, los pacientes que alcanzan una RVS presentan menores niveles de expresión de ISGs antes del tratamiento que aquellos que no responden, seguido de un incremento de la expresión sobre todo en los RVS23. Nuestro trabajo apoya estos hallazgos previos ya que cuando distribuimos a los pacientes de acuerdo a la RVS, observamos menores niveles de ISGs basales en los pacientes que responden y posteriormente, con el tratamiento, una amplia activación de ISGs en las primeras doce semanas frente a un escaso número de ISGs que son activados en el grupo de no respondedores.
Además, en el grupo NRVPC se observó la sobreexpresión de genes con actividad inhibitoria sobre la ruta del interferón como USP18. Chen et al describieron un aumento de la expresión de USP18 en no respondedores antes del tratamiento en biopsias de hígado13. Según nuestros resultados, la expresión de este gen casi triplica su nivel en las primeras doce semanas de tratamiento en sangre periférica. La inhibición de la actividad del interferón por este gen podría ser crítica para eludir los efectos tanto del interferón endógeno como terapéutico en pacientes no respondedores. Hasta ahora, hemos descrito el aumento en la expresión de genes relacionados con la ruta del interferón cuya implicación en la infección por el VHC ha sido ampliamente descrita en la literatura. Nosotros hemos observado en NRVPC un aumento de la expresión de otros genes como son NDUFA4, GYPB y FMNL3 cuyo posible papel en la infección y tratamiento de la hepatitis C es aún desconocido.
El estrés oxidativo es un mecanismo clave en la patogénesis de la infección por el VHC24,25. Algunos estudios han sugerido que el VHC induce la generación de especies reactivas de oxígeno causando daños en el DNA. En nuestro trabajo, observamos que el tratamiento indujo un descenso de la expresión de genes implicados en procesos de oxido-reducción y defensa al estrés oxidativo (SOD2, MT-ATP8, MT-CYB) que probablemente habían sido activados antes del tratamiento. El descenso de estos genes antioxidantes podría ser debido a una menor presión ejercida por especies reactivas de oxígeno como consecuencia del tratamiento.
Debido a que distribuyendo a los pacientes estudiados en función de su evolución final (RVS y NRVS) se obtuvieron valores de sobreexpresión y represión de la expresión génica con una tendencia similar a los obtenidos en los grupos RVPC y NRVPC, los comentarios realizados hasta el momento en RVPC y NRVPC podrían ser trasladables a los grupos RVS y NRVS en las primeras doce semanas de tratamiento.
Por otra parte, la interpretación de los resultados de expresión génica en función del genotipo del polimorfismo rs1297860 de la IL28B es difícil, debido al limitado número de pacientes de nuestra cohorte. Son necesarios estudios con una «n» mayor para aclarar el papel de los cambios en el gen del interferón-λ (IL-28) en los niveles de expresión de ISGs y en la respuesta al tratamiento.
Otra limitación del estudio es que la expresión génica se ha determinado en sangre periférica y no directamente en tejido hepático, y podría quedar la incógnita de si los resultados serían trasladables a los hepatocitos infectados por el VHC. Aunque los dos aspectos mencionados son ciertamente limitantes, el hecho de que algunos de los resultados obtenidos en este trabajo hayan sido previamente corroborados por otros trabajos previos garantiza la fiabilidad global de nuestro estudio.
En conclusión, nuestros resultados muestran que el tratamiento combinado con interferón pegilado+ribavirina en pacientes con infección crónica por el VHC induce un aumento coordinado de genes de respuesta al interferón a las doce semanas de tratamiento, siendo mayor el número de genes activados en el grupo de pacientes respondedores. Esta mayor activación de genes de respuesta al interferón podría por tanto explicar el mejor control de la carga viral por parte de estos pacientes. Por otra parte, este trabajo ha mostrado por primera vez la participación en la respuesta al tratamiento de genes cuya implicación en la infección VHC no había sido descrita hasta el momento presente. Estos hallazgos podrían abrir nuevas vías de estudio para una mayor compresión de la patogénesis de la enfermedad y el efecto del tratamiento.
FinanciaciónEste trabajo fue posible gracias a la ayuda obtenida de «Fondo de Investigaciones Sanitarias» (FIS), Ministerio de Ciencia e Innovación, España y Consejería de Sanidad Junta de Castilla y León («Programa para favorecer la incorporación de grupos de investigación en las Instituciones del Sistema Nacional de Salud, EMER07/050» y «Proyectos de investigación en salud» PI081236).
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Los autores quieren agradecer el apoyo técnico ofrecido por Lucía Rico y Verónica Iglesias. También agradecen la colaboración ofrecida por el equipo de enfermeras del Servicio de Medicina Digestiva de nuestro hospital por la recogida de las muestras, y a Antonio Orduña y José de la Higuera, de la sección de Inmunología del Servicio de Microbiología por realizar el diagnóstico viral.