El cáncer de próstata (CaP) muestra una agresividad variada, lo que complica las decisiones de tratamiento personalizadas. Los sistemas tradicionales de estratificación del riesgo se basan en parámetros clínicos, pero pueden pasar por alto aspectos genéticos cruciales. El P-score de Prostatype® integra la expresión genética con los datos clínicos para optimizar la precisión de la evaluación del riesgo del CaP.
ObjetivosValidar el rendimiento predictivo del P-score para la mortalidad específica por cáncer de próstata (MECP) y la metástasis en una cohorte española, y compararlo con los sistemas de NCCN, D’Amico y EAU.
Materiales y métodosSe realizó un estudio multicéntrico y retrospectivo que incluyó 7 hospitales españoles. De un total de 154 biopsias con aguja gruesa, 93 cumplieron los criterios de calidad del ARN. En estas muestras se calculó el P-score en función de la expresión génica de IGFBP3, VGLL3 y F3, junto con los datos clínicos correspondientes. El objetivo principal fue la MECP, mientras que los objetivos secundarios incluyeron el desarrollo de metástasis, la presencia de enfermedad adversa (PA) y el grado según la clasificación de la International Society of Urological Pathology (ISUP).
ResultadosEl P-score demostró una mayor precisión en la predicción de la MECP a 10 años, con un AUC de 0,81 y un índice C de 0,75, superando a los sistemas NCCN (AUC 0,77; índice C 0,69) y D’Amico/EAU (AUC 0,70; índice C 0,62). Para la predicción de metástasis, el P-score alcanzó un índice C de 0,77, significativamente superior a NCCN, D’Amico y EAU (0,58). El análisis de Kaplan-Meier destacó la capacidad mayor del P-score para estratificar el riesgo, especialmente en los grupos de alto riesgo. Además, el P-score se correlacionó con la carga tumoral, con asociaciones significativas con el número de cilindros positivos en la biopsia (p=0,017) y el grado ISUP en la prostatectomía radical (p=0,0028).
ConclusionesEn esta cohorte española, el P-score superó a los sistemas clinicopatológicos tradicionales en la predicción de la MECP, el desarrollo de metástasis y los marcadores patológicos, lo que respalda su utilidad clínica para un manejo más personalizado del CaP.
Prostate cancer (PCa) shows varied aggressiveness, complicating personalised treatment decisions. Traditional risk stratification systems rely on clinical parameters but may miss crucial genetic insights. The Prostatype® score (P-score) integrates gene expression with clinical data to improve PCa risk assessment precision.
ObjectivesTo validate the P-score's predictive performance for prostate cancer-specific mortality (PCSM) and metastasis in a Spanish cohort, comparing it with NCCN, D’Amico, and EAU systems.
Materials and MethodsThis study was multicentre, retrospective and included seven Spanish hospitals. Of 154 core needle biopsies, 93 met RNA criteria, and for those, P-score was calculated based on IGFBP3, VGLL3, and F3 genes expression and clinical data.
The primary endpoint was PCa-specific mortality (PCSM), with secondary endpoints being development of metastasis, adverse pathology (AP), and International Society of Urological Pathology (ISUP) grading.
ResultsThe P-score demonstrated superior accuracy in predicting 10-year PCSM, with an AUC of 0.81 and a C-index of 0.75, outperforming NCCN (AUC 0.77, C-index 0.69) and D’Amico/EAU (AUC 0.70, C-index 0.62). For metastasis prediction, the P-score achieved a C-index of 0.77, significantly higher than NCCN, D’Amico, and EAU (0.58). Kaplan-Meier analysis underscored the P-score's ability to better stratify patients by risk, especially high-risk groups. Additionally, the P-score correlated with tumour burden, showing significant associations with positive biopsy cores (p=0.017) and ISUP grade at radical prostatectomy (p=0.0028).
ConclusionsIn this Spanish cohort, the P-score outperformed traditional clinicopathological systems in predicting PCSM, development of metastasis, and pathological markers, supporting its clinical utility for more personalised PCa management.
El cáncer de próstata (CaP) es el segundo cáncer más frecuente en el varón a nivel mundial y presenta un amplio espectro de comportamientos clínicos, desde casos indolentes que pueden requerir solo seguimiento hasta formas muy agresivas que exigen una intervención inmediata. España se ha convertido en uno de los países europeos con las tasas de mortalidad por CaP más bajas (13,2 muertes por 100.000 personas/año)1, con la mayoría (89,8% en 20102) de los diagnósticos iniciales de CaP en España correspondiendo a enfermedad localizada no metastásica. Las opciones de tratamiento habitual para el CaP incluyen la prostatectomía radical (PR), la radioterapia (RT) y la terapia hormonal, así como la vigilancia activa (VA). Cada método, aparte de la VA, se asocia a efectos adversos potenciales como la disfunción eréctil y la incontinencia urinaria, entre otros, mientras que la VA está relacionada con la incertidumbre sobre la enfermedad3. Por lo tanto, una estratificación precisa del riesgo en el momento del diagnóstico es crucial para orientar las decisiones terapéuticas individualizadas4,5. Existen varios sistemas de estratificación del riesgo. El sistema D’Amico utiliza nomogramas que combinan el antígeno prostático específico (PSA), el estadio clínico del tumor (estadio cT) y el Gleason Score (GS), y estratifica a los pacientes en grupos de riesgo bajo, intermedio y alto6. El sistema de la Asociación Europea de Urología (EAU) incorpora el porcentaje de infiltración tumoral en las muestras de biopsia7. El sistema de la National Comprehensive Cancer Network (NCCN) añade al PSA el estadio cT y el GS, el volumen tumoral y la extensión de la enfermedad. Además, divide el grupo intermedio en favorable y desfavorable, y añade los grupos de riesgo muy bajo y muy alto, lo que lo convierte en un sistema más granular8. Actualmente, un porcentaje considerable de pacientes con CaP reciben sobretratamientos, lo que conduce a un deterioro de la calidad de vida y a una carga económica para el sistema de salud9.
El test genético P-score de Prostatype® es un sistema de estratificación del riesgo que integra una firma génica de 3 genes (IGFBP3, que codifica para la proteína 3 de unión al factor de crecimiento similar a la insulina, VGLL3, que codifica el miembro 3 de la familia vestigial-like, y F3, que codifica el factor III de coagulación) junto con parámetros clinicopatológicos (GS, PSA, estadio cT) para una evaluación precisa del riesgo de mortalidad específica del CaP (MECP). Al incorporar biomarcadores genéticos, el P-score demostró una mejor capacidad de estratificación en los pacientes con CaP y, lo que podría facilitar una toma de decisiones terapéuticas más personalizada10–12.
Este estudio tuvo como objetivo validar la utilidad clínica del P-score en la población española, comparando su rendimiento en la predicción de la mortalidad específica por cáncer de próstata (MECP) y el desarrollo de metástasis con los sistemas de clasificación de riesgo establecidos (NCCN, D’Amico y EAU). Al evaluar si el P-score proporciona una estratificación del riesgo y un poder pronóstico superiores, el estudio buscó generar evidencia que respalde la incorporación de marcadores génicos en la práctica clínica habitual, con el fin de favorecer un manejo más personalizado del CaP.
Material y métodosCohorte del estudio y recogida de muestrasEste estudio retrospectivo multicéntrico incluyó a los pacientes con CaP procedentes de siete hospitales españoles. Los pacientes elegibles (50-100 años) presentaban un CaP localizado (grupo de riesgo de la EAU) diagnosticado entre 2000-2017, sin metástasis en el momento del diagnóstico, sometidos a tratamientos curativos (PR, RT±TPA) y disponían de biopsias con aguja gruesa (BAG) e historias clínicas con datos clinicopatológicos relevantes. Criterios de exclusión: historias clínicas o material de BAG no disponibles, tejido insuficiente (<2mm), calidad inadecuada del ARN, metástasis en el momento del diagnóstico o muertes no relacionadas con el cáncer. Se examinó a un total de 154 pacientes y 93 fueron elegibles para el análisis final. Se obtuvieron BAG de tejido fijado en formol e incluido en parafina (FFIP) de cada hospital y se enviaron al Hospital Universitario La Paz para su análisis. La selección de la cohorte y la manipulación de las muestras se describen detalladamente en el material suplementario.
Análisis de la expresión génica y cálculo del P-scoreLos niveles de expresión de los genes IGFBP3, F3 y VGLL3 se evaluaron con el kit Prostatype® RT-qPCR, y el P-score se calculó como se describe en el material suplementario y en Söderdahl et al.12. Los pacientes se clasificaron en 3 grupos de riesgo P-score (riesgo bajo, intermedio y alto) en función de la puntuación calculada. Para el análisis comparativo, los pacientes también se clasificaron mediante los sistemas de estratificación del riesgo de la NCCN, D’Amico y EAU.
Criterios de valoraciónEl criterio de valoración principal fue la MECP. Los criterios de valoración secundarios fueron el desarrollo de metástasis, el porcentaje de muestras de biopsia positivas (PPB), la presencia de enfermedad adversa (PA) y el grado según la International Society of Urological Pathology (ISUP).
Análisis estadísticoLos análisis estadísticos se detallan en el material suplementario. Las características basales se compararon utilizando la prueba de Chi-cuadrado (para variables categóricas) o la prueba de Kruskal-Wallis (para variables continuas).
Las curvas de supervivencia de Kaplan-Meier y las pruebas de rangos logarítmicos evaluaron la MECP en los grupos de P-score, NCCN, D’Amico y EAU.
La exactitud predictiva para MECP y PA se evaluó mediante curvas ROC (AUC con IC 95%).
La capacidad de predicción de cada sistema de estratificación tanto para la MECP como para el desarrollo de metástasis a lo largo del tiempo se evaluó mediante el índice de concordancia (índice C); un valor de 0,5 corresponde a «no mejor que el azar», un valor de 1 corresponde a «predicción perfecta».
El impacto del sistema de estratificación en la predicción del tratamiento se evaluó mediante puntuaciones de intensidad del tratamiento. Para la reclasificación, se analizaron las asignaciones de riesgo iniciales en los sistemas NCCN, D’Amico y EAU con la clasificación basada en el P-score y los cambios en las decisiones de tratamiento de los pacientes que habrían sido reclasificados. La probabilidad de cambios en el tratamiento se estimó mediante un modelo de simulación MonteCarlo (10.000 iteraciones).
Los diagramas de Sankey se generaron utilizando el paquete JavaScript® D3.
Todos los análisis se realizaron con la versión 2024-04-2+764 de RStudio®. Un valor p<0,05 se consideró estadísticamente significativo.
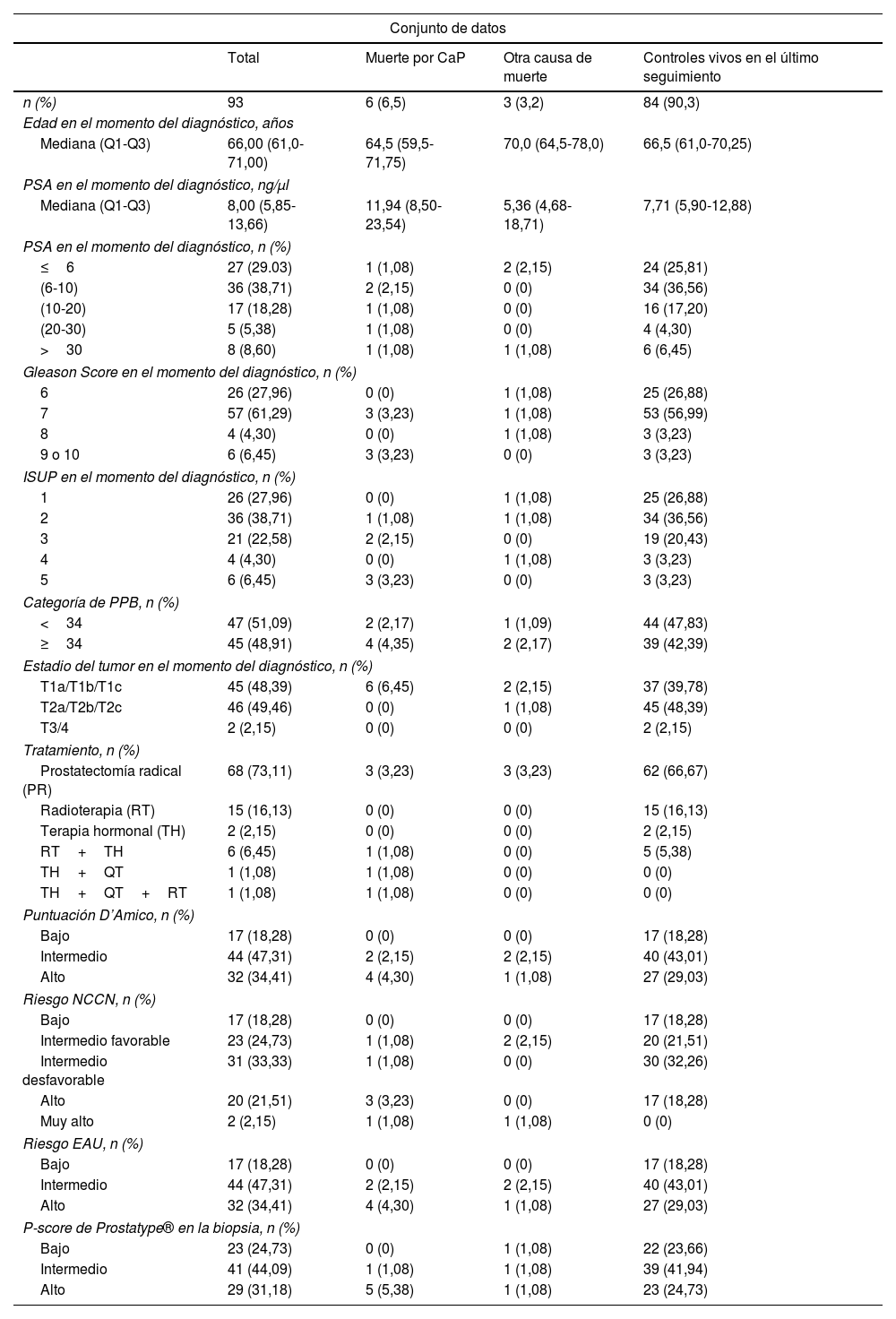
ResultadosCaracterísticas de los pacientesDe 154 casos de CaP identificados inicialmente y elegibles para su inclusión en el estudio, 14 casos no tenían células tumorales en la biopsia, un paciente tenía metástasis en el momento del diagnóstico y 46 casos tenían ARN de mala calidad y no pudieron analizarse. Finalmente, 93 casos tuvieron resultados válidos (tabla 1). La mediana de edad en el momento del diagnóstico de los pacientes incluidos fue de 66 años (Q1-Q3: 61,0-71,0) y la mediana del tiempo de seguimiento fue de 8,51 años. Del total, 23 casos (24,73%) tenían un P-score de riesgo bajo, 41 (44,09%) un P-score de riesgo intermedio y 29 (31,18%) un P-score de riesgo alto. El P-score osciló entre 0 y 10, con una mediana de 4,0 (Q1-Q3: 3,0-6,0) (fig. S1 del material suplementario). Los datos clinicopatológicos y la asignación a los sistemas de puntuación de riesgo D’Amico, NCCN y EAU figuran en la tabla 1. De los 93 casos incluidos en el análisis final, 6 murieron por CaP.
Características clínicas de los pacientes incluidos en el conjunto de datos, n=93
| Conjunto de datos | ||||
|---|---|---|---|---|
| Total | Muerte por CaP | Otra causa de muerte | Controles vivos en el último seguimiento | |
| n (%) | 93 | 6 (6,5) | 3 (3,2) | 84 (90,3) |
| Edad en el momento del diagnóstico, años | ||||
| Mediana (Q1-Q3) | 66,00 (61,0-71,00) | 64,5 (59,5-71,75) | 70,0 (64,5-78,0) | 66,5 (61,0-70,25) |
| PSA en el momento del diagnóstico, ng/μl | ||||
| Mediana (Q1-Q3) | 8,00 (5,85-13,66) | 11,94 (8,50-23,54) | 5,36 (4,68-18,71) | 7,71 (5,90-12,88) |
| PSA en el momento del diagnóstico, n (%) | ||||
| ≤6 | 27 (29.03) | 1 (1,08) | 2 (2,15) | 24 (25,81) |
| (6-10) | 36 (38,71) | 2 (2,15) | 0 (0) | 34 (36,56) |
| (10-20) | 17 (18,28) | 1 (1,08) | 0 (0) | 16 (17,20) |
| (20-30) | 5 (5,38) | 1 (1,08) | 0 (0) | 4 (4,30) |
| >30 | 8 (8,60) | 1 (1,08) | 1 (1,08) | 6 (6,45) |
| Gleason Score en el momento del diagnóstico, n (%) | ||||
| 6 | 26 (27,96) | 0 (0) | 1 (1,08) | 25 (26,88) |
| 7 | 57 (61,29) | 3 (3,23) | 1 (1,08) | 53 (56,99) |
| 8 | 4 (4,30) | 0 (0) | 1 (1,08) | 3 (3,23) |
| 9 o 10 | 6 (6,45) | 3 (3,23) | 0 (0) | 3 (3,23) |
| ISUP en el momento del diagnóstico, n (%) | ||||
| 1 | 26 (27,96) | 0 (0) | 1 (1,08) | 25 (26,88) |
| 2 | 36 (38,71) | 1 (1,08) | 1 (1,08) | 34 (36,56) |
| 3 | 21 (22,58) | 2 (2,15) | 0 (0) | 19 (20,43) |
| 4 | 4 (4,30) | 0 (0) | 1 (1,08) | 3 (3,23) |
| 5 | 6 (6,45) | 3 (3,23) | 0 (0) | 3 (3,23) |
| Categoría de PPB, n (%) | ||||
| <34 | 47 (51,09) | 2 (2,17) | 1 (1,09) | 44 (47,83) |
| ≥34 | 45 (48,91) | 4 (4,35) | 2 (2,17) | 39 (42,39) |
| Estadio del tumor en el momento del diagnóstico, n (%) | ||||
| T1a/T1b/T1c | 45 (48,39) | 6 (6,45) | 2 (2,15) | 37 (39,78) |
| T2a/T2b/T2c | 46 (49,46) | 0 (0) | 1 (1,08) | 45 (48,39) |
| T3/4 | 2 (2,15) | 0 (0) | 0 (0) | 2 (2,15) |
| Tratamiento, n (%) | ||||
| Prostatectomía radical (PR) | 68 (73,11) | 3 (3,23) | 3 (3,23) | 62 (66,67) |
| Radioterapia (RT) | 15 (16,13) | 0 (0) | 0 (0) | 15 (16,13) |
| Terapia hormonal (TH) | 2 (2,15) | 0 (0) | 0 (0) | 2 (2,15) |
| RT+TH | 6 (6,45) | 1 (1,08) | 0 (0) | 5 (5,38) |
| TH+QT | 1 (1,08) | 1 (1,08) | 0 (0) | 0 (0) |
| TH+QT+RT | 1 (1,08) | 1 (1,08) | 0 (0) | 0 (0) |
| Puntuación D’Amico, n (%) | ||||
| Bajo | 17 (18,28) | 0 (0) | 0 (0) | 17 (18,28) |
| Intermedio | 44 (47,31) | 2 (2,15) | 2 (2,15) | 40 (43,01) |
| Alto | 32 (34,41) | 4 (4,30) | 1 (1,08) | 27 (29,03) |
| Riesgo NCCN, n (%) | ||||
| Bajo | 17 (18,28) | 0 (0) | 0 (0) | 17 (18,28) |
| Intermedio favorable | 23 (24,73) | 1 (1,08) | 2 (2,15) | 20 (21,51) |
| Intermedio desfavorable | 31 (33,33) | 1 (1,08) | 0 (0) | 30 (32,26) |
| Alto | 20 (21,51) | 3 (3,23) | 0 (0) | 17 (18,28) |
| Muy alto | 2 (2,15) | 1 (1,08) | 1 (1,08) | 0 (0) |
| Riesgo EAU, n (%) | ||||
| Bajo | 17 (18,28) | 0 (0) | 0 (0) | 17 (18,28) |
| Intermedio | 44 (47,31) | 2 (2,15) | 2 (2,15) | 40 (43,01) |
| Alto | 32 (34,41) | 4 (4,30) | 1 (1,08) | 27 (29,03) |
| P-score de Prostatype® en la biopsia, n (%) | ||||
| Bajo | 23 (24,73) | 0 (0) | 1 (1,08) | 22 (23,66) |
| Intermedio | 41 (44,09) | 1 (1,08) | 1 (1,08) | 39 (41,94) |
| Alto | 29 (31,18) | 5 (5,38) | 1 (1,08) | 23 (24,73) |
CaP: carcinoma de próstata; EAU: European Association of Urology; ISUP: International Society of Uropathology; NCCN: National Comprehensive Cancer Network; PPB: porcentaje de cilindros positivos en la biopsia; PSA: antígeno específico prostático.
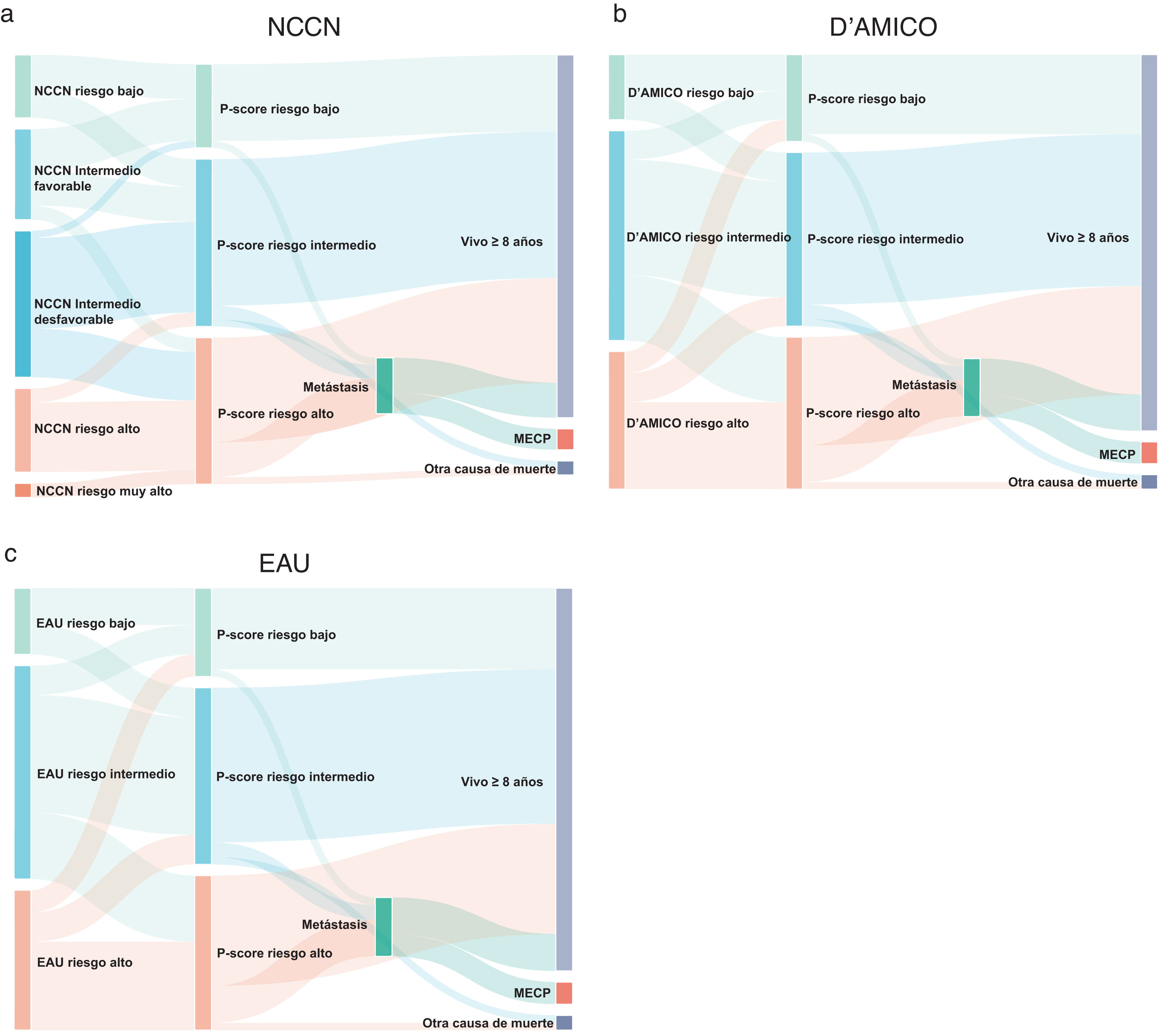
La reclasificación se basó en 57 pacientes con ≥8 años de seguimiento y sin metástasis en el momento del diagnóstico. La NCCN clasificó a los pacientes en 5 grupos (fig. 1a). De 9 pacientes de bajo riesgo, 5 (56%) siguieron teniendo un P-score de bajo riesgo (uno desarrolló metástasis), mientras que 4 (44%) fueron reclasificados como de riesgo intermedio (2 desarrollaron metástasis). Todos sobrevivieron más de 8 años.
Entre los 13 pacientes con riesgo intermedio favorable según la NCCN, 6 (46%) fueron reclasificados a riesgo bajo según el P-score, 5 (38%) a riesgo intermedio y 2 (16%) a riesgo alto. Un paciente de bajo riesgo y otro de alto riesgo murieron por causas ajenas a al CaP, mientras que un paciente de alto riesgo murió de CaP. De los 21 pacientes de riesgo intermedio desfavorable según la NCCN, uno (5%) fue reclasificado como de bajo riesgo, 13 (62%) como de riesgo intermedio y 7 (33%) como de alto riesgo. Un paciente de riesgo intermedio desarrolló metástasis y murió de CaP. De los 12 pacientes de alto riesgo según la NCCN, 2 (17%) fueron reclasificados como de riesgo intermedio, mientras que 10 (83%) siguieron siendo de alto riesgo. Cuatro desarrollaron metástasis y 3 murieron de CaP. Ningún paciente fue reclasificado como de muy alto riesgo según la NCCN.
Los sistemas de clasificación de D’Amico y de la EAU estratificaron a los pacientes en 3 grupos (riesgo bajo, riesgo intermedio, riesgo alto) (figs. 1b y c). En los grupos de bajo riesgo, los 9 pacientes sobrevivieron al menos 8 años. De estos 9 pacientes, 4 (44%) fueron reclasificados a riesgo intermedio según el P-score, y 2 de ellos desarrollaron metástasis.
Entre los 29 pacientes de riesgo intermedio D’Amico/EAU, 4 (14%) pasaron a riesgo bajo (una muerte no relacionada con el CaP) y 9 (31%) pasaron a riesgo alto (2 desarrollaron metástasis).
De los 19 pacientes de alto riesgo D’Amico/EAU, 3 (16%) fueron reclasificados a riesgo bajo según el P-score y 4 (21%) a riesgo intermedio. Todos sobrevivieron más de 8 años. De los 12 pacientes de (63%) asignados al grupo de alto riesgo según P-score, 4 desarrollaron metástasis (3 de los cuales murieron de CaP).
Impacto en la decisión de tratamientoAnalizamos la puntuación referida a la intensidad del tratamiento para evaluar el impacto de la clasificación de riesgo en las decisiones de tratamiento. La PR y la RT fueron los tratamientos principales en todas las clasificaciones, con intensidades de tratamiento variadas (tabla S1 del material suplementario). Los pacientes de riesgo bajo (puntuación=2,00) recibieron principalmente PR o RT. Los pacientes de riesgo intermedio (puntuación=2,02-2,03) se sometieron a intervenciones ligeramente más agresivas (por ejemplo, RT+TPA). Los pacientes de alto riesgo (puntuación=2,38-2,41) tenían más probabilidades de recibir terapia multimodal. Para evaluar el impacto de la reclasificación de los sistemas NCCN, D’Amico y de la EAU al P-score en las decisiones de tratamiento, realizamos una simulación Monte Carlo, modelando los ajustes de tratamiento basados en probabilidades. En este análisis, el uso del P-score condujo a una probabilidad estimada de intensificación del tratamiento del 10,06% para la NCCN y del 9,94% para D’Amico/EAU, mientras que la probabilidad de desintensificación fue del 9,55% para la NCCN, del 9,49% para D’Amico y del 9,48% para el sistema de la EAU (fig. S2 del material suplementario). Los pacientes reclasificados de riesgo intermedio a alto por el P-score tenían más probabilidades de recibir un tratamiento intensificado, como la adición de TPA a RT. Sin embargo, el impacto global sobre la intensidad del tratamiento fue modesto, y la PR o la RT siguieron siendo las modalidades de tratamiento principales. Los pacientes cuyo grado se redujo de alto riesgo a riesgo intermedio o bajo por el P-score tuvieron resultados clínicos favorables, sin que se registraran metástasis durante el seguimiento. Los hallazgos sugieren que el uso del P-score en el momento del diagnóstico podría ayudar a prevenir el sobretratamiento al tiempo que se mantiene una gestión eficaz de la enfermedad.
Mortalidad específica por cáncer de próstataDe los 93 pacientes incluidos en el conjunto de datos, 6 (6,5%) murieron de CaP en los 10 años siguientes al diagnóstico. Uno de estos pacientes (1,1%) fue estratificado según el P-score como riesgo intermedio y los 5 pacientes restantes (5,4%) como de alto riesgo. Tres pacientes (3,2%) de este conjunto de datos fallecieron por causas ajenas al CaP. Todos los pacientes restantes (n=84) seguían vivos en el último seguimiento. Diez pacientes desarrollaron metástasis; según el P-score, 6 de ellos se había clasificado de alto riesgo, 3 en riesgo intermedio y uno en riesgo bajo. La clasificación según los sistemas NCCN, D’Amico y de la EAU figura en la tabla 1.
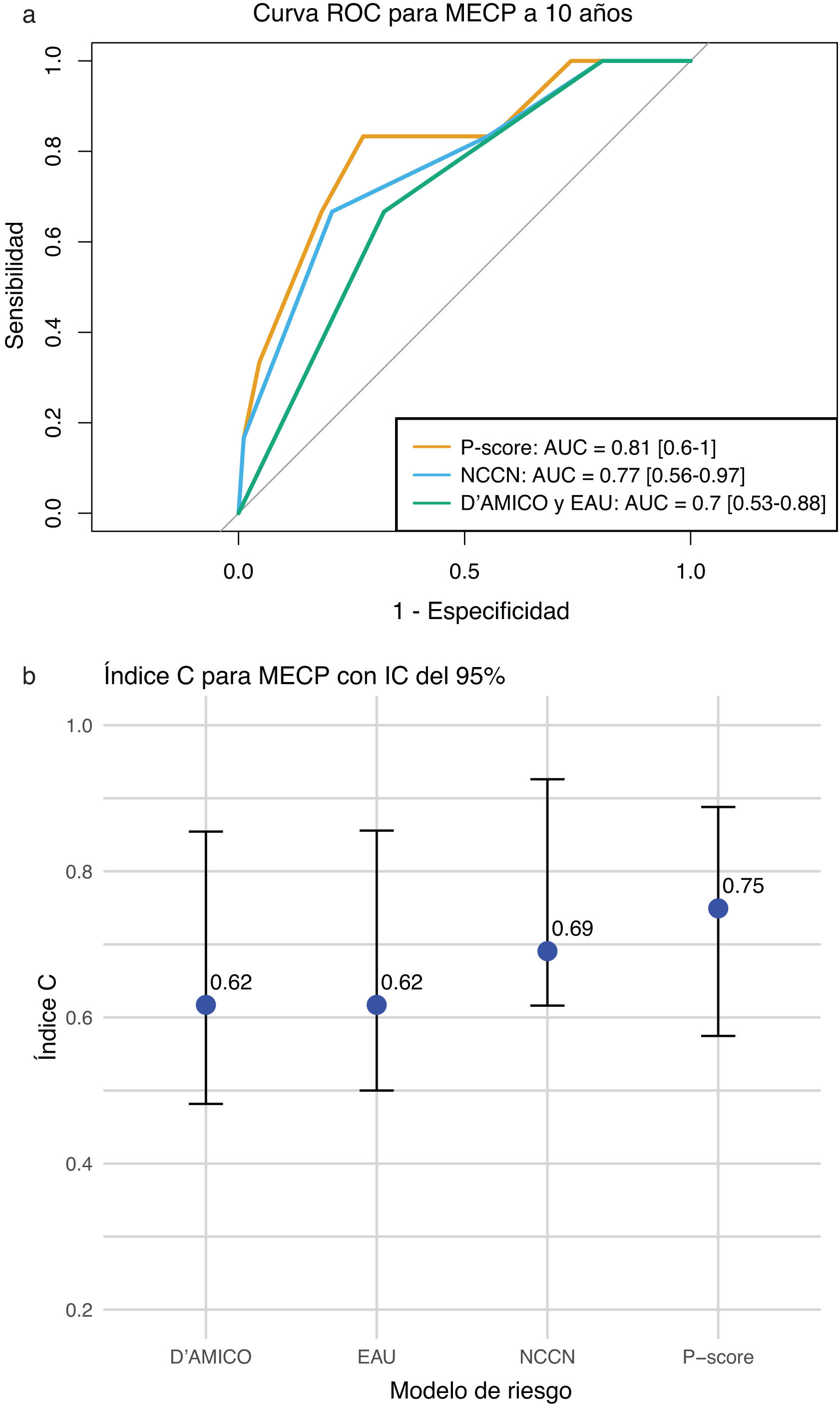
Entre todos los sistemas de estratificación del riesgo incluidos en este estudio, P-score demostró la mayor precisión en la predicción de la MECP a los 10 años, con un AUC de 0,81 (IC 95%: 0,61-1,00), mientras que el AUC para NCCN fue de 0,77 (IC 95%: 0,56-0,97), y tanto la clasificación de D’Amico como la de EAU tuvieron un AUC de 0,70 (IC 95%: 0,53-0,88) (fig. 2a). El índice de concordancia (índice C) fue de 0,75 (IC 95%: 0,58-0,89) para la puntuación P, de 0,69 (IC 95%: 0,62-0,93) para la NCCN, de 0,62 (IC 95%: 0,48-0,85) para D’Amico y de 0,62 (IC 95%: 0,50-0,86) para EAU (fig. 2b).
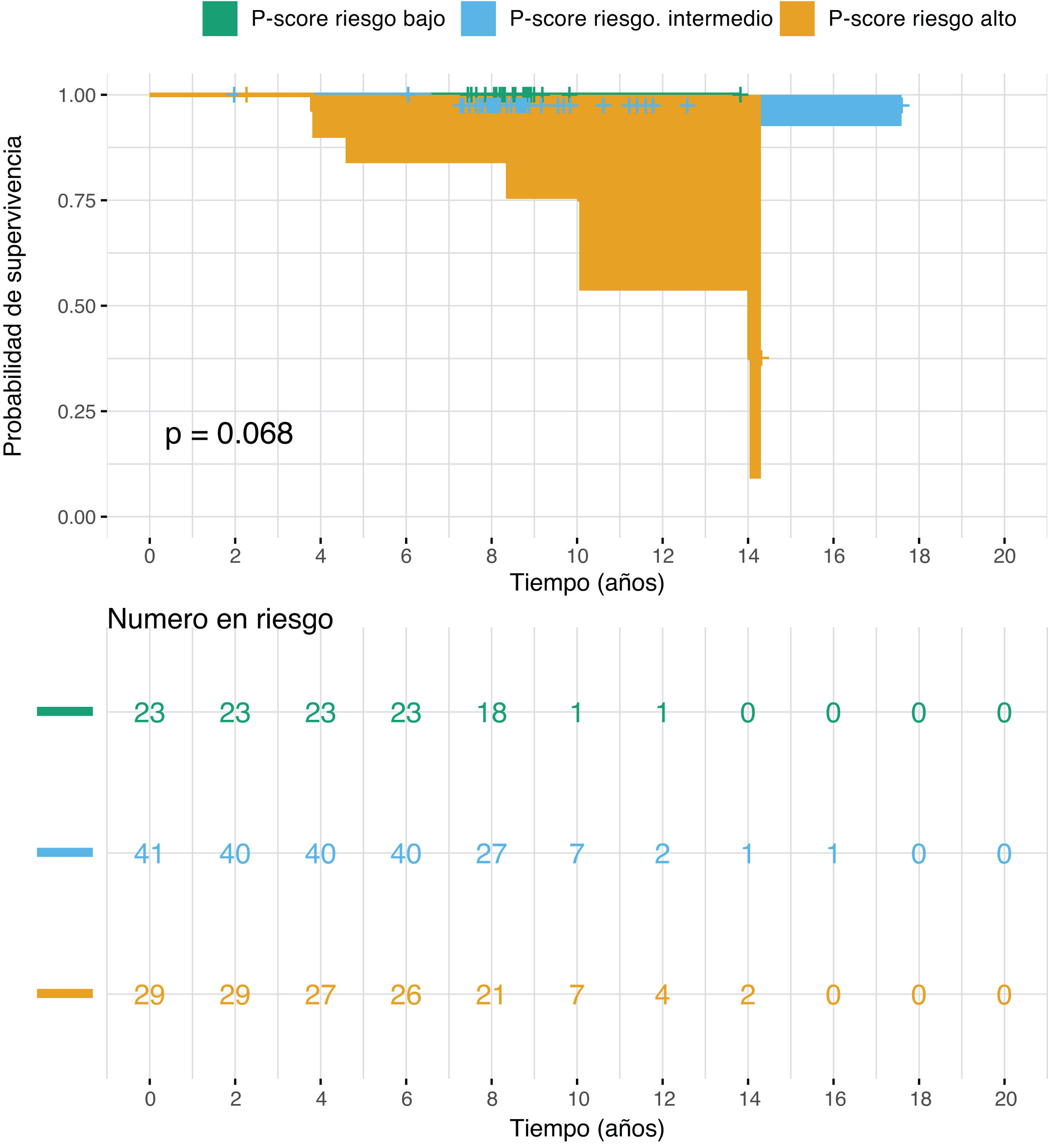
Los gráficos de Kaplan-Meier (KM) ilustran la supervivencia específica del CaP, en la que la muerte refleja el MECP. El P-score (fig. 3) proporcionó la mejor diferenciación entre los grupos de riesgo alto e intermedio durante los primeros 5 años posteriores al diagnóstico. En la clasificación según el P-score, 2 pacientes de alto riesgo murieron de CaP durante este periodo, sin que se produjera ninguna muerte en los grupos de riesgo intermedio o bajo. Según la clasificación de la NCCN (fig. S3 del material suplementario), estos 2 pacientes se clasificaron en el grupo de alto riesgo y en el grupo de riesgo intermedio favorable, mientras que, según los sistemas D’Amico (fig. S4 del material suplementario y EAU (fig. S5 del material suplementario), se clasificaron en los grupos de riesgo intermedio y alto, respectivamente. Las diferencias de supervivencia no fueron estadísticamente significativas, con p=0,068 para el P-score y p≥0,1 para los demás sistemas. Sin embargo, la combinación de los grupos de riesgo bajo e intermedio en el P-score arrojó un valor p estadísticamente significativo de 0,023 (fig. S6 del material suplementario).
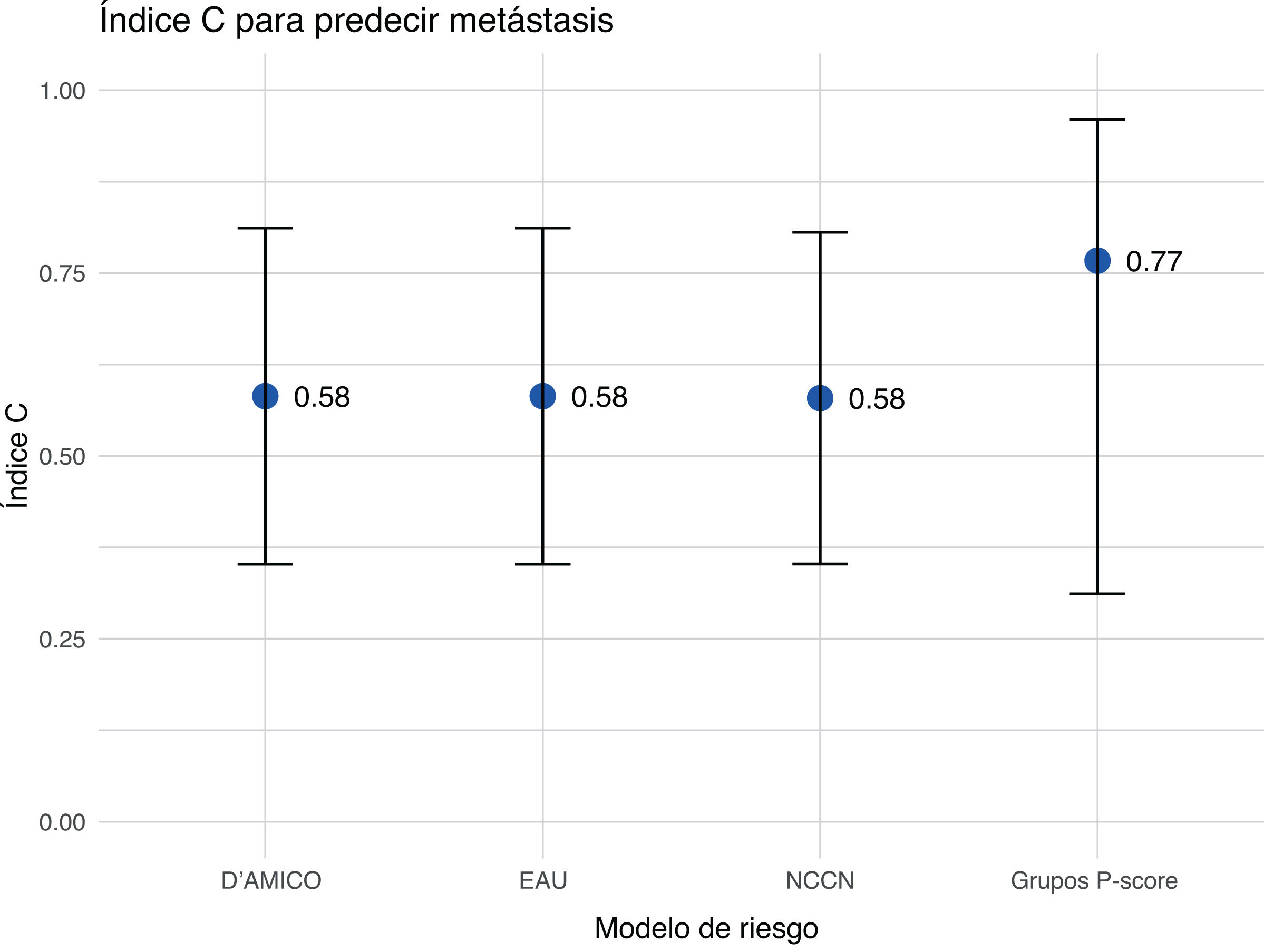
MetástasisAlrededor del 10% de los pacientes con CaP recién diagnosticados presentan metástasis óseas, y este porcentaje aumenta hasta el 80% en estadios avanzados13. La diseminación metastásica dificulta el tratamiento y afecta al pronóstico. La evaluación de la precisión del modelo de riesgo mostró que el P-score tenía la mejor precisión predictiva de metástasis con un índice C de 0,77 (IC 95%: 0,31-0,96), mientras que el índice C de 0,58 (IC 95%: 0,35-0,81) era el mismo para los sistemas NCCN, D’Amico y EAU (fig. 4).
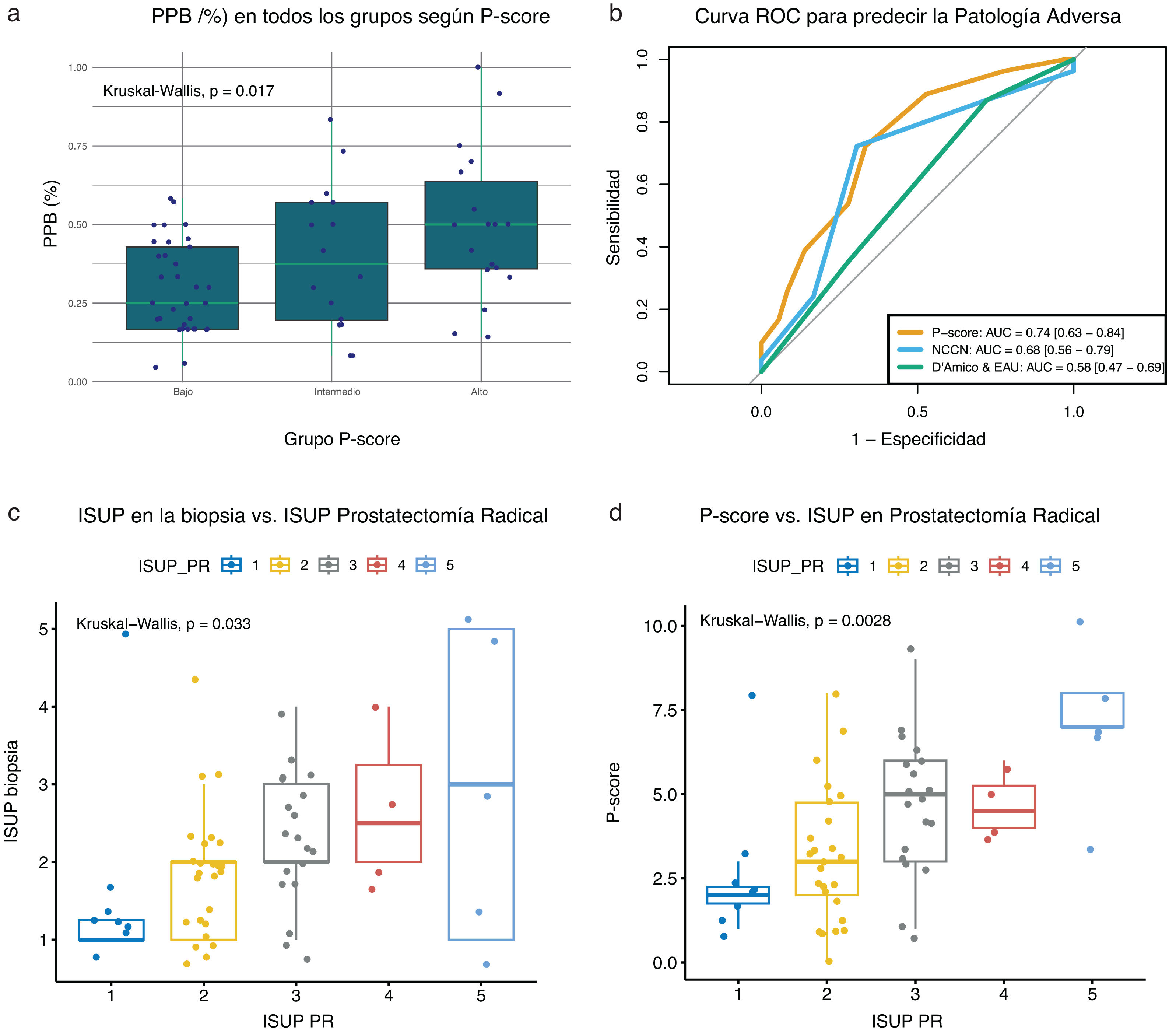
Porcentaje de cilindros positivos en la biopsiaEl porcentaje de cilindros positivos en la biopsia (PPB) proporciona información sobre la extensión y la agresividad del CaP. En consonancia con ello, el PPB aumentó a medida que los grupos pasaban de bajo a alto riesgo (p=0,017; fig. 5a) según el P-score. Esto sugiere que el P-score puede predecir la carga tumoral en los cilindros de la biopsia.
Análisis de las características patológicas, n=68: a) Porcentaje de cilindros positivos en la biopsia (PPB) en todos los grupos de P-score, b) comparación de la enfermedad adversa entre los sistemas P-score, NCCN, D’Amico y EAU en el análisis AUC, c) relación entre el grado ISUP en las muestras de biopsia y el grado ISUP en la PR, d) relación entre los grupos de riesgo según P-score en la biopsia y el grado ISUP en la PR.
Se evaluaron la enfermedad adversa (definida como GS≥4 o estadio patológico ≥T3 en las muestras quirúrgicas) y el grado ISUP en los pacientes sometidos a RP (n=66). El rendimiento predictivo para la PA se representa en curvas ROC (fig. 5b), que muestran que el P-score (AUC=0,74; IC 95%: 0,63-0,84) tiene una capacidad mayor para predecir la PA que los sistemas NCCN (AUC=0,68; IC 95%: 0,56-0,79) y D’Amico y EAU (AUC=0,58; IC 95%: 0,47-0,69).
El sistema de clasificación ISUP es una herramienta histopatológica utilizada para evaluar la agresividad tumoral. Consta de 5 grados, donde el grado 1 representa los cánceres menos agresivos y el grado 5, los más agresivos. La figura 5c muestra que la clasificación ISUP en las muestras de biopsia discrimina entre los grados observados en la PR, con un valor de p=0,033. Por su parte, el P-score categoriza los grados ISUP en la PR con una mayor significación estadística (p=0,0028; fig. 5d).
DiscusiónEl objetivo de este estudio era validar el rendimiento predictivo del P-score en una cohorte española analizando la expresión de IGFBP3, VGLL3 y F3. Utilizando los datos de expresión y la información clinicopatológica, calculamos los valores del P-score a partir de muestras históricas de BAG (≥10 años). Del tejido de biopsia recogido de 154 pacientes, 93 se incluyeron en el análisis, 46 muestras no fueron elegibles debido a la degradación del ARN.
Se realizó un análisis de reclasificación, basado en los pacientes con al menos 8 años de datos de seguimiento, para 57 casos. Al reclasificar las puntuaciones D’Amico/EAU y NCCN según el P-score, 24 casos cambiaron de categoría de riesgo establecida según los sistemas D’Amico/EAU, y 22 casos cambiaron de categoría de riesgo en comparación con el sistema NCCN. La mayoría de las reclasificaciones resultaron en el incremento a una categoría de riesgo superior. Ninguno de los pacientes reclasificados a un grupo de riesgo inferior desarrolló metástasis, mientras que se observaron metástasis en algunos casos reclasificados a un grupo de riesgo superior. Entre los pacientes que se sometieron a PR, el P-score demostró una exactitud predictiva superior para el desarrollo de metástasis en comparación con las clasificaciones NCCN, D’Amico y EAU, lo que sugiere que el uso del P-score es valioso en la identificación de los pacientes con alto riesgo de progresión del CaP.
La PR y la RT siguieron siendo los tratamientos principales. Según el P-score, un ∼10% de los pacientes requería intensificación del tratamiento, lo que estuvo en consonancia con peores resultados de supervivencia, y un ∼9,5% que requería la desintensificación, donde no hubo metástasis. Igual que en otros estudios que informan de una mediana de supervivencia superior a 10 años para los pacientes con CaP1,14, el número de muertes relacionadas con el CaP en este estudio fue bajo (n=6). No obstante, pudimos evaluar la precisión de la predicción de la MECP. Tanto el análisis AUC como el del índice C demostraron que el P-score proporcionaba una mayor precisión en la predicción de la MECP a 10 años en comparación con otros sistemas de puntuación incluidos en este estudio. Los gráficos de Kaplan-Meier para la supervivencia específica del CaP también ilustraron una mayor precisión del P-score, en particular durante los primeros 5 años de seguimiento. En conjunto, estos resultados indican que el P-score es una herramienta más fiable para predecir los resultados específicos del CaP que los sistemas NCCN, D’Amico o EAU.
El P-score demostró un fuerte poder predictivo al evaluar la carga tumoral en muestras de biopsia, lo que resulta esencial para identificar a los pacientes que pueden beneficiarse de tratamientos agresivos o de un seguimiento más estrecho. Al mejorar la precisión predictiva, el P-score permite un enfoque más personalizado del tratamiento. Además, aunque tanto el P-score como el grado ISUP de la biopsia se correlacionaron significativamente con el grado ISUP en la PR, el P-score mostró una asociación significativamente más fuerte, indicada por un valor p más bajo. La validación en una cohorte más amplia podría respaldar aún más su utilidad para identificar a los pacientes con mayor riesgo de progresar a fenotipos de cáncer agresivos antes de la cirugía.
Una limitación importante es el tamaño modesto de la muestra (n=93), con exclusiones debidas a la falta de tejido (9,2%) o a la mala calidad del ARN (33,1%). Estos factores pueden introducir sesgos, aunque la variabilidad en la calidad de la muestra es habitual en los estudios retrospectivos15. Además, aunque se extrajo de múltiples hospitales españoles, es posible que la cohorte de pacientes utilizada en este estudio no represente plenamente la diversidad de la población española de CaP en general, ni tenga en cuenta las variaciones en las prácticas clínicas. Es esencial realizar más estudios a futuro con cohortes más amplias y diversas para confirmar la exactitud predictiva del P-score y mejorar su aplicabilidad en diferentes entornos clínicos.
Este estudio confirma la precisión mayor del P-score en la predicción de la MECP y la metástasis en una cohorte española. Al integrar datos genéticos y clínicos, el P-score ayuda a tomar decisiones de tratamiento personalizadas, distinguiendo a los pacientes de alto riesgo de los candidatos a VA.
Responsabilidades éticasEl estudio cumplió la Declaración de Helsinki y fue aprobado por el Comité de Ética Regional de España. El material de los pacientes se anonimizó y se obtuvo su consentimiento.
Conflicto de interesesEB trabaja para Prostatype Genomics AB. Los autores restantes no declaran ningún otro conflicto de intereses en este trabajo.
Agradecemos enormemente las contribuciones del científico de datos Vladimir Bykov (consultor independiente) y de la redactora médica Agata Wasik (LINK Medical AB, Suecia).