. La espectrometría de masas (EM) MALDI-TOF se ha convertido en un recurso de referencia para la identificación de microorganismos en los servicios de microbiología clínica. No obstante, los datos relativos a algunos grupos de microorganismos son todavía controvertidos. En el presente estudio se ha determinado la fiabilidad de la EM MALDI-TOF para la identificación de aislamientos clínicos de bacterias anaerobias, en comparación con técnicas bioquímicas convencionales, y usando como referencia en caso de discrepancias las secuenciación de ARNr 16S.
Material y métodosSe analizaron 126 aislamientos clínicos de bacterias anaerobias mediante el sistema API 20A (bioMérieux, Marcy l’Étoile, Francia) y mediante EM MALDI-TOF (Autoflex II, Bruker Daltonics, Alemania), utilizando la base de datos BioTyper 2.0 (Bruker Daltonics, Alemania). Cuando se produjeron discrepancias, o la EM MALDI-TOF no fue capaz de identificar microorganismo alguno, se usó como método de identificación de referencia la secuenciación del ARNr 16S.
ResultadosEl método bioquímico y la EM MALDI-TOF coincidieron, a nivel de especie, en el 60,9% de los casos, y solo a nivel de género en el 20,3%. De las 48 identificaciones discrepantes, la secuenciación respaldó la identificación por EM MALDI-TOF a nivel de especie en 32 casos (66,7%), y a nivel de género en 8 (16,7%). Dicha secuenciación apoyó la identificación bioquímica a nivel de especie solamente en 2 casos (4,2%), y a nivel de género en 26 casos (54,2%). En los 8 casos en que la EM MALDI-TOF no obtuvo identificación alguna o la obtenida fue refutada a nivel de género por la secuenciación de ARNr 16S, la especie a la que correspondía en realidad el microorganismo no se encontraba incorporada a la base de datos BioTyper II.
ConclusionesLos datos obtenidos en este estudio demuestran que, en conjunto, la espectrometría de masas ofrece una identificación de bacterias anaerobias más fiable que la identificación bioquímica convencional (un 24% más de identificaciones correctas a nivel de especie), con un número de errores mayores (identificación incorrecta a nivel de género) 2,5 veces inferior. Puesto que todas las identificaciones fallidas a nivel de género derivaron de la no incorporación de varias especies a la base de datos, a medida que esta base de datos se vaya completando, las diferencias probablemente se incrementarán.
MALDI-TOF mass spectrometry (MS) is becoming a major resource in the Clinical Microbiology laboratory. Results on some groups of microorganisms are still controversial. We have studied the reliability of MALDI-TOF MS for the identification of anaerobic clinical isolates was studied compared to conventional biochemical methods, with rRNA 16S sequencing being used as a reference when discrepancies arose.
Material and methodsA total of 126 anaerobic bacteria clinical isolates were studied by using API20A kits (bioMérieux, Marcy l’Étoile, France) and MALDI-TOF MS (Autoflex II, Bruker Daltonics, Germany), and using the data library BioTyper 2.0 (Bruker Daltonics, Germany). When discrepancies arose, or MALDI-TOF MS was not able to identify any microorganism, rRNA 16S sequencing was used as the reference standard.
ResultsThe biochemical method and MALDI-TOF MS agreed in identifying 60.9% of isolates at species level, and 20.3% of isolates at genus level. Among the 48 discrepancies observed, rRNA 16S sequencing supported MALDI-TOF MS identification, at species level, in 32 isolates (66.7%), and in 8 isolates (16.7%) at genus level. rRNA 16S sequencing supported biochemical identification in only two isolates (4.2%) at species level, and in 26 isolates (54.2%) at genus level. The eight isolates for which MALDI-TOF MS did not manage to identify, or the identification obtained was rejected by sequencing, belonged to species that are still not added to the BioTyper II data library.
ConclusionsResults obtained in this study show that, overall, MALDI-TOF MS identification of anaerobic bacteria is more reliable than identification obtained by conventional biochemical methods (24% more correct identifications at species level). The number of major errors (incorrect identification at the genus level) is also 2.5-times lower. Moreover, all the major errors obtained by MALDI-TOF MS were due to the absence of some species in the data library. Thus, when data libraries are more complete, reliability differences between both methods will probably be even higher.
La espectrometría de masas (EM) MALDI-TOF ha revolucionado la metodología de identificación de microorganismos en microbiología clínica por su rapidez, fiabilidad y bajo coste en consumibles. Diversos estudios demuestran su gran fiabilidad en la identificación de diferentes grupos de microorganismos (enterobacterias, bacilos gramnegativos no fermentadores, estafilococos, levaduras, etc.). Sin embargo, algunos grupos de microorganismos presentan todavía limitaciones, como es el caso de los estreptococos del grupo viridans, o han sido estudiados de manera menos detallada, como ocurre, entre otros grupos, con las bacterias anaerobias. Mientras algunos estudios muestran porcentajes de identificación correcta muy satisfactorios1–5, otros ofrecen porcentajes que no van más allá del 60-70%. Por otra parte, existen estudios que demuestran que, probablemente, la identificación bioquímica clásica de estos microorganismos tenía una resolución insuficiente para la identificación de la gran variedad de bacterias anaerobias potencialmente patógenas para el hombre. De este modo, agrupaba dentro de la misma especie a microorganismos en realidad diferentes, y era incapaz de identificar a otros, llevando en conjunto a una considerable simplificación de la etiología de las infecciones por bacterias anaerobias. La disponibilidad de técnicas de mayor resolución, como la secuenciación del ARNr 16S, y sobre todo la EM MALDI-TOF, mucho más aplicable en la rutina diaria del Laboratorio de Microbiología Clínica, probablemente dará una visión más exacta de la complejidad real del grupo de los microorganismos anaerobios patógenos y de la importancia como patógenos de especies que, hasta ahora, estaban probablemente infradiagnosticadas.
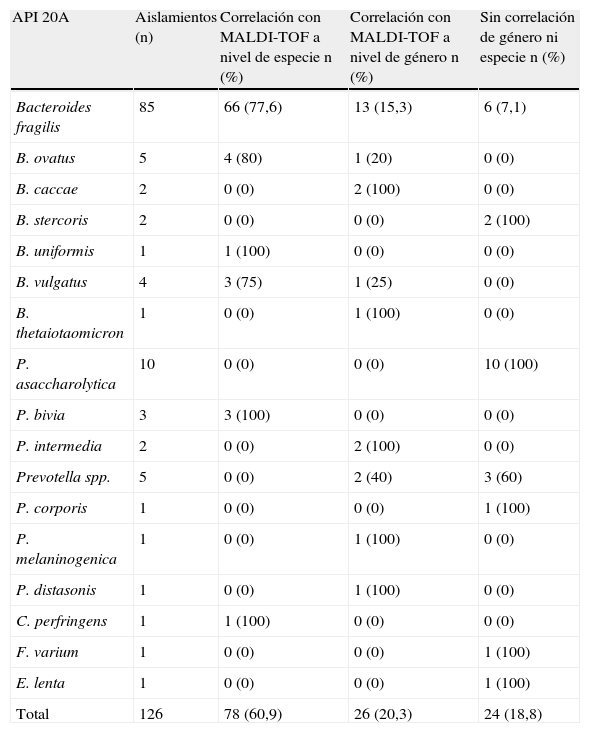
Material y métodosSe analizaron 126 aislamientos clínicos de bacterias anaerobias que fueron identificados mediante el sistema API 20A® (bioMérieux, Marcy l’Étoile, Francia) siguiendo las instrucciones del fabricante. La identificación bioquímica de estos microorganismos aparece reflejada en la tabla 1.
Correlación entre la identificación bioquímica y la identificación por EM MALDI-TOF en bacterias anaerobias
| API 20A | Aislamientos (n) | Correlación con MALDI-TOF a nivel de especie n (%) | Correlación con MALDI-TOF a nivel de género n (%) | Sin correlación de género ni especie n (%) |
| Bacteroides fragilis | 85 | 66 (77,6) | 13 (15,3) | 6 (7,1) |
| B. ovatus | 5 | 4 (80) | 1 (20) | 0 (0) |
| B. caccae | 2 | 0 (0) | 2 (100) | 0 (0) |
| B. stercoris | 2 | 0 (0) | 0 (0) | 2 (100) |
| B. uniformis | 1 | 1 (100) | 0 (0) | 0 (0) |
| B. vulgatus | 4 | 3 (75) | 1 (25) | 0 (0) |
| B. thetaiotaomicron | 1 | 0 (0) | 1 (100) | 0 (0) |
| P. asaccharolytica | 10 | 0 (0) | 0 (0) | 10 (100) |
| P. bivia | 3 | 3 (100) | 0 (0) | 0 (0) |
| P. intermedia | 2 | 0 (0) | 2 (100) | 0 (0) |
| Prevotella spp. | 5 | 0 (0) | 2 (40) | 3 (60) |
| P. corporis | 1 | 0 (0) | 0 (0) | 1 (100) |
| P. melaninogenica | 1 | 0 (0) | 1 (100) | 0 (0) |
| P. distasonis | 1 | 0 (0) | 1 (100) | 0 (0) |
| C. perfringens | 1 | 1 (100) | 0 (0) | 0 (0) |
| F. varium | 1 | 0 (0) | 0 (0) | 1 (100) |
| E. lenta | 1 | 0 (0) | 0 (0) | 1 (100) |
| Total | 126 | 78 (60,9) | 26 (20,3) | 24 (18,8) |
La EM se llevó a cabo siguiendo métodos previamente descritos6. Cada microorganismo fue procesado por duplicado, según las instrucciones del fabricante (Bruker Daltonics GmbH, Leipzig, Alemania). La identificación se llevó a cabo a partir de colonias crecidas en agar Wilkins-Chalgren con 5% de sangre desfibrinada, preparado a partir de medio deshidratado Wilkins Chalgren anaerobe agar (Oxoid, Thermo Scientific, Basingstoke, Reino Unido), o en agar Brucella con 5% de sangre desfibrinada suplementado con gentamicina y vancomicina (bioMérieux, La Balme Les Grottes, Francia), tras 48h de incubación en condiciones de anaerobiosis. La identificación se llevó a cabo sobre una sola colonia de cada microorganismo.
- 1.
Preparación de la muestra. Se depositó una pequeña cantidad de una colonia directamente sobre la placa ground steel del espectrómetro de masas, formando una delgada película. Sobre ella se aplicó 1μl de la solución matriz (solución saturada de ácido alfa-ciano-4-hidroxicinámico en acetonitrilo al 50% y ácido trifluoroacético al 2,5%) y se dejó secar a temperatura ambiente.
- 2.
Espectrometría de masas. Las medidas se realizaron en un espectrómetro de masas MALDI-TOF-TOF Autoflex III (Bruker Daltonics GmbH, Leipzig, Alemania). Se obtuvo el espectro entre 2 y 20kDa de forma automática, trabajando en modo lineal positivo a una frecuencia de 200hertzios. Los parámetros que se fijaron para el espectrómetro fueron IS1 a 20kV, IS2 a 18,6kV, lente a 6kV, PIE a 40ns. El protocolo de trabajo del MALDI Biotyper proporciona la adquisición óptima de la muestra (acumulación de 500disparos del láser en diferentes lugares de la muestra). Los espectros se calibraron externamente utilizando una mezcla de calibrantes comercial (extracto de Escherichia coli DH5 α más 2 proteínas adicionales: RNAasa A y mioglobina para cubrir un intervalo de 4 a 17kDa).
- 3.
Análisis de los espectros. Para la identificación de los microorganismos, el espectro obtenido de los microorganismos problema se procesó con el programa MALDI Biotyper 2.0. La lista de picos generada se comparó con la biblioteca de referencia del MALDI Biotyper 2.0, usando un algoritmo de comparación integrado en el software. Tras importar el espectro al programa, todo el proceso, desde su análisis hasta su identificación, se lleva a cabo de forma automática, sin intervención alguna por parte del usuario.
- 4.
Valoración de resultados. Las identificaciones mediante MALDI-TOF se clasificaron como fiable a nivel de especie (puntuación en la comparación entre el perfil del microorganismo problema y la base de datos ≥2), fiable a nivel de género (puntuación ≥1,7 y <2) y no fiable (puntuación <1,7). Las identificaciones que entraron en esta última categoría se consideraron fallidas. En cuanto a la comparación entre MALDI-TOF y los sistemas clásicos, se diferenció según hubiera correlación de género y especie, correlación solo de género (bien porque el sistema solo identificara a este nivel, o porque hubiera discrepancia en la especie identificada), o no hubiera correlación de especie ni género. En este último caso, la identificación se consideró asimismo fallida.
Cuando no existió correlación entre la identificación bioquímica y la obtenida por MALDI-TOF, o este último no ofreció identificación fiable alguna, la identificación se corroboró mediante secuenciación del ARNr 16S7.
ResultadosLos resultados obtenidos mediante identificación convencional y mediante EM MALDI-TOF aparecen reflejados en la tabla 1. El método bioquímico y la EM MALDI-TOF coincidieron, a nivel de especie, en el 60,9% de los casos, y a nivel de género en el 20,3%, de modo que, en conjunto, las identificaciones coincidieron, al menos a nivel de género, en el 81,2% de los casos. En los aislamientos identificados por métodos bioquímicos dentro del género Bacteroides, las identificaciones fueron coincidentes a nivel de especie en el 74% de los casos, y a nivel de género en el 18%, de modo que, en conjunto, las identificaciones fueron coincidentes como mínimo a nivel de género en el 92% de los casos.
Sin embargo, en los aislamientos identificados bioquímicamente dentro del género Porphyromonas, la identificación fue discordante a nivel de especie y a nivel de género en el 100% de los casos, y en los identificados dentro del género Prevotella, la identificación fue coincidente solo en el 25% de los casos a nivel de especie y en el 41,7% a nivel de género. Por el contrario, el 33,3% de los aislamientos mostraron una identificación discordante a nivel de género y especie.
Cuando se compararon las divergencias con la identificación obtenida por secuenciación de ARNr 16S (tabla 2) se observó que, de 48 identificaciones discrepantes, la secuenciación respaldó la identificación por EM MALDI-TOF a nivel de género y especie en 32 casos (66,7%), y a nivel de género en ocho (16,7%). De estos 8 casos, en tres (2 Prevotella oris y un Bacteroides dorei) el microorganismo identificado por la secuenciación de ARNr 16S no estaba recogido en la base de datos Biotyper 2.0, por lo que fueron identificados como Prevotella spp. (ninguna especie alcanzó el score de 2.0), Prevotella buccae y Bacteroides uniformis. De los 5 restantes, en 2 la especie que obtuvo mayor score fue la misma que posteriormente se corroboró por secuenciación de ARNr 16S (Prevotella buccae, score: 1.914; Prevotella melaninogenica, score: 1,705), mientras que en otros 3, la especie con mayor score fue distinta (Prevotella nigrescens por EM MALDI-TOF [score 1,726-1,771] en 3 casos identificados por secuenciación como Prevotella intermedia).
Identificación por ARNr 16S de las discrepancias entre la identificación convencional y la EM MALDI-TOF
| API 20A (n) | Id. EM MALDI-TOF (n) | Id. rRNA 16S (n) |
| Bacteroides fragilis (19) | Id. no fiable (score <1,7) (2) | Bacteroides cellulosilyticusa (2) |
| Bacteroides thetaiotaomicron (4) | Bacteroides thetaiotaomicron (4) | |
| Parabacteroides distasonis (3) | Parabacteroides distasonis (3) | |
| Bacteroides nordii (1) | Bacteroides nordii (1) | |
| Bacteroides vulgatus (3) | Bacteroides vulgatus (3) | |
| Bacteroides ovatus (3) | Bacteroides ovatus (3) | |
| Prevotella spp.b (1) | Prevotella buccae (1) | |
| Prevotella buccae (1) | Prevotella orisa (1) | |
| Prevotella bivia (1) | Prevotella bivia (1) | |
| Bacteroides ovatus (1) | Bacteroides thetaiotaomicron (1) | Bacteroides thetaiotaomicron (1) |
| Bacteroides. caccae (2) | Bacteroides fragilis (1) | Bacteroides fragilis (1) |
| Bacteroides thetaiotaomicron (1) | Bacteroides thetaiotaomicron (1) | |
| Bacteroides stercoris (2) | Prevotella nigrescens (1) | Prevotella nigrescens (1) |
| Fusobacterium varium (1) | Fusobacterium varium (1) | |
| Bacteroides vulgatus (1) | Bacteroides uniformis (1) | Bacteroides doreia (1) |
| Bacteroides thetaiotaomicron (1) | Bacteroides fragilis (1) | Bacteroides fragilis (1) |
| Porphyromonas asaccharolytica (10) | Prevotella sppc (3) | Prevotella intermedia (3) |
| Id. no fiable (score <1.7) (2) | Bacteroides ureolyticusa (2) | |
| Prevotella nigrescens (1) | Prevotella nigrescens (1) | |
| Prevotella melaninogenica (1) | Prevotella melaninogenica (1) | |
| Prevotella bivia (1) | Prevotella bivia (1) | |
| Id. no fiable (score <1,7) (1) | Dialister pneumosintesa (1) | |
| Bacteroides fragilis (1) | Bacteroides fragilis (1) | |
| Prevotella intermedia (2) | Prevotella nigrescens (1) | Prevotella nigrescens (1) |
| Prevotella spp.d (1) | Prevotella orisa (1) | |
| Prevotella spp (5) | Id. no fiable (score <1,7) (1) | Negativicoccus succinivoransa (1) |
| Id. no fiable (score <1,7) (1) | Olsenella spp.a (1) | |
| Bacteroides fragilis (1) | Bacteroides fragilis (1) | |
| Prevotella oralis (1) | Prevotella oralis (1) | |
| Prevotella melaninogenica (1) | Prevotella melaninogenica (1) | |
| Prevotella corporis (1) | Id. no fiable (score <1,7) (1) | Prevotella corporisa (1) |
| Prevotella melaninogenica (1) | Prevotella spp.e (1) | Prevotella melaninogenica (1) |
| Parabacteroides distasonis (1) | Bacteroides thetaiotaomicron (1) | Bacteroides thetaiotaomicron (1) |
| Fusobacterium varium (1) | Prevotella bivia (1) | Prevotella bivia (1) |
| Eggerthella lenta (1) | Bifidobacterium longum (1) | Bifidobacterium longum (1) |
En los 8 casos (16,7%) en que la EM MALDI-TOF no fue capaz de identificar el aislamiento ni siquiera a nivel de género, ello se debió a que, según la secuenciación de ARNr 16S, se trataba de especies no incluidas en la base de datos Biotyper 2.0 (2 Bacteroides cellulosilyticus, 2 Bacteroides ureolyticus, un Dialister pneumosintes, un Negativicoccus succinivorans, un Olsenella spp., un Prevotella corporis).
Por el contrario, de las 48 identificaciones discrepantes, la secuenciación de ARNr 16S apoyó la identificación bioquímica a nivel de especie solamente en 2 casos (4,2%) (un Prevotella corporis, un Prevotella melaninogenica), y a nivel de género en 26 casos (54,2%). La comparación global entre ambos métodos, tomando como referencia la secuenciación de ARNr 16S, aparece en la tabla 3.
Comparación de errores a nivel de género y especie de la identificación convencional y la espectrometría de masas MALDi-TOF, tomando como referencia la secuenciación de rRNA 16S
| Rapid 32A (n) | Id. EM MALDI-TOF (n) | ||||
| Id. correctaan (%) | Error especie n (%) | Error género n (%) | Id. correcta n (%) | Error especie n (%) | Error género n (%) |
| 80 (63,5) | 26 (20,6) | 20 (15,9) | 110 (87,3) | 8 (6,3) | 8 (6,3)b |
Los estudios sobre aplicación de EM-MALDI-TOF a la identificación de microorganismos muestran, en general, resultados muy positivos respecto a su fiabilidad6,8–11. Sin embargo, existen grupos concretos de microorganismos en los que existe menos experiencia, o los resultados publicados son más heterogéneos. Uno de ellos es el de las bacterias anaerobias. Algunos estudios muestran buenos resultados en la identificación de determinados géneros. Este es el caso de algún estudio relativo a la identificación de Clostridium, aunque, como ocurre en general en relación con las bacterias anaerobias, la rentabilidad diagnóstica del método está muy condicionada por la calidad de la base de datos usada como referencia1. En nuestro caso solo se incluye un aislamiento de C. perfringens, que fue correctamente identificado, pero la casuística es excesivamente baja para obtener conclusiones. Un estudio reciente muestra también buenos resultados en la identificación de especies de Prevotella aisladas de cuadros de periodontitis2. En el presente estudio se incluyeron 20 aislamientos de Prevotella, de acuerdo con la identificación por secuenciación. De ellos, 12 se identificaron por EM MALDI-TOF correctamente a nivel de especie (60%) y 8 a nivel de género (40%). No se identificó incorrectamente como Prevotella ningún aislamiento perteneciente a otros géneros. Los resultados son inferiores a los obtenidos en otros géneros, como Bacteroides (80,8% de identificaciones correctas a nivel de especie), pero son sin embargo muy superiores a los obtenidos con la identificación bioquímica, que han resultado mediocres en la identificación de Prevotella. El método bioquímico identificó correctamente 5 de 20 aislamientos a nivel de especie (25%) y 4 a nivel de género (16%), errando la identificación a nivel de género en el 59% de los casos. Además, identificó erróneamente como Prevotella a 4 aislamientos de los 106 anaerobios no pertenecientes al género Prevotella incluidos en el estudio (3,8%).
Un estudio más amplio sobre 270 aislamientos clínicos de bacterias anaerobias, no restringido a géneros concretos, muestra que el 97,5% de los aislamientos fueron identificados a nivel de especie (score >2,00) por MALDI-TOF. Además, en 10 de los 11 aislamientos en que se dieron discrepancias con respecto a la identificación convencional, la secuenciación del ARNr 16S confirmó la identificación obtenida por MALDI-TOF3. Del mismo modo, otra publicación realizada en 2011 muestra un 95% de identificaciones correctas en cocos grampositivos anaerobios, usando también como referencia la secuenciación de ARNr 16S4, una vez confeccionada una base de datos a medida que incluía el perfil de cepas tipo de las principales especies de cocos grampositivos anaerobios.
Sin embargo, otras publicaciones recientes ofrecen resultados menos favorables. En un estudio comparativo de 2 sistemas comerciales de EM MALDI-TOF sobre 79 aislamientos pertenecientes a 19 géneros, usando como referencia la secuenciación de ARNr 16S, Shimadzu identificó correctamente el 71% de los aislamientos a nivel de género, y el 61% a nivel de especie. Bruker Daltonics obtuvo resultados inferiores, con el 61 y el 51%, respectivamente. No obstante, cuando el mismo estudio se realizó con una versión actualizada del software de Bruker Daltonics, los resultados se equipararon6. Otro estudio posterior, también publicado en 2011, sobre más de 500 aislamientos de bacterias anaerobias muestra también porcentajes de identificación correcta del 61%8, y el estudio más reciente publicado hasta el momento muestra porcentajes similares (67% de identificaciones correctas con Bruker frente a 49% con Shimadzu)9. Estos estudios muestran resultados inferiores al nuestro, en el que la EM MALDI-TOF identificó correctamente a nivel de especie al 87,6% de los aislamientos y, a nivel de género, al 93,7%. La mayor parte de los estudios referidos inciden en la importancia de la base de datos para obtener buenos resultados con este sistema10. Esta circunstancia es ratificada por otros autores, que afirman que la utilidad clínica de estos sistemas para la identificación de bacterias anaerobias pasa por una optimización de las bases de datos disponibles11. Los resultados de nuestro estudio corroboran esta afirmación, ya que el 100% de las identificaciones fallidas (score <1,7) por EM MALDI-TOF corresponde a microorganismos cuyos perfiles no estaban incluidos en la versión de la base de datos utilizada.
Un estudio reciente demuestra que, si se elaboran bases de datos de referencia amplias de los grupos fenotípicos, basadas en una identificación inequívoca por secuenciación de ARNr 16S, los resultados posteriores de la EM MALDI-TOF en muestras clínicas mejoran sensiblemente4. Del mismo modo, un estudio reciente limitado al género Bacteroides muestra que la correlación con la secuenciación del ARNr 16S es mucho mayor en el caso de la EM MALDI-TOF que en el de la identificación bioquímica convencional (87% frente a 52,3%), y que en la mayoría de los casos las identificaciones erróneas derivan de la no inclusión de algunas especies descritas recientemente en las bases de datos12. Esto es especialmente evidente, en el caso de este estudio, en Bacteroides dorei, que es también incorrectamente identificado como B. uniformis en nuestro estudio al no estar presente B. dorei en la base de datos Biotyper 2.0.
Hace casi 10 años, Shah auguraba que la EM MALDI-TOF iba a tener un notable impacto en el diagnóstico de los numerosos microorganismos anaerobios que crecen mal en los medios de cultivo habituales y se identifican de manera poco fiable por los sistemas de identificación convencionales13. Los datos que van estando disponibles, al igual que los obtenidos en este estudio, demuestran que, en conjunto, la EM ofrece una identificación de bacterias anaerobias sensiblemente más fiable que la identificación bioquímica convencional (un 24% más de identificaciones correctas a nivel de especie en el presente estudio), con un número de errores mayores (identificación incorrecta a nivel de género) muy inferior (2,5 veces inferior en este estudio). Además, todas las identificaciones fallidas a nivel de género derivan de la no inclusión de diferentes especies en la base de datos, de modo que, a medida que esta base de datos se vaya completando, las diferencias serán probablemente incluso mayores.