En la actualidad, tanto las imágenes como los datos que generan y los informes que se emiten son digitales y constituyen una fuente de datos fiable. Los informes pueden clasificarse, en función de su contenido, formato y tipo de datos, como organizado (texto libre en lenguaje natural), predefinido (con plantillas y guías construidas con campos previamente determinados con lenguaje natural tipo BI-RADS y PI-RADS) y estructurado (con desplegables en forma de preguntas, con diversas posibles respuestas, previamente pactadas en el equipo multidisciplinario, con léxicos estandarizados y estructurado como base de datos, con información trazable y explotable mediante herramientas estadísticas y de minería de datos). Este informe estructurado, compatible con MRRT (Management of Radiology Report Templates), permite incorporar información cuantitativa relacionada con el análisis digital de los datos de las imágenes adquiridas para describir, mediante radiómica (características y parámetros), las propiedades y el comportamiento de los tejidos con exactitud y veracidad. En conclusión, los datos digitales estructurados (imágenes, texto, mediciones, radiómica, biomarcadores de imagen) deben integrarse en un informe informatizado que permita su indexación en grandes repositorios. Estos bancos de datos radiológicos son fundamentales en la medicina personalizada para explotar la información sanitaria, fenotipificar las lesiones y enfermedades, y extraer conclusiones.
Nowadays, the images and information generated in imaging tests, as well as the reports that are issued, are digital and represent a reliable source of data. Reports can be classified according to their content and to the type of information they include into three main types: organized (free text in natural language), predefined (with templates and guidelines elaborated with previously determined natural language like that used in BI-RADS and PI-RADS), or structured (with drop-down menus displaying questions with various possible answers that have been agreed on with the rest of the multidisciplinary team, which use standardized lexicons and are structured in the form of a database with data that can be traced and exploited with statistical tools and data mining). The structured report, compatible with Management of Radiology Report Templates (MRRT), makes it possible to incorporate quantitative information related with the digital analysis of the data from the acquired images to accurately and precisely describe the properties and behavior of tissues by means of radiomics (characteristics and parameters). In conclusion, structured digital information (images, text, measurements, radiomic features, and imaging biomarkers) should be integrated into computerized reports so that they can be indexed in large repositories. Radiologic databanks are fundamental for exploiting health information, phenotyping lesions and diseases, and extracting conclusions in personalized medicine.
El objetivo común de los radiólogos en su práctica diaria es realizar el procedimiento de imagen correcto para una justificación clínica dada, de forma adecuada y protocolizada, además de segura para el paciente. Los radiólogos asumen que cuando se solicita una exploración no solo se piden las imágenes, sino que, mucho más relevante para el proceso del paciente, se espera una evaluación adecuada, exhaustiva y completa de su situación concreta en un momento dado. El informe radiológico es, pues, el principal elemento de comunicación de los radiólogos con la comunidad sanitaria y con los pacientes. En este informe, la conclusión emitida como juicio sitúa al radiólogo como consultor especialista cuyo objetivo principal es informar del estado del paciente con coherencia y precisión, eliminando las posibles variabilidades e incertidumbres innecesarias.
Sin embargo, pese a su importancia, los informes radiológicos no están lo suficientemente estandarizados ni consensuados con respecto a la información que deben contener. Hasta la llegada del informe estructurado, los esfuerzos por organizarlos, generalizarlos y controlar su calidad han sido poco ambiciosos. En la actualidad, tanto las imágenes que se adquieren como los datos que generan y los informes que se emiten son digitales y constituyen una fuente de datos que debe ser completa y fiable, clínicamente más útil y científicamente explotable. No solo las imágenes son datos; los informes, también. Estos informes radiológicos expresan la calidad global del servicio de radiología y no únicamente la opinión de las personas que los emiten1,2.
Como criterios básicos de calidad, la comunicación que representa el informe debe cumplir con los criterios de utilidad para el prescriptor y el paciente, pertinencia al proceso clínico y a la salud del paciente, y claridad, es decir, debe ser inteligible y preciso, conciso y claro, organizado y completo. Por lo tanto, tiene que expresar el grado de confianza del radiólogo en las observaciones y en los juicios, mencionando las limitaciones que pudiera tener la exploración y evitando en la medida de lo posible el uso de indefiniciones (tales como compatible con, parece tratarse de, no hay signos de, es posible que)1,2.
Pueden distinguirse varios tipos principales de informes radiológicos en función de la disposición, el contenido y el formato de su información1–3 (fig. 1). El primer modelo utiliza texto libre organizado en distintas secciones, donde el radiólogo introduce en lenguaje natural la información pertinente de forma organizada por apartados generales. Estos incluyen los datos clínicos como justificación de la prueba de estudio, resumiendo sus aspectos más relevantes; la técnica realizada con el protocolo de exploración empleado, incluyendo el medio de contraste empleado y los efectos indeseados que pudieran haberse presentado; así como los problemas en la adquisición y las fechas de los estudios previos que se hubieran empleado para comparar los hallazgos y evaluar la evolución. El cuerpo principal de este informe, V1 u organizado, son los hallazgos anormales, donde el radiólogo describe las lesiones y alteraciones detectadas y su apariencia, con las comparaciones oportunas, además de citar lo normal que sea relevante al juicio clínico. Finalmente se incluye el cuerpo de la conclusión, con un resumen conciso de la enfermedad (detectar, localizar, gradar, estadificar, evaluar respuesta) y los consejos sobre el proceder diagnóstico y terapéutico que considere pertinentes y fundados. Actualmente, este tipo de informe en texto plano y lenguaje natural es el más utilizado por los facultativos. Este documento expresa las habilidades clínicas desarrolladas por los radiólogos para que los pacientes tengan un diagnóstico preciso y una terapia adecuada a su situación1,2.
El segundo modelo, V2, se desarrolla para facilitar el trabajo clínico de los radiólogos, minimizando la variabilidad y promoviendo la completitud y la relevancia de los informes que emite. Esta versión de informe predefinido utiliza plantillas y guías construidas con campos previamente determinados en los que se definen aquellos aspectos que el radiólogo debe completar con sus observaciones. Estos informes son útiles tanto en situaciones de baja probabilidad de alteraciones, en los que hay una guía sistematizada de lectura, como en aquellas enfermedades en las que deben recogerse una serie de hallazgos para que la lectura sea completa, como por ejemplo en los estudios de endometriosis4. Además, estos campos pueden estar delimitados por su tipología y por el número de opciones que puede seleccionar el facultativo5. En este sentido, la Radiological Society of North America (RSNA) promueve, entre sus mejores prácticas, adaptar este informe predefinido a cada centro6. Estos informes en texto libre y lenguaje natural están organizados en librerías de acceso por el radiólogo, organizadas por técnicas de estudio y por la enfermedad objeto de evaluación, con una completitud basada en la evidencia científica disponible, incorporando análisis multimodalidad y una semántica estandarizada. Este tipo de informes ha permitido el desarrollo de las guías tipo BI-RADS para las lesiones de mama, PI-RADS para el cáncer de próstata, TI-RADS para el tiroides, LI-RADS para el carcinoma hepatocelular sobre hígado cirrótico y CAD-RADS para las estenosis coronarias en la coronariografía por tomografía computarizada7.
El informe de cáncer de mama BI-RADS (Breast Imaging Reporting and Data System) es el mejor ejemplo de este tipo de informes, y el primero que se implementó. Su relevancia está basada en la necesidad de estandarizar y normalizar la información emitida por el radiólogo, dada la prevalencia de esta patología y la influencia clínica de los posibles hallazgos. BI-RADS cuenta con cinco ediciones distintas: 1993, 1995, 1998, 2003 y 20138. En ellas se incluyen guías específicas para mamografía, ecografía y resonancia magnética, en las que todos los términos definidos están codificados en RADLEX. La mejora más importante es la aportación de una mayor claridad y una mejor consistencia en la información generada, repercutiendo finalmente en la atención al paciente. Como ejemplo, un estudio realizado entre residentes de radiología de América del Norte y Canadá concluía que el 98% de ellos utilizaron BI-RADS en sus informes mamográficos9.
Una aproximación estructurada relacionada con el informe predefinido BI-RADS es la realizada con la herramienta de trabajo TRENCADIS10. En ella se genera una solución Web a través de una interfaz gráfica de usuario con plantillas DICOM-SR para que el radiólogo realice los informes integrando además toda la información de las pruebas de imagen realizadas. Esta herramienta informática se ha validado, concluyendo que para todas las modalidades y grupos de facultativos la realización del informe BI-RADS estructurado es más completa y requiere menos tiempo, de forma estadísticamente significativa para todos los estudios, independientemente de la experiencia del facultativo.
A raíz de la repercusión de BI-RADS, el American College of Radiology (ACR) incentivó el desarrollo de otros léxicos a medida como plantillas predefinidas de informes. Se desarrollaron así el Liver Imaging Reporting and Data System (LI-RADS), el Lung CT Screening Reporting and Data System (Lung-RADS) y el Head Injury Imaging Reporting and Data System (HI-RADS)7,11–13. El último desarrollo está relacionado con el informe del cáncer de próstata Prostate Imaging Reporting and Data System (PI-RADS), en el que se establece una interpretación inequívoca y consistente en el informe radiológico del carcinoma prostático para el diagnóstico mediante resonancia magnética14,15. Este proyecto fue iniciado y promovido por la European Society of Urogenital Radiology. En la actualidad ya existe demostración científica de la eficiencia y la eficacia de PI-RADS, con un aumento significativo del número de biopsias positivas cuando el radiólogo utiliza el informe estructurado para localizar, estratificar y gradar las lesiones16,17.
El tercer tipo de informe, más evolucionado y complejo, que hemos descrito como informe estructurado V3, utiliza desplegables en forma de preguntas con diversas posibles respuestas, previamente pactadas en el equipo multidisciplinario responsable de la enfermedad o situación clínica objeto del estudio. Emplea un lenguaje con léxicos estandarizados, como puede ser RADLEX, con el fin de dotar a los campos definidos en la estructura de un identificador inequívoco para que el intercambio de información y lectura del informe radiológico sea entendible y completo, de forma independiente del radiólogo que lo ha realizado18,19. Una característica principal de este informe es su estructura como base de datos, con información trazable, integrada en los sistemas de información, con posibilidad de búsqueda informática avanzada, eficiente y explotable mediante herramientas estadísticas y de minería de datos20. Esta aproximación mejora además la eficiencia del trabajo radiológico21.
Evolución histórica del informe digitalEl ACR desarrolló el primer sistema para organizar y definir informes de imagen médica a través de su indexación con una finalidad docente, ofreciendo identificadores anatómicos y patológicos mediante números decimales. Actualmente se encuentran accesibles en Internet distintos materiales didácticos que se ayudan de léxicos tipo SNOMED-CT para organizar la información en los expedientes médicos digitales, dado que disponen de un catálogo de términos más amplio que el ACR.
Por otro lado, la RSNA generó el proyecto MIRC con el propósito de elaborar una herramienta accesible en Internet para la creación de archivos de docencia a través de bibliotecas de imágenes22. MIRC utiliza RADLEX como léxico para proporcionar una terminología utilizable para anotar, indexar y recuperar el contenido de las imágenes médicas y sus informes. RADLEX incluye los códigos anatómicos y patológicos originariamente presentes en el ACR, integrando además otros tipos de términos, tales como equipos, procedimientos y técnicas de adquisición de imágenes, dificultad de interpretación y calidad de la imagen. Otras organizaciones de normalización, como la DICOM (Digital Imaging and Communication in Medicine) y la Integrating the Healthcare Enterprise (IHE) participaron también en su elaboración22,23.
Para la utilización de estos léxicos se plantean iniciativas como la llevada a cabo por la RSNA y la European Society of Radiology (www.radreport.org), en la que se facilitan plantillas de informes estructurados específicas para diferentes modalidades de adquisición de imágenes y patologías. Estas plantillas están compuestas por una serie de campos delimitados por su tipología, los valores a introducir en él, si se trata de un campo opcional o no, y si depende del contenido de otro campo de la plantilla. Cada uno de estos datos tiene asociado además un término de los léxicos detallados anteriormente.
Para conseguir la interoperabilidad de estas plantillas con los sistemas de información hospitalarios, la IHE desarrolló, a través de su grupo de trabajo radiológico Management of Radiology Report Templates (MRRT), un estándar para la presentación de informes estructurados, nuestro V324. En él se especifica qué tecnología debe utilizarse para la elaboración de las plantillas, y se describen cómo debe ser la administración de estas plantillas y su integración y migración en los sistemas de información radiológicos (RIS). Por el contrario, el MRRT no define explícitamente cómo se transmiten estos informes desde un sistema de información radiológico a un sistema de registro de salud electrónico.
Por todo ello, el propio estándar DICOM se presenta como la tecnología para el almacenamiento de los informes estructurados. DICOM-SR permite la integración de los léxicos más importantes en la imagen médica, pudiendo además añadir léxicos propios. Este estándar establece las pautas de preparación del documento estructurado DICOM y su codificación estándar de la información en formato DICOM.
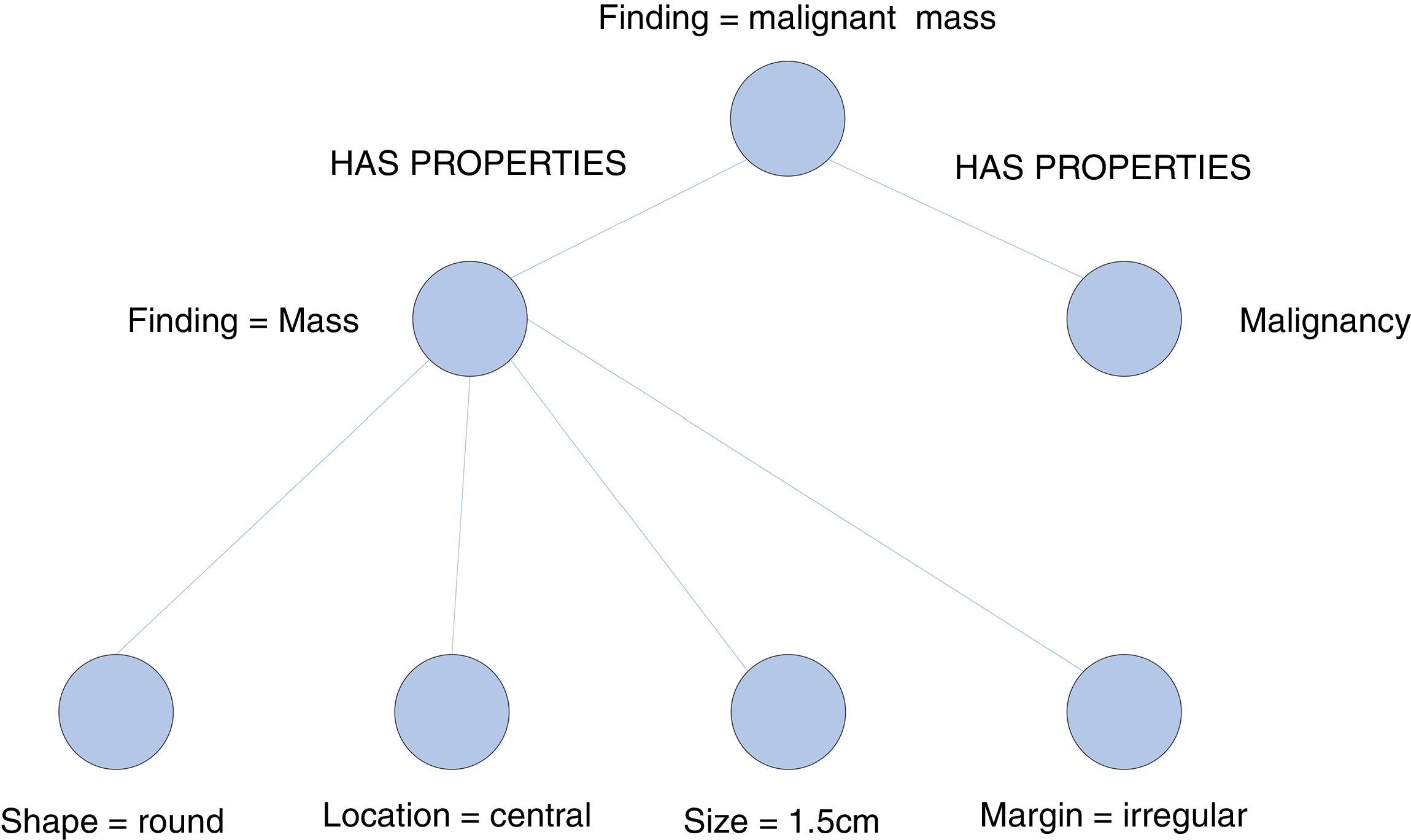
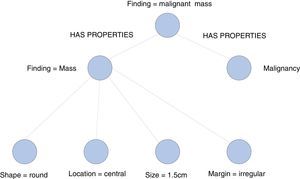
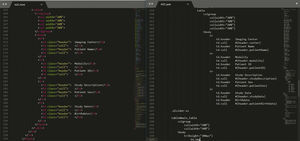
Los objetos DICOM-SR presentan una estructura de árbol, que representa la información de informe como Data Elements. Esta estructura, conocida como SR Document Content, contiene la definición del informe, donde cada nodo mantiene una relación con su nodo padre, con un tipo definido para el valor que almacena. En la figura 2 se muestra un ejemplo de DICOM-SR que describe el hallazgo de un tumor maligno.
DICOM-SR permite estructurar el contenido semántico de los datos clínicos a través de la inclusión del estándar HL7, utilizando la gestión de documentos denominada CDA23,24. Todo el contenido de un CDA se define mediante la codificación de la información utilizando el estándar XML. Sin embargo, DICOM-SR tiene un enfoque complementario para los informes de imagen médica. Con la inclusión de HL7, DICOM-SR permite al facultativo enriquecer el informe con la información clínica relevante para su diagnóstico a través de las imágenes obtenidas y sus datos asociados25,26.
DICOM-SR permite también la inclusión en el informe de las medidas tomadas por el facultativo sobre las imágenes, utilizando para ello las herramientas de medida del visor del PACS. Estas anotaciones y mediciones proporcionan una información crítica que sustenta con frecuencia las observaciones cualitativas reflejadas en el informe. El propósito principal de integrar estas mediciones en DICOM-SR es evitar expresiones propias de un informe no estructurado, tales como «masa de 5cm que se observa mejor en la imagen 62»27.
Implantación del informe estructurado en la prácticaEl Servicio de Diagnóstico e Intervencionismo Radiológico del Hospital Universitario de Gutenberg, de la universidad de Mainz, Alemania, desarrolló una herramienta Web con el objetivo de integrar el informe estructurado en la práctica clínica de los servicios de radiología28. Esta herramienta está conectada con los sistemas de información hospitalarios, permitiendo añadir automáticamente desde la propia aplicación Web nuevas plantillas, escritas en HTML5 y JavaScript. Las características fundamentales de la aplicación son:
- •
Conexión con RIS y PACS para la obtención de estudios.
- •
Listado de estudios a informar.
- •
Gestión de usuarios, perfiles de usuarios y privilegios de cada perfil.
- •
Inclusión de plantillas personalizadas para la realización del informe estructurado (HTML5 + JavaScript).
- •
Búsqueda de informes.
- •
Validación de informes una vez terminados (esta característica es importante en el caso de que informen residentes).
- •
Generación del informe en diferentes formatos: HTML, XML, PDF, DICOM-PDF, DICOM-SR y HL7 CDA.
- •
Envío del informe al PACS (DICOM-PDF o DICOM-SR) y al RIS (HL7 CDA).
- •
Gráficas interactivas que muestran los valores introducidos por los radiólogos en los informes.
- •
Exportación de los datos introducidos por los radiólogos en un fichero CSV.
Otra particularidad es su universalidad, ya que es posible la importación de cualquier modelo compatible con MRRT, permitiendo a los servicios de radiología elegir o crear las plantillas que deseen utilizar para cada pregunta clínica para la que se desarrolla un informe específico. Esta característica supone un incentivo para su uso, al ser totalmente personalizable. Además, la capacidad de exportar los datos introducidos en los informes de forma estructurada posibilita el cálculo de parámetros epidemiológicos, como por ejemplo la incidencia de una determinada lesión o hallazgo en un momento dado. También posibilita el análisis para determinar un patrón o tendencia en los resultados del paciente para una determinada condición.
Esta aplicación se ha puesto en funcionamiento en el Hospital Universitari i Politècnic La Fe de Valencia, diseñándose inicialmente como prueba piloto las plantillas de informe radiológico para cáncer de hígado, cáncer de recto y cáncer de pulmón. En todas las plantillas se sigue el mismo esquema general (fig. 3):
- •
Información clínica que justifica la prueba.
- •
Técnica de adquisición con los parámetros técnicos pertinentes.
- •
Informe con los hallazgos y posibles categorías de respuestas.
- •
Conclusiones con estadificación.
- •
Resumen exportable.
El recurso «Resumen» genera automáticamente el informe finalizado por el facultativo en formato lenguaje natural para facilitar la comunicación con el médico prescriptor y el paciente. Su contenido simula el texto de un informe radiológico predefinido más tradicional, pero siempre con la misma estructura y con todos los aspectos relevantes del informe estructurado generado. Este texto será el que visualizará el facultativo peticionario de la prueba. Además, en las plantillas Web se han añadido elementos de apoyo o ayuda para la realización del informe, sea como texto descriptivo, como imágenes estáticas o como diagramas interactivos.
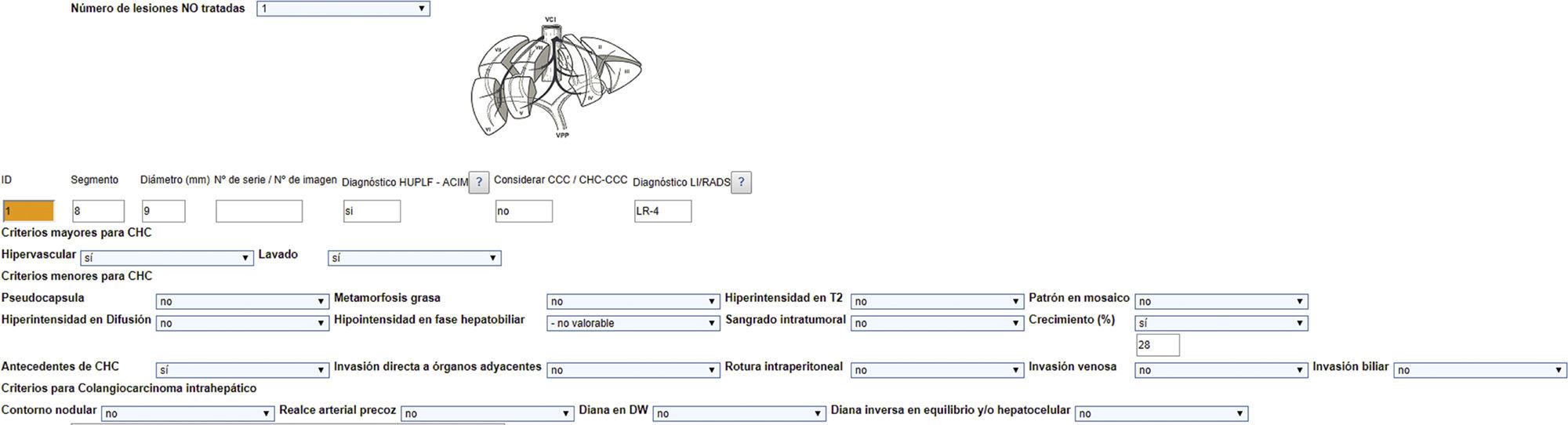
Cuando se informa un caso, la plantilla muestra u oculta campos al facultativo dependiendo de las respuestas que este vaya realizando. Los informes permiten también el cálculo automático de parámetros, como la estadificación y la gradación de la respuesta al tratamiento, según la información introducida por el radiólogo. Así, por ejemplo, en el informe estructurado de cáncer de hígado se obtienen de manera automática, y para cada una de las nuevas lesiones, la probabilidad de hepatocarcinoma, la categoría LI-RADS, el diagnóstico según los criterios propios ACIM-HUPLF29 y la posibilidad de que se trate de un colangiocarcinoma intrahepático o un hepatocolangiocarcinoma (ICC, CHC-CCC). Todos estos clasificadores emplean los criterios mayores y auxiliares para las lesiones y la consideración de pseudolesiones (fig. 4).
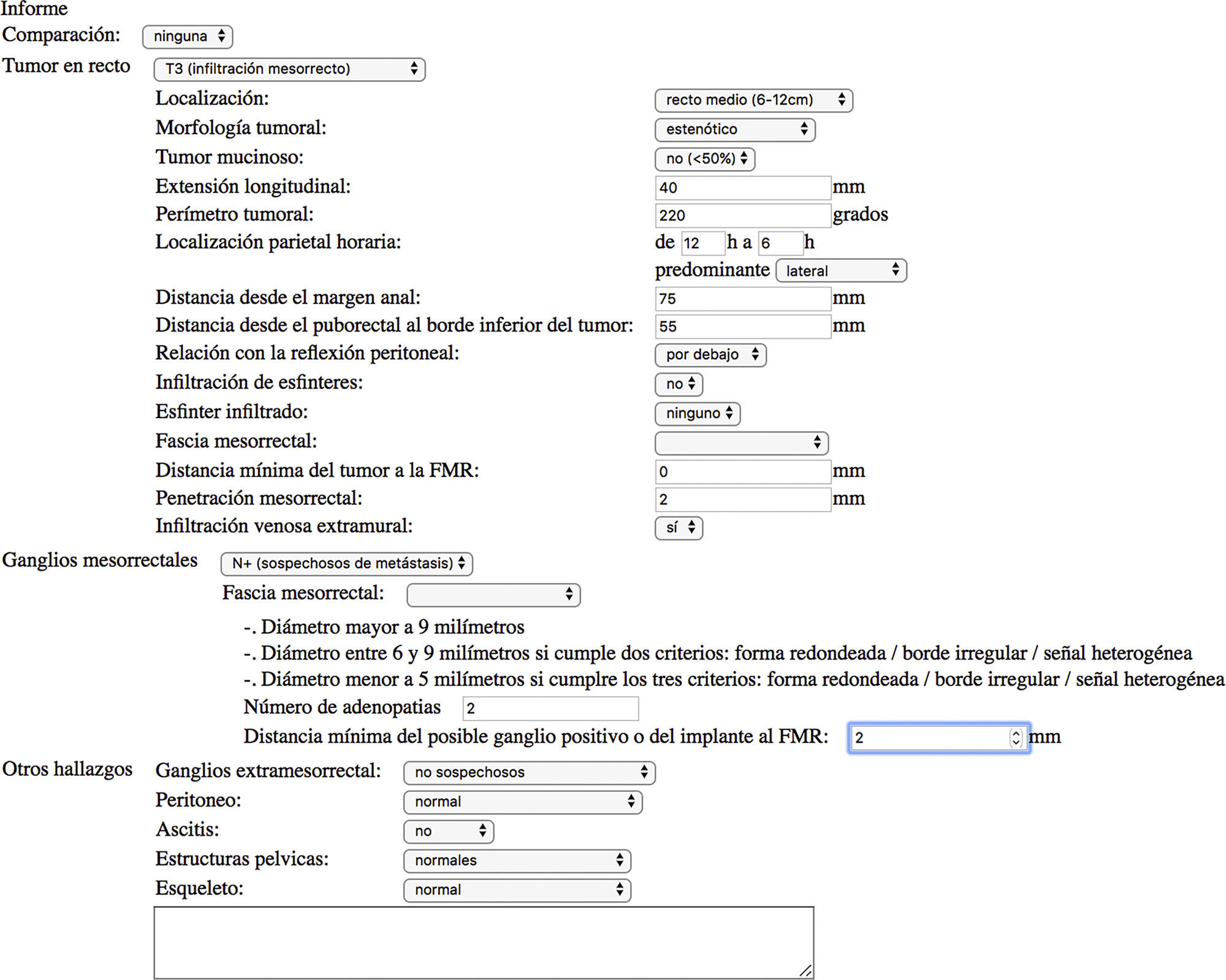
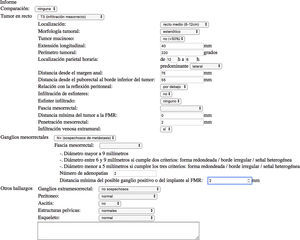
De igual modo, en el informe estructurado de cáncer de recto y en el informe de cáncer de pulmón se obtiene la categoría TNM de forma automática basándose en las características señaladas por el facultativo en los distintos campos del informe (fig. 5).
Informe estructurado con información cuantitativaEn la actualidad se genera una gran cantidad de resultados cuantitativos mediante la aplicación de algoritmos computacionales a partir del análisis de las imágenes médicas. Entendemos como imagen médica computacional a la ciencia que estudia el análisis digital de los datos de las imágenes adquiridas, las propiedades y el comportamiento de los tejidos, en un intento de describir con exactitud y veracidad los fenómenos relevantes para una enfermedad. Es la intención de esta imagen computacional evaluar la predisposición para padecer una enfermedad, detectar de manera precoz las lesiones cuando no se han expresado clínicamente, establecer un diagnóstico preciso de la naturaleza y la agresividad de las lesiones existentes, evaluar su extensión local y su capacidad de invasión a distancia, conocer su probable respuesta a las diferentes terapias y el pronóstico del paciente, y valorar el efecto del tratamiento y el estado de las lesiones en el tiempo de seguimiento.
Para alcanzar estos hitos se extraen características y parámetros de las lesiones y los tejidos. Estas características, conocidas como radiómica, dan información del dominio de la señal (histograma, distribución espacial, descriptores geométricos) y del modelado biológico (ajustes dinámicos). Con ello se obtiene información de, por ejemplo, la heterogeneidad, el espesor, la forma, la celularidad y la angiogénesis de una lesión tumoral. Estos datos objetivos se definen como biomarcadores cuando se ha demostrado su reproducibilidad y su relación subrogada con procesos biológicos normales, cambios patológicos o respuestas a una terapia. Además, estos biomarcadores de imagen extraídos proporcionan una información cuantitativa de la distribución regional y de la magnitud del cambio mediante las imágenes paramétricas, donde la información está resuelta en el espacio (por la distribución del parámetro) y en el tiempo (por el estudio de los cambios longitudinales)30.
La radiómica permite además el análisis multivariante y la extracción de clases tisulares características, incluyendo los datos obtenidos por técnicas de inteligencia artificial, como las redes neuronales convolucionales y el aprendizaje profundo.
Sin embargo, aunque estos datos radiómicos y sus biomarcadores de imagen estuvieran disponibles en un entorno de investigación e innovación, los sistemas de información radiológica existentes en la práctica clínica no permiten el manejo de esta información cuantitativa desde la perspectiva de su explotación de datos, divulgación del conocimiento relevante validado y aplicación a una mejora en los procesos de diagnóstico, valoración pronóstica y evaluación de la respuesta al tratamiento. El resultado final del análisis radiómico debe poder ser visualizado y revisado por los radiólogos y los médicos prescriptores en el informe estructurado empleando los sistemas de información disponibles30,31. Así pues, es necesario integrar los biomarcadores de imagen, como datos objetivos relacionados con una propiedad o característica biológica, en el informe radiológico y en los sistemas de información hospitalarios, tales como el PACS, el RIS y la historia clínica electrónica.
Aunque una solución para la gestión integrada de estos resultados es el informe estructurado DICOM-SR, los sistemas PACS y RIS actuales no integran visores DICOM-SR que permitan explotar la información adicional agregada en las cabeceras y que contienen los resultados cuantitativos. Por lo tanto, no podría realizarse una minería de datos a los biomarcadores de imagen obtenidos actualmente en un grupo de pacientes. Una posible solución para este problema es la utilización de una arquitectura software complementaria al PACS y desarrollada específicamente para el posprocesado de imagen, la generación de informes estructurados cuantitativos y el almacenamiento de los biomarcadores para así poder explotarlos y analizarlos estadísticamente en un momento posterior.
El módulo de generación de informes estructurados cuantitativos (qSR) debe incorporar plantillas con campos desplegables para los informes que puedan finalmente generarse en diferentes formatos, como HTML, Jade, JasperReports o Crystal Reports, y así permitir crear informes flexibles31–34. Estos formatos permiten personalizar la información e incorporar datos de entrada con los biomarcadores de imagen extraídos de un estudio. Los tipos de entrada con los que es posible alimentar estas plantillas para la generación del informe específico del paciente son ficheros CSV, ficheros XML, ficheros JSON y bases de datos tanto SQL (MySQL, Oracle, PostgreSQL) como no-SQL (Mongo DB). El informe final generado, que contiene los resultados de los biomarcadores de imagen, puede leerse y almacenarse en el PACS como un archivo DICOM-SC (captura secundaria) o DICOM-SR (informe estructurado) asociado al estudio del paciente. Si se ha realizado el proceso de extracción radiómica de los biomarcadores relevantes con anterioridad, el radiólogo podrá disponer de los datos cuantitativos durante su lectura de las imágenes en el mismo entorno de trabajo31.
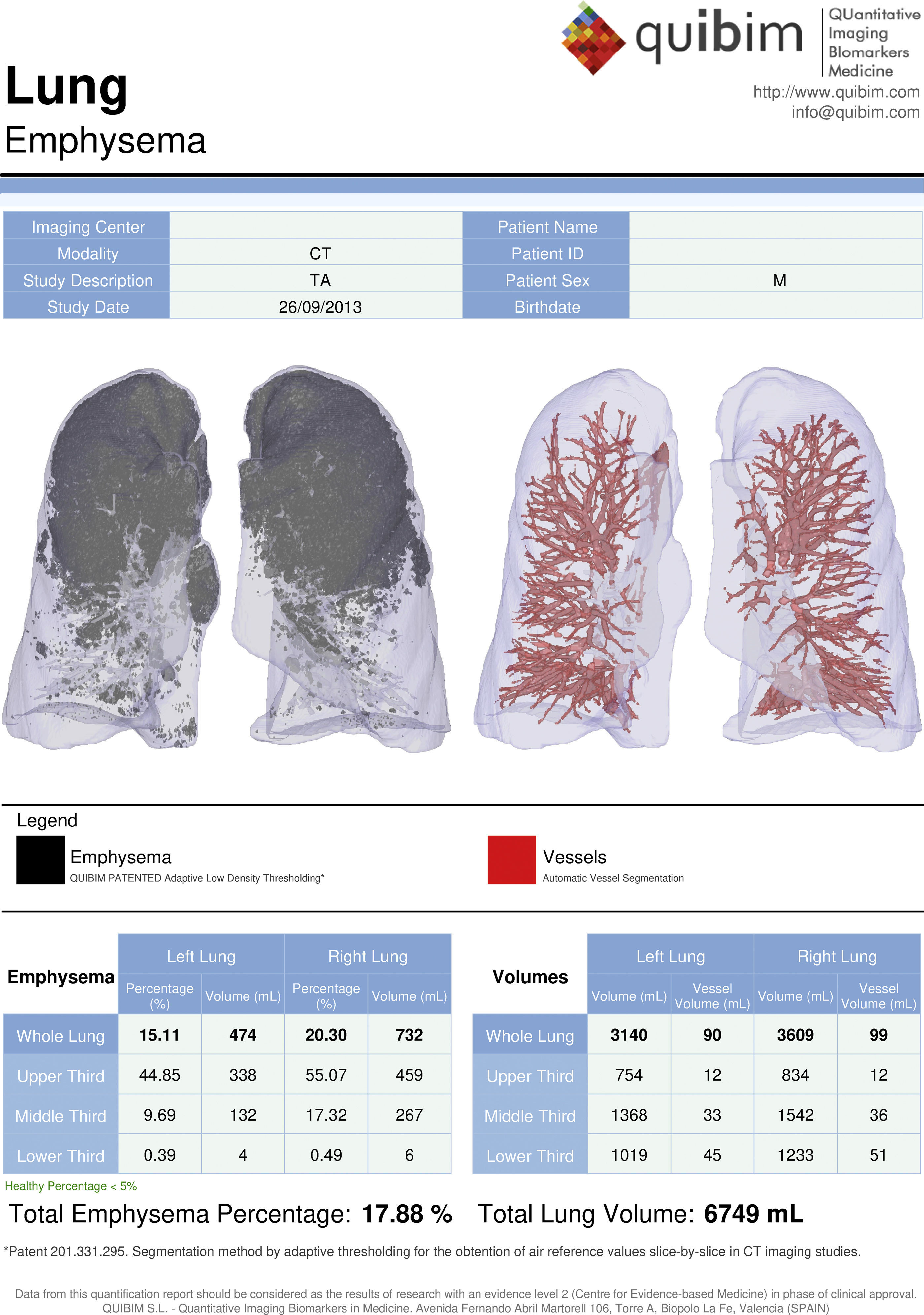
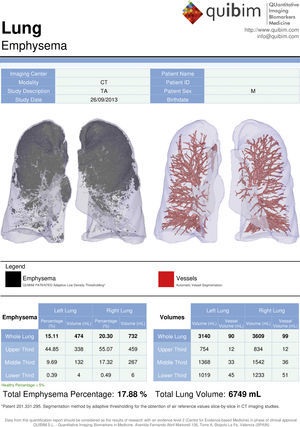
El primer paso para la generación de este informe cuantitativo estructurado qSR (fig. 6) es establecer un grupo de trabajo con los principales actores involucrados, tales como radiólogos, clínicos prescriptores e ingenieros biomédicos, para así determinar el contenido de los campos del informe y la mejor metodología para su implementación. Es importante incluir datos del paciente, fecha de procesamiento, modalidad de adquisición de imágenes, imágenes paramétricas significativas, valores resultantes de los biomarcadores de imagen analizados y valores de normalidad ajustados, si fuera necesario, por sexo y edad.
Dado que los biomarcadores de imágenes pueden generar una gran cantidad de resultados, es necesario decidir qué imágenes y parámetros se incluirán en el informe. Este paso es de especial relevancia, ya que los biomarcadores de imagen elegidos deben aportar valor al radiólogo en el diagnóstico y al clínico en la decisión de la mejor estrategia terapéutica o de seguimiento.
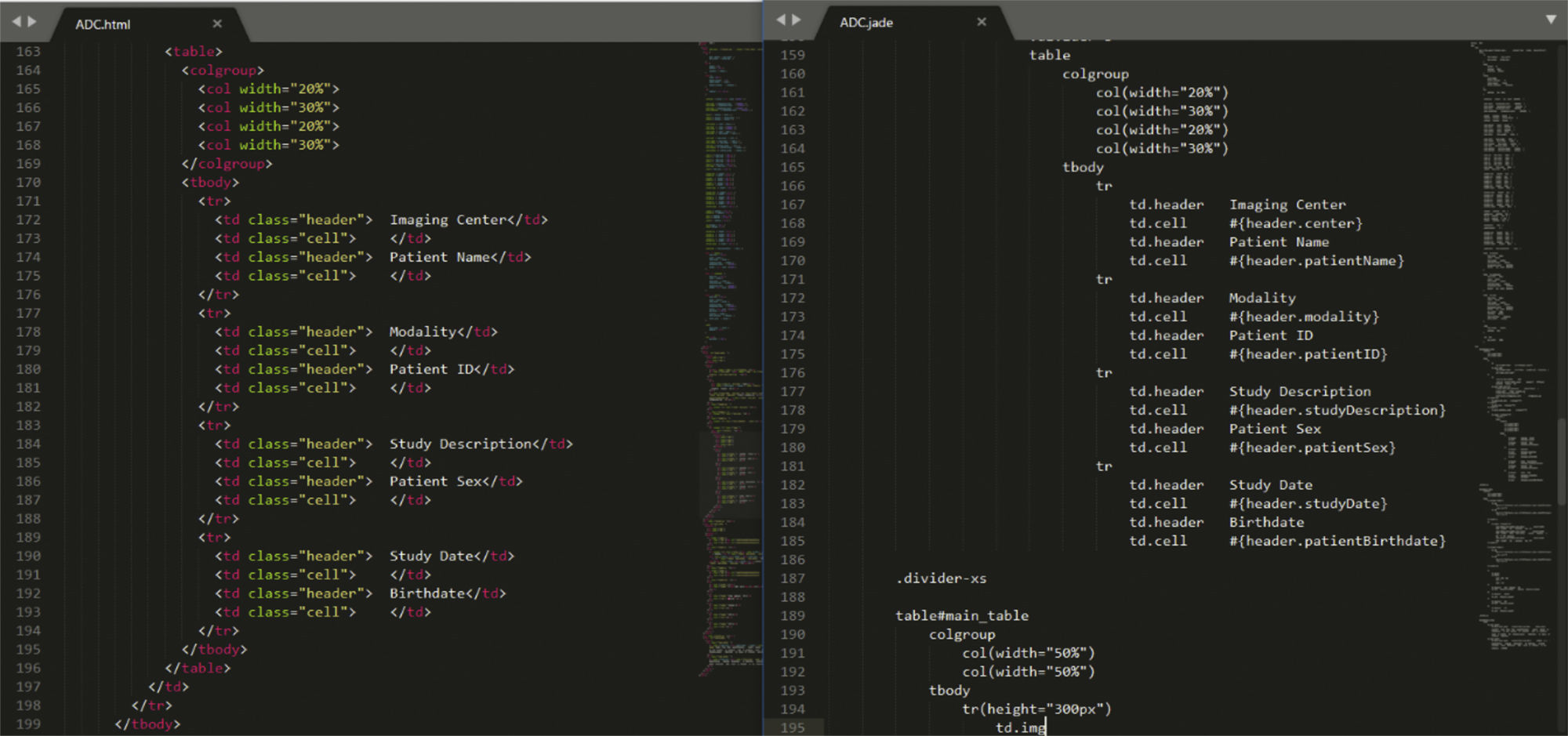
A continuación, debe crearse un borrador tipo mock-up del informe y validarse por todas las partes implicadas. Tras la validación del borrador definitivo hay que elegir una tecnología para llevar a cabo la implementación. La propuesta de los autores es la utilización de JADE, un motor de generación de plantillas en lenguaje HTML que permite optimizar el proceso de creación, evitando dedicar tiempo a cumplir con las reglas de estructura de los ficheros HTML (fig. 7).
La aceptación de estos informes qSR es alta en un entorno hospitalario complejo con importante actividad clínica de referencia. En la actualidad ya están implementados en nuestro hospital los informes de hepatocarcinoma, cáncer de colon, cáncer de próstata, cáncer de pulmón, esclerosis lateral amiotrófica, esclerosis múltiple y código ictus.
En conclusión, los datos digitales estructurados (imágenes, texto, mediciones, radiómica, biomarcadores de imagen) deben integrarse en un informe informatizado que permita su indexación en grandes repositorios. Estos bancos de datos radiológicos son fundamentales en la medicina personalizada para explotar la información sanitaria, fenotipificar las lesiones y enfermedades, y extraer conclusiones. Estos biobancos y sus biomarcadores de imagen asociados son nuestra mejor aproximación a la medicina de precisión y los big data. Los radiólogos debemos implementar en nuestro quehacer diario el informe estructurado, mejorando su completitud (con plantillas de referencia), validez (sin términos erróneos), efectividad (con éxito en la comunicación de datos relevantes) y disponibilidad de datos radiómicos (registro de biomarcadores), de forma global en todas las técnicas de imagen y enfermedades.
Autoría- 1.
Responsable de la integridad del estudio: LMB, AAB y ERM.
- 2.
Concepción del estudio: LMB y ERM.
- 3.
Diseño del estudio: LMB, ERM, AT y AAB.
- 4.
Obtención de los datos: N/A
- 5.
Análisis e interpretación de los datos: N/A
- 6.
Tratamiento estadístico: N/A
- 7.
Búsqueda bibliográfica: LMB, ERM, AT y AAB.
- 8.
Redacción del trabajo: LMB, ERM, AT y AAB.
- 9.
Revisión crítica del manuscrito con aportaciones intelectualmente relevantes: LMB, ERM, AT y AAB
- 10.
Aprobación de la versión final: LMB, ERM, AT y AAB.
LMB y AAB son socios fundadores de Quibim, S.L. Ninguno de los autores ha recibido financiación relacionada con este trabajo.