Se analizaron 200 aislamientos de Acinetobacter correspondientes a igual cantidad de pacientes atendidos en el Hospital de Clínicas José de San Martín entre marzo de 2013 y junio de 2014. La identificación se realizó mediante espectrometría de masa y se confirmó con métodos moleculares. La sensibilidad a los antimicrobianos se determinó mediante el sistema Vitek-2. La correlación entre la identificación obtenida con la espectrometría de masa y las técnicas moleculares fue del 94 %. Acinetobacter baumannii multirresistente fue la genoespecie predominante (92,6 %) en la infección intrahospitalaria, y la frecuencia de aislamiento de Acinetobacter pitti y de Acinetobacter nosocomialis fue de 3,5 % y 0,5 %, respectivamente. En la infección extrahospitalaria se observó una mayor presencia de otras genoespecies. Acinetobacter johnsonii y A. baumannii fueron las más frecuentes y juntas representaron el 45,9 % de los hallazgos. La resistencia a carbapenems y a minociclina solo se observó en A. baumannii. La espectrometría de masa resultó ser una herramienta útil en la identificación de las diferentes genoespecies.
Two-hundred Acinetobacter isolates belonging to 200 patients admitted to Hospital de Clínicas José de San Martín during the period March 2013-June 2014 were analyzed. The identification was performed by mass spectrometry and was confirmed by molecular methods. Susceptibility to antimicrobials was studied by the Vitek-2 system. A 94% correlation of both identification methods was found. Multidrug resistant Acinetobacter baumannii was the predominant genomic species (92.6%) in hospital-acquired infections, whereas Acinetobacter pitti and Acinetobacter nosocomialis accounted for 3.5% and 0.5% of the isolates recovered, respectively. In community-acquired infections a major predominance of the different genomic species was observed. Acinetobacter johnsonii and A. baumannii are the most frequent species, accounting for 45.9% of the isolates recovered. Resistance to carbapenems and minocycline was only observed in A. baumannii. Mass spectrophotometry was an effective tool for the identification of the different genomic species.
El género Acinetobacter spp. está constituido por 33 genoespecies. La imposibilidad de distinguirlas mediante pruebas bioquímicas ha dificultado el estudio de la epidemiología del género10.
La utilización de técnicas moleculares capaces de identificar las genoespecies de Acinetobacter ha permitido evidenciar grandes diferencias en la incidencia de estas, así como en sus perfiles de resistencia a los antimicrobianos1,4,12. Asimismo, la mayor disponibilidad de equipos de espectrometría de masa posibilitaría a los laboratorios de microbiología clínica de mediana complejidad identificar dichas genoespecies.
Si bien Acinetobacter baumannii es la genoespecie aislada con mayor frecuencia, asociada en forma predominante a infecciones hospitalarias, recientemente se han publicado varios trabajos en diferentes países en los cuales Acinetobacter nosocomialis y Acinetobacter pitti están presentes en altos porcentajes, desplazando incluso a A. baumannii3,5,7,13.
La multirresistencia es característica de A. baumannii, en este microorganismo son las carbapenemasas de clase D, fundamentalmente OXA-23, las responsables de la resistencia a carbapenems en las cepas endémicas10. Sin embargo, en genoespecies diferentes a A. baumannii se han descrito otras carbapenemasas de manera esporádica en nuestro país y frecuentemente en otras regiones11,13.
El objetivo de este trabajo fue estudiar en forma prospectiva la frecuencia de aislamiento de Acinetobacter spp., las características clínicas de los afectados y la resistencia a los antimicrobianos de los aislamientos recuperados de pacientes atendidos en un hospital universitario de la ciudad de Buenos Aires, incluyendo internados y ambulatorios. También se quiso evaluar la capacidad de la espectrometría de masa comparada con las técnicas de biología molecular para identificar genoespecies de Acinetobacter spp.
Se estudiaron en forma prospectiva todos los aislamientos de Acinetobacter spp. recuperados de pacientes internados o atendidos por consultorio externo en el Hospital de Clínicas “José de San Martín”, Universidad de Buenos Aires, entre marzo de 2013 y junio de 2014.
Los aislamientos fueron identificados por características culturales y mediante la espectrometría de masa con un equipo MALDI-TOF, Bruker, Daltonics. Los resultados fueron analizados utilizando la base de datos de Biotyper (versión 3.1, BD, Bruker Daltonik, Bremen, Alemania). Se seleccionó un 50 % representativo de las diferentes genoespecies para corroborar la identificación de la espectrometría de masa con métodos moleculares6,14.. La técnica de rpoB fue utilizada en 27 casos, A. baumannii 5/159, A. johnsonnii 3/9, A. pitti 5/7, Acinetobacter lwoffii 3/7, Acinetobacter junii 2/5, Acinetobacter ursingii 2/4, Acinetobacter guillouiae 2/3, Acinetobacter baylyi 1/2 y 1/1 en Acinetobacter haemolyticus, Acinetobacter 15TU, A. nosocomialis y Acinetobacter gyllenbergii. La presencia de blaOXA-51-like fue investigada en 98 aislamientos, 80/159 de A. baumannii y en 18 aislamientos de otras genoespecies. La técnica de rpoB siempre se consideró definitoria, y la detección de blaOXA-51-like se consideró definitoria cuando fue posterior a la identificación de la genoespecie A. baumannii por MALDI-TOF. La sensibilidad a los antimicrobianos fue determinada mediante el equipo VITEK-2 y la actividad de minociclina fue determinada mediante dilución en agar. Se utilizaron las recomendaciones del Clinical and Laboratory Standards Institute (CLSI) en la interpretación de los resultados2.
La presencia de blaOXA-51, blaOXA-23, blaOXA-58 y blaOXA-24/40 se investigó mediante cebadores específicos de acuerdo con lo publicado por Woodford et al.14.
Para cada paciente se elaboró una ficha clínica en donde se incluyeron los siguientes datos: espécimen clínico, tratamiento previo con carbapenems, fecha y servicio de internación (si correspondiera); enfermedad de base o diagnóstico.
El porcentaje de identificaciones correctas, de acuerdo con las consideraciones antes explicitadas, fue del 94 %. Solo se detectó blaOXA-51 en A. baumannii. Se observó falta de correlación entre los resultados obtenidos por espectrometría de masa y por detección de rpoB o blaOXA-51-like genes (o ambos) en 4 aislamientos de A. baumannii, 1 de A. gyllenbergii y 1 de Acinetobacter 15TU, los cuales fueron identificados por MALDI-TOF como A. nosocomialis, Acinetobacter calcoaceticus y A. baumannii, respectivamente.
La frecuencia de aislamiento de A. baumannii fue de 79,5 %, mientras que la de A. pitti y de A. nosocomialis fue de 3,5 % y 0,5 %, respectivamente. El 81,5 % de los aislamientos correspondieron a infecciones intrahospitalarias (IH), entre estos, el 92,6 % se identificó como A. baumannii.
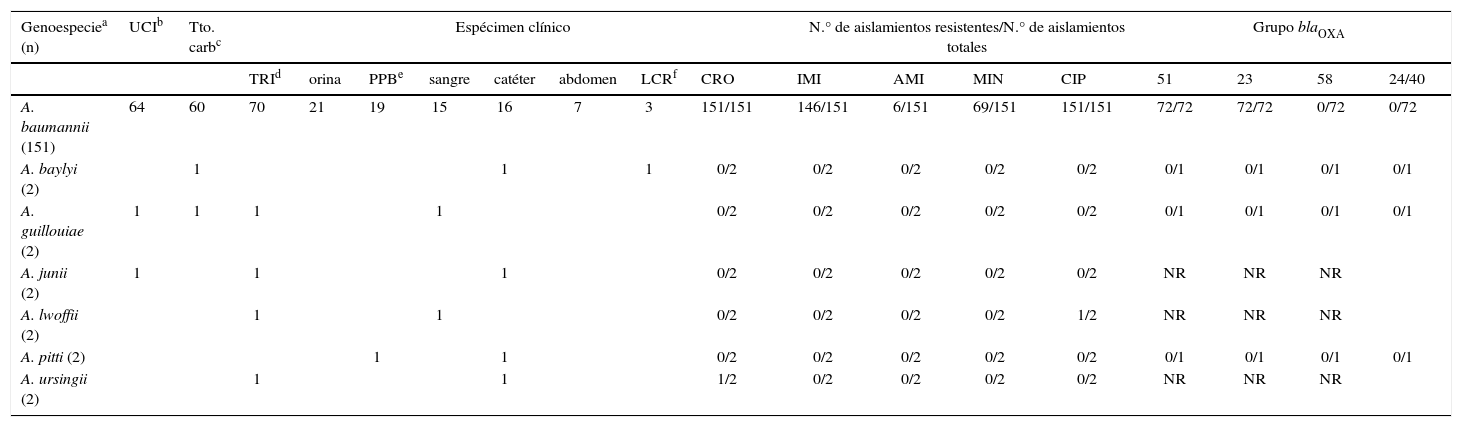
La distribución de las diferentes genoespecies entre los especímenes clínicos, el perfil de resistencia a los antimicrobianos y la presencia de carbapenemasas se detallan en las tablas 1 y 2. En las infecciones IH, la infección respiratoria baja fue el principal diagnóstico (45,4 %), seguido de la infección de herida quirúrgica (12,3 %). La infección urinaria predominó entre los pacientes no internados. El 92 % de las infecciones respiratorias bajas fueron producidas por A. baumannii. No se observó ninguna otra asociación entre genoespecie y especímenes clínicos, aunque el bajo número de aislamientos impidió el tratamiento estadístico de los datos. Se hallaron aislamientos con resistencia a carbapenems y a minociclina solamente en A. baumannii.
Características microbiológicas y resistencia a los antimicrobianos de las genoespecies de Acinetobacter recuperadas de infecciones intrahospitalarias
| Genoespeciea (n) | UCIb | Tto. carbc | Espécimen clínico | N.° de aislamientos resistentes/N.° de aislamientos totales | Grupo blaOXA | |||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| TRId | orina | PPBe | sangre | catéter | abdomen | LCRf | CRO | IMI | AMI | MIN | CIP | 51 | 23 | 58 | 24/40 | |||
| A. baumannii (151) | 64 | 60 | 70 | 21 | 19 | 15 | 16 | 7 | 3 | 151/151 | 146/151 | 6/151 | 69/151 | 151/151 | 72/72 | 72/72 | 0/72 | 0/72 |
| A. baylyi (2) | 1 | 1 | 1 | 0/2 | 0/2 | 0/2 | 0/2 | 0/2 | 0/1 | 0/1 | 0/1 | 0/1 | ||||||
| A. guillouiae (2) | 1 | 1 | 1 | 1 | 0/2 | 0/2 | 0/2 | 0/2 | 0/2 | 0/1 | 0/1 | 0/1 | 0/1 | |||||
| A. junii (2) | 1 | 1 | 1 | 0/2 | 0/2 | 0/2 | 0/2 | 0/2 | NR | NR | NR | |||||||
| A. lwoffii (2) | 1 | 1 | 0/2 | 0/2 | 0/2 | 0/2 | 1/2 | NR | NR | NR | ||||||||
| A. pitti (2) | 1 | 1 | 0/2 | 0/2 | 0/2 | 0/2 | 0/2 | 0/1 | 0/1 | 0/1 | 0/1 | |||||||
| A. ursingii (2) | 1 | 1 | 1/2 | 0/2 | 0/2 | 0/2 | 0/2 | NR | NR | NR | ||||||||
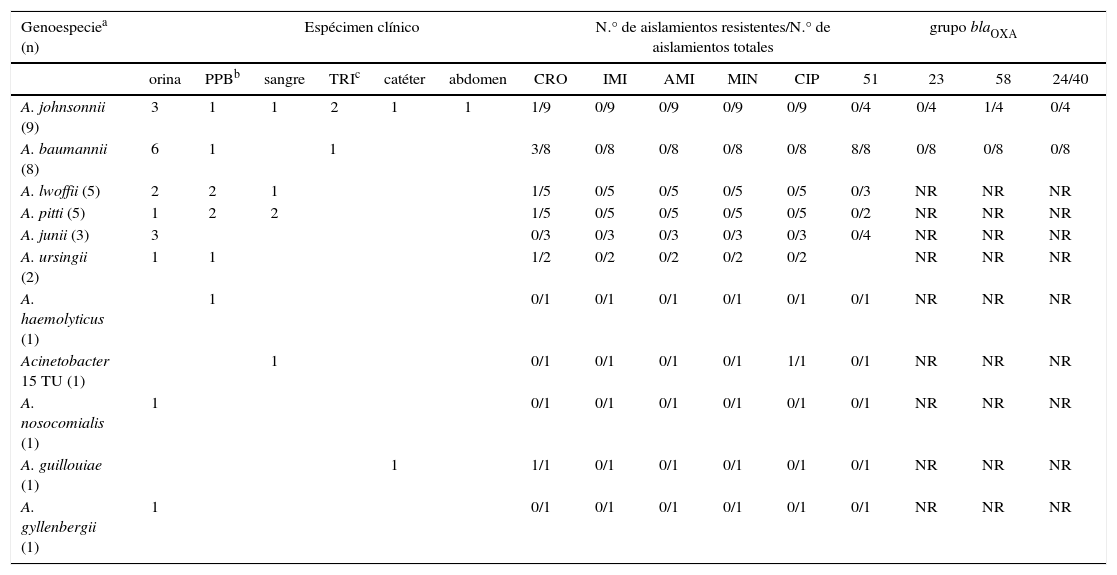
Características microbiológicas y resistencia a los antimicrobianos de las genoespecies de Acinetobacter recuperadas de infecciones extrahospitalarias
| Genoespeciea (n) | Espécimen clínico | N.° de aislamientos resistentes/N.° de aislamientos totales | grupo blaOXA | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| orina | PPBb | sangre | TRIc | catéter | abdomen | CRO | IMI | AMI | MIN | CIP | 51 | 23 | 58 | 24/40 | |
| A. johnsonnii (9) | 3 | 1 | 1 | 2 | 1 | 1 | 1/9 | 0/9 | 0/9 | 0/9 | 0/9 | 0/4 | 0/4 | 1/4 | 0/4 |
| A. baumannii (8) | 6 | 1 | 1 | 3/8 | 0/8 | 0/8 | 0/8 | 0/8 | 8/8 | 0/8 | 0/8 | 0/8 | |||
| A. lwoffii (5) | 2 | 2 | 1 | 1/5 | 0/5 | 0/5 | 0/5 | 0/5 | 0/3 | NR | NR | NR | |||
| A. pitti (5) | 1 | 2 | 2 | 1/5 | 0/5 | 0/5 | 0/5 | 0/5 | 0/2 | NR | NR | NR | |||
| A. junii (3) | 3 | 0/3 | 0/3 | 0/3 | 0/3 | 0/3 | 0/4 | NR | NR | NR | |||||
| A. ursingii (2) | 1 | 1 | 1/2 | 0/2 | 0/2 | 0/2 | 0/2 | NR | NR | NR | |||||
| A. haemolyticus (1) | 1 | 0/1 | 0/1 | 0/1 | 0/1 | 0/1 | 0/1 | NR | NR | NR | |||||
| Acinetobacter 15 TU (1) | 1 | 0/1 | 0/1 | 0/1 | 0/1 | 1/1 | 0/1 | NR | NR | NR | |||||
| A. nosocomialis (1) | 1 | 0/1 | 0/1 | 0/1 | 0/1 | 0/1 | 0/1 | NR | NR | NR | |||||
| A. guillouiae (1) | 1 | 1/1 | 0/1 | 0/1 | 0/1 | 0/1 | 0/1 | NR | NR | NR | |||||
| A. gyllenbergii (1) | 1 | 0/1 | 0/1 | 0/1 | 0/1 | 0/1 | 0/1 | NR | NR | NR | |||||
El género Acinetobacter ha sido objeto de varios trabajos en nuestro país, sin embargo, estos estuvieron centrados en A. baumannii9,11. La utilidad de la espectrometría de masa en la identificación de las diferentes genoespecies de Acinetobacter ha sido relativamente poco estudiada1,4,8. Los mayores problemas comunicados se vinculan con la composición de la base de datos y con la falta de especificidad de los picos en el espectro de masa obtenido, principalmente en el complejo Acinetobacter baumannii calcoaceticus1,4. En las genoespecies menos frecuentes, el bajo número de aislamientos analizados hace más difícil establecer las causas de los errores9. La principal dificultad encontrada en nuestro trabajo fue la identificación errónea de 4 aislamientos de A. baumannii como A. nosocomialis. El porcentaje de los resultados de correlatividad entre los métodos (94 %) fue similar a lo publicado previamente1,4,9.
La gran dispersión en cuanto a la incidencia de las diferentes genoespecies del género Acinetobacter al comparar los distintos estudios es llamativa. En el caso de A. baumannii, el rango comunicado es de 8,8 a 97,8 %, en el de A. nosocomialis, de 0 a 47 %, en A. pitti, de 1,7 a 19,5 %. Una situación similar se observa con las genoespecies relativamente infrecuentes3,5,7,13. Sin causas aparentes que justifiquen tal amplitud de datos, se podría correlacionar un mayor porcentaje de A. baumannii con tasas más altas de infecciones IH. Nuestros datos son comparables con lo publicado por Fernández-Cuenca et al., en un estudio multicéntrico realizado en España5.
La fuerte asociación de A. baumannii con la infección respiratoria baja IH en pacientes con tratamiento previo con carbapenems ya ha sido extensamente descrita10. Estos aislamientos se recuperaron en forma continua en todo el período estudiado (dato no mostrado), lo que evidencia la endemia de las infecciones IH por A. baumannii en nuestro hospital, principalmente en la unidad de cuidados intensivos, donde esta genoespecie representó el 96,9 % de los aislamientos de Acinetobacter spp. En cambio, los aislamientos pertenecientes a las otras genoespecies fueron siempre esporádicos.
La resistencia a carbapenems en A. baumannii estuvo relacionada con la presencia de bla-OXA-23 en el 100 % de los casos, lo que supera el dato comunicado por este mismo grupo hace 6 años. Este incremento en la presencia de OXA-23 ya fue descrito por otros autores en diferentes países10,11. La amicacina fue el único antimicrobiano respecto del cual se verificó un aumento en el porcentaje de aislamientos sensibles con respecto a un estudio previo: 96 % en la presente investigación versus 25 % de sensibilidad en la publicada en 200911. Dicha variación en la actividad reflejaría un cambio en los clones circulantes en el hospital.
En las infecciones extrahospitalarias (EH) se observa claramente una mayor frecuencia de aislamiento de otras genoespecies, las que resultaron ampliamente sensibles a los antimicrobianos ensayados. A. johnsonii y A. baumannii fueron las más frecuentes y juntas representaron el 45,9 % de los hallazgos. La infección más frecuente fue la urinaria (48,6 %) y estuvo asociada, en la mayoría de los casos, a la presencia de sonda urinaria.
Solo A. ursingii fue siempre resistente a las cefalosporinas, lo cual es coincidente con lo comunicado por otros autores12. A diferencia de lo que se informa en otros trabajos, no encontramos resistencia a los carbapenems en genoespecies diferentes de A. baumannii12, salvo en un aislamiento de A. johnsonii con sensibilidad disminuida a carbapenems (CIM = 2μg/ml), en el cual se detectó la presencia de blaOXA-58-like.
En conclusión, la espectrometría de masa resultó ser una herramienta útil en el estudio de las diferentes genoespecies de Acinetobacter, pero algunos resultados deben ser analizados con precaución, principalmente en el complejo Acinetobacter baumannii calcoaceticus. En nuestro estudio se ratifica la presencia endémica de A. baumannii en la infección IH, y no se observa por el momento una creciente relevancia de A. nosocomialis y A. pitti. En las infecciones EH se describe una mayor dispersión de genoespecies con la característica común de la amplia sensibilidad a los antimicrobianos.
Responsabilidades éticasProtección de personas y animales. Los autores declaran que para esta investigación no se han realizado experimentos en seres humanos ni en animales.
Confidencialidad de los datos. Los autores declaran que en este artículo no aparecen datos de pacientes.
Derecho a la privacidad y consentimiento informado. Los autores declaran que en este artículo no aparecen datos de pacientes.
FinanciaciónEste trabajo fue subsidiado mediante el Proyecto UBACYT 01/W296 de la Universidad de Buenos Aires.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.