El incremento en las últimas dos décadas en la incidencia de fungemias causadas por hongos levaduriformes en pacientes inmunodeprimidos susceptibles y la poca sensibilidad del cultivo de sangre convencional hacen necesario el desarrollo de enfoques alternativos para la detección temprana y la identificación de las especies responsables. El objetivo de este trabajo ha sido comparar la utilidad de la prueba molecular de la reacción en cadena de la polimerasa (PCR) y métodos convencionales para identificar aislamientos clínicos de diferentes especies, incluyendo el sistema ATB ID32C (bioMérièux, Francia), el cultivo cromogénico Chromagar Candida® (Chromagar, Francia) y la morfogénesis en agar harina de maíz. Se estudiaron 79 aislamientos clínicos en los cuales la especie más prevalente usando el sistema ATB ID32C y la PCR fue C. albicans, seguida por C. tropicalis, C. glabrata y C. krusei. Los patrones de PCR obtenidos para la identificación de aislamientos clínicos fueron estables y consistentes en los diferentes ensayos independientes y mostraron una buena reproducibilidad. Se concluye que la PCR con los cebadores específicos para cada especie, que amplifican los genes ITS1 e ITS2 del ARNr o del gen de la topoisomerasa ii, demostró ser un método sensible y específico para la identificación de los aislamientos de C. glabrata C. krusei, C. albicans y, con menor especificidad, para C. tropicalis.
The increase in the incidence of yeast species causing fungemia in susceptible immunocompromised patients in the last two decades and the low sensitivity of conventional blood culture has led to the need to develop alternative approaches for the early detection and identification of causative species. The aim of this study was to compare the usefulness of molecular testing by the polymerase chain reaction (PCR) and conventional methods to identify clinical isolates of different species, using the ID32C ATB system (bioMérieux, France), chromogenic culture Chromagar Candida® (CHROMagar, France) and morphogenesis in corn meal agar. We studied 79 isolates, in which the most prevalent species using the system ID32C and PCR was C. albicans, followed by C. tropicalis, C. glabrata and C .krusei. PCR patterns obtained for the identification of clinical isolates were stable and consistent in the various independent studies and showed good reproducibility, concluding that PCR with species-specific primers that amplify genes ITS1 and ITS2 for rRNA or topoisomerase ii primers is a very specific and sensitive method for the identification of C. glabrata, C. krusei, C. albicans, and with less specificity for C. tropicalis.
El notable incremento en las últimas dos décadas en la incidencia de fungemias causadas por especies levaduriformes en pacientes inmunodeprimidos susceptibles, y la poca sensibilidad del cultivo de sangre convencional para detectar el crecimiento levaduriforme ha hecho necesario el desarrollo de enfoques alternativos para la detección temprana y la identificación de las especies causantes más importantes, que incluyen a Candida albicans, Candida glabrata, Candida parapsilosis, Candida tropicalis y Candida krusei, las cuales suman entre el 80 y 90% de las causas de fungemias diagnosticadas3,19,36. De estas C. albicans ha sido reconocida como la principal y la más común de las especies relacionadas a las candidiasis, sin embargo, cada vez se observa más que otras especies menos comunes y más resistentes incrementan su prevalencia9. Candida spp. es usualmente susceptible a los antifúngicos de uso clínico, pero se han reportado especies con resistencia intrínseca a la anfotericina B (Candida lusitaniae) o al fluconazol, como C. krusei y C. glabrata36. Adicionalmente, la colonización de Candida en las mucosas orales o en otras partes del organismo se ha determinado como un factor importante para desarrollar infecciones profundas, sistémicas o diseminadas cuando las condiciones favorecen su diseminación34. Por lo anterior, un diagnóstico rápido y preciso de estas especies en las infecciones fúngicas invasivas es crítico para iniciar el tratamiento adecuado1,27.
En la actualidad se utilizan numerosos métodos para la identificación de los aislamientos clínicos, entre los que destacan el sistema API 20C o el sistema ATB ID32C, sin embargo estos requieren varios días para lograr dichas identificaciones11,31,37. Otros métodos propuestos incluyen el uso de medios diferenciales como el Chromagar Candida® que ha sido ampliamente validado; este contiene sustratos cromogénicos y provee una identificación presuntiva de manera rápida de las principales especies de importancia médica incluyendo C. albicans, C. tropicalis y C. krusei; sin embargo, hay que resaltar que diversos autores recomiendan practicar pruebas fenotípicas adicionales para la confirmación de estas especies29. Los métodos basados en biología molecular son excelentes herramientas para la identificación de especies de importancia médica y su uso ha sido propuesto desde la década de los 905. En la actualidad, debido a su gran sensibilidad y especificidad, el método molecular de la PCR (de sus siglas en inglés Polymerase Chain Reaction) está resultando exitoso18, algunos laboratorios lo han implementado para la tipificación de aislamientos de Candida9,35, señalándolo como una rápida y segura herramienta para la identificación de varios hongos patógenos, incluyendo diferentes especies de Candida, ya que además es capaz de identificar a aquellas especies que pudieran no ser observadas en el cultivo debido al sobrecrecimiento de una especie sobre otra36.
El objetivo de este trabajo fue comparar el método molecular de la PCR con los métodos convencionales usados ampliamente para la tipificación de aislamientos clínicos tales como cultivos clásicos, filamentación, el cultivo cromógeno Chromagar Candida® y el sistema basado en la asimilación de carbohidratos ATB ID32C de BioMerieux®.
Material y métodosAislamientos clínicosSe estudiaron aislamientos provenientes de la colonización o infección de sujetos de estudio que procedían de las clínicas de atención dental de la Universidad Autónoma de Ciudad Juárez (UACJ) y del hospital regional de zona no. 6 del Instituto Mexicano del Seguro Social (IMSS) en Ciudad Juárez, Chihuahua. En la UACJ se estudiaron sujetos adultos con factores predisponentes locales y sistémicos de padecer candidiasis, la mayoría de ellos pacientes portadores de prótesis parcial o total, personas adultas mayores y diabéticos e hipertensos que acuden a recibir atención dental en dichas clínicas. En el hospital del IMSS, que es un hospital público de tercer nivel de atención, se estudiaron sujetos adultos con factores predisponentes sistémicos principalmente diabéticos de la consulta externa, pacientes del área de cuidados intensivos u hospitalizados en las unidades de medicina interna, trasplantes o endocrinología. Se recolectaron muestras clínicas de la mucosa oral, de la superficie de materiales protésicos o catéteres y expectoraciones o fluidos de pacientes con sospecha de candidiasis sistémica o diseminada. Cada muestreo se codificó y se colocó en medio de Stuart o frascos estériles cerrados para su transporte al laboratorio, según el tipo de muestra.
Cultivo primario e identificación de especies por métodos convencionales y Chromagar Candida®Cada muestra de mucosa oral recolectada se sembró en placas de ChromAgar Candida® y se incubó a 36°C durante dos días; para descartarlos como negativos se mantuvieron incubados por un periodo adicional de siete días a una temperatura de 30±1°C. Con este cultivo cromogénico se hizo una identificación presuntiva de especies de Candida, de acuerdo a las características colorimétricas que indica el fabricante para cada especie; además, el uso de este medio permitió separar dos o más cepas juntas de diferentes especies en un mismo aislamiento. Para el cultivo primario e identificación primaria de muestras de fluidos o materiales protésicos se siguieron los métodos descritos en la guía práctica de Identificación y Diagnóstico en Micología Clínica dependiendo de la muestra a estudiar29. Los cultivos positivos a una o más especies se resembraron y purificaron en placas de agar glucosado de Sabouraud incubándolos a 36°C (± 1°C) por dos días y fueron confirmados microscópicamente. Posteriormente las especies levaduriformes fueron identificadas por medio del sistema de asimilación de carbohidratos ID32C AUX y la base de datos de APIWeb (BioMerieux, Lyon, Francia). Para las pruebas de filamentación cada aislamiento se sembró en placas de agar harina de maíz incubándolas a 36°C (± 1°C) durante 7 días y fue monitoreado el desarrollo de sus colonias mediante microscopia invertida. En nuestros ensayos se incluyeron cepas de referencia obtenidas de la American Type Culture Collection (ATCC) y los aislamientos ya fenotipificados fueron almacenados en agua estéril a temperatura ambiente para su uso posterior.
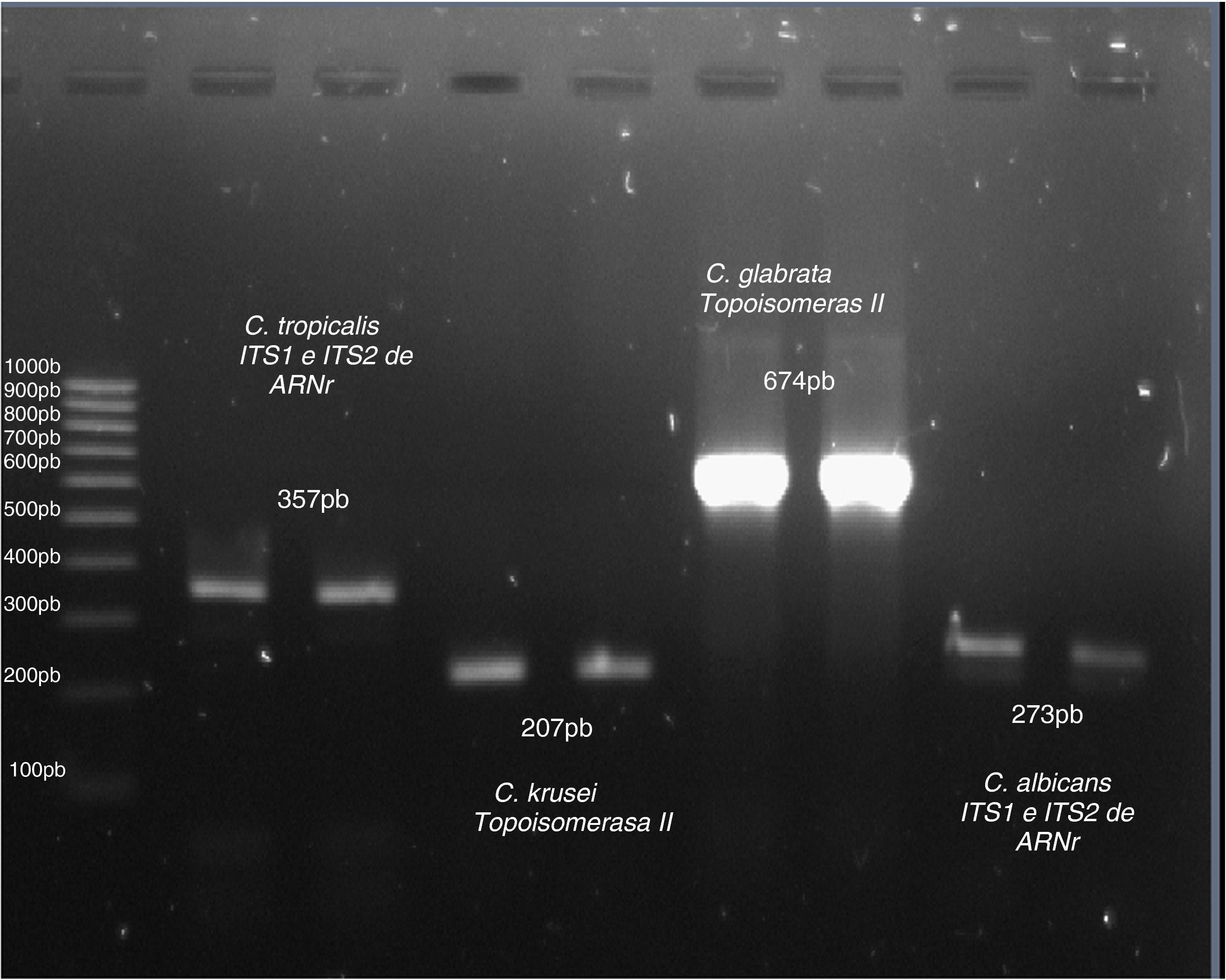
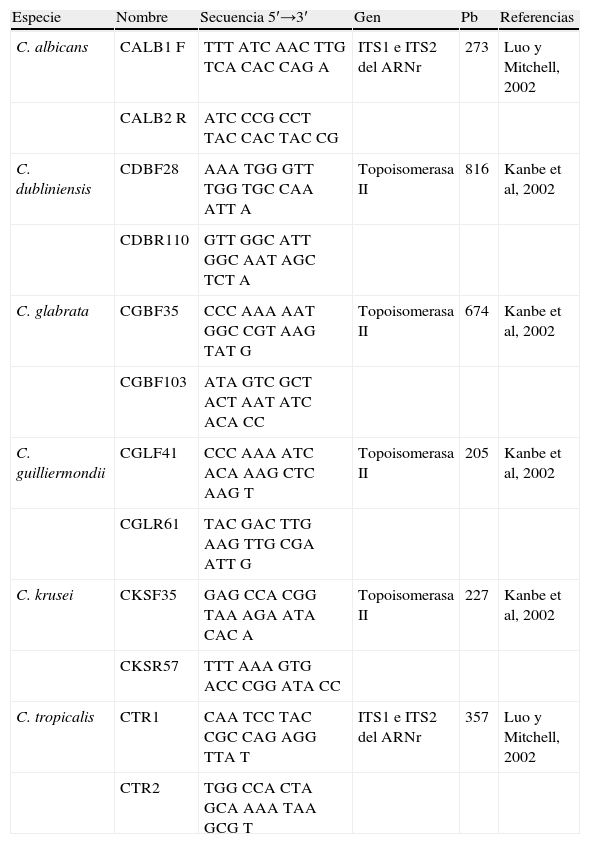
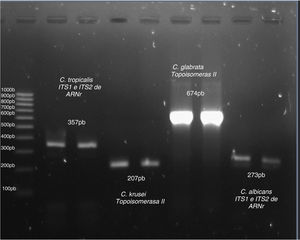
Identificación de especies mediante el método de la reacción en cadena de la polimerasaLa caracterización molecular se realizó utilizando la prueba de la reacción en cadena de la polimerasa4,41 a partir de los cultivos primarios con el objeto de comparar este método molecular con otros métodos de identificación de especies que se utilizan convencionalmente con fines diagnósticos, y medir mediante una prueba diagnóstica la especificidad y sensibilidad de dicha prueba molecular en la identificación de las especies más frecuentes, previa optimización del método utilizando cepas control de cada especie. Para cada reacción, fue tomada una colonia de Candida directamente de un cultivo puro de cada aislamiento a ensayar crecido en agar dextrosa Sabouraud por 24h a 37°C y esta fue resuspendida en un vial con 300μL de amortiguador de fosfatos (1,4M NaCl, 50mM KCl, 90mM Na2PO4·2H2O, 20mM KH2PO4, con pH 7,4); para degradar completamente la pared de los hongos se añadieron 125 U de liticasa (Sigma, EE.UU.), y se incubó por 30min a 37°C agregando posteriormente proteinasa K (25 a 50μL de una solución a 20mg/mL, Promega, EE.UU.) y 100μL de solución de extracción (400mM Tris-Cl, 1M NaCl, 20mM EDTA, 2% dodecil sulfato de sodio) incubando nuevamente durante 12h a 65°C. Para la extracción y purificación del ADN fue utilizada la técnica de remoción de proteínas con fenol-cloroformo y para la precipitación del ADN fue añadido y lavado con 1mL de isopropanol absoluto frío seguido de etanol frío al 75%, centrifugando entre cada lavado a 14.000rpm durante 10min a 4°C. El etanol se retiró, se desecó el ADN al aire libre durante 30min y se resuspendió, cuantificó y ajustó a una concentración de 0,25 a 1mg/μL para su posterior amplificación. Las muestras de ADN obtenidas fueron conservadas a –80°C por tiempo indefinido hasta su utilización. En la reacción de PCR fueron empleados cebadores específicos para seis especies, que amplifican los genes ITS1 e ITS2 del ARNr o el gen de la topoisomerasa ii, los cuales se presentan en la tabla 116,24. El cóctel para cada reacción de PCR fue preparado con 12,5μL de 2X Go Taq Green Master Mix (Promega, EE.UU.), 5μL de primer (Invitrogen, EE.UU.) mix (sentido y antisentido a una concentración final de 0,5mM para cada especie) y 1 a 5μL del ADN previamente extraído; dicha reacción fue aforada a 25μL. Previo a las identificaciones de aislamientos clínicos se realizó una estandarización que incluyó ensayos donde se probó la PCR desde 10 ng hasta 1μg de ADN. En cada ensayo, se incluyó un control negativo que contenía únicamente agua en lugar de ADN. El termociclador (Multigene, Labnet Internacional, Inc. EE.UU.) fue programado para las reacciones de PCR para amplificación con una fase inicial de desnaturalización a 96°C por 5min, seguida por 38 ciclos, iniciando con una temperatura de 96°C por 30s, seguida por la fase de alineación a 54°C por 30s y una fase de elongación a 72°C por 30s; finalmente, fue programada una fase de elongación final a 72°C por 10min. El análisis electroforético de los productos de la PCR de las especies de Candida fue visualizado en un gel de agarosa al 1,7% con 1μg/mL de bromuro de etidio. Cada pozo del gel de agarosa fue cargado con 5μL del producto de la PCR, y solo un pozo fue cargado con 5μL del marcador de pares de bases (Mpb), pBR322 digerido con HaeIII (Sigma, EE.UU.). A la cámara le fue aplicado un voltaje de 100V entre 25 y 30min. Los amplicones y los marcadores de pares de bases fueron visualizados a través de su interacción con bromuro de etidio y la exposición a luz ultravioleta (Labnet International, Inc., EE.UU.), mientras que la documentación fotográfica de los geles fue realizada en el sistema VersaDoc con el programa Quantity One (Bio-RAD, Inc., EE.UU.). Los productos de la PCR fueron analizados evaluando el tamaño en pares de bases de cada una de las bandas obtenidas tomando como positivas aquellas que presentaban el tamaño esperado para cada especie (tabla 1) y comparando con la banda obtenida de la amplificación del ADN de cepas control de cada especie.
Cebadores específicos empleados para la identificación molecular de las especies más frecuentes de Candida
| Especie | Nombre | Secuencia 5′→3′ | Gen | Pb | Referencias |
| C. albicans | CALB1 F | TTT ATC AAC TTG TCA CAC CAG A | ITS1 e ITS2 del ARNr | 273 | Luo y Mitchell, 2002 |
| CALB2 R | ATC CCG CCT TAC CAC TAC CG | ||||
| C. dubliniensis | CDBF28 | AAA TGG GTT TGG TGC CAA ATT A | Topoisomerasa II | 816 | Kanbe et al, 2002 |
| CDBR110 | GTT GGC ATT GGC AAT AGC TCT A | ||||
| C. glabrata | CGBF35 | CCC AAA AAT GGC CGT AAG TAT G | Topoisomerasa II | 674 | Kanbe et al, 2002 |
| CGBF103 | ATA GTC GCT ACT AAT ATC ACA CC | ||||
| C. guilliermondii | CGLF41 | CCC AAA ATC ACA AAG CTC AAG T | Topoisomerasa II | 205 | Kanbe et al, 2002 |
| CGLR61 | TAC GAC TTG AAG TTG CGA ATT G | ||||
| C. krusei | CKSF35 | GAG CCA CGG TAA AGA ATA CAC A | Topoisomerasa II | 227 | Kanbe et al, 2002 |
| CKSR57 | TTT AAA GTG ACC CGG ATA CC | ||||
| C. tropicalis | CTR1 | CAA TCC TAC CGC CAG AGG TTA T | ITS1 e ITS2 del ARNr | 357 | Luo y Mitchell, 2002 |
| CTR2 | TGG CCA CTA GCA AAA TAA GCG T |
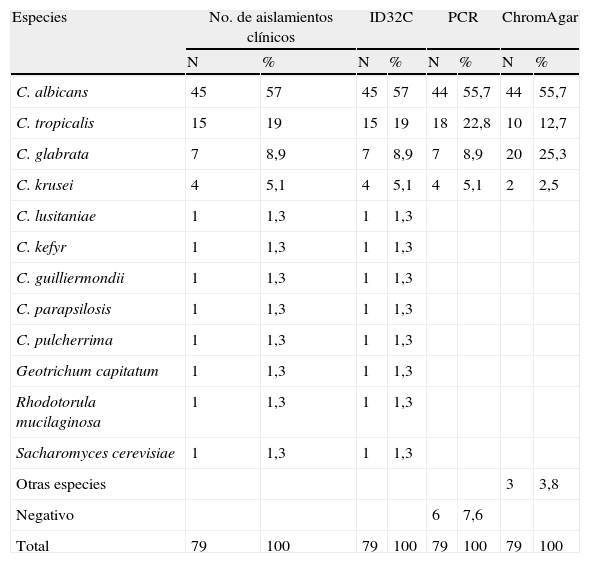
Se estudiaron 79 aislamientos clínicos provenientes de la colonización o infección de diversos grupos de pacientes; la mayoría de los aislamientos (39,2%) provenían de la mucosa oral de pacientes desdentados parciales o totales, en un 58,2% provenían de la mucosa oral de pacientes diabéticos (n=31) y dos aislamientos (2,5%) se obtuvieron a partir de expectoraciones de pacientes con enfermedad respiratoria aguda. Algunas muestras estudiadas de otras regiones, materiales o fluidos fueron negativas a la presencia de levaduras. En la tabla 2 se muestran los resultados y las diferencias obtenidas en la identificación de especies por varios métodos. La identificación de especies por medio de la prueba molecular de la PCR y por medio de los cultivos en Chromagar Candida® mostró algunas diferencias en las frecuencias de especies con respecto a la identificación por el sistema ID32C AUX, el cual fue considerado en este ensayo como el estándar de oro ya que es el método validado universalmente en la actualidad. La especie más prevalente utilizando el sistema ID32C y la PCR fue C. albicans, seguida por C. tropicalis, C. glabrata y C. krusei, las cuales han sido identificadas como las principales asociadas a las candidiasis locales, sistémicas o diseminadas por sus factores de virulencia y resistencia a los antifúngicos.
Comparación de los métodos de identificación ID32C, reacción en cadena de la polimerasa y ChromAgar Candida®
| Especies | No. de aislamientos clínicos | ID32C | PCR | ChromAgar | ||||
| N | % | N | % | N | % | N | % | |
| C. albicans | 45 | 57 | 45 | 57 | 44 | 55,7 | 44 | 55,7 |
| C. tropicalis | 15 | 19 | 15 | 19 | 18 | 22,8 | 10 | 12,7 |
| C. glabrata | 7 | 8,9 | 7 | 8,9 | 7 | 8,9 | 20 | 25,3 |
| C. krusei | 4 | 5,1 | 4 | 5,1 | 4 | 5,1 | 2 | 2,5 |
| C. lusitaniae | 1 | 1,3 | 1 | 1,3 | ||||
| C. kefyr | 1 | 1,3 | 1 | 1,3 | ||||
| C. guilliermondii | 1 | 1,3 | 1 | 1,3 | ||||
| C. parapsilosis | 1 | 1,3 | 1 | 1,3 | ||||
| C. pulcherrima | 1 | 1,3 | 1 | 1,3 | ||||
| Geotrichum capitatum | 1 | 1,3 | 1 | 1,3 | ||||
| Rhodotorula mucilaginosa | 1 | 1,3 | 1 | 1,3 | ||||
| Sacharomyces cerevisiae | 1 | 1,3 | 1 | 1,3 | ||||
| Otras especies | 3 | 3,8 | ||||||
| Negativo | 6 | 7,6 | ||||||
| Total | 79 | 100 | 79 | 100 | 79 | 100 | 79 | 100 |
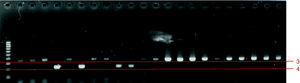
Una vez estandarizada la amplificación de especies utilizando la PCR, la cual incluyó varios ensayos de gradiente de concentración de ADN y gradientes de la temperatura de alineación, se realizó la identificación analizando los productos de la amplificación del ADN de los aislamientos clínicos y de cepas ATCC (fig. 1). En la fig. 2 se incluyen las identificaciones de algunos aislamientos clínicos de C. albicans y C. glabrata, mientras que en la fig. 3 se incluyen las identificaciones de algunos aislamientos clínicos de C. tropicalis y C. krusei.
Respecto a la identificación de especies presuntiva que nos proporciona el uso del medio cromogénico ChromAgar Candida®, la identificación fue excelente para C. albicans y las mayores discrepancias se presentaron para C. glabrata, donde observamos casos falsos positivos, mientras que para C. tropicalis y C. krusei fue menos sensible. Adicionalmente se observaron aislamientos identificados como otras especies (3,8%) ya que no se pudo identificar plenamente un color y textura característicos, siguiendo las instrucciones del fabricante.
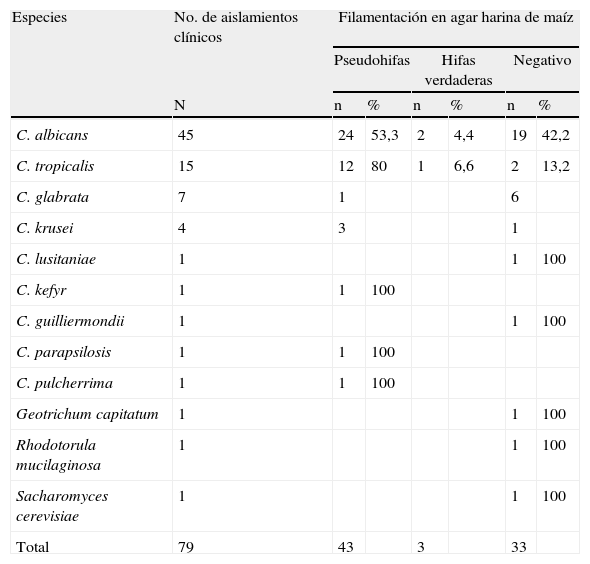
En la producción de formas filamentosas en agar harina de maíz (tabla 3) se observó que de los 45 aislamientos de C. albicans, 24 (53,3%) fueron positivos para pseudohifas, dos (4,4%) produjeron hifas verdaderas y 19 (42,2%) fueron negativos para filamentación; de los 15 aislamientos de C. tropicalis, 12 (80%) fueron positivos para pseudohifas, uno (6,6%) produjo hifas verdaderas y dos (13,3%) fueron negativos para filamentación; los aislamientos de C. kefyr, C. parapsilosis y C. pulcherrima fueron positivos para pseudohifas (100%, con un aislamiento respectivamente); tres aislamientos de C. krusei fueron positivos para filamentación con formación de pseudohifas, mientras que un aislamiento fue negativo; los aislamientos C. glabrata fueron negativos para filamentación en todos los casos; C. lusitaniae y C. guillermondii también fueron negativos para filamentación (100%, con un aislamiento respectivamente). El análisis estadístico demostró que las formas filamentosas se asocian (chi cuadrado p<0,0001) a las especies C. albicans y C. tropicalis y que las especies negativas a filamentación (Chi cuadrado p<0,0001) fueron C. glabrata y C. krusei.
Filamentación en agar harina de maíz de las diferentes especies de Candida
| Especies | No. de aislamientos clínicos | Filamentación en agar harina de maíz | |||||
| Pseudohifas | Hifas verdaderas | Negativo | |||||
| N | n | % | n | % | n | % | |
| C. albicans | 45 | 24 | 53,3 | 2 | 4,4 | 19 | 42,2 |
| C. tropicalis | 15 | 12 | 80 | 1 | 6,6 | 2 | 13,2 |
| C. glabrata | 7 | 1 | 6 | ||||
| C. krusei | 4 | 3 | 1 | ||||
| C. lusitaniae | 1 | 1 | 100 | ||||
| C. kefyr | 1 | 1 | 100 | ||||
| C. guilliermondii | 1 | 1 | 100 | ||||
| C. parapsilosis | 1 | 1 | 100 | ||||
| C. pulcherrima | 1 | 1 | 100 | ||||
| Geotrichum capitatum | 1 | 1 | 100 | ||||
| Rhodotorula mucilaginosa | 1 | 1 | 100 | ||||
| Sacharomyces cerevisiae | 1 | 1 | 100 | ||||
| Total | 79 | 43 | 3 | 33 | |||
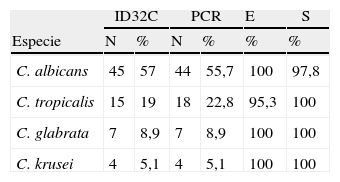
La identificación de especies utilizando el método molecular de la PCR según la prueba diagnóstica (intervalo de confianza de 95%) mostró una especificidad y sensibilidad del 100% para C. krusei y C. glabrata. La identificación de C. albicans por PCR mostró una especificidad del 100%, con una sensibilidad del 97,8% ya que identificó un falso negativo, mientras que para C. tropicalis se observó una sensibilidad del 100%, con una especificidad del 95,3% ya que fueron identificados 3 falsos positivos por la prueba molecular de la PCR para esta especie (tabla 4). Los casos falsos positivos fueron confirmados como verdaderos negativos mediante un ensayo con enzimas de restricción, para lo que obtuvimos las secuencias ITSI e ITS2 de C. tropicalis (Acceso AB534606 GenBank, NCBI); con esta secuencia y con los cebadores específicos utilizados se corrió una PCR in silico utilizando el programa Fast PCR Professional Versión 6.0 (Helsinki, Finlandia 2010) para obtener la secuencia del producto de amplificación. El producto obtenido de la amplificación se copió y mediante el programa Webcutter versión 2.0 se realizó un análisis de sitios para enzimas de restricción. El producto de amplificación de los aislamientos de C. tropicalis fue de 356 pb y la enzima seleccionada fue la Bbul que corta en la posición 217. Las amplificaciones falsas positivas no fueron cortadas.
Prueba diagnóstica que evalúa la sensibilidad y especificidad de la prueba molecular de la reacción en cadena de la polimerasa comparada con el sistema ID32C AUX
| ID32C | PCR | E | S | |||
| Especie | N | % | N | % | % | % |
| C. albicans | 45 | 57 | 44 | 55,7 | 100 | 97,8 |
| C. tropicalis | 15 | 19 | 18 | 22,8 | 95,3 | 100 |
| C. glabrata | 7 | 8,9 | 7 | 8,9 | 100 | 100 |
| C. krusei | 4 | 5,1 | 4 | 5,1 | 100 | 100 |
E: especificidad; S: sensibilidad.
La mortalidad de las candidiasis invasoras está condicionada por factores tanto diagnósticos como terapéuticos25 y disminuye de manera importante cuando existe un diagnóstico oportuno12. Se estima que la tasa de mortalidad atribuible a la candidemia antemortem oscila entre el 22 y el 33% de pacientes con infecciones diseminadas y puede llegar a ser hasta del 71% en enfermos inmunodeficientes. No obstante, una elevada proporción de diagnósticos de candidiasis sistémica ocurre en las necropsias (67-88%) de personas cuya causa de muerte no fue atribuida a Candida39. La más alta incidencia de estas candidiasis invasoras está asociada a pacientes ingresados en Unidades de Cuidados Intensivos30,32,33 y se ha convertido en un importante problema de salud pública, ya que Candida es el tercer o cuarto patógeno aislado del torrente sanguíneo en infecciones hospitalarias en los Estados Unidos13, mientras que en México se ha reportado que un 10% de estas infecciones son causadas por hongos, siendo el más frecuente C. albicans8, seguido de C. parapsilosis, C. tropicalis, C. glabrata y C. krusei14; así, los métodos de identificación deben ser lo suficientemente sensibles, específicos y rápidos en el diagnóstico e identificación de especies de Candida, a partir de muestras clínicas diversas o a partir de aislamientos primarios. Para ello, durante algunas décadas las pruebas bioquímicas como el API 20C o el ATB ID32C han sido elegidos como el estándar de oro por diversos autores debido a su amplio número de pruebas de asimilación de carbohidratos y porque evitan el uso complementario de pruebas microscópicas7, sin embargo, estas y las pruebas de filamentación en agar requieren de 18 a 72 horas o más, lo que puede entorpecer las decisiones terapéuticas, mientras que la prueba de producción del tubo germinativo y la PCR son las que requieren el mínimo de tiempo (2 a 16 horas), aunque las pruebas de filamentación son poco confiables especialmente en la identificación de especies diferentes a C. albicans. Nuestro estudio da evidencia de ello, ya que diferentes especies incluyendo C. albicans mostraron diversa capacidad de filamentación utilizando un medio ampliamente validado para este fin que es el agar harina de maíz, lo que hace a esta prueba poco específica de especies.
Más recientemente se están utilizando los medios cromógenos diferenciales como el Chromagar Candida® que permiten una identificación presuntiva rápida de C. albicans directamente al observar el color de las colonias aisladas. Cárdenes et al7 determinaron que la sensibilidad para Chromagar Candida® excede un 97% y la especificidad es mayor al 92%. A este respecto coincidimos en que el color único característico de los aislamientos de C. albicans permite una confiable identificación de esta especie sin necesidad de más estudios de laboratorio; esta característica por sí sola hace a Chromagar Candida® de valiosa consideración para los aislamientos de rutina, aunque en México no se ha extendido su comercialización y resultan excesivamente costosos. Adicionalmente, coincidimos con otros autores en que C. tropicalis es la principal fuente de falsas identificaciones con estos medios cromógenos6.
Los métodos moleculares basados en ADN tienen el potencial de disminuir el tiempo del laboratorio en la identificación de los patógenos que crecen de manera lenta o que son difíciles de cultivar; así, las ventajas de la PCR son su corto tiempo de procesamiento con una alta sensibilidad y especificidad para las especies más frecuentes como lo pudimos observar (tabla 4). Nuestros resultados determinaron que la amplificación por PCR es ideal para detectar levaduras en volúmenes mínimos con concentraciones de ADN desde 10 ng/μl, apoyando la idea de otros autores que resaltan que la PCR puede detectar el genoma de Candida en concentraciones que van desde 3 células/0,1mL26 hasta una célula/mL de muestra17. Sin embargo, en este estudio realizamos la identificación molecular de especies a partir de ADN extraído de colonias aisladas con el fin de comparar y validar el método de la PCR contra métodos convencionales, lo que permitirá definir futuras pautas de identificación en nuestro medio hospitalario y proponer su posterior uso e incluso mejorarlo a partir de muestras clínicas independientes al cultivo. Diversos estudios de identificación simultánea de especies de Candida por PCR reportan excelentes sensibilidades, directamente de muestras clínicas orales22, vaginales40, o a partir de sangre2, con la ventaja adicional de que no existen reacciones cruzadas con bacterias19, otros hongos o virus, situación importante ya que frecuentemente, en la población de pacientes inmunosuprimidos, estos tienen el mayor riesgo de candidiasis diseminadas y presentan cultivos sanguíneos mixtos levaduras/bacterias. Estas ventajas dependen en mucho de los fragmentos amplificados; algunos marcadores usados para la PCR incluyen el citocromo P450, los genes de choque térmico y los genes de regulación de pH, sin embargo los cebadores que amplifican los genes de rRNA (18S, 28S y 5.8S) son los más frecuentemente usados por su naturaleza universal y por su largo número de copias42, además se ha demostrado que son los más conservados y proveen una base molecular para el establecimiento de relaciones filogenéticas, no obstante, entre esas secuencias se encuentran las regiones de los espaciadores internos transcritos 1 y 2 (ITS1 e ITS2) que evolucionan rápidamente y por lo tanto pueden variar entre las diferentes especies dentro de un género10,21. La identificación a partir de colonias aisladas utilizando los cebadores ITS1 e ITS2 también ha sido propuesta con rangos de identificación del 96,8% (ITS1) y del 99,7% (ITS2)21, mientras que el gen de la topoisomerasa ii es un blanco que también se ha utilizado para ver relaciones filogenéticas entre las especies de Candida y para la identificación de C. albicans, C.tropicalis, C. parapsilosis, C. glabrata, C. dubliniensis, C. kefyr, C. krusei, C. parapsilosis, C.guillermondi y C. lusitaniae con una excelente especificidad y sensibilidad aun en presencia de varias especies a la vez17,15.
Nuestros resultados revelan que el ensayo de la PCR es altamente discriminatorio para identificar a C. krusei y C. glabrata, donde la especificidad y sensibilidad fue del 100% con respecto al sistema ID32C AUX, mientras que C. albicans mostró una especificidad del 100% y una alta sensibilidad (97,8%) y C. tropicalis, de manera inversa, mostró una sensibilidad del 100% y una especificidad menor (95%). A este respecto, Luo y Mitchell24 proponen el uso de los cebadores CTR1 y CTR2 para la identificación de C. tropicalis, ellos probaron en un ensayo de PCR múltiple 35 aislamientos previamente identificados con un 100% de sensibilidad y especificidad; en nuestros ensayos obtuvimos tres identificaciones falsas positivas, donde pudimos observar bandas que correspondían al tamaño esperado pero de baja intensidad y como resultado de un ensayo con enzimas de restricción se determinó que estas amplificaciones eran negativas para esta especie; a través de un Blast pudimos percatarnos de que estos cebadores pueden flanquear en regiones similares intergénicas de otras especies.
Los patrones obtenidos de la PCR son consistentes usando los cebadores que amplifican los genes ITS1 e ITS2 o el gen de la topoisomerasa ii y muestran resultados confiables, rápidos y repetibles, además con una metodología simple para la identificación de estas especies de Candida. Sin embargo, hay que señalar que la prueba molecular de la PCR a pesar de ser un método rápido y eficaz en la identificación de especies, no puede señalar si se trata de infección o colonización en el caso de muestras que provienen de mucosas, por lo que hasta hoy, la PCR deberá estar acompañada de evidencia clínica como radiología, cultivos, histopatología u otras pruebas diagnósticas, así, sugerimos validar su eficacia a partir de muestras clínicas con futuras manipulaciones de los productos de la amplificación para consensuar las secuencias, realizando ensayos con enzimas de restricción38, pruebas con marcadores enzimáticos o radioactivos42 o la secuenciación de los fragmentos del ADN1,28 debiendo considerar en la validación de los métodos otras alternativas como la PCR cuantitativa, la PCR Microsatelite (MSP-PCR), el fragmento amplificado de longitud de polimorfismo (AFLP)23 y la PCR multiplex-tándem20, que están entre los más nuevos y prometedores métodos de caracterización microbiana.
Concluimos que la prueba molecular de la PCR utilizando los cebadores que amplifican los genes ITS1 e ITS2 o el gen de la topoisomerasa ii es una prueba altamente específica y sensible para la identificación de importantes especies patógenas de Candida incluyendo C. krusei, C. glabrata, C. albicans y menos específica para C. tropicalis. En lo que se refiere al medio cromógeno CromAgar Candida®, este es excelente para identificar a C. albicans ofreciendo identificaciones presuntivas de otras especies y resulta muy útil para identificar la presencia de dos o más especies en una muestra clínica, mientras que en lo que se refiere a la capacidad de filamentación, C. albicans, C. tropicalis y C. krusei filamentan de manera variable y lenta, por lo que este método solo puede orientarnos en el laboratorio respecto a la capacidad de patogenicidad de las especies dimorfas.
FinanciaciónEste proyecto ha sido financiado parcialmente por la UACJ en su convocatoria de Investigación 2008-2009, por el apoyo PROMEP SEP 2008-2009.
Agradecemos la asesoría y colaboración de la Dra. María de la Luz Macías Argüelles por su apoyo en los análisis bioinformáticos.
Comparativo entre ChromAgar Candida®, PCR e ID32C AUX para la identificación de especies de Candida.