En la actualidad existen notables diferencias en el envejecimiento de los individuos de las poblaciones modernas. Mientras que algunos de ellos disfrutan de un prolongado envejecimiento saludable, otros desarrollan enfermedades neurodegenerativas como la enfermedad de Alzheimer (EA). Los factores ambientales son decisivos en este hecho, pero la genética puede contribuir a explicar las diferencias observadas. Recientemente se ha postulado que los genes de la longevidad podrían ser también neuroprotectores.
ObjetivosEvaluar si determinadas variantes genéticas relacionadas con la longevidad pueden tener un carácter neuroprotector.
MétodosLos sujetos a estudio son las personas con una edad superior a 90 años. De cada participante se realizará la recogida de datos sociodemográficos, clínicos y múltiples valoraciones: cognitiva, funcional, antropométrica, nutricional, sensorial y física. Además, se realizará el análisis de 64 loci SNPs, distribuidos en 13 genes candidatos FOXO3, SIRT1, TOMM40, APOE, PICALM, COMT, CETP, CLU, CR1, IL-6, PCK-1, ZNF224 y ACE mediante Taqman array.
ResultadosObtener un mayor conocimiento sobre los alelos infra/sobre representados en las personas nonagenarias. Además, la comparación de las características genéticas de los nonagenarios con EA con aquellos libres de enfermedad permitirá observar vinculaciones entre determinados alelos con la protección o el riesgo de EA.
La información asociada de los participantes permitirá crear subgrupos mostrando las interacciones entre el ambiente y las variaciones genéticas en relación al envejecimiento saludable y la EA.
ConclusiónEl estudio de la variabilidad genética de las personas nonagenarias nos puede dar información sobre los alelos relacionados con la longevidad y la neuroprotección.
Currently there are notable differences in the aging of individuals in modern populations. While some of them enjoy a long healthy aging, others develop neurodegenerative diseases, such as Alzheimer's disease (AD). Environmental factors are critical, but genetics could explain the differences observed. It has recently been postulated that longevity genes might also be neuroprotective.
ObjectivesTo assess whether certain genetic variants associated with longevity might have a neuroprotective effect.
MethodsThe subjects of this study are people older than 90 years. We will collect sociodemographic and clinical data and multiple assessments, cognitive, functional, anthropometric, nutritional, sensory and physical each participant. In addition, 64 SNPs loci distributed in 13 candidate genes FOXO3, SIRT1, TOMM40, APOE, PICALM, COMT, CETP, CLU, CR1, IL-6, PCK-1, ZNF224 and ACE will be analysed by Taqman array.
ResultsIt is hoped to gain more knowledge about under/over-represented alleles in nonagenarians. Furthermore, comparison of the genetic characteristics of nonagenarians with AD with those free of disease will enable links to be seen between certain alleles with protection or the risk of AD. Associated information on the participants will create subgroups showing the interactions between environment and genetic variation in relation to healthy aging and AD.
ConclusionThe study of the genetic variability of nonagenarians can give us information on the alleles associated with longevity and neuroprotection.
El importante desarrollo de las sociedades occidentales ha propiciado el envejecimiento de la población. En contrapartida, este hecho está relacionado con el incremento de la incidencia de las enfermedades neurodegenerativas, lo que origina un importante impacto social y económico.
Sin embargo, esta realidad afecta de forma desigual a los individuos que componen las sociedades modernas. Mientras algunos disfrutan de una longevidad acompañada de un envejecimiento saludable, con sus capacidades físicas y cognitivas conservadas, otros en mayor o menor medida, sufren un deterioro físico y enfermedades neurodegenerativas que condicionan el envejecimiento, produciendo discapacidad y acortando la supervivencia.
El componente ambiental parece tener un papel importante en la longevidad, pero resulta insuficiente para explicar las diferencias entre los individuos que envejecen en un entorno común. Estas diferencias obedecen a múltiples factores (hábitos de vida, enfermedades, situación económica, accidentes, etc.) entre los que la genética contribuye a explicar el 15-30% de nuestra supervivencia y envejecimiento1–4. La influencia de los factores genéticos es aún mayor en el último tercio de la vida (pasados los 60 años de edad)5 y en algunos aspectos relevantes del envejecimiento como el rendimiento físico, la función cognitiva y el envejecimiento óseo1–10.
A lo largo de la última década se han realizado importantes avances sobre la relación existente entre la genética y la longevidad. Las investigaciones realizadas en organismos modelo han permitido asociar genes candidatos con rutas biológicas vinculadas a la longevidad. Por una parte, los trabajos en invertebrados son diversos, aunque el organismo que más ha aportado al conocimiento del envejecimiento, la longevidad y los procesos de reparación del ADN es Caenorhabditis elegans (C. elegans)11–13. Igualmente, los trabajos realizados en la mosca de la fruta Drosophila melanogaster (D. melanogaster) son destacables, habiéndose encontrado nexos con C. elegans. Así, se ha podido observar que los factores de crecimiento de insulina tipo 1 (IGF-1) y la influencia del estrés oxidativo afectan de forma determinante a la longevidad14–18. Con el objetivo de intentar crear un puente entre el hombre y los invertebrados se ha estudiado el ratón como modelo de envejecimiento, y algunos de los datos obtenidos parecen ser correlativos a los reflejados en lo estudios de C. elegans y D. melanogaster16. Estos estudios en diferentes modelos de envejecimiento, han puesto de manifiesto la existencia de genes homólogos entre humanos y animales, por lo que su estudio tiene el potencial para proporcionar información sobre la longevidad19–21, en lo que parece un cruce entre los mecanismos de regulación de la vía de señalización Ins/IGF-1 (insulina/factor de crecimiento de insulina tipo 1) y la regulación de las vías de señalización de las especies oxígeno reactivas (ROS)22,23.
Las tecnologías de genotipado masivo, los estudios de asociación de genoma completo (Genome Wide Association Studies [GWAS]) desarrolladas en los últimos años, han supuesto un gran avance en el análisis de la relación existente entre los polimorfismos encontrados en todo el genoma y la prolongación de la vida humana. Varias publicaciones recogen los metaanálisis realizados con estos estudios sin haber encontrado una asociación significativa de ningún gen con la longevidad24–26.
Sin embargo, Sebastiani et al publican en 2010 un trabajo no incluido en los metaanálisis anteriores, ofreciendo datos diferentes. Tras estudiar el genoma de 1.055 centenarios y 1.267 controles, encuentran 150 polimorfismos de un solo nucleótido (SNPs) relacionados con la longevidad y con la supuesta capacidad para predecirla27, aunque estos resultados deben ser replicados en poblaciones diferentes.
Respecto a las enfermedades neurodegenerativas, destaca el incremento de la incidencia de la enfermedad de Alzheimer (EA) al aumentar la edad. Las formas familiares están vinculadas con alteraciones en la proteína precursora de amiloide (APP), presenilina 1 (PSEN1) y presenilina 2 (PSEN2), como han recogido múltiples trabajos realizados en la última década. La vinculación entre ciertos genes y EA esporádica queda recogida en la website Alzgene, en forma de metaanálisis de la mayoría de los trabajos publicados hasta la fecha en relación a la EA, inclusive los últimos GWAS. Como resultado de estos análisis, la website propone una lista, encabezada por el gen APOE28.
Sanders et al (2010) han propuesto recientemente la hipótesis de que las variantes génicas que tienen una vinculación con la longevidad guardan una relación con la neuroprotección29. Sería posible, por tanto, que las personas longevas sin EA sean portadoras de ciertas variantes génicas de longevidad y de neuroprotección, no presentes en longevos con Alzheimer.
Con el objetivo de evaluar que variaciones genéticas pueden estar relacionadas con la longevidad y las enfermedades neurodegenerativas, particularmente la EA, hemos realizado, en primer lugar, una selección de 13 genes candidatos a partir de la literatura. Así, se han incluido genes relacionados con la regulación transcripcional (FOXO3 [Gen ID: 2309] y SIRT1 [Gen ID: 23411]), el movimiento de proteínas al interior de la mitocondria (TOMM40 [Gen ID:10452]), el transporte de lípidos (APOE [Gen ID: 348], CETP [Gen ID: 1071] y CLU [Gen ID: 1191]), endocitosis (PICALM [Gen ID: 8301]), metabolismo de neurotransmisor (COMT [Gen ID: 1312]), respuesta inmune (CR1 [Gen ID: 1378] e IL-6 [Gen ID: 3569]), metabolismo glucosa (PCK-1 [Gen ID:5105] y ZNF224 [Gen ID:7767]), vasoconstricción (ACE [Gen ID: 1636]).
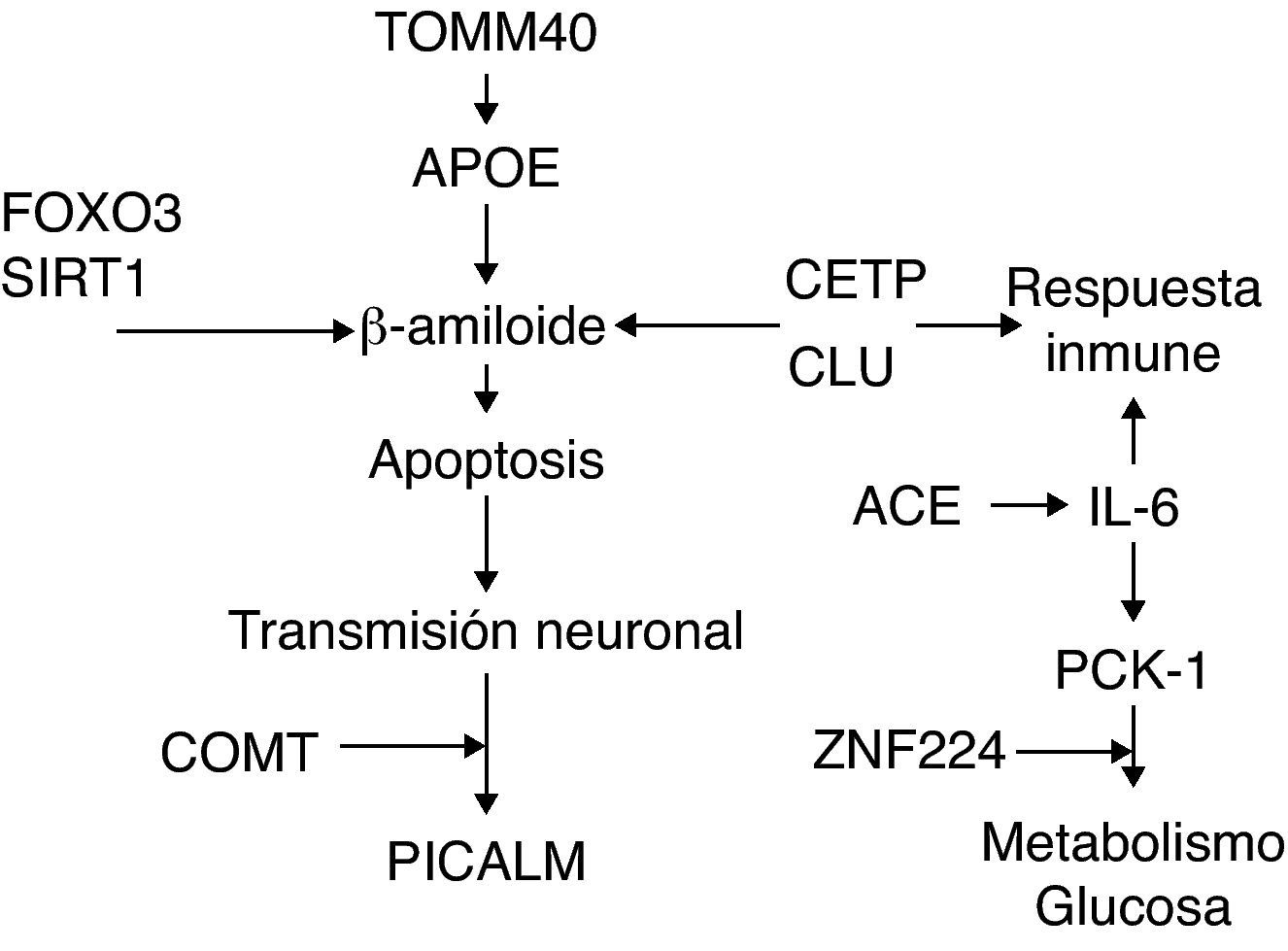
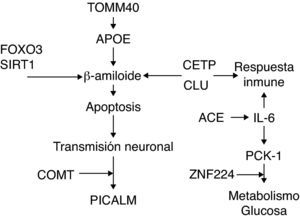
Estos genes están interconectados funcionalmente con la longevidad, la EA o con ambas, como se señala en la figura 1.
FOXO3, es regulado por SIRT-1 como respuesta al estrés oxidativo, lo que incrementa la capacidad de FOXO3 para detener el ciclo celular y aumentar la resistencia al estrés oxidativo, así como la inhibición de inducir la apoptosis30. Polimorfismos en TOMM40 parecen estar condicionando la edad de inicio de la EA, independientemente del alelo 4 de riesgo de APOE (APOE¿4), el mayor factor de riesgo para la EA esporádica31,32. APOE está relacionado con un mayor depósito de placa de beta-amiloide y deterioro de los mecanismos de reparación33,34, mientras que SIRT-1 parece estar relacionado con la supresión de la producción de beta-amiloide y la neuroprotección35,36, por lo que tienen efectos opuestos sobre la apoptosis. La apoptosis neuronal, la liberación y acción de neurotransmisores afecta a la transmisión de lo estímulos nerviosos. PICALM esta relacionado con el tráfico vesicular37, esencial en la liberación de neurotransmisores como la dopamina que es degradada por la enzima codificada por COMT38,39.
CETP y CLU, al igual que APOE, pueden interaccionar con lípidos. CETP esta relacionado con los niveles de HDL en plasma40, mientras que CLU participa en el intercambio de lípidos entre las apolipoproteínas41,42. Además, ha sido relacionado con la protección contra la toxicidad de beta-amiloide in-vitro, a diferencia de APOE, y la respuesta inmune al igual que CR1 e IL-643–45. IL-6 está relacionado con la disminución de la expresión de PCK1, represión que afecta al metabolismo de la glucosa, al igual que hace ZNF22446–51. La expresión de IL-6 parece estar condicionada en algunos casos por la acción proinflamatoria de ACE52–54.
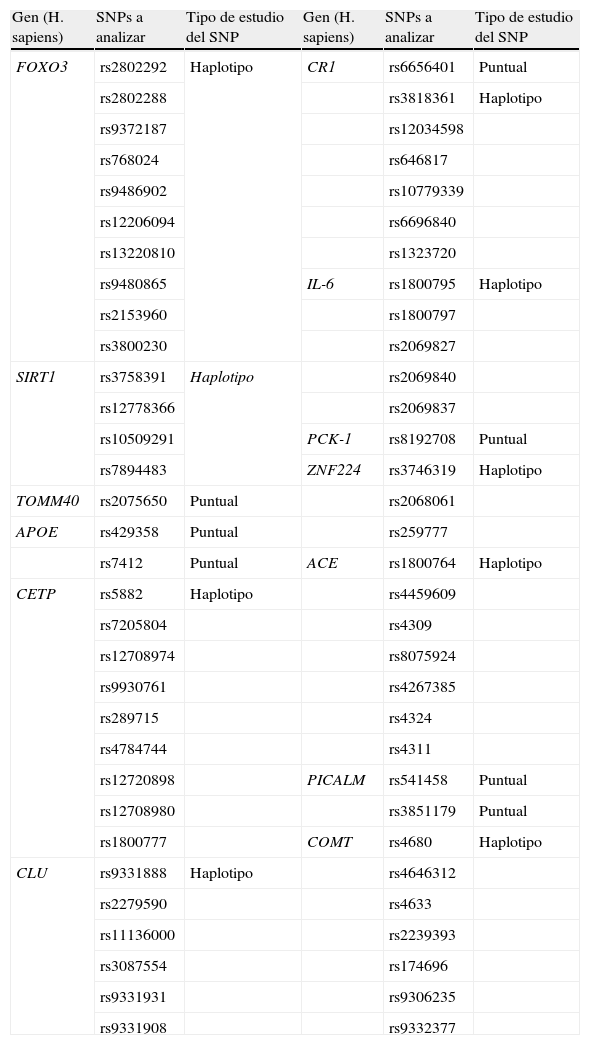
En segundo lugar, con objeto de caracterizar el grado de variación genética en estos genes, se ha establecido un conjunto de SNPs representativos de una región del genoma con alto desequilibrio de ligamiento (tagSNPs) que incorpora la información genética poblacional extraída del proyecto HapMap55, así como las variantes descubiertas que parecen relacionarse con la longevidad y demencia, que recogemos en la tabla 1.
Relación de SNPs a analizar dentro de los genes candidatos
| Gen (H. sapiens) | SNPs a analizar | Tipo de estudio del SNP | Gen (H. sapiens) | SNPs a analizar | Tipo de estudio del SNP |
| FOXO3 | rs2802292 | Haplotipo | CR1 | rs6656401 | Puntual |
| rs2802288 | rs3818361 | Haplotipo | |||
| rs9372187 | rs12034598 | ||||
| rs768024 | rs646817 | ||||
| rs9486902 | rs10779339 | ||||
| rs12206094 | rs6696840 | ||||
| rs13220810 | rs1323720 | ||||
| rs9480865 | IL-6 | rs1800795 | Haplotipo | ||
| rs2153960 | rs1800797 | ||||
| rs3800230 | rs2069827 | ||||
| SIRT1 | rs3758391 | Haplotipo | rs2069840 | ||
| rs12778366 | rs2069837 | ||||
| rs10509291 | PCK-1 | rs8192708 | Puntual | ||
| rs7894483 | ZNF224 | rs3746319 | Haplotipo | ||
| TOMM40 | rs2075650 | Puntual | rs2068061 | ||
| APOE | rs429358 | Puntual | rs259777 | ||
| rs7412 | Puntual | ACE | rs1800764 | Haplotipo | |
| CETP | rs5882 | Haplotipo | rs4459609 | ||
| rs7205804 | rs4309 | ||||
| rs12708974 | rs8075924 | ||||
| rs9930761 | rs4267385 | ||||
| rs289715 | rs4324 | ||||
| rs4784744 | rs4311 | ||||
| rs12720898 | PICALM | rs541458 | Puntual | ||
| rs12708980 | rs3851179 | Puntual | |||
| rs1800777 | COMT | rs4680 | Haplotipo | ||
| CLU | rs9331888 | Haplotipo | rs4646312 | ||
| rs2279590 | rs4633 | ||||
| rs11136000 | rs2239393 | ||||
| rs3087554 | rs174696 | ||||
| rs9331931 | rs9306235 | ||||
| rs9331908 | rs9332377 |
Búsqueda de factores genéticos relacionado con la longevidad y la neuroprotección.
Objetivos específicosPara alcanzar este objetivo general se proponen los siguientes objetivos concretos:
- 1.
Selección de al menos 200 personas con una edad igual o superior a 90 años.
- 2.
Dividir la muestra entre nonagenarios con un estado cognitivo sano y otros con demencia tipo Alzheimer.
- 3.
Análisis en todos los sujetos de 64 loci SNPs, distribuidos en 13 genes candidatos FOXO3, SIRT1, TOMM40, APOE, PICALM, COMT, CETP, CLU, CR1, IL-6, PCK-1, ZNF224, ACE mediante Taqman array.
- 4.
Validación de los resultados de los SNPs significativos.
El estudio está planteado como multicéntrico, prospectivo, descriptivo y analítico, en el ámbito del País Vasco, con la incorporación de muestras de Biobancos a nivel nacional.
Población elegibleIndividuos con una edad superior a 90 años.
Cálculo del tamaño muestralEl tamaño muestral se ha calculado teniendo en cuenta los datos de las frecuencias alélicas y haplotípicas de los genes candidatos seleccionados de Hapmap. Si bien Hapmap no recoge toda la variabilidad génica observada en personas longevas56, resulta la mejor aproximación disponible.
Las personas con una edad superior a los 90 años en la CAPV representan el 0,5% de total de la población. Por lo que las 200 personas con una edad superior a 90 años propuesta para recoger representan el 2% de la población nacida antes de 1920.
Criterios éticos de reclutamientoEste estudio ha sido evaluado por el comité de Ética e Investigación de la Universidad del País Vasco UPV/EHU. El estudio cumple con la Declaración de Helsinki en lo que respecta a la investigación en sujetos humanos.
Criterios de exclusiónNo disponer del historial médico del participante.
Variables del estudioValoración cognitiva- –
Screening de deterioro cognitivo: MMSE de Folstein, adaptado según edad, sexo y escolaridad.
- –
Función ejecutiva: test el reloj.
- –
Fluidez verbal: test de animales en un minuto.
- –
Praxias ideomotoras: Imitación de figuras del test de Barcelona revisado.
- –
Memoria: Memory Impairment Screen (MIS).
- –
Criterios de Demencia: DSM-IV.
- –
Criterios de enfermedad de Alzheimer: criterios NICDS-ADRA.
- –
Despistaje de demencia vascular: escala de Hachinsky.
- –
Estadiaje de la demencia: escala de GDS-FAST de Reisberg.
Índice de Barthel de las actividades de la vida diaria.
Antropometría y valoración nutricionalPeso, talla, Índice de masa corporal (IMC) y el test Mini Nutritional Assessment (MNA-SF)57.
Valoración sensorialCapacidad auditiva, visual y de comunicación.
Estado físicoTA, enfermedades relevantes, tratamientos e Índice de comorbilidad de Charlsson. Parámetros analíticos: glucemia basal, perfil lipídico, TSH, B-12, colesterol.
Variables sociodemográficasEdad, género, escolaridad, profesión, antecedentes familiares de demencia y factores de riesgo cardiovascular, hábitos tóxicos, estilo de vida.
NeuroimagenEn los casos en los que se disponga se recogerá los comentarios generales de TAC.
Variables biológicasA cada participante se le extraerán 2 tubos de EDTA de 10ml de sangre periférica. Mediante el proceso de Salting-out se obtendrá el ADN con el que realizar los estudios genéticos.
Variables genéticasEn total se estudiarán 64 SNPs en los genes candidatos. Siete de los SNPs se estudiarán de forma independiente, en base a los datos obtenidos en la revisión bibliográfica que muestran su relación con la longevidad o la demencia. Para el resto de los 57 SNPs que se estudiarán se han elegido aquellos tagSNPs disponibles en la base de datos de HapMap. Los criterios de selección fueron: 1) Menor frecuencia alélica (MAF) ≥ 5%, y 2) Una r2≥0,8, tabla 1.
Análisis estadísticoEstadística descriptiva general; media, mediana, desviación estándar, valores mínimos y máximos de las variables de escala con el paquete estadístico SPSS versión 17.0.
Para la comparación entre grupos (con demencia o sin demencia) se utilizará el test χ2, G-test (variable cualitativa) y ANOVA (variables cuantitativas).
Los datos obtenidos del ensayo TaqMan array serán analizados mediante los programas estadísticos PHASE, P-LINK y Haploview. Entre los diferentes estadísticos que se aplicarán se incluirán:
Comprobación del equilibrio de las poblaciones (Hardy Weinberg Equilibrium) y desequilibrio de ligamiento (LD).
Tests estadísticos para la determinación de asociaciones significativas alelo/genotipo, utilizando como controles los participantes sin demencia y como casos los pacientes afectados por la EA. Estratificación y cálculo por presencia/ausencia del alelo APOE¿4.
Clustering estadístico atendiendo a los correspondientes genotipos.
El cálculo de riesgo para los alelos asociadas a la demencia se establecerá mediante el cálculo del Odds ratio (OR), utilizando el cálculo de la regresión logística multinomial con un intervalo de confianza del 95%. Se considerará como diferencias estadísticamente significativas valores de p<0,05.
Esquema del estudio y plan de trabajoEl período máximo de realización del estudio está establecido en 36 meses.
Las fases en las que se divide el estudio son:
Fase 1. Fase de iniciaciónInclusión de los sujetos en el estudio. Recogida de muestras de sangre y recogida de datos. Esta fase se llevará a cabo durante la primera anualidad.
Fase 2. Fase de ejecuciónExtracción de ADN de las muestras de sangre. Normalización de las concentraciones de ADN para la ejecución del análisis mediante Taqman array. Se realizará en la Universidad del País Vasco UPV-EHU. Validación de los datos mediante el diseño de Snap-Shot. Esta fase se llevará a cabo durante la segunda anualidad.
Fase 3. Fase de entrega de resultadosTratamiento estadístico de los datos. Análisis de los genotipos. Elaboración del manuscrito y memoria final de resultados. Esta fase se llevará a cabo durante la tercera anualidad.
Fase 4: Fase de controlRecogida de la totalidad de la muestra en la primera anualidad y de la memoria final en la tercera anualidad.
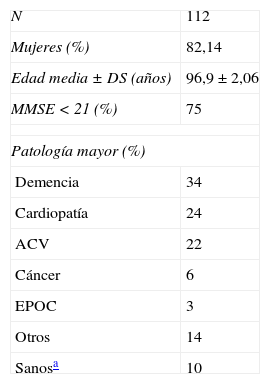
Resultados y discusiónActualmente el proyecto cuenta con 112 participantes. Se están incluyendo sujetos activamente por dos investigadores de dos residencias diferentes del País Vasco. Las características de los individuos incluidos se presentan en la tabla 2.
Análisis preliminar de las características demográficas más relevantes de los sujetos participantes
| N | 112 |
| Mujeres (%) | 82,14 |
| Edad media ± DS (años) | 96,9±2,06 |
| MMSE < 21 (%) | 75 |
| Patología mayor (%) | |
| Demencia | 34 |
| Cardiopatía | 24 |
| ACV | 22 |
| Cáncer | 6 |
| EPOC | 3 |
| Otros | 14 |
| Sanosa | 10 |
DS: desviación estándar; MMSE: Mini Mental State Examination.
El grupo de personas con edad superior a los 90 años es cada vez más numeroso en las sociedades desarrolladas. El factor de riesgo más importante para el desarrollo de una enfermedad neurodegenerativa es la edad. Esto origina que entre las personas nonagenarias las enfermedades neurodegenerativas afecten al 50% de los sujetos, siendo la más frecuente la EA.
Frente a esta realidad se encuentra un verdadero paradigma, las personas longevas que disfrutan de un envejecimiento saludable. Con objeto de dar explicación a los procesos que rodean a la longevidad, diversos estudios realizados en modelos animales han permitido obtener una valiosa información sobre la implicación de algunos genes. En algunos casos esta vinculación se ha podido trasladar a estudios en humanos, si bien, son necesarios más estudios específicos para encontrar nuevos genes relacionados con la longevidad.
Paralelamente, el estudio de los genes implicados en el desarrollo de las enfermedades neurodegenerativas centra actualmente un importante número de recursos científico-tecnológicos. Fruto de estos esfuerzos se ha podido observar que ciertos genes asociados a la longevidad son protectores de procesos neurodegenerativos, como la EA.
Por ello, el estudio de un grupo de personas nonagenarias representa una oportunidad de conocer la influencia de la dotación genética en la longevidad y los procesos neuroprotectores/neurodegenerativos. Conocer qué alelos pueden estar infra/sobre representados en este grupo, puede poner de manifiesto alguna posible vinculación con la longevidad. De igual modo, al comparar los nonagenarios con EA con aquellos libres de enfermedad, se pueden observar vinculaciones entre alelos con la protección o el riesgo. Asimismo, la información asociada de los participantes permitirá crear subgrupos, según factores ambientales, lo que permitirá observar interacciones entre ambiente y variaciones genéticas en el desarrollo de un envejecimiento saludable.
El presente proyecto de investigación se encuentra en ejecución. Los datos de asociación con la longevidad que sean obtenidos podrán servir de base para futuros estudios de comparación con otras poblaciones. En el caso de confirmarse será necesario por último, estudios in vitro o in vivo para demostrar la pérdida o ganancia de función debido a la variación genética identificada.
En cuanto a las limitaciones de este tipo de estudio se concretan en dos aspectos principales. Por una parte, la dificultad de obtener una muestra de personas longevas bien caracterizada. En parte esta limitación se subsana gracias al conocimiento que les proporciona a los investigadores participantes el seguimiento continuado de sus pacientes en las residencias. Sin embargo, esto genera un sesgo en la recogida de muestras al reducirlo al ámbito institucional, máxime cuando la mayoría de las personas nonagenarias no están institucionalizadas. Por ello, los investigadores del presente proyecto intentarán extender la recogida a sujetos nonagenarios no institucionalizados, supeditada a la obtención del historial clínico.
Por otra parte, aunque se ha utilizado la información del proyecto HapMap55, las frecuencias alélicas y haplotípicas en personas nonagenarias tienen un carácter prospectivo, por lo que las estimaciones en la proporción de las frecuencia haplotípicas son solo orientativas.
ConclusionesEl estudio de la variabilidad genética de las personas nonagenarias nos puede dar información sobre la implicación de determinados alelos en la longevidad y la neuroprotección.
Consideraciones éticasCon objeto de cumplir la Ley Orgánica 15/1999 de 13 de diciembre de protección de datos de carácter personal, (LOPD), desde el momento de la recogida del consentimiento informado se realizara la codificación del consentimiento informada, el cuestionario y las muestras biológicas que se tomen del sujeto. El consentimiento se incluirá en el fichero de investigación de nivel alto, con código de inscripción en el Registro de Protección de Datos 2080310015, cuyo titular es la Universidad del País Vasco UPV/EHU. La utilización de las muestras biológicas de los sujetos participantes estarán supeditadas a lo expuesto en Ley 14/2007, de 3 de julio, de investigación biomédica. El presente estudio ha sido evaluado por el comité de ética e investigación de la Universidad del País Vasco UPV-EHU.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Los autores agradecen el apoyo técnico y humano de los SGIker (UPV/EHU, MICINN, GV/EJ, FEDER y FSE) y la colaboración el Banco Nacional de ADN (Salamanca). Además, destacamos el apoyo y colaboración del Foro Agure (Vitoria-Gasteiz).
Este estudio está siendo financiado por el Departamento de Sanidad y Consumo del Gobierno Vasco (2009111083).