Existe un interés creciente por el carcinoma hepatocelular (CHC) debido a su elevada mortalidad y aumento en su incidencia. A diferencia de otros cánceres, el CHC surge mayoritariamente en el contexto de daño hepático crónico, complicando tanto el manejo clínico como la estimación de su pronóstico. Actualmente la clasificación BCLC proporciona una guía eficiente para el manejo de estos pacientes. Sin embargo, sería útil poder identificar preoperatoriamente los pacientes con mayor riesgo de recurrencia después de la resección, o aquellos que se beneficiarían del trasplante hepático excediendo los Criterios de Milán. El avance en técnicas genómicas de alta densidad, aplicables a tejido parafinado, ha favorecido el desarrollo de múltiples firmas genéticas y otros biomarcadores con fines pronósticos. Ninguno de estos biomarcadores, basados en el transcriptoma, micro-RNAs o metiloma, son utilizados en la práctica clínica, aunque es previsible que en el futuro puedan complementar la capacidad pronóstica que proporcionan las variables clínico-patológicas.
Recently, interest in hepatocellular carcinoma (HCC) has grown due to its high mortality and increased incidence. Unlike other malignancies, HCC mainly arises in the context of chronic liver injury, complicating its management and the prediction of prognosis. The Barcelona Clinic Liver Cancer (BCLC) staging classification currently offers an efficient decision-making guide in these patients. However, preoperative identification of patients with a higher risk of recurrence after resection and of those who could benefit from liver transplantation despite not meeting the Milan criteria would be useful. New high-throughput genomic technologies that can be applied to paraffin-embedded tissue have facilitated the identification of gene signatures and other biomarkers able to predict prognosis in HCC patients. None of these biomarkers, based on transcriptome, microRNAs or metilome, has been incorporated into clinical practice, although in future they may be able to complement the prognostic value of clinical and pathologic variables.
El carcinoma hepatocelular (CHC) es el cáncer hepático primario más frecuente. Además, es el tumor que ha experimentado mayor crecimiento en términos de mortalidad en los EEUU en las últimas dos décadas1. Las áreas geográficas que concentran mayor incidencia se localizan en el África subsahariana y en Asia, particularmente en China y Mongolia2. Esta elevada incidencia se asocia a elevadas tasas de infección por el virus de la hepatitis B y exposición al carcinógeno aflatoxina B1. A diferencia de la mayoría de tumores sólidos, más del 80% de los pacientes con CHC presentan lesión hepática subyacente, principalmente cirrosis o hepatitis crónica. En EE.UU. y Europa occidental, la infección por el virus de la hepatitis C es el principal factor etiológico para el desarrollo de CHC. Diferentes estimaciones sugieren que la esteatohepatitis no alcohólica, enfermedad asociada a la obesidad y al síndrome metabólico, tendrá un papel etiológico muy relevante en las próximas décadas3.
A pesar de la recomendación de implementar programas de cribado4,5, la mayoría de los pacientes con CHC se siguen diagnosticando en estadios intermedio/avanzado según la clasificación del grupo Barcelona Clinic Liver Cancer (BCLC)6. Esto imposibilita la aplicación de terapias potencialmente curativas (por ejemplo, resección, trasplante o ablación tumoral percutánea) y se traduce en un reducido pronóstico vital. Recientemente, el ensayo clínico en fase 3 con el inhibidor multiquinasa sorafenib demostró un incremento significativo en la supervivencia en pacientes con CHC en estadio avanzado7. Su aprobación por parte de la Food and Drug Administration (FDA) y la European Medicines Agency marcó un hito, ya que es el primer fármaco sistémico aprobado para esta indicación. El impacto de este estudio fue doble: por un lado estableció un nuevo tratamiento estándar para los pacientes en estadio avanzado; y por otro sirvió para afianzar el diseño racional de fármacos, concepto que prima el conocimiento de la patogenia molecular del cáncer como base para el desarrollo de nuevos fármacos8.
En 2001 se publicó el primer borrador del Proyecto Genoma Humano9. Este ambicioso estudio perseguía obtener la secuencia nucleotídica completa del ADN humano, y suposo el inicio de una nueva era en la investigación biomédica. Esta nueva etapa se caracteriza, entre otros, por un modelo de obtención de datos genómicos basado en tecnologías de alta densidad (por ejemplo, microarrays). Estas plataformas permiten conocer los niveles de expresión génica, la secuencia de nucleótidos o los cambios epigenéticos de forma simultánea en decenas de miles de genes que, en ocasiones, pueden llegar a abarcar el genoma completo del individuo. Su aplicación en medicina translacional ha significado el desarrollo de firmas genéticas que caracterizan un determinado fenotipo. En el CHC se han descrito numerosas firmas genéticas con potencial impacto pronóstico, pero hasta la fecha ninguna ha sido incorporada en las guías de práctica clínica. Esta revisión analizará el estado actual de los marcadores pronósticos genómicos en el CHC, y proporcionará algunas claves para su futura implementación en el manejo de estos pacientes.
Evaluación pronóstica en el carcinoma hepatocelularEn general, un sistema ideal de estadificación debería proporcionar información pronóstica, permitir una óptima asignación terapéutica y facilitar la homogeneización de criterios de inclusión en protocolos de investigación. De manera singular con respecto a otros tumores, en la mayoría de pacientes con CHC coexisten 2 enfermedades potencialmente letales: cáncer y cirrosis. Ello obliga a que cualquier algoritmo pronóstico en CHC deba contener variables que capten información referente al grado de disfunción hepatocelular. Existen diferentes sistemas pronósticos en el CHC, que incluyen variables relacionadas con la carga tumoral, la disfunción hepática y la sintomatología10. Un estudio comparativo demostró que el sistema BCLC, que clasifica a los pacientes en 5 estadios, presentaba la mayor capacidad discriminativa pronóstica en pacientes con CHC en países occidentales11. Actualmente, este algoritmo diagnóstico y terapéutico ha sido adoptado por el Ministerio de Sanidad como guía de práctica clínica12.
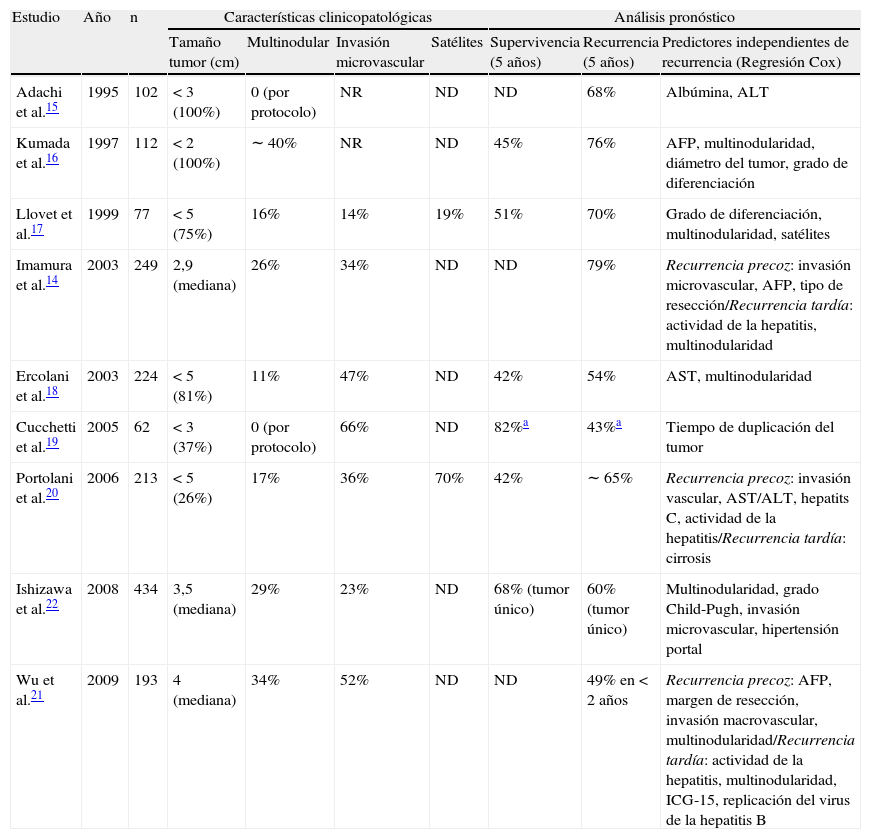
Según el algoritmo BCLC, las terapias con intención curativa (por ejemplo, resección, trasplante y ablación percutánea) se reservan a pacientes en estadios iniciales (BCLC 0-A), obteniendo supervivencias a los 5 años en torno al 70%13. La resección está indicada en pacientes con tumores únicos y ausencia de hipertensión portal clínicamente significativa. La recurrencia tumoral es un factor limitante ya que alcanza el 70% a los 5 años. Existen 2 patrones de recurrencia en el CHC. Uno es debido a la presencia de metástasis no detectadas tras la resección del tumor principal (recurrencia temprana) y otro relacionado con el desarrollo de un nuevo tumor metacrónico en el contexto de la cirrosis hepática (recurrencia tardía). Se ha establecido un criterio temporal arbitrario de 2 años tras la resección para diferenciarlas, y existen diferentes variables clínico-patológicas que orientan hacia el desarrollo de una u otra14. De todas formas, el criterio de clonalidad que permitiría diferenciarlas de forma inequívoca no se aplica en la práctica clínica. No obstante, la distinción entre ambos tipos de recurrencia no tiene fines exclusivamente académicos. Es muy probable que la aplicación de terapias personalizadas exija conocer con precisión la naturaleza de la recurrencia, ya que la patogenia molecular responsable de cada una puede ser diferente. Numerosos estudios han evaluado las variables clinicopatológicas que determinan el riesgo de recurrencia tumoral (tabla 1)14–22. Como predictores independientes destacan la multinodularidad, la invasión microvascular y la satelitosis. La mayoría de estos estudios se realizaron sobre pieza quirúrgica, lo que imposibilita la evaluación de muchas de estas variables antes de la cirugía (por ejemplo, invasión microvascular). A pesar de que gran parte de las firmas genéticas pronósticas en CHC se desarrollaron sobre muestras de tejido fresco congelado, actualmente es factible realizar estudios genómicos de alta densidad en muestras de tejido parafinado posibilitando su aplicación sistemática en biopsias de rutina. Este salto cualitativo permitirá realizar pronósticos basados en firmas genéticas antes de la intervención quirúrgica. Una aplicación interesante podría ser la posibilidad de predecir preoperatoriamente la presencia de invasión microvascular mediante firmas genéticas23.
Predictores clinicopatológicos de recurrencia tumoral tras resección quirúrgica del carcinoma hepatocelular
| Estudio | Año | n | Características clinicopatológicas | Análisis pronóstico | |||||
| Tamaño tumor (cm) | Multinodular | Invasión microvascular | Satélites | Supervivencia (5 años) | Recurrencia (5 años) | Predictores independientes de recurrencia (Regresión Cox) | |||
| Adachi et al.15 | 1995 | 102 | < 3 (100%) | 0 (por protocolo) | NR | ND | ND | 68% | Albúmina, ALT |
| Kumada et al.16 | 1997 | 112 | < 2 (100%) | ∼ 40% | NR | ND | 45% | 76% | AFP, multinodularidad, diámetro del tumor, grado de diferenciación |
| Llovet et al.17 | 1999 | 77 | < 5 (75%) | 16% | 14% | 19% | 51% | 70% | Grado de diferenciación, multinodularidad, satélites |
| Imamura et al.14 | 2003 | 249 | 2,9 (mediana) | 26% | 34% | ND | ND | 79% | Recurrencia precoz: invasión microvascular, AFP, tipo de resección/Recurrencia tardía: actividad de la hepatitis, multinodularidad |
| Ercolani et al.18 | 2003 | 224 | < 5 (81%) | 11% | 47% | ND | 42% | 54% | AST, multinodularidad |
| Cucchetti et al.19 | 2005 | 62 | < 3 (37%) | 0 (por protocolo) | 66% | ND | 82%a | 43%a | Tiempo de duplicación del tumor |
| Portolani et al.20 | 2006 | 213 | < 5 (26%) | 17% | 36% | 70% | 42% | ∼ 65% | Recurrencia precoz: invasión vascular, AST/ALT, hepatits C, actividad de la hepatitis/Recurrencia tardía: cirrosis |
| Ishizawa et al.22 | 2008 | 434 | 3,5 (mediana) | 29% | 23% | ND | 68% (tumor único) | 60% (tumor único) | Multinodularidad, grado Child-Pugh, invasión microvascular, hipertensión portal |
| Wu et al.21 | 2009 | 193 | 4 (mediana) | 34% | 52% | ND | ND | 49% en < 2 años | Recurrencia precoz: AFP, margen de resección, invasión macrovascular, multinodularidad/Recurrencia tardía: actividad de la hepatitis, multinodularidad, ICG-15, replicación del virus de la hepatitis B |
ND: no descrito.
Los pacientes no candidatos a resección que presenten un tumor único o 3 de menos de 3 cm (criterios de Milán) son candidatos a trasplante hepático. Estos pacientes, en estadio inicial según la clasificación BCLC, tienen supervivencias del 70% a los 5 años tras el trasplante24. Ha habido diferentes intentos de expandir estos criterios incrementando el umbral de carga tumoral25. Dado que los resultados son inferiores cuando se comparan con los obtenidos con los criterios de Milán, la expansión de criterios todavía no se ha generalizado26. A pesar de que existen datos indirectos que sugieren que algunos tumores excediendo los criterios de Milán pueden tener un comportamiento biológico más benigno, no es posible detectarlos siguiendo criterios basados en el tamaño o número de lesiones. Es probable que el conocimiento de los determinantes moleculares que favorecen la progresión tumoral permita estimar de forma más fiable la agresividad de estos tumores. Los pacientes en estadio inicial también pueden recibir terapias de ablación tumoral percutánea (por ejemplo, inyección de alcohol o radiofrecuencia), cuando no sean candidatos a tratamiento quirúrgico.
El estadio intermedio (BCLC-B) viene definido por enfermedad multinodular asintomática, sin invasión macrovascular ni diseminación a distancia. La historia natural de estos pacientes se corresponde con una mediana de supervivencia de 16 meses que se incrementa a 22-25 con la aplicación de quimioembolización transarterial (TACE). Existen otras modalidades de tratamiento locorregional, como la radioembolización con microesferas de ytrium-9027, pero faltan estudios para determinar el potencial beneficio de su incorporación en el algoritmo terapéutico del CHC. Los pacientes en estadio avanzado (BCLC-C), que se define por la presencia de enfermedad tumoral fuera del parénquima hepático (invasión macrovascular o metástasis a distancia), son candidatos a recibir sorafenib (400mg/12 h). La mediana de supervivencia en pacientes tratados con sorafenib es de aproximadamente 11 meses7. Por último, los pacientes en estadio terminal (BCLC-D), con una expectativa vital inferior a una mediana de 3 meses son subsidiarios de medidas paliativas.
Firmas genéticas en el carcinoma hepatocelularUna firma o huella genética es un conjunto de genes capaces de clasificar o caracterizar una determinada condición, y abarca desde firmas para el diagnóstico y pronóstico de enfermedad, hasta las empleadas para predecir respuesta terapéutica28. En 2003, Ye et al. publicaron la primera firma genética con impacto pronóstico en CHC29. El estudio analizaba muestras obtenidas en 40 pacientes y describió una firma asociada al riesgo de desarrollar metástasis intrahepáticas tras la resección del tumor primario. Entre los genes presentes en la firma destacaba la osteopontina (SPP1). Desde entonces, se han reportado más de 20 firmas genéticas capaces de predecir pronóstico en CHC. A pesar de que todas ellas fueron obtenidas en pacientes tratados con resección quirúrgica, existe cierta heterogeneidad en términos de estadio, etiología de la enfermedad hepática subyacente y plataformas genómicas empleadas. Según lo comentado previamente, no es de extrañar que algunas de estas firmas se generaron en el tejido cirrótico no tumoral30,31, dada la relevancia pronóstica de la enfermedad hepática subyacente. La progresión en la disfunción hepática y el riesgo de desarrollar un recurrencia tardía viene determinado por las alteraciones genómicas codificadas en el tejido cirrótico no tumoral. La importancia de integrar información pronóstica genómica de ambos compartimentos (tumor y tejido cirrótico no tumoral) quedó de manifiesto en un reciente estudio que determinó el grado de solapamiento en términos de predicción pronóstica de las firmas publicadas hasta la fecha32. Este estudio evidenció un alto grado de concordancia entre la mayor parte de las firmas genéticas evaluadas. Además, el trabajo identificaba cómo la combinación de 2 firmas genéticas, G333 (del tejido tumoral) y poor-survival31 (del tejido cirrotico adyacente), en asociación con la presencia de satelitosis mejoraba de forma significativa la predicción de recurrencia tumoral. No obstante, se debe tener en cuenta que todos estos estudios se realizaron en pacientes tratados mediante resección hepática, por lo que se desconoce el comportamiento pronóstico de las firmas genéticas en pacientes con estadios más avanzados. Es más, no se puede descartar que la diseminación de la enfermedad se acompañe de una modificación sustancial de las alteraciones genómicas que determinan el pronóstico en pacientes con enfermedad avanzada.
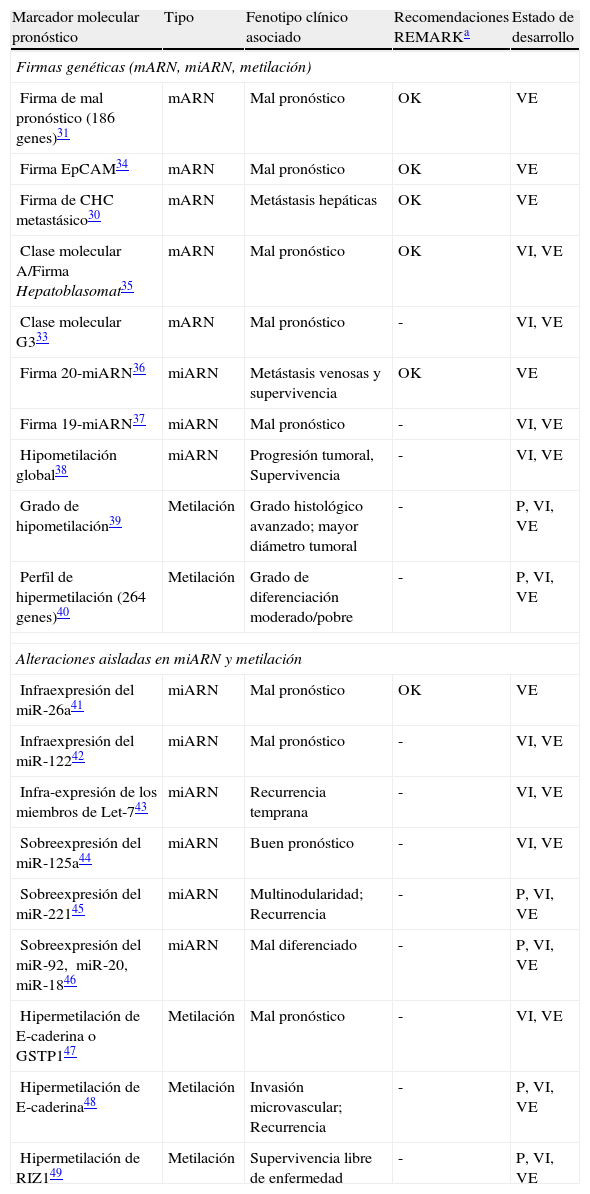
La predicción pronóstica basada en parámetros moleculares de alta densidad no se restringe al análisis del ácido ribonucleico mensajero (ARNm). Numerosos trabajos han evaluado el rol de los micro-ARN (miARN) o de los perfiles de metilación del ADN para clasificar tumores y su pronóstico (tabla 2)30,31,33–49. Los miARN son moléculas de ARM monocatenario que regulan la expresión génica celular mediante fenómenos de ribointerferencia. Un estudio sugiere que los miARN son capaces de clasificar tumores incluso con mayor precisión que el mARN50. Al igual que ocurre en otros tumores, existe una marcada desregulación de miARN en el CHC51,52. De hecho, la disminución en los niveles de miR-26 en CHC se ha correlacionado con peor supervivencia de los pacientes a los que se ha realizado resección quirúrgica41. La identificación de perfiles de miARN capaces de predecir el pronóstico tiene una potencial vertiente terapéutica, ya que se están desarrollando moléculas que permiten modular la expresión de los niveles de miARN53. Otro mecanismo de regulación de la expresión génica es la metilación del ADN, que consiste en la adición de un grupo metilo a determinadas bases nitrogenadas. Numerosos procesos celulares están controlados por fenómenos de metilación (por ejemplo, desarrollo embrionario) y es una de las fuentes principales de plasticidad fenotípica. A pesar de que el desarrollo de firmas pronósticas basadas en perfiles de metilación ha sido menor en comparación con el transcriptoma, hay evidencia clara de su capacidad para clasificar tumores54. Asimismo, existen fármacos capaces de revertir cambios en la metilación que están en evaluación clínica en fases iniciales en algunos tumores.
Firmas genéticas/alteraciones aisladas en miARN y metilación con impacto pronóstico en el carcinoma hepatocelular
| Marcador molecular pronóstico | Tipo | Fenotipo clínico asociado | Recomendaciones REMARKa | Estado de desarrollo |
| Firmas genéticas (mARN, miARN, metilación) | ||||
| Firma de mal pronóstico (186 genes)31 | mARN | Mal pronóstico | OK | VE |
| Firma EpCAM34 | mARN | Mal pronóstico | OK | VE |
| Firma de CHC metastásico30 | mARN | Metástasis hepáticas | OK | VE |
| Clase molecular A/Firma Hepatoblasomat35 | mARN | Mal pronóstico | OK | VI, VE |
| Clase molecular G333 | mARN | Mal pronóstico | - | VI, VE |
| Firma 20-miARN36 | miARN | Metástasis venosas y supervivencia | OK | VE |
| Firma 19-miARN37 | miARN | Mal pronóstico | - | VI, VE |
| Hipometilación global38 | miARN | Progresión tumoral, Supervivencia | - | VI, VE |
| Grado de hipometilación39 | Metilación | Grado histológico avanzado; mayor diámetro tumoral | - | P, VI, VE |
| Perfil de hipermetilación (264 genes)40 | Metilación | Grado de diferenciación moderado/pobre | - | P, VI, VE |
| Alteraciones aisladas en miARN y metilación | ||||
| Infraexpresión del miR-26a41 | miARN | Mal pronóstico | OK | VE |
| Infraexpresión del miR-12242 | miARN | Mal pronóstico | - | VI, VE |
| Infra-expresión de los miembros de Let-743 | miARN | Recurrencia temprana | - | VI, VE |
| Sobreexpresión del miR-125a44 | miARN | Buen pronóstico | - | VI, VE |
| Sobreexpresión del miR-22145 | miARN | Multinodularidad; Recurrencia | - | P, VI, VE |
| Sobreexpresión del miR-92, miR-20, miR-1846 | miARN | Mal diferenciado | - | P, VI, VE |
| Hipermetilación de E-caderina o GSTP147 | Metilación | Mal pronóstico | - | VI, VE |
| Hipermetilación de E-caderina48 | Metilación | Invasión microvascular; Recurrencia | - | P, VI, VE |
| Hipermetilación de RIZ149 | Metilación | Supervivencia libre de enfermedad | - | P, VI, VE |
P: evaluación pronóstica preliminar; VE: carece de validación externa; VI: carece de validación interna
Adaptada de Villanueva et al.65.
No existe un consenso uniforme sobre los requisitos que debe cumplir un biomarcador pronóstico para ser incluido en guías de práctica clínica y recomendado para ser utilizado de forma sistemática. En el caso de nuevos fármacos, se han desarrollado sistemas de jerarquización de la evidencia científica y propuestas de gradación en las recomendaciones55. Hay poca controversia cuando los datos son generados en ensayos clínicos que tienen los más altos estándares de calidad en el diseño: estudio aleatorizado, controlado, en fase III, con una correcta estratificación de pacientes y cuyo objetivo primario es la supervivencia global. El debate surge cuando el diseño del estudio no permite alcanzar este nivel de evidencia. Existen una serie de recomendaciones sobre el proceso de publicación o comunicación de nuevos biomarcadores pronósticos en cáncer, englobadas dentro del consenso REMARK (Reporting recommendations for tumor marker prognostic studies)56. El objetivo de las recomendaciones es establecer un marco general sobre el que uniformizar estos estudios y facilitar un análisis más riguroso de los resultados. El consenso incluye recomendaciones a diferentes niveles: diseño, características de los pacientes, recogida de muestras, análisis de datos, presentación de resultados, etc. (tabla 3). Cuando se evalúa un nuevo biomarcador, se recomienda la realización de un diseño tipo training-validation, así como incluir en el análisis multivariado todas aquellas variables clinicopatológicas con impacto pronóstico conocido en el tumor a estudio. Además, para incrementar la solidez del predictor, es muy recomendable que haya sido validado de forma independiente por un grupo externo al que generó la hipótesis. Hasta el momento, ninguna de las firmas genéticas o biomarcadores basados en datos genómicos se han incorporado en las guías de práctica clínica en el CHC, aunque alguno de ellos se encuentre en avanzado estado de desarrollo preclínico (tabla 2).
Resumen de las recomendaciones para publicación de estudios con biomarcardores pronóstico en cáncer según el consenso REMARK
| 1. Diseño del estudio: |
| a) Características de los pacientes: criterios de inclusión/exclusión, comorbilidades, datos epidemiológicos, estadio tumoral y tratamientos recibidos. Consideraciones éticas (consentimiento informado y aprobación del comité ético) |
| b) Descripción de las muestras: tipo de material, forma de preservación, protocolo de procesamiento (kits de extracción, reactivos, reproducibilidad, controles). Estimación de calidad del material |
| c) Metodología preespecificada: descripción de hipótesis y objetivos. Inclusión retrospectiva o prospectiva. Estratificación. Emparejamiento. Periodo y tiempo de seguimiento. Número y descripción de eventos. Variables clinicopatológicas incluidas. Cálculo tamaño muestral |
| d) Plan de análisis: descripción de todos los métodos estadísticos, asunciones en los modelos, manejo de datos perdidos, especificación de subanálisis. Justificación de valores de corte para el biomarcador |
| 2. Descripción de resultados: |
| a) Presentación de datos: diagrama de flujo, causas de exclusión de pacientes, eventos y seguimiento por grupo según biomarcador. Estadísticos descriptivos globales y agrupados según biomarcador. Predictores pronósticos por grupo |
| b) Análisis estadístico: relación entre el biomarcador y otras variables pronósticas. Diferencias basales entre los grupos según biomarcador. Análisis univariado con estimación de efecto (HR o probabilidad de presentar el evento). Figura Kaplan-Meier incluyendo pacientes en riesgo. Análisis multivariado con estimación de efecto (HR con intervalo de confianza) y variables incluidas en el modelo final. Estimación de efecto combinando biomarcador y variables pronóstico estándar. Descripción de análisis de sensibilidad y validación interna |
Adaptada y modificada de McShane et al.56.
Un factor limitante a tener en cuenta en la estimación pronóstica basada en firmas genéticas obtenidas en biopsias es la heterogeneidad molecular intratumoral. Datos preliminares generados con ultrasecuenciación en CHC y otros cánceres demuestran cierta variabilidad intratumoral en las alteraciones genéticas. Esto afecta fundamentalmente a mutaciones, pero es factible que también influya en otras aberraciones cromosómicas (por ejemplo, inserciones, deleciones, translocaciones, etc.). Totoki et al. analizaron la secuencia completa de ADN y del exoma de un paciente con CHC y, entre otras alteraciones, identificaron mutaciones en el gen TSC1, que actúa inhibiendo la señal de la vía de mTOR57. Mediante técnicas de genotipificación digital estimaron que dicha mutación afectaba a cerca del 15% de la masa celular del tumor. Esto plantea 2 cuestiones que deben tenerse en cuenta cuando se interpreten resultados de firmas genéticas sobre muestras de biopsia: a) la posibilidad de no identificar alteraciones poco prevalentes debido al error de muestreo inherente a la biopsia, y b) el desconocimiento del rol específico de dichas alteraciones en la progresión tumoral. Un estudio piloto sugiere que el impacto de la heterogeneidad tumoral en las predicciones de firmas genéticas en tumores resecables puede ser limitada32, pero son necesarios estudios diseñados ad hoc para establecerlo de forma definitiva.
Biomarcardores de respuesta terapéutica: addicción oncogénicaLas terapias moleculares dirigidas están generando un nuevo modelo para el desarrollo de fármacos en oncología. Desde que en 2001 se introdujo el imatinib para el tratamiento de pacientes con leucemia mieloide crónica, el uso de las terapias moleculares se ha extendido a numerosos tumores sólidos8. Los resultados preliminares en ensayos clínicos fase 2 generaron grandes expectativas, y prueba de ello fue la aprobación de algunos de estos fármacos por la FDA por vía acelerada: gefitinib (inhibidor EGFR) en el cáncer de pulmón, bevacizumab (anticuerpo monoclonal contra VEGF). Sin embargo, los resultados a medio plazo no han sido acordes con las expectativas iniciales. Prueba de ello es la reciente retirada de la indicación del bevacizumab para el tratamiento del cáncer de mama por parte de la FDA, tras los resultados en fase 3, un hecho sin precedentes hasta la fecha58. El error conceptual radica en considerar el cáncer como una entidad homogénea. Hoy sabemos que los pacientes con cáncer de pulmón con mutaciones en el receptor del EGF son los que mejor responden a los inhibidores selectivos gefitinib o erlotinib59. Ocurre algo similar con las mujeres con cáncer de mama que presentan amplificaciones de HER2 y responden a un anticuerpo monoclonal que bloquea selectivamente dicho receptor (trastuzumab)60. Estas aberraciones genómicas que dominan la proliferación tumoral y cuyo bloqueo selectivo impacta sobre la progresión (adicción oncogénica) son una fuente de nuevas dianas terapéuticas. El diseño de ensayos clínicos usando subpoblaciones enriquecidas en pacientes con una determinada adicción oncogénica ya son una realidad. El vemurafenib, que inhibe selectivamente una forma mutada de BRAF, ha demostrado incrementar de forma significativa la supervivencia en pacientes con melanoma metastásico61. Este ensayo clínico en fase 3 fue diseñado para incluir solo pacientes que presentasen este biomarcador de respuesta al vemurafenib (BRAF mutado).
Las terapias moleculares también han mostrado su eficacia en el CHC. El sorafenib, un inhibidor con actividad bloqueante sobre diferentes quinasas (por ejemplo, BRAF, VEGFR, PDGFR), incrementa de forma significativa la supervivencia de los pacientes con CHC avanzado7,62. No hay datos concluyentes sobre el mecanismo principal que condiciona la respuesta al sorafenib. Se especula sobre el perfil antiangiogénico y el bloqueo sobre la vía RAS/MAPK como 2 de los mecanismos más destacados. El estudio en fase 2 identificó mayor respuesta a sorafenib en aquellos tumores con fosforilación incrementada de ERK, considerado un marcador de activación de la vía RAS/MAPK63. Estos resultados están pendientes de ser confirmados en el ensayo clínico en fase 3. Desgraciadamente, todavía no se ha identificado ningún fenómeno de adición oncogénica en CHC. Además, los estudios realizados hasta la fecha muestran una baja tasa mutacional de receptores tirosin-quinasa en el CHC. Los genes más frecuentemente mutados en CHC son TP53 y CTNNB1 (que codifica para ß-catenina)64, cuyos efectos son de difícil modulación farmacológica. A priori puede ser complicado identificar nuevas mutaciones recurrentes susceptibles de intervención farmacológica. No ocurre lo mismo con las aberraciones cromosómicas (muy marcadas en el CHC en comparación con otros tumores), las alteraciones epigenéticas o los fenómenos de fusión, ambos infraevaluados hasta la fecha en el CHC.
Conflicto de interesesAugusto Villanueva ha recibido honorarios como consultor de Bayer Pharmaceuticals.