Las sustituciones asociadas a resistencia (RAS) a elbasvir, el nuevo inhibidor de la NS5A del virus de la hepatitis C (VHC), pueden presentar relevancia al limitar su eficacia y conducir al fracaso virológico en pacientes infectados por VHC genotipo 1a (GT1a) a diferencia de lo observado en GT1b y GT4. No existen datos fuera de ensayos clínicos que evalúen la prevalencia y el impacto de grazoprevir/elbasvir en pacientes infectados con GT1a en España. Se llevó a cabo un estudio transversal multicéntrico en 632 pacientes iniciales, en 13 de los cuales no se consiguió amplificar la muestra o no fue válida para alcanzar una secuencia consenso mediante secuenciación de Sanger. Finalmente, se analizaron 617 individuos infectados con VHC-GT1a atendidos en 84 hospitales distribuidos por las 17 comunidades autónomas más las 2 ciudades autónomas que conforman el territorio español. La población de VHC secuenciada se ha usado para identificar RAS a elbasvir, mientras que el patrón mutacional y la sensibilidad farmacológica se confirmaron mediante geno2pheno[HCV]. Los virus portadores de RAS a elbasvir se observaron en el 6,2% de los pacientes infectados con el VHC-G1a. Las RAS más comunes fueron Y93C/H/N y Q30E/H/R (2,4 y 2,3%, respectivamente). Solo el 3,4% de las RAS a elbasvir identificadas confirieron susceptibilidad reducida al fármaco mediante geno2pheno[HCV] que identificó exclusivamente como principales RAS a elbasvir las posiciones Q30H/R (n=7) y Y93C/H/N (n=8) como mutaciones simples y Q30H+Y93H (n=4) y Q30R+Y93H (n=2) como mutaciones dobles. En nuestra cohorte española con VHCG1a se observó una menor prevalencia de RAS a elbasvir que la reportada previamente en ensayos clínicos realizados en pacientes norteamericanos. Esta información puede ser esencial para el manejo de los pacientes infectados con G1a y guiar la implementación de grazoprevir/elbasvir en España.
Resistance-associated substitutions (RASs) to the new HCV NS5A inhibitor elbasvir may limit its efficacy and lead to virological failure in HCV-GT1a-infected patients. There are no data outside clinical trials evaluating their prevalence and impact in grazoprevir/elbasvir in GT1a-infected patients in Spain. A multicentre cross-sectional study of 632 initial patients was conducted. In 13 of these patients, the sample could not be amplified or a consensus sequence by Sanger sequencing could not be performed. Ultimately, 617 HCV-G1a-infected individuals treated at 84 Spanish hospitals from the 17 autonomous communities plus the 2 autonomous cities of Spain were analysed. HCV population sequencing was used to identify RAS to elbasvir and the mutational pattern and drug sensitivity were confirmed by geno2pheno[HCV]. Viruses bearing RASs to elbasvir were present in 6.2% of HCV-G1a infected patients. The most common RASs were the Y93C/H/N and Q30E/H/R (2.4% and 2.3%, respectively). Only 3.4% of the identified RASs to elbasvir conferred reduced susceptibility to elbasvir by geno2pheno[HCV], which exclusively identified the positions Q30H/R (n=7) and Y93C/H/N (n=8) as single mutations and Q30H+Y93H (n=4) and Q30R+Y93H (n=2) as double mutations as the major RASs to elbasvir. A lower prevalence of RASs to elbasvir was observed in our HCV-G1a Spanish cohort than reported previously in clinical trials evaluating patients from the USA. This information may be essential to guide the implementation of grazoprevir/elbasvir in Spain and to manage G1a-infected patients.
Grazoprevir/elbasvir (EBR) fue aprobado por la Food and Drug Administration en enero de 20161. En la Unión Europea, la Agencia Europea de Medicamentos lo aprobó en julio de 2016 y fue incluido en la lista de medicamentos sujetos a seguimiento adicional2. En España, a 1 de septiembre de 2016 el Ministerio de Sanidad aprobó la financiación pública de este medicamento. Este fármaco representa el nuevo producto de combinación de antivirales de acción directa (AAD) para tratar el virus de la hepatitis C (VHC) incorporado al arsenal terapéutico y ha sido aprobado para su uso en GT1 y GT41. El uso de grazoprevir/EBR no está indicado en pacientes con infección por los genotipos 2, 3, 5 y 6. Grazoprevir pertenece a la familia de inhibidores de la proteasa NS3/4A del VHC y EBR representa el primer inhibidor de segunda generación de la NS5A del VHC. Sin embargo, la presencia de sustituciones asociadas a resistencia (RAS) puede reducir la sensibilidad a los AAD, limitando su eficacia. Estudios previos han mostrado que la presencia de RAS en la región NS3 no afecta a la eficacia de grazoprevir en pacientes previamente no tratados con los nuevos AAD o pacientes que nunca recibieron tratamiento3. Sin embargo, RAS en la región NS5A tienen un fuerte impacto en la eficacia de EBR.
El impacto de las variantes preexistentes del VHC en la respuesta a la terapia con grazoprevir/EBR en sujetos infectados con VHC que nunca han recibido tratamiento ha sido evaluado en el estudio C-EDGE TN, en el cual se identificaron RAS a EBR en el 12% (19/154) de los participantes portadores del genotipo 1a (GT1a) y se han asociado a una reducción del 58% (11/19) de la respuesta viral sostenida a las 12 semanas3. La recomendación de extender a 16 semanas el tratamiento con grazoprevir/EBR con la inclusión de ribavirina para pacientes VHC-GT1a nunca tratados y con RAS en NS5A se basa en los resultados del estudio C-EDGE TE4. Específicamente, las variantes de base NS5A con un cambio 5 veces mayor a EBR redujeron la respuesta viral sostenida a las 12 semanas al 52,4% (11/21) en sujetos infectados con el VHC-GT1a, mientras que la respuesta viral sostenida a las 12 semanas se incrementó en el 94,8% (55/58) en el análisis por intención de tratar cuando se añadió ribavirina a grazoprevir/EBR y el tratamiento se extendió a las 16 semanas. En pacientes infectados con VHC-GT1b la eficacia de grazoprevir/EBR fue del 99 y del 100%, respectivamente, en individuos infectados con VHC-GT43.
El fallo en la terapia con EBR en pacientes infectados con VHC-GT1a se ha asociado a la presencia de sustituciones específicas M28A/G/T, Q30D/E/G/H/K/L/R, L31F/M/V, H58D y Y93C/H/N/S, de acuerdo con las Guías Europeas de 20165, las cuales representan un subconjunto de RAS en NS5A6. Se ha documentado resistencia cruzada entre las RAS en NS5A a EBR y los inhibidores de NS5A de primera generación aprobados6,7, así como la alta persistencia de estas sustituciones en la población viral una vez establecida8,9. Ambos fenómenos podrían limitar el uso de la familia de inhibidores de NS5A. Con base en este problema, se recomienda el estudio de las resistencias en NS5A en pacientes infectados con VHC-GT1a candidatos a tratamiento con grazoprevir/EBR10.
En España, a 1 de septiembre de 2016 el Ministerio de Sanidad aprobó la financiación pública de este medicamento, y el 3 de febrero de 2017, la Agencia Española de Medicamentos y Productos Sanitarios publicó el Informe de Posicionamiento Terapéutico de EBR y grazoprevir (Zepatier®) en hepatitis C cro¿nica, comenzando de ese modo su implantación oficial11. El genotipo VHC-GT1a representa aproximadamente el 23% de los genotipos circulantes en nuestro país12 y, por tanto, el conocimiento previo de la presencia de RAS en NS5A a EBR en la población española infectada con VHC-GT1a ayudaría a identificar la proporción de pacientes que se pueden beneficiar de este nuevo tratamiento. En el presente estudio se ha realizado un análisis de RAS mediante secuenciación de Sanger en pacientes españoles infectados con VHC-GT1a para evaluar la prevalencia y el impacto de RAS en NS5A clínicamente relevantes al nuevo fármaco inhibidor de NS5A.
MétodosDiseño del estudio y pacientesSe llevó a cabo un estudio transversal y multicéntrico con 632 individuos infectados de forma crónica con VHC-GT1a y nunca tratados con inhibidores NS5A. Como ayuda para el diseño y la dirección del estudio se empleó la lista de verificación STROBE13.
Los pacientes fueron atendidos en 84 centros de salud españoles distribuidos por todo el territorio nacional (ver Apéndice, datos adicionales). Las muestras fueron seleccionadas de manera aleatoria y se recogieron antes del inicio de la terapia anti-NS5A, entre octubre de 2014 y febrero de 2015. Los especímenes de plasma se enviaron junto con un conjunto mínimo de datos a la Unidad de Infección Viral e Inmunidad del Laboratorio de Referencia e Investigación en Hepatitis Víricas del Centro Nacional de Microbiología (Instituto de Salud Carlos III [ISCIII]) a través del Área de Orientación Diagnóstica de dicho centro (ISCIII). Tanto los datos como las muestras se anonimizaron y transfirieron al Biobanco Nacional del ISCIII (Ref.: B.0000984).
ÉticaEl estudio fue llevado a cabo de acuerdo con la Declaración de Helsinki. Fue aprobado por la Junta de Revisión Institucional y el Comité de Ética de la Investigación del ISCIII (CEI PI 43_2015).
Amplificación y secuenciación de NS5AEl ARN viral se extrajo del plasma mediante el kit QIAsymphony DSP Virus/Pathogen (Qiagen, Hilden, Alemania) a partir de 500μl, y la amplificación completa del gen NS5A se realizó con el kit RT-PCR OneStep (Qiagen, Hilden, Alemania) usando los oligonucleótidos NS4BFW (5′TGAGGCGACTVCACCAGTGG3′) y NS5BRV (5′TCTTCCGCGGCRCACGGGGTGA3′). El protocolo de amplificación fue: 30min a 54°C; 15min a 95°C; 35 ciclos repetitivos de 30seg a 94°C, 30seg a 60°C y 2min a 72°C en el termociclador Applied Biosystems Verity TM. En todas las amplificaciones se incluyeron controles positivos y negativos. Los productos de PCR positivos se visualizaron con GelRed (Biotium, Estados Unidos) como una banda específica VHC de ∼1,343pb. Los amplicones se purificaron (illustra™ GFX™ PCR DNA y Gel Band Purification Kit, GE Healthcare, Estados Unidos) y diluyeron 1:2 usando agua libre de nucleasas (Roche). A continuación, la reacción de secuenciación se llevó a cabo con los siguientes oligonucleótidos: FwSc2 (5′CGACTRCACCAGTGGATAAGC3′); FwSc3 (5′CTRCACCAGTGGATAAGCTCG3′); FwSc5 (5′CCCATTAACGCCTACACCACG3′); FwSc7 (5′CCTGACGCCGAGCTCATAGAG3′); RvSc3 (5′AGCGAGTGTGCATGATGCCAT3′); RvSc7 (5′GTGCGCCTGTCCAGGAATAAGA3′) y se realizó la secuenciación de Sanger mediante ABI PRISM® 377 DNA sequencer (Applied Biosystems, Foster City, Estados Unidos).
Análisis bioinformáticoPara la obtención de las secuencias consenso se usó el programa SeqMan (Lasergene DNASTAR Inc., Madison, WI, Estados Unidos), mientras que para alinear las secuencias NS5A con la secuencia representativa VHC-1a H77 se usó el programa MEGA6 (Molecular Evolutionary Genetics Analysis Version 6.0; http://www.megasoftware.net/). El punto de corte de detección de RAS utilizado fue del 15% de acuerdo con las recomendaciones de las Guías Europeas mediante el software Minor Variant Finder (Applied Biosystems). Para determinar la prevalencia de RAS en NS5A a EBR en pacientes que nunca recibieron inhibidores NS5A y con VHC-GT1a se analizó el gen NS5A según las últimas Guías Europeas5. El patrón mutacional de NS5A y la sensibilidad farmacológica se confirmaron mediante geno2pheno[HCV] (g2p[HCV]) (Bonn, Alemania; http://hcv.geno2pheno.org/). Los linajes VHC-GT1a (clado i y clado ii) se identificaron usando g2p[HCV]. El aislado H77 (número de acceso GenBank AF009606) se usó como secuencia de referencia del G1a. El nivel de resistencia a EBR (en número de veces) de cada RAS identificada en nuestra cohorte se evaluó de acuerdo con Black et al. (2015)8 y Cento et al. (2015)14.
Análisis estadísticoLas variables categóricas se analizaron usando la prueba chi-cuadrado o el test exacto de Fisher, mientras que las variables continuas se compararon mediante la prueba U de Mann-Whitney. Los valores de p se obtuvieron mediante prueba bilateral, definiendo la significación estadística como p<0,05. El análisis estadístico se realizó con la versión 22.0 del software SPSS® (IBM Corp, Chicago, Armonk, NY, Estados Unidos).
ResultadosDe los 632 pacientes incluidos en el estudio, 13 (2,1%) tuvieron una muestra que no amplificó o no fue válida para alcanzar una secuencia consenso mediante secuenciación de Sanger. En total, 617 pacientes con VHC-GT1a fueron válidos para el análisis estadístico. En total, el 80,1% (n=494) de los sujetos eran hombres de 50 años de media (47-53). Además, el 52,8% (n=326) eran pacientes monoinfectados con VHC y el 47,2% (n=291) eran pacientes coinfectados VIH/VHC. El clado ii de VHC-GT1a tuvo más representación que el clado i (82,5%, n=509, frente a 17,5%, n=108), y fue más prevalente en individuos monoinfectados con VHC que en coinfectados con VIH/VHC (87,4 frente a 77,0%, p=0,001).
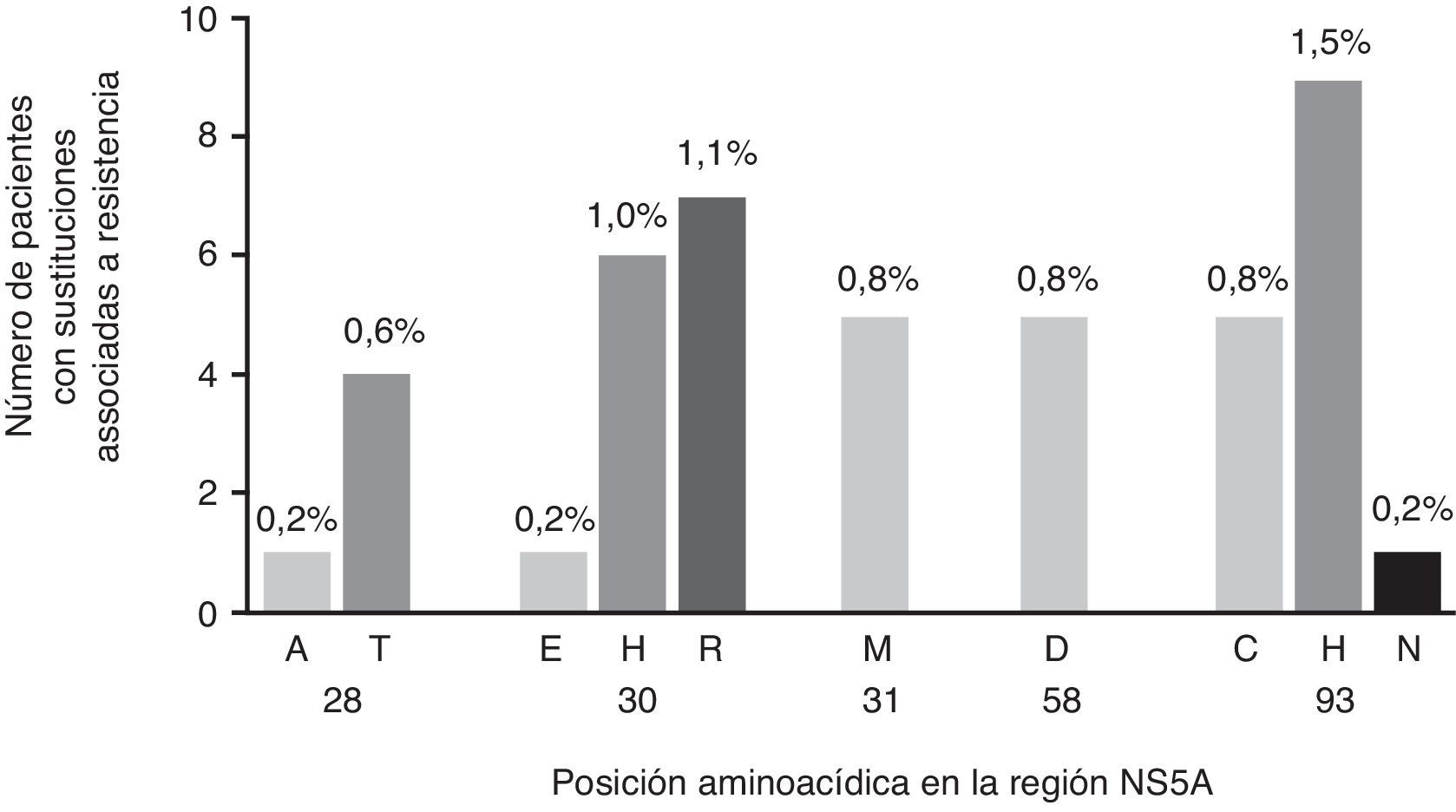
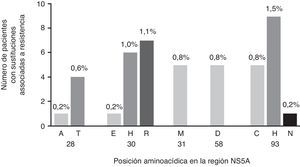
Los virus portadores de RAS a EBR estuvieron presentes en 38 muestras (6,2%) de los pacientes infectados con VHC-GT1a (fig. 1), 6 de las cuales contenían virus con RAS doble. Las RAS más comunes fueron la Y93C/H/N (2,4%; n=15) y la Q30E/H/R (2,3%; n=14), mientras que M28A/T, L31M y H58D tuvieron menor representación (0,8%, n=5, cada una). Las mutaciones dobles Q30H+Y93H y Q30R+Y93H mostraron una frecuencia baja (0,6%, n=4, y 0,3%, n=2, respectivamente) (fig. 1). Curiosamente, solo se identificaron RAS que conferían sensibilidad o susceptibilidad reducida a EBR en 21 de las 38 (55,2%) cepas identificadas con RAS a EBR cuando se analizó mediante g2p[HCV]. Este algoritmo identificó exclusivamente como principales RAS a EBR las posiciones Q30H/R (n=7) y Y93C/H/N (n=8) como mutaciones individuales y Q30H+Y93H (n=4) y Q30R+Y93H (n=2) como mutaciones dobles.
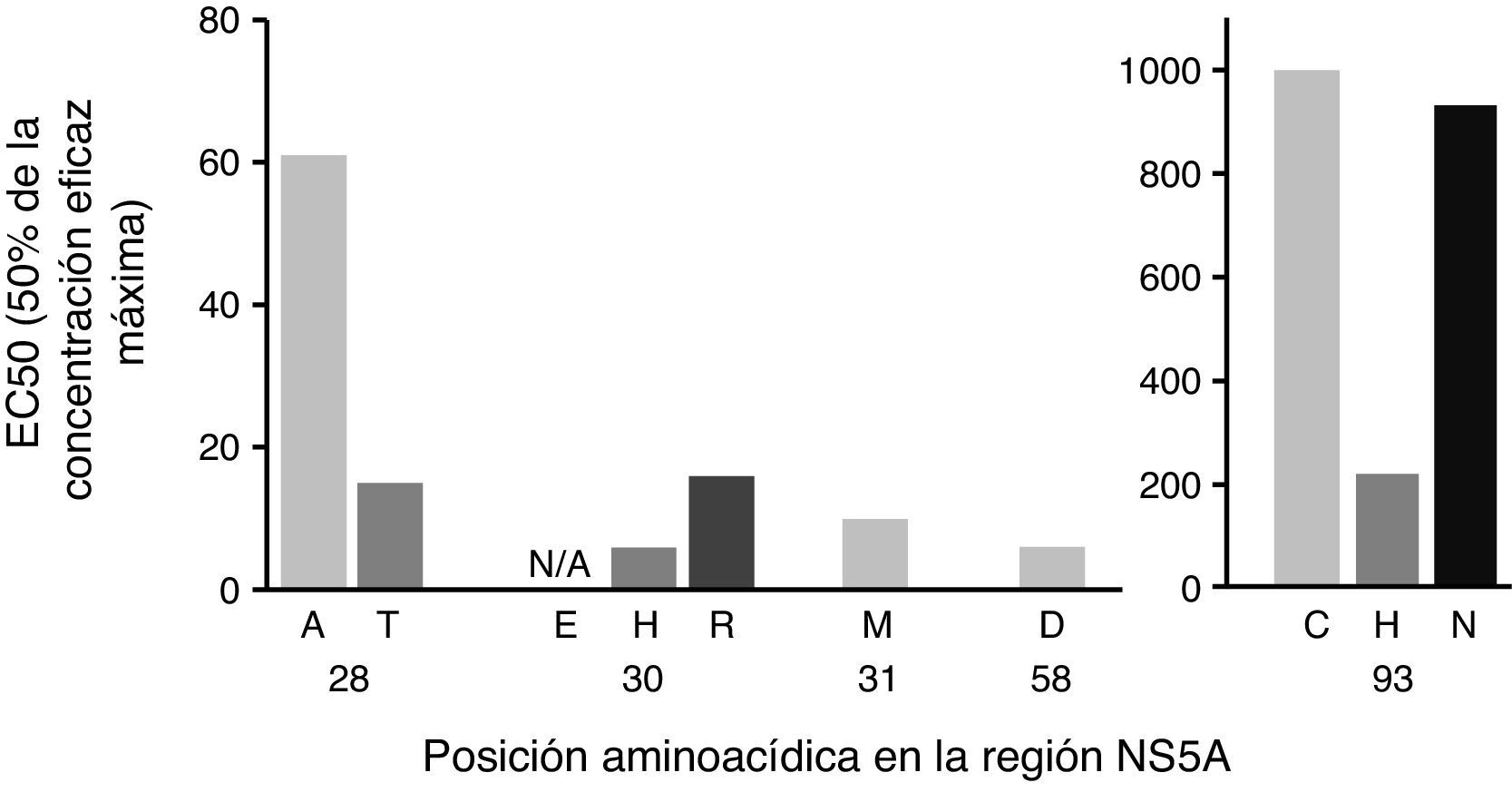
A continuación, se evaluó el impacto de las variantes identificadas de acuerdo con el cambio en el número de veces con el fin de evaluar el peso específico de cada mutación y, por tanto, la relevancia clínica de las variantes encontradas en nuestra cohorte (fig. 2). Las mutaciones identificadas en las secuencias analizadas presentaron un patrón de resistencia a EBR diferente, oscilando desde 6x a 1235x (fig. 2). En particular, identificamos las mutaciones en las posiciones Q30H/R y Y93C/H/N como las que tuvieron el mayor nivel de resistencia a EBR (fig. 2).
Impacto de las variantes de sustitución de NS5A según el cambio en el número de veces. El gen completo de NS5A se secuenció mediante secuenciación de Sanger. La sensibilidad farmacológica se confirmó mediante geno2pheno[HCV]. En 6 individuos se encontraron las mutaciones dobles Q30H+Y93H y Q30R+Y93H. El análisis del cambio en el número de veces se basó en Black et al. 20158 y Cento et al. 201614.
EC50: concentración necesaria para alcanzar el 50% de la replicación viral; NS5A: proteína no estructural 5A.
Posteriormente, se evaluó la influencia de los distintos clados en la presencia de RAS a EBR, pero no se observaron diferencias (clado i 8,3%, n=9/108, frente a clado ii 5,7%, n=29/509; p=0,278). Sorprendentemente, los pacientes infectados con virus que poseían RAS en NS5A que les conferían una susceptibilidad reducida a EBR estaban 2 veces más presentes entre los individuos coinfectados con VIH/VHC que en los monoinfectados con VHC (8,2%, n=24/291, frente a 4,3%, n=14/326; p=0,045).
DiscusiónEn este estudio observamos una baja prevalencia de RAS en NS5A a EBR en pacientes VHC-GT1a no tratados previamente con fármacos de la familia de los inhibidores NS5A. Por lo que sabemos, este es el primer estudio que evalúa la prevalencia de RAS en NS5A en el genotipo VHC-GT1a en España. Nuestros datos de prevalencia de RAS en NS5A a EBR contrastan con el 12% presentado recientemente por Zeuzem et al. (2015) en pacientes infectados con VHC-GT1a nunca tratados, involucrados en el ensayo fase iii C-EDGE3.
El efecto en la respuesta al tratamiento del VHC de las RAS en NS5A a EBR en el mundo real no está claro todavía y su caracterización e impacto en la terapia de primera línea y estrategias de tratamiento siguen siendo un gran desafío. Además, la falta de datos en la práctica clínica nos impide comparar nuestros resultados con los de otros países.
Estudios recientes han dado a conocer las distintas distribuciones de los 2 clados del VHC-GT1a, presentes de forma igualitaria en Europa y siendo el clado i más prevalente en Estados Unidos15,16. En nuestro estudio, observamos una prevalencia mucho mayor del clado ii que del i, representando el primero aproximadamente 4 de cada 5 secuencias analizadas; el segundo es significativamente menos prevalente (17%) que en otros países europeos (48% para secuencias italianas, 53% para alemanas). Esta diferencia filogenética puede indicar la posible diferencia en la propagación temporal de los clados del VHC-GT1a en España en comparación con otros países europeos, donde ambos clados se distribuyen de forma igualitaria. Además, se observó una asociación inversa del clado ii con la presencia de coinfección con VIH, en contraste con los datos publicados por De Luca et al. en 201515. Las diferencias en la prevalencia de RAS en NS5A a EBR según los clados del VHC-GT1a pueden proporcionar alguna explicación de por qué las variantes en pacientes europeos infectados con VHC-GT1a son menos frecuentes que los norteamericanos, donde el clado i es más prevalente15. En nuestra población de estudio, la baja prevalencia de RAS en NS5A a EBR y del clado i del VHC-GT1a no permitió establecer ninguna asociación entre el clado y la presencia de RAS en NS5A. Por lo tanto, se necesitan estudios adicionales con cohortes más grandes para clarificar la emergencia de RAS en NS5A a EBR en relación con los clados del VHC-GT1a.
Las últimas Guías Americanas recomiendan realizar pruebas de resistencia para identificar NS5A RAS en pacientes infectados con VHC-GT1a que inician tratamiento con grazoprevir/EBR10. De hecho, la presencia de RAS en NS5A y su efecto en la respuesta al tratamiento influirán en la futura selección de la terapia. De acuerdo con la baja prevalencia observada en nuestra cohorte, quizás actualmente en España no sea necesario realizar estas pruebas de forma rutinaria. Sin embargo, es probable que este escenario cambie y, por tanto, habría que implantar un sistema de monitorización que nos permita evaluar la necesidad de realizar pruebas de RAS en NS5A en un futuro cercano.
Es importante mencionar que grazoprevir/EBR puede ser el tratamiento más coste-efectivo de los AAD aprobados hasta el momento, lo que probablemente influirá en la elección de la terapia17. Este hecho se suma a la baja prevalencia de RAS en NS5A a EBR observada en nuestra cohorte, lo que sin duda repercutirá en la introducción de grazoprevir/EBR como terapia de primera línea en España.
Se deben tener en cuenta algunas limitaciones del estudio. En primer lugar, la ausencia de datos clínicos no nos ha permitido evaluar el posible impacto de las RAS en NS5A a EBR en la respuesta virológica. No obstante, las RAS clínicamente relevantes se extrapolaron de las Guías Europeas recientemente revisadas5. En segundo lugar, los datos clínicos y epidemiológicos (por ejemplo, el modo de transmisión del VHC, la carga de ARN viral) eran limitados y serían necesarios para la interpretación de los resultados. Por ejemplo, a pesar de que el plan estratégico para combatir el VHC en el Sistema Nacional de Salud español más reciente estableció los criterios generales para el tratamiento con AAD, considerando de alta prioridad a los pacientes con un grado de fibrosis hepática avanzado (F2-F4) y aquellos incluidos en lista de espera para trasplante o ya trasplantados18, no podemos garantizar que todos los pacientes incluidos en el estudio presenten un grado de fibrosis hepática avanzado. A pesar de ello, es importante mencionar que nuestro estudio usa una muestra nacional representativa de toda la población que vive con infección crónica por VHC en España. Esto puede ayudarnos a tener una visión global de la situación actual en la población española, a diferencia de los estudios realizados en hospitales individuales. En tercer lugar, se usó la secuenciación de Sanger (secuenciación de la población de VHC) en lugar de secuenciación masiva para identificar las RAS en NS5A. Aunque la secuenciación de Sanger puede infraestimar ligeramente la prevalencia de RAS en NS5A a EBR, el impacto de las RAS en la eficacia de grazoprevir/EBR parece desaparecer tras administrar durante 16 semanas EBR/grazoprevir+RBV6. Además, la necesidad de un diagnóstico rápido de las RAS en NS5A puede disputar como opción a la secuenciación masiva debido al mayor tiempo de respuesta y coste.
En conclusión, nuestros datos muestran que, en España, entre los pacientes infectados con VHC-GT1a no tratados con inhibidores de la NS5A previamente existe una baja prevalencia de RAS en NS5A a EBR. Esta información puede ser esencial para guiar la implementación de este AAD en nuestro país y para el manejo de los pacientes infectados con VHC-GT1a en un futuro próximo.
FinanciaciónEste trabajo ha sido financiado por el Instituto de Salud Carlos III (Ref. PI14CIII/00011 a SR, MPY 1039/14 y PI15CIII/00031 a VB). CP cuenta con el apoyo de la Fundação para a Ciência e Tecnologia portuguesa (número de subvención SFRH/BPD/77448/2011, parte del programa EDCTP2 financiado por la Unión Europea). VB es financiada por el programa Miguel Servet dirigido por el Fondo de Investigación Sanitaria (Instituto de Salud Carlos III) (número de subvención CP13/00098).
AutoríaVB concibió y diseñó el estudio; MSC, BEC y VB obtuvieron los datos; CP, IMC y VB analizaron e interpretaron los datos; CP, BEC y VB redactaron el manuscrito; SR e IMC realizaron una revisión crítica del artículo; CP, SR y VB aprobaron la versión final. SR y VB contribuyeron de igual modo.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Los autores agradecen la participación a todos los pacientes, y a los centros colaboradores, las muestras clínicas proporcionadas.
Las siguientes personas e instituciones participaron en esta encuesta epidemiológica:
- 1.
Área Sanitaria de Ferrol: P. Ordoñez Barrosa.
- 2.
Centro Médico de Asturias: R. Vilches Vilches.
- 3.
Complejo Asistencial Universitario de Burgos: Gonzalo F. Saez-Royuela.
- 4.
Complejo Hospitalario de Navarra: I. Polo Vigas.
- 5.
Complejo Hospitalario de Orense: G. Esteban Meruéndano.
- 6.
Complejo Hospitalario de Pontevedra: M. Trigo Daporta.
- 7.
Complejo Hospitalario Universitario de Santiago: A. Aguilera Guirao.
- 8.
Complejo Hospitalario Universitario La Coruña: M. A. Cañizares Castellanos.
- 9.
Complejo Hospitalario Xeral-Calde: A. Coira Nieto.
- 10.
Fundación Hospital Alcorcón: M. L. Casas Losada.
- 11.
Fundación Hospital de Jove: E. Hidalgo Pérez.
- 12.
Fundación Jiménez Díaz-Ute: R. Fernández Roblas.
- 13.
Gerencia del Área de Salud de Badajoz, Llerena y Zafra: G. Sánchez Alor.
- 14.
Gestión Sanitaria de Mallorca (GESMA): V. Fernández Baca Gutiérrez del Álamo.
- 15.
Hospital Arnau de Vilanova de Valencia: R. Giner Duran.
- 16.
Hospital Can Misses: A. Hurtado Fernandez.
- 17.
Hospital Central de Asturias: S. Melón Garcia.
- 18.
Hospital Central de la Defensa Gómez Ulla: M. Mateo Maestre.
- 19.
Hospital Clínic i Provincial de Barcelona: F. Marco Reverte.
- 20.
Hospital Clínico San Carlos: E. Culebras Lopez.
- 21.
Hospital Clínico Universitario de Salamanca: M. N. Gutierrez Zufiaurre.
- 22.
Hospital Clínico Universitario de Valladolid: C. Hinojosa Mena Bernal.
- 23.
Hospital Clínico Universitario Lozano Blesa: C. Rubio Calvo.
- 24.
Hospital Comarcal de Inca: J. Saurina Gomila.
- 25.
Hospital de Basurto: S. Hernaez Crespo.
- 26.
Hospital de Cabueñes: L. Otero Guerra.
- 27.
Hospital de Cruces: L. López Soria.
- 28.
Hospital de Donostia: C.G. Cilla Eguiluz.
- 29.
Hospital de Especialidades de Jerez de la Frontera: M. D. López Prieto.
- 30.
Hospital de Especialidades de Puerto Real: I. Jesús de la Calle.
- 31.
Hospital de Gran Canaria Dr. Negrín: M. J. Pena López.
- 32.
Hospital de Hellín: C. Romero Portilla.
- 33.
Hospital de la Agencia Valenciana de Salud Vega Baja: N. Gonzalo Jiménez.
- 34.
Hospital de la Línea de la Concepción: F. J. Casas Ciria.
- 35.
Hospital de Móstoles: F. López Fabal.
- 36.
Hospital de Palamós: N. Guinart Sola.
- 37.
Hospital de Poniente: T. Cabezas Fernández.
- 38.
Hospital do Meixoeiro: S. Pérez Castro.
- 39.
Hospital Don Benito-Villanueva de la Serena: A. Valle Valencia.
- 40.
Hospital El Bierzo: C. Raya Fernández.
- 41.
Hospital Ernest Lluch Martin: B. Fortuño Cebamanos.
- 42.
Hospital Galdakao-Usansolo: M. J. Lopez de Goicoechea San Román.
- 43.
Hospital García Orcoyen: L. Barrado Blanco.
- 44.
Hospital General de Castellón: B. Gomila Sard.
- 45.
Hospital General de Lanzarote: R. Copado Carretero.
- 46.
Hospital General de Segovia: J. Elizaga Corrales.
- 47.
Hospital General Juan Ramón Jiménez: J. Saavedra Martin.
- 48.
Hospital General Río Carrión: T. García Valero.
- 49.
Hospital General San Jorge: L. Torres Sopena.
- 50.
Hospital General Universitario de Alicante: A. Gimeno Gascón.
- 51.
Hospital General Universitario de Elche: M. Ruiz García.
- 52.
Hospital General Universitario Gregorio Marañón: T. Vicente Rangel.
- 53.
Hospital General Universitario Reina Sofía: A. J. Marín Cervantes.
- 54.
Hospital Infanta Cristina (Badajoz): R. Sánchez Silos.
- 55.
Hospital Infanta Elena: M. de la Iglesia Salgado.
- 56.
Hospital J. M. Morales Meseguer: C. Guerrero Gómez.
- 57.
Hospital Mateu Orfila: L. Carbo.
- 58.
Hospital Nuestra Señora de Sonsoles: A. Gómez del Campo Dechado.
- 59.
Hospital Povisa S. A.: M. D. Martínez Otero.
- 60.
Hospital Puerta de Hierro (Majadahonda): F. Portero Azorin.
- 61.
Hospital Rafael Méndez: E. Cascales Alcolea.
- 62.
Hospital Reina Sofía (Córdoba): F. Rodríguez Cantalejo.
- 63.
Hospital Reina Sofía (Tudela): J. J. García Irure.
- 64.
Hospital San Agustín (Asturias): G. Sierra Dorado.
- 65.
Hospital San Pedro: C. Martínez Gil.
- 66.
Hospital Santa Maria Nai: J. Garcia Costa.
- 67.
Hospital Txagorritxu: M. J. Lezaun Bugui.
- 68.
Hospital Universitario Marqués de Valdecilla: J. Crespo García.
- 69.
Hospital Universitario de Canarias: F. Díaz-Flores Estevez.
- 70.
Hospital Universitario de Ceuta: J. López Barba.
- 71.
Hospital Universitario de La Princesa: L. Cardeñoso Domingo.
- 72.
Hospital Universitario Dr. Peset: J. Alberola Enguidanos.
- 73.
Hospital Universitario Insular de Gran Canaria: E. Santana Rodriguez.
- 74.
Hospital Universitario Miguel Servet: A. Martínez Sapiña.
- 75.
Hospital V. Alvarez Buylla: M. C. Galarraga Gay.
- 76.
Hospital Virgen de la Concha: R. Martínez González.
- 77.
Hospital Virgen de la Salud (Toledo): P. Zamarrón Fuertes.
- 78.
Hospital Virgen de la Victoria: E. Clavijo Frutos.
- 79.
Hospital Virgen del Castillo: M. L. López Yepes.
- 80.
Hospital Virgen del Puerto: C. García Tejero.
- 81.
Hospital Virgen del Rocío: L. Merino Díaz.
- 82.
Laboratorio de Referencia del Camp de Tarragona i Terres de l’Ebre: M. J. Puerta Martínez.
- 83.
Laboratorio de Referencia de Catalunya (El Prat de Llobregat): M. Salvado Costa M; G. Soria Guerrero.
- 84.
Laboratorio BR Salud: E. Aznar Cano.





![Impacto de las variantes de sustitución de NS5A según el cambio en el número de veces. El gen completo de NS5A se secuenció mediante secuenciación de Sanger. La sensibilidad farmacológica se confirmó mediante geno2pheno[HCV]. En 6 individuos se encontraron las mutaciones dobles Q30H+Y93H y Q30R+Y93H. El análisis del cambio en el número de veces se basó en Black et al. 20158 y Cento et al. 201614. EC50: concentración necesaria para alcanzar el 50% de la replicación viral; NS5A: proteína no estructural 5A. Impacto de las variantes de sustitución de NS5A según el cambio en el número de veces. El gen completo de NS5A se secuenció mediante secuenciación de Sanger. La sensibilidad farmacológica se confirmó mediante geno2pheno[HCV]. En 6 individuos se encontraron las mutaciones dobles Q30H+Y93H y Q30R+Y93H. El análisis del cambio en el número de veces se basó en Black et al. 20158 y Cento et al. 201614. EC50: concentración necesaria para alcanzar el 50% de la replicación viral; NS5A: proteína no estructural 5A.](https://static.elsevier.es/multimedia/0213005X/0000003600000005/v1_201805030425/S0213005X1730126X/v1_201805030425/es/main.assets/thumbnail/gr2.jpeg?xkr=ue/ImdikoIMrsJoerZ+w96p5LBcBpyJTqfwgorxm+Ow=)


