El incremento de Salmonella enterica multirresistente a los antibióticos, incluidos β-lactámicos y fluoroquinolonas, es un problema de importancia clínica. La propagación de Salmonella Typhimurium resistente a ampicilina (AMP)-cloranfenicol (CHL)-estreptomicina (STR)-sulfamidas (SUL)-tetraciclina (TET) portadoras de la Isla Genómica de Salmonella de tipo 1 (SGI1) y la captación de material genético transferible han favorecido la multirresistencia en este género.
MétodosSe estudiaron 114aislados clínicos de S.enterica (período 2009-2010). Se determinó la sensibilidad a 20antibióticos por difusión en disco y microdilución. Los mecanismos de resistencia e integrones se analizaron por PCR y secuenciación en los aislados AMPR. En los aislados portadores del gen blaPSE-1 se determinó la relación clonal mediante PFGE, y la presencia de la SGI1 y 29genes de virulencia mediante PCR.
ResultadosEntre los 114aislados analizados se detectaron 18serotipos distintos, destacando entre ellos Typhimurium (61%) y Enteritidis (16%). Se observaron altos porcentajes de resistencia a SUL (68%), TET (58%), AMP (55%) y STR (46%). El 92% de los 63 aislados AMPR fueron multirresistentes, siendo el más frecuente el fenotipo AMP-STR-TET-SUL (19aislados) asociado al genotipo blaTEM-1b+strA-strB+tet(B)+sul2. El 48% de los aislados presentaron integrones de clase1 (7estructuras distintas), destacando la estructura blaOXA-1+aadA1 (8aislados), un integrón vacío e integrones no clásicos (5aislados). El gen blaPSE-1 se detectó dentro de la SGI1 clásica en 13aislados clonalmente relacionados y portadores del mismo perfil de virulencia.
ConclusionesEl alto porcentaje de S.enterica multirresistentes, especialmente asociado a S.Typhimurium, al fenotipo AMP, STR, TET y SUL y al genotipo blaTEM-1b+strA-strB+tet(B)+sul2 evidencia un riesgo importante de posibles fracasos en el tratamiento de infecciones graves producidas por este serotipo.
The increase of Salmonella enterica isolates multi-resistant to different antibiotics, including β-lactams and fluoroquinolones, is a problem of clinical importance. The dissemination of Salmonella Typhimurium resistant to ampicillin (AMP)-chloramphenicol (CHL)-streptomycin (STR)-sulphonamides and (SUL)-tetracycline (TET), that harbour the Salmonella Genomic Island type1 (SGI1), and the acquisition of transferable genetic material have favoured the multi-resistance in this genus.
MethodsA total of 114clinical S.enterica isolates were studied (period 2009-2010). The susceptibility to 20antibiotics was determined by disc diffusion and microdilution. The antimicrobial resistance mechanisms and the integrons were analysed by PCR, and sequencing in the AMPR isolates. In all the blaPSE-1-positive isolates, the clonal relationship was determined by PFGE, as well as the presence of SGI1 and 29 virulence genes by PCR.
ResultsEighteen different serotypes were found among the 114isolates studied, Typhimurium (61%) and Enteritidis (16%) being the most prevalent. High percentages of resistance to SUL (68%), TET (58%), AMP (55%) and STR (46%) were observed. The great majority (92%) of 63 AMPR isolates were multi-resistant, with the AMP-STR-TET-SUL phenotype (19 isolates) being the most frequent one and associated with the blaTEM-1b+strA-strB+tet(B)+sul2 genotype. Class1 integrons (7different structures) were observed in 48% AMPR isolates, highlighting the blaOXA-1+aadA1 structure (8isolates), one empty integron and non-classical integrons (5isolates). The blaPSE-1 gene was detected inside the classical SGI1 structure in 13clonally-related isolates that showed the same virulence profile.
ConclusionsThe high percentage of multi-resistant S.enterica isolates, especially associated to S.Typhimurium, to the AMP, STR, TET and SUL phenotype, and to the blaTEM-1b+strA-strB+tet(B)+sul2 genotype, shows an important risk of possible failures in the treatment of serious infections caused by this serotype.
La salmonelosis es una de las infecciones alimentarias más importantes en Europa, donde alcanza un ratio de 29,8casos por 100.000habitantes cada año y afecta principalmente a niños entre 0 y 4años1. Aunque se ha reducido el número de salmonelosis durante los últimos años, Salmonella enterica sigue siendo una de las primeras causas de brotes de toxiinfección alimentaria2. Enteritidis y Typhimurium son las serovariedades de Salmonella enterica subespecie enterica (S.Enteritidis y S.Typhimurium, respectivamente) más prevalentes en el ámbito clínico, representando más del 80% de los aislados obtenidos2-4.
Las infecciones por salmonelas gastroentéricas cursan como una gastroenteritis autolimitada, de manera que el tratamiento antibiótico solo es requerido en casos graves, en pacientes inmunodeprimidos, con factores predisponentes de riesgo o en edades extremas de la vida. No obstante, es preocupante el incremento de aislados de Salmonella enterica resistentes a algunos de los antibióticos utilizados para el tratamiento empírico, en particular amoxicilina/ácido clavulánico (AMC), cefalosporinas de tercera generación o fluoroquinolonas. La resistencia a β-lactámicos en S.enterica se debe principalmente a la adquisición de enzimas β-lactamasas, siendo TEM-1, PSE-1 y OXA-1 las más frecuentemente detectadas y relacionadas con la resistencia a ampicilina (AMP) y AMC5-7. La diseminación de este tipo de resistencias de manera horizontal está mediada por elementos genéticos móviles o movilizables, como plásmidos, transposones e integrones.
Los integrones son elementos genéticos capaces de adquirir y expresar casetes génicos mediante un mecanismo de recombinación sitio-específica8. Los integrones de clase1 son los más frecuentemente descritos y presentan habitualmente los genes qacEΔ1 y sul1 en su región 3′-conservada (3′-CS), aunque también se han descrito integrones noclásicos carentes de dicha región8-10.
La aparición a escala mundial de cepas de S.Typhimurium con fenotipo de pentarresistencia ACSSuT (resistencia a ampicilina, cloranfenicol, estreptomicina, sulfamidas y tetraciclina), especialmente del fagotipo DT104 y fagotipos relacionados, se ha asociado principalmente con la presencia de los genes blaPSE-1, floR, aadA2, sul1 y tet(G) localizados en un integrón complejo de clase1 denominado In10411,12. Este integrón forma parte de una isla genómica de 43kb denominada «Salmonella Genomic Island type 1» (SGI1)11,12. Sin embargo, también se han descrito variantes de la SGI1 surgidas en eventos de recombinación y asociadas a distintos fenotipos de resistencia11,13.
Se han estudiado diversos factores de virulencia, incluso su relación con determinantes de resistencia, en aislados del género Salmonella de distintos hospedadores y localizaciones geográficas14,15. Estos factores de virulencia pueden encontrarse tanto formando parte de islas de patogenicidad (SPI) como codificados en cromosoma o en plásmidos15,16. Así, algunos estudios inciden en la co-integración de determinantes de virulencia y de resistencia a antibióticos en plásmidos híbridos virulencia-resistencia que pueden ser fácilmente seleccionados bajo presión antibiótica14,17,18.
Tras conocer el fenotipo de resistencia a distintos antibióticos en una colección de aislados de S.enterica identificados en pacientes asistidos en el Hospital Clínico Universitario Lozano Blesa de Zaragoza, los objetivos de nuestro trabajo fueron caracterizar los mecanismos de resistencia a β-lactámicos y a otros antibióticos, así como los integrones en los aislados resistentes a ampicilina. Asimismo, en los aislados portadores del gen blaPSE-1 se llevó a cabo la determinación de la relación clonal, el perfil de virulencia y la presencia de la isla genómica de Salmonella (SGI1).
MétodosAislamientos clínicosEntre septiembre de 2009 y noviembre de 2010 se procesaron 7.606coprocultivos en el Laboratorio de Microbiología del Hospital Clínico Universitario Lozano Blesa (Zaragoza). Durante el periodo de tiempo estudiado se obtuvieron 247aislados de S.enterica, entre los que se seleccionó un aislado por paciente, incluyéndose así un total de 114aislados en el presente trabajo. La identificación se llevó a cabo mediante pruebas bioquímicas (WIDER®, Francisco Soria Melguizo, Madrid, España) y el serotipado mediante el estudio del antígeno somáticoO (lipopolisacárido) y de los factores antigénicos flagelares H (proteínas) (Biorad, Marne-la-Coquette, Francia), siguiendo el esquema de Kauffmann y White19.
Estudios de sensibilidad a antibióticosSe estudió la sensibilidad a 20antibióticos (AMP, AMC, cefalotina, ceftazidima, cefotaxima, cefepima, aztreonam, cefoxitina, gentamicina, tobramicina, kanamicina, amikacina, estreptomicina, ácido nalidíxico, ciprofloxacino, tetraciclina, cloranfenicol, sulfamidas, trimetoprim y trimetoprim/sulfametoxazol) mediante microdilución (WIDER®) y/o difusión en disco20. La producción de β-lactamasas de espectro extendido (BLEE) se determinó fenotípicamente utilizando el test de sinergia de doble disco y el fenotipo AmpC se estableció por comparación de la zona de inhibición del disco de cefoxitina (30μg), en presencia o en ausencia de cloxacilina (200μg)7.
Detección molecular de genes de resistenciaEn los aislados resistentes a ampicilina (AMPR), se evaluó la presencia de los genes implicados en la resistencia a β-lactámicos (blaTEM, blaSHV, blaOXA-1 y blaPSE-1), tetraciclina [tet(A)-tet(E), tet(G)], aminoglucósidos [aadA, strA-strB, aac(3)-I, aac(3)-II, aac(3)-IV, ant(2′’) y aac(6′)-Ib], sulfamidas (sul1, sul2, sul3), trimetoprim (dfrA), cloranfenicol (cmlA, catA y floR) y quinolonas (qnrA, qnrB, qnrS, qepA, oqxAB), así como el entorno genético de los genes sul2 y sul3 mediante PCR y secuenciación7.
Detección y caracterización de integronesLa presencia de los genes codificantes de integrasas de tipo1, 2 y 3 (intI1, intI2, intI3), así como la región 3′-CS de los integrones de clase 1 (qacEΔ1-sul1), fueron analizados por PCR en los aislados AMPR. La región variable del integrón fue estudiada por PCR y posterior secuenciación para conocer el conjunto de casetes génicos que portaban7. En los integrones de clase1 que contenían el casete génico blaOXA-1 se caracterizaron los promotores implicados en su expresión mediante PCR y secuenciación21.
Detección y caracterización de la Isla Genómica de Salmonella, factores de virulencia y estudio de clonalidad en aislados blaPSE-1-positivosEn los aislados blaPSE-1-positivos se evaluó la presencia de la SGI1 y su localización en el cromosoma mediante PCR y secuenciación con cebadores anclados a las uniones al cromosoma, así como a los determinantes de resistencia e integrones localizados en ella12.
Asimismo, en estas cepas portadoras del gen blaPSE-1 se estudiaron por PCR y secuenciación 29genes de virulencia: los genes invE/A, orgA, avrA, ttrC, ssaQ, sugR, rhuM, rmbA, misL, mgtC, spi4R, spi4D, sopB y pipA localizados en las SPI de tipo1-5; los genes phoP/Q, hin/H2, iroB, slyA, sodC1, sopE1, sopE2, bcfC y gipA en el cromosoma y los genes spvC, rck, pefA, pefB, pefC y pefD localizados en plásmidos22. La presencia o ausencia de los genes de virulencia avrA, ssaQ, mgtC, spi4D, sopB, gipA, sodC1, sopE1, spvC y bcfC se utilizó para clasificar las cepas en perfiles de virulencia15.
Se estudió la relación clonal de los aislados blaPSE-1-positivos mediante electroforesis en campo pulsado (PFGE) tras digestión del ADN genómico con las enzimas XbaI y SpeI (New England Biolabs Inc., EE.UU.). Se utilizó el método de preparación de insertos del ADN genómico y las condiciones de electroforesis previamente descritas22. Los geles de agarosa se analizaron e interpretaron según el criterio de Tenover et al. (1995)23. La finalidad además de este estudio fue comparar los patrones de PFGE detectados con los patrones determinados en un estudio previo22.
ResultadosAislados bacterianos y estudios de sensibilidad a antibióticosDurante el período analizado (2009-2010), entre los 114aislados de Salmonella estudiados se identificaron una Salmonella enterica subesp. arizonae y 113aislados de Salmonella enterica subesp. enterica serotipos Typhimurium (70 aislados), Enteritidis (18), Rissen (3), Infantis (3), Mikawashima (2), Ohio (2), Riggil (1), Nigeria (1), Manhattan (1), Newport (1), Lindenburg (1), Bovis-morbificans (1), Neasden (1), Dublin (1), Moscow (1), Anatum (1), Salmonella spp. (5). S. Typhimurium y S. Enteritidis fueron los más prevalentes (61 y 16%, respectivamente).
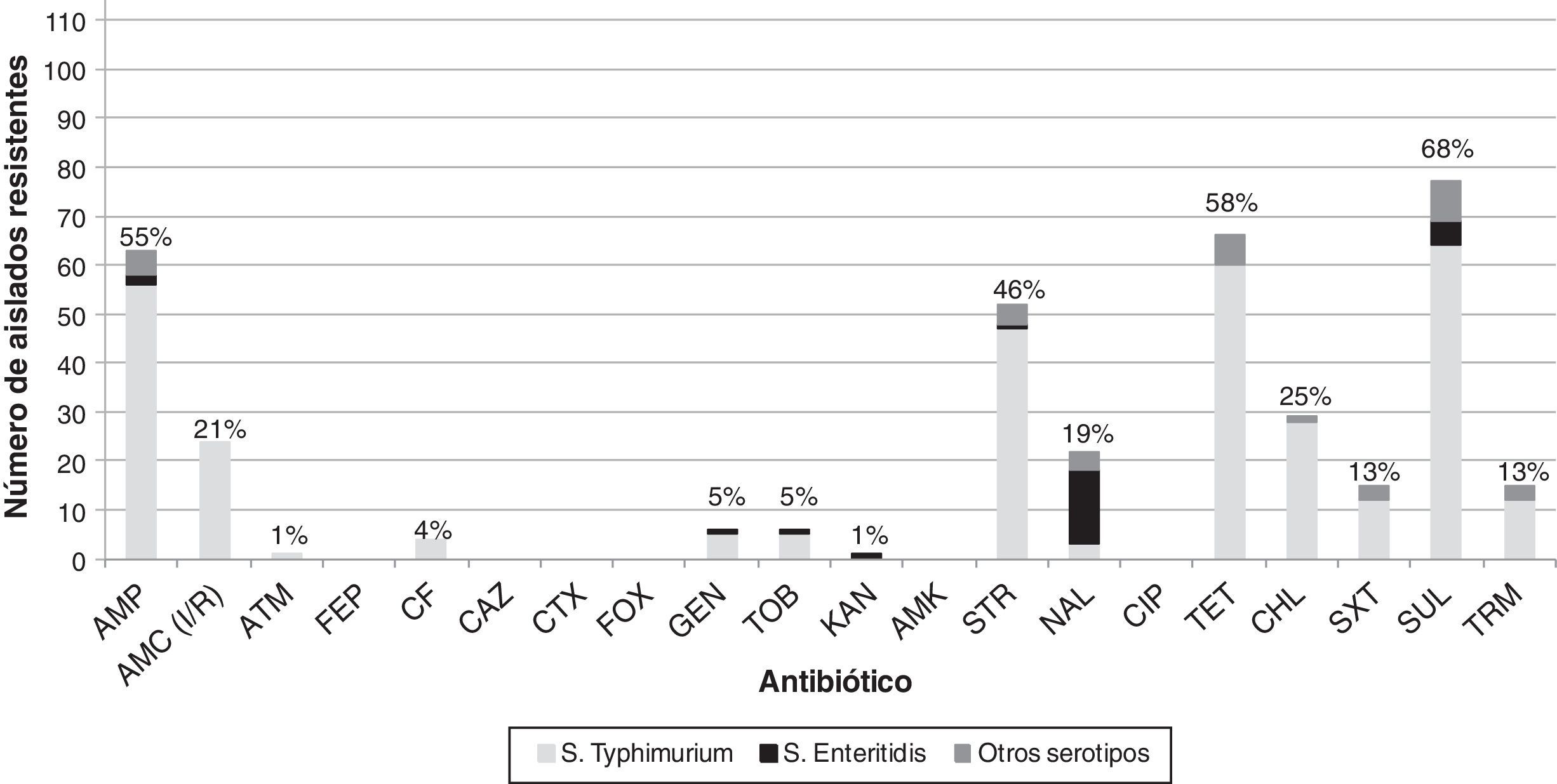
Se observó un alto porcentaje de resistencia a sulfamidas (68%), tetraciclina (58%), AMP (55%) y estreptomicina (46%) en los 114aislados estudiados (fig. 1). Los mayores porcentajes de resistencia se observaron entre los aislados del serotipo Typhimurium, con la excepción del ácido nalidíxico, para el que se detectó que 15 de los 22aislados resistentes a dicho antibiótico (68%) eran del serotipo Enteritidis (fig. 1). Por otra parte, considerando únicamente los 63aislados AMPR, se encontraron altos porcentajes de co-resistencia a sulfamidas (94%), tetraciclina (90%) y estreptomicina (76%), así como a cloranfenicol y AMC (38%) (tabla 1). En 58 de los 63aislados AMPR (mayoritariamente pertenecientes al serotipo Typhimurium, 55aislados) se observaron 17fenotipos distintos de multirresistencia (resistencia a 3 o más familias de antibióticos), siendo las asociaciones más comunes las que presentaban resistencia a AMP-estreptomicina-tetraciclina-sulfamidas (19aislados) y AMP-AMC-estreptomicina-tetraciclina-cloranfenicol-sulfamidas (14aislados) (tabla 1). Se detectó el fenotipo ACSSuT en el 30% de los aislados de S.enterica AMPR, y en ningún caso se detectaron los fenotipos AmpC o BLEE. Todos los aislados fueron sensibles a cefepima, ceftazidima, cefotaxima, cefoxitina, amikacina y ciprofloxacino.
Número de aislados resistentes a los 20antibióticos analizados en las 114Salmonella estudiadas según sus serotipos (porcentaje de resistencia marcado sobre las barras del histograma). Para AMC se consideraron tanto los valores de resistencia como los de resistencia intermedia al antibiótico según CLSI20.AMC: amoxicilina/ácido clavulánico; AMK: amikacina; AMP: ampicilina; ATM: aztreonam; CAZ: ceftazidima; CF: cefalotina; CHL: cloranfenicol; CIP: ciprofloxacino; CTX: cefotaxima; FEP: cefepima; FOX: cefoxitina; GEN: gentamicina; KAN: kanamicina; NAL: ácido nalidíxico; STR: estreptomicina; SUL: sulfamidas; SXT: trimetoprim/sulfametoxazol; TET: tetraciclina, TOB: tobramicina; TRM: trimetoprim.
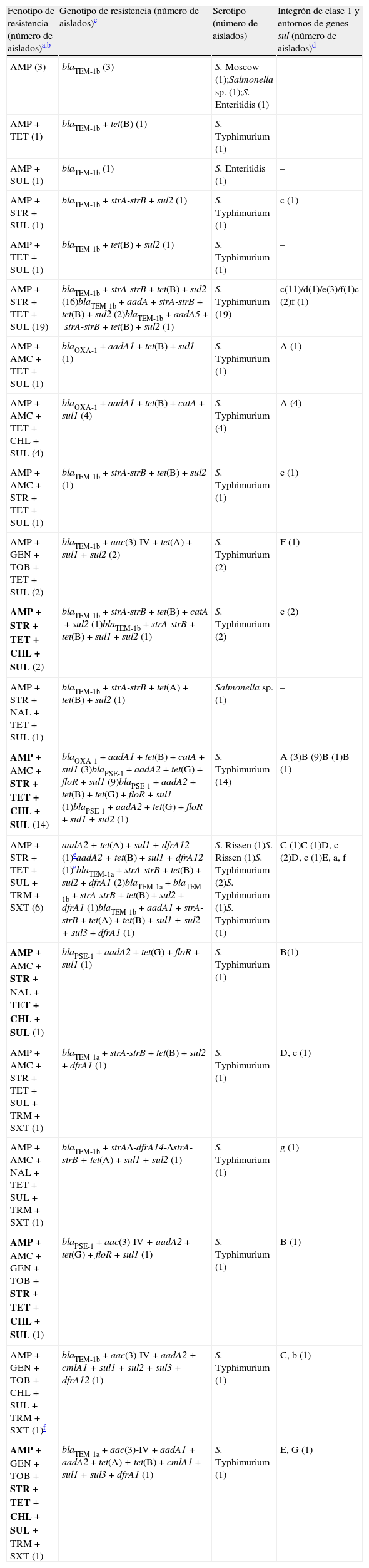
Fenotipos y mecanismos de resistencia detectados en los 63 aislados de Salmonella AMPR
| Fenotipo de resistencia (número de aislados)a,b | Genotipo de resistencia (número de aislados)c | Serotipo (número de aislados) | Integrón de clase 1 y entornos de genes sul (número de aislados)d |
| AMP (3) | blaTEM-1b (3) | S. Moscow (1);Salmonella sp. (1);S. Enteritidis (1) | – |
| AMP+TET (1) | blaTEM-1b+tet(B) (1) | S. Typhimurium (1) | – |
| AMP+SUL (1) | blaTEM-1b (1) | S. Enteritidis (1) | – |
| AMP+STR+SUL (1) | blaTEM-1b+strA-strB+sul2 (1) | S. Typhimurium (1) | c (1) |
| AMP+TET+SUL (1) | blaTEM-1b+tet(B)+sul2 (1) | S. Typhimurium (1) | – |
| AMP+STR+TET+SUL (19) | blaTEM-1b+strA-strB+tet(B)+sul2 (16)blaTEM-1b+aadA+strA-strB+tet(B)+sul2 (2)blaTEM-1b+aadA5+strA-strB+tet(B)+sul2 (1) | S. Typhimurium (19) | c(11)/d(1)/e(3)/f(1)c (2)f (1) |
| AMP+AMC+TET+SUL (1) | blaOXA-1+aadA1+tet(B)+sul1 (1) | S. Typhimurium (1) | A (1) |
| AMP+AMC+TET+CHL+SUL (4) | blaOXA-1+aadA1+tet(B)+catA+sul1 (4) | S. Typhimurium (4) | A (4) |
| AMP+AMC+STR+TET+SUL (1) | blaTEM-1b+strA-strB+tet(B)+sul2 (1) | S. Typhimurium (1) | c (1) |
| AMP+GEN+TOB+TET+SUL (2) | blaTEM-1b+aac(3)-IV+tet(A)+sul1+sul2 (2) | S. Typhimurium (2) | F (1) |
| AMP+STR+TET+CHL+SUL (2) | blaTEM-1b+strA-strB+tet(B)+catA+sul2 (1)blaTEM-1b+strA-strB+tet(B)+sul1+sul2 (1) | S. Typhimurium (2) | c (2) |
| AMP+STR+NAL+TET+SUL (1) | blaTEM-1b+strA-strB+tet(A)+tet(B)+sul2 (1) | Salmonella sp. (1) | – |
| AMP+AMC+STR+TET+CHL+SUL (14) | blaOXA-1+aadA1+tet(B)+catA+sul1 (3)blaPSE-1+aadA2+tet(G)+floR+sul1 (9)blaPSE-1+aadA2+tet(B)+tet(G)+floR+sul1 (1)blaPSE-1+aadA2+tet(G)+floR+sul1+sul2 (1) | S. Typhimurium (14) | A (3)B (9)B (1)B (1) |
| AMP+STR+TET+SUL+TRM+SXT (6) | aadA2+tet(A)+sul1+dfrA12 (1)eaadA2+tet(B)+sul1+dfrA12 (1)eblaTEM-1a+strA-strB+tet(B)+sul2+dfrA1 (2)blaTEM-1a+blaTEM-1b+strA-strB+tet(B)+sul2+dfrA1 (1)blaTEM-1b+aadA1+strA-strB+tet(A)+tet(B)+sul1+sul2+sul3+dfrA1 (1) | S. Rissen (1)S. Rissen (1)S. Typhimurium (2)S. Typhimurium (1)S. Typhimurium (1) | C (1)C (1)D, c (2)D, c (1)E, a, f |
| AMP+AMC+STR+NAL+TET+CHL+SUL (1) | blaPSE-1+aadA2+tet(G)+floR+sul1 (1) | S. Typhimurium (1) | B(1) |
| AMP+AMC+STR+TET+SUL+TRM+SXT (1) | blaTEM-1a+strA-strB+tet(B)+sul2+dfrA1 (1) | S. Typhimurium (1) | D, c (1) |
| AMP+AMC+NAL+TET+SUL+TRM+SXT (1) | blaTEM-1b+strAΔ-dfrA14-ΔstrA-strB+tet(A)+sul1+sul2 (1) | S. Typhimurium (1) | g (1) |
| AMP+AMC+GEN+TOB+STR+TET+CHL+SUL (1) | blaPSE-1+aac(3)-IV+aadA2+tet(G)+floR+sul1 (1) | S. Typhimurium (1) | B (1) |
| AMP+GEN+TOB+CHL+SUL+TRM+SXT (1)f | blaTEM-1b+aac(3)-IV+aadA2+cmlA1+sul1+sul2+sul3+dfrA12 (1) | S. Typhimurium (1) | C, b (1) |
| AMP+GEN+TOB+STR+TET+CHL+SUL+TRM+SXT (1) | blaTEM-1a+aac(3)-IV+aadA1+aadA2+tet(A)+tet(B)+cmlA1+sul1+sul3+dfrA1 (1) | S. Typhimurium (1) | E, G (1) |
AMC: amoxicilina/ácido clavulánico; AMP: ampicilina; CHL: cloranfenicol; FOX: cefoxitina; GEN: gentamicina; NAL: ácido nalidíxico; STR: estreptomicina; SUL: sulfamidas; SXT: trimetoprim/sulfametoxazol; TET: tetraciclina; TOB: tobramicina; TRM: trimetoprim.
En la tabla 1 se observan los genes de resistencia detectados en los 63aislados de S.enterica AMPR. Entre los genes codificantes de β-lactamasas, el más frecuente fue blaTEM-1 (40aislados, 64%), seguido de blaPSE-1 (13aislados, 21%) y blaOXA-1 (8aislados, 13%), destacando que en los aislados AMPR-AMCI/R (n=24) el gen más frecuentemente detectado fue blaPSE-1 (13aislados, 54%), seguido de blaOXA-1 (8aislados) y blaTEM-1 (3aislados). No se detectaron los genes codificantes de β-lactamasas estudiados (blaTEM, blaOXA-1 y blaPSE) en 2aislados AMPR (CMI>16mg/l y sensibles al resto de β-lactámicos analizados). En los aislados blaOXA-1-positivos se observó un fenotipo de sensibilidad disminuida a cefepima.
Se encontraron distintas asociaciones genéticas responsables del fenotipo ACSSuT detectado en 18aislados de S.enterica AMPR (tabla 1): a)blaOXA-1+catA+aadA1+sul1+tet(B); b)blaPSE-1+floR+aadA2+sul1+tet(G), y c)blaTEM-1+catA o cmlA+aadA o strA-strB+sul2 o sul3+ tet(A) o tet(B).
Se detectaron los genes dfrA1 (6aislados), dfrA14 (un aislado) y dfrA12 (3aislados) entre los 10aislados trimetoprim-resistentes, y el gen aac(3)-IV en los 5aislados resistentes a gentamicina (tabla 1). Ningún aislado de S.enterica testado amplificó qnrA, qnrB, qnrS, qepA, oqxAB ni aac(6′)-Ib-cr.
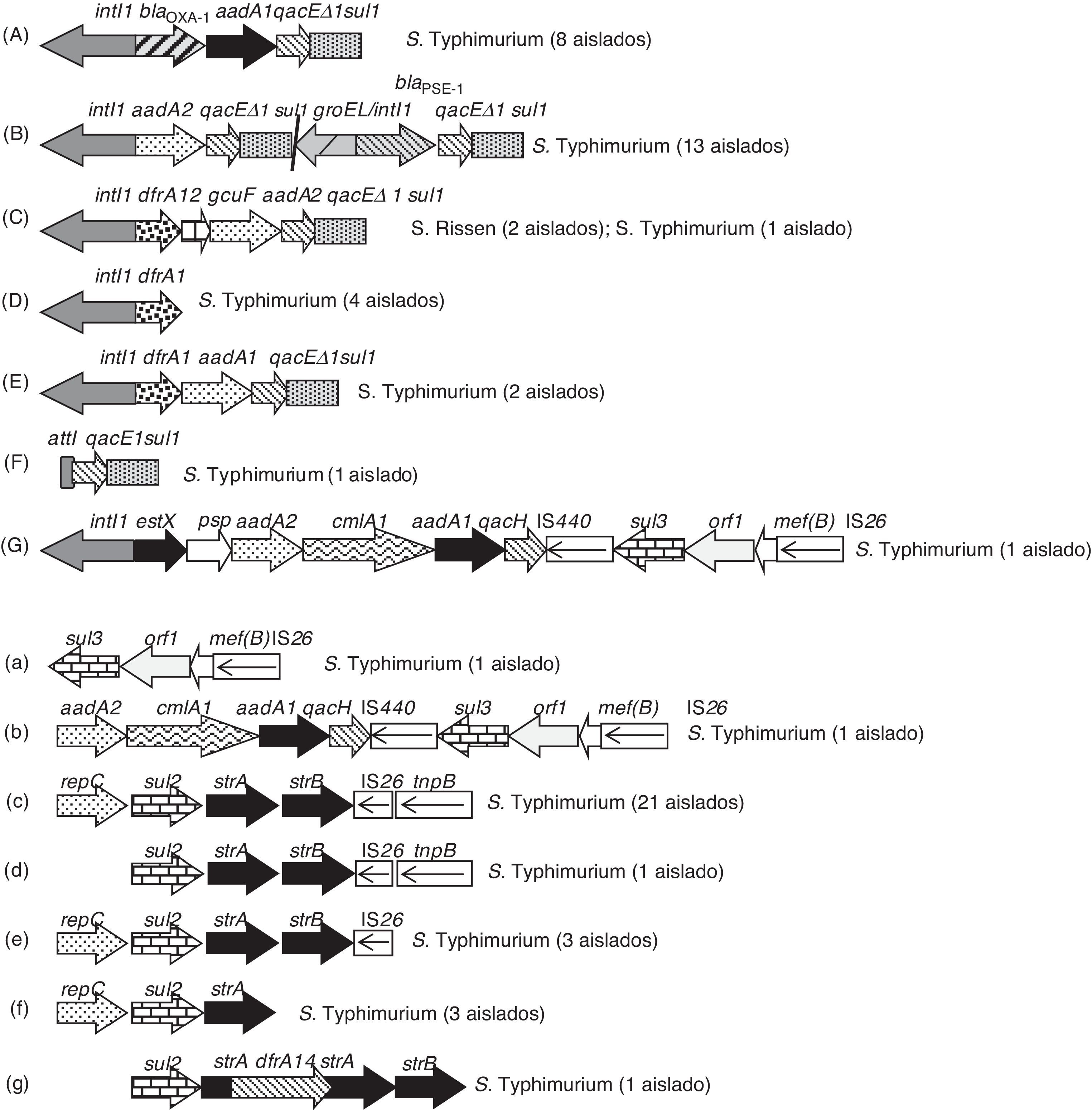
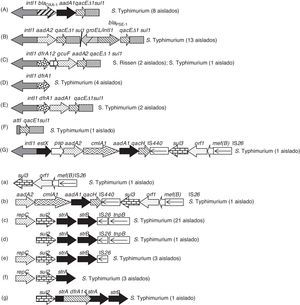
Detección y caracterización de integronesTreinta aislados de S.enterica AMPR (48%) amplificaron el gen intI1, sin embargo no se detectaron integrones de clase 2 o 3. Se observaron 7estructuras distintas de integrones de clase1 (tabla 1 y fig. 2). Los 13aislados de S.Typhimurium blaPSE-1-positivos mostraron 2integrones, con regiones variables de 1.000 y 1.200pb, que albergaban los genes aadA2 y blaPSE-1, respectivamente. La estructura genética blaOXA-1+aadA1, asociada a un promotor PcW-P2, se encontró en los 8aislados blaOXA-1-positivos.
Se observó la presencia de un integrón vacío en un aislado y de integrones no clásicos (defectivos en su región 3′-CS) en 5 aislados, con las siguientes organizaciones (número de aislados): dfrA1 (4) y estX+psp+aadA2+cmlA1+aadA1+qacH+IS440+sul3 (1) (fig. 2).
Entorno genético de genes sulUn total de 59aislados (94%) con fenotipo AMPR presentaron resistencia a sulfamidas, en los que se detectaron las siguientes combinaciones de genes de resistencia: sul1 (22aislados), sul2 (28), sul1+sul2 (5), sul1+sul3 (1) y sul1+sul2+sul3 (2) (tabla 1). Se encontraron 5entornos distintos para el gen sul2 asociados a los genes de resistencia a estreptomicina strA-strB (fig. 2). En uno de los aislados, el gen strA se encontraba truncado por el gen dfrA14, dando lugar a un fenotipo de sensibilidad a estreptomicina (tabla 1, fig. 2). El gen sul3 (detectado en 3aislados) se relacionó en su entorno genético con el gen de resistencia a eritromicina (mef(B), truncado en todos los casos por una IS26) y adicionalmente a los genes aadA1, aadA2, cmlA y qacH en 2aislados, uno de los cuales era portador de un integrón defectivo de clase1 (fig. 2).
Detección y caracterización de la Isla Genómica de Salmonella, factores de virulencia y estudio de clonalidad en aislados blaPSE-1-positivosLos 13aislados de S.Typhimurium blaPSE-1-positivos mostraron el fenotipo de multirresistencia ACSSuT asociado con los genes blaPSE-1, floR, aadA2, sul1 y tet(G), y presentaban el doble integrón de 1.000 y 1.200pb (fig. 2, estructura B). Toda esta zona de resistencia se encontró junto con las uniones a cromosoma en la estructura de SGI1, mapeada por PCR y secuenciación en los 13aislados.
Todos los aislados fueron positivos para los genes albergados en las islas de patogenicidad SPI1-SPI5, para los genes codificados en cromosoma (phoP/Q, hin/H2, iroB, slyA, sodC1, sopE2 y bcfC) y para los genes plasmídicos spvC, rck, pefA, pefB, pefC y pefD. Los genes gipA y sopE1 no se detectaron en ningún aislado. Todos los aislados se agruparon en un único perfil de virulencia, compuesto por los genes avrA-ssaQ-mgtC-spi4D-sopB-sodC1-spvC-bcfC.
El PFGE tras digestión con la enzima XbaI agrupó a los 13aislados en 3perfiles (X1a, X1b y X1g), mientras que el PFGE-SpeI los clasificó en 4patrones (S1a, S1d, S1k y S1l). Teniendo en cuenta ambas digestiones, se encontraron 6combinaciones de patrones: X1a-S1a (2 cepas), X1a-S1d (4), X1a-S1k (1), X1b-S1a (1), X1b-S1d (4) y X1g-S1l (1). Los patrones mostraban entre 1 y 3bandas de diferencia con respecto al primer patrón de cada digestión (X1a y S1a para PFGE-XbaI y PFGE-SpeI, respectivamente) y se clasificaron como cepas clonalmente relacionadas.
DiscusiónEntre los antibióticos más utilizados para el tratamiento de infecciones graves producidas por S.enterica figuran AMC, cefalosporinas de tercera generación y fluoroquinolonas, pero la aparición de mecanismos de resistencia a dichos antibióticos limita las opciones terapéuticas. La resistencia a fluoroquinolonas resulta todavía infrecuente en S.enterica, quizás debido a que supone un alto coste energético para la bacteria24, mientras que el creciente incremento de la prevalencia del serotipo Typhimurium durante los últimos años se asocia a mayores porcentajes de resistencia a AMP y AMC, en detrimento del serotipo Enteritidis, menos relacionado con fenotipos de multirresistencia2-4,25. Este fenómeno se está observando también en nuestro entorno clínico, en el que a principios del período 2001-2008 el serotipo Enteritidis (56%) dominaba sobre Typhimurium (26%), mientras que se observó una inversión en esta tendencia al final del mismo (27% Enteritidis versus 41% Typhimurium)26. Además, el aumento en el número de S.Typhimurium estuvo acompañado por un porcentaje mayor de aislados de S.enterica resistentes a AMP (del 39 al 52% al avanzar el período) y a AMC (0,4-16%), por unos porcentajes de resistencia a cotrimoxazol y ciprofloxacino mantenidos (13 y 0,5%, respectivamente) y por una disminución en la resistencia a ácido nalidíxico (del 42,5 al 20%). Durante el período 2009-2010 continuó la tendencia creciente de Typhimurium frente a Enteritidis (61% vs 16%) y el porcentaje de aislados AMPR que se encontró fue del 55%, con el 87% de ellos del serotipo Typhimurium, valores acordes con otros estudios previos5-7.
Entre los 63aislados AMPR estudiados se encontraron 17fenotipos distintos de multirresistencia, siendo el más común la corresistencia a AMP-estreptomicina-tetraciclina-sulfamidas detectada en 19 aislados que portaban los genes de resistencia blaTEM-1b-strA-strB-tet(B)-sul2. Esta asociación fenotipo-genotipo había sido previamente descrita por nuestro grupo7. Por otra parte, el fenotipo ACSSuT de S.Typhimurium ha sido frecuentemente asociado a la presencia de SGI1, que contiene los genes blaPSE-1, floR, aadA2, sul y tet(G)11,12, aunque en nuestro caso solo 13 de 19aislados con fenotipo ACSSuT presentaron el gen blaPSE-1 y sensibilidad disminuida a AMC. Otros perfiles que incluyen los genes blaTEM-1 y blaOXA-1 han sido previamente referidos y relacionados con la presencia de plásmidos transferibles17,18. Al contrario que otros estudios y conforme a trabajos realizados en nuestro grupo, no se detectó la presencia de más de una β-lactamasa en el mismo aislado5-7. Todos los aislados que portaban los genes blaPSE-1 y blaOXA-1 eran AMPR-AMCI/R, mientras que el gen blaTEM-1 estuvo relacionado con el fenotipo AMPR-AMCS en la mayoría de los casos, al igual que en trabajos previamente descritos5,7.
A semejanza de estudios previos5,11, se encontraron integrones de clase1 en el 48% de los 63aislados AMPR, siendo los más numerosos los que contenían en su región variable los genes blaOXA-1+aadA1 (8aislados) y la presencia de cepas con el doble integrón característico de la SGI1 (blaPSE-1/aadA1, 13aislados). Aunque el gen blaTEM-1 no se encontró dentro de integrones, 10 de los aislados blaTEM-1-positivos albergaron genes dfrA en estructuras de tipo integrón previamente descritas, como la estructura intI1+dfrA12+gcuF+aadA2+qacEΔ1+sul1 y relacionado con el serotipo Rissen9, el gen dfrA1, presente en 2estructuras distintas27, o el integrón de clase1, carente de su región 3′-CS, asociado al gen sul3 previamente descrito en Salmonella y E.coli9,10.
Algunos autores sugieren que dentro de la subespecieI de Salmonella existe una baja clonalidad28, mientras que otros estudios muestran por PFGE la alta clonalidad existente en un mismo serotipo29. Las 13cepas de S.Typhimurium blaPSE-1-positivas estaban clonalmente relacionadas, y sus patrones de PFGE eran coincidentes con los de aislados procedentes de otros hospitales españoles geográficamente distantes22. En este trabajo se vuelve a demostrar que además de la alta clonalidad existente entre las cepas de S.Typhimurium blaPSE-1, todas ellas mostraron el mismo perfil de virulencia, conteniendo los genes avrA-ssaQ-mgtC-spi4D-sopB-sodC1-spvC-bcfC, muy prevalente entre las cepas de S.Typhimurium según datos previos15,22. Además, en nuestros aislados se encontró el gen blaPSE-1 dentro de la estructura clásica de la SGI1 en cepas S.Typhimurium, serotipo frecuentemente asociado a dicha estructura11,12.
En resumen, se ha encontrado una alta prevalencia de cepas resistentes a AMP, estreptomicina, tetraciclina y sulfamidas. El serotipo Typhimurium resultó el más prevalente, tanto en el total de aislados como entre los AMPR (87%). El 92% de los aislados AMPR mostraron un fenotipo de multirresistencia, siendo el gen blaTEM-1 el más frecuentemente detectado (63,5%). Las cepas que albergaban el gen blaPSE-1 mostraron una baja diversidad clonal, así como homogeneidad en cuanto a la presencia y estructura de la SGI1 detectada y a los determinantes de virulencia contenidos. La asociación de genes de virulencia y resistencia en cepas clonalmente relacionadas plantea la conveniencia de vigilar su evolución y establecer los reservorios y las vías de propagación que podrían explicar su aparición en aislados clínicos.
El alto porcentaje de S.enterica multirresistentes detectadas, especialmente asociado a S.Typhimurium, dificulta la elección de alternativas seguras para el tratamiento empírico de infecciones graves ocasionadas por Salmonella y precisa estudios epidemiológicos continuos que permitan vigilar la evolución de la resistencia y la aparición de clones que aúnan resistencia y virulencia.
FinanciaciónM. de T. posee una beca de investigación del Ministerio de Sanidad y Consumo. Instituto de Salud Carlos III (ref. FI08/00508).
Conflicto de interesesLos autores declaran que no existe ningún conflicto de intereses.
Parte de este trabajo fue presentado en el XVI Congreso de la Sociedad Española de Enfermedades Infecciosas y Microbiología Clínica (comunicación 692). Bilbao, 9-11 de mayo de 2012.