La sepsis neonatal es una importante causa de morbimortalidad. Un diagnóstico precoz y el inicio rápido del tratamiento son fundamentales en la evolución del recién nacido. El hemocultivo, considerado técnica de referencia para el diagnóstico de sepsis, presenta una baja sensibilidad en este grupo de pacientes. Se planteó evaluar la utilidad de la PCR múltiple LightCycler® SeptiFast (LC-SF) en el diagnóstico de la sepsis neonatal, comparándola con el hemocultivo tradicional.
MétodosSe recogieron 42 muestras de sangre correspondientes a 35 recién nacidos con episodios febriles ingresados en la unidad de cuidados intensivos neonatal del Hospital Universitario Virgen de las Nieves. Por cada episodio febril se procesaron 2 muestras de sangre venosa periférica para la realización del ensayo LC-SF y del hemocultivo, respectivamente.
ResultadosLa sensibilidad y la especificidad de LC-SF, comparada con el diagnóstico clínico de sepsis, fueron del 79 y del 87%, respectivamente. La tasa de hemocultivos contaminados fue del 16,7%, detectándose Staphylococcus coagulasa negativa (SCN) y Streptococcus grupo viridans. En LC-SF la tasa de SCN contaminantes fue del 2,4%. La concordancia entre las 2 técnicas diagnósticas fue moderada (índice kappa: 0,369). La técnica LC-SF demostró una mayor concordancia con el diagnóstico clínico final (índice kappa: 0,729) que el hemocultivo (índice kappa: 0,238).
ConclusiónLC-SF podría ser una herramienta útil, empleada en paralelo con el hemocultivo en recién nacidos, al confirmar o descartar los casos que el hemocultivo no resuelve, además de disminuir el tiempo de obtención de resultado a 7h.
Neonatal sepsis is a significant cause of morbidity and mortality. Early diagnosis and prompt antimicrobial therapy are crucial for a favorable outcome of the newborn child. Blood culture, the current “gold standard” method for diagnosing bloodstream infections, has a low sensitivity in newborns. We evaluated the multiplex real-time PCR LightCycler® SeptiFast (LC-SF) for detection of bloodstream infections in newborns, compared with conventional blood culture.
MethodsA total of 42 blood samples were obtained from 35 subjects presenting with a febrile episode and hospitalized in neonatal intensive care unit at Hospital Universitario Virgen de las Nieves. Two samples were collected during each febrile episode in order to carry out LC-SF assay and blood culture, respectively.
ResultsSensitivity and specificity of 79% and 87%, respectively, compared with clinical diagnosis, were obtained for LC-SF. Contamination rate of blood cultures was 16.7%, mainly due to coagulase-negative staphylococci (CoNS) and viridans groups of streptococci. Contamination rate of LC-SF by CoNS was 2.4%. Concordance between LC-SF and blood culture was moderate (kappa index: 0.369). LC-SF demonstrated a higher concordance (kappa index: 0.729) with the final clinical diagnosis than blood culture (kappa index: 0.238).
ConclusionLC-SF assay could be a useful diagnostic tool, along with a conventional blood culture, in newborn, for confirming or ruling out those cases that blood culture could not determine, shortening the time to result to 7hours.
Las infecciones son la principal causa de morbilidad y mortalidad en el periodo neonatal, y en la mayoría de los casos prolongan la estancia hospitalaria en las unidades de cuidados intensivos neonatal (UCIN). Según los estudios del Grupo Castrillo, la incidencia en España de sepsis de transmisión vertical es de 2,5 por 1.000 recién nacidos (RN) vivos, y la de sepsis nosocomial del 2,1% de los ingresos en unidades neonatales, con una mortalidad del 8,7 y del 11,8%, respectivamente1,2.
Un diagnóstico precoz y el inicio rápido del tratamiento son fundamentales en la evolución del RN. La obtención de este diagnóstico precoz sigue siendo un desafío, ya que la mayoría de síntomas y signos de infección neonatal son inespecíficos3,4. Los marcadores biológicos como la proteína C reactiva, la procalcitonina o algunas interleucinas se pueden elevar durante las primeras horas de vida en neonatos no infectados5. El hemocultivo está considerado como la técnica de referencia para el diagnóstico de sepsis. Sin embargo, la necesidad de instaurar de forma precoz un tratamiento antibiótico, generalmente de amplio espectro, hace que el hemocultivo tenga un elevado porcentaje de falsos negativos, dado que en muchos casos la toma de muestra se realiza tras la instauración de la antibioterapia6-8. Otro factor importante es el volumen de sangre extraído en los RN, que suele ser significativamente inferior al obtenido en adultos (<1ml frente a 10-30ml). Todo esto, junto con el hecho de que las bacteriemias en RN son a menudo transitorias o interminentes, limita mucho el valor del hemocultivo en el diagnóstico de sepsis en este grupo de pacientes9.
En la última década se han descrito distintas técnicas moleculares para la detección de ADN bacteriano y fúngico directamente de sangre10-13. El ensayo LightCycler® SeptiFast Test Mgrade (LC-SF; Roche Diagnostics GmbH, Mannheim, Alemania) es una prueba de reacción en cadena de la polimerasa (PCR) múltiple en tiempo real, para la detección de ADN bacteriano y fúngico (región ITS [internal transcriptor spacer], situada entre las secuencias ADNr 16S-23S y ADNr 18S-5,8S, respectivamente). Permite la identificación de 25 microorganismos en sangre humana, representando aproximadamente el 90% de los patógenos implicados en bacteriemia en la población adulta. El objetivo principal de este estudio es evaluar la utilidad de la técnica LC-SF en el diagnóstico de la sepsis neonatal, en comparación con el hemocultivo tradicional.
MétodosSe recogieron un total de 42 muestras de sangre de otros tantos episodios febriles de 35 RN y lactantes ingresados en la UCIN del Hospital Universitario Virgen de las Nieves entre abril de 2007 y abril de 2009.
Se procesaron 2 muestras de sangre venosa periférica de cada episodio, con un volumen recomendado de 1ml para cada técnica, y un volumen final que osciló entre 350 y 1.000μl según las posibilidades de extracción dadas las características de estos pacientes. Una muestra se inoculó en un tubo de hemograma con K-EDTA, para realización del ensayo LC-SF, y la otra, en frasco pediátrico para hemocultivo por el sistema automático Bactec-9240® (Becton Dickinson, Franklin Lakes, NJ, EE.UU.). LC-SF se llevó a cabo en las 24h siguientes a la recepción de la muestra. La sangre se conservó a 4°C hasta la realización del ensayo. La extracción del ADN, la realización del ensayo LC-SF y la interpretación de los resultados de la PCR se llevaron a cabo siguiendo las recomendaciones del fabricante.
El tiempo mínimo necesario para la obtención de resultados tras el hemocultivo fue de 48h. El método de extracción de ácido nucleico en el ensayo LC-SF recomendado por el fabricante fue la extracción manual, llevándose a cabo en aproximadamente 4h. Esto hace un tiempo total de procesamiento de muestras hasta la obtención de resultados con la técnica LC-SF de 7h.
El registro de datos y análisis de resultados se llevó a cabo usando PASW Statistics version 18.0 (IBM SPSS Inc., Chicago, EE.UU). Se realizó análisis retrospectivo de los datos clínicos y de laboratorio junto con los resultados obtenidos por ambas técnicas microbiológicas.
El diagnóstico clínico final fue de sepsis clínica, sepsis probada o sepsis descartada. Se definió como sepsis clínica cuando se cumplían los criterios clínicos de mala perfusión, mala tolerancia oral, distrés respiratorio e hipotensión y/o los criterios analíticos de leucopenia o leucocitosis con desviación a la izquierda y proteína C reactiva elevada (>1mg/dl). La sepsis probada se definió como la presencia de sepsis clínica junto con el aislamiento del microorganismo en el hemocultivo. Se definió como sepsis descartada cuando solo se cumplía uno de los criterios clínicos y/o analíticos de sepsis clínica debido a otras causas, bien porque el hemocultivo se había realizado como control de tratamiento de un episodio previo o porque el paciente estaba afectado por otras patologías no infecciosas.
Se realizó estadística descriptiva de los datos clínicos y de laboratorio. Así mismo se determinaron los valores de sensibilidad, especificidad, valor predictivo positivo y valor predictivo negativo de la técnica LC-SF tomando como referencia el diagnóstico clínico final. Se determinó el índice kappa14 y el acuerdo porcentual para calcular el grado de concordancia entre las técnicas diagnósticas, así como el grado de concordancia entre cada técnica y el diagnóstico clínico final. Para cada parámetro estadístico analizado se calcularon los intervalos de confianza al 95% (IC95%).
ResultadosLa población estaba constituida por pacientes con edades comprendidas entre 0 y 151 días, con una mediana de 8 días (desviación cuartilíca: 6 días), de los cuales 13 eran mujeres (37,1%) y 22 varones (62,9%). La mediana del peso al nacer de los RN fue de 2.400g (rango: 615-3.760g; desviación cuartilíca: 1.090g), siendo el 54,3% (19/35) RN pretérmino y el 31,4% (11/35) RN de muy bajo peso (RNMBP) (<1.500g).
Se consideró sepsis descartada en 23 episodios (54,8%) y sepsis clínica en los otros 19 episodios (45,2%), de los cuales 10 se confirmaron por el hemocultivo (sepsis probada).
Entre los 19 RN pretérmino, en 10 de ellos (52,6%) hubo sepsis clínica, de los cuales 5 (26,3%) fueron sepsis probada. De los 11 RNMBP, 6 (54,5%) fueron sepsis clínica, de los cuales 3 (27,3%) fueron sepsis probada.
El número de episodios positivos por el hemocultivo y el LC-SF fue de 17 y 18, respectivamente. Se obtuvieron 25 episodios negativos por hemocultivo y 24 por LC-SF.
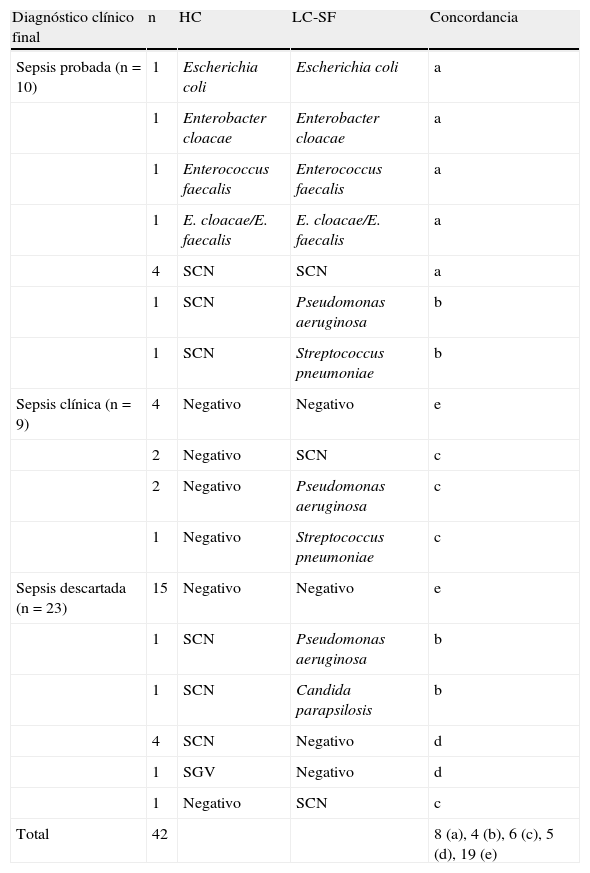
En la tabla 1 se muestran los resultados del hemocultivo y el LC-SF en relación con el diagnóstico clínico final, así como la concordancia y discordancia entre ambas técnicas.
Resultados de las técnicas de hemocultivo y LightCycler® SeptiFast Test Mgrade en relación con el diagnóstico clínico final
| Diagnóstico clínico final | n | HC | LC-SF | Concordancia |
| Sepsis probada (n = 10) | 1 | Escherichia coli | Escherichia coli | a |
| 1 | Enterobacter cloacae | Enterobacter cloacae | a | |
| 1 | Enterococcus faecalis | Enterococcus faecalis | a | |
| 1 | E. cloacae/E. faecalis | E. cloacae/E. faecalis | a | |
| 4 | SCN | SCN | a | |
| 1 | SCN | Pseudomonas aeruginosa | b | |
| 1 | SCN | Streptococcus pneumoniae | b | |
| Sepsis clínica (n = 9) | 4 | Negativo | Negativo | e |
| 2 | Negativo | SCN | c | |
| 2 | Negativo | Pseudomonas aeruginosa | c | |
| 1 | Negativo | Streptococcus pneumoniae | c | |
| Sepsis descartada (n = 23) | 15 | Negativo | Negativo | e |
| 1 | SCN | Pseudomonas aeruginosa | b | |
| 1 | SCN | Candida parapsilosis | b | |
| 4 | SCN | Negativo | d | |
| 1 | SGV | Negativo | d | |
| 1 | Negativo | SCN | c | |
| Total | 42 | 8 (a), 4 (b), 6 (c), 5 (d), 19 (e) |
Concordancia. a: positivos ambas técnicas con identificación concordante; b: positivos ambas técnicas con identificación discordante; c: HC negativo/LC-SF positivo; d: HC positivo/LC-SF negativo; e: negativos ambas técnicas.
HC: hemocultivo; LC-SF: LightCycler® SeptiFast Test Mgrade; SCN: Staphylococcus coagulasa negativa; SGV: Streptococcus grupo viridans.
Entre los 10 casos de sepsis probada, 8 fueron resultados positivos con identificación concordante entre ambas técnicas. En los 2 casos discordantes, se aisló Staphylococcus coagulasa negativa (SCN) en el hemocultivo. En uno de estos casos, LC-SF detectó Pseudomonas aeruginosa (P. aeruginosa). Se trataba de un paciente que tambien dio positivo a P. aeruginosa en otra muestra de sangre mediante LC-SF, con resultado negativo de hemocultivo y diagnóstico de sepsis clínica. A este paciente se le habían tomado otras tantas muestras coincidiendo con las 2 muestras de sangre, 3 aspirados endotraqueales y un líquido peritoneal, aislándose P. aeruginosa en un aspirado traqueal y en el líquido peritoneal. En el otro caso discordante, LC-SF detectó Streptococcus pneumoniae (S. pneumoniae); en este caso, el pediatra consideró sepsis tardía por SCN, a consecuencia de un cuadro febril con elevación de PCR.
LC-SF fue capaz de detectar un microorganismo en 5 de los 9 casos de sepsis clínica con hemocultivo negativo, 2 P. aeruginosa (una corresponde al caso descrito anteriormente), un S. pneumoniae y 2 SCN.
Entre los 23 episodios en los que la sepsis se consideró descartada, 16 dieron negativo el hemocultivo y 20 el ensayo LC-SF, lo que supone el 69,5 y el 87% de las sepsis descartadas, siendo concordante el resultado negativo en 15 casos. Los 7 casos de sepsis descartada con hemocultivo positivo correspondían a 6 SCN y un Streptococcus grupo viridans, por lo que se consideraron contaminaciones de la toma. SCN solo se detectó por LC-SF en uno de los casos de sepsis descartada. Por tanto, la tasa de contaminación por SCN del hemocultivo y del LC-SF fue del 14,3 y del 2,4%, respectivamente. Además, LC-SF detectó P. aeruginosa en un caso y Candida parapsilosis (C. parapsilosis) en otro caso de sepsis descartada.
Los valores de sensibilidad, especificidad, valor predictivo negativo y valor predictivo positivo de la técnica LC-SF fueron de 79% (IC95%: 57-91%), 87% (IC95%: 68-95%), 83% (IC95%: 64-93%) y 83% (IC95%: 61-94%), respectivamente.
Al analizar la concordancia entre las 2 técnicas diagnósticas se obtuvieron 5 grupos de resultados: 8 casos positivos por ambas técnicas con identificación concordante, 4 casos positivos por ambas técnicas con identificación diferente, 6 casos con hemocultivo negativo y LC-SF positivo, 5 casos con hemocultivo positivo y LC-SF negativo y 19 casos negativos por ambas técnicas.
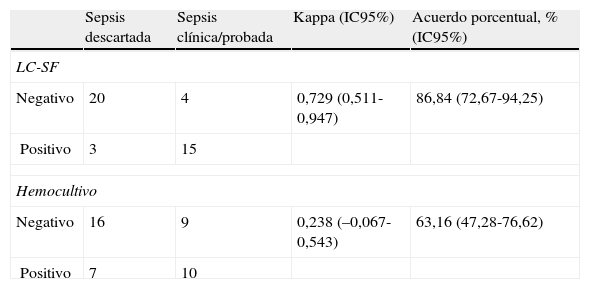
El estudio de concordancia entre las 2 técnicas diagnósticas demostró un índice kappa de 0,369 (IC95%: 0,062-0,675), equivalente a una concordancia moderada, y un acuerdo porcentual del 71,1% (IC95%: 55,2-83%).
Asimismo, se determinó el grado de concordancia de cada técnica con el diagnóstico realizado por el pediatra. Se demostró una mayor concordancia del diagnóstico clínico con los resultados del LC-SF que con los del hemocultivo (tabla 2).
Concordancia entre el diagnóstico clínico y los resultados del ensayo LightCycler® SeptiFast Test Mgrade y del hemocultivo
| Sepsis descartada | Sepsis clínica/probada | Kappa (IC95%) | Acuerdo porcentual, % (IC95%) | |
| LC-SF | ||||
| Negativo | 20 | 4 | 0,729 (0,511-0,947) | 86,84 (72,67-94,25) |
| Positivo | 3 | 15 | ||
| Hemocultivo | ||||
| Negativo | 16 | 9 | 0,238 (–0,067-0,543) | 63,16 (47,28-76,62) |
| Positivo | 7 | 10 | ||
IC95%: intervalo de confianza al 95%; LC-SF: LightCycler® SeptiFast Test Mgrade.
La sepsis neonatal es una causa importante de morbilidad y mortalidad, fundamentalmente en RN pretérmino15. De todos los casos de sepsis en el RN, más del 25% afecta a RNMBP16. En nuestro estudio, teniendo en consideración los 35 RN, de 14 casos de sepsis clínica, 10 afectaron a RN pretérmino (71,4%) y 6 a RNMBP (42,8%).
La detección e identificación de microorganismos en sangre por técnicas moleculares se consiguió en un tiempo medio de 7h, tiempo muy inferior a los del hemocultivo, de 48-72h. No obstante, el tiempo requerido para el procesamiento y realización de la técnica es superior para LC-SF. Recientemente se han publicado trabajos17 que han optimizado esta técnica con extracción automatizada, que reducen el tiempo de manipulación durante el proceso de extracción de ácidos nucleicos, que era, por otra parte, el más laborioso de todo el procedimiento de LC-SF. Estos sistemas automáticos de extracción reducen el tiempo total para obtener un resultado en 4h, y minimizan el riesgo de contaminación.
En los episodios donde la sospecha clínica de sepsis fue confirmada con el aislamiento del microorganismo en el hemocultivo, LC-SF detectó 8 de los 10 casos. Estos microorganismos fueron E. coli, E. cloacae, E. faecalis y SCN. Esto muestra el valor de la técnica LC-SF en la identificación de enterobacterias y de E. faecalis, además de confirmar el papel patógeno de SCN en ciertos casos, dado que la interpretación del aislamiento a partir del hemocultivo con una sola toma (generalmente el tipo de muestra disponible en RN) suele generar controversia.
De los 2 episodios restantes con sepsis clínica, en uno de ellos LC-SF detectó P. aeruginosa, microorganismo que se aisló en otras muestras del mismo paciente. Por el contrario, en el hemocultivo se aisló SCN. P. aeruginosa es uno de los principales probables contaminantes generados en el flujo de trabajo en la realización de PCR universal18, y SCN forma parte de la microbiota epitelial y por tanto puede contaminar la sangre extraída. El hecho de que P. aeruginosa se detectara en otras muestras del paciente puede hacer suponer que una posible contaminación del hemocultivo con SCN impidiera la detección del otro patógeno en la sangre. Realmente es difícil determinar cuál es el microorganismo causante de sepsis en estos casos, ya que se dispone solo de una toma. Lo mismo pudo ocurrir en el caso discordante en que LC-SF detectó S. pneumoniae y el hemocultivo detectó SCN.
La determinación de la sensibilidad y especificidad del hemocultivo es difícil, ya que no existe una regla de oro independiente para compararla19. Por ello, aunque sigue considerándose la técnica de referencia para el diagnóstico de sepsis neonatal, tiene un valor muy limitado, debido al elevado número de falsos negativos y falsos positivos. En los RN es frecuente obtener hemocultivos negativos en presencia de fuertes indicios de sepsis clínica16. El factor más importante causante de esto es el volumen de sangre20, que en este grupo de pacientes suele ser inferior a 0,5ml21. Otras causas que pueden influir en estos falsos negativos son las bacteriemias de bajo nivel (aquellas con una concentración de patógenos ≤10UFC/ml en sangre), que se pueden presentar en el 68% de los casos de sepsis22, y el uso de antibióticos intraparto, que pueden estar presentes en la sangre del RN, lo que da lugar a falsos negativos del hemocultivo pero que no impiden el desarrollo de sepsis23. En nuestro estudio, 5 de los 9 casos de sepsis clínica con hemocultivo negativo fueron positivos mediante LC-SF. Son varios los estudios realizados donde queda patente la mayor sensibilidad del LC-SF sobre el hemocultivo en distintos ámbitos clínicos, como son en las unidades de cuidados intensivos12,24-26, en urgencias27, pacientes con neutropenia febril28, y especialmente pacientes en tratamiento antimicrobiano12,13,26.
Por ello, la exactitud diagnóstica de la prueba LC-SF se determinó basándose en el diagnóstico clínico final del pediatra teniendo en cuenta datos clínicos, de laboratorio y microbiológicos, debido a la inferior sensibilidad del hemocultivo con respecto al LC-SF, tal y como queda reflejado en varios estudios11-13.
Se consideraron contaminaciones por SCN los episodios en los que se aisló o detectó el microorganismo que fueron clasificados finalmente como sepsis descartada. Según esto, se demostró una tasa de contaminación del hemocultivo con SCN del 14,3% frente al 2,4% con LC-SF. Teniendo en cuenta que habitualmente solo vamos a disponer de una sola toma en RN, LC-SF probablemente es de mayor utilidad que el hemocultivo para descartar sepsis por SCN. Además, los patógenos importantes son detectados en las primeras 24h de incubación del hemocultivo29,30. Por tanto, se podría pensar que un tiempo de detección corto en el hemocultivo, junto con un LC-SF positivo a SCN u otro microorganismo potencial contaminante (como SGV), podría confirmar una sepsis por los mismos. La detección de P. aeruginosa mediante LC-SF en un caso de sepsis descartada con aislamiento de SCN en el hemocultivo podría deberse a contaminación asociada a la técnica molecular. Sin embargo, la detección de C. parapsilosis por LC-SF en el otro caso es difícil de interpretar. La revisión de la historia del paciente en donde se dio esta circunstancia no reflejó ningún dato adicional que permitiera interpretar el resultado del estudio microbiológico.
Un inconveniente de LC-SF para el manejo de la sepsis neonatal es que está diseñada para sepsis en adultos, y por tanto el espectro de microorganismos detectados no es el óptimo para RN. LC-SF no detecta de forma específica microorganismos comúnmente implicados en sepsis neonatal como Streptococcus agalactiae (aunque está incluido en Streptococcus spp. junto con Streptococcus pyogenes) y Listeria monocytogenes.
Los actuales métodos automáticos de extracción de ácidos nucleicos simplifican el método molecular, pero a pesar de esto, el tiempo requerido de procesamiento de la muestra es muy superior al necesario para procesar un hemocultivo. Por otra parte, las técnicas moleculares permiten obtener la identificación del microorganismo causal en un tiempo menor. Las técnicas moleculares tendrán realmente mayor difusión como método diagnóstico de sepsis cuando se consiga la automatización total del proceso.
Además, para el empleo de técnicas de PCR en el diagnóstico de sepsis neonatal se deben incluir dianas para detectar específicamente los patógenos más frecuentemente implicados en este grupo poblacional. No obstante, LC-SF podría confirmar o descartar sepsis clínica en determinados casos que el hemocultivo no resuelve.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.