El diagnóstico de infección por Chlamydia trachomatis es difícil en recién nacidos; sin embargo, este se realiza mediante el cultivo celular o por la detección de anticuerpos IgM anti-C. trachomatis (anti-CT). La detección de ADN de C. trachomatis en leucocitos de sangre mediante reacción en cadena de la polimerasa (PCR) podría ser una mejor herramienta para el diagnóstico de infección por este patógeno.
Material y métodosSe analizaron 44 recién nacidos, todos ellos prematuros y con peso menor de 2.500g. De cada paciente se obtuvieron muestras de sangre y de lavado nasofaríngeo. El ADN de los leucocitos fue obtenido mediante la técnica de fenol-cloroformo. La detección de C. trachomatis fue llevada a cabo mediante la amplificación del gen ompA utilizando el PCR de punto final. Además, se realizaron las pruebas de cultivo celular y la detección de anticuerpos IgM anti-CT mediante la técnica de microinmunofluorescencia.
ResultadosVeinte pacientes fueron PCR-positivo (45,5%), y la prueba se asoció significativamente con la presencia de neumonía (RR=2,28; IC95%: 1,01-5,17; p=0,035). El cultivo celular de lavado nasofaríngeo solo fue positivo en 7 muestras y no hubo asociación significativa con algún dato clínico o de laboratorio. El título de anticuerpos anti-CT asociado al PCR-positivo fue 1:32 (RR=2,74; IC95%: 1,21-6,23; p=0,008); sin embargo, este título no se asoció a la presencia de neumonía.
ConclusiónLa detección de ADN en leucocitos de sangre periférica podría ser útil para el diagnóstico de infección por C. trachomatis.
Diagnosis of Chlamydia trachomatis infection in newborns is difficult; however, this diagnosis is performed by cell culture or by detection of IgM antibodies against C. trachomatis. Detection of C. trachomatis DNA in peripheral blood leukocytes using polymer chain reaction (PCR) may be a better tool for the diagnosis of infection by this pathogen.
Material and methodsA total of 44 premature newborns, all weighing less than 2500g, were included in the study. A blood sample and nasopharyngeal lavages were obtained from each newborn. Leukocyte DNA was obtained by phenol-chloroform extraction technique. Detection of C. trachomatis was performed by amplifying the ompA gene using the PCR endpoint. Cell culture tests and the detection of IgM antibodies against C. trachomatis by microimmunofluorescence assay were also performed.
ResultsTwenty newborns were PCR-positive (45.5%), with this test being significantly associated with the presence of pneumonia (RR=2.28; 95%CI: 1.01 to 5.17; P=.035). The cell culture of nasopharyngeal lavage was positive in only 7 samples and no significant association was observed with any clinical or laboratory data. The titer of IgM antibodies against C. trachomatis associated with PCR-positive was 1:32 (RR=2.74; 95%CI: 1.21 to 6.23; P=.008), however this titer was not associated with the presence of pneumonia.
ConclusionDNA detection in peripheral blood leukocytes could be useful for diagnosis of C. trachomatis infection.
Chlamydia trachomatis es considerada la bacteria que se aísla con mayor frecuencia en las infecciones de transmisión sexual1. En la mujer embarazada puede provocar embarazos ectópicos, partos pretérmino, rotura prematura de membranas, corioamnionitis, aborto espontáneo y mortalidad perinatal2. El recién nacido puede adquirir la infección cuando pasa a través del canal de parto o durante la rotura prematura de membranas, y posiblemente también in utero3-5. Las manifestaciones clínicas más comunes en el recién nacido son la conjuntivitis o la neumonía, las cuales aparecen entre las 4 y las 11 semanas después del nacimiento3,5-7. El diagnóstico de infección por C. trachomatis en el recién nacido es difícil y en algunas ocasiones tardío, y los métodos empleados con mayor frecuencia son la detección de anticuerpos IgM anti C. trachomatis mediante la prueba de microinmunofluorescencia (MIF)7-9 y la detección de antígeno mediante el cultivo celular e inmunofluorescencia directa10,11. Se ha informado que uno o más órganos de los recién nacidos pueden ser colonizados por esta bacteria; esta infección generalizada posiblemente es mediante vía hematógena12,13. Los monocitos son considerados como los susceptibles a la infección por esta bacteria, los cuales en el tejido se transformarán en macrófagos14,15. Desde el año 2001 se ha detectado Chlamydia trachomatis y/o Chlamydia pneumoniae en leucocitos de sangre periférica de personas adultas y niños en edad preescolar y escolar15,16. El objetivo de esta investigación fue determinar la utilidad de la técnica de reacción en cadena de la polimerasa (PCR) para la detección de ADN de C. trachomatis en sangre de recién nacidos con dificultad respiratoria.
Material y métodosPacientes y muestrasCincuenta recién nacidos con datos clínicos de neumonía atípica o sepsis fueron evaluados para la detección de ADN de C. trachomatis en leucocitos de sangre periférica. Se extrajeron 500μl de sangre en tubos microtainer con EDTA mediante la punción de la vena basílica del dorso de la mano. De igual forma se obtuvieron muestras de lavado nasofaríngeo, las cuales fueron depositadas en medio de transporte 2SP. Las muestras de sangre fueron centrifugadas a 3.000g×10min a 10°C, posteriormente se recuperaron tanto la capa de células blancas (Buffy coat) como el plasma. Los leucocitos fueron utilizados para la extracción de ácidos nucleicos y la identificación de ADN de Chlamydia mediante PCR de punto final. El plasma fue guardado en tubos Eppendorf a −70°C para determinar la presencia de anticuerpos IgM anti C. trachomatis mediante MIF. Las muestras de lavado nasofaríngeo fueron utilizadas para el diagnóstico de C. trachomatis mediante cultivo celular e inmunofluorescencia directa.
Obtención de ADNAntes de la obtención del ADN de los leucocitos, se eliminaron los eritrocitos mediante 2 lavados con 1ml de la solución de lisis de glóbulos rojos (Tris-HCl 10mM, MgCl 5mM y NaCl 10mM) por centrifugación a 1.500g×7min a temperatura ambiente. Ya eliminados los eritrocitos, los leucocitos fueron incubados con 200μl de la solución de lisis para glóbulos blancos (Tris base 10mM, EDTA 10mM, NaCl 50mM, SDS 0,2%, pH 7,6 con 10μl de proteinasaK a una concentración de10mg/ml) en baño maría a 55°C por 1h. Posteriormente el ADN fue obtenido mediante la técnica de fenol-cloroformo.
Detección de ChlamydiaSe emplearon 2 técnicas de reacción en cadena de la polimerasa (PCR) de punto final para confirmar la presencia del ADN de C. trachomatis. La primera de ellas fue para amplificar un producto del gen ompA de 129pb y la segunda, para obtener un producto del mismo gen de 1.142pb. Los iniciadores empleados para la primer PCR fueron los reportados por Dutilh et al.17: Momp1 (5′-GCCGCTTTGAGTTCTGCTTCCTC-3′); Momp2 (5′-CCAAGTGGTGCAAGGATCGCA-3′), y en la segunda PCR se utilizaron los iniciadores reportados por Yang et al.18: OMP1 (5′-GCCGCTTTGAGTT CTGCTTCCTC-3′) y OMP2 (5′-ATTTACGTGAGCAGCTCTCT CAT-3′). En la mezcla de reacción se utilizaron las siguientes concentraciones: 1,75mM de MgCl2, 0,2mM de dNTP, 25pM de cada uno de los iniciadores, 2,5U de Taq polimerasa (GoTaq ® Flexi ADN polimerasa de Promega©, Madison, WI, EE.UU.) y 5μl del ADN de la muestra para un volumen final de 25μl. En la primera PCR, la mezcla de reacción se incubó durante 5min a 95°C, seguido por 35 ciclos de 1min a 95°C para la desnaturalización, seguido de 2min a 61°C para la alineación y 1min a 70°C para la extensión. La elongación final fue de 5min a 70°C. La segunda PCR se realizó bajo las mismas condiciones, solo cambió el tiempo y la temperatura de alineación, las cuales fueron: 1min a 59°C (termociclador: MJ Research, Inc.Mod. PTC-100. Watertown, Mass, EE.UU.).
Detección de Mycoplasma spp.Para la detección de Mycoplasma se utilizó la técnica de PCR descrita por Van Kuppeveld et al.19, y los iniciadores utilizados fueron: MGSO (5′-GCACCATCTGTCACTCTGTTAACCTC-3′ y GPO-1 (5′-ACTCCTACGGGAGGCAGC AGTA-3′). La mezcla y las condiciones de reacción fueron utilizadas tal como se describió previamente13.
MicroinmunofluorescenciaLa técnica de microinmunofluorescencia (MIF) se llevó a cabo de acuerdo con el procedimiento descrito en el instructivo de los proveedores (Chlamydia pneumoniae IgG/IgM Micro-IF Test kit, Ani Labsystems Ltd. Oy, Vantaa, Finlandia). La presencia de fluorescencia y la intensidad de esta se evaluaron mediante un microscopio de epifluorescencia Axio Scope.A1 (Carl Zeiss, Alemania).
Cultivo celular para Chlamydia trachomatisPara el cultivo celular se emplearon células McCoy, y la detección de Chlamydia trachomatis se llevó a cabo mediante inmunofluorescencia directa empleando el kit comercial Chlamydial Direct IF (BioMérieux SA. F-69280 Marcy l’Etoile, Francia); posteriormente se siguió el procedimiento tal y como se describió previamente20.
Cultivo celular para virus sincitial respiratorioLas muestras de lavado nasofaríngeo fueron depositadas en el medio de transporte, el cual fue utilizado para infectar células Hep-2 y Vero. La identificación del virus se realizó mediante el uso de anticuerpos monoclonales anti-virus sincitial respiratorio (RSV3-FITC Novocastra, Leica Biosystem, Balliol Business Park West, Reino Unido). El procesamiento de las muestra se realizó tal y como se describió previamente10.
Cultivo microbiológicoSe realizaron cultivos microbiológicos de aspirado bronquial. Se utilizaron medios artificiales selectivos y diferenciales para el aislamiento de las bacterias y levaduras; la identificación de estos se realizó mediante pruebas bioquímicas en un equipo automatizado Vitek 2 compact (BioMérieux)
Análisis estadísticoLos resultados fueron sometidos a análisis estadístico mediante la prueba t de Student (a un nivel de significación de p=0,05) para pruebas paramétricas. Para pruebas no paramétricas se utilizó el ji-cuadrado. La magnitud de las asociaciones entre las variables fue expresada como riesgo relativo (RR) en un intervalo de confianza del 95%. Las diferencias fueron consideradas estadísticamente significativas cuando el valor de p fue menor de 0,05. La información obtenida fue analizada con el programa SPSS para Windows (versión 17.0.1, 2008; IBM Corporation, New Orchard Road. Armonk, NY, EE.UU.). También se determinaron la sensibilidad y la especificidad de cada una de las pruebas para la detección de C. trachomatis. Para ello se consideraron pacientes positivos a la infección por esta bacteria cuando 2 o más de las pruebas de diagnóstico fueron positivas (amplificación positiva del gen ompA, cultivo celular positivo o título de anticuerpos IgM mayor o igual a 1:32).
ResultadosEn la tabla 1 se muestra la información referida a los datos clínicos y microbiológicos de la población estudiada. Se analizaron solo 44 recién nacidos de un total de 50, todos ellos con dificultad respiratoria. Seis pacientes fueron excluidos debido a la falta de datos clínicos.
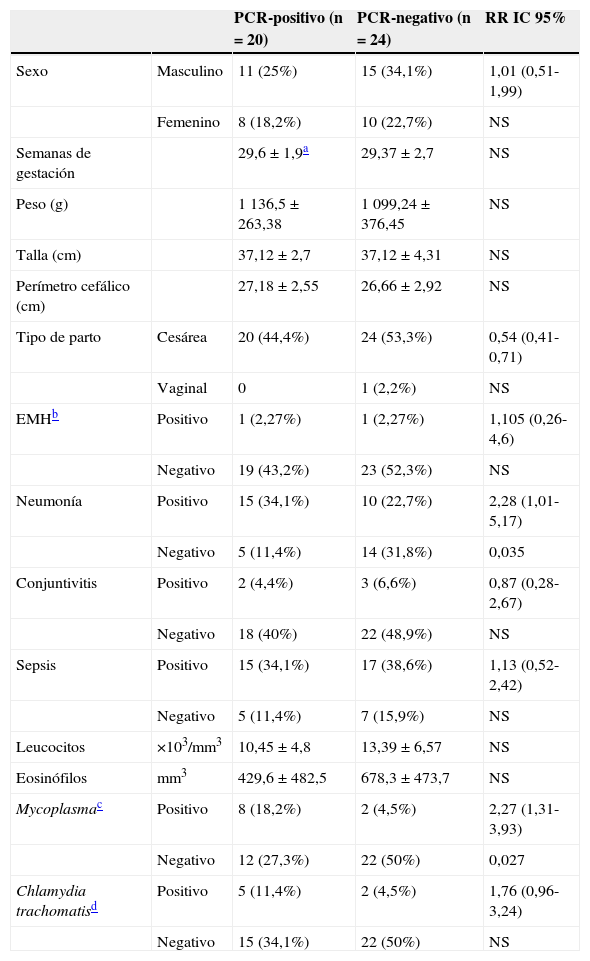
Características clínicas y microbiológicas de neonatos positivos y negativos a PCR para la detección de ADN de Chlamydia trachomatis
| PCR-positivo (n=20) | PCR-negativo (n=24) | RR IC 95% | ||
|---|---|---|---|---|
| Sexo | Masculino | 11 (25%) | 15 (34,1%) | 1,01 (0,51-1,99) |
| Femenino | 8 (18,2%) | 10 (22,7%) | NS | |
| Semanas de gestación | 29,6±1,9a | 29,37±2,7 | NS | |
| Peso (g) | 1 136,5±263,38 | 1 099,24±376,45 | NS | |
| Talla (cm) | 37,12±2,7 | 37,12±4,31 | NS | |
| Perímetro cefálico (cm) | 27,18±2,55 | 26,66±2,92 | NS | |
| Tipo de parto | Cesárea | 20 (44,4%) | 24 (53,3%) | 0,54 (0,41-0,71) |
| Vaginal | 0 | 1 (2,2%) | NS | |
| EMHb | Positivo | 1 (2,27%) | 1 (2,27%) | 1,105 (0,26-4,6) |
| Negativo | 19 (43,2%) | 23 (52,3%) | NS | |
| Neumonía | Positivo | 15 (34,1%) | 10 (22,7%) | 2,28 (1,01-5,17) |
| Negativo | 5 (11,4%) | 14 (31,8%) | 0,035 | |
| Conjuntivitis | Positivo | 2 (4,4%) | 3 (6,6%) | 0,87 (0,28-2,67) |
| Negativo | 18 (40%) | 22 (48,9%) | NS | |
| Sepsis | Positivo | 15 (34,1%) | 17 (38,6%) | 1,13 (0,52-2,42) |
| Negativo | 5 (11,4%) | 7 (15,9%) | NS | |
| Leucocitos | ×103/mm3 | 10,45±4,8 | 13,39±6,57 | NS |
| Eosinófilos | mm3 | 429,6±482,5 | 678,3±473,7 | NS |
| Mycoplasmac | Positivo | 8 (18,2%) | 2 (4,5%) | 2,27 (1,31-3,93) |
| Negativo | 12 (27,3%) | 22 (50%) | 0,027 | |
| Chlamydia trachomatisd | Positivo | 5 (11,4%) | 2 (4,5%) | 1,76 (0,96-3,24) |
| Negativo | 15 (34,1%) | 22 (50%) | NS |
Todos los pacientes analizados fueron menores de 36semanas de gestación (rango de 24,7-35±2,34) y con peso al nacimiento menor de 2.500g (rango de 600-2.065±326,7). En un solo paciente se aisló C. albicans del aspirado bronquial, y en ningún caso se evidenció infección por virus sincitial respiratorio. A 20 de los recién nacidos (45,5%) se les detectó ADN de C. trachomatis (PCR-positivos) en sus leucocitos de sangre periférica (tabla 1). La neumonía y la sepsis fueron los 2 padecimientos que se reportaron con mayor frecuencia al momento de la toma de las muestras; sin embargo, solo la neumonía se asoció significativamente con los pacientes que fueron PCR-positivo (RR=2,28; IC95%: 1,01-5,17; p=0,035). En cuanto a los pacientes que mostraron sepsis, 13/15 fueron PCR-positivo con neumonía (RR=2,17; IC95%: 0,73-6,46; p=NS, datos no mostrados) y solo 2 de estos tuvieron hemocultivo positivo, uno a S. aureus y otro a Enterobacter cloacae (datos no mostrados). El peso, la talla y la circunferencia cefálica se asociaron directamente con la edad gestacional (coeficiente de correlación Sperman rho 0,44; 0,57; 0,50; p=0,003, respectivamente; datos no mostrados) y no con la detección del ADN de C. trachomatis.
Otro de los patógenos identificados en 10 muestras de ADN en sangre fue Mycoplasma spp. Ocho de estos se asociaron con la presencia de ADN de C. trachomatis (RR=2,27; IC95%: 1,31-3,93; p=0,027). En cuanto a los niveles de leucocitos totales y de eosinófilos, no estuvieron asociados con la PCR-positiva.
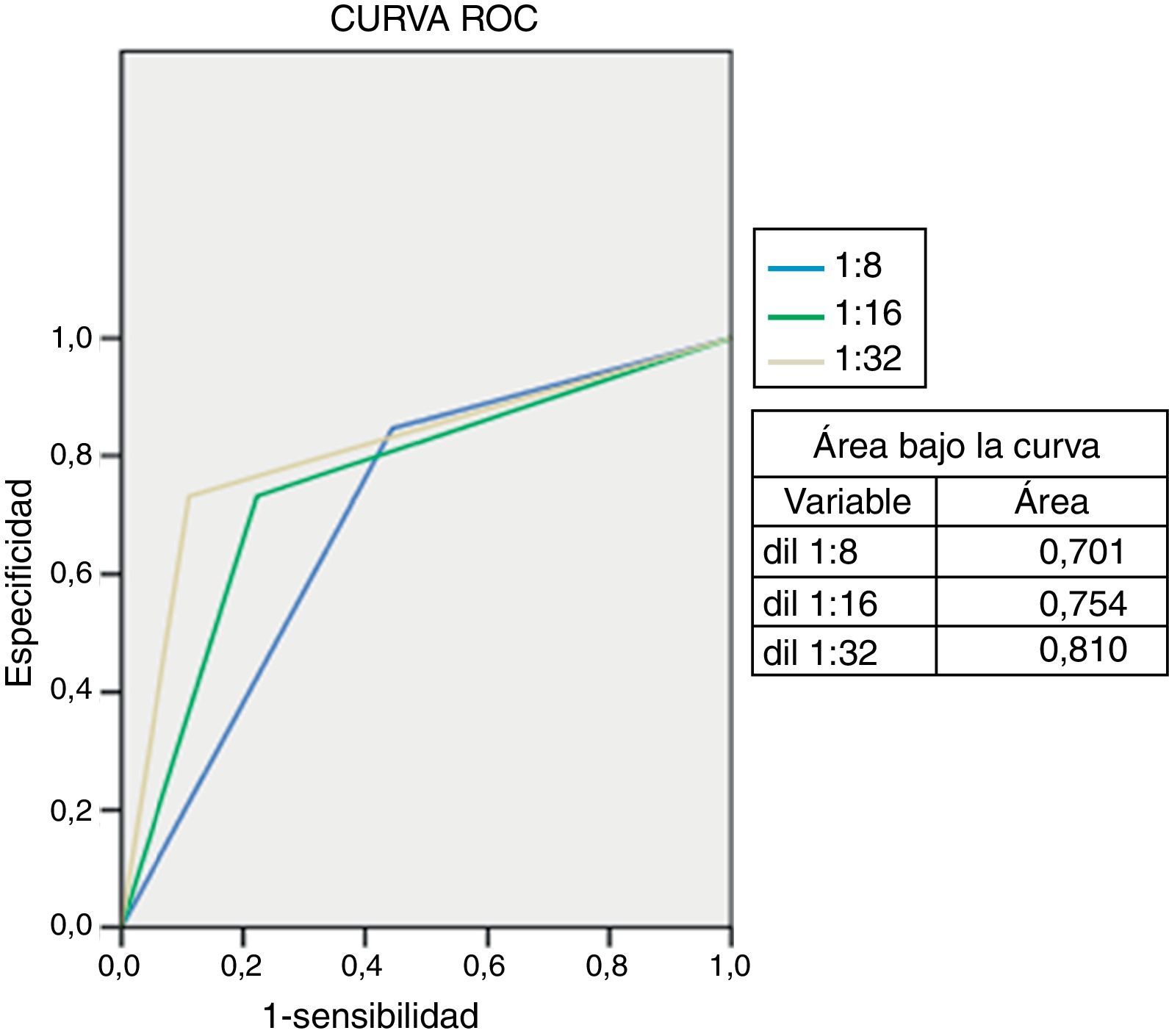
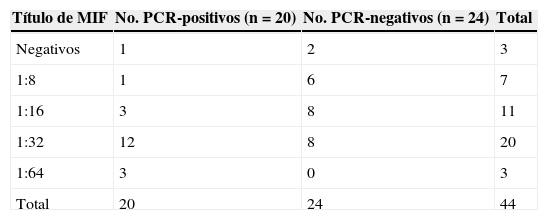
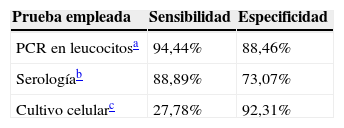
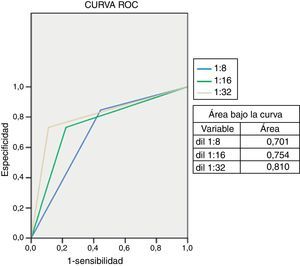
Otro aspecto estudiado fue la serología, mediante la cual se llevó a cabo la detección de anticuerpos IgM anti-C. trachomatis; los resultados obtenidos se muestran en la tabla 2. En 23 pacientes se identificaron títulos mayores o iguales a 1:32; de ellos, 15 fueron PCR-positivo (RR=2,74; IC95%: 1,21-6,23; p=0,008). Al identificar la sensibilidad y la especificidad de la prueba serológica se encontró que estas eran del 88,89 y del 73,07%, respectivamente. El análisis de la curva ROC indica que la prueba es menos sensible para identificar a neonatos con infección por C. trachomatis (fig. 1). En cuanto al cultivo celular, se identificaron 7 muestras positivas, de las cuales solo 5 fueron PCR-positivo, mostrando una sensibilidad y una especificidad del 27,78 y del 92,31%, respectivamente (tabla 3).
Distribución del título de anticuerpos IgM por microinmunofluorescencia (MIF) contra pacientes positivos o negativos a PCR para Chlamydia trachomatis
| Título de MIF | No. PCR-positivos (n=20) | No. PCR-negativos (n=24) | Total |
|---|---|---|---|
| Negativos | 1 | 2 | 3 |
| 1:8 | 1 | 6 | 7 |
| 1:16 | 3 | 8 | 11 |
| 1:32 | 12 | 8 | 20 |
| 1:64 | 3 | 0 | 3 |
| Total | 20 | 24 | 44 |
Sensibilidad y especificidad de las pruebas de diagnóstico para la detección de Chlamydia trachomatis
| Prueba empleada | Sensibilidad | Especificidad |
|---|---|---|
| PCR en leucocitosa | 94,44% | 88,46% |
| Serologíab | 88,89% | 73,07% |
| Cultivo celularc | 27,78% | 92,31% |
La sensibilidad y la especificidad se calcularon en función a que 2 pruebas o más fueran positivas.
Las afecciones que se producen durante una infección por C. trachomatis en el recién nacido son la neumonía y la conjuntivitis, las cuales aparecen entre la cuarta y la undécima semana de vida21. El diagnóstico de infección por este patógeno es difícil; sin embargo, se realiza mediante el cultivo celular o por la detección de anticuerpos IgM anti-C. trachomatis.
El cultivo celular es considerado como el estándar de oro debido a que se pueden observar los cuerpos elementales y reticulares cuando se utiliza para la detección una técnica de inmunofluorescencia directa10,22. Sin embargo, diversos estudios han informado que esta técnica tiene una sensibilidad del 50 al 85%22,23. En este estudio solo 7 pacientes fueron positivos a la infección por este patógeno, y el análisis estadístico no mostró ninguna asociación con los datos clínicos y microbiológicos que mostraron los pacientes. Solo por mencionar, 5 de estos pacientes mostraron neumonía y fueron PCR-positivos. Aunque la especificidad que mostró el cultivo celular en este estudio fue aceptable, la sensibilidad fue baja; posiblemente esto se debió a que la muestra fue mal tomada o por la presencia de sustancias que inhiben el crecimiento de la bacteria, o que dañen las células del cultivo celular.
Actualmente la detección de anticuerpos no es considerada como una prueba eficiente para identificar infecciones activas por C. trachomatis en pacientes adultos. Sin embargo, la identificación de anticuerpos IgM anti-C. trachomatis es apreciada como una prueba de diagnóstico en aquellos niños que muestran neumonía3,7. La MIF, que fue desarrollada en los años setenta y consiste en la reacción de los anticuerpos del paciente con los cuerpos elementales de Chlamydia, es considerada como el mejor método para el diagnóstico de infección por C. pneumoniae, reportándose una sensibilidad hasta del 92%9,22,24. En esta investigación se encontró que solamente el título de anticuerpos 1:32 se asoció significativamente con la detección de ADN de C. trachomatis obtenido de los leucocitos de estos pacientes y con una asociación negativa con aquellos que mostraron conjuntivitis (RR=−2,43; IC95%: 1,67-3,55; p=0,019), es decir, que la infección en la conjuntiva posiblemente no induce la producción de anticuerpos. Además se demostró que esta prueba tiene una sensibilidad buena pero una especificidad baja, lo cual corrobora la reactividad cruzada observada entre C. pneumoniae y C. psittaci; esta reactividad probablemente está dirigida hacia el antígeno de género que es el lipopolisacárido22.
En cuanto a las técnicas de amplificación de ácidos nucleicos, se reporta que tienen una sensibilidad del 90 al 95% y que en teoría pueden detectar hasta un cuerpo elemental22,23. Además, la adición experimental de cuerpos elementales en muestras de esputo ha demostrado que estas técnicas son capaces de detectar hasta 5 cuerpos elementales25. También se ha informado que la detección ADN de C. pneumoniae y C. trachomatis en sangre es posible15,25. En este estudio se demostró que efectivamente se puede identificar el ADN de C. trachomatis en muestras de sangre, ya que se logró amplificar el gen ompA de este patógeno mediante 2 técnicas diferentes de PCR.
El análisis estadístico mostró que la técnica de PCR-positiva estuvo asociada significativamente con la presencia de neumonía, con la dilución del suero y con la presencia de ADN de Mycoplasma.
Se ha informado que C. trachomatis está involucrado en el desarrollo de parto pretérmino, rotura prematura de membranas, corioamnionitis y bajo peso al nacimiento2. En esta investigación no se logró demostrar asociación con parto pretérmino, ya que todos los recién nacidos estudiados fueron menores de 35semanas de gestación. En cuanto a la rotura prematura de membranas, estuvo presente en 12 pacientes; sin embargo, no hubo asociación con la PCR-positiva, la corioamnionitis solo estuvo presente en un paciente y el bajo peso estuvo más asociado a las semanas de gestación que a la PCR-positiva.
Otros hallazgos de laboratorio importantes que se han descrito en los recién nacidos infectados con C. trachomatis es la presencia de una eosinofilia. Numazaki et al.21 han reportado eosinofilia de 400células/mm3 o más. Sin embargo, en este estudio 11/20 PCR-positivo mostraron eosinofilia menor a este valor. Otros microorganismos que pueden desarrollar signos clínicos de neumonía semejantes a los de C. trachomatis son las infecciones por virus sincitial respiratorio, citomegalovirus y Ureaplasma urealiticum13,21,26. En este estudio se observó que en ningún paciente hubo infección por virus sincitial respiratorio. Para citomegalovirus no se realizó el cultivo celular; no obstante, sus anticuerpos IgM anti-citomegalovirus fueron negativos. A pesar de lo anterior, una de las limitaciones del estudio es que no se identificaron otros virus, tales como virus de la parainfluenza, coronavirus y rinovirus27. Aunque cabe señalar que el virus más frecuente en niños menores de 2 meses de edad es el virus sincitial respiratorio, siendo hasta del 3% en recién nacidos prematuros menores de 32semanas de gestación28. Otra limitante del estudio es que no se evaluó mediante PCR la presencia de C. trachomatis en las muestras de lavados nasofaríngeos. Actualmente existen pocos estudios que indican que hay buena significación estadística al emplear estas muestras29; sin embargo, no existe una evaluación sobre la sensibilidad y la especificidad con respecto a otras técnicas que se utilizan para el diagnóstico de infección por esta bacteria, además de que hasta el 13% de las muestras respiratorias pueden contener sustancias que inhiben la PCR, como la sangre o el moco30,31. Debido a lo anterior se decidió utilizar la serología como la prueba de diagnóstico estándar de infección por C. trachomatis en los recién nacidos y compararla con la detección del ADN de C. trachomatis de los leucocitos de los recién nacidos por la técnica de PCR.
Por otro lado, la identificación del ADN de Mycoplasma en muestras PCR-positivas a C. trachomatis sugiere una co-infección con ambas bacterias. Cabe señalar que Mycoplasma hominis y Ureaplasma urealyticum son consideradas como parte de la flora normal de las mujeres con vida sexual activa, y en la actualidad existen evidencias clínicas y experimentales de que estos organismos contribuyen al desarrollo de corioamnionitis, de una respuesta inflamatoria fetal, de nacimientos pretérmino, de displasia broncopulmonar, de hemorragias intraventriculares y de enterocolitis necrosante32.
Además, la co-infección entre C. trachomatis y especies de Mycoplasma o Ureaplasma ha sido reportada en pacientes con VIH33 y en mujeres de 15 a 64años de edad, que muestran infecciones genitales hasta en el 9% de los casos34. De manera similar, se ha reportado la co-infección entre Chlamydia pneumoniae y Mycoplasma pneumoniae en infecciones de vías respiratorias en niños menores de 15años35. Debido a lo anterior, no es extraño que ambos patógenos hayan sido detectados en estas muestras y la presencia de ambos pudiese haber exacerbado la enfermedad respiratoria en estos pacientes. Sin embargo, estos pacientes no mostraron datos clínicos y de laboratorio diferentes a los infectados solamente con C. trachomatis.
Las infecciones respiratorias pueden ser producidas por otras bacterias o levaduras diferentes a Chlamydia o Mycoplasma; en esta investigación todos los cultivos microbiológicos de los aspirados bronquiales fueron negativos, excepto en un paciente, que mostró Candida albicans; esta levadura pudo haberse adquirido de manera congénita, aunque este neonato no presentó manifestaciones clínicas en la piel, por lo que esta infección se consideró de adquisición nosocomial36,37.
Finalmente, se concluye que el uso de PCR de punto final en leucocitos de sangre periférica es útil para la detección de ADN de C. trachomatis y muestra mayor sensibilidad para la detección de recién nacidos que han sido infectados por este patógeno.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.