Seleccionar individuos cuya obesidad mórbida (OM) se pueda atribuir preferentemente al perfil genético individual. Tras descartar pacientes con posibles síndromes monogénicos y otras enfermedades con obesidad asociada, evaluar la asociación de la variabilidad del gen FTO (asociado con la masa grasa y la obesidad), sobre la base de los polimorfismos de un solo nucleótido (SNP) con rs1861868 y rs9939609, con la predisposición heredada a padecer OM.
Pacientes y métodosTras evaluar a 270 pacientes con OM instaurada antes de los 14 años, seleccionamos a 194 por su fenotipo y la historia familiar referida; se incluyó a 289 individuos controles. Se genotiparon los cambios en el SNP rs1861868 y rs9939609 del gen FTO, y comparamos sus frecuencias genotípicas y haplotípicas en ambos grupos poblacionales.
ResultadosSe confirmó la asociación del alelo A del SNP rs9939609 con la obesidad grave instaurada en la infancia en la población española. Los individuos portadores del haplotipo rs1861868 G/rs9939609 A del gen FTO mostraron un incremento del riesgo (odds ratio de 3,03; intervalo de confianza del 95%: 1,74–5,27) a padecer obesidad mórbida en nuestra población.
ConclusiónAnalizar las bases genéticas de la obesidad precisa una rigurosa selección de los casos. La asociación del SNP rs9939609 con la obesidad, ampliamente descrita en distintas poblaciones, se confirma en la población española. Identificamos el primer haplotipo de riesgo al analizar el SNP rs1861868 del bloque haplotípico contiguo al que contiene el primero. Es preciso estudiar en profundidad la variabilidad interindividual del gen FTO para identificar la causa de su capacidad deletérea a la enfermedad.
To select individuals whose morbid obesity can be attributed mainly to their individual genetic profile. After excluding patients with potential monogenic syndromes or diseases associated with obesity, we evaluated the association of the single nucleotide polymorphisms (SNPs) rs1861868 and rs9939609 of the fat-mass and obesity-associated FTO gene with an inherited predisposition to morbid obesity.
Patients and methodsWe evaluated 270 patients with morbid obesity and onset before the age of 14 years and selected 194 due to their phenotypes and family history; 289 control individuals were included. The rs1861868 and rs9939609 variants, located in the FTO gene, were genotyped. Genotype and haplotype frequencies were compared between cases and controls.
ResultsThe A allele of rs9939609 was associated with severe obesity starting in childhood among the Spanish population. The rs1861868 G/rs9939609 A haplotype of the FTO gene was also significantly associated with severe obesity in our population, with an odds ratio of 3.03 (95% confidence interval, 1.74–5.27).
ConclusionAnalysis of the genetic basis of obesity requires rigorous selection of cases. In this study, the association of the rs9939609 SNP with obesity widely described in distinct populations was confirmed among overweight Spanish children. Genotyping rs1861868 allowed us to identify the first risk haplotype in the FTO gene, which is located in the adjacent haplotype block containing rs9939609. In-depth study of the variability of the FTO gene is essential to define its deleterious capacity.
La epidemiología genética constituye en la actualidad una de las disciplinas fundamentales en el abordaje de la obesidad. La complejidad y la diversidad del diseño de sus análisis favorecen la identificación de nuevos loci que puedan contener genes con variantes de susceptibilidad. La obesidad es un ejemplo perfecto de enfermedad en la que interaccionan factores no genéticos y genéticos, ajustados a modelos de riesgo difíciles de definir y calcular1. Los distintos aspectos de su etiología han creado la necesidad de diferenciar con rigurosidad las alteraciones fisiológicas capaces de desequilibrar los mecanismos de ajuste entre el aporte de sustratos combustibles y las demandas de energía, los cuales tienen por objeto mantener una masa corporal estable. A pesar del importantísimo avance que han supuesto la identificación y la caracterización de los síndromes monogénicos de obesidad mórbida (OM) no sindrómica, el porcentaje de casos de obesidad manifiesta que se explica por su conjunto es difícilmente superior al 5%2. A partir del interés por aplicar estrategias de prevención primaria, surge la prioridad de identificar el máximo porcentaje de individuos cuya susceptibilidad heredada a padecer OM sea el agente causal principal para producirla. Para esto se desarrollan inversiones de gran magnitud para el estudio en profundidad de las bases poligénicas de la enfermedad.
Una de las herramientas más interesantes es el genome wide association study (GWAS, ‘estudio de asociación sobre el genoma completo’). A pesar de que la heterogeneidad y el coste-beneficio de los proyectos de GWAS son un tema en debate y generan una controversia patente entre los expertos3, la avalancha de publicaciones de prestigio basadas en sus resultados ha permitido la descripción de un importantísimo listado de factores genéticos de riesgo a enfermedades comunes, entre ellas la obesidad4. Su utilidad crece al combinarse con los resultados de los estudios tradicionales de casos y controles, que tratan de replicar y definir con exactitud los riesgos de las variantes sugeridas, así como de sus combinaciones, en los genes candidatos identificados.
El gen FTO (asociado con la masa grasa y la obesidad) se localiza en el locus 16q12.2, el cual ha mostrado un evidente ligamiento con la obesidad en diversos estudios, como los mencionados5–10. A pesar de que las revisiones bibliográficas señalan cierta controversia respecto a ciertos polimorfismos de un solo nucleótido (SNP) del gen FTO, sobre todo entre diferentes etnias, su variabilidad interindividual sigue considerándose de enorme interés. Se han corroborado los primeros resultados extraídos de los GWAS, utilizando diferentes estrategias de análisis, confirmándose ampliamente la relación de la variante rs9939609 con la existencia de sobrepeso grave y algunas de sus comorbilidades en poblaciones europeas11–14.
Las funciones del gen FTO parecen relacionarse, al igual que las de los genes responsables de los síndromes de obesidad monogénica no sindrómica15–17, con el control hipotalámico de la saciedad, la hiperfagia y la ansiedad manifiesta ante la restricción de comida; también existen trabajos que han estudiado su relación con la susceptibilidad a comportamientos adictivos. Se ha demostrado su expresión en los núcleos cerebrales que ejercen el control de la alimentación, modificándose ante situaciones de ayuno. En los individuos portadores del alelo A se ha descrito, en los núcleos cerebrales donde se expresa la proteína que codifica FTO, una mayor resistencia a la insulina, así como la preferencia por ingestas hipercalóricas13.
Consideramos la variante genética rs9939609, candidata a estudio en la población española y a un análisis exhaustivo en pacientes afectados de obesidad manifiesta desde la infancia. Entre éstos suelen existir claros patrones de agregación familiar, con otros casos de obesidad grave de aparición temprana. Dadas las características y la localización del polimorfismo seleccionado, así como la no asociación con el riesgo a desarrollar obesidad que se ha descrito en poblaciones no caucásicas18,19, se precisó una mejor descripción, valoración y análisis de otros cambios genéticos que definen la variabilidad interindividual del gen FTO. También es posible que la existencia de una variante genética de ancestro caucásico, aún no identificada, sea el agente causal del efecto observado para el rs9939609, en desequilibrio de ligamiento con éste.
Por esto, propusimos la construcción de haplotipos que lo contuvieran, analizando otros SNP que aportaran información de bloques de desequilibrio de ligamiento contiguos. Seleccionamos el SNP con rs1861868 por estar localizado en una zona de la secuencia del gen FTO, con la que segrega en desequilibrio de ligamiento, situada previamente a la región donde se encuentra el rs9939609, de características similares. Estudiamos la distribución de los cambios seleccionados (rs1861868 y rs9939609), así como la de sus combinaciones, en ambos grupos de nuestra serie. Se sugirió que podrían contribuir a la existencia de diferentes perfiles de riesgo heredado y transmisible para el incremento de peso grave.
Fue preciso describir el impacto de las bases genéticas en la etiopatogenia de la enfermedad, así como su modulación por otros factores derivados de hábitos, estilo de vida, estatus socioeconómico y cultural. El conjunto de antecedentes personales y familiares de obesidad recopilados en cada caso nos permitió la aproximación de la posible correlación genotipo-fenotipo en los individuos portadores de las combinaciones de riesgo identificadas.
Pacientes y métodosDurante el bienio 2007–2009 se evaluó a 270 pacientes por parte del grupo de profesionales de la Unidad de Genética del Hospital Infanta Cristina de Badajoz, procedentes de las consultas de Endocrinología del Complejo Hospitalario Universitario de Badajoz (Nutrición y Dietética, y Endocrinología Pediátrica) y de los centros de salud de Valdepasillas, Montijo y Puebla de Obando. Se trataba de adultos con antecedentes de OM establecida antes de los 16 años (índice de masa corporal [IMC] >40kg/m2) y menores de esa edad con peso superior a 3 desviaciones estándares (DE). Recopilamos a 289 individuos controles de nuestro ámbito, pareados por sexo y no relacionados con los casos.
Completamos un cuestionario de datos personales y familiares, refiriendo comorbilidades y enfermedades previas. Se realizó la construcción de un árbol genealógico amplio. Atendimos al origen y a la posible consanguinidad en cada familia. Realizamos una exploración física, prestando especial atención al fenotipo del paciente y su familia. Ante cualquier sospecha diagnóstica de un posible caso de obesidad monogénica, sindrómica o no sindrómica, así como de enfermedades con obesidad asociada, se procedió a derivar a consulta de Genética Clínica, Endocrinología Pediátrica, Endocrinología o Nutrición, según se considerase en cada caso. Se recopilaron en el conjunto de pacientes los datos de edad de aparición del sobrepeso (>3 DE), el IMC, la medida de la cintura y la cadera y las concentraciones séricas de glucosa, el colesterol total, el colesterol unido a lipoproteínas de alta densidad, el colesterol unido a lipoproteínas de baja densidad y los triglicéridos.
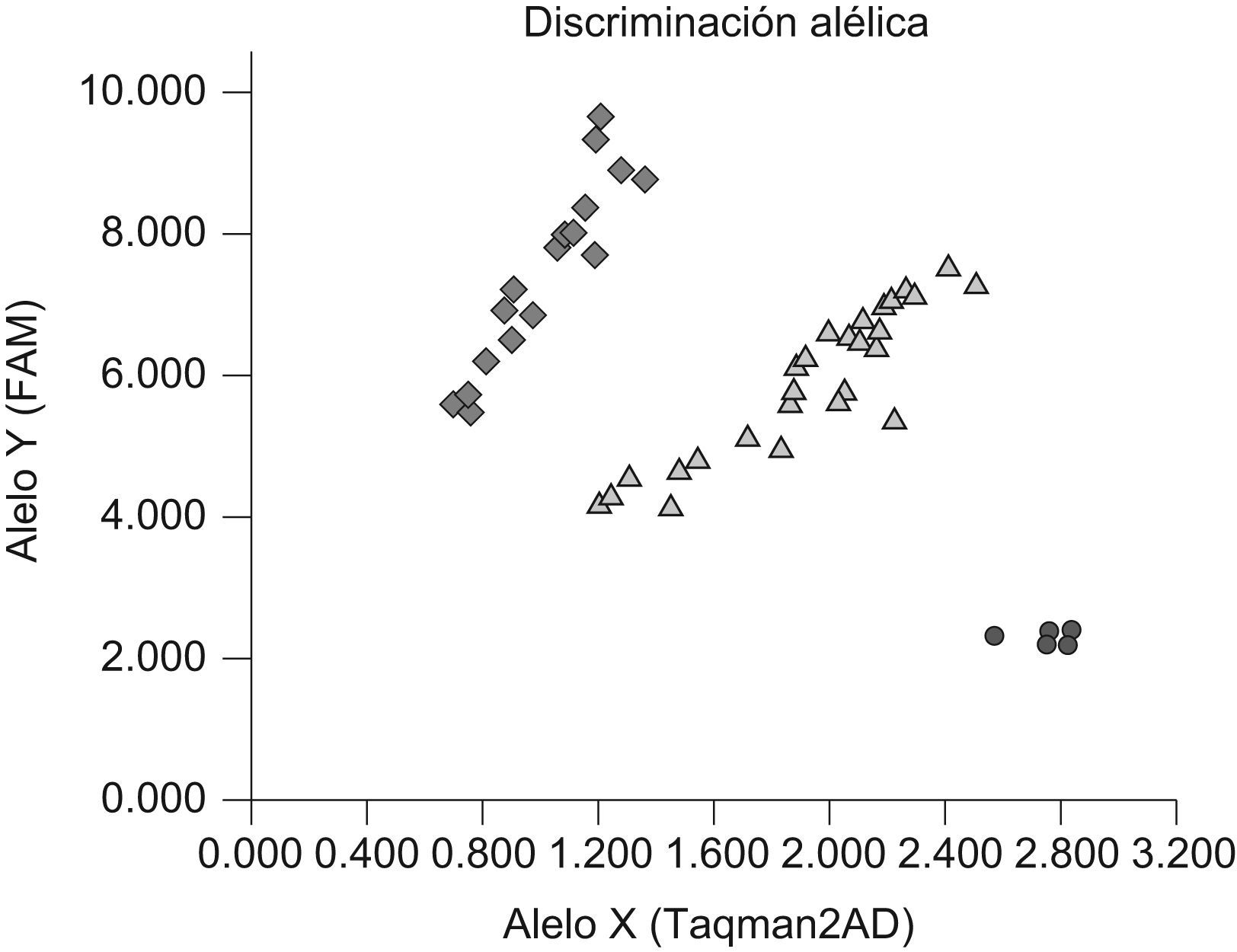
Previo consentimiento informado se obtuvo una muestra de sangre periférica (10ml) anticoagulada con EDTA de los individuos seleccionados a partir del árbol genealógico por haber alcanzado el mayor peso a la edad más temprana. Se extrajo el ADN mediante los reactivos incluidos en el QIAamp DNA Blood Kit. El genotipado se realizó mediante las sondas de discriminación alélica Custom TaqMan® SNP Genotyping Assays de Applied Biosystems, con los códigos de identificación C__11717119_10 y C__30090620_10 para los SNP con rs1861868 y rs9939609, respectivamente (7300 AB) (fig. 1). Se analizó a un total de 194 pacientes y a 289 individuos controles. Aplicamos la ley de Hardy-Weinberg y comparamos las frecuencias genotípicas y haplotípicas en ambos grupos poblacionales. Descripción, valoración y selección del conjunto de SNP del gen FTO detalladas en las bases de datos National Center for Biotechnology Information y Ensembl, así como la diversidad haplotípica descrita en HapMap. Realizamos la predicción bioinformática de la posible capacidad funcional del SNP rs9939609 a partir de la secuencia del SNP y su región flanqueante descrita en Ensembl. La predicción de los sitios de regulación en estas regiones se hizo usando el programa MatInspector sobre la base de datos de TRANSFAC de sitios de unión de factores de transcripción, que compara la secuencia de interés con una matriz de homología de sitios de unión comprobados para cada factor de transcripción.
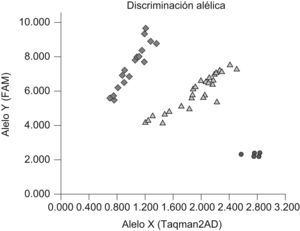
Representación de los resultados obtenidos de un ensayo de discriminación alélica correspondiente al genotipado del polimorfismo de un solo nucleótido con rs9939609 (G/A). Los puntos en rojo representan a individuos homocigotos para el polimorfismo de un solo nucleótido (AA), los triángulos verdes a individuos heterocigotos para el cambio (GA) y los rombos azules a individuos homocigotos normales (GG).
Para los análisis de las frecuencias alélicas, genotípicas y de haplotipos utilizamos el programa SNPStats. Corroboramos la estadística descriptiva y determinados estudios de asociación de los SNP analizados con variables fenotípicas mediante el programa SPSS v.11.5.
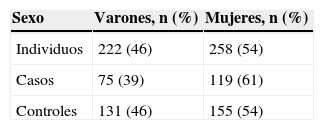
ResultadosLa serie incluyó a un 40% de pacientes (n=194) frente a un 60% de individuos controles (n=289). La distribución por sexos no mostró diferencias significativas (tabla 1). La edad media de los pacientes en la fecha en que fueron incluidos en el estudio fue de 24 años (límites de 6-54 años), siendo 6 años la media de edad en que refirieron haber alcanzado un peso superior a 3 DE. La edad media de los individuos controles se situó en 42 años (límites de 29–81 años).
Tras la evaluación de los 270 pacientes que consultaron por obesidad manifiesta por parte del equipo sanitario multidisciplinario, se atribuyeron criterios a 227 para una valoración exhaustiva por posible origen genético. Cincuenta y tres pacientes fueron evaluados por sospecha de otras enfermedades asociadas con un grave incremento de peso, obesidad monogénica sindrómica o monogénica no sindrómica. El resto fueron considerados candidatos para inclusión en estudios de casos y controles para la búsqueda de perfiles genéticos de susceptibilidad heredada.
Se incluyó a 194 casos en el estudio de asociación diseñado para el genotipado de los SNP rs1861868 y rs9939609 del gen FTO, frente a 289 individuos controles representativos de nuestra población.
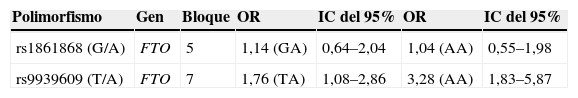
Tras genotipar y analizar la distribución de las variantes en ambas series, así como la prevalencia de sus combinaciones, observamos que el SNP rs1861868 (con frecuencias alélicas descritas en caucásicos: G;0.521/A;0.479) mostró una distribución alélica similar entre pacientes y controles. Los análisis estadísticos sugirieron que la variante no conllevaba riesgo a la enfermedad (tabla 2).
Cálculo de riesgos para los genotipos de los polimorfismos analizados
| Polimorfismo | Gen | Bloque | OR | IC del 95% | OR | IC del 95% |
| rs1861868 (G/A) | FTO | 5 | 1,14 (GA) | 0,64–2,04 | 1,04 (AA) | 0,55–1,98 |
| rs9939609 (T/A) | FTO | 7 | 1,76 (TA) | 1,08–2,86 | 3,28 (AA) | 1,83–5,87 |
IC: intervalo de confianza; OR: odds ratio; SNP: polimorfismo de un solo nucleótido.
El SNP rs9939609 (caucásicos: T;0.580/A;0.410) mostró diferencias estadísticamente significativas entre pacientes heterocigotos y homocigotos (OR de 1,76 y 3,28, respectivamente).
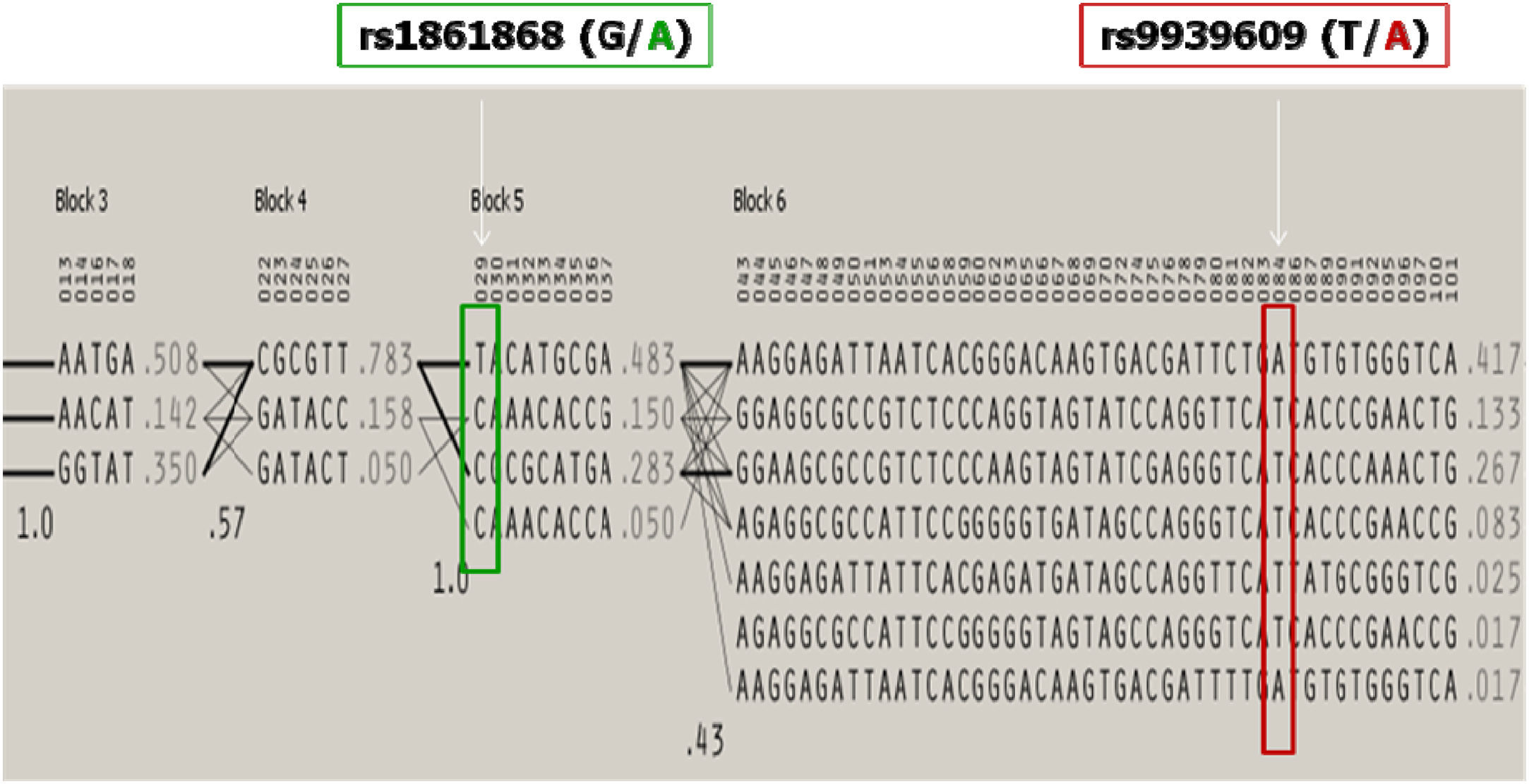
En cambio, el SNP con rs9939609 (frecuencias alélicas en caucásicos: T;0.580/A;0.410) mostró diferencias estadísticamente significativas entre pacientes. Para individuos portadores heterocigotos T/A y homocigotos A/A, definimos una odds ratio (OR) de 1,76 (intervalo de confianza [IC] del 95%: 1,08–2,86) y una OR de 3,28 (IC del 95%: 1,83–5,87), respectivamente (tabla 2). Este polimorfismo segregó en desequilibrio de ligamiento con un conjunto de SNP que se localizan a lo largo de la secuencia del gen FTO, de la cual ofrece información. Dado que se sitúa en una zona intrónica, no codificante, el efecto observado se atribuyó en principio a la región completa (fig. 2).
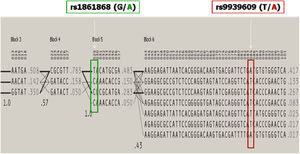
Representación de la distribución de bloques haplotípicos del gen FTO. El haplotipo es un conjunto de polimorfismos de un solo nucleótido existentes de manera contigua en una sola cromátida. Se encuentran asociados en porcentajes identificados y/o calculados de individuos, heredándose en grupos con índices conocidos.
A pesar de su localización y sus características, los resultados de la predicción bioinformática de la posible capacidad funcional del rs9939609 sugirieron un posible efecto per se. El alineamiento de la secuencia del gen a ese nivel del intrón mostró una importantísima conservación entre especies (el 100% de conservación con el gorila, el 99% con el mono rhesus y el 91% con el caballo). La variante A del SNP generó un sitio de unión al factor de transcripción SRY, mientras que la variante G mantuvo este sitio de unión y creó de novo un sitio de unión al factor de transcripción Ets1. Estos hallazgos sugirieron que este polimorfismo podría modificar la regulación transcripcional del gen FTO.
El análisis de la diversidad haplotípica de ambos cambios reveló que la combinación rs1861868 A/rs9939609 A se identificó en el 27,3% de los controles frente a un 37,9% de los casos, y que la combinación rs1861868 G/rs9939609 A aparecía en el 9,5% de los controles frente al 18,6% de los pacientes. El estudio de la distribución de las combinaciones extendió la información obtenida al bloque haplotípico previo y contiguo al SNP de riesgo rs9939609 (fig. 3).
El haplotipo rs1861868 A/rs9939609 A resultó conferir un OR de 2,14 (IC del 95%: 1,38–3,32) frente a la combinación G/T. Las diferencias en la distribución del haplotipo rs1861868 G/rs9939609 A identificaron un incremento del riesgo a padecer OM (OR de 3,03) para los individuos portadores al menos en heterocigosis (IC del 95%: 1,74–5,27) (tabla 3).
Cálculo de riesgos para los haplotipos descritos
| rs1861868 (G/A) | rs9939609 (T/A) | Controles, % | Casos, % | OR | IC del 95% |
| G | T | 33 | 21 | – | – |
| A | T | 31 | 22 | 1,12 | 0,70–1,79 |
| G | A | 9 | 19 | 3,03 | 1,74–5,27 |
| A | A | 27 | 38 | 2,14 | 1,38–3,32 |
IC: intervalo de confianza; OR: odds ratio.
El análisis de diversidad haplotípica reveló que la combinación rs1861868 G/rs9939609 A aparecía en el 9% de los controles frente al 19% de los pacientes y que la combinación rs1861868 A/rs9939609 A aparecía en el 27% entre controles frente al 38% de los casos. Ambos haplotipos mostraron un incremento del riesgo a padecer obesidad mórbida (OR de 3,03 y 2,14, respectivamente).
Los perfiles de riesgo identificados en nuestro estudio se hallaron, solos o en combinación, en el 56,5% de los 194 pacientes frente al 36,8% con el que aparecieron en el grupo control.
No detectamos ninguna asociación de los genotipos de los SNP analizados con el conjunto de las variables y los parámetros bioquímicos disponibles, tampoco con los haplotipos descritos. El análisis de correlaciones sí mostró aun mayor sobrerrepresentación del alelo A en el rs9939609 entre pacientes controles mujeres (p=0,011).
Observamos que un 4,7% de los individuos afectados de obesidad (8 individuos) eran homocigotos para el haplotipo de mayor riesgo (rs1861868GG/rs9939609AA), siendo el porcentaje entre individuos controles del 3,3% (10 individuos). Esta comparación no alcanzó la significación estadística, pero resultó llamativo que el 88% de los casos homocigotos GG/AA (n=7) fueran mujeres, frente al 20% entre los controles (n=2). Los 8 pacientes mostraron una media de edad de 4 años para la instauración de la obesidad grave (peso superior a 3 DE), 5 de ellos relacionaron su obesidad con una actitud ansiosa y compulsiva ante la comida, habiendo precisado dos individuos tratamiento por psiquiatría infantil. Resultó sorprendente que, ante la pregunta de si usaron chupete y cuánto tiempo, las familias de 4 de los pacientes reconocieron un apego llamativo por el chupete, alargando su uso tras el primer año de vida y manteniendo uno de ellos durante años la succión del pulgar para conciliar el sueño. La revisión de los árboles genealógicos mostró en 5 de los 8 casos un patrón evidente de agregación familiar, observándose en todos ellos la existencia de al menos otro caso de OM entre los parientes de primer grado del caso índice, de 1–2 familiares afectados de segundo grado y al menos otros 2 entre los de tercer grado.
DiscusiónNuestra serie pone de manifiesto la heterogeneidad de las bases genéticas de la OM. El análisis en profundidad de cada caso, previo a su inclusión o descarte del estudio, evidencia la necesidad de establecer criterios que permitan una clasificación de los pacientes sobre la base de la etiopatogenia predominante. Estudios como el que presentamos pueden ser de gran ayuda para tal objetivo, y señalan la importancia de la selección correcta de casos para el diseño de estudios genéticos de riesgo a enfermedades complejas20,21. Pretendimos identificar variantes genéticas que, de manera individual o en combinación, definieran perfiles heredados capaces de explicar un porcentaje del riesgo a padecer OM.
Estableciendo como criterios de alto riesgo que el peso alcanzado por edad indicara OM antes de los 14 años y que la valoración del árbol genealógico mostrara una patente agregación familiar, observamos un alto índice de heredabilidad de la enfermedad entre los individuos incluidos en la serie analizada. Se insiste en la bajísima prevalencia de la obesidad monogénica, tanto sindrómica como no sindrómica, en la población general, que, sin embargo, no es despreciable en el contexto de una consulta de genética humana.
La heterogeneidad entre los estudios de los resultados obtenidos, tanto de los GWAS como de los estudios de casos y controles para el análisis de la distribución de los polimorfismos candidatos, es un dato por tener en cuenta para construir definitivamente un perfil genético de riesgo a una enfermedad22. Sin embargo, es evidente que variables como los fallos en el genotipado, la estratificación de poblaciones, la selección adecuada de la serie de individuos controles y, sobre todo, la diversidad fenotípica de los sujetos considerados como casos en los diferentes estudios pueden sugerir falsos negativos.
La variabilidad interindividual del gen FTO es de gran interés en la búsqueda de alelos de susceptibilidad a padecer OM. Corroboramos la asociación del SNP rs9939609 con la enfermedad, identificando en nuestra serie riesgos para los individuos con genotipo heterocigoto (T/A) y homocigoto para el cambio (A/A) (OR de 1,76 y OR de 3,28, respectivamente). Tales resultados corroboran lo publicado en la población del norte de Europa13, respecto a la relación del SNP rs9939609 con elevados IMC; los riesgos calculados en otros estudios para los mismos genotipos son inferiores, inferidos a partir de series más amplias, pero cuyos casos fueron seleccionados únicamente por desarrollar obesidad grave a cualquier edad.
El alelo de riesgo definido apareció con una frecuencia alélica del 55% entre los individuos controles de la población extremeña, frente al 46% descrito en los controles del norte de Europa o al 16% en la población asiática; estudios epidemiológicos podrían revelar la implicación de las diferencias observadas en las distintas tasas de prevalencia de la OM en estas poblaciones.
El análisis de la distribución de la diversidad haplotípica del gen FTO potencia la significación de los hallazgos obtenidos en el análisis univariante. La variabilidad interindividual, sobre la base de los SNP seleccionados, identifica el primer haplotipo de riesgo en la población extremeña; la combinación rs1861868G/rs9939609A muestra un incremento del riesgo (OR de 3,03; IC del 95%: 1,74–5,27) a padecer OM en los individuos portadores, al menos en un alelo.
Aparte del genotipado de tales polimorfismos genéticos se debe disponer de un amplio conjunto de datos clínicos y biológicos, personales y familiares relacionados con su proceso. Nuestro estudio no encontró asociación entre el genotipo del gen FTO y las características específicas de la obesidad desarrollada, aunque la temprana edad de los pacientes puede enmascarar posibles alteraciones bioquímicas diferenciales. Sí reveló resultados preliminares respecto a la mayor capacidad deletérea del gen en la fisiología de la mujer, con un fenotipo basado en la ansiedad de ciertos pacientes, homocigotos para el haplotipo de mayor riesgo, cuya hiperfagia puede tener un efecto sedante para combatirla23. Se ha sugerido ampliamente que el efecto observado del SNP rs9939609 en cuanto a la aparición de la obesidad se relaciona con una respuesta inadecuada a la sensación de hambre e, incluso, a un trastorno de la sensación de saciedad11. La disfunción en el gen FTO podría aumentar la susceptibilidad de ciertos individuos a padecer un estado de ansiedad, no reactivo a ningún factor exógeno, mejorable mediante hábitos como el uso persistente del chupete en la infancia. El inicio de la autonomía para la ingesta en el niño puede buscar, complementar e incluso sustituir tal efecto sedante por el que también genera la ingesta. La fisiología de la mujer podría ser más vulnerable a dicha situación.
La genética clínica puede ser esencial para establecer las pautas y los protocolos derivados del consejo genético a las familias con casos de alto riesgo y agregación familiar de obesidad manifiesta. Teniendo en cuenta el coste global de la enfermedad, la baja proporción de curaciones y los decepcionantes resultados de las estrategias desarrolladas en el ámbito de la prevención por los sistemas sanitarios, proponemos el diseño de nuestro estudio como un abordaje por tener en cuenta para el control y el manejo multidisciplinario y especializado de la enfermedad. Los análisis de perfiles genéticos de riesgo aplicados a la población general ofrecerían la posibilidad de identificar un porcentaje importante de personas sobre las que ejercer las máximas medidas de prevención primaria, única estrategia de futuro en el abordaje de la enfermedad. El diagnóstico a través de estudios genéticos que se apoyen en las estrategias nutricionales y de tratamientos conductuales tradicionales permitiría, además, individualizar la práctica clínica en cada caso24.
A pesar del desarrollo biotecnológico y la accesibilidad a análisis genéticos masivos, así como las herramientas bioinfomáticas cada vez más capaces y complejas, la búsqueda de variantes genéticas y, por tanto, de perfiles genéticos de susceptibilidad no parte de una correcta selección de casos. No se tiene en cuenta la edad ni la gravedad de la aparición del sobrepeso, ni los antecedentes familiares, hechos traducidos en la evidente heredabilidad de la enfermedad que ya se acepta superior a la de procesos como los neoplásicos.
Sugerimos la necesidad de un mayor aprovechamiento de los resultados significativos y corroborados de variantes genéticas de riesgo, los cuales se han estimado en un 2% del total publicado en las revistas de mayor impacto. Proponemos el seguimiento de los individuos portadores de los haplotipos de riesgo identificado en FTO, tratando de integrar las conclusiones obtenidas en una estrategia de abordaje multidisciplinario de la enfermedad.
FinanciaciónEste trabajo ha sido financiado por la Junta de Extremadura (PRI08OB007) y por el Instituto de Salud Carlos III (07PI0594).
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.