Estimar la correlación entre los índices de calidad dietética (DQI), de sensibilidad (QUICKI) y de resistencia a la insulina (HOMA-IR), el índice cintura-cadera (ICC) y el índice de masa corporal (IMC) con los alelos y genotipos del SNP rs2291166 de TJP1 y el VNTR de ATXN2 en pacientes adolescentes.
Sujetos y métodoEl estudio se incluyó 85 sujetos con rango de edad entre 10-20años, originarios de la ciudad de Tuxtepec, Oaxaca, México, captados en el periodo 2017-2018. Se determinó el DQI, el IMC, el ICC, el índice HOMA-IR, el QUICKI y el índice de calidad dietética. El polimorfismo rs2291166 de TJP1 se determinó por PCR-alelo específico y la expansión (CAG)n de ATXN2 se determinó por PCR-Hot Start. Los productos de PCR se analizaron por electroforesis PAGE al 8% y tinción con nitrato de plata.
ResultadosSe encontró la correlación entre los índices DQI, HOMA-IR, ICC e IMC con el genotipo heterocigoto del SNP rs2291166 de TJP1 y los repetidos largos así como cortos del repetido CAG de ATXN2 en pacientes adolescentes con obesidad, con una correlación positiva muy fuerte entre el SNP de TJP1 y el índice HOMA-IR (p<0,05).También se encontró correlación positiva entre el repetido CAG de ATXN2 con el índice QUICKI (p=0,000) (p<0,05), mientras que el DQI correlaciona más con el IMC y el ICC.
ConclusionesEl DQI, el SNP rs2291166 de TJP1 y el repetido CAG de ATXN2 son factores determinantes de los parámetros de riesgo relacionados con la obesidad tales como el IMC, el ICC, el QUICKI y el HOMA-IR en la población adolescente analizada.
To estimate the correlation between indices of diet quality (DQIs), insulin sensitivity (QUICKI) and resistance (HOMA-IR), waist circumference (WHR) and body mass (BMI) and the alleles and genotypes of the TJP1 SNP rs2291166 and the VNTR of ATXN2 in adolescent patients.
Subjects and methodThe study enrolled 85 subjects aged 10-20years, from the city of Tuxtepec, Oaxaca, Mexico, recruited in the period 2017-2018. DQIs, BMI, WHR, HOMA-IR, QUICKI, and diet quality index were measured. The rs2291166 polymorphism in TJP1 was determined by allele-specific PCR and the (CAG)n expansion in ATXN2 was determined by hot start PCR. PCR products were analyzed using 8% PAGE electrophoresis and silver nitrate staining.
ResultsA correlation was found of indices DQIs, HOMA-IR, WHR and BMI with the heterozygous genotype of the TJP1 SNP rs2291166 and the long and short repeats of the ATXN2 CAG repeat in obese adolescent patients. A very strong positive correlation was seen between the TJP1 SNP and the HOMA-IR index (P<.05). A positive correlation was also found between the ATXN2 CAG repeat and the QUICKI index (P=.000) (P<.05), while the DQIs index correlated more closely with BMI and WHR.
ConclusionsDQIs, TJP1 SNP rs2291166, and ATXN2 CAG repeat are determinants of obesity-related risk parameters such as BMI, WHR, QUICKI, and HOMA-IR in the adolescent population analyzed.
En la actualidad, el aumento en la prevalencia de obesidad se ha convertido en un problema de salud pública a nivel mundial. La obesidad se define como la acumulación anormal o excesiva de tejido adiposo en el organismo, que cuando se concentra en la parte troncal del cuerpo durante la adolescencia se asocia de manera positiva e independiente con múltiples enfermedades metabólicas crónicas en la edad adulta1. En los menores de 19años puede diagnosticarse cuando su índice de masa corporal (IMC) se encuentra por encima del percentil 95, según las tablas por edad y género de la Organización Mundial de la Salud (OMS)2. En México, la prevalencia de obesidad en adolescentes se ha incrementado en 1,4 puntos porcentuales de 2012 a 2016, alcanzando un 36,3% de prevalencia combinada de sobrepeso/obesidad3.
Existen diversos estudios que confirman que la etiología de la obesidad es multifactorial, determinada por factores ambientales y genéticos, con lo que su abordaje también debe encaminarse en este sentido. Dentro de las causas ambientales están los hábitos alimentarios, el sedentarismo, los factores económicos, el ambiente obesogénico, los factores sociales, la genética, la microbiota y/o las alteraciones endocrinas. La genética ha sido la menos estudiada en México4, con lo que es de suma importancia realizar investigaciones que diseccionen los factores genético-ambientales, su interacción, su resultado en forma de fenotipo clínico y sus comorbilidades asociadas. En este tópico, una de las vertientes más estudiadas es la caracterización de la obesidad monogénica, ocasionada por mutaciones en un solo gen; en este rubro se encuentran los genes que codifican proteínas encargadas de la regulación del apetito, tal es el caso de la leptina (LEP), el receptor de leptina (LEPR), la pro-opiomelanocortina (POMC), la pro-convertasa tipo1 (PC1) y el receptor tipo4 de melanocortina (MC4R)5,6. En un estudio reciente realizado en la región central de México (3.938 adultos y 636 escolares) se buscó una influencia de la interacción entre los polimorfismos rs9939609 de FTO y rs9282541 de ABCA1 (miembro1 de la subfamiliaA de los casetes de las ATPasa) sobre los valores del IMC y del índice cintura-cadera (ICC), hallando una asociación significativa entre la variante de FTO en estado heterocigoto con un mayor IMC (p=0,043) y ligeramente con un mayor ICC (p=0,067)7. Sin embargo, hacen falta más estudios de este tipo debido a la diversidad étnica que caracteriza la población mexicana, la cual se relaciona directamente con las diferencias metabólicas en los individuos.
En la actualidad existen dos genes cuya relación con el estado nutricional y la obesidad ha sido poco estudiada.
El primer gen es el que codifica para la proteína de unión estrecha tipo1 (Tight Junction Protein1 [TJP1]), que se localiza en el cromosoma15 y es el encargado de codificar para las proteínas de la zona occludens tipo1 (Zonula Occludens type1 [ZO-1]) que forman parte de las uniones estrechas, regulando el transporte lateral y basolateral en las membranas de nutrientes incluyendo las de las células epiteliales intestinales8.
El segundo gen es el que codifica para la ataxina-2 (Spinocerebellar Ataxia Type2 Protein [ATXN2]), tiene su locus en el cromosoma12, banda q24.1, y codifica para la ataxina-2, una proteína de unión al ARN que tiene interacción con la proteína de unión citoplasmática poly[A] (PABPC1) y que pertenece a un grupo de genes que está asociado con las enfermedades de expansión de microsatélites (trastornos neurológicos y neuromusculares causados por la expansión de tramos cortos de ADN repetitivo)8-10. La ataxina-2 también es un regulador del estrés celular en el aparato de Golgi, de la vía de señalización de la insulina (vía GRB2), regula la expresión de enzimas del ciclo de Krebs, de la oxidación de los ácidos grasos, de transportadores en la membrana celular para ácidos grasos, aminoácidos, minerales, entre otros nutrientes, por lo cual influencia el estado nutricional, y cuando está deficiente se produce obesidad y resistencia a la insulina10.
Así, la presente investigación tiene por objetivo estimar la correlación entre los índices de calidad dietética (DQI), de sensibilidad (QUICKI) y de resistencia a la insulina (HOMA-IR), el ICC y el IMC con los alelos y genotipos del SNP rs2291166 de TJP1 y el VNTR de ATXN2 en pacientes adolescentes
Material y métodosSujetos de estudioEl presente trabajo es un estudio transversal analítico. En él se incluyeron 85 pacientes adolescentes (170 cromosomas) de entre 10 y 20años de la ciudad de Tuxtepec, Oaxaca, México, de origen étnico Chinanteca, que representan todos los usuarios de la jurisdicción número3 de los Servicios de Salud del Estado de Oaxaca, Sistema de Información en Crónicas (SIC), del Centro Nacional de Programas Preventivos y/o del Control de Enfermedades (CENAPRECE). Los sujetos incluidos o sus tutores no tenían conocimiento de padecer enfermedades o rasgos asociados al síndrome metabólico, presentaban presión arterial <120/85mmHg, niveles séricos de colesterol total <180mg/dl, triglicéridos totales <170mg/dl, colesterol HDL entre 35-45mg/dl, colesterol LDL <140mg/dl, hemoglobina glucosilada A1C <6,0%. Se establecieron como variables dependientes: IMC, CC, HOMA-IR, QUICKI, y como variables independientes: polimorfismos rs2291166 de TJP1 así como (CAG)n de ATXN2 y DQI.
Aspectos de éticaEl estudio se llevó a cabo en consonancia con el código ético de la OMS y la Declaración de Helsinki revisada. Como primer requisito de inclusión en el proyecto, cada uno de los participantes (padres o tutores en el caso de los menores de edad) firmó un consentimiento informado legal de participación. Todos los datos se manejaron de manera anónima, respetando la privacidad de cada uno de los integrantes. El estudio fue aprobado por los comités de ética e investigación de la Jurisdicción Sanitaria No.3 de Tuxtepec, Servicios de Salud del Estado de Oaxaca, con folio de autorización J3/EP/106/2019.
Análisis antropométricoPara la estimación del peso se utilizó una báscula (TANITA modelo UM-081, Zapopan, Jalisco, México), para la talla se utilizó un estadímetro (SECA modelo 206, Hamburgo, Alemania) y la circunferencia de cintura se midió con una cinta de fibra de vidrio (SECA modelo 201, Hamburgo, Alemania). Todas las medidas anteriores se tomaron mediante las pautas establecidas por la Secretaría de Salud de México11. Se calculó el IMC a partir de la fórmula descrita por Quetelet en 1833, y se analizó mediante las curvas así como por las tablas de IMC para la edad de la OMS 20072.
Análisis de la presión arterialLa medición de la presión arterial se realizó con baumanómetro mercurial de caja MI-300 Marca MEDDEX FAB NACIONAL Modelo 030, fabricado bajo los estándares de la Norma Oficial Mexicana 009 SFCI1993 y certificado por la American Heart Association (AHA).
Análisis bioquímicoA todos los sujetos del estudio se les tomaron 5ml de sangre periférica por punción venosa en tubos BD Vacutainer para las pruebas bioquímicas. Las determinaciones de glucosa se realizaron mediante kits enzimáticos-colorimétricos de Human Co. por espectrofotometría en el equipo BeckamCoulter DU730, Brea, CA, EE.UU.; la insulina sérica se realizó por RIA mediante el Kit 80-INSHU-E01.1 de Alpco (Salem, NH, EE.UU.) mediante el lector Chromate Microplate Reader de Awareness-Technology, Inc. (Palm City, FL, EE.UU.). Posteriormente, los índices de resistencia y sensibilidad a la insulina fueron determinados mediante las fórmulas de HOMA-IR y QUICKI. Los índices de resistencia a la insulina fueron determinados mediante las siguientes fórmulas: HOMA-IR=(insulina en ayuno (μU/ml)*glucosa en ayuno [mmol/l])/22,5; índice QUICKI=1/(log insulina plasmática [uU/ml] + log glucosa plasmática en ayuno 6 [mg/dl])12.
Análisis del perfil de lípidosLos niveles de colesterol y triglicéridos totales, colesterol HDL (lipoproteína de alta densidad) y colesterol LDL (lipoproteína de baja densidad) se determinaron por fotometría directa mediante el equipo automatizado (ES-3000, Guadalajara, Jalisco, México, Laboratorios Tolsa).
Análisis de hemoglobina glucosiladaLos niveles de la hemoglobina glucosilada A1C se analizaron mediante inmunofluorescencia con el equipo CHROMA II, Bodea, Corea (Laboratorios Tolsa de Guadalajara, Jalisco, México).
Análisis genético molecularA partir de 5ml de sangre periférica por punción venosa se extrajo ADN genómico por el método de perlas magnéticas del kit Genecatcher (Invitrogene) para la reacción de amplificación13-14.
Amplificación PCR-Hot Start para el VNTR (CAG)n de ATXN2Se utilizaron los iniciadores ATXN2-1 5́-GGGCCCCTCACCATGTCG-3́, ATXN2-2 5’-CGGGCTTGCGGACATTGG-3’, los cuales fueron sintetizados por la compañía Sigma Aldrich™, previamente diseñados y probados9,15. El programa de amplificación que se realizó en el Termociclador 2720 Thermal Cycler, Applied Biosystem (Foster City, California, EE.UU.) fue el siguiente: 1, 96°C 3min; 2, 96°C 60s; 3, 59°C 30s; 4, 72°C 1min; 5, 28veces del ciclo 2 al 4. Mezcla de reacción: Buffer10X 2μl/por reacción, iniciadores ATXN2-1 y ATXN2-2 4pmol/μl/por reacción, dNTP 10mM 2μl/por reacción, MgCl2 2mM 2μl/por reacción, DMSO al 8% 2μl/por reacción, Taq DNA Polymerase Recombinan (Invitrogene, Sao Paulo, Brazil) 5U/μl, ADN 100ng/μl. Volumen final de la reacción: 25μl. El producto de PCR se mezcló con formamida desionizada y se desnaturalizó en baño maría durante 10min, posteriormente se colocó en hielo frapé durante 3min para su corrimiento electroforético. Los productos amplificados fueron sometidos a electroforesis en geles de poliacrilamida al 8% (19:1) 180V durante 2h. Posteriormente, los geles fueron teñidos con nitrato de plata 0,1g, 0,5ml de ácido acético y 10ml de etanol aforados a 100ml. La solución reveladora usada fue a base de hidróxido de sodio al 3% con 270μl de formaldehído al 37% aforado a 100ml9,15. La identificación de las repeticiones se realizó con base en la clasificación de los productos de PCR, 106pb corresponde al alelo de 14 repeticiones, 22 repeticiones a 130pb, y que corresponde al alelo de mayor frecuencia; 154pb para el alelo de 30 repeticiones16.
Amplificación PCR-PASA para el SNP rs2291166del gen TJP1La detección del polimorfismo rs2291166 se realizó mediante los iniciadores: FW1G 5́-CTTCATCTTCTTCAGGTT-3́, FW2A 5́-ATATTCTTCATCTTCTTC AGG TǴ-3, RW3 5́-GTCATTCATTATCTGTTAGG-3́ (Genosys Sigma-Aldrich) previamente reportados17. El programa de amplificación consistió en desnaturalización inicial a 95°C durante 5min, seguido de 30ciclos con las siguientes características: 1, 30s de desnaturalización a 95°C; 2, hibridación durante 45s a 48°C, y 3, polimerización por 30s a 72°C, con una extensión final a 72°C durante 5min, de acuerdo a lo previamente reportado17. La mezcla de reacción fue: Buffer KCl 2,5μl (1X), MgCl2 1,5μl (25mM), 0,5μl de dNTP (0,2mM), 0,5μl de cada iniciador (25pmoles), 2μl de templado de ADN (200ng), Taq DNA Polymerase Recombinant 0,3μl (3U/μl) (Invitrogen, Sao Paulo, Brazil), y finalmente 17,20 μl de agua, reacción final 25ml. Los productos de PCR se analizaron mediante electroforesis en poliacrilamida en la proporción 19:1 al 7%17 con corrimiento electroforético buffer TBE1X por 1,5h a 200volts, 80-84mA. Para revelar los geles se usó nitrato de plata. Los productos se diferenciaron por los tamaños: el de 102pb corresponde al aleloT, mientras que el de 107pb corresponde al alelo G17. Las reacciones de PCR de los genes TJP1 y ATXN2, así como las electroforesis, fueron realizadas e interpretadas por triple ciego.
Análisis dietéticoPara evaluar la ingesta nutricional se aplicaron dos recordatorios de 24h no consecutivos a cada uno de los sujetos de estudio; posteriormente se determinó el promedio individual de ingesta y la ingesta en gramos de cada uno de los nutrimentos ingeridos.
A partir de las 8 recomendaciones de ingesta saludable de nutrimentos emitidas por el Comité de Expertos FAO/OMS para la prevención de enfermedades crónicas18 se construyó un índice de calidad de la dieta, o DQI, de 8ítems. Tomando como referencia las recomendaciones de la FAO/OMS, se determinó el porcentaje de los nutrimentos ingeridos.
Los componentes positivos del índice fueron: consumo diario de ≥400g de frutas y hortalizas; ingesta diaria de proteína ≥10% de la energía total; ingesta diaria de grasa total <30% de la energía total; ingesta diaria de grasa saturada <10% de la energía total; ingesta diaria de grasa poliinsaturada 6-10% de la energía total; ingesta diaria de colesterol <300mg de la ingesta diaria total, y ≥25g de fibra dietética total al día18.
A partir de estos ítems se asignó un valor de1 al cumplimiento de cada uno y un valor de0 al incumplimiento, con lo que el valor máximo para el índice fue de 8 y los valores observados oscilarían entre 2 y 8. Para su interpretación se utilizaron tertiles de índice en los análisis (2-4, 5-6 y 7-8), lo que correspondió a una puntuación preventiva dietética baja, media y alta, respectivamente18.
Análisis estadísticoEl tamaño de la muestra fue por conveniencia. La mínima calculada era de 100 cromosomas (50 personas) por las proporciones de los alelos analizados acorde a los criterios de Neale; se incluyeron 85 pacientes (170 cromosomas), que representan todos los usuarios adolescentes de la jurisdicción sanitaria No.3 del programa SIC-CENAPRECE.
Las frecuencias alélicas y genotípicos de los dos polimorfismos se establecieron por conteo directo y se aplicaron a través de la razón como medida de frecuencia. Las pruebas para comparar variables no paramétricas, como en el caso del equilibrio Hardy-Weinberg o la correlación con los biomarcadores y los polimorfismos (CAG)n en ATXN2 y rs2291166 de TJP1, fueron la χ2 de Pearson, el cociente de Spearman y un análisis de regresión logística múltiple binominal. Las diferencias entre los parámetros antropométricos clínicos y bioquímicos se validaron mediante la t de Student (muestras independientes), considerando un resultado estadísticamente significativo a valores p<0,05; χ2≥6,99. Posteriormente se procedió a la realización de un ANOVA y la prueba de Dunnet entre los grupos con las variables antropométricas, bioquímicas y los genotipos significativos para la prueba t de Student. Para la realización de estos procedimientos estadísticos se utilizó el apoyo del programa estadístico SPSS versión 25.0 (Chicago, IL, EE.UU.).
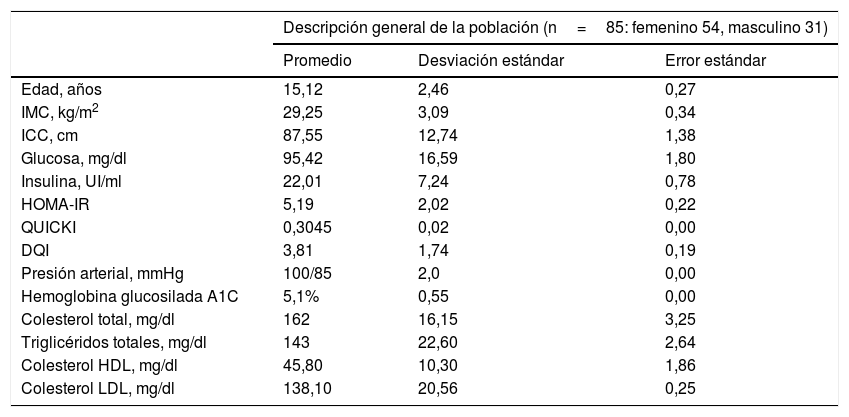
ResultadosEn la tabla 1 se muestra una descripción comparativa del promedio, la desviación estándar y el error estándar de las características antropométricas, bioquímicas y dietéticas de la población estudiada, siendo relevante que la población adolescente estudiada muestra niveles de glucosa dentro del rango normal y presenta hiperinsulinemia, resistencia a la insulina así como alteración en la sensibilidad de la insulina; sin embargo, ninguno era diabético al momento de ser incluido en el estudio, lo que se refleja en los valores de hemoglobina glucosilada A1C (HbA1C) menores del 6,5% (tabla 1). Además, ninguno de los casos índice presentó hipertensión arterial o alguna dislipidemia, ya que mostraron valores de colesterol total, triglicéridos totales, colesterol LDL y colesterol HDL dentro de los rangos normales (tabla 1).
Descripción general de la población de estudio
| Descripción general de la población (n=85: femenino 54, masculino 31) | |||
|---|---|---|---|
| Promedio | Desviación estándar | Error estándar | |
| Edad, años | 15,12 | 2,46 | 0,27 |
| IMC, kg/m2 | 29,25 | 3,09 | 0,34 |
| ICC, cm | 87,55 | 12,74 | 1,38 |
| Glucosa, mg/dl | 95,42 | 16,59 | 1,80 |
| Insulina, UI/ml | 22,01 | 7,24 | 0,78 |
| HOMA-IR | 5,19 | 2,02 | 0,22 |
| QUICKI | 0,3045 | 0,02 | 0,00 |
| DQI | 3,81 | 1,74 | 0,19 |
| Presión arterial, mmHg | 100/85 | 2,0 | 0,00 |
| Hemoglobina glucosilada A1C | 5,1% | 0,55 | 0,00 |
| Colesterol total, mg/dl | 162 | 16,15 | 3,25 |
| Triglicéridos totales, mg/dl | 143 | 22,60 | 2,64 |
| Colesterol HDL, mg/dl | 45,80 | 10,30 | 1,86 |
| Colesterol LDL, mg/dl | 138,10 | 20,56 | 0,25 |
DQI: índice de calidad dietética; HDL: lipoproteína de alta densidad; HOMA-IR: índice de resistencia a la insulina, IMC: índice de masa corporal; ICC: índice cintura-cadera; LDL: lipoproteína de baja densidad; QUICKI: índice de sensibilidad a la insulina.
No se encontraron diferencias significativas por género y edad para el IMC, el ICC, los niveles de glucosa, de insulina, el HOMA-IR, el QUICKI, el DQI, la HbA1C, la presión arterial, el colesterol total, los triglicéridos totales y el colesterol HDL y LDL (p>0,05).
Valoración nutricionalEn la muestra total (n=85), 21 masculinos se clasificaron con sobrepeso (24,7%) y 10 con obesidad (40%); del género femenino, 34 se clasificaron con sobrepeso (11,8%) y 20 con obesidad (23,5%).
Frecuencias alélicas y genotípicas del polimorfismo rs2291166 del gen TJP1 de la poblaciónEn cuanto a la determinación de las frecuencias alélicas del polimorfismo rs2291166 del gen TJP1 en la población estudiada, se observó mayor presencia del aleloT (88,8%; n=75) que del aleloG (11,2%; n=10). Con respecto a la frecuencia genotípica del polimorfismo rs2291166 del gen TJP1, se encontró mayor presencia del genotipoT/T (80%; n=68) que del genotipoT/G (17,6%; n=15) y del genotipo G/G (2,4%; n=2).
Equilibrio HW para el polimorfismo rs2291166 del gen TJP1En el análisis del equilibrio de HW se compararon las frecuencias obtenidas para cada genotipo del polimorfismo rs2291166 del gen TJP1 (T/T=68; T/G=15; G/G=2) con las frecuencias esperadas (T/T=67,3; T/G=16,7; G/G=1) y se validaron utilizando la χ2 con un valor de 5,99 y una p=0,55, pudiendo describir que la población se encuentra en equilibrio HW.
Frecuencias alélicas y genotípicas así como la prueba de HW para el VNTR de ATXN2En el caso de la frecuencia alélica de alelos del VNTR (CAG)n del gen para la ataxina-2, se obtuvo que el alelo más frecuente en la población analizada fue el de 22 repetidos con un 87%, el segundo más frecuente fue el de 23 repetidos con 5,88%, 18 y 29 repetidos con 2,35% para ambos y por último 20 y 24 repetidos con 1,18% para ambos. Los alelos 18, 20, 22, 23, 24 y 29 son polimórficos en la población estudiada debido a que sus frecuencias son superiores al 1%.
En la muestra total (n=85) se encontraron seis genotipos homocigotos para el alelo 22 repeticiones y los heterocigotos para 18/22, 20/22, 22/24 y 22/29. El genotipo observado con mayor frecuencia fue el homocigoto para el alelo de 22 repetidos (87%), seguido del genotipo 22/23 (5,88%), luego el 18/22 y 22/29 (ambos con 2,35%) y por último los genotipos 20/22 y 22/24, con 1,17% cada uno. La distribución de estos genotipos se encontró en equilibrio HW.
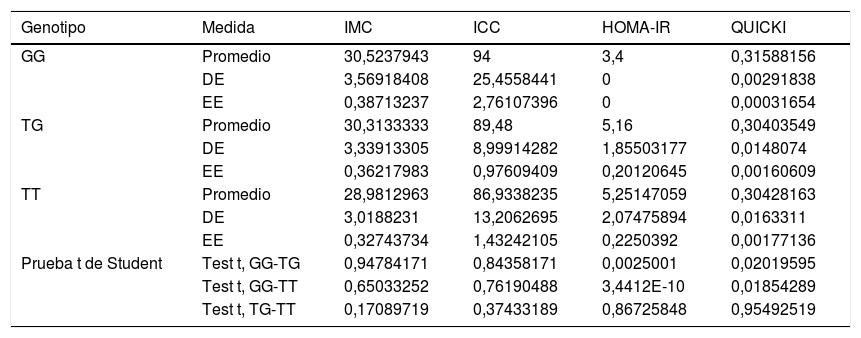
Asociación entre marcadores de riesgo nutricional con los genotipos del SNP rs2291166 de TJP1 y del VNTR de ATXN2Al comparar la media del IMC, del ICC, del HOMA-IR y del QUICKI con los genotipos se encontraron diferencias significativas con la prueba t de Student: en los homocigotosG (G/G) contra los heterocigotos son más bajas la resistencia a la insulina y el deterioro de la sensibilidad a la insulina. Al comparar los homocigotosT (T/T) contra los homocigotosG (G/G), los homocigotosT presentan valores más altos para la resistencia a la insulina y más deterioro de la sensibilidad a la insulina. Por otra parte, no se encontraron diferencias significativas con el IMC y el ICC en relación con la distribución de medias por genotipos (tabla 2). El análisis multivariado (ANOVA) del polimorfismo rs2291166 de TJP1 muestra diferencias significativas inter e intragrupos (p=0,0001), particularmente mediante la prueba de Dunnet; se encontró significativo el estado heterocigoto T/C comparado con los homocigotosG (G/G) (p=0,031) respecto al índice HOMA-IR. Las medias del IMC, del ICC y del QUICKI no presentaban diferencias significativas en el ANOVA, así como los valores de la presión arterial, colesterol total, triglicéridos totales y colesterol HDL y LDL en relación con los genotipos del SNP de TJP1 (p=0,10, 0,33, 0,44, 0,98 y 0,73, respectivamente).
Nivel de significancia con la prueba t de Student para variables dependientes con genotipos del gen TJP1
| Genotipo | Medida | IMC | ICC | HOMA-IR | QUICKI |
|---|---|---|---|---|---|
| GG | Promedio | 30,5237943 | 94 | 3,4 | 0,31588156 |
| DE | 3,56918408 | 25,4558441 | 0 | 0,00291838 | |
| EE | 0,38713237 | 2,76107396 | 0 | 0,00031654 | |
| TG | Promedio | 30,3133333 | 89,48 | 5,16 | 0,30403549 |
| DE | 3,33913305 | 8,99914282 | 1,85503177 | 0,0148074 | |
| EE | 0,36217983 | 0,97609409 | 0,20120645 | 0,00160609 | |
| TT | Promedio | 28,9812963 | 86,9338235 | 5,25147059 | 0,30428163 |
| DE | 3,0188231 | 13,2062695 | 2,07475894 | 0,0163311 | |
| EE | 0,32743734 | 1,43242105 | 0,2250392 | 0,00177136 | |
| Prueba t de Student | Test t, GG-TG | 0,94784171 | 0,84358171 | 0,0025001 | 0,02019595 |
| Test t, GG-TT | 0,65033252 | 0,76190488 | 3,4412E-10 | 0,01854289 | |
| Test t, TG-TT | 0,17089719 | 0,37433189 | 0,86725848 | 0,95492519 |
DE: desviación estándar; EE: error estándar; HOMA-IR: índice de resistencia a la insulina; ICC: índice cintura-cadera; IMC: índice de masa corporal; QUICKI: índice de sensibilidad a la insulina.
Fuente: elaboración propia.
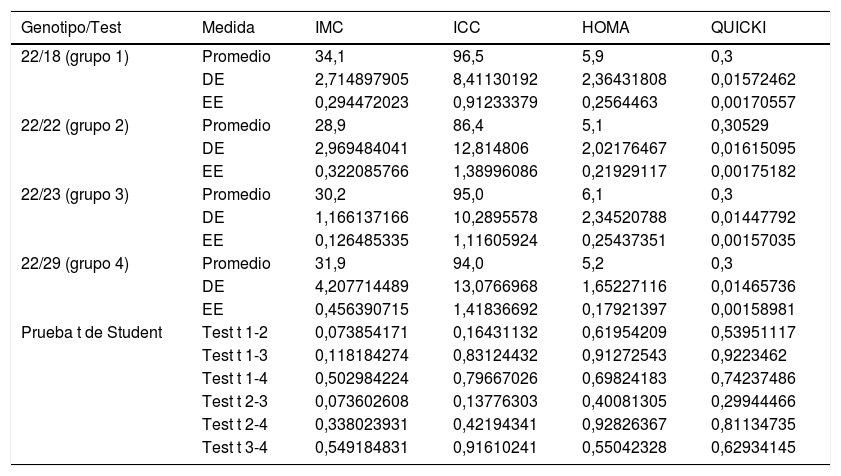
Por otra parte, al comparar la media del IMC, del ICC, del HOMA-IR y del QUICKI con los cuatro genotipos del VNTR de ATXN2 no se encontraron diferencias significativas con la prueba t referente a los valores del ICC, del HOMA-IR y del QUICKI; sin embargo, existe una relación directa e inversa entre el incremento o la disminución de la longitud de la expansión del repetido CAG con los valores altos del IMC (portadores del genotipo 18/22 y 22/23), al compararlos con los valores de los portadores del genotipo más común, el homocigoto de 22 repeticiones (tabla 3). Las medias no presentaban diferencias significativas para la prueba t y ANOVA con los valores de la presión arterial, colesterol total, triglicéridos totales, colesterol HDL y LDL en relación con los genotipos del VNTR (CAG)n del gen ATXN2 (p=0,21, 0,64, 0,56, 0,71 y 0,35, respectivamente).
Nivel de significancia con la prueba t de Student para variables dependientes con genotipos del gen ATXN2
| Genotipo/Test | Medida | IMC | ICC | HOMA | QUICKI |
|---|---|---|---|---|---|
| 22/18 (grupo 1) | Promedio | 34,1 | 96,5 | 5,9 | 0,3 |
| DE | 2,714897905 | 8,41130192 | 2,36431808 | 0,01572462 | |
| EE | 0,294472023 | 0,91233379 | 0,2564463 | 0,00170557 | |
| 22/22 (grupo 2) | Promedio | 28,9 | 86,4 | 5,1 | 0,30529 |
| DE | 2,969484041 | 12,814806 | 2,02176467 | 0,01615095 | |
| EE | 0,322085766 | 1,38996086 | 0,21929117 | 0,00175182 | |
| 22/23 (grupo 3) | Promedio | 30,2 | 95,0 | 6,1 | 0,3 |
| DE | 1,166137166 | 10,2895578 | 2,34520788 | 0,01447792 | |
| EE | 0,126485335 | 1,11605924 | 0,25437351 | 0,00157035 | |
| 22/29 (grupo 4) | Promedio | 31,9 | 94,0 | 5,2 | 0,3 |
| DE | 4,207714489 | 13,0766968 | 1,65227116 | 0,01465736 | |
| EE | 0,456390715 | 1,41836692 | 0,17921397 | 0,00158981 | |
| Prueba t de Student | Test t 1-2 | 0,073854171 | 0,16431132 | 0,61954209 | 0,53951117 |
| Test t 1-3 | 0,118184274 | 0,83124432 | 0,91272543 | 0,9223462 | |
| Test t 1-4 | 0,502984224 | 0,79667026 | 0,69824183 | 0,74237486 | |
| Test t 2-3 | 0,073602608 | 0,13776303 | 0,40081305 | 0,29944466 | |
| Test t 2-4 | 0,338023931 | 0,42194341 | 0,92826367 | 0,81134735 | |
| Test t 3-4 | 0,549184831 | 0,91610241 | 0,55042328 | 0,62934145 |
DE: desviación estándar; EE: error estándar; HOMA-IR: índice de resistencia a la insulina; ICC: índice cintura-cadera; IMC: índice de masa corporal; QUICKI: índice de sensibilidad a la insulina.
Fuente: elaboración propia.
El análisis multivariado (ANOVA) para los genotipos de ATXN2 no muestra diferencias significativas inter e intragrupo para los valores del ICC, del HOMA-IR y del QUICKI (p=0,5671, 0,3621 y 0,4438, respectivamente). Para el IMC mostró un valor de p=0,0507 entre los grupos 1, 2 y 3. La prueba de Dunnett mostró significativo el incremento del IMC en los portadores que no son homocigotos del genotipo más frecuente (el 22/22) (p=0,01).
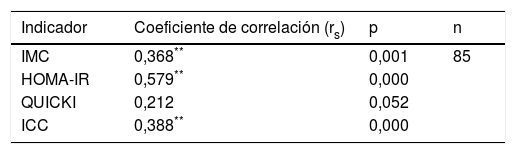
Correlación entre polimorfismo rs2291166 del gen TJP1 con IMC, HOMA-IR, ICC y QUICKIEl grado de significancia de correlación de Spearman mostró una correlación positiva estadísticamente significativa entre el estado heterocigoto del polimorfismo rs2291166 del gen TJP1 y el IMC (p=0,001; significación media), también para el indicador HOMA-IR (p=0,000; significación considerable) y el indicador ICC (p=0,000; significación media), mientras que con el indicador de la sensibilidad a la insulina QUICKI no se halló correlación (p=0,052), concluyendo que el IMC, el HOMA-IR y el ICC podrían estar correlacionados al polimorfismo rs2291166 del gen TJP1, pero no en el caso de QUICKI (tabla 4).
Correlación de Spearman para el polimorfismo del gen TJP1
| Indicador | Coeficiente de correlación (rs) | p | n |
|---|---|---|---|
| IMC | 0,368** | 0,001 | 85 |
| HOMA-IR | 0,579** | 0,000 | |
| QUICKI | 0,212 | 0,052 | |
| ICC | 0,388** | 0,000 |
HOMA-IR: índice de resistencia a la insulina; ICC: índice cintura-cadera; IMC: índice de masa corporal; QUICKI: índice de sensibilidad a la insulina.
El grado de significancia de correlación de Spearman entre el repetido (CAG)n del gen ATXN2 y las variables de riesgo nutricional mostraron una correlación positiva media con el IMC (rs=0,435; p=0,000), con el HOMA-IR (rs=0,419; p<0,01) y con el ICC (rs=0,421; p<0,01), y una correlación positiva considerable con el índice QUICKI (rs=0,675; p<0,01) (tabla 5).
Correlación de Spearman para el polimorfismo del repetido (CAG)n del gen ATXN2 (n=85)
| Indicador | Coeficiente de correlación (rs) | p |
|---|---|---|
| IMC | 0,435** | 0,000 |
| HOMA-IR | 0,419** | 0,000 |
| QUICKI | 0,675** | 0,000 |
| ICC | 0,421** | 0,000 |
HOMA-IR: índice de resistencia a la insulina; ICC: índice cintura-cadera; IMC: índice de masa corporal; QUICKI: índice de sensibilidad a la insulina.
El grado de significancia de correlación de Spearman para el caso del DQI con los indicadores asociados a obesidad mostró asociación positiva perfecta para el IMC (rs=0,914), una correlación positiva media para el HOMA-IR (rs=0,186), una correlación positiva considerable para el QUICKI (rs=0,673) y una correlación positiva muy fuerte para el ICC (rs=0,868) (tabla 6).
Correlación de Spearman para el índice de calidad dietética, IMC, HOMA-IR, ICC y QUICKI (n=85)
| Indicador | Coeficiente de correlación (rs) | p |
|---|---|---|
| IMC | 0,914** | 0,000 |
| HOMA-IR | 0,186** | 0,000 |
| QUICKI | 0,673** | 0,000 |
| ICC | 0,868** | 0,000 |
HOMA-IR: índice de resistencia a la insulina; ICC: índice cintura-cadera; IMC: índice de masa corporal; QUICKI: índice de sensibilidad a la insulina.
El presente trabajo es el primer estudio que muestra la interactómica entre el rs2291166 del gen TJP1 y el VNTR (CAG)n del ATXN2, así como el DQI, con los parámetros de riesgo relacionados con la obesidad en población adolescentes, como lo son los índices IMC, ICC y HOMA-IR. El SNP rs2291166 de TJP1 es el que tiene mayor efecto en la resistencia a la insulina, lo cual se ve reflejado en los altos valores del índice HOMA-IR, mientras que el VNTR (CAG)n de ATXN2 tiene un mayor efecto en la sensibilidad a la insulina, lo cual se ve reflejado en los valores del índice QUICKI, mientras el DQI tiene mayor influencia en los índices relacionados directamente con la obesidad, como lo son el IMC y el ICC. Los efectos patogénico y fisiológico del SNP rs2291166 TJP1 ya se han demostrado previamente17,19, lo cual se debe a que conduce un cambio conformacional de ZO-1, debido a la sustitución de alanina por aspartato en el residuo 1334 de ZO-1, el cual se traduce en una modificación de la permeabilidad celular apico-lateral o basal-lateral, que puede afectar el flujo de nutrientes a los intestinos y en los tejidos, así como a los depósitos en el tejido adiposo20.
Por otra parte, el gen ATXN2 es un sensor de estrés celular y nutricional, es regulador de la sensibilidad a la insulina en los tejidos y sensor de nutrientes a través TORC-1 y de la activación de genes transportadores de nutrientes para diferentes moléculas en las membranas celulares, como SLC5A6, SLC16A1, BTD, SGLT4, a través de la vía GRB2 que regula al sustrato del receptor de insulina (SUR-1), lo cual se relaciona con el desarrollo de diabetes mellitus tipo2 y con obesidad, actividad vinculada con la actividad transcripcional de alelos largos y cortos del repetido (CAG)n10. A nivel mundial existen muy pocos estudios en humanos sobre el rol de ATXN2 en el metabolismo de hidratos de carbono y lípidos; sin embargo, Lv et al.21 y Meierhofer et al.22 coinciden en que las mutaciones que involucren pérdida de funciones o desactivación del ATXN2 (que se relacionan con mayores o menores repeticiones CAG) pueden provocar susceptibilidad a padecer obesidad, resistencia a la insulina y dislipidemias. Por otro lado, Figueroa et al.23, Ma et al.24 y Gardiner et al.25 coinciden con el presente estudio en que mientras más repeticiones del polimorfismo CAG de ATXN2 existan, habrá un mayor riesgo genético de adiposidad y obesidad. Esto, sumado a la alta frecuencia del alelo ancestral del SNP rs2291166 de TJP1, el cual es muy frecuente en la población mexicana10, incrementa más dicho riesgo26-27.
Recientemente la metagenómica ha tomado auge para entender el papel que juegan en las enfermedades metabólicas la microbiota comensal; en el caso de la obesidad es una frontera de la investigación entre las variantes genéticas y ña expresión genética con los factores genéticos del huésped y el medio ambiente en el que están inmersos ambos. En este sentido se ha reportado que los pacientes obesos tienen una microbiota intestinal distinta a la de los individuos con normopeso28, lo cual tiene repercusiones en la extracción energética de los alimentos, en el metabolismo de ácidos grasos, en la regulación de la síntesis de hormonas intestinales (incretinas) involucradas en la homeostasis energética y en la regulación de los depósitos corporales de tejido adiposo, ya que las bacterias participan directamente en el catabolismo de nutrientes para facilitar su absorción28. Esta simbiosis fue adquirida durante el tiempo de la cuarta glaciación, cuando los humanos debían cazar sus alimentos, correr rápido para no ser predados y pasaban días de ayuno prolongado; así, el hecho de presentar una microbiota intestinal que permitiera extraer al máximo la energía de los alimentos —cuando los había— favorecía el almacenamiento de grasa corporal; esto era una ventaja selectiva en aquel entonces, y es un factor del genotipo ahorrador, que proponía Neel en 196229-31. El humano probablemente ha sido seleccionando a lo largo de la evolución microorganismos con estas propiedades. Sin embargo, en las sociedades poscoloniales actuales con alto contenido energético el genotipo ahorrador y el fenotipo ahorrador de la microbiota favorecen la adipogénesis y la obesidad, ya que tanto los hábitos nutricionales como los tipos de alimentos no son los mismos, por lo que el fenotipo ahorrador de la microbiota intestinal se convierte en una desventaja, contribuyendo a la acumulación de grasa y al desarrollo de patologías relacionadas con la resistencia a la insulina28.
Partiendo de las observaciones anteriores en las que se puede ver francamente interacción entre factores nutricionales y genéticos, es importante considerar la interactómica entre la microbiota, los genes ahorradores de los microorganismos y del humano, con la alteración de la permeabilidad de las células de la mucosa intestinal por disrupción de las uniones estrechas, particularmente ZO-1 (codificada por TJP1), el cambio en la permeabilidad celular relacionado con el SNP rs2291166 de TJP1 que conduce a un cambio conformacional, como se ha visto en modelos experimentales de uveítis autoinmune o por toxicidad celular en el caso de la uremia20,32,33. Sumado a esto se ha descrito incremento en la permeabilidad endotelial mediada por ZO-1 en los niveles circulantes de la proteína de unión a lipopolisacárido (LBP) o en la endotoxemia metabólica causada por el LPS proveniente de la microbiota, parámetros de origen intestinal asociados al desarrollo de la obesidad28. Sin duda los ensayos en humanos, así como experimentales in vitro y en vivo para entender estas interacciones de la relación TJP1-homeostasis-microbiota, son la vía para relacionar a TJP1 como un mecanismo patogénico en la obesidad, lo cual escapa a los alcances del presente estudio por cuestiones legales (edad de los sujetos: son pediátricos), de ética y de usos así como costumbres de la comunidad donde se incluyeron, ya que no son muy permisivos para investigaciones.
A nivel de demografía genética y poblacional, el presente trabajo es el primero en aportar a nivel de Oaxaca, México, y a la diversidad poblacional nacional, las frecuencias de alelos y genotipos del SNP rs2291166 del gen TJP1 y el VNTR (CAG)n del ATXN2 en población con ancestria Chinanteca de Tuxtepec, así como su relación con parámetros de la obesidad, lo cual es muy importante desde la salud pública, ya que los sujetos de estudio son usuarios atendidos por los Servicios de Salud del Estado de Oaxaca a través del sistema de vigilancia epidemiológica, por lo cual los polimorfismos estudiados podrían ser utilizados como marcadores predictivos y preventivos de la obesidad. Ciertamente, como en otros estudios, los dos polimorfismos se encuentran en equilibrio de Hardy Weinberg y las frecuencias alélicas y genotípicas son muy similares, lo cual es congruente con la teoría de que los dos loci donde se hallan estos polimorfismos son loci muy conservados durante la evolución y entre los grupos étnicos amerindios9,15-18.
El presente estudio ofrece una ventaja sobre otros estudios realizados con obesidad y/o sobrepeso, porque se puede ver el efecto directo que tienen los polimorfismos analizados y el DQI sobre la obesidad y la resistencia a la insulina, cuyo fenotipo, en este trabajo, por lo menos no está traslapado con el síndrome metabólico o con los factores de riesgo cardiovascular, por lo cual son altamente sugestivos los efectos de los polimorfismos analizados en TJP1, ATXN2 y el DQI sobre el HOMA-IR y el QUICKI, lo cual se puede corroborar al ver los promedios de las variables bioquímicas de los sujetos incluidos (tabla 1), las cuales reflejan valores dentro del rango normal para glucosa, colesterol, triglicéridos, colesterol HDL y colesterol LDL, y por lo tanto se descartó un sesgo de información por el efecto o la relación que pudieran tener con la dislipidemia, la diabetes mellitus o la hipertensión arterial. Estos resultados deben replicarse en un estudio epidemiológico con más casos índices de la región, incluyendo población adulta, para validarlos como biomarcadores a nivel estatal o regional. Esto representa un reto, porque la zona de Tuxtepec y otros pueblos del Estado de Oaxaca tienen factores ambientales —como la alimentación regional altamente calórica— y culturales que conducen a la obesidad34.
A nivel de nutrición son limitados los estudios entre el DQI de la FAO y su relación directa con el IMC y el ICC, lo cual corrobora los resultados similares en población adulta de Valles Centrales, Oaxaca, México, con el síndrome metabólico35.
Cabe señalar que el presente estudio presenta limitantes; la principal es que no es un estudio de causalidad, el cual demuestre directamente la relación entre los polimorfismos y el DQI sobre parámetros de riesgo nutricional relacionados con la obesidad. Solo es un estudio de asociación que requiere ser corroborado por estudios posteriores experimentales moleculares in vivo e in vitro que demuestren el efecto a nivel fisiológico de los polimorfismos y del DQI, entre ellos analizar parámetros de inflamación relacionados con la permeabilidad intestinal, como los niveles de LBP o de LPS20. En este sentido sería útil la biopsia a través de la endoscopia para analizar la expresión a nivel de ARNm de los genes de la barrera intestinal, como son el caso de TJP1 y ATXN2, entre otros, así como cuantificar por inmunohistoquímica las proteínas y medir el efecto de la permeabilidad celular, lo cual es muy difícil en nuestro medio comunitario, ya que las prácticas y las investigaciones invasivas son muy restringidas, sobre todo si se trata de pacientes pediátricos o adolescentes.
La segunda limitante es que el número de sujetos incluidos en comparación con otros estudios con polimorfismos es muy pequeño5-7, pero tiene más de 50 cromosomas, lo mínimo requerido de muestra para análisis genético36, lo que obliga a hacer estudios posteriores de réplica epidemiológicos en otras poblaciones; sin embargo, el pequeño grupo Chinanteco de Tuxtepec que fue analizado es un grupo bien controlado, en el cual se ha logrado eliminar el sesgo que pudiera tener si se incluyeran obesos con parámetros de riesgo cardiovascular o síndrome metabólico. Además, la población analizada es un grupo vulnerable que no había sido metabólicamente estudiado, lo cual es una aportación de la presente investigación. Sin embargo, se sugiere aumentar el número de individuos en estudios similares futuros.
En conclusión, el presente estudio mediante pruebas de asociación y correlación muestra una relación directa entre los polimorfismos y el DQI con parámetros de riesgo de la obesidad: SNP rs2291166 del gen TJP1 influye más sobre la resistencia a la insulina (HOMA-IR), mientras que VNTR (CAG)n de ATXN2 influye sobre la sensibilidad a la insulina (QUICKI), y el DQI, sobre el IMC y el ICC.
Agradecimientos y financiaciónAl CONACYT por la beca de posgrado no. 2018 a favor del Lic. Arturo Caballero Avendaño. Por el financiamiento al Cuerpo Académico CA-UDG-701 Inmuno-metabolismo y envejecimiento de la Universidad de Guadalajara, con el número de financiamiento 01-34-2019 y a la Jurisdicción Sanitaria número 3, Servicios de Salud del Estado de Oaxaca, número financiamiento J3/EP/106/2019.
Conflicto de interesesNinguno.