La posible implicación de patógenos en el desarrollo de la aterosclerosis se ha estudiado durante décadas. Algunos estudios previos han logrado identificar la presencia de patógenos en la placa de ateroma. Sin embargo, muchas de estas determinaciones, se basan en técnicas estándar, son dirigidas y están encaminadas a detectar la presencia de una especie en particular. El empleo de tecnologías ómicas permite analizar el perfil microbiano completo de una determinada muestra. En el caso concreto de la placa de ateroma, el estudio de la composición carga bacteriana, ayudaría a esclarecer la posible relación entre infección y aterosclerosis e identificar si existe un perfil bacteriano asociado con placas inestables y, por lo tanto, con el consecuente riesgo de eventos isquémicos.
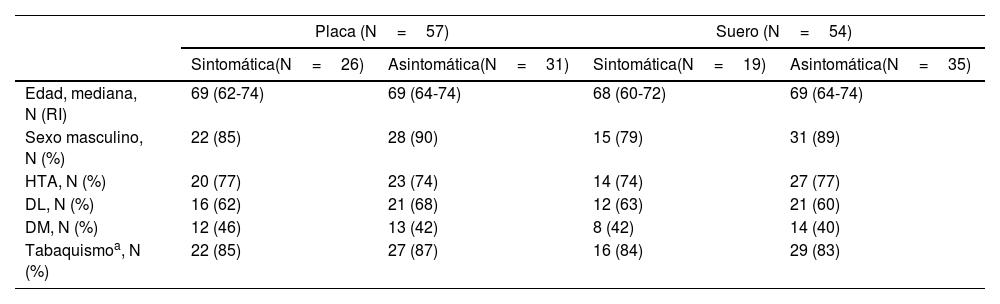
MetodologíaAnalizamos fragmentos transversales de placa de ateroma carotídeo (N=57) y suero (N=54) de los pacientes con sintomatología neurológica reciente y asintomáticos (grupo control). Se extrajeron los ácidos nucleicos de las muestras mediante digestión enzimática y homogeneización, previo tratamiento adicional con colagenasa tipo I en el caso de las placas. Se amplificó el RNA ribosomal bacteriano (gen 16S-rRNA) y se sometió a secuenciación masiva mediante a plataforma Illumina® Miseq. El análisis bioinformático, para identificar la composición taxónomica, y bioestadístico, para determinar los taxones significativos, del 16S-rRNA se realizó en el entorno R. Como control de contaminación se consideraron significativas ratios de especies bacterianas ≥10 respecto a controles negativos.
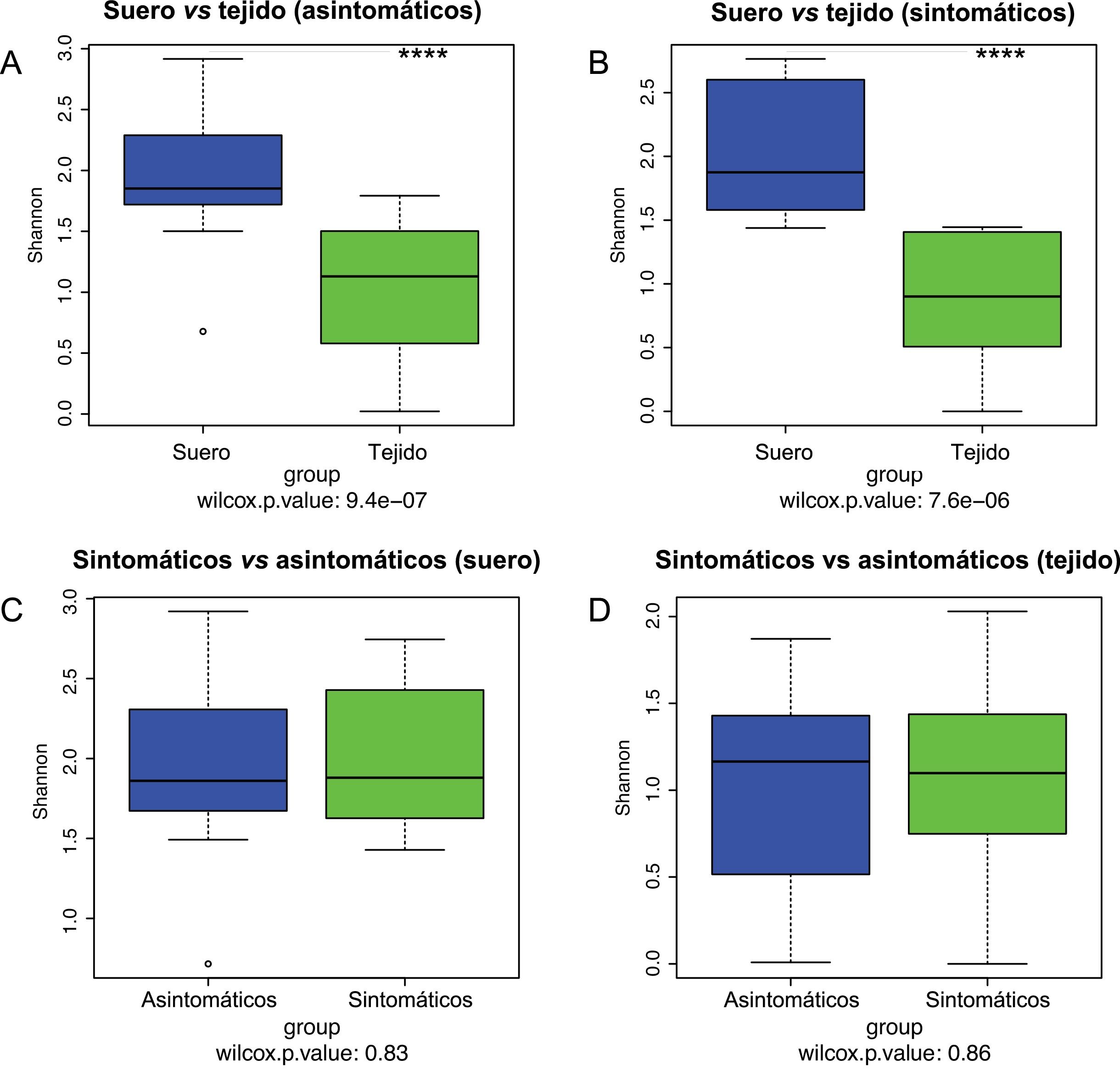
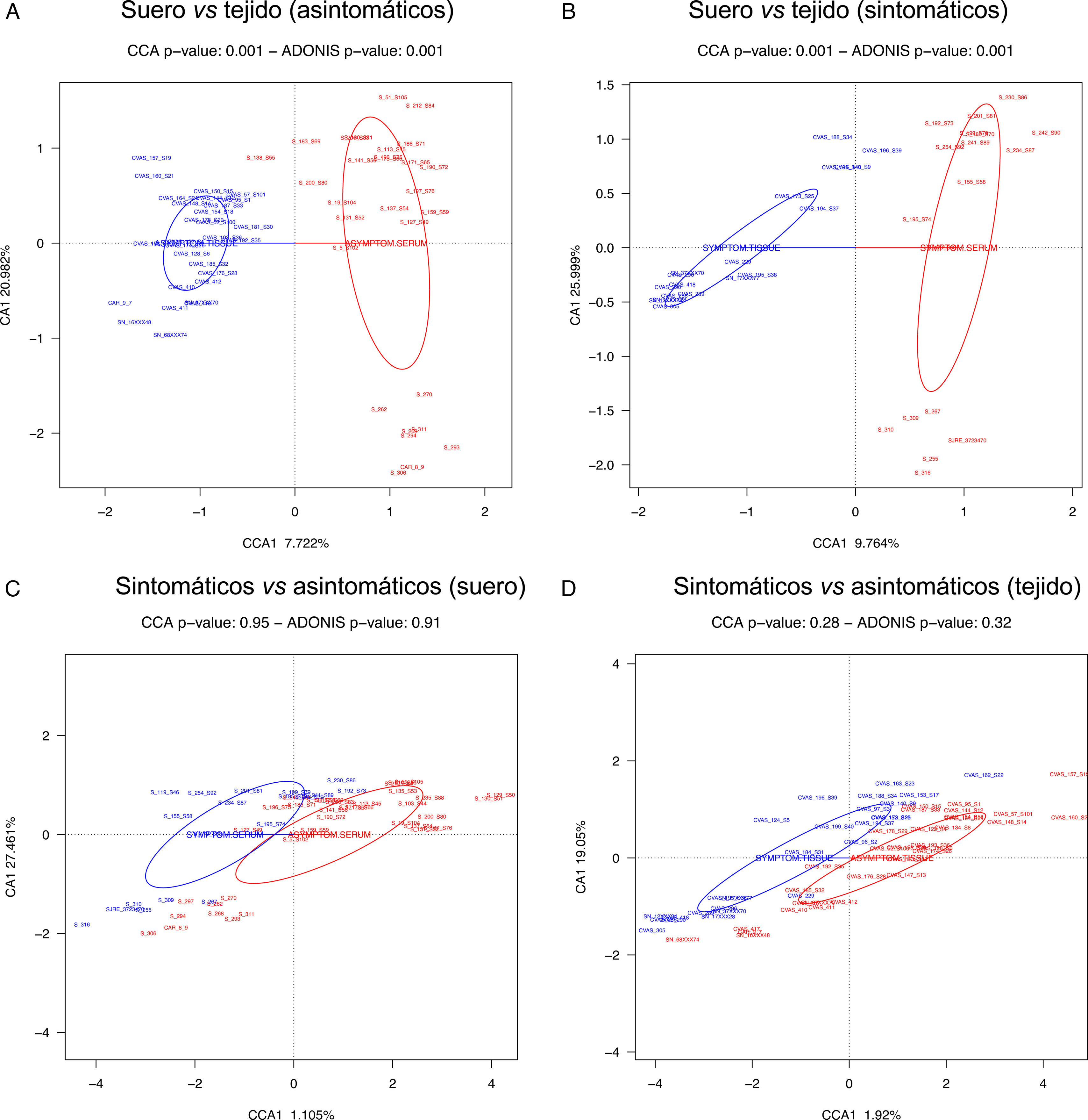
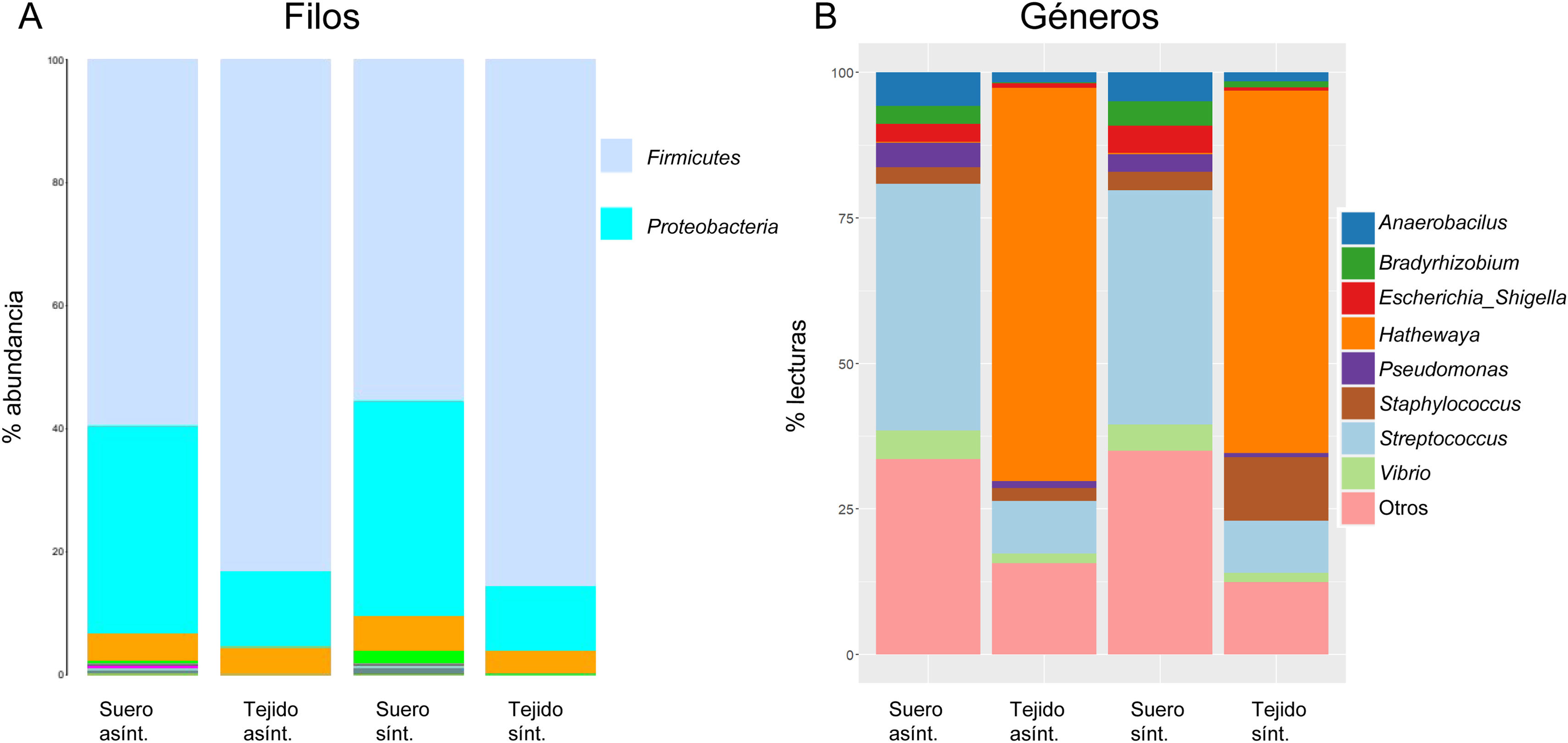
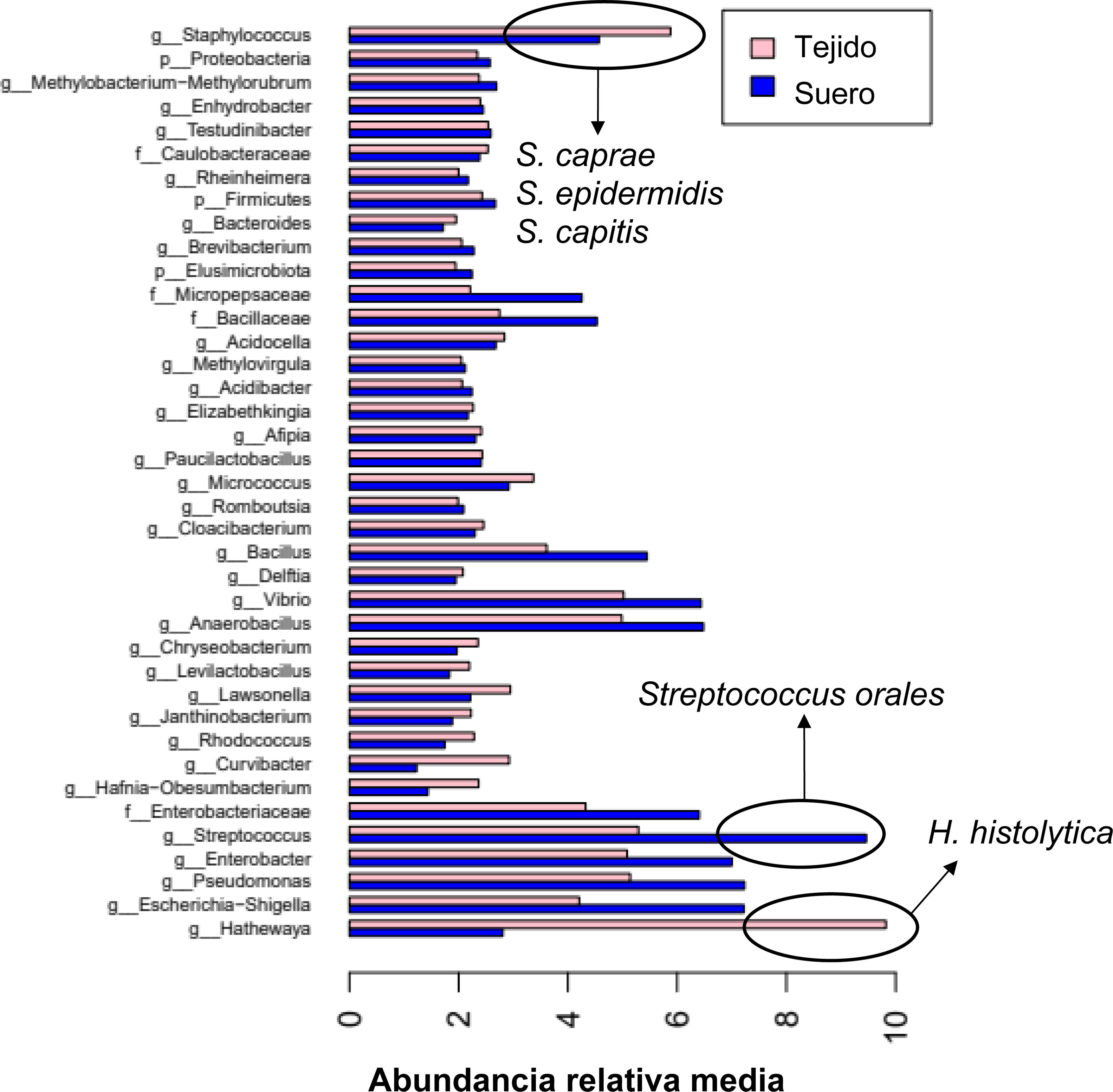
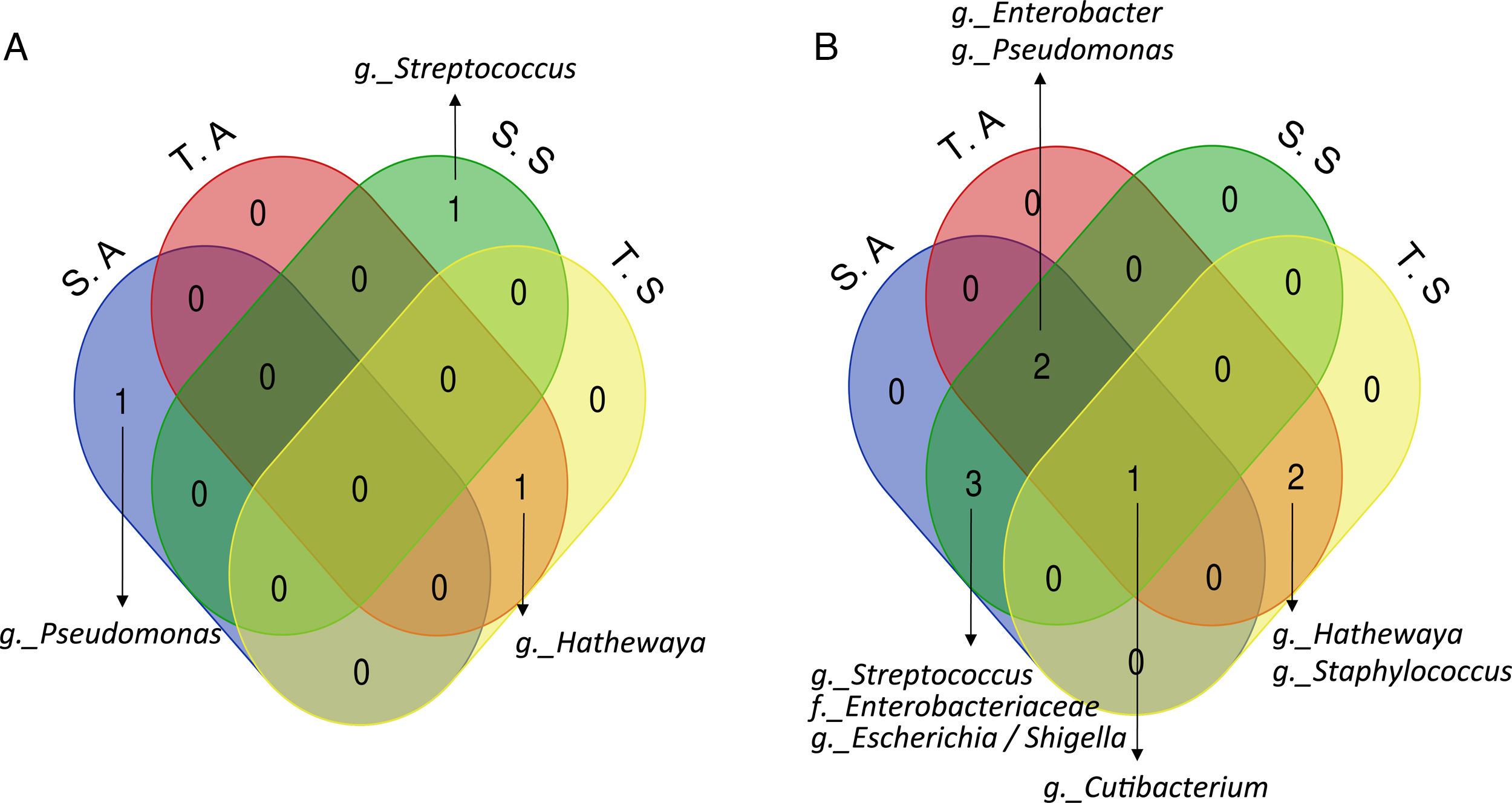
ResultadosLa presencia de 16S-rRNA bacteriano fue muy baja en ambos tipos de muestra. La composición bacteriana en términos de diversidad α y diversidad β difirió entre placa y suero; sin embargo, no observamos diferencias significativas entre muestras de los pacientes sintomáticos y asintomáticos. El filo y género más abundante en placa fueron, respectivamente, Firmicutes y Staphylococcus. Para Staphylococcus encontramos homología con el 100% de similitud del 16S-rRNA con 3 especies (S. epidermidis, S. caprae y S. capitis). En suero, los filos más abundantes fueron, Firmicutes y Proteobacteria, siendo Streptococcus el género dominante, para el que encontramos 100% de homología del 16S-rRNA con 20 especies de estreptococos orales.
ConclusionesHemos aplicado con éxito técnicas de secuenciación masiva para determinar la presencia y abundancia relativa de especies bacterianas en placas de ateroma y suero de los pacientes sometidos a endarterectomía carotídea. No hemos observado diferencias significativas entre los pacientes sintomáticos y asintomáticos en cuanto a los principales géneros, por lo que no podemos establecer una conexión directa entre la composición bacteriana y la vulnerabilidad de la placa de ateroma. Sin embargo, no se descarta una posible asociación entre la aterosclerosis y la presencia de estafilococos y estreptococos en placa y suero, respectivamente.
The potential involvement of pathogens in the development of atherosclerosis has been studied for decades. Some previous studies have successfully identified the presence of pathogens in the atheromatous plaque. However, many of these determinations are aimed at detecting the presence of a particular species. The use of omics technologies allows for the analysis of the complete microbial profile of a given sample. In the specific case of atheromatous plaque, the study of the bacterial load composition would help to clarify the possible relationship between infection and atherosclerosis and identify whether there is a bacterial profile associated with unstable plaques and, therefore, with the consequent risk of ischemic events.
MethodologyWe analyzed cross-sectional fragments of carotid atheromatous plaque (N=57) and serum (N=54) from patients with recent neurological symptoms and asymptomatic patients (control group). Nucleic acids were extracted from the samples by enzymatic digestion and homogenization, with additional treatment with type I collagenase in the case of plaques. Bacterial ribosomal RNA (16S-rRNA gene) was amplified and subjected to massive sequencing using the Illumina® Miseq platform. The bioinformatic analysis, to identify the taxonomic composition, and biostatistical analysis, to determine the significant taxa, of the 16S-rRNA was performed in the R environment. As contamination control, bacterial species ratios ≥10 with respect to negative controls were considered significant.
ResultsThe presence of bacterial 16S-rRNA was very low in both types of samples. The bacterial composition in terms of α diversity and β diversity differed between plaque and serum; however, we did not observe significant differences between samples from symptomatic and asymptomatic patients. The most abundant phylum and genus in plaque were Firmicutes and Staphylococcus, respectively. For Staphylococcus, we found 100% similarity homology of the 16S-rRNA with 3 species (S. epidermidis, S. caprae, and S. capitis). In serum, the most abundant phyla were Firmicutes and Proteobacteria, with Streptococcus being the dominant genus, for which we found 100% homology of the 16S-rRNA with 20 species of oral streptococci.
ConclusionsWe have successfully applied massive sequencing techniques to determine the presence and relative abundance of bacterial species in atheromatous plaques and serum of patients undergoing carotid endarterectomy. We have not observed significant differences between symptomatic and asymptomatic patients regarding the main genera, so we cannot establish a direct connection between bacterial composition and atheromatous plaque vulnerability. However, a possible association between atherosclerosis and the presence of staphylococci and streptococci in plaque and serum, respectively, cannot be ruled out.