analizar el valor pronóstico del sistema «TNM» y en concreto del estado «TN», frente al perfil de expresión génica tumoral, para evaluar el riesgo de enfermedad metastásica.
Materiales y métodosrevisión retrospectiva de 224 casos de cáncer infiltrante de mama. El riesgo genómico se estableció mediante la plataforma de expresión génica de 70 genes (MammaPrint®). Se ha considerado gold standard el resultado obtenido con la evaluación del riesgo genómico.
Resultados126 casos (56,25%) mostraron un perfil genómico de riesgo «bajo» y 98 casos (43,75%) de riesgo «alto». Entre los de riesgo genómico bajo, se observaron un 26,19% de tumores clasificados «T2», «T3» y «T4». Por el contrario, entre los de riesgo «alto», se observaron un 67,35% de tumores clasificados como «T1». En cuanto al estado «N», entre los tumores de riesgo «bajo», se observaron un 26,98% de casos clasificados como «N1», «N2» y «N3». En cambio, entre los de riesgo genómico «alto» se observaron un 77,14% de casos clasificados como «N0». Entre un total de 131 tumores «T1N0», 57 (43,51%) mostraron un riesgo genómico «alto». No se han observado diferencias significativas, en cuanto a evaluación del riesgo de enfermedad metastásica (alto vs. bajo), entre las diversas categorías «TN» estudiadas.
Conclusionesla evaluación del riesgo de enfermedad metastásica mediante el sistema «TN» no resulta acertada. La detección de tumores precoces no supone, necesariamente, un buen pronóstico. Por el contrario, la detección de tumores «T2» o «N1» no supone un mal pronóstico en todos los casos.
To analyze the prognostic value of the “TNM” system, and specifically the “TN” status, compared to the tumor gene expression profile, when evaluating the risk of metastatic disease.
Materials and methodsRetrospective review of 224 cases of infiltrating breast cancer. Genomic risk was established using the 70-gene gene expression platform (MammaPrint®). The result obtained with the genomic risk assessment has been considered “gold standard”.
Results126 cases (56.25%) showed a “low” risk genomic profile and 98 cases (43.75%) showed a “high” risk. Among those with low genomic risk, 26.19% of tumors classified “T2”, “T3” and “T4” were observed. On the contrary, among those with “high” risk, 67.35% of tumors classified as “T1” were observed. Regarding the “N” status, among the “low” risk tumors, 26.98% of cases classified as “N1”, “N2” and “N3” were observed. On the other hand, among those with “high” genomic risk, 77.14% of cases classified as “N0” were observed. Among a total of 131 “T1N0” tumors, 57 (43.51%) showed “high” genomic risk. No significant differences have been observed in terms of risk assessment of metastatic disease (high vs. low) between the various “TN” categories studied.
ConclusionsThe evaluation of the risk of metastatic disease using the “TN” system is not accurate. The detection of early tumors does not necessarily mean a good prognosis. On the contrary, the detection of “T2” or “N1” tumors does not imply a poor prognosis in all cases.
Con arreglo a los criterios pronósticos de sistema TNM, a su vez basados en el «paradigma halstediano»1, viene clásicamente aceptándose que los pequeños tumores (pT1) sin afectación ganglionar (pN0) suponen siempre «buenas noticias» para la paciente, y bajo esta premisa se defienden los beneficios de los programas de cribado. Por el contrario, el diagnóstico de tumores de mayor tamaño, con afectación ganglionar, invitan al pesimismo.
Sin embargo, los avances acaecidos durante los últimos años en biología molecular y genética (con la aparición de las plataformas de expresión génica tumoral)1, han venido a cuestionar, no solo el valor pronóstico del sistema TNM, sino también el de los subtipos moleculares subrogados. Así, algunos autores2 encuentran entre 19 y 34% de tumores de alto riesgo génico entre los casos detectados en el cribado, considerados «precoces». Y, por otro lado, existen claras discrepancias entre los subtipos moleculares subrogados y los determinados mediante técnicas de microarrays3.
Además, la presencia de células tumorales circulantes, y en la médula ósea, en los pacientes con cáncer precoz parece reforzar el paradigma «fisheriano» de diseminación1, aunque el verdadero impacto de tales células tumorales no sea aún bien conocido4.
Tales hechos, sin embargo, no invalidan algunos aspectos beneficiosos del cribado, puesto que la mayoría de casos detectados, especialmente en ronda prevalente, presentan un bajo riesgo genómico2 y, además, posibilitan las actuaciones locorregionales menos agresivas5.
De modo que no existe una completa correlación entre los sistemas clásicos (TNM y subtipos moleculares subrogados) de evaluación pronóstica y los basados en las plataformas de expresión génica6. Como consecuencia, pueden darse casos de sobretratamiento e infratratamiento entre las pacientes7, por lo que se hacen necesarias herramientas más precisas, como las citadas plataformas. De hecho, en la última edición del sistema TNM de evaluación pronóstica8, se ha incluido la información proporcionada por las plataformas de expresión génica tumoral, no descartando que estas resulten fundamentales, a corto plazo, a la hora de evaluar un pronóstico y de planificar correctamente una terapia sistémica en todos los casos de cáncer infiltrante de mama.
En el presente trabajo original presentamos nuestra experiencia al respecto, poniendo de manifiesto que, si se admite la superioridad del valor pronóstico de las plataformas de expresión génica tumoral, el sistema TNM debiera perder el valor pronóstico del que aún goza.
Material y métodosRevisión retrospectiva de 224 tumores infiltrantes de mama, observados entre 2013 y 2023, que disponen de perfil de expresión génica tumoral y estadificación anatomopatológica «pT» y «pN».
La plataforma utilizada ha sido, en todos los casos, MammaPrint® (AGENDIA), con resultados obtenidos en un único laboratorio centralizado, a partir de muestras quirúrgicas o procedentes de biopsias percutáneas, indistintamente. Dicha plataforma clasifica el riesgo genómico en «alto» y «bajo».
Dentro del sistema «TN» se han contemplado las siguientes situaciones consideradas clásicamente de buen pronóstico: tumores pT1, tumores pN0 y tumores T1N0. También se han contemplado los datos «TN» del resto de tumores (pT2/3/4, pN1/2), clásicamente considerados de peor pronóstico.
Se ha determinado el valor predictivo positivo (VPP) de la afectación ganglionar axilar positiva para alto riesgo genómico, así como de los parámetros «T1N0» y «pN0» para bajo riesgo genómico.
Se ha considerado «test de referencia» (gold standard) el resultado obtenido con la plataforma de expresión génica.
Análisis estadísticoSe ha utilizado el test de chi cuadrado para evaluar la asociación entre las variables de estadificación «TN» y genómicas de riesgo, considerando significativo un valor de p<0,05.
ResultadosSe estudiaron 126 tumores (56,25%) de bajo riesgo genómico y 98 casos (43,75%) de alto riesgo genómico en pacientes con los siguientes intervalos de edad: menores de 40 años (7 casos), entre 40 y 49 años (58 casos), entre 50 y 69 años (109 casos) y mayores de 69 años (50 casos). No se observaron diferencias significativas, en cuanto a los grupos de edad y riesgo genómico.
Entre los 224 tumores revisados, 159 casos (70,98%) fueron clasificados como pT1, 164 casos clasificados como pN0 (73,21%) y 131 casos (58,48%) cumplieron criterios T1N0.
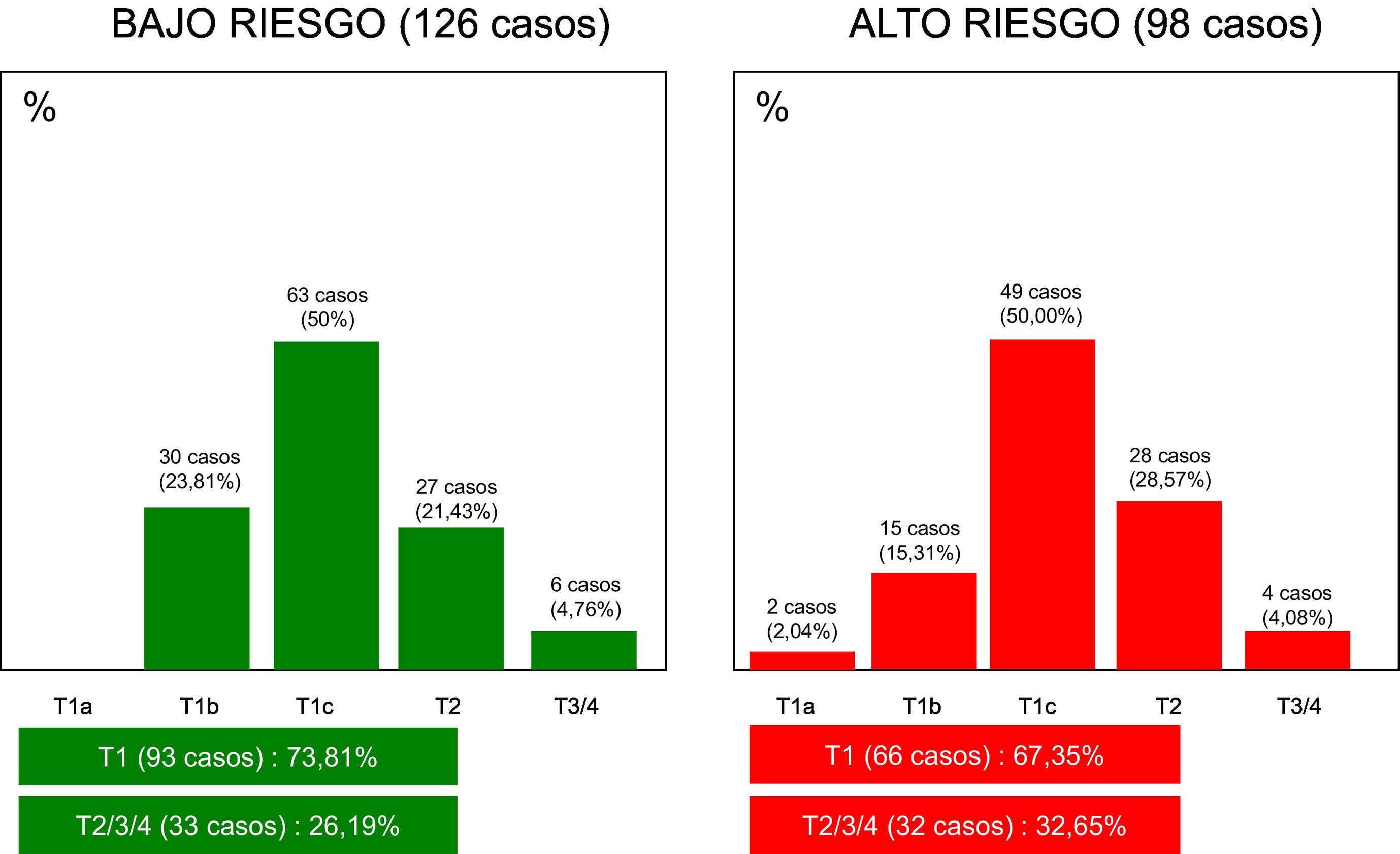
La distribución de casos, en cuanto al tamaño tumoral (pT) y riesgo genómico (alto vs. bajo) puede apreciarse en la figura 1. A destacar que un 26,19% de tumores pT2/3/4 mostraron bajo riesgo genómico, mientras que un 67,35% de tumores pT1 mostraron alto riesgo genómico. No se han observado diferencias significativas entre el parámetro «T» y el riesgo genómico (es decir, riesgo de enfermedad metastásica).
Estado «T» y perfil de expresión génica tumoral (224 casos). En la gráfica de la izquierda se representan los tumores de bajo riesgo. En la gráfica de la derecha, los tumores de alto riesgo. Obsérvese que, entre los de bajo riesgo, figura un 26,19% de tumores T2/T3/T4. Por el contrario, entre los altos riesgos, figuran un 67,35% de tumores T1.
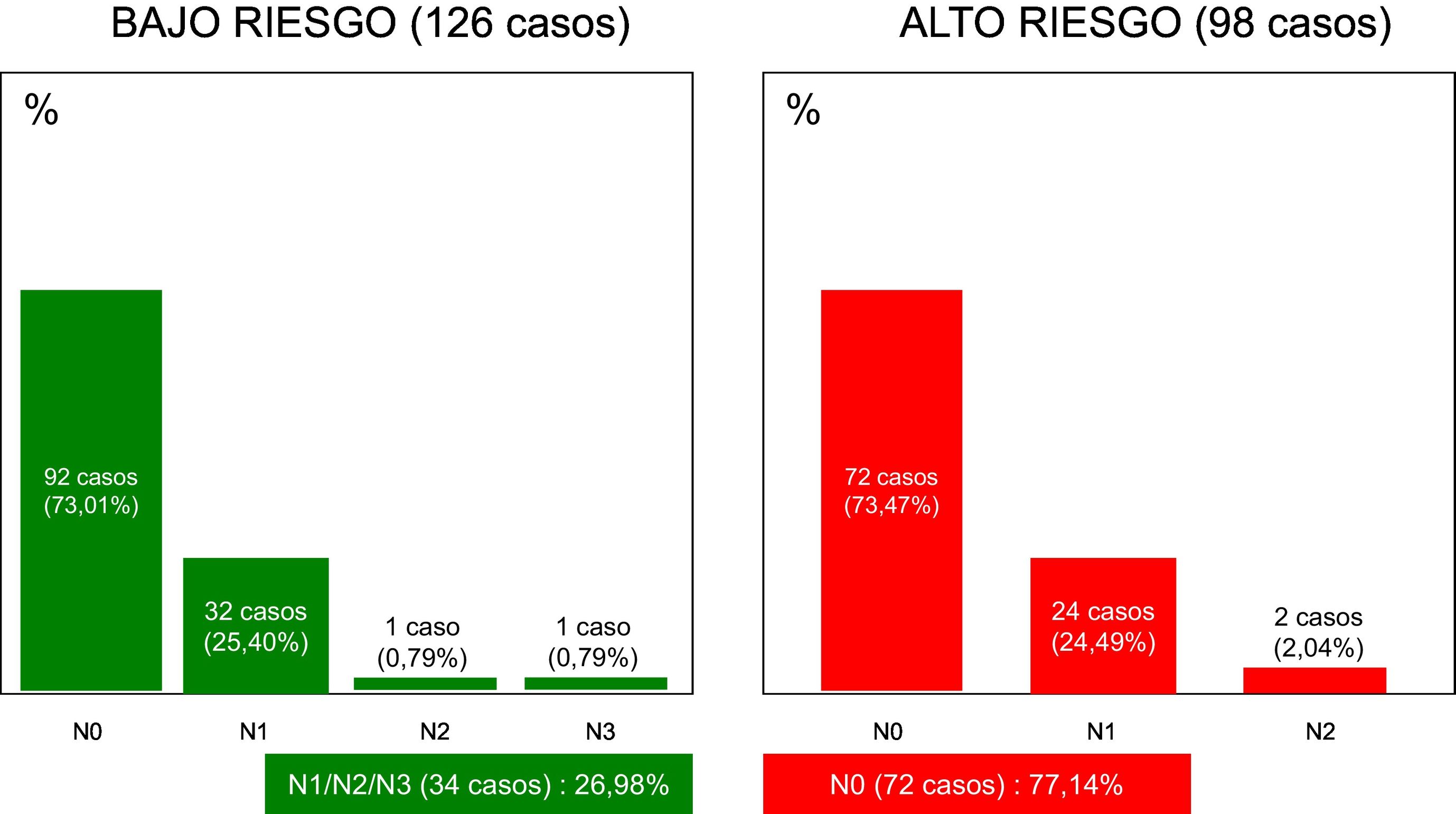
La distribución de casos, en cuanto al estado ganglionar axilar (pN) puede apreciarse en la figura 2. A destacar que un 26,98% de casos clasificados como pN1/2/3 mostraron bajo riesgo genómico, mientras que un 77,14% de los clasificados como pN0 mostraron alto riesgo genómico. No se han observado diferencias significativas entre el parámetro «N» y el riesgo genómico (es decir, riesgo de enfermedad metastásica).
Estado «N» y perfil de expresión génica tumoral (224 casos). En la gráfica de la izquierda se representan los tumores de bajo riesgo. En la gráfica de la derecha, los tumores de alto riesgo. Obsérvese que, entre los de bajo riesgo, figura un 26,98% de tumores N1/N2/N3. Por el contrario, entre los altos riesgo, figuran un 77,14% de tumores N0.
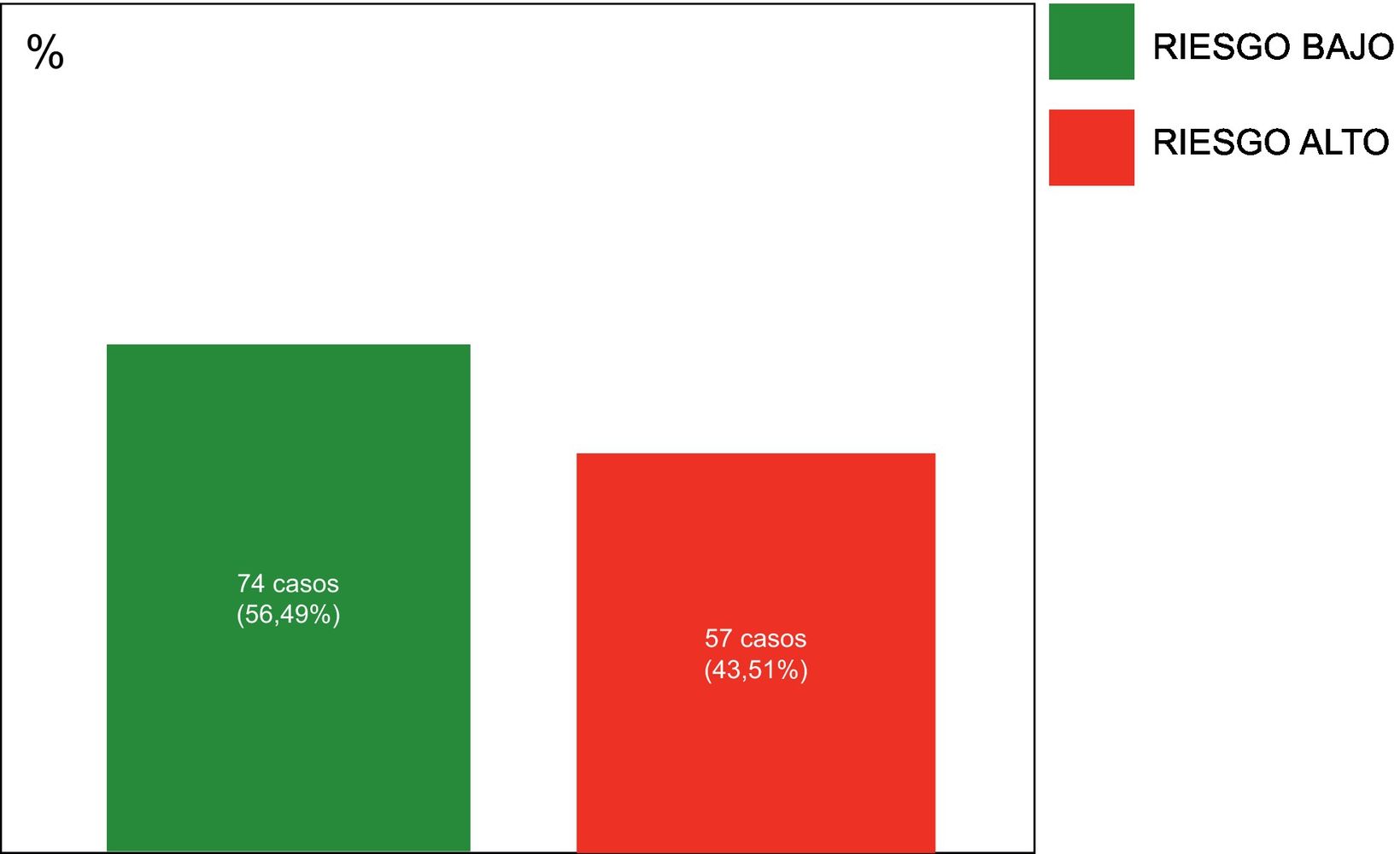
Entre los 131 tumores T1N0, 57 (43,51%) mostraron alto riesgo genómico (fig. 3).
El VPP de la afectación ganglionar para predecir alto riesgo genómico, es de 26,53%. Los VPP de los factores «T1N0» y «pN0», para predecir bajo riesgo genómico, son de 56,49 y 56,10%, respectivamente.
DiscusiónResulta habitualmente aceptado que la detección precoz es fundamental para hacer frente al cáncer de mama y que el mejor método para ello es la realización periódica de mamografías, que contribuyen a la reducción de las tasas de mortalidad entre las mujeres que se las realizan periódicamente, (resultados básicamente reportados entre mujeres de 40-74 años9). Sin embargo, nunca han faltado voces críticas10 que cuestionan tales aspectos beneficiosos de las mamografías y el impacto positivo de la detección precoz en la supervivencia de las pacientes y en la reducción de la mortalidad, voces progresivamente amplificadas gracias al asentamiento del paradigma de Fisher11–13, que postula que el cáncer de mama es una enfermedad sistémica desde su inicio y, por lo tanto, la detección precoz de pequeños tumores, sin afectación ganglionar axilar (objetivo perseguido con arreglo al paradigma de Halsted), no tendría la gran importancia que se le viene atribuyendo, de cara al pronóstico de las pacientes.
La evidencia de que casi un tercio de pacientes con tumores «pN0» (sin afectación ganglionar) desarrollaba enfermedad metastásica, y de que un tercio de pacientes con afectación ganglionar permanecía libre de enfermedad sistémica al menos 10 años tras la terapia local11, invitó a cuestionar la validez del paradigma halstediano, la validez pronóstica del sistema TNM y, por lo tanto, el papel «curativo» de la detección precoz.
Desde 1960 y durante la década de los 70, tuvieron lugar diversas observaciones clínicas y de laboratorio, en especial el ensayo randomizado NSABP (National Surgical Adjuvant Breast and Bowell Project, B-04), después de que el cirujano norteamericano Bernard Fisher y su hermano, el patólogo Edwin R. Fisher, iniciaran un extenso estudio acerca de las metástasis del cáncer de mama que desembocaron en formular la teoría, que abanderó B. Fisher (1918-2019), de que el cáncer de mama es en realidad una enfermedad sistémica desde el principio, y de que existe la probabilidad de diseminación de células tumorales (por lo tanto, probabilidad de metástasis) incluso antes del diagnóstico clínico/radiológico del tumor primario13. De este modo, el denominado paradigma de Fisher parecía explicar mejor el comportamiento biológico tumoral en algunos casos. Y no solo eso, también permitió cuestionar la utilidad de los procedimientos quirúrgicos locales radicales para el control sistémico de la enfermedad.
Y es que, como se viene demostrando durante los últimos años, el cáncer de mama es en realidad una enfermedad heterogénea y los tradicionales marcadores pronósticos (basados en el sistema TNM) tan solo son capaces de estimar atinadamente el pronóstico en aproximadamente un 30% de pacientes. Para identificar el 70% restante se necesitan nuevos biomarcadores11.
Era necesario avanzar más y mejor para conocer de manera más fiable el comportamiento biológico del tumor (en especial el riego de desarrollar enfermedad metastásica), por lo que pronto aparecieron en escena las determinaciones de material genético tumoral (a partir del ADN/ARN, principalmente) mediante diversas técnicas, fundamentalmente PCR (reacción en cadena de la polimerasa) y microarrays11,14.
En este sentido, en el año 2002 van de Vijver MJ, et al.15 dieron a conocer el primer estudio de validación clínica de una conocida y pionera plataforma de expresión génica (la denominada por algunos, firma holandesa de 70 genes), como método para evaluar el comportamiento biológico tumoral, que confirmó su importancia práctica años después, tras la realización y posterior análisis de uno de los proyectos prospectivos de referencia obligada (MINDACT). Entre las conclusiones del proyecto MINDACT7, figuraba el dato de que la clásica evaluación clínica del riesgo de enfermedad metastásica (basada en parámetros histopatológicos, inmunohistoquímicos y subtipos moleculares subrogados) sobrestimaba el riesgo de enfermedad metastásica en más del 46% de los pacientes, y lo subestimaba en el 17,74%, con el consiguiente riesgo de sobretratamiento e infratratamiento sistémico quimioterápico, respectivamente.
En efecto, diversos trabajos16 parecen demostrar las limitaciones del factor de proliferación Ki67 como parámetro discriminador de pacientes a la hora de prescribir/no prescribir terapia sistémica con quimioterapia. Entre los tumores de bajo riesgo genómico, el valor del factor Ki67 es igual/mayor del 14% en 52% de casos, igual/mayor de 20% en 43%, e igual/mayor de 30% en 17%. Por otro lado, 15% de tumores de alto riesgo genómico presentan valores Ki67 menores de 14%. Ello puede motivar sobretratamiento o infratratamiento16 sistémico.
Nuestros resultados reflejan las limitaciones pronósticas del sistema TNM, tanto en los tumores «precoces», considerados clásicamente de buen pronóstico, como en los tumores con afectación ganglionar axilar, considerados clásicamente como de mal pronóstico, algo ya descrito previa y recientemente por nosotros16. Por otra parte, la presencia de células tumorales circulantes en la sangre de pacientes con cáncer de mama, ya fue confirmada hace varios años a nivel molecular, así como su contribución a la diseminación metastásica del cáncer17. De hecho, usando técnicas de secuenciación de célula individualizada, se ha demostrado que la generación espontánea de estas células tumorales circulantes con una alta propensión a metastatizar no ocurre continuamente, sino que se concentra en la fase de reposo del individuo afectado, lo que sugiere un debate sobre el tratamiento controlado en el tiempo de los cánceres propensos a metástasis18. A pesar de los avances en las técnicas de secuenciación, la toma de decisiones sobre el tratamiento guiadas por perfiles genómicos integrales sigue siendo un desafío en la práctica oncológica habitual. Sin embargo, ensayos clínicos están empezando a mostrar evidencias de que las decisiones lideradas por la genómica presentan beneficios en pacientes con cáncer de mama metastásico19.
Parece demostrado que los programas de cribado posibilitan la detección de tumores que, frecuentemente, muestran un riesgo genómico bajo. Sin embargo y con arreglo a nuestros resultados aquí presentados, el hecho de que un 43,51% de tumores T1N0 muestren alto riesgo genómico debería hacer reflexionar acerca de los profusamente divulgados beneficios de los programas de cribado, y las mujeres que participan en los mismos deberían ser conscientes de ello, tras recibir una información más ajustada al comportamiento biológico tumoral.
Podría concluirse que, en la actualidad, el sistema TNM sigue siendo útil para la planificación terapéutica local/regional, pero la planificación de la terapia sistémica debiera fundamentarse, principalmente, en la información suministrada por una plataforma de expresión génica eficaz7, incluso en pacientes con afectación ganglionar axilar (fenómeno no siempre de significado infausto), como contempla la firma holandesa de 70 genes. De hecho, la última edición (8ª) del sistema TNM8 (en la que, por cierto, han desaparecido de la portada las siglas TNM), recoge la aportación predictiva y pronóstica aportada por las plataformas de expresión génica20, especialmente en el denominado «cáncer precoz»21.
ConclusionesLa evaluación del riesgo de enfermedad metastásica mediante el sistema «TN» no resulta acertada en muchos casos y debiera limitarse a la estadificación locorregional. La detección de tumores precoces no supone, necesariamente, un buen pronóstico. Por el contrario, la detección de tumores con afectación ganglionar no supone un mal pronóstico en todos los casos.
Consideraciones éticasLas muestras objeto de estudio son las habitualmente obtenidas durante los protocolos rutinarios de trabajo, en los cuales se incluyen los correspondientes consentimientos informados (biopsia percutánea, cirugía y determinación del perfil de expresión génica tumoral). Se han observado las elementales normas de protección de datos.
Consentimiento informadoLos autores declaran que han obtenido el consentimiento de los pacientes para la publicación de este artículo.
FinanciaciónNinguna.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
AgradecimientosEn el contexto sanitario privado, donde ejercemos nuestro trabajo cotidiano casi todos/as los/las autores/as del presente trabajo, no ha sido fácil recabar los datos necesarios, correspondientes a múltiples pacientes, atendidos en diversos centros, consultas y laboratorios (generalmente «incomunicados» digital y electrónicamente). Por ello, deseamos agradecer la impagable colaboración e infinita paciencia de diversas personas (incluidos algunos colegas), aunque principalmente a personal no sanitario y perfil administrativo, en especial a Vicky Peláez (Departamento de Soporte de la empresa Healthincode, Málaga, España) y a Javier Jiménez (Laboratorio de Anatomía Patológica, Hospital Zorrotzaurre, Bilbao, España).