Evaluar la utilidad de la biopsia ecoguiada con aguja gruesa diagnóstica (BAG1) para la determinación del perfil de expresión génica tumoral (PEG), en los tumores malignos de mama.
Materiales y métodosRevisión de 130 biopsias ecoguiadas con aguja gruesa (BAG), con resultado de malignidad. Se consideraron «aptas» las muestras con, al menos, un 30% de células tumorales. Se estudió la influencia del tamaño tumoral (menor de 1 cm, entre 1-2 cm y mayor de 2 cm) y se analizaron las causas que motivaron muestras no aptas. Se utilizó la plataforma MammaPrint® (70 genes). Se evaluó la influencia del grado histológico y del riesgo genómico en los resultados.
ResultadosEn la BAG1 se obtuvieron muestras aptas en 100 biopsias (76,92%). Entre los 36 casos en los que se utilizó la BAG para obtener el PEG, en 32 (88,89%) se realizó a partir de la BAG1. Entre los 30 casos en los que la BAG1 no resultó apta, en 26 casos no se obtuvo el porcentaje mínimo de células tumorales en la muestra. Ni el grado histológico ni el riesgo genómico influyeron en los resultados.
ConclusionesLas muestras diagnósticas de la biopsia ecoguiada con aguja gruesa (BAG1) pueden ser válidas para la determinación del perfil de expresión génica. Ello facilita y acelera el proceso de evaluación pronóstica en los tumores infiltrantes de mama. Por ello, proponemos que, de manera rutinaria y ante el diagnóstico de tumor maligno infiltrante, conste el porcentaje de células tumorales en los informes anatomopatológicos.
To assess the utility of diagnostic ultrasound-guided core needle biopsy (CNB1) for determining tumour gene expression profile (GEP) in malignant breast tumours.
Materials and MethodsReview of 130 diagnostic ultrasound-guided core needle biopsies (CNB1) indicating malignancy. Samples with at least 30% tumour cells were considered suitable. The influence of tumour size (less than 1 cm, between 1-2 cm and greater than 2 cm) was studied and the causes of unsuitable samples were analysed. The MammaPrint® platform (70 genes) was used. The influence of histological grade and genomic risk was evaluated.
ResultsSuitable CNB1 samples were obtained in 100 biopsies (76.92%). Among the 36 cases in which CNB was used to obtain the GEP, in 32 (88.89%) it was performed using the CNB1. Among the 30 cases in which CNB1 was not suitable, the minimum percentage of tumour cells in the sample was not obtained in 26 cases. Neither histological grade nor genomic risk influenced the results.
ConclusionsDiagnostic samples from ultrasound-guided biopsy (CNB1) can be valid to determine GEP. This facilitates and accelerates the prognostic evaluation process in infiltrating breast tumours. Therefore, we propose that, when diagnosing an infiltrating malignant tumour, the percentage of tumour cells should be routinely recorded in the pathology reports.
La biopsia percutánea con aguja gruesa (BAG) se ha convertido en una herramienta fundamental para el diagnóstico del cáncer de mama. Permite alcanzar un diagnóstico no quirúrgico de malignidad, así como obtener una importante información predictiva y pronóstica mediante determinaciones inmunohistoquímicas (en especial: receptores de estrógenos, receptores de progesterona, oncogén HER2 y factor de proliferación celular Ki67) y sus correspondientes subtipos moleculares subrogados (comúnmente, Luminal A, Luminal B, HER2 y Triple Negativo).
Con la aparición de los Perfiles de Expresión Génica (PEG), al principio sólo posibles a partir de muestras quirúrgicas «en fresco», pronto se desarrollaron técnicas que permitieron su determinación a partir de bloques de parafina, utilizando muestras de menor volumen, tales como las obtenidas mediante la BAG1–3. De este modo, las muestras de biopsia percutánea han ampliado notablemente sus utilidades en el manejo del cáncer de mama, de tal manera que hoy, gracias a la información inmunohistoquímica y genómica obtenida, permiten una evaluación predictiva y pronóstica muy certera.
Las referencias bibliográficas con respecto al uso de la BAG para la determinación de los PEG son escasas4–10, en especial las referentes al uso de las muestras de la BAG realizada durante el proceso diagnóstico inicial (BAG1)6,8,9, por lo que nuestro objetivo es determinar la potencial validez de la biopsia percutánea ecoguiada, con diagnóstico de malignidad, para la determinación del PEG mediante una plataforma de 70 genes.
Material y métodosRevisión retrospectiva de 130 casos de cáncer de mama diagnosticados mediante BAG ecoguiada, en los que se dispone de información sobre el porcentaje de células tumorales en la muestra de BAG1, distribuidos en dos periodos. Desde enero de 2013, en que nuestro equipo multidisciplinar se planteó el uso de la plataforma de 70 genes recurriendo a las muestras de la BAG1, tras indicación clínica para la determinación del PEG y cuando nuestro patólogo habitual (primero) y el laboratorio centralizado (después) las consideraban aptas o válidas. Durante este primer periodo se reunieron 19 casos. Desde noviembre de 2018, en que nuestro habitual laboratorio de Anatomía Patológica acordó informar, adicionalmente, sobre el porcentaje de células tumorales en las muestras correspondientes a carcinomas infiltrantes, en el informe anatomopatológico rutinario, por si fuese necesaria la determinación del PEG a partir de la BAG1. A este segundo periodo corresponden 111 casos.
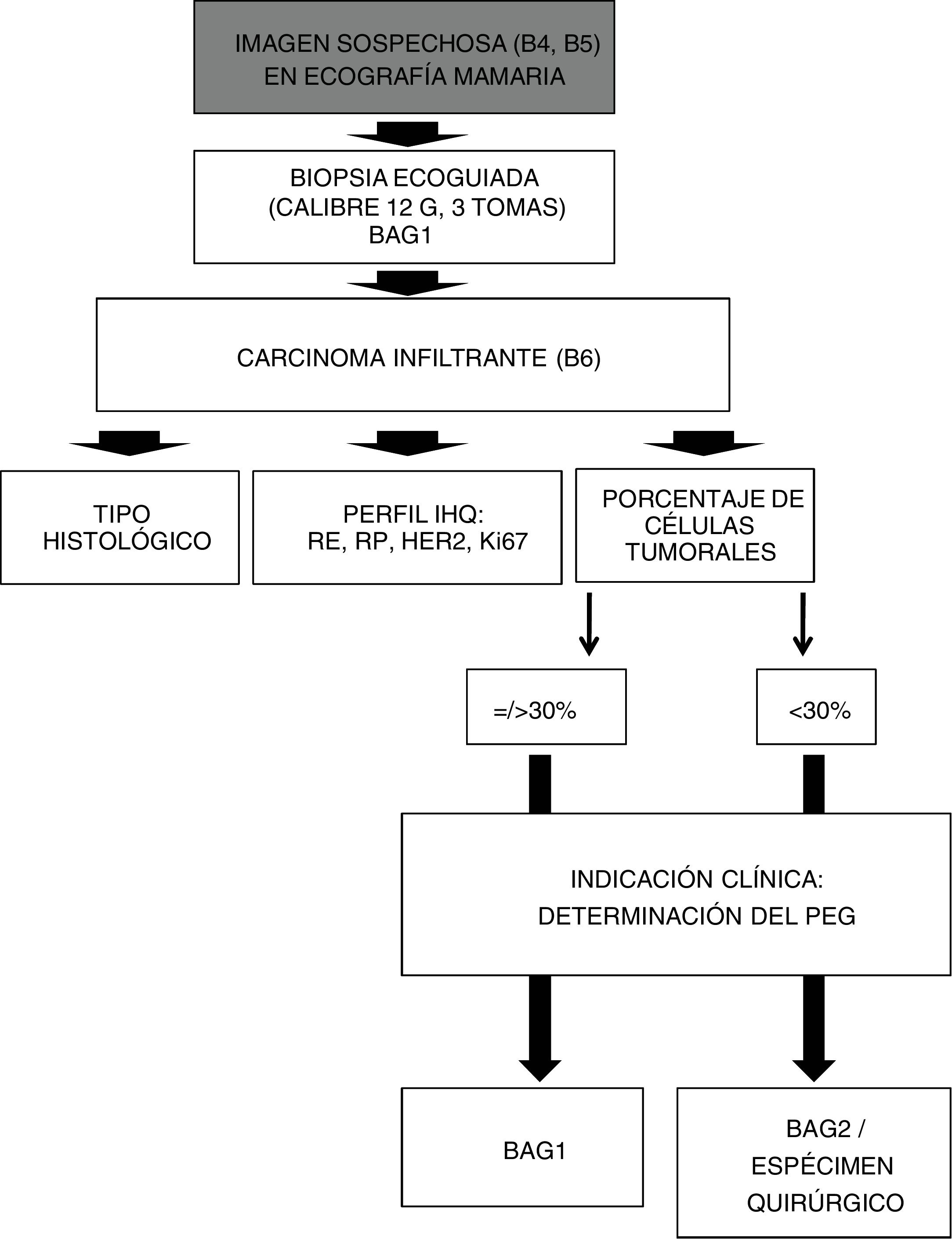
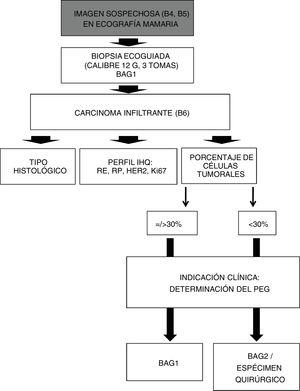
Con arreglo a nuestro actual protocolo de actuación (fig. 1), toda lesión sospechosa de malignidad (categorías BI-RADS 4 y 5), visible en ecografía, se somete a BAG ecoguiada (BAG1). En el caso de confirmarse su naturaleza maligna, el anatomopatólogo procede a las habituales determinaciones inmunohistoquímicas: receptores de estrógeno y progesterona, onocogén HER2 (incluyendo técnicas FISH/SISH, si fuera necesario) y factor de proliferación celular Ki67.
Algoritmo de actuación, ante imágenes sospechosas de malignidad, visibles en ecografía mamaria.
B4 = BI-RADS 4. B5 = BI-RADS 5. BAG1: Biopsia con Aguja Gruesa diagnóstica. B6 = BI-RADS 6 (lesión de malignidad probada).
IHQ = InmunoHistoQuímica. RE = Receptores de Estrógeno. RP = Receptores de Progesterona. HER2 = Oncogén. Ki67 = Factor de proliferación celular. BAG2 = Biopsia con Aguja Gruesa, segunda toma, en caso de BAG1 no válida.
En el caso de que hubiera indicación clínica para la determinación del PEG, nuestro equipo multidisciplinar utiliza habitualmente la plataforma de 70 genes MammaPrint® (Agendia Inc., Amsterdam, The Netherlands), a partir de los bloques de parafina, considerando válidas las muestras con al menos un 30% de células tumorales. MammaPrint® considera dos posibles resultados: alto riesgo o bajo riesgo.
En este trabajo se han considerado válidas las muestras de BAG1 cuando así lo ha validado finalmente el laboratorio centralizado (en el caso de determinación del PEG), o cuando nuestro laboratorio de anatomía patológica determinaba un porcentaje de células tumorales igual o superior al 30% (en el caso de no indicación clínica para la determinación del PEG).
En el caso de que la BAG1 no fuese apropiada para la determinación del PEG, se toma la decisión clínica acerca de realizar una segunda biopsia percutánea (BAG2), dedicada exclusivamente a obtener muestras para la determinación del PEG, o bien de recurrir al espécimen quirúrgico (en el caso de pacientes no candidatas a terapia neoadyuvante).
Todas las BAG fueron realizadas por radiólogos con experiencia en intervencionismo de Mama, mediante agujas automáticas «de corte lateral», utilizando agujas de calibre 12 G (seleccionando una longitud de disparo de 22 mm), con una media de tres pases.
La evaluación del porcentaje de células tumorales se realizó en el mismo laboratorio de Anatomía Patológica, en todos los casos, a partir de los bloques de parafina. La estimación del volumen tumoral (equivalente al porcentaje de células tumorales) con respecto al volumen tisular total se ha efectuado de forma aproximada a partir de los cortes histológicos teñidos con hematoxilina/eosina, sin utilizar sistemas de digitalización, desechando las áreas de necrosis y del componente inflamatorio.
Una vez verificada la validez de la muestra y la indicación clínica de la determinación del PEG, el anatomopatólogo seleccionó el bloque de parafina a enviar al laboratorio centralizado.
En los casos en los que se realizó correlación con el estadio «TN» y el Grado Histológico, éstos se han obtenido tras confirmación quirúrgica, excepto en pacientes candidatas a iniciar terapia neoadyuvante, en cuyo caso el estadio se ha estimado de manera radiológica (mediante ecografía y/o resonancia magnética mamaria) o a través de la BAG (Grado Histológico), respectivamente.
Se han evaluado el tamaño tumoral y el porcentaje reportado de células tumorales, se realiza una revisión bibliográfica al respecto y se discute acerca de las causas que han dado lugar a las muestras de BAG1 no válidas.
Ética en la publicaciónDado que el material y los datos manejados en el presente trabajo no suponen haber realizado procedimientos adicionales a los ya rutinarios (incluyendo el consentimiento informado en los casos de determinación del perfil de expresión génica), entendemos que cumplimos los requisitos contemplados en el código ético de la Organización Mundial de la salud (OMS) (Declaración de Helsinki) y las normas sobre Ética de la publicación científica.
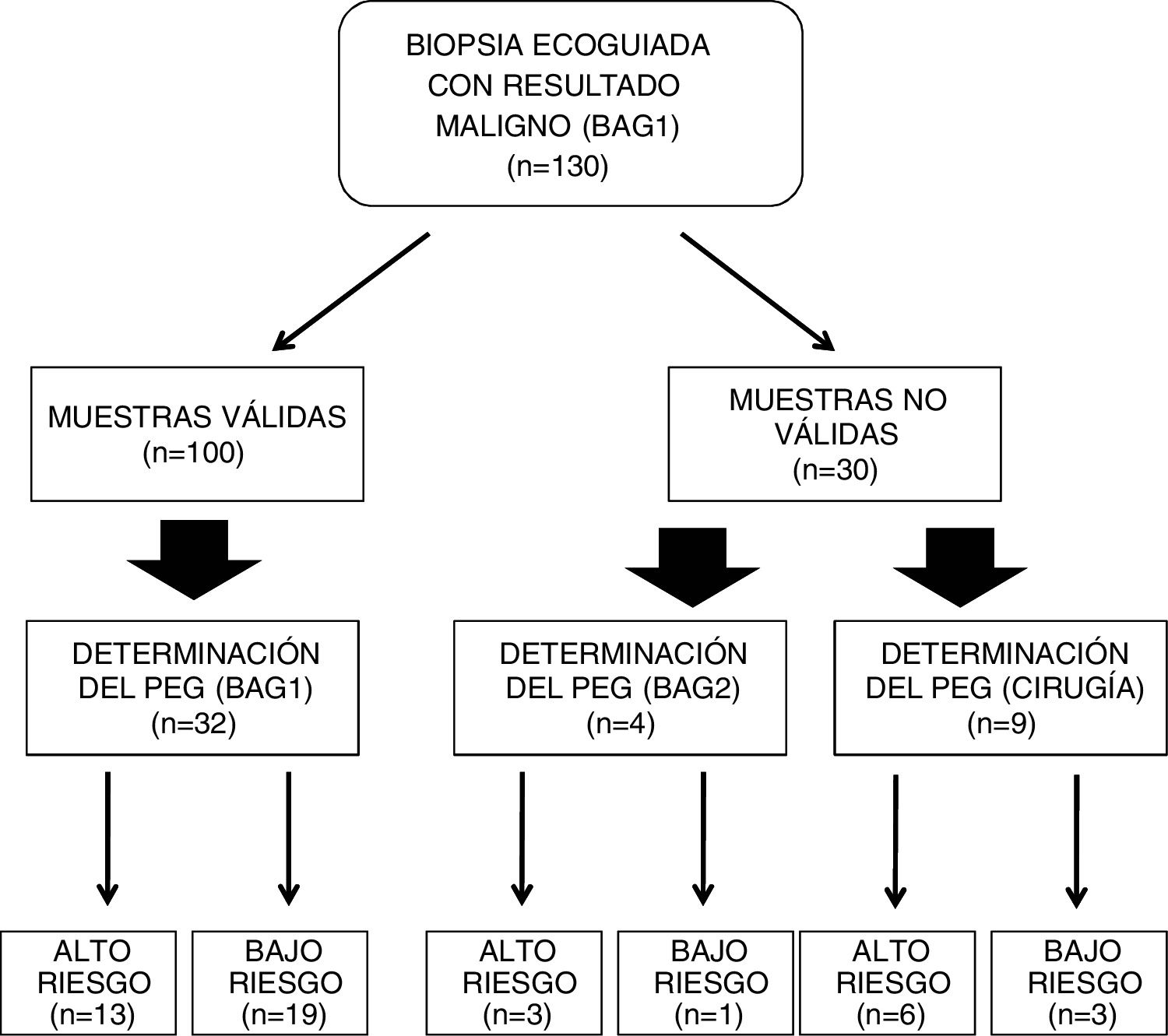
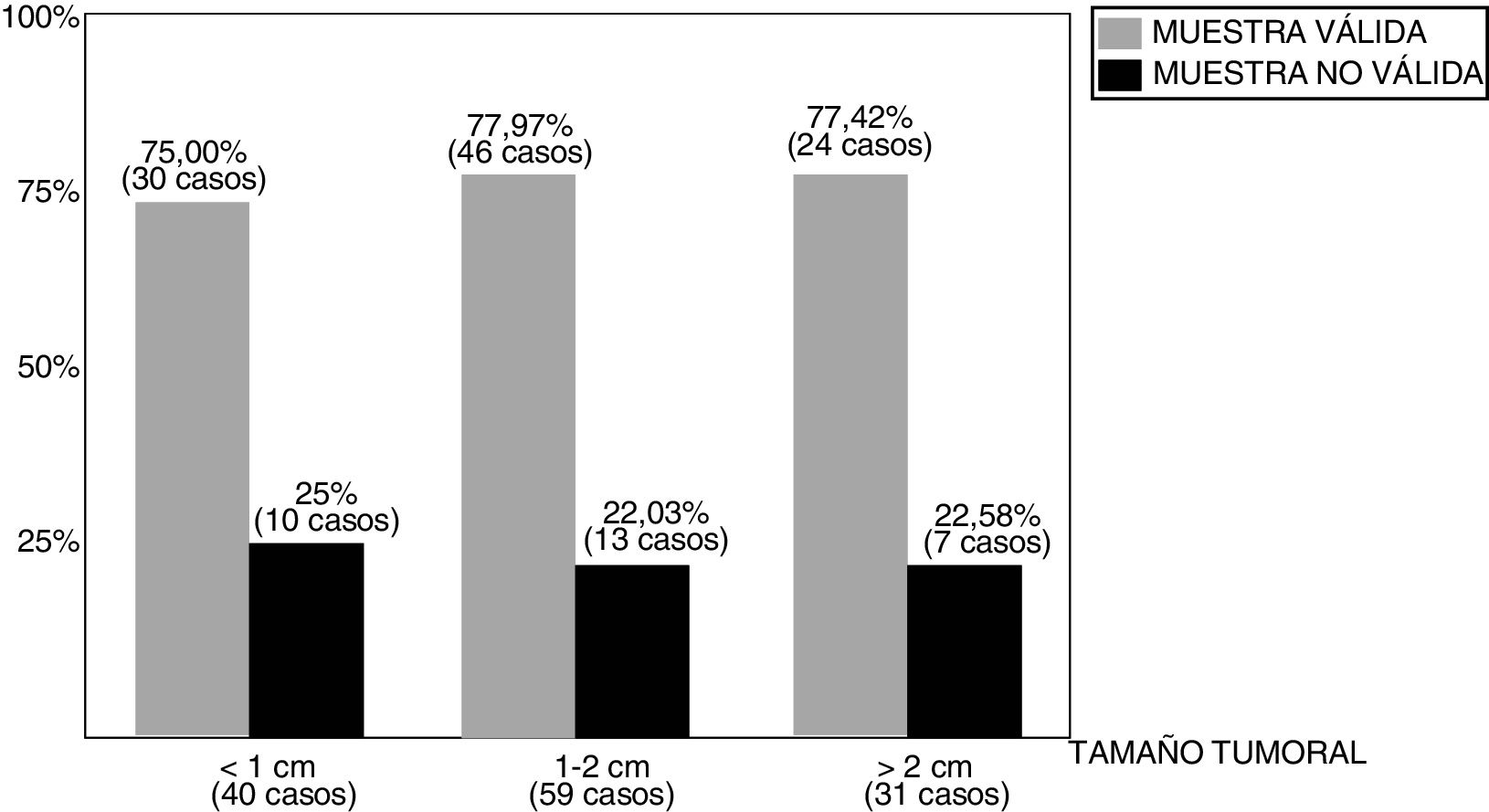
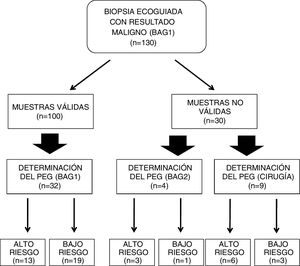
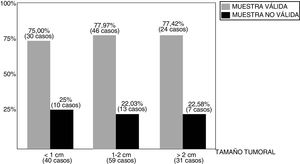
ResultadosLa distribución del número de muestras y respectivos resultados puede observarse en la fig. 2. Entre los 130 casos revisados, la BAG1 fue considerada válida para la determinación del PEG en 100 casos (76,92%), si bien el porcentaje de validez de las muestras varía según el tamaño de las lesiones biopsiadas (fig. 3), de tal modo que los mejores resultados (77,97% de muestras válidas) se han observado entre los tumores de tamaño comprendido entre 1 y 2 cm.
Entre las muestras consideradas válidas (100 casos con porcentajes celulares tumorales iguales o mayores al 30%), en 82 casos (82%) se obtuvieron porcentajes celulares tumorales mayores o iguales al 50%.
En 36 casos (fig. 2 y tabla 1) se indicó la determinación del PEG a partir de las muestras de biopsia percutánea, 32 a partir de la BAG1 (88,89%) y cuatro a partir de la BAG2.
BAG, estadificación «TN», grado histológico y riesgo genómico
| CASO | EDAD | BAG | T | N | G | RIESGO |
|---|---|---|---|---|---|---|
| 005 | 47 | BAG2 | T2* | N2* | 2 | ALTO |
| 006 | 58 | BAG1 | T1c | N0 | 2 | BAJO |
| 010 | 43 | BAG1 | T1c | N1 | 2 | BAJO |
| 013 | 41 | BAG1 | T1c | N0 | 2 | BAJO |
| 014 | 61 | BAG1 | T2 | N1 | 2 | BAJO |
| 021 | 44 | BAG1 | T1b | N0 | 2 | BAJO |
| 022 | 59 | BAG1 | T1c | N0 | 3 | ALTO |
| 023 | 42 | BAG1 | T1c | N0 | 2 | BAJO |
| 025 | 82 | BAG1 | T2 | N0 | 2 | ALTO |
| 027 | 46 | BAG1 | T2 | N0 | 2 | BAJO |
| 036 | 40 | BAG1 | T2* | N1* | 2 | BAJO |
| 055 | 57 | BAG1 | T1c | N0 | 2 | BAJO |
| 059 | 57 | BAG1 | T1c | N0 | 1 | ALTO |
| 068 | 74 | BAG1 | T1c | N0 | 2 | ALTO |
| 069 | 67 | BAG1 | T2 | N2 | 2 | BAJO |
| 070 | 40 | BAG1 | T1c | N1 | 2 | ALTO |
| 076 | 47 | BAG1 | T1c | N0 | 2 | ALTO |
| 077 | 40 | BAG1 | T1c | N1 | 2 | BAJO |
| 080 | 74 | BAG1 | T1c | N0 | 2 | BAJO |
| 086 | 56 | BAG1 | T1b | N1 | 2 | BAJO |
| 088 | 55 | BAG2 | T2* | N1* | - | ALTO |
| 091 | 52 | BAG1 | T1c | N0 | 2 | BAJO |
| 092 | 53 | BAG2 | T2* | N1* | - | ALTO |
| 093 | 82 | BAG1 | T2 | N0 | 2 | ALTO |
| 094 | 53 | BAG1 | T1c | N1 | 3 | BAJO |
| 095 | 67 | BAG1 | T1c | N0 | 2 | ALTO |
| 103 | 64 | BAG2 | T2* | N1* | - | BAJO |
| 104 | 67 | BAG1 | T1c | N0 | - | ALTO |
| 105 | 65 | BAG1 | T1c | N0 | 3 | ALTO |
| 106 | 58 | BAG1 | T1c | N0 | 2 | ALTO |
| 108 | 61 | BAG1 | T1b | N1 | 1 | BAJO |
| 109 | 61 | BAG1 | T1b | N0 | 2 | BAJO |
| 112 | 48 | BAG1 | T1b | N0 | 2 | ALTO |
| 113 | 77 | BAG1 | T1b | N0 | 2 | BAJO |
| 114 | 39 | BAG1 | T2 | N1 | 2 | ALTO |
BAG: Biopsia con Aguja Gruesa; T, N: Datos quirúrgicos según el sistema «TNM»
G: Grado histológico; T*N*: Estadificación clínica (previa a terapia neoadyuvante)
BAG1: BAG inicial, diagnóstica
BAG2: BAG consecutiva por muestra no válida en BAG1
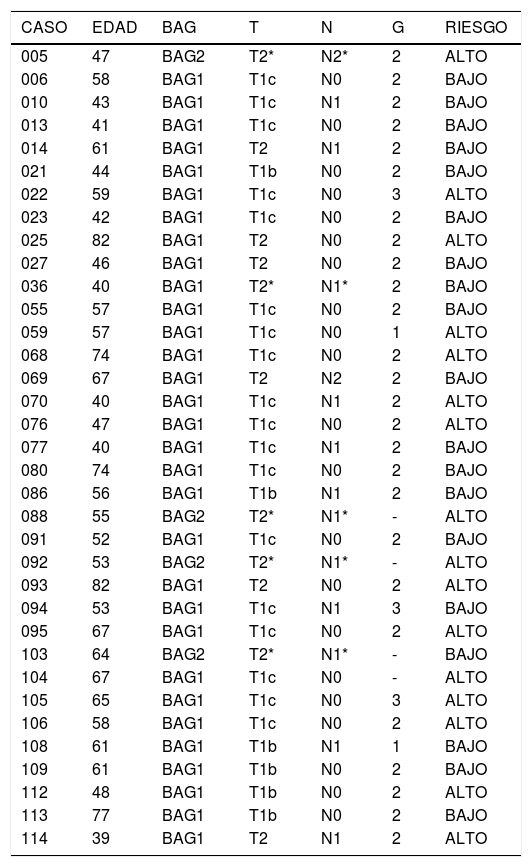
Entre los 36 casos, en 16 se obtuvo un riesgo genómico «alto», y en 20 casos un riesgo genómico «bajo». Entre los cuatro casos en los que fue necesario recurrir a una segunda biopsia (BAG2), tres mostraron un riesgo genómico «alto» (tabla 1). El tamaño de las lesiones en los cuatro casos en los que fue necesario recurrir a una segunda biopsia (BAG2), era mayor de 2 cm (tres casos) y entre 1-2 cm (un caso).
Entre los 32 casos en los que se realizó la determinación del PEG a partir de la BAG1, hubo 19 (59,38%) que resultaron ser bajo riesgo (fig. 2). Entre los dos casos con Grado histológico alto (G3), uno de ellos resultó ser bajo riesgo (tabla 1).
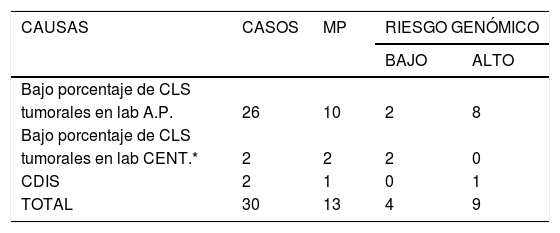
Entre los 30 casos en los que la BAG1 no resultó apropiada para la determinación del PEG (tabla 2), la causa más frecuente fue el bajo porcentaje de células tumorales en la muestra (26 casos), seguida de la discrepancia entre nuestro laboratorio de patología y el centralizado con respecto al porcentaje de células tumorales en la muestra (dos casos) y la obtención del diagnóstico de carcinoma ductal in situ (dos casos). Entre los 30 casos, en 13 existió indicación clínica para la determinación del PEG, ya sea a partir de una segunda biopsia percutánea o bien a partir de una muestra quirúrgica (fig. 2). Tras la cirugía, uno de los dos casos de carcinoma ductal in situ correspondió, finalmente, a carcinoma ductal infiltrante con un PEG de riesgo alto.
Causas de muestras no válidas en BAG1 y riesgo genómico obtenido a partir de BAG2 o muestras quirúrgicas
| CAUSAS | CASOS | MP | RIESGO GENÓMICO | |
|---|---|---|---|---|
| BAJO | ALTO | |||
| Bajo porcentaje de CLS | ||||
| tumorales en lab A.P. | 26 | 10 | 2 | 8 |
| Bajo porcentaje de CLS | ||||
| tumorales en lab CENT.* | 2 | 2 | 2 | 0 |
| CDIS | 2 | 1 | 0 | 1 |
| TOTAL | 30 | 13 | 4 | 9 |
BAG1: Biopsia diagnóstica
BAG2: Biopsia repetida
MP = Indicación of MammaPrint®
LAB A.P. = Laboratorio de Anatomía Patológica
LAB CENT. = Laboratorio centralizado
El uso de la BAG en el diagnóstico no quirúrgico del cáncer de mama está plenamente aceptado en la inmensa mayoría de guías de actuación, considerándose un estándar de atención. Por otro lado, la información inmunohistoquímica (IHQ) obtenida a partir de las muestras de BAG (conservadas en bloques de parafina) presenta alta correlación con la obtenida a partir del espécimen quirúrgico, correlación también alcanzada en lo concerniente a la determinación del PEG2,3,8.
Se estima que bastarían cantidades de 1 μg de ácido desoxirribonucleico (ADN), o dos muestras de BAG (con agujas de calibre 18G) de al menos 1 cm de tumor, para poder realizar la determinación del PEG, aunque en la práctica y bajo tales condiciones el 20% de muestras no serían válidas. Si se utiliza ácido ribonucleico (ARN), se estima que bastarían 100 células tumorales, si bien el grado de degradación del ARN parece superior en las muestras de BAG, y de hecho algunos postulan la superioridad de las muestras obtenidas mediante punción aspirativa con aguja fina (PAAF) en este sentido11. Pero incluso disponiendo de alto contenido celular, tan sólo el 50% de las muestras permitirían obtener el PEG11.
De este modo, la posibilidad de la determinación del PEG, a partir de muestras de BAG conservadas en parafina, permitió engrosar los usos potenciales de la misma, en especial y junto con la IHQ, el de una evaluación predictiva y pronóstica sumamente fiable.
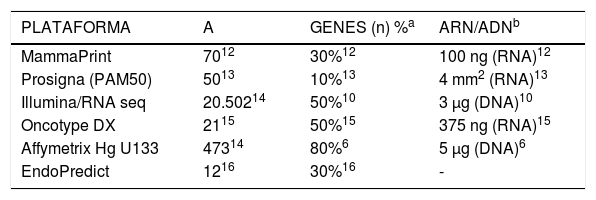
En la actualidad se dispone de varias plataformas comerciales (tabla 3) para la determinación del perfil de expresión génica tumoral (PEG). Con arreglo a la información obtenida a partir de algunos autores y de algunas empresas del sector (tabla 3)6,10,12–16, el porcentaje celular/cantidad de ARN/ADN mínimo exigido para la determinación del PEG varía según la plataforma utilizada, siendo la menos «exigente» Prosigna (PAM50), con sólo un 10%, y la más Affymetrix Hg U133, con un 80% de contenido celular tumoral mínimo. La plataforma utilizada en este trabajo (MammaPrint®), requiere de un porcentaje celular tumoral mínimo del 30% para proceder a la determinación del PEG12. Por otro lado, su uso ha sido validado en pacientes con afectación ganglionar axilar12.
Plataformas de expresión génica y mínimo porcentaje requerido de células tumorales y contenido de ARN/ADN*
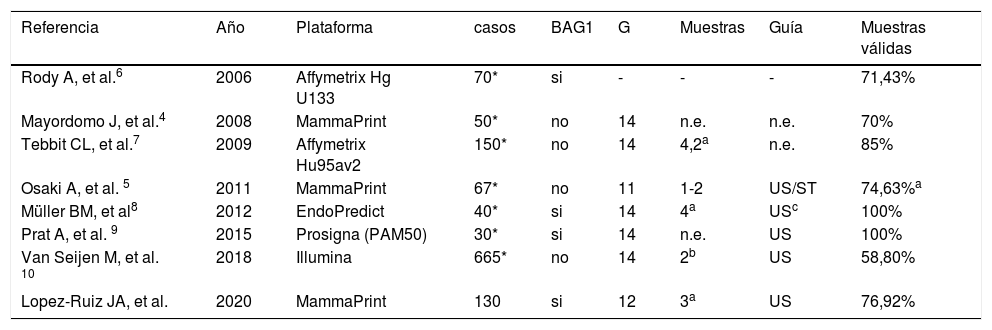
El uso de las muestras obtenidas en BAG de mama para la determinación del PEG ha sido abordado previamente, generalmente focalizado en pacientes seleccionados (que reúnen determinados criterios clínicos y patológicos), y en general a partir de muestras obtenidas en una segunda toma, dirigida exclusivamente a la determinación del PEG4-6,8–10,17. Son muy escasas las referencias bibliográficas relativas a la posible validez de las muestras obtenidas en la BAG16,8,9, de modo que nuestros resultados son difícilmente comparables a la mayoría de series publicadas (tabla 4). Por otro lado, las plataformas utilizadas por otros autores son diversas, con metodología diferente, con calibres de aguja variados y con un número de muestras también diferente, lo cual dificulta aún más realizar un estudio comparativo de nuestros resultados. Desde el punto de vista metodológico (uso de bloques de parafina pertenecientes a la BAG1 ecoguiada), podrían considerarse las series de Rody A, et al.6, de Müller BM et al.8, y de Prat A, et al.9, con porcentajes reportados de muestras válidas de 71,43%, 100% y 100%, respectivamente, si bien los casos estudiados en tales series han sido el fruto de algún de tipo de selección, en base a determinados parámetros clínicos, terapéuticos o inmunohistoquímicos, con un número de pacientes bastante inferior y con plataformas menos exigentes en algunos casos9.
Perfil de expresión génica mediante BAG: resultados comparativos
| Referencia | Año | Plataforma | casos | BAG1 | G | Muestras | Guía | Muestras válidas |
|---|---|---|---|---|---|---|---|---|
| Rody A, et al.6 | 2006 | Affymetrix Hg U133 | 70* | si | - | - | - | 71,43% |
| Mayordomo J, et al.4 | 2008 | MammaPrint | 50* | no | 14 | n.e. | n.e. | 70% |
| Tebbit CL, et al.7 | 2009 | Affymetrix Hu95av2 | 150* | no | 14 | 4,2a | n.e. | 85% |
| Osaki A, et al. 5 | 2011 | MammaPrint | 67* | no | 11 | 1-2 | US/ST | 74,63%a |
| Müller BM, et al8 | 2012 | EndoPredict | 40* | si | 14 | 4a | USc | 100% |
| Prat A, et al. 9 | 2015 | Prosigna (PAM50) | 30* | si | 14 | n.e. | US | 100% |
| Van Seijen M, et al. 10 | 2018 | Illumina | 665* | no | 14 | 2b | US | 58,80% |
| Lopez-Ruiz JA, et al. | 2020 | MammaPrint | 130 | si | 12 | 3a | US | 76,92% |
BAG1 = Muestras diagnósticas en todos los casos; G = calibre de aguja
En cuanto a las series que utilizaron la misma plataforma comercial (tabla 4), Mayordomo et al.4 utilizaron muestras «frescas» (50 casos), exclusivamente dedicadas a la determinación del PEG, conservadas en un medio apropiado y con un porcentaje de muestras válidas del 70%, mientras que Osaki A et al.5 (67 casos) utilizaron biopsia asistida por vacío, con agujas de calibre 11G, separando a priori dos muestras exclusivas para la determinación del PEG, con un 74,63% de muestras válidas, resultado discretamente inferior al nuestro (76,92%), con la importante diferencia de que, en nuestra serie, no hubo separación de muestras para la determinación del PEG y el calibre de aguja fue menor.
En general, y en todas las series citadas (tabla 4), ha sido posible (aunque con desigual acierto) obtener el PEG en la mayoría de los casos. Algunos autores10 opinan que la posibilidad de obtener muestras aptas para la determinación del PEG a partir de la BAG depende del Grado histológico (mayor en tumores Grado 3) y del perfil de riesgo genómico (mayor en tumores de «alto» riesgo), algo que nosotros no hemos podido confirmar (tabla 1). Por otro lado, resulta evidente que «tumor precoz» no presupone buen pronóstico, puesto que entre los 18 casos «T1N0» (tabla 1), nueve (50%) presentaron un PEG de alto riesgo.
Otros autores11,18 evalúan la influencia del tamaño de las muestras de la BAG (con arreglo al calibre de las agujas utilizadas) en la probabilidad de obtener la determinación del PEG, sin claras conclusiones. En principio, parece bastar el uso de agujas de calibre 18G, aunque la mayoría de autores4,8–10,17 reportan el uso de agujas de calibre 14G en sus series, con porcentajes variables en la validez de las muestras. Otros autores18 encuentran una relación directa entre el volumen del tumor obtenido mediante la BAG y la fiabilidad de Grado histológico resultante, argumentando que, de ese modo, las limitaciones de una posible heterogenicidad tumoral son menores. En consonancia con tal argumento, nosotros pensamos que un calibre de 12G garantiza un mayor porcentaje de muestras válidas (para una determinación del PEG), permiten la obtención de porcentajes celulares tumorales elevados (más del 50% en el 82% de las muestras válidos) y, por lo tanto, una mejor correlación de resultados con el verdadero comportamiento biológico tumoral.
En 30 casos (25,66%) no nos ha resultado posible obtener muestras consideradas válidas para la determinación del PEG, debido sobre todo a la presencia de un bajo contenido celular (tabla 2), no totalmente dependiente del tamaño de la lesión (fig. 3). Otras causas adicionales descritas19 son la presencia única o predominante de componente «in situ» (dos de nuestros casos), o de zonas periféricas de lesión, con presencia de tejido no tumoral.
El porcentaje de muestras válidas en la BAG1 difiere, comprensible aunque discretamente, según el tamaño tumoral (fig. 3), entre el 75% (tumores menores de 1 cm) y el 77,97% (tumores entre 1 y 2 cm). Pero no encontramos una explicación plausible al porcentaje encontrado (77,42%) entre los tumores mayores de 2 cm, en teoría más «proclives» para la obtención de muestras válidas. Tal vez la presencia de fenómenos de necrosis tumoral (en algunos casos) o bien la dificultad de visualizar nítidamente los límites del tumor (en otros casos) a la hora del abordaje ecográfico, pudieran justificarlo.
Aunque el número de casos aquí presentado pudiera parecer modesto (aun así resulta el tercero de todas las series publicadas hasta la fecha, y el más numeroso con la misma plataforma) (tabla 4), parece clara la alta probabilidad (76,92%) de obtener muestras válidas en la BAG 1 para la determinación del PEG con la plataforma de 70 genes, aunque tal vez la probabilidad pudiera ser más alta con el uso de plataformas menos «exigentes» (tabla 3). En todo caso, creemos que sería conveniente que se hiciera constar el porcentaje de células tumorales en el informe anatomopatológico de la BAG1 (como venimos haciendo nosotros desde hace algún tiempo), con objeto de que el oncólogo conociera la posibilidad de solicitar la determinación del PEG mediante la plataforma comercial adecuada, con arreglo a sus preferencias, el porcentaje celular tumoral y al margen de la posible afectación axilar (caso de MammaPrint®), lo cual agilizaría el proceso de evaluación predictiva y pronóstica.
ConclusionesEn la mayoría de los casos presentados (76,92%), es posible la determinación del PEG (70-genes) incluso en lesiones menores de 1 cm, mediante la BAG1. Lesiones de mayor tamaño (mayores de 2 cm), sin embargo, no garantizan un mayor porcentaje de éxito.
La determinación del PEG a partir de la BAG1 facilita y acelera el proceso de evaluación predictiva/pronóstica, por lo que proponemos que, de manera rutinaria y ante el diagnóstico de tumores malignos infiltrantes de mama, conste el porcentaje de células tumorales en los informes anatomopatológicos, en especial en el contexto de equipos multidisciplinares en los que resulte frecuente la indicación de un PEG a partir de la BAG1, al margen de una posible afectación ganglionar axilar.
Confidencialidad de los datosLos autores declaran que han seguido los protocolos de su centro de trabajo sobre la publicación de datos de pacientes y han obtenido los permisos correspondientes.
FinanciaciónEste trabajo no ha recibido ningún tipo de financiación.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.