En este artículo se revisan los aspectos microbiológicos de la infección COVID-19 y se presentan las recomendaciones sobre los análisis que deben realizarse en casos forenses. En primer lugar se analizan las características taxonómicas del virus, su relación con la familia Coronaviridae y su estructura genética. Se presentan brevemente las características clínicas y patológicas de la infección COVID-19, así como las coinfecciones que pueden asociarse a este virus. En el diagnóstico de laboratorio se describen la PCR —técnica de elección en la fase aguda de la infección—, los estudios antigénicos y los estudios serológicos. Finalmente se detallan los principales objetivos para los estudios microbiológicos en fallecidos en relación con la pandemia COVID-19 y se describen los principales análisis microbiológicos post mortem a realizar en fallecidos en el ámbito forense. Los estudios microbiológicos deben estar dirigidos tanto a la detección del SARS-CoV-2 como a la de las coinfecciones, que también podrían contribuir a la causa de muerte.
We review the microbiological aspects of COVID-19 infection and present the microbiological studies that should be performed in forensic cases. We describe the taxonomic characteristics of the virus, its relationship with the Coronaviridae family and its genetic structure. We briefly present the clinical and pathological characteristics of COVID-19 infection, as well as the co-infections that could be associated with this virus. In the laboratory, PCR is a first-choice technique in the acute phase of the infection, together with antigen and serological studies. Finally, we describe the main objectives of microbiological studies in the deceased in relation to the COVID-19 pandemic, as well as the main post-mortem microbiological analysis to be carried out in the medico-legal context. The microbiological analysis should aim to detect both SARS-CoV-2 and coinfections, which may also contribute to the cause of death.
Coronavirus (CoV) es el nombre común con el que se conoce a los miembros de la subfamilia Orthocoranavirinae. Taxonómicamente pertenecen a la familia Coronaviridae, orden Nidovirales. Son virus ampliamente distribuidos que se descubrieron como patógenos humanos en la década de los sesenta. Son virus zoonóticos capaces de transmitirse entre animales y humanos, por lo que tienen un importante impacto económico y social. El nombre de coronavirus es consecuencia de la estructura que presentan estos virus al microscopio electrónico, puesto que parecen tener una especie de corona en su parte externa similar a la apariencia del sol1.
Existen cuatro géneros de coronavirus que se diferencian en la secuencia de sus proteínas: Alfacoronavirus, Betacoronavirus, Gammacoronavirus y Deltacoronavirus. Los dos primeros infectan a mamíferos y suelen tener a los murciélagos como reservorio, mientras que el género Gammacoronavirus incluye a todos los coronavirus aviares. Los Deltacoronavirus afectan tanto a mamíferos como a aves2.
Hay siete tipos de coronavirus que infectan al ser humano, y con anterioridad al brote de SARS-CoV del año 2003 se les consideraba responsables de infecciones respiratorias leves y autolimitadas. Los coronavirus humanos que se conocen en la actualidad son: coronavirus 229E (HCoV-229E), coronavirus OC43 (HCoV-OC43), SARS CoV, coronavirus NL63 (HCoV-NL63), coronavirus humano HKU1 (HCoV-HKU1), síndrome respiratorio por coronavirus de Oriente Medio (MERS-CoV) y Wuhan coronavirus o SARS-CoV-2. Cuatro de estos siete virus (HCoV-229E, HCoV-OC43, HCoV-NL63, HCoV-HKU1) afectan específicamente a la especie humana y causan entre el 15 y el 30% de las infecciones del tracto respiratorio cada año, con cuadros más graves en los recién nacidos, los ancianos y las personas con enfermedades subyacentes, y afectación principal del tracto respiratorio inferior3,4.
Los tres coronavirus restantes (SARS-CoV, MERS-CoV y el SARS-CoV-2 de 2019) son altamente patogénicos y causan infecciones severas del tracto respiratorio inferior provocando dificultad respiratoria aguda y manifestaciones extrapulmonares. El brote de SARS ocurrido en 2003 supuso un replanteamiento de la capacidad patogénica de estos virus y de su papel en las infecciones humanas5,6. Diez años después de este primer brote surgió otro en la península Arábiga (MERS), desde donde se propagó de manera esporádica al resto del mundo7-9. El nuevo coronavirus SARS-CoV-2 es el responsable de la pandemia actual y ha provocado una crisis sanitaria y económica sin precedentes en la edad moderna.
La secuencia completa del genoma del SARS-CoV-2, determinada mediante secuenciación masiva, estableció diferencias significativas con los coronavirus anteriores responsables de brotes (SARS y MERS). El análisis detallado de la secuencia10 permitió establecer un 96,2% de homología con un coronavirus del murciélago (Bat-SARS RaTG13), por lo que se incluyó, junto con este virus, dentro de un linaje distinto del subgénero del Sarbecovirus11. Aunque no puede establecerse con certeza la secuencia exacta de transmisión entre murciélago (principal reservorio) u otros mamíferos, parece innegable su origen zoonótico.
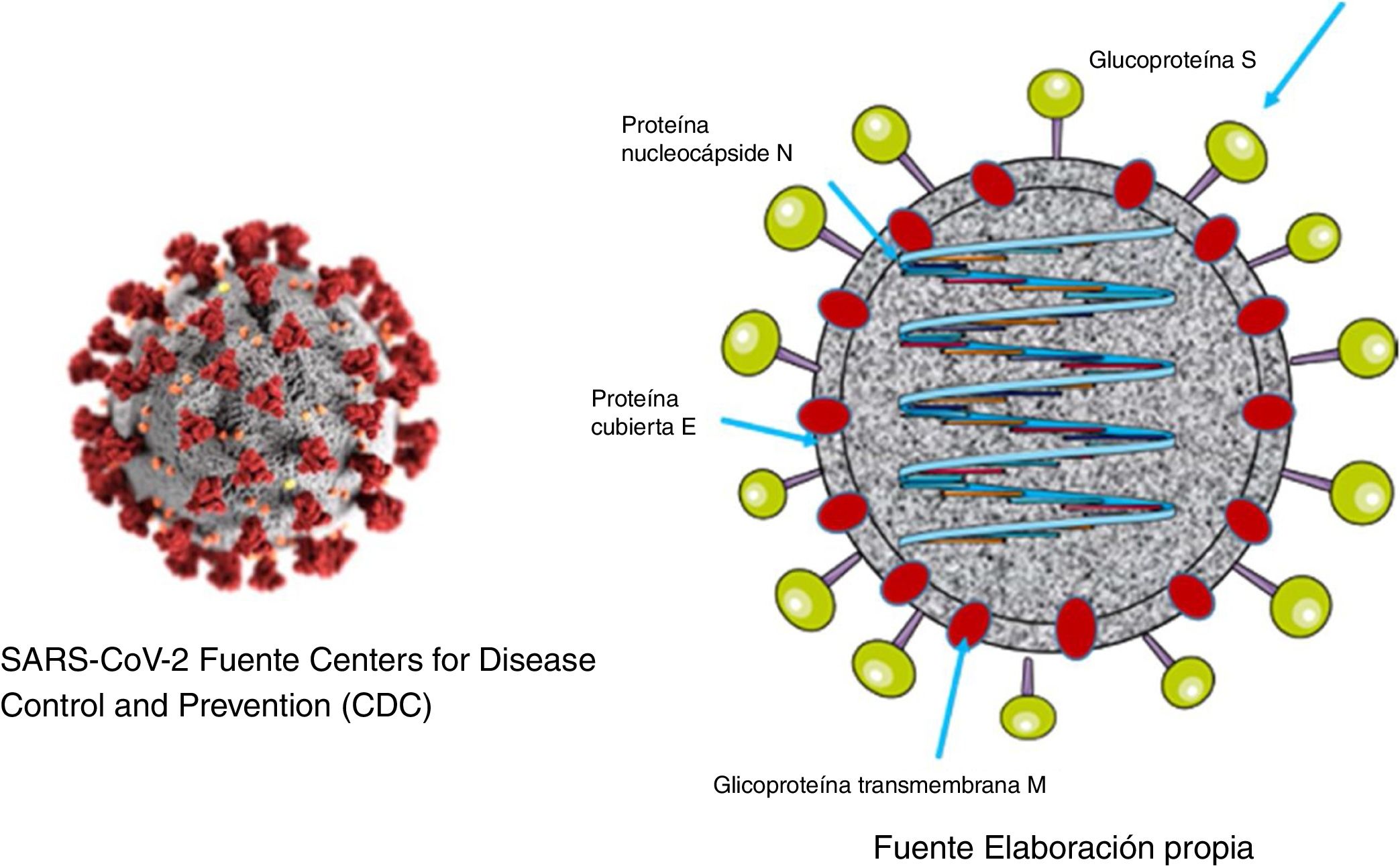
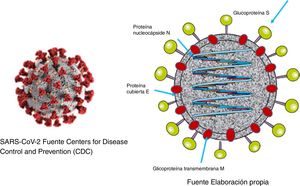
Al igual que el SARS y el MERS, el SARS-CoV-2 es un virus con envoltura de tamaño aproximado de 100-160nanómetros de diámetro con envuelta de bicapa lipídica. La envoltura presenta unas espículas que se proyectan hacia el exterior y que están formadas por trímeros de la glicoproteínaS.
El genoma viral es una secuencia sencilla de polaridad positiva. En el extremo 5’ del mismo se encuentran los genes que codifican para las proteínas reguladoras que van a dar lugar, entre otras, a la proteasa, a la ARN polimerasa ARN dependiente y a la helicasa. Los genes que codifican para las proteínas estructurales (S [spike protein], E [envelope], M [membrane] y N [nucleocapsid]) se sitúan en el extremo 3’12. Además, el virus codifica proteínas no estructurales1,13 (fig. 1).
La proteínaS forma estructuras que sobresalen de la envuelta del virus. Contiene el dominio de unión al receptor de las células que infecta (RBD), y por tanto es la proteína determinante del tropismo del virus. Consta de dos dominios diferentes: el dominio S1, responsable de la unión al receptor, y el dominio S2, responsable de la fusión con la membrana celular14.
La secuencia y el análisis de la proteínaS del SARS-CoV-2 indican que, al igual que sucedía con el SARS-CoV-1, utiliza la enzima convertidora de angiotensina2 (ECA2, más conocida por sus siglas del inglés angiotensin-converting enzyme2 [ACE2]) como receptor para la entrada en la célula huésped15. La ACE2 está situada en la superficie de una amplia variedad de células de mucosas, pulmones, arterias, intestino, etc., donde se encarga de convertir la angiotensinaI en angiotensinaII, aumentando así su acción vasoconstrictora. El virus emplea esta molécula para su internalización en la célula, donde los ribosomas celulares utilizarán el ARN viral como ARN mensajero sintetizando a partir de él las proteínas del virus. Esto, junto con la replicasa viral, permitirá hacer múltiples copias del virus que favorecerán su diseminación.
La proteína N está en el interior del virión asociada al ARN viral y juega un importante papel en la replicación del virus y en el ensamblaje de nuevas partículas virales. La proteínaM es la más abundante y la responsable de la forma final del virión. La proteínaE es de pequeño tamaño y se encuentra en pequeñas cantidades en la cubierta. Las proteínas no estructurales desempeñan importantes funciones específicas en el proceso de replicación del virus.
El hecho de que SARS-CoV-2 haya llegado a los seres humanos a partir de un origen animal implica que la probabilidad de futuros brotes con virus similares es alta, ya que este tipo de virus sigue circulando en la población animal. Es por tanto prioritario conocer las características (transmisibilidad, patogenicidad, tasa evolutiva, etc.) que van a condicionar su propagación y determinar la extensión de la pandemia. También es de especial interés conocer si el SARS-CoV-2 puede exhibir estacionalidad como la mayor parte de los coronavirus que infectan a humanos.
La enfermedad COVID-19Presentación clínicaLa unión del SARS-CoV-2 al receptor ACE2, localizado, entre otras, en las células alveolares tipoI yII, puede causar lesiones a nivel de estas células, particularmente en las que tienen una elevada cantidad del receptor. Este daño puede desencadenar una liberación masiva de citoquinas, lo que, unido a la capacidad del virus de evadir la respuesta inmunitaria, juega un papel importante en la patogenia y la severidad de la enfermedad16.
Las manifestaciones clínicas en los casos de COVID-19 son muy variables, desde casos asintomáticos hasta shock séptico, neumonía y fallo multiorgánico. La enfermedad se clasifica en diferentes tipos según la gravedad. Los síntomas más comunes son fiebre, fatiga, tos seca y diarrea. Tras un periodo de incubación de 5 a 14días, el 80% de los casos aparecen como un cuadro leve con síntomas locales en vías respiratorias altas acompañado de síntomas inespecíficos como fiebre, astenia, anosmia, ageusia y mialgias. Algunos pacientes pueden deteriorarse rápidamente y evolucionar a un cuadro más grave con disnea, taquipnea, disminución de la saturación e infiltrados alveolares bilaterales, que puede requerir ingreso hospitalario. Un porcentaje variable del total de casos pueden a su vez requerir ingreso en unidades de cuidados intensivos (UCI) por neumonía severa, síndrome de distrés respiratorio severo, shock séptico o fallo multiorgánico17. Estos pacientes presentan disnea severa, taquipnea (>30/min), SpO2<93%, PaO2/FiO2<300 y/o un aumento del 50% de los infiltrados pulmonares en 24 o 48h. Las complicaciones extrapulmonares más frecuentes son cardiovasculares, hepáticas y renales. En los parámetros de laboratorio son característicos la linfopenia y niveles muy elevados de dímero-d, ferritina, urea y creatinina. El fallo multiorgánico es consecuencia de una respuesta excesiva del propio sistema inmune del paciente. El CDC chino sugiere que la mortalidad en pacientes críticos puede ser hasta del 49%, principalmente ante la presencia de comorbilidades como diabetes, enfermedad respiratoria, enfermedad cardiovascular, hipertensión y complicaciones oncológicas18.
La mortalidad general en la serie de Wuhan fue del 5,8%16, aunque diferentes estudios cuestionan esta cifra. En cualquier caso, las cifras reales de mortalidad son difíciles de calcular por la elevada heterogeneidad de algunos datos.
PatologíaPulmonesMacroscopia: aumentados de tamaño, con pleura opaca. La superficie de corte muestra consolidación y/o edema. En caso de infección bacteriana secundaria puede agregarse un exudado purulento19.
Microscopia: en fases tempranas de la infección se observa edema intraalveolar, hiperplasia de neumocitos de tipo II, células gigantes, inflamación focal y generalmente sin presencia de membranas hialinas; en etapas más avanzadas de la infección se observa daño alveolar difuso, con membranas hialinas, exudados de fibrina, infiltrado linfocitario intersticial (neumonitis), presencia de células gigantes y atipia de neumocitos descamados en el espacio alveolar y con prominencia de nucléolos. Los linfocitos corresponden al tipoT (principalmente CD4, y menos CD8). Recientemente se han detectado partículas virales en el pulmón de pacientes afectados por la COVID-1920, si bien otros autores no las han detectado21-24. Algunos describen la presencia de microtrombos en los capilares alveolares. Si hay confección bacteriana, al cuadro histológico se pueden añadir los abscesos20.
CorazónMacroscopia: esta puede mostrar áreas de hemorragia y signos de infarto.
Microscopia: se puede observar daño endotelial, con acumulación de células inflamatorias en relación con las células endoteliales (endotelitis linfocitaria) y apoptosis en el miocardio. También pueden encontrarse signos histológicos de miocarditis linfocitaria. El virus se ha detectado en el citoplasma de las células endoteliales afectadas.
Otros órganosSe ha descrito el desarrollo de isquemia (macroscópica) y/o endotelitis linfocitaria (microscópica) en colon, intestino delgado, mesenterio, riñones e hígado25.
Muy recientemente se han descrito casos de encefalitis en enfermos de COVID-19, incluyendo encefalitis hemorrágica necrosante26,27.
Coinfecciones en la enfermedad COVID-19La neumonía es una de las principales causas de ingreso hospitalario. Este cuadro clínico puede ser producido tanto por bacterias como por virus, hongos y parásitos. Lo habitual es que un único microorganismo sea responsable del cuadro, pero no siempre es así, y gracias a los métodos de diagnóstico actuales se ha podido comprobar que en muchos casos hay más de un microorganismo presente en estas patologías y que la presencia de varios agrava los cuadros clínicos, complicando la recuperación de los pacientes. Este hecho es aplicable no solo a la neumonía tradicional, sino también a la ocasionada por la COVID-19.
Coinfecciones bacterianas y fúngicasLos pacientes ingresados, y especialmente aquellos en las UCI, tienen un alto riesgo de desarrollar infecciones nosocomiales o neumonías asociadas a ventilación mecánica, además de coinfecciones secundarias no relacionadas con su presentación respiratoria (p.ej., infección urinaria). Las infecciones bacterianas y fúngicas son complicaciones comunes de la neumonía viral, especialmente en pacientes críticos. Sin embargo, en pacientes con COVID-19 los estudios realizados sobre este tipo de infecciones son escasos, y en la mayoría de los trabajos publicados no se incluyen en el análisis de pronóstico, aun cuando se ha encontrado infección secundaria en el 50% de los pacientes con COVID-19 no supervivientes28.
La revisión de la literatura existente indica que un 8% del total de los pacientes ingresados han sufrido coinfecciones fúngicas o bacterianas. La presentación más habitual de estas coinfecciones es la bacteriemia, y entre los microorganismos encontrados aparecen tanto bacterias grampositivas (Staphylococcus aureus) como gramnegativas (Acinetobacter baumannii, Klebsiella pneumoniae); en muestras respiratorias se detectan también bacterias atípicas (Legionella sp, Chlamydophila pneumoniae)29.
La sobreinfección por Aspergillus también se ha encontrado en varios sujetos con distrés respiratorio e indica la necesidad de que los médicos que atienden a pacientes con esta patología, aunque sea producida por la COVID-19, consideren la posibilidad de aspergilosis pulmonar invasiva y, en consecuencia, procesar las muestras respiratorias con análisis específicos que permitan detectar la coinfección30.
Aunque puede ser difícil distinguir la infección bacteriana o fúngica y la neumonía viral existente en función de la apariencia clínica y radiológica, el examen microbiológico —especialmente el cultivo de esputo— puede ofrecer una información muy valiosa siempre que el personal de laboratorio de análisis mantenga las medidas de protección adecuadas para el manejo de estas muestras de alto potencial infeccioso.
El porcentaje de pacientes con COVID-19 en los que se ha detectado este tipo de coinfecciones es pequeño en comparación con las que se observaron con otros coronavirus. Sin embargo, esto se podría explicar porque durante esta pandemia la administración de antibióticos ha sido elevada y en muchos casos ha implicado a antibióticos de amplio espectro; no obstante, la identificación de microorganismos multirresistentes no ha sido frecuente.
Una elevada proporción de las coinfecciones bacterianas que aparecen en la literatura sobre coronavirus está asociada a la asistencia sanitaria, incluyendo las bacteriemias y la infección asociada a ventilación mecánica. En estas condiciones deben extremarse las medidas de control de la infección y la prescripción de antimicrobianos, dado que los antibióticos juegan un papel importante en el manejo de la COVID-19. La terapia combinada con azitromicina e hidroxicloroquina se ha utilizado de manera empírica para tratar a los pacientes con neumonía.
Coinfecciones viralesLos síntomas ya mencionados para catalogar a un paciente como posible COVID-19 (fiebre, astenia, tos seca, mialgia, dolor de cabeza, mareos, dolor abdominal, diarrea, náuseas y vómitos) son comunes a muchas infecciones respiratorias agudas, incluidas las virales31,32. Aunque las imágenes radiológicas pueden ser más orientativas, no sirven para descartar con total certeza la presencia de otros virus en los pacientes con sospecha de infección por SARS-CoV-2.
En este sentido se han realizado diversos estudios para detectar coinfecciones virales en pacientes con clínica COVID. Así, Lin et al.33, en un trabajo que incluye 186 individuos con clínica compatible con COVID-19, encontraron en 6pacientes (3,2%) coinfecciones con otros virus respiratorios y vieron que en otros 18pacientes uno o varios virus respiratorios distintos al SARS-CoV-2 (rinovirus, virus respiratorio sincitial, adenovirus, influenzaA yB, parainfluenza2 y3, coronavirus HKU1 y metapneumovirus) eran los responsables del cuadro clínico observado.
Datos similares se han encontrado en otras series realizadas en distintos países. En Estados Unidos, en un estudio que incluye más de 5.000 pacientes del área de Nueva York, se observó una tasa de coinfección por virus respiratorios del 2,1%34.
Hay descritas coinfecciones del SARS-CoV-2 con otros coronavirus35, con metapneumovirus36 y con virus influenza37,38. Todos estos datos destacan la importancia de mantener la sospecha de posible coinfección y de no excluir ningún virus respiratorio (SARS-CoV-2 incluido) cuando se detecte la presencia de otro. Este hecho es de especial importancia, dado que se desconoce la posible estacionalidad del coronavirus responsable de la actual pandemia.
No está claro el papel que el estado inmunitario del huésped puede jugar en el desarrollo de la infección por SARS-CoV-2, y dado que en el mundo existen más de 37millones de personas infectadas por el VIH, es importante conocer cómo esta infección puede afectar al desarrollo de la COVID-19. Además, el uso de antirretrovirales se ha propuesto como terapia alternativa, y en la actualidad el remdesivir se presenta como una opción de tratamiento prometedora. Junto con algunos datos provenientes de China39,40, un estudio llevado a cabo en nuestro país41 parece indicar que la evolución de estos pacientes es similar a la de la población general, si bien es cierto que el número de casos no tiene el tamaño suficiente como para considerar estas conclusiones como definitivas.
Las comorbilidades agravan el pronóstico de la COVID-19. Los pacientes con edad avanzada, diabetes, complicaciones respiratorias graves, hipertensión, enfermedades cardiovasculares, recuento de leucocitos/linfocitos en sangre alterados, etc., tienen mayor riesgo de malos resultados clínicos. Las coinfecciones, tanto víricas como bacterianas y fúngicas, son otro factor más que tiene que considerarse como riesgo añadido.
Diagnóstico microbiológico del SARS-CoV-2El diagnóstico rápido de laboratorio de COVID-19 es relevante para identificar, aislar y tratar rápidamente a los pacientes infectados con SARS-CoV-2, para limitar la transmisión del virus, así como para la descongestión de las urgencias o la valoración del estado de los clínicos expuestos al virus.
La muestra de elección para este diagnóstico es el hisopado nasofaríngeo (HNF), ya que la carga viral es superior en las fosas nasales que en la orofaringe. La técnica más útil es la detección de ácidos nucleicos de SARS-CoV-2 en muestra de exudado nasofaríngeo, ya que el virus puede detectarse desde las primeras fases de la infección. Es fundamental una correcta toma de la muestra para evitar falsos negativos; para ello se debe inclinar hacia atrás la cabeza del paciente, introducir el hisopo a través de la fosa nasal hasta la nasofaringe, rotar enérgicamente el hisopo y luego retirar. También se pueden utilizar muestras faríngeas, de aspirado endotraqueal, broncoaspirado y lavado broncoalveolar42.
La PCR a tiempo real y otros análisis molecularesLas técnicas de amplificación de ácidos nucleicos, y particularmente la RT-PCR (reacción en cadena de la polimerasa-transcripción inversa, en adelante PCR), se consideran de elección para la fase aguda de la enfermedad COVID-19. En los pacientes con síntomas de infección estas técnicas tienen un elevado valor predictivo positivo (VPP).
La mayoría de los ensayos en uso son comerciales, aunque algunos laboratorios de referencia utilizan test in house. Entre las regiones que detectan estos ensayos se hallan la ORF1ab y las regiones codificantes de las proteínasS yN. La selección de dichas regiones se ha hecho con el fin de maximizar la especificidad de la amplificación para evitar reacciones cruzadas con otros coronavirus relacionados. Algunos ensayos incluyen como cribado la detección del gen codificante de la proteínaE, que es común con otros coronavirus del grupo SARS, por lo que daría resultados positivos con otros coronavirus relacionados como SARS-CoV de 2003 y SARS-CoV de murciélago, virus ambos que no circulan, y en el caso del segundo, que no infecta al ser humano. Algunos de estos kits analizan dos o más de las mencionadas regiones en formato multiplex, esto es, en la misma reacción, utilizando un control que se añade antes de la extracción para verificar la eficiencia del proceso completo —extracción y amplificación— y la ausencia de inhibidores en la reacción.
Entre las PCR comerciales destacan las comercializadas por Altona, Thermofisher, Seegene, Genesig, Biomerieux, Bio-Speedy, Bioeksen Turkey, y las fabricadas en España, como las de Vircell, Genomica o Genetics PCR Solution. También existen otros formatos, como la PCR-hibridación en microarray, aunque la PCR a tiempo real es la más utilizada. Datos no publicados sugieren una diferente sensibilidad de las técnicas, aunque, en general, el límite de detección suele ser de 10 copias de genoma viral por reacción.
La PCR ha permitido conocer que en los primeros días con síntomas —y probablemente también en la fase presindrómica— los infectados tienen una alta carga viral (entre 104 y 108 copias de genoma/ml por muestra nasofaríngea o de saliva)43. Cuando el curso de la infección es leve, el pico de carga viral aparece durante los primeros 5-6días tras el inicio de los síntomas, desapareciendo alrededor del décimo día. Aunque en estas muestras puede haber virus después de ese día, la carga viral es entre 100-1.000 veces menor, lo que indicaría una muy baja transmisibilidad del virus en ese período. Por el contrario, en los infectados con infección grave la carga viral es de hasta 60 veces mayor que en los que cursan con síntomas leves44 y la excreción viral puede ser más duradera. En un estudio se pone de manifiesto que en pacientes fallecidos por infección grave por COVID-19 la PCR seguía siendo positiva hasta el fallecimiento, no negativizándose en ningún momento45. En cuanto a la transmisibilidad del virus, se piensa que en los casos más leves comenzaría uno o dos días antes del inicio de los síntomas y duraría unos 5 o 6días, mientras que en los casos más graves la transmisión sería más larga e intensa debido a la mayor carga viral43, e incluso nuevos estudios describen la posibilidad de alargar hasta 6días el periodo asintomático en el que hay transmisibilidad del virus46.
La interpretación de la PCR se debe hacer siempre dentro del contexto clínico, especialmente en los casos negativos. Los falsos negativos podrían deberse principalmente a una toma inadecuada de la muestra, al transporte inadecuado o a baja carga viral en el momento de la toma, que haga que esta se halle debajo del límite de sensibilidad de la técnica.
Técnicas de detección de antígenoLa utilización de técnicas de inmunocromatografía para la detección de antígeno de SARS-CoV-2 en muestras respiratorias (principalmente en exudado nasofaríngeo) podría ser el primer paso en la detección del patógeno en pacientes sintomáticos e incluso asintomáticos en los primeros momentos de la infección, puesto que la carga viral del virus es elevada en esos momentos. Además, la simplicidad de la técnica, así como el no utilizar equipos específicos, la convertían en un candidato ideal43.
Sin embargo, en el mercado mundial no hay casi ningún kit de estas características, tan solo el BIOEASY™ Diagnostic Kit for Novel Coronavirus (COVID-19) Ag Test Kit, que fue comprado por el Ministerio de Sanidad y Consumo y evaluado antes de su uso por las autoridades sanitarias para su validación. Tanto el primer kit estudiado —una inmunocromatografía convencional con oro coloidal de lectura visual simple— como la segunda versión remitida, que era una inmunofluorescencia rápida, mostraron cifras de sensibilidad muy bajas (<30%), que en ningún caso permitirían su utilización como herramienta diagnóstica útil42.
Técnicas de detección de anticuerposPara el estudio de los anticuerpos existen diferentes test con características de sensibilidad y especificidad variables, y en algunos casos con validaciones limitadas por el carácter urgente de la pandemia. En estos momentos existen evaluaciones independientes de algunos de ellos con el fin de unificar los criterios, así como validar clínicamente estos test42. En España, concretamente la SEIMC y el CNM están recabando información para disponer de datos fidedignos a nivel nacional sobre la sensibilidad y la especificidad de estos kits, datos estos que en un futuro próximo estarán disponibles47.
Para el estudio serológico se emplean métodos para la detección de la respuesta humoral frente a la proteínaS y la proteínaN, puesto que ambas han mostrado un elevado carácter inmunogénico. La proteínaS se une a un receptor celular, la enzima convertidora de angiotensina2 (ACE2), tanto en el caso del SARS-CoV-1 como en el caso del SARS-CoV-2. Algunos anticuerpos anti-S, incluyendo aquellos frente al RBD, tienen actividad neutralizante, pero falta por determinar su proporción relativa real frente al total de anticuerpos anti-SARS-CoV-2 durante la infección por el virus48.
Una vez producida la infección y tras la aparición de los síntomas, a los 5días aproximadamente se produce la aparición de IgM e IgA; con posterioridad, a los 14días aparece la IgG (10-18días)45,50. La seroconversión para anticuerpos totales se produce en el día1147,49.
Algunos test que combinan IgM e IgG pueden proporcionar resultados poco definitorios, puesto que puede existir una reactividad cruzada potencial con otros anticuerpos frente a otros coronavirus, así como la frecuente baja especificidad de la IgM50.
Dado que se ha detectado una respuesta variable a ambas inmunoglobulinas en el curso de la enfermedad, un resultado negativo de IgM y de IgG no excluiría que el paciente esté infectado por SARS-CoV-2, especialmente si se trata de inmunodeprimidos. Por tanto, aunque el estudio de los anticuerpos es una herramienta útil para estimar la proporción de individuos que han estado expuestos al SARS-CoV-2, es de menor utilidad para el diagnóstico y la valoración clínica de un paciente, salvo en circunstancias muy concretas. Un ejemplo de esto sería la detección de pacientes infectados con PCR-negativa, en particular en los que presentan resultados con carga viral baja por debajo del límite de detección de la PCR, o cuando no sea posible tomar muestras del tracto respiratorio inferior51.
Inmunocromatografíaa. Point of care (POC)Conocidas en muchos casos como pruebas rápidas, puesto que la lectura del resultado se obtiene en 10-15min, en realidad sirven para detectar la respuesta de anticuerpos IgM e IgG, por lo que desde el momento de la infección deberá haber transcurrido un periodo de tiempo suficiente para que se produzcan los citados anticuerpos47. En la actualidad en España hay más de 60kits con marcado CE para la detección de anticuerpos. Una de sus ventajas es que se pueden emplear en sangre periférica (incluso por punción capilar), no solo en suero o plasma. Sin embargo, existen otras técnicas, de ELISA o de quimioluminiscencia, que son más sensibles y específicas.
Una parte de los kits en el mercado detectan la presencia de anticuerpos IgG e IgM totales frente a diferentes antígenos del virus, mientras que otra parte detectan de forma separada la presencia de anticuerpos IgM e IgG.
El kit comprado en primera instancia por el Ministerio de Sanidad y Consumo para realizar el cribado detectaba de forma conjunta IgM e IgG frente a la proteínaS (COVID-19 IgG/IgM Rapid Test Cassette, Zhejiang Orient Gene Biotech Co Ltd, Huzhou, Zhejiang, China)52 a partir de muestras de suero o plasma.
Métodos automatizadosRecientemente se han puesto en marcha diversas técnicas para la detección rápida de SARS-CoV-2 mediante detección de anticuerpos automatizada (menos de una hora). Algunas de estas pruebas ya tienen la aprobación de la FDA y se espera en un futuro inmediato el marcado CE.
SARS-CoV-2 IgG Abbott. El inmunoanálisis quimioluminiscente de micropartículas (CMIA) que detecta anticuerpos IgG frente a la proteínaN de la nucleocápside en suero y plasma es un sistema automatizado en 2etapas. En la primera fase las micropartículas paramagnéticas recubiertas de antígeno del SARS-CoV-2 se unen a los anticuerpos IgG presentes en la muestra, se lava la mezcla y se añade un conjugado de anticuerpos anti-IgG humana marcado con acridinio y se incuba. Posteriormente, tras la adición de solución preactivadora y activadora, se procede a la lectura de la reacción quimioluminiscente resultante.
LIAISON SARS CoV-2 IgG. En este caso es un ensayo cualitativo de tecnología quimioluminiscente (CLIA) que detecta anticuerpos IgG frente a los dominios S1 y S2 de la proteínaS del SARS-CoV-2.
Otros laboratorios están desarrollando nuevos ensayos automatizados para el estudio de anticuerpos IgG frente al SARS-CoV-2.
MAGLUMI 2019-nCoV IgG e IgM. Es un ensayo cuantitativo de tecnología quimioluminiscente (CLIA) que detecta anticuerpos IgG e IgM frente a la proteínaS y a la proteínaN del virus53.
Existen otros ensayos disponibles en el mercado con presentación monotest en los que se incluyen todos los reactivos necesarios, como el COVID-19 VIRCLIA® IgM +IgA MONOTEST, que además detecta la presencia de IgA.
Métodos semiautomatizadosExisten pocos kits comercializados para el estudio de IgM e IgG frente al SARS-CoV-2, y la mayor parte de ellos son ELISA (enzyme-linked immunoassay) para realizar en microplacas de 96 pocillos tapizados con proteínaS del virus y posterior revelado colorimétrico (COVID-19 Human IgM IgG Assay Kit Abnova, COVID-19 ELISA IgG e IgM +IgA Vircell).
Estudios epidemiológicos para conocer el virusEstudio de sus propiedades biológicas: cultivo celularEl aislamiento del SARS-CoV-2 en cultivos celulares se ha realizado desde el inicio del brote en China y es de enorme interés para poder determinar si un determinado valor de ciclo umbral o Ct obtenido mediante métodos de amplificación genómica PCR correlaciona con virus capaces de transmitirse entre el ser humano. La PCR positiva en muestra nasofaríngea puede durar hasta 22días o más tras el inicio de los síntomas54,55. Sin embargo, el virus infectivo (determinado por aislamiento en cultivo) solamente es detectable hasta los 8días tras la aparición de síntomas en pacientes leves55. Se ha comprobado que la carga viral mínima necesaria para aislar el virus a partir de saliva es de 106 copias de ARN/ml. El contagio se ha estimado que se produce entre 2-8días antes de los síntomas y 7días después54.
Las principales aplicaciones del aislamiento en cultivos celulares de SARS-CoV-2 incluyen: a)investigación de antivirales mediante la capacidad experimental de que las nuevas sustancias puedan inhibir el crecimiento del virus; b)investigación de la patogenia viral mediante el estudio de las cadenas de transmisión en el ser humano, la gravedad de la infección, la carga viral que se deriva de dichas infecciones y la capacidad de infección de los diferentes órganos, y c)investigación de la estabilidad del virus demostrando la capacidad de mantener la infectividad en superficies o en determinadas condiciones ambientales, temperatura y luz (https://www.cdc.gov/coronavirus/2019-ncov/php/grows-virus-cell-culture.html).
Estudio de sus características genómicas: secuenciación masivaLa secuenciación genética es una técnica que se desarrolla en prácticamente todos los laboratorios de investigación biológica y biomédica. El método de referencia es la secuenciación Sanger, que mediante las técnicas fluorescentes y la introducción de la electroforesis capilar permitió automatizar el proceso de secuenciación y que se aplica al conocimiento genómico de los virus, y concretamente a los virus respiratorios. A lo largo de los años han ido surgiendo nuevas tecnologías que permiten obtener millones de reacciones de secuenciación de forma simultánea y de manera muy rápida, utilizando las tecnologías Illumina e IonTorrent, y se han considerado parte de la segunda generación de secuenciación genómica. La tercera generación de técnicas la componen métodos que son capaces de secuenciar moléculas de ADN sin amplificación previa, desarrolladas por Pacific BioSciences y Oxford Nanopore56,57.
La secuenciación del genoma del SARS-CoV-2 supone un enorme desafío para llegar a conocer su genoma y su variabilidad. De esta manera se ha realizado desde el inicio de la pandemia un trabajo de secuenciación sin precedentes para obtener secuencias de manera muy rápida que se pueden analizar en tiempos muy cercanos a su detección y diagnóstico en los pacientes. El objetivo de los análisis genómicos es el de generar conocimiento para la investigación de fármacos para un tratamiento y de vacunas para una prevención. Los datos de secuencias también generan resultados que permiten evaluar los kits diagnósticos, trazar el origen del virus, estudiar las rutas de transmisión e identificar las opciones para una intervención estratégica. Se ha publicado en la plataforma protocols.io (https://www.protocols.io/) un protocolo para la secuenciación masiva del SARS-CoV-2 utilizando la tecnología Oxford Nanopore, que se va actualizando por los diferentes usuarios (labworm.com/tool/protocolsio)58. Otras aproximaciones se realizan mediante tecnologías Illumina e IonTorrent.
Actualmente se encuentran almacenadas en la base de datos GISAID59 un total de 16.000 secuencias genómicas. Los análisis de las secuencias de los diferentes países se analizan utilizando como base la cepa considerada de referencia, Wuhan-Hu-1/2019, secuenciada el 10 de enero 2020 y depositada con número de acceso MN908947 en la base de secuencias GenBank, y EPI-ISL-402119 en la base de secuencias GISAID. Esta cepa, junto con otras coetáneas, sirve como base para los análisis filogenéticos. Además, se utilizan como base para los estudios estructurales de las proteínas asociadas a las nuevas secuencias. Se ha calculado con datos de secuencias españolas que la evolución del SARS-CoV-2 presenta un rango de 1,08×10−3 a 1,87×10−3 sustituciones por sitio por año60.
Funcionamiento de la Red Nacional de Vigilancia Epidemiológica en relación con la COVID-19La vigilancia epidemiológica de enfermedades transmisibles es una de las actividades que tradicionalmente ha ido ligada a la salud pública. Dichas enfermedades se estudian en un departamento cuya misión es la de mejorar el conocimiento de estas y de los brotes de cualquier etiología mediante la vigilancia y la investigación epidemiológica y de salud pública. El objetivo final es proporcionar información de calidad para guiar el control y la prevención de las enfermedades transmisibles, así como orientar el diseño, la implementación y la evaluación de políticas sanitarias.
La vigilancia se estructura gracias a la actividad de la Red Nacional de Vigilancia Epidemiológica (RENAVE), que gestiona el Centro Nacional de Epidemiología en el Instituto de Salud CarlosIII (ISCIII). Se trata de una estructura descentralizada que refleja la organización autonómica de nuestro país. La integran profesionales y técnicos de salud pública de los departamentos de salud local, autonómica y estatal. La Red da respuesta a las necesidades de información de las autoridades de salud y de todos los profesionales que, desde distintos ámbitos y responsabilidades, necesitan conocer la presentación, los patrones de riesgo y la distribución de las enfermedades transmisibles en la población. La RENAVE articula la vigilancia integrando: a)la notificación de casos y b)la investigación epidemiológica de dichos casos de enfermedades transmisibles.
En la actual pandemia, el Ministerio de Sanidad, a través del Centro de Coordinación de Alertas y Emergencias Sanitarias (CCAES), se encuentra en permanente contacto con las comunidades autónomas (CCAA) a través de la Ponencia de Alertas y Planes de Preparación y Respuesta. Además, intervienen el Centro Nacional de Epidemiología (CNE) y el Centro Nacional de Microbiología (CNM) en el ISCIII. La coordinación con los organismos internacionales (OMS, Centro de Control de Enfermedades Europeo [ECDC] y Comisión Europea [UEC]) es un punto básico para evaluar los riesgos de las situaciones y coordinar las medidas de respuesta.
Desde el inicio de la crisis se ha realizado un protocolo de actuación ante la aparición de casos en España que se ha ido actualizando a lo largo de 2020. Este protocolo recoge el Formulario de Notificación de casos por el nuevo coronavirus, cuya información se envía desde las CCAA al CNE y al CCAES, así como las indicaciones para el envío de muestras al CNM.
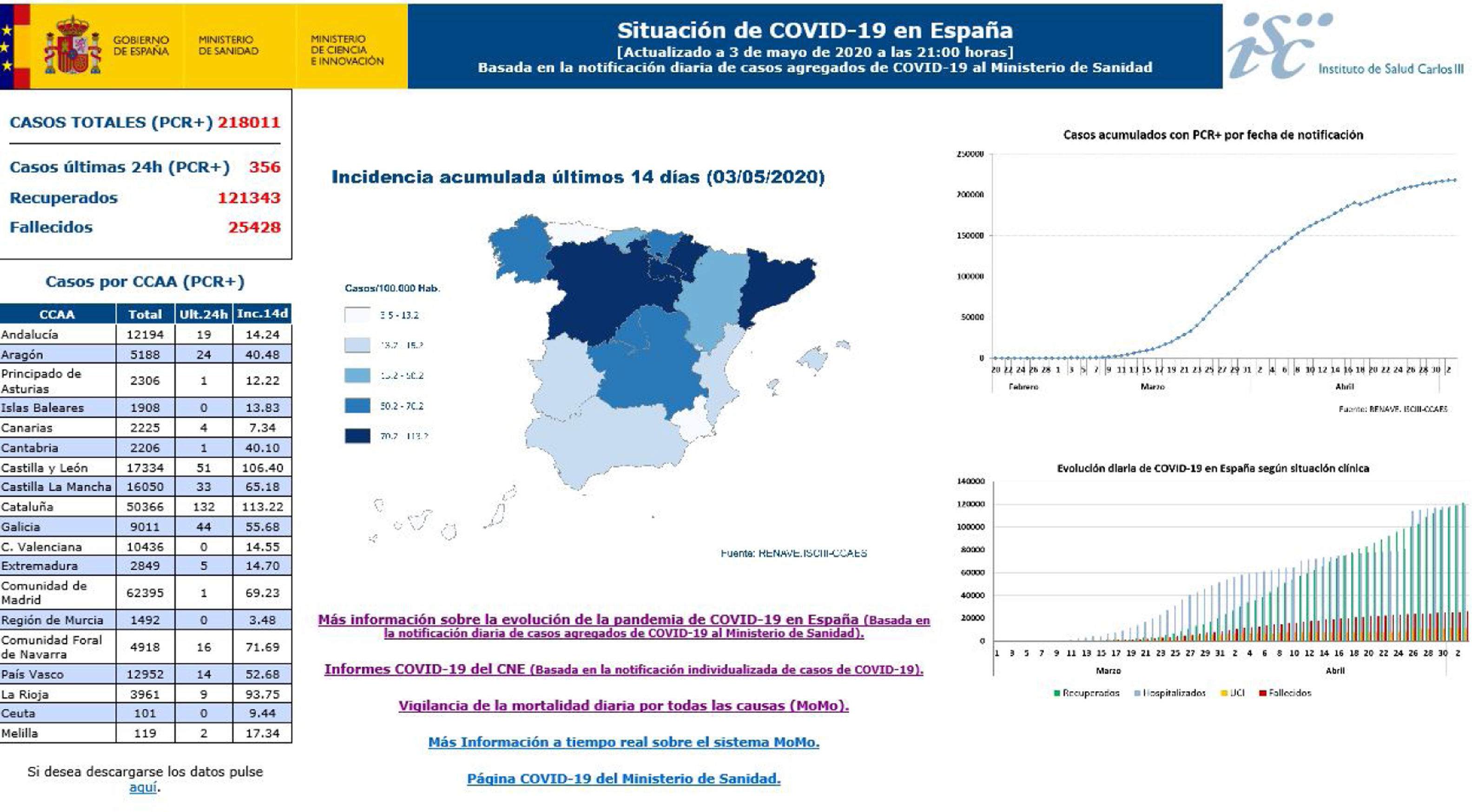
Hasta el 30 de abril se han realizado 27 informes de situación en donde quedan recogidos los datos de las CCAA, que notifican diariamente al Ministerio de Sanidad las cifras de casos confirmados acumulados de COVID-19: total de casos, casos en profesionales sanitarios, hospitalizaciones, ingresos en UCI, fallecidos y casos recuperados. Las CCAA completan una encuesta individualizada para cada uno de dichos casos que incluye información clínico-epidemiológica consensuada y aprobada por la Ponencia de Alertas y Planes de Preparación y Respuesta y la RENAVE, y la notifican mediante la plataforma informática SiViEs (Sistema para la Vigilancia en España), que gestiona el CNE. Desde el principio de la pandemia de COVID-19 la vigilancia de los casos de esta enfermedad en España se basa en la notificación universal de todos los casos confirmados de COVID-19 que se identifican en cada CCAA. Los datos agregados de COVID-19 (fig. 2) se pueden consultar en https://covid19.isciii.es/
Es importante señalar que la vigilancia de los excesos de mortalidad por todas las causas se realiza mediante un sistema de vigilancia llamado MoMo, que registra los excesos de mortalidad por todas las causas, evaluando el impacto en la mortalidad de la población de todo evento que pueda suponer una amenaza para la salud pública. Sus resultados pueden apoyar las evaluaciones de riesgo de dichos eventos y contribuir a guiar adecuadamente la respuesta de salud pública y el desarrollo de políticas de control. Se pueden consultar en Informes MoMo 2020, en https://cnecovid.isciii.es/
Estudios microbiológicos post mortem con referencia al SARS-CoV-2: aplicabilidadInterés y objetivos de la detección post mortem del SARS-CoV-2Los principales supuestos medicolegales en los que es de interés la detección de COVID-19 como técnica diagnóstica en muestras forenses procedentes de autopsia son: a)casos sospechosos (no confirmados) de COVID-19 en muertes naturales; b)muertes súbitas-inesperadas de adultos o niños en las que sea necesario descartar el COVID-19 como posible causa de muerte; c)denuncias por mala praxis en las que la infección pudiera estar involucrada en la causa de muerte; d)muertes violentas en las que las circunstancias hagan conveniente esta determinación (p. ej., suicidio, posibilidad de crimen encubierto), y e)cualquier otra situación en que el forense lo considere necesario.
Además de las cuestiones de importancia medicolegal, el médico forense se enfrenta a una enfermedad infecciosa nueva. Como tal, está en una posición óptima para:
- -
Identificar la fisiopatología y el mecanismo de acción de este nuevo agente infeccioso.
- -
Identificar patrones de la infección en las distintas poblaciones de riesgo y etarias.
- -
Contribuir al estudio epidemiológico de la pandemia.
- -
Optimización de las técnicas de diagnóstico post mortem.
Hasta el momento, la experiencia práctica en la detección post mortem de la infección por COVID-19 es limitada debido al bajo número de autopsias realizadas en estos pacientes en los países afectados24. En los escasos artículos publicados en los que se recogen resultados de autopsias de COVID-19, estas son dirigidas, limitadas a determinados órganos y/o tejidos, o mínimamente invasivas, solo refiriendo autopsias completas en algunos casos20,24,25,61,62. Salvo excepciones, en la mayoría de dichas publicaciones no se aportan muchos detalles sobre las investigaciones microbiológicas realizadas. En la mayoría de estos estudios la detección de este patógeno se había realizado ante mortem con las muestras usuales en clínica (HNF o hisopo faríngeo), o bien no se especifica cuándo ni a partir de qué muestra se ha realizado este diagnóstico. En las publicaciones que indican que el diagnóstico microbiológico se ha realizado post mortem la técnica usada es la PCR21,63,64. En estos casos, la muestra analizada ha sido el hisopo faríngeo64, empleando solo en algunos casos muestras de vías bajas respiratorias, tales como hisopado de parénquima pulmonar62 o necropsias de pulmón fijadas en parafina21. Solo excepcionalmente se utiliza la microscopia electrónica para verificar la presencia de partículas de inclusión viral en pulmón20 o en riñón61.
En la bibliografía consultada no se ha encontrado ningún estudio que utilice en paralelo más de un tipo de muestra para el análisis microbiológico, por lo que no se puede establecer basándose en ellos ninguna recomendación respecto a qué muestra puede ser más recomendable emplear en cadáveres.
Recomendaciones para la determinación de COVID-19 en cadáveresTécnicas molecularesEn España los protocolos forenses para la investigación de la COVID-1965 indican la recogida de muestras previa a la autopsia, concretamente de HNF e hisopo faríngeo. Estas serían las muestras más adecuadas si se pretende realizar un cribado de COVID-19 para decidir sobre la realización de la autopsia, dependiendo de que el resultado sea negativo o positivo, respectivamente. Estas muestras deben recogerse en medio de transporte de virus, o en medio de inactivación; este último, además de garantizar la estabilidad del virus, permite también su inactivación para una mayor bioseguridad en el transporte y el inicio del análisis. Estas muestras se destinarán al análisis molecular.
En cualquier caso, si se decide realizar la autopsia completa, dirigida o mínimamente invasiva, se podrían tomar adicionalmente otras muestras de vías bajas respiratorias para el diagnóstico microbiológico. Estas muestras pueden ser hisopos bronquiales o de parénquima pulmonar, e incluso una pequeña biopsia pulmonar en fresco. Estas muestras pueden ser de gran utilidad cuando el virus no sea detectable en nasofaringe —lo que puede ocurrir en determinadas fases de la enfermedad— pero sí en vías bajas respiratorias, al igual que ocurre en ocasiones con el virus de la gripe y otros virus respiratorios. Además, datos no publicados de estudios post mortem en COVID-19 (comunicación personal Nihan Ziyade, Neval Elgormus) ponen de manifiesto que las torundas traqueales pueden ser positivas cuando el HNF es negativo. Todo esto hay que tenerlo en cuenta especialmente en los casos con sospecha de COVID-19 en los que se obtiene un resultado negativo en los HNF o hisopos faríngeos.
Otra muestra que puede ser interesante sería el hisopado rectal, pues la excreción del virus en heces es más prolongada (hasta 30días), pero habrá que tener en cuenta en la interpretación de este resultado que este hallazgo no tiene por qué indicar necesariamente que el fallecido se encuentra en la fase aguda de la enfermedad, sino que evidencia una infección pasada.
Como se ha indicado anteriormente, la interpretación de la PCR en cadáveres deberá hacerse cuidadosamente. Ante un resultado claramente positivo en HNF, en muestra faríngea o en muestras de vías bajas respiratorias, se asumirá que el fallecido tiene la infección COVID-19. No obstante, esto no siempre implica que el paciente se encuentre en la fase aguda de la enfermedad, puesto que se han publicado series de pacientes en donde existe persistencia de ARN viral de este patógeno en muestras clínicas sin que el virus sea viable.
Por el contrario, ante un resultado negativo, la negatividad puede no deberse a la ausencia de la infección, sino a otras razones, siendo las principales:
- -
La muestra no se ha tomado bien.
- -
El virus ya no se detecta en faringe ni nasofaringe, pero podría estar en vías bajas respiratorias debido a la evolución de la enfermedad.
- -
La infección está remitiendo y la carga viral, al ser muy baja, está por debajo del límite de detección de la técnica.
No existen en la actualidad datos fundados para el uso como cribado de los llamados test rápidos serológicos para recomendar de manera rutinaria su uso en cadáveres, pues este puede dar lugar a errores según lo indicado anteriormente.
En lo que respecta a la realización de test serológicos clásicos, semiautomatizados y automatizados, estos tendrían sentido en las siguientes circunstancias:
- -
Fallecidos con resultado negativo de PCR y sospecha de infección por COVID-19, es decir, para confirmar la infección en ausencia de una PCR positiva. En estos casos la evolución de la infección debería ser superior a 7días.
- -
Cuando haya que confirmar una «infección pasada» mediante la determinación de la IgG, en relación o no con la negativización de una PCR realizada ante-mórtem.
Por estas razones los protocolos forenses a nivel nacional han incluido la recogida de suero, con el fin de realizar estudios serológicos. Además, estos protocolos también han incluido la recogida de sangre periférica con EDTA, con objeto de detectar una posible viremia, si bien la utilidad diagnóstica sería muy limitada, pues no existen estudios que hayan comprobado la presencia del virus en sangre u orina66.
En cualquier caso, la interpretación final de los resultados obtenidos deberá hacerse siempre teniendo en cuenta la situación clínica del fallecido, por lo que sería conveniente disponer de datos de la historia clínica del mismo si la hubiese y de la entrevista con los convivientes, sobre todo con objeto de trazar el inicio de la sintomatología si la hubiese habido; no olvidemos que, sobre todo en pacientes de edad avanzada, algunos síntomas pueden confundirse con manifestaciones propias de la edad (tos crónica, comorbilidad, deterioro cognitivo, etc.).
Otros estudios microbiológicos de interés en fallecidos por COVID-19Aunque desde el punto de vista forense los fallecidos por COVID-19 presentan características fácilmente distinguibles, el hecho demostrado de que estos pacientes pueden haber padecido distintos tipos de coinfecciones en el momento de su muerte hace necesarios estudios microbiológicos complementarios que aclaren esta posibilidad. Si no se realiza la apertura del cadáver, lógicamente la posibilidad de detectar estas coinfecciones va a ser menor. Siempre se pueden realizar tomas adicionales de HNF e hisopado faríngeo para dedicarlas al cultivo bacteriano y a los análisis moleculares para virus respiratorios. No obstante, su negatividad no implica que no haya una infección en vías bajas respiratorias. Por ello sería conveniente la toma adicional de hisopo bronquial y biopsia de pulmón, si se realiza la autopsia tradicional67, o de un aspirado con jeringa de pulmón si se hiciera una autopsia mínimamente invasiva, siguiendo recomendaciones del ESGFOR (European Study Group of Forensic and Post-mortem Microbiology de la ESCMID, European Society of Clinical Microbiology and Infectious Diseases)68.
Como en cualquier otro estudio post mortem, los análisis más frecuentes son las técnicas de cultivo bacteriológico y fúngico, las técnicas de detección de antígeno y las técnicas moleculares69.
Para la detección de las bacterias que pueden aparecer en coinfecciones de la COVID-19 se puede recurrir al cultivo bacteriológico de las muestras en medios convencionales sólidos y líquidos con condiciones y tiempos de incubación similares a los de las muestras en vivos. Las bacterias atípicas (Legionella sp. y C. pneumoniae) pueden analizarse mediante técnicas antigénicas (enzimoinmunoanálisis o inmunocromatografía) o técnicas moleculares. Cuando se sospeche de una infección fúngica las muestras se deben inocular en agar Sabouraud con antibiótico, si bien es cierto que Aspergillus crece bien en casi todos los medios de cultivo tanto para bacterias como para hongos. Para detectar la presencia de este hongo son útiles los antígenos fúngicos como el galactomanano, y sobre todo la búsqueda de su ADN. La detección de ácidos nucleicos por técnicas moleculares es de utilidad para poner de manifiesto cualquiera de los microorganismos que pueden acompañar al SARS-CoV-2, pero en el caso de los estudios virológicos adquiere una especial relevancia.
Cualquier estudio molecular tiene como punto de partida la extracción de ácidos nucleicos de la muestra. Esta suele hacerse con plataformas automáticas que garanticen la adecuada calidad del material obtenido. Sobre este material se realizan PCR de distinta naturaleza que pueden combinarse con otras técnicas, como los microarrays. Para detectar la presencia de otros con virus respiratorios diferentes del SARS-CoV-2 es de gran utilidad el uso de microarrays comerciales que permiten en un solo ensayo el análisis de los 17 virus respiratorios más frecuentes en humanos (Clart Pneumovir. Genómica S.A.U.). Otras técnicas, como el NxTAG de Luminex o el Filmarray de Biomerieux (PCRs multiplex), permiten la detección simultánea de manera fácil y fiable tanto de los principales virus respiratorios como de las bacterias responsables de las neumonías atípicas, e incluso su cuantificación en algunos casos.
Conflicto de interesesLos autores no tienen conflicto de intereses.
Los autores quieren expresar su agradecimiento a los miembros del grupo ESGFOR (European Study Group of Forensic and Post-mortem Microbiology de la ESCMID, European Society of Clinical Microbiology and Infectious Diseases) por su colaboración y la experiencia aportada con respecto a la pandemia de COVID-19, y especialmente a las Dras. Nihan Ziyade y Neval Elgormus (Ministry of Justice, Council of Forensic Medicine, Postmortem Microbiology Laboratory, Estambul, Turquía).