El análisis de ADN circulante a partir de sangre periférica ha demostrado ser de utilidad en campos clínicos tan diferentes como la oncología, los trasplantes y el cribado prenatal. Para su incorporación al laboratorio clínico es necesario asegurar protocolos preanalíticos adecuados, reproducibles y estandarizados. En este documento se pretenden dar unas recomendaciones preanalíticas para la obtención de ADN circulante a partir de sangre periférica. Incluyen el tipo de espécimen, el tipo de tubo de extracción, el modo de centrifugación de la muestra, la extracción del ADN circulante y cuantificación, así como su conservación.
Cell-free DNA analysis in peripheral blood has been shown to be useful in oncology, organ transplantation, and prenatal screening. For its introduction into the clinical laboratory, it is necessary to ensure appropriate, reproducible and standardised pre-analytical protocols are in place. The aim of this document is to provide pre-analytical recommendations for obtaining of cell free DNA from peripheral blood. These recommendations include the type of sample and extraction tube, the method of centrifugation, the method for cell free DNA extraction, and measurement and storage conditions.
Se considera ADN circulante (ADNc) al que ha sido liberado en los fluidos biológicos y, por tanto, se encuentra fuera de las células. La presencia de ADN extracelular en sangre ya fue descrita por Mandel y Metais en 19481. Las células pueden liberar el ADNc a la circulación por diversos mecanismos, destacando entre ellos la apoptosis2. Por ello, este ADNc está formado por fragmentos de pequeño tamaño, de aproximadamente 145-200 pares de bases (pb), que corresponde al tamaño de ADNc incluido en los nucleosomas liberados en la apoptosis3. En ocasiones se encuentran también fragmentos de ADNc que provienen de la necrosis celular, los cuales en general tienen tamaños mucho mayores, incluso superiores a 10.000 pb.
El ADNc no está libre en los fluidos, sino que se encuentra asociado a lípidos, proteínas e incluso a microvesículas. Es importante conocer esto, ya que estas uniones podrían interferir en la extracción y análisis posterior del ADNc4. En la literatura científica en inglés el ADNc se denomina frecuentemente cell-free DNA, o ADN libre celular, refiriéndose a que no está incluido dentro de las células. Sin embargo, esta denominación puede ser equívoca teniendo en cuenta lo anteriormente indicado.
El ADNc se encuentra habitualmente en concentraciones muy bajas, habitualmente entre 1 y 100μg/l, que varían ampliamente de unos individuos a otros. Los niveles del ADNc dependen de diversos factores, como la velocidad de liberación al medio, la degradación y el aclaramiento. El ADNc en sangre tiene una vida media corta, entre 4 y 30 min, y se elimina fundamentalmente en el riñón, bazo y en el hígado5,6. La concentración de ADNc puede aumentar especialmente en algunas situaciones patológicas, como procesos inflamatorios o cáncer, en donde se produce una mayor apoptosis o necrosis celular.
Desde el descubrimiento del ADNc, numerosos trabajos han puesto de manifiesto la utilidad de su medición en campos tan diferentes como la oncología, los trasplantes y el cribado prenatal (revisado en2). Así, se ha demostrado que la detección y cuantificación del ADNc tumoral en pacientes con cáncer de pulmón es útil en la elección de terapias dirigidas y la monitorización del tratamiento2. También la detección del ADNc procedente del donante en el plasma de pacientes trasplantados de riñón, hígado o corazón permite una detección temprana del rechazo2. Finalmente, en el campo de la obstetricia, el análisis del ADN fetal en sangre materna permite detectar la trisomía 21, 18 y 13 con mayor eficiencia que las pruebas bioquímicas clásicas7.
La existencia de protocolos estandarizados es fundamental para la incorporación de las tecnologías basadas en el análisis del ADNc en el laboratorio clínico, así como para la obtención de resultados comparables entre diferentes laboratorios. En este contexto, la estandarización de la fase preanalítica, si bien es muy importante en el análisis de las magnitudes bioquímicas en general, aquí cobra una importancia crucial8. Es importante definir el espécimen más adecuado y las condiciones para su obtención, así como la metodología de extracción del ADNc y su almacenamiento previo al análisis.
Además de la baja concentración del ADNc, la fracción de interés analítico puede tener una abundancia relativa muy baja respecto a ADNc total. Así por ejemplo, la fracción de ADNc fetal en sangre materna es de aproximadamente el 10% en el primer trimestre9; sin embargo, la fracción de ADNc tumoral puede ser mucho menor, incluso inferior al 0,1%2. Todo ello hace que los requerimientos de sensibilidad y especificidad analítica sean muy exigentes10. Frecuentemente incluso se realiza la analogía entre el análisis de estas fracciones de ADNc y la búsqueda de una aguja en un pajar. En algunas ocasiones, sin embargo, la fracción de interés analítico es mayoritaria respecto al ADNc y puede llegar, incluso, al 90% del total.
Objeto y campo de aplicaciónEn este documento se pretenden dar una serie de recomendaciones preanalíticas para la obtención de ADNc a partir de sangre periférica. Incluyen el tipo de espécimen, el tipo de tubo de extracción, el modo de centrifugación de la muestra, la extracción del ADNc y cuantificación, así como su conservación. Si bien estas recomendaciones se podrían aplicar asimismo a otros fluidos biológicos, los aspectos más concretos de estos quedan fuera del ámbito de la presente guía.
Tipos de espécimen para el análisis de ADN circulanteEn relación con el espécimen de sangre, la extracción del ADNc puede realizarse a partir de suero o de plasma11. En ambos casos es necesaria una adecuada separación de la fracción celular para evitar al máximo la contaminación con ADN celular. Normalmente es preferible el plasma al suero, ya que el proceso de coagulación causa una liberación de ADN genómico por parte de los leucocitos4. Así pues, el suero tiene una mayor concentración de ADNc respecto del plasma, debido sobre todo al ADN genómico liberado en la coagulación12. Ello resulta en que, a igualdad de copias mutantes, la fracción alélica será menor en suero que en plasma. Por ejemplo, la presencia de tres copias mutadas frente a 300 copias del alelo silvestre resulta en una frecuencia alélica del 1%. Si la muestra está enriquecida en ADN genómico, esas mismas tres copias pueden estar acompañadas de, por ejemplo, 3.000 copias del alelo silvestre, por lo que la frecuencia alélica sería de solamente el 0,1%. De ello también se deduce que una mayor riqueza de ADNc no implica necesariamente una mayor capacidad de identificar copias mutadas2. Esto es especialmente importante cuando se trabaja con muestras que tienen concentraciones de ADNc de interés bajas, como suele suceder en el ámbito de la oncología. Así pues, el uso de plasma proporciona mejores resultados que el suero para el estudio de alteraciones moleculares o secuencias que están en muy baja proporción13.
Otra desventaja del suero respecto al plasma se refiere al hecho de que en el proceso de coagulación se pueden retirar porciones del ADNc asociado a las partículas (como virtosomas, HDL, argonauta 2 o exosomas) que se pueden unir a la fibrina y o el fibrinógeno14.
Además de la sangre, que es el espécimen más usado, el ADNc se puede analizar a partir de otros líquidos biológicos. Estos pueden ser más interesantes, por ejemplo, cuando las barreras semipermeables causen un incremento local del ADNc, impidiendo que se difunda a circulación general. Así, por ejemplo, el líquido cefalorraquídeo puede ser el espécimen de elección para el análisis del ADN liberado por un tumor cerebral. También el ADNc fetal estará más concentrado en el líquido amniótico que en la circulación materna, aunque en este caso la concentración en sangre es suficiente para realizar la mayoría de los estudios genéticos fetales. Ya que el ADNc se puede filtrar en el riñón, también la orina puede ser un espécimen adecuado para analizarlo, pero este se encuentra en fragmentos más pequeños debido a la acción de las ADNasas15.
Elección del tubo de extracción del espécimenComo ya se ha mencionado anteriormente, el plasma es la muestra más adecuada para analizar el ADNc, y la elección del tipo de tubo en el que se va a extraer la sangre es también una cuestión preanalítica relevante. Existen dos tipos de tubos en el mercado que pueden utilizarse para el estudio del ADNc:
Tubos sin conservante: requieren una rápida centrifugación y separación del plasma durante las primeras 2-4 h tras la extracción de la sangre para evitar la lisis celular con la consiguiente dilución del ADNc11,13,16. Esto limita su aplicación únicamente a las muestras que se extraen en lugares muy próximos al laboratorio en donde se va a realizar el análisis y, además, requiere de un mayor control por parte del personal para llevar a cabo su procesamiento rápidamente. Dentro de los tubos de plasma, aquellos que contienen etilendiaminotetraacetato potásico (K2EDTA) como anticoagulante resultan de elección para la realización de estos estudios17, habiendo demostrado ampliamente su utilidad. Existen algunos modelos que incorporan un gel separador que facilita la obtención del plasma. Se desaconseja el empleo de heparina ya que inhibe la polimerasa, lo que restringe bastante el uso posterior del ADNc. Pese a que algunos autores han observado una mayor estabilidad del ADNc en tubos con citrato sódico al 3,2%18, el uso de este anticoagulante está mucho menos extendido. En el caso de usar este tipo de tubos hay que tener en cuenta el efecto de dilución debido al propio anticoagulante.
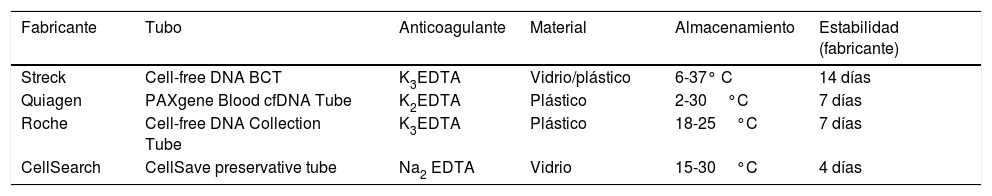
Tubos con conservante: reducen el efecto de la lisis celular19,20 y conservan el ADNc en la sangre durante varias h, e incluso días, a temperatura ambiente sin necesidad de centrifugación21. Por ello resultan idóneos para el envío de especímenes sin procesar a centros externos. En este grupo destacan los tubos Cell-free DNA BCT (Streck) y PAXgene Blood DNA Tube (Qiagen), ambos disponibles con marcado CE. Existen otros modelos que se usan habitualmente para el aislamiento de células tumorales circulantes, como los tubos Cell-free DNA Collection Tube (Roche Diagnostics) o los CellSave Preservative Tube (Janssen Diagnostics), pero que también estabilizan y conservan el ADNc22,23 (tabla 1). Una alternativa más económica a estos tubos es la adición de formaldehído a los tubos con EDTA26.
Características de los tubos comerciales con conservante para el estudio de ADNc
| Fabricante | Tubo | Anticoagulante | Material | Almacenamiento | Estabilidad (fabricante) |
|---|---|---|---|---|---|
| Streck | Cell-free DNA BCT | K3EDTA | Vidrio/plástico | 6-37° C | 14 días |
| Quiagen | PAXgene Blood cfDNA Tube | K2EDTA | Plástico | 2-30°C | 7 días |
| Roche | Cell-free DNA Collection Tube | K3EDTA | Plástico | 18-25°C | 7 días |
| CellSearch | CellSave preservative tube | Na2 EDTA | Vidrio | 15-30°C | 4 días |
Pese a la gran cantidad de tubos disponibles en el mercado, existen todavía pocos estudios que determinen de forma robusta la capacidad de conservación del ADNc de cada uno de ellos. Por este motivo, cada laboratorio debe implantar sus propios protocolos de extracción según sus necesidades, tratando de estandarizar al máximo el proceso. Sin embargo, la muestra que más rendimiento está obteniendo en la detección de porcentajes de ADN mutado entre 1-0,01% (dependiendo del método de amplificación) es la sangre obtenida en tubos con K2 EDTA13.
Obtención del plasmaLa concentración de ADNc en la muestra aumenta a medida que transcurre el tiempo entre la venopunción y su procesamiento. Si no se usan tubos con estabilizantes de la integridad celular, se recomienda procesar cuanto antes las muestras de sangre con anticoagulante, siendo preferible realizarlo en las 2-4 h tras su obtención24. Otro factor a evitar es la hemólisis ya que se puede liberar ADN genómico que puede interferir en la extracción y disminuir la proporción de ADNc. Es necesario tener en cuenta el transporte de la muestra para que no suponga un riesgo de lisis celular. En relación a ello, no se han encontrado diferencias en la concentración de ADNc cuando se emplea tubo neumático en el transporte25. También es conveniente evitar temperaturas elevadas.
Se han evaluado distintas velocidades de centrifugación, obteniéndose más contaminación de ADN genómico a velocidades inferiores a 800g, ya que el sobrenadante obtenido puede no estar completamente libre de células. Un problema similar puede suceder si se centrifuga a una velocidad excesivamente alta, por la posible rotura de células y la liberación de ADN genómico. Se ha comprobado que una primera centrifugación del plasma a 1.200 o 1.600g es suficiente para obtener plasma libre de células evitando el daño celular14.
Se concluye como protocolo óptimo realizar un primer paso de centrifugación durante 10min a 1.600g, luego proceder a la separación del plasma y, posteriormente, centrifugar este a 16.000g durante 5min. En relación a la utilización de plasmas congelados, no influye en el rendimiento si la segunda centrifugación del plasma se hace antes o después de congelarse13.
Extracción del ADN circulanteLos métodos de extracción de ADNc han de estar adaptados a las características especiales relacionadas con su baja concentración en los fluidos biológicos y al hecho de que se encuentre como pequeños fragmentos. Por tanto, la elección de un método adecuado de extracción de ADNc es esencial para conseguir cuatro aspectos importantes:
- a)
Una buena recuperación de ADNc.
- b)
Capacidad de obtener fragmentos cortos de ADNc.
- c)
Que sea reproducible.
- d)
Que se pueda utilizar en análisis posteriores.
El ADNc se puede extraer empleando métodos convencionales manuales, como el de fenol-cloroformo o de precipitación con alcohol, que han demostrado ser eficientes y con capacidad de extraer fragmentos pequeños de ADN26. Sin embargo, estos procedimientos son complejos, requieren un personal especializado, son difíciles de estandarizar y no son adecuados para un número elevado de muestras26. Debido a esto, es más recomendable usar kits específicos, ya que son más sencillos de usar y se pueden emplear con un número de muestras elevado (tabla 2). Con el fin de garantizar la integridad y recuperación del ADNc, y la reproducibilidad de la extracción, el subproyecto de SPIDIA (Standardisation and improvement of generic pre-analytical tools and procedures for in vitro diagnostics) SPIDIA-DNAplas EQA recomienda el uso de procedimientos de extracción específicamente desarrollados para ADNc27.
Ejemplos de kits comerciales para aislamiento de ADNc
| Método | Fabricante | Kit | Volumen de plasmarequerido (mL) |
|---|---|---|---|
| Qiagen | QIAamp Circulating Nucleic Acid Kit | 1-5 | |
| Columna | Analytik Jena | Polymer Mediated Enrichment (PME) free-circulating DNA Kit-IPC16 | 1-5 |
| Epigentek | FitAmp Circulating DNA Kit | 0,3 | |
| Macherey-Nagel | Nucleospin Plasma XS Kit | 0,25 | |
| Norgen Biotek | Plasma/Serum Cell-Free Circulating DNA kit | 0,2-0,5 | |
| Roche Molecular Systems | cobas® DNA Sample Preparation kit | 2 | |
| Zymo Research | Quick-cfDNA Serum & Plasma Kit | 0,2-10 | |
| Partículas magnéticas | Promega | The Maxwell RSC cADNc Plasma Kit | 0,2-1 |
| Qiagen | QIAamp MinElute ccfDNA | 4-10 | |
| Perkin Elmer | Chemagic cfNA 5k Kit | 2-5 | |
| Thermo Fisher | MagMAX Cell-Free DNA Isolation Kit | 0,5-10 |
Existen numerosos kits de extracción de ADNc que se basan fundamentalmente en la unión del ADN a membranas de sílice en presencia de agentes caotrópicos o a bolitas magnéticas recubiertas de sílice, presentando estas últimas la ventaja de evitar los pasos de centrifugación26 (tabla 2). Además, algunos protocolos de extracción de los kits incluyen, o recomiendan, una etapa previa de incubación con proteinasa K, lo que permite incrementar la recuperación de ADNc11,26. Hay que considerar que incluso empleando kits comerciales específicos para ADNc, la eficiencia de la extracción de ADNc a partir de una misma muestra puede diferir en varios órdenes de magnitud28. Numerosos autores han comparado métodos de extracción de ADNc (revisado en26). En un estudio del SPIDIA-DNAplas EQA, 56 laboratorios europeos recibieron muestras de plasma y extrajeron el ADNc, que posteriormente enviaron para analizar a un centro y se comprobó que la concentración de ADNc variaba entre 2,87 y 224,02μg/l plasma27. Además, el aislamiento de ADNc se puede realizar empleando kits de extracción manuales o automáticos. Un estudio reciente de comparación entre laboratorios observó que, si bien los kits automáticos eran más precisos, los manuales eran más eficientes, extrayendo una mayor cantidad de ADNc por mL29.
Aparte de la diferencia en la cantidad de ADNc extraído, los kits presentan una recuperación variable de los fragmentos cortos y largos de ADNc30. Así por ejemplo, se ha observado analizando mutaciones de KRAS en pacientes con cáncer de colon que los métodos basados en membrana de sílice favorecen la extracción de fragmentos largos de ADNc, mientras que los sistemas basados en bolas magnéticas son más eficientes recuperando fragmentos cortos de ADNc31. Esta diferente capacidad de extracción podría causar un enriquecimiento de unas copias respecto a otras, causando variaciones en la fracción alélica.
Finalmente, hay que tener en cuenta que los kits de extracción concentran el ADNc en pequeños volúmenes a partir de largos volúmenes iniciales, por lo que en el proceso puede haber un arrastre de posibles compuestos interferentes en los análisis posteriores, como puede ser la PCR cuantitativa28. Por ello es necesario asegurarse que el kit empleado en la extracción de ADNc es óptimo para los procesos de análisis posteriores.
Análisis del ADN circulanteEs recomendable realizar un análisis de la calidad del ADNc tras la extracción y como paso previo a su procesamiento analítico. En primer lugar, es necesario determinar la concentración del ADNc y, en segundo lugar, comprobar que este corresponde al ADN fragmentado y que está libre de ADN genómico.
Concentración del ADN circulantePara la medida de la concentración del ADN circulante se pueden emplear métodos espectrofotométricos, fluorimétricos o de PCR cuantitativa32. De estos, el más habitual y recomendable es el método fluorimétrico, que utiliza compuestos fluorescentes que se intercalan entre las hebras de ADN emitiendo fluorescencia de forma proporcional a la concentración de esta molécula5. Los ensayos «double-stranded DNA» son idóneos para la medida del ADNc, ya que, dependiendo del ensayo, pueden detectar específicamente el ADN intacto de doble cadena y con una sensibilidad inferior a 0,1 ng/μL33–35. El principal inconveniente de esta técnica es que realiza una cuantificación relativa en función de una recta de calibrado con patrones de concentración conocida, lo que requiere de un mantenimiento periódico por parte del personal. Existen equipos comerciales que se basan en estos métodos fluorimétricos, como el Qubit® fluorometer (Thermo Fisher Scientific), que es ampliamente utilizado, y Quantus® fluorometer (Promega corporation).
Los otros dos sistemas que se han empleado para cuantificar ADNc son los métodos espectrofotométricos y los basados en PCR cuantitativa. Los sistemas de medida por espectrofotometría UV miden la absorbancia de la muestra a 260nm. El equipo más utilizado es el NanoDrop® (Thermo Scientific). Aunque es un método barato y que requiere solamente unos pocos segundos de análisis, su sensibilidad es limitada, lo que supone el principal problema para la cuantificación del ADNc. Además, estos sistemas suelen producir una sobreestimación de los resultados al detectar también otras moléculas como el ADN degradado, de cadena simple o nucleótidos libres34. Sin embargo, el equipo NanoDrop® proporciona información de la presencia de contaminantes, como RNA o proteínas32. La relación de la densidad óptica a 260nm y 280nm da información de la pureza del ADN, considerándose una ratio superior a 1,8 como un indicador de pureza. La absorbancia a 230nm puede ser útil para determinar contaminaciones por solutos durante el proceso de extracción. De hecho, Nakayama et al. sugieren un análisis inicial con NanoDropTM para averiguar la posible presencia de contaminantes, seguida de una cuantificación más específica mediante Qubit®34. Por último, la PCR cuantitativa de un gen diana resulta útil para la medida del ADNc amplificable36, pero el elevado consumo de tiempo y recursos que supone esta metodología limita considerablemente su utilidad en la rutina clínica. Se han empleado diversos genes dianas, como el del precursor del beta amiloide32 o el de la gliceraldehido-3-fosfato deshidrogenasa34.
Distribución de tamaños moleculares del ADNcEs importante asegurarse de que con el método de extracción del ADNc utilizado se obtienen fragmentos pequeños y que se encuentra libre de ADN genómico. Esta comprobación se puede realizar usando técnicas de electroforesis, que permiten ver la distribución de tamaños de los fragmentos de ADN obtenidos. Mediante este método se puede distinguir entre el ADN genómico, que aparece como una banda de elevado peso molecular, y el ADNc, que aparece como bandas del tamaño aproximado de múltiplos de nucleosomas, esto es, en el rango de 100-300 pb. Este análisis de fragmentación del ADN se realiza, sobre todo, mediante electroforesis capilar o en microfluido, que además proporciona una estimación de su concentración5. Si bien la electroforesis capilar permite un análisis continuo, la electroforesis en microfluido tiene la ventaja de ser más exacta en la determinación del tamaño de ADNc y más sensible. Existen proveedores que disponen de sistemas automatizados de sistemas de microfluido con una sensibilidad inferior a 0,1 ng/μL, que los hacen muy adecuados para el ADNc.
Almacenamiento de la muestra que no se va a usarEl plasma puede almacenarse a -20°C si se va a proceder a extraer el ADNc durante los 30 días siguientes. Si la extracción se va a realizar más tarde, es recomendable el almacenamiento a -80°C13. Es aconsejable alicuotar el plasma para evitar los ciclos de congelación y descongelación de la muestra, ya que pueden interferir en su estabilidad37. Si se almacena de forma adecuada, las alteraciones moleculares presentes en el ADNc pueden detectarse en muestras de plasma congeladas durante años38. No obstante, después de unos 7 años puede observarse una reducción del 35-40% ADNc, lo cual es importante para muestras almacenadas en biobanco18.
El ADNc también puede almacenarse una vez extraído a -20°C13. Como en el caso anterior, la conservación durante 7 años produce un deterioro del 20-25% del ADNc18. El deterioro es mayor cuando la frecuencia alélica es baja. Es recomendable no superar 3 ciclos de congelación y descongelación para evitar problemas de estabilidad en la muestra.
RecomendacionesLa determinación de alteraciones en el ADNc es un proceso muy exigente que requiere unas condiciones preanalíticas muy estrictas para proporcionar unos resultados correctos y comparables. Por ello, es necesario establecer recomendaciones para la obtención y aislamiento del ADNc a partir de sangre periférica:
- a)
Se recomienda utilizar plasma obtenido con EDTA para la extracción de ADNc a partir de sangre. Se recomienda emplear tubos especiales de recogida que eviten la lisis celular si la muestra no se va a procesar en las 2-4 h siguientes.
- b)
Se recomienda obtener el plasma mediante una doble centrifugación, consistente en una primera a 1.200-1.600g durante 10min y, tras la separación del plasma, una segunda a 16.000g durante 5min.
- c)
Se recomienda usar kits comerciales de extracción ADNc que hayan sido diseñados específicamente para ese propósito.
- d)
Se recomienda realizar la cuantificación del ADNc extraído mediante métodos fluorimétricos o PCR cuantitativa. Asimismo, es recomendable evaluar la contaminación del ADNc extraído con ADN genómico usando métodos electroforéticos.
- e)
Se recomienda conservar tanto el plasma a -80°C como el ADNc ya extraído a -20°C, evitando repetir ciclos de congelación y descongelación.
Este documento tiene la conformidad de las tres Sociedades como Recomendación profesional en el ámbito del Laboratorio Clínico.