Clostridium difficile es un bacilo gram positivo, anaerobio estricto, capaz de formar esporas que le permiten su supervivencia en aguas, suelos y en ambientes hospitalarios, donde puede permanecer hasta años. Inicialmente, fue descrito en 1935 como un agente normal de la microbiota intestinal de recién nacidos sanos.
El presente escrito pretende revisar las generalidades del microorganismo y de la enfermedad asociada a C. difficile, enfatizando la prevalencia de ambos en nuestro medio, la problemática, el vacío del conocimiento que se presenta y los métodos de laboratorio que permiten su diagnóstico y estudio.
Clostridium difficile is a gram-positive, strictly anaerobic, bacillus capable of forming spores that enable it to survive in waters, soils, and in hospital environments, where it can remain for years. It was initially described in 1935 as a normal microorganism of the intestinal microbiota of healthy newborns.
This article reviews the general features of the microorganism and the disease associated with C. difficile, emphasising the prevalence of both in our environment, the problems, the lack of knowledge on it, and the laboratory methods that help in its diagnosis and study.
Clostridium difficile (C. difficile) es un bacilo grampositivo, anaerobio estricto, capaz de formar esporas que le permiten su supervivencia en aguas, suelos y en ambientes hospitalarios, donde puede permanecer hasta años. Inicialmente, fue descrito en 1935 como un agente normal de la microbiota intestinal de recién nacidos sanos.
Su transmisión por vía fecal-oral convierte al personal de la salud, objetos médicos y superficies infectadas en una importante fuente de infección intrahospitalaria1,2, llegando a considerarse, actualmente, el principal agente causal de diarrea nosocomial asociada al tratamiento con antibióticos, donde además de la diarrea, puede causar colitis seudomembranosa (CSM), enfermedad atribuida, en la mayoría de los casos, a C. difficile, e incluso complicaciones como la colitis fulminante, todas ellas englobadas en el termino de enfermedades asociadas a C. difficile (EACD).
Con la aparición en 2002 del brote causado por la cepa hipervirulenta BI/NAP1/027/toxinotipo III, las cifras de morbimortalidad en el ámbito mundial aumentaron, viéndose afectados, principalmente, los países de Norteamérica y Europa. Al reconocer a C. difficile como un patógeno importante para la salud, con alto potencial epidémico, se han desarrollado herramientas diagnósticas y de estudio que han permitido conocer a fondo la fisiopatología de la enfermedad, factores de riesgo asociados, prevalencias, resistencia bacteriana y medidas adecuadas de promoción y prevención.
La implementación de métodos de tipificación molecular, como la electroforesis de campo pulsado en gel (PFGE: Pulsed field gel electrophoresis), el análisis con endonucleasas de restricción (REA: Restriction Endonuclease Analysis), la toxinotipificación, el análisis de repeticiones en tándem de número variable en locus múltiples (MLVA: Multiple-locus variable-number), la ribotipificación y el análisis de secuencias de locus múltiples (MLST: Multilocus sequence typing) han permitido el estudio a fondo de esas características de C. difficile, enfocados principalmente al estudio de brotes.
El presente escrito pretende revisar las generalidades del microorganismo y de la EACD, enfatizando la prevalencia de ambos en nuestro medio, la problemática, el vacío del conocimiento que se presenta y los métodos de laboratorio que permiten su diagnóstico y estudio.
Reseña históricaLos primeros reportes de C. difficile como causante de enfermedad se dieron en 1893, cuando Finney describió el caso de una mujer joven con diarrea hemorrágica y seudo-membranas a nivel del colon3. Sin embargo, la bacteria fue aislada por primera vez en 1935 por Hall y O’toóle como microbiota normal del tracto gastrointestinal (TGI) de neonatos, denominándola en ese momento Bacillus difficilis por su dificultad para crecer en medio in vitro4.
Bartlett et al. (1978) determinaron que C. difficile era el principal agente causal de CSM en pacientes en tratamiento con antibióticos5; en este estudio, encontraron que estos pacientes presentaban altos niveles de citotoxicidad en las muestras de materia fecal, atribuyéndolo en un principio a un agente viral, Mycoplasma, o a la toxina de algún otro tipo de bacteria, sin poderlos aislar en el laboratorio. Utilizando un pool de antisueros para varios clostridios, se logró en los pacientes en estudio la neutralización de la actividad citotóxica con suero anti-C. sordelli, logrando aislar con posterioridad a C. difficile de la materia fecal de esos pacientes, y gracias a esto, hoy en día, se puede asociar con el 90 a 100% de los casos de CSM, donde la población más susceptible son los ancianos mayores de 65 años6–10.
PatogénesisComo mecanismo de infección, la forma esporulada de C. difficile, la cual es resistente a la desecación, químicos y temperaturas extremas, ingresa por vía oral, atraviesa el sistema digestivo y se establece en el colon, donde encuentra el ambiente propicio para adoptar su forma vegetativa. La bacteria se adhiere a la capa mucosa del enterocito y la atraviesa con ayuda del flagelo, adhesinas y proteasas que produce11.
En condiciones normales, el TGI del humano posee una microbiota indígena característica que le proporciona el equilibrio necesario para poder inhibir el crecimiento de ciertos patógenos para él, lo que se ha llamado “resistencia a la colonización”12. Sin embargo, ciertas condiciones (ver factores de riesgo asociados al desarrollo de EACD), como el tratamiento con antibióticos de amplio espectro, pueden ocasionar en el individuo una alteración de esta microbiota permitiendo que C. difficile aproveche este desequilibrio para replicarse en grandes cantidades y causar daño a través de la producción de sus toxinas.
ToxinasFue a finales de los años setenta cuando varios autores empezaron a describir la presencia de una toxina en un hámster infectado por la bacteria con un efecto diferente, enterotóxica, a la citotoxina antes mencionada por otros autores13,14, y a principios de los ochenta fueron aisladas de materia fecal de humanos enfermos por C. difficile las 2 clases de toxinas15,16. Hoy en día, se conoce que la bacteria, a nivel del colon, produce una toxina A o enterotoxina y toxina B o citotoxina, ambas tienen efecto citotóxico, causando permeabilidad vascular y hemorragias, pero además, la enterotoxina induce la acumulación de líquidos y células inflamatorias a través de la activación de la respuesta inflamatoria, mientras que la citotoxina causa destrucción del citoesqueleto del enterocito, siendo más potente que la primera16–18.
Las cepas de C. difficile que no producen toxinas no son patógenas18; pero las que tienen los genes tcdA y tcdB, que codifican la expresión de la toxina A y B, respectivamente, sí lo son. Estos genes se encuentran ubicados en una región del cromosoma llamada locus de patogenicidad (PaLoc), el cual contiene también 3 genes accesorios: tcdR, tcdE y tcdC. tcdR ha demostrado tener la capacidad de modular positivamente la expresión de los genes de las toxinas, mientras que tcdC modula negativamente tal expresión, al interferir con la capacidad de la ARN polimerasa de reconocer los promotores de tcdA y tcdB. Esto se ha sugerido al encontrar que tcdA, tcdB y tcdR se transcriben durante la fase de crecimiento estacionaria, mientras que tcdC se expresa ya en la fase de crecimiento exponencial. En cuanto a tcdE, su función aún no está bien definida19–21 (verfig. 1).
Distribución de genes en el PaLoc que codifican y regulan la expresión de las toxinas de Clostridium difficile20.
El receptor al cual se unen las toxinas a nivel del epitelio intestinal aún no está bien definido. Se presume que cada toxina tiene tropismo por diferentes sitios en la célula hospedera: la toxina A se enlaza más efectivamente hacia el lado apical de la célula, mientras que la toxina B se enlaza mejor a un receptor aún desconocido en la zona basolateral de la célula. En animales, la toxina A se ha evidenciado que se puede enlazar al trisacárido Gal1(a1-3)Gal(B1-4) GlcNac, mientras que en humanos se ha propuesto a la glicoproteína gp96 como correceptor para la adherencia de la toxina A a la célula22,23.
Desde que Hall y O’toole, en 1935, describieron por primera vez a C. difficile como microbiota comensal del TGI de recién nacidos, se ha establecido que esta población puede estar infectada sin desarrollar la enfermedad hasta en un 80%, mientras que en los adultos esta cifra es menor del 5%18,24. Se han planteado varias hipótesis de por qué los recién nacidos se infectan pero no padecen la enfermedad: a) el calostro proveniente de la madres puede contener anticuerpos que contribuyan a neutralizar las toxinas A y B25; b) las células intestinales fetales son mucho menos sensibles al efecto de las toxinas que las células intestinales de los adultos26, y c) los recién nacidos pueden carecer de receptores específicos para la unión de las toxinas a nivel de los enterocitos27,28.
Cepa BI/NAP1/027/toxinotipo IIIHacia 2002, la Universidad de Pittsburg Medical Center, en los Estados Unidos, reportó un aumento de los casos de EACD en Norteamérica, inicialmente en Canadá, posteriormente, en EE. UU. y Europa29–32. Tales casos llevaron a la aparición de un brote causado por una cepa hipervirulenta de C. difficile, que en 2005 fue caracterizada como BI/ NAP1/027/toxinotipo III, BI por REA, NAP1 por PFGE y 027 por ribotipificación, y cuyas características principales son:
- a)
Deleción en el gen tcdC, por lo cual no se puede controlar la expresión de los genes, lo que conlleva la hiperproducción toxigénica33,34.
- b)
Hiperproducción de toxina binaria. Esta toxina es codificada en una región llamada locus CDT (CdtLoc), separada del PaLoc, y comprende 3 genes en lugar de 5. Las toxinas que producen, CdtA y CdtB, se encuentran reguladas por el gen CtdR y no poseen relación alguna con las toxinas del PaLoc. La CdtB es la encargada de unirse a la célula hospedera, permitiendo la traslocación de CdtA al interior22,35,36 (verfig. 2).
Figura 2.Distribución de genes en el CdtLoc que codifican y regulan la expresión de las toxinas binarias Clostridium difficile22.
- c)
Resistencia a las fluoroquinolonas30,34,37,38. Se ha sugerido que un factor importante que potenció el desarrollo del brote por este ribotipo 027 resistente a las fluoroquinolonas fue que al mismo tiempo, en los hospitales de Canadá, se empezaba a utilizar comúnmente estos antibióticos39.
Además, in vitro, se ha demostrado que estas cepas tienen alta capacidad de esporulación, en comparación con las cepas que no causan brotes40.
PrevalenciaEl sistema nacional de estadísticas vitales del CDC publicó, en marzo de 2011, el reporte preliminar para muertes en 2009, siendo la enterocolitis la causa número 19 de muerte en la población mayor de 65 años41; en 1999, se presentaron 793 muertes causadas por C. difficile, mientras que en 2009, se reportaron 7.285 muertes, con una tasa de muerte ajustada a la edad de 2,2 muertes por 100.000 personas42. Según la Guía práctica para la infección por C. difficile en adultos, publicada por la Sociedad Americana de Epidemiología para el Cuidado de la Salud y la Sociedad Americana de Enfermedades Infecciosas, C. difficile es el principal patógeno causante de la colitis asociada a antibióticos y del 20–30% de los casos de diarrea nosocomial asociada a antibióticos43.
Después de la aparición de los brotes en Canadá por la cepa BI/NAP1/027/toxinotipo III, las cifras de incidencia y la severidad de la EACD en el ámbito mundial han ido en aumento: en los hospitales canadienses, entre 1997 y 2005, las tasas de incidencia aumentaron de 3,8 a 9,5 casos por cada 10.000 pacientes diarios, o 3,4 a 8,4 casos por cada 1.000 ingresos en cuidados intensivos43; mientras en niños hospitalizados en Estados Unidos, entre 2001 y 2006, la incidencia anual de EACD tuvo un incremento de 2,6 a 4,0 casos por 1.000 admisiones, o de 4,4 a 6,5 casos por 10.000 pacientes diarios44. Según McFarland et al. (2008), la incidencia de EACD a partir del brote en Canadá cambió drásticamente, de 35,6 casos por 100.000 pacientes en 1991 a 156,3 casos por 100.000 pacientes en el 2004, con un aumento en la mortalidad de 4,5% de casos en 1991 a 22% de casos en el 200445. Loo et al. (2004), describieron en varios hospitales de Canadá una incidencia de 22,5 por 1.000 casos de diarrea nosocomial asociada a C. difficile, con una tasa de mortalidad de 6,9% a los 30 días y de 16,7% al año de haber sido diagnosticados30.
Tal vez el ribotipo más ampliamente descrito como causante de infección por C. difficile, especialmente en Europa, es el ribotipo 001, el cual ha sido asociado a altas tasas de resistencia a antibióticos como eritromicina, ciprofloxacina y moxifloxacina46,47. En los últimos años, ha aumentado el reporte de casos de EACD causada por el ribotipo 017, el cual expresa toxinas A−/B+ debido a una deleción del gen tcdA46,48,49, lo cual limita el diagnóstico del laboratorio para aquellas técnicas que detecten la expresión de este gen, resaltando la importancia de implementar o complementar con técnicas moleculares.
Goorhuis et al. (2008), describieron en los Países Bajos una nueva cepa hipervirulenta, ribotipo 078, causante de una EACD de similar severidad a la enfermedad causada por la cepa 027, con un incremento en la incidencia entre 2005 y 2008 del 3 al 13%, afectando principalmente a la población joven y asociada a la comunidad50.
En Latinoamérica, en Costa Rica, también se ha descrito a C. difficile como el principal agente causal de diarrea nosocomial, con una tasa de infección cercana al 30% de los casos de diarrea51,52, mientras que la cepa NAP1 fue encontrada, por primera vez, en otro centro hospitalario de este país con una prevalencia del 54% del total de los aislamientos53.
En Brasil, Souza Dias et al. (2010) describieron un seudobrote en su país en el 2002–2003, donde evaluaron 138 casos de EACD, con una incidencia de 3,3 por 1.000 pacientes hospitalizados, y obteniendo 16 tipos diferentes de C. difficile por reacción en cadena de la polimerasa (PCR: Polymerase Chain Reaction)54. En un estudio de casos y controles, se determinó por ensayo inmunoenzimático (EIA) que 22 casos de 49 estudiados eran positivos para las toxinas de C. difficile55. Balassiano et al. (2011), aislaron y caracterizaron cepas de C. difficile de un hospital de Rio de Janeiro, encontrando a través de EIA, una prevalencia de 27,1% (19 de 70) en los pacientes sujetos a estudios, donde la mayoría de las cepas presentaban los genes tcdA y tcdB, ninguna cepa deleción en el gen tcdC, y el ribotipo 133 fue aislado en el 50% de esas cepas56. El mismo autor, en el 2010, ya había descrito la prevalencia de infección por C. difficile en un grupo de pacientes hospitalizados en una unidad de cuidados intensivos (UCI) en Rio de Janeiro, de los cuales, 43 (19,7%) de los pacientes presentaron EACD, con una incidencia de 1,8 casos por 1.000 pacientes diarios que ingresan en la UCI57.
En Argentina, Legaria et al. (2003), describieron que de 87 pacientes con sospecha de EACD, el 40% eran positivos para la determinación toxigénica de C. difficile a través de EIA58. En Chile, Gardilcic et al. (2000), evaluaron por EIA 27 pacientes hospitalizados con 31 episodios de diarrea asociada a C. difficile durante 4 meses, de los cuales solo murió 1 (4%) paciente por megacolon tóxico59; mientras que en Uruguay, evaluaron 78 pacientes de UCI con diarrea, a los cuales se les evaluó la presencia de C. difficile por aglutinación en látex y EIA, encontrando una prevalencia del 26%60.
Willingham et al. (1998), determinaron por EIA, que de 126 pacientes peruanos con sida, con y sin diarrea, 27 (21,4%) estaban infectados por C. difficile, de los cuales 11 (42%) murieron en el transcurso del estudio61. Más recientemente, en 2007, García et al. (2007), reportaron que de 156 pacientes de un hospital de Lima con diarrea nosocomial, 55 (35,2%) fueron diagnosticados de EACD62.
En Colombia, hasta la fecha, se han realizado pocos estudios que permitan determinar la prevalencia de la infección y enfermedad por C. difficile. En 2008, se publicó una revisión bibliográfica realizada por Otero-Rengino, la cual actualiza la epidemiología, patogénesis y tratamiento de la infección por C. difficile10, y en 2009, el mismo autor determinó mediante un estudio observacional la prevalencia y la causa de colitis en adultos mayores, atribuyéndole a C. difficile el 40% de las causas infecciosas de la enfermedad63.
En Medellín, recientemente Becerra et al. (2011), publicaron el estudio “Factores epidemiológicos y clínicos asociados a infección por C. difficile”, el cual estudia los principales factores clínicos y epidemiológicos asociados al desarrollo de EACD64; y Escobar Díaz et al. (2011), desarrollaron el estudio “Diagnóstico de Clostridium difficile por toxina y cultivo en pacientes con diarrea hospitalizados en la IPS Universitaria Clínica León XIII”, encontrando que de 48 muestras, 8 (16,7%) fueron positivas para toxina y cultivo (estudio aún sin publicar).
Formas clínicasLos individuos que padecen la EACD pueden desarrollar cualquiera de estos estados:
Colitis simpleLa colitis se define como un proceso inflamatorio del colon que puede ser causado por diferentes etiologías: isquémica, infecciosa, ulcerativa, entre otras. En este caso, se presenta una diarrea abundante, de 3 o más deposiciones diarias, liquida, con moco y sangre, acompañada de dolor abdominal; generalmente, autolimitada, que se controla con hidratación y la suspensión de la terapia antimicrobiana18,63.
Colitis seudomembranosa (CSM)Causada por C. difficile se caracteriza por lesiones con eritema, edema, pérdida del patrón vascular, sangrado, placas amarillentas elevadas de 2 a 10mm18,63.
En el estudio de Martin-Alva et al. (2007), sobre la asociación de la toxina A de C. difficile y el daño histopatológico causado en los pacientes, se clasificaron los resultados de las biopsias colónicas y se dividieron en 3 tipos de lesiones65:
- •
Tipo I: se pueden apreciar acumulaciones focales de polimorfonucleares con áreas de necrosis epitelial focalizada y exudación de fibrina y neutrófilos en la luz colónica.
- •
Tipo II: presenta un exudado más prominente que se origina en un área de ulceración epitelial, manteniéndose intacta la mucosa circundante.
- •
Tipo III: consiste en la necrosis epitelial difusa (no focal, como sucedía en los tipos anteriores), cubierta por una seudomembrana formada por polimorfo nucleares, fibrina y detritus celulares.
Manifestación grave y altamente letal de la enfermedad, que aparece aproximadamente en el 3% de los infectados con C. difficile. Se caracteriza, además de lo descrito en la CSM, por presentar una alteración del estado general del individuo, acompañado por letargo, fiebre, leucocitosis, acidosis láctica, taquicardia, dolor abdominal, tono muscular ausente, íleo paralítico, megacolon tóxico, entre otras manifestaciones, hasta llegar a la colectomía e incluso la muerte18,24,66,67.
RecurrenciasAproximadamente entre un 15–45% de los pacientes tratados de EACD sufren recaídas en un período de 2 meses. Esto se puede deber, además de las constantes alteraciones de la microbiota a nivel del TGI, a la posible presencia de esporas de la bacteria no erradicadas por el tratamiento y a que la inmunidad que se genera a partir de la primera exposición no es especifica, ya que estas reinfecciones pueden ser por cepas diferentes a la inicial68.
Infecciones extraintestinalesRara vez, se han reportado casos de infecciones extraintestinales; se han descrito casos de bacteremias, peritonitis, osteomielitis, abscesos viscerales y cerebrales, infección en piel y tejidos, entre otros. En la bacteremia, el daño que las toxinas, especialmente la enterotoxina A, causan a nivel del epitelio del TGI puede llevar a una perforación del mismo y al paso de las toxinas al torrente sanguíneo, donde la acción de la citotoxina puede ser potencialmente letal69,70.
Factores de riesgo asociados al desarrollo de enfermedad asociada al Clostridium difficileEl principal factor de riesgo que influye en el desarrollo de la EACD es la terapia prolongada con antibióticos de amplio espectro. Esto se empezó a evidenciar en 1968, cuando Small demostró en hámster inyectados con lincomicina el desarrollo de enterocolitis, y posteriormente su muerte71, mientras otros estudios, en los setenta, encontraron en materia fecal de hámster tratados con antibióticos, especialmente con clindamicina, y que murieron por inflamación del ciego, un alto número de toxinas de C. difficile72–74. Posteriormente, otras investigaciones determinaron que otros antibióticos, amino-glicósidos y betalactámicos, especialmente ampicilina y cefalosporinas, también incidían en el desarrollo de la EACD en pacientes hospitalizados75–77.
La edad avanzada, especialmente en ancianos hospitalizados, juega un papel muy importante en el desarrollo de la EACD. Este factor de riesgo se ha visto muy asociado con la respuesta del sistema inmune, especialmente la de tipo humoral, que pueda presentar cada individuo ante la infección; en pacientes con edad avanzada, se presenta una falla en la producción de inmunoglobulina G antitoxina A, como respuesta inicial a la infección78–80, además de otras afecciones que se presentan con la edad y que le dificultan la respuesta contra esta clase de patógenos. Karas et al. (2010), en un revisión de la literatura estableció que en 27 estudios, con 10.975 casos de EACD, la mortalidad calculada era menor de 5,99% en esos pacientes con 3 meses de diagnóstico, siendo esta cifra asociada fuertemente a la edad avanzada, donde el 13,5% de los pacientes tenían más de 80 años de edad81.
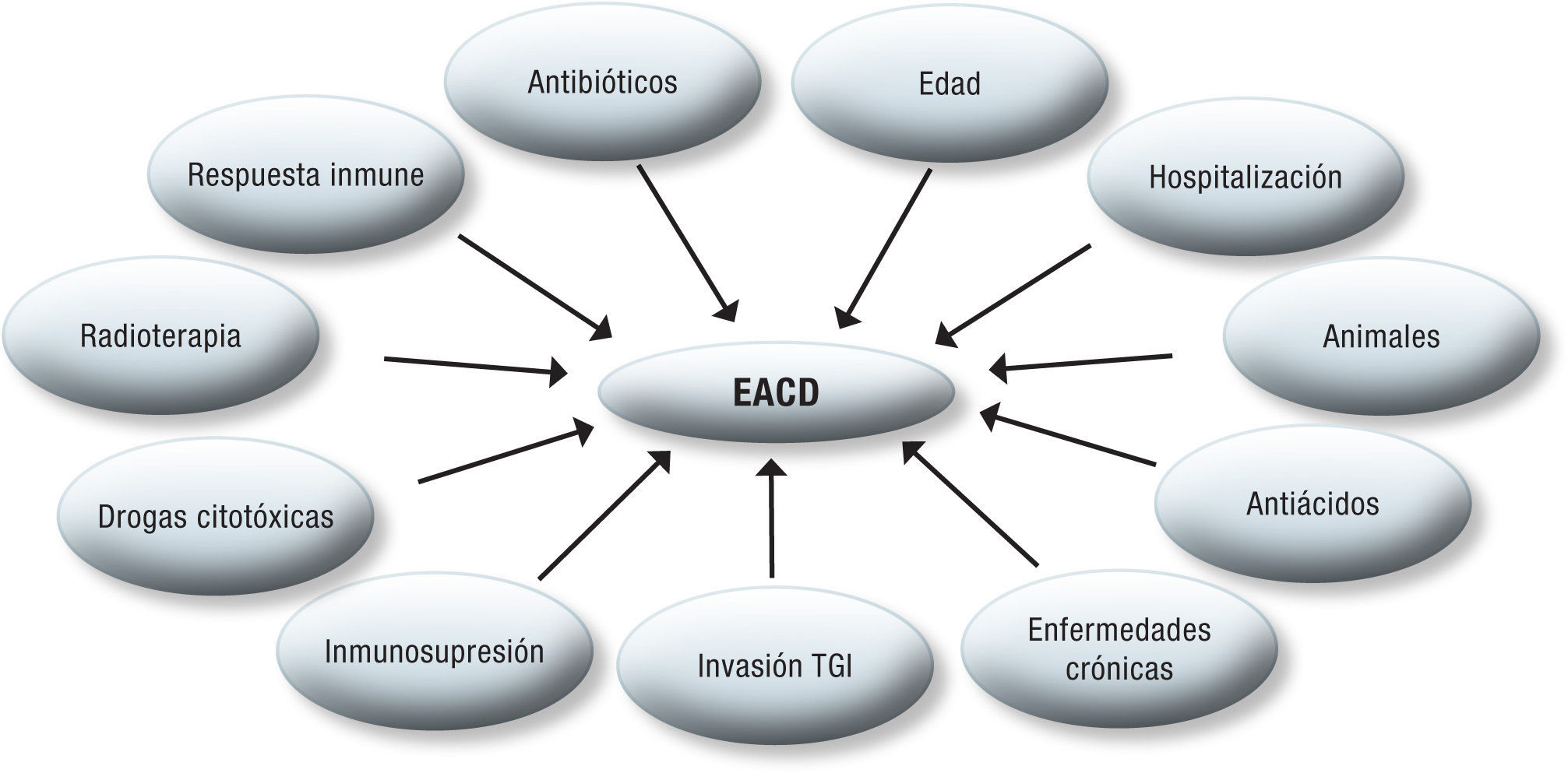
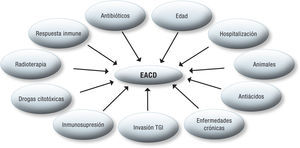
Entre otros factores de riesgo que participan en el desarrollo de la EACD están: estancia prolongada en hospitales, especialmente en centros para personas de la tercera edad y en las UCI, pacientes con enfermedades crónicas severas (renales y hepáticas), estados de inmunosupresión y quimio/radioterapia, drogas citotóxicas, antiácidos, inhibidores de la bomba de protones, cirugías gastrointestinales y otros procedimientos invasivos en el TGI1,8,24.
Como enfermedad mayormente asociada al cuidado de la salud, la presencia de esporas en el medio ambiente hospitalario y su transmisión a los pacientes se ve influida por las inadecuadas practicas de desinfección de espacios físicos, instrumentos médicos y de lavado de manos del personal de salud82–84.
Los animales también establecen una importante ruta de infección para el humano, ya sea que padezcan de la enfermedad o como portadores asintomáticos; se han descrito animales domésticos como gatos y perros, caballos, terneros y lechones como posibles agentes diseminadores de la enfermedad85–88. Se ha descubierto, por técnicas moleculares, que algunos ribotipos de C. difficile encontrados tanto en humanos como en animales se encuentran genéticamente relacionados50 (ver fig. 3).
Diagnóstico por laboratorio y tipificación bacterianaEl diagnóstico de la EACD debe estar basado en una combinación de la clínica del paciente, exámenes de rutina como la colonoscopia o estudios histológicos y el laboratorio. En el laboratorio, existen diferentes métodos para establecer el diagnóstico de la enfermedad: ensayos de neutralización de citotoxina por cultivo celular, aglutinación en látex, cultivo anaerobio más pruebas de identificación y EIA.
En cuanto a las pruebas moleculares, estas pueden ser utilizadas, ya sea como herramientas diagnósticas, siendo el caso de la PCR convencional, o como herramientas de estudio epidemiológico y filogenético, como se va a describir más adelante.
Cultivo celularEl ensayo de neutralización de citotoxina es la prueba de referencia para la identificación de la toxina B en materia fecal, gracias a que en más del 90% de los pacientes con CSM se puede identificar el efecto citopático que la toxina produce sobre tejido humano in vitro. La técnica posee una sensibilidad y una especificidad de casi el 100%. Entre sus limitaciones, encontramos su elevado costo, el tiempo para la entrega de los resultados (2–3 días), la escasez de centros especializados en la técnica y que no existe relación entre la presencia de la toxina y la gravedad de la enfermedad42,43,89.
Tricota-Lee et al. (1987), determinaron que los fibroblastos humanos, en comparación con otras líneas celulares, son los más sensibles para la detección de la toxina90.
Cultivo anaerobioPermite el crecimiento de cepas morfológicamente compatibles con la bacteria; se utilizan medios selectivos y diferenciales incubados en anaerobiosis, el medio más ampliamente utilizado es el de agar cicloserina-cefoxitina-fructosa, descrito por primera vez por George et al. (1979)89,91,92. Está técnica permite el aislamiento de la colonia y su posterior manipulación para utilizarla en la identificación de fenotipos, y el estudio de su perfil de resistencia. Por ejemplo, la identificación confirmatoria se puede realizar mediante las pruebas de indol negativo, L-prolina-aminopeptidasa positiva y métodos comerciales (Crystal™ ID Systems Becton Dickinson, Loveton Circle Sparks, MD)92. Sin embargo, esta técnica puede ser ineficaz para fines diagnósticos debido la dificultad para que este tipo de bacterias crezcan, desarrollo de cepas no toxigénicas o que no expresen la toxina in vitro.
También se han utilizado otros medios de cultivo para el aislamiento de C. difficile: agar cicloserina manitol, agar sangre manitol cicloserina, agar cefoxitina cicloserina con suplementos de sangre, entre otros. Mundy et al. (1995), compararon el agar cicloserina-cefoxitina-fructosa con el agar sangre manitol cicloserina, donde este último presentó menos inhibición de la flora normal presente en la materia fecal, siendo el agar cicloserina-cefoxitina-fructosa más indicado para el asilamiento de C. difficile93.
Ensayos inmunoenzimáticosDesde los años ochenta hasta la fecha, los EIA han sido ampliamente utilizados y modificados para detectar, dependiendo de la casa comercial, la presencia de la toxina A, la toxina B o ambas en materia fecal.
Son pruebas diagnósticas comerciales, rápidas y económicas, con una sensibilidad cercana al 63–94% y una especificidad del 75–100%; los resultados deben ser comparados a la luz de la historia clínica, debido a que hay cepas que pueden no expresar la toxina que se está evaluando, siendo los EIA que aplican ambas toxinas en su detección las mejores pruebas diagnósticas42,43,89,94–96.
Aglutinación en látexInicialmente, se pensó que la aglutinación en látex para detectar C. difficile detectaba la toxina A en las muestras de material fecal; sin embargo, estudios posteriores demostraron que cepas bacterianas no toxigénicas o a las cuales se les ha extraído la toxina son positivas igualmente con el test, sugiriendo que es otra la proteína la causante de la reacción97. Lyerly et al. (1991), comprobaron que la proteína causante de la aglutinación era un antígeno común en C. difficile: la glutamato deshidrogenasa98.
Esta técnica tiene una especificidad del 94–98% y una baja sensibilidad, 58–68%43,89, lo cual la hace poco idónea para ser utilizada como test diagnóstico de rutina en el laboratorio.
Técnicas molecularesEn el caso de C. difficile, las técnicas de biología molecular pueden ser utilizadas para fines epidemiológicos, entender los modos de transmisión, evaluar los factores de virulencia y los mecanismos de resistencia bacteriana.
Los primeros pasos para detectar cepas toxigénicas de C. difficile se dieron a principios de los noventa, mediante la implementación de la PCR como método más sensible y rápido que permitió detectar los genes que codificaban para la expresión de las toxinas A y B99–101.
Los métodos, y algunas técnicas, más utilizados para la tipificación de cepas de C. difficile son:
- •
Métodos basados en restricción: PFGE102,103, REA103,104, toxinotipificación105.
- •
Métodos basados en amplificación: análisis de repeticiones en tándem de MLVA106, ribotipificación103,107–109.
- •
Métodos basados en secuenciación: MLST106.
Diferentes estudios han comparado varias de estas metodologías para determinar cuál de ellas presenta un mayor poder discriminatorio en cepas de C. difficile aisladas en pacientes con diarrea asociada a antibióticos provenientes de diversos hospitales. La mayoría de trabajos coinciden en que MLVA y PFGE presentan el mayor poder discriminatorio, seguidos de REA y MLST106,110–112.
La electroforesis en campo pulsado en gelLa PFGE es uno de los primeros métodos implementados para la tipificación de C. difficile, siendo considerada la técnica estándar en Canadá y Estados Unidos103.
Esta técnica emplea una enzima que se encarga de cortar y fragmentar el genoma bacteriano; se han utilizado enzimas como la Sma-I o Sac-II, dependiendo del autor. Estos fragmentos son separados en un gel de poliacrilamida que es sometido a un campo eléctrico, lo que permite a los fragmentos migrar a través del gel de acuerdo con su tamaño y ser analizados visualmente o a través de un software. Patrones de banda con una similaridad ≥ 80% les permite clasificar como un solo pulsotipo; en Norteamérica, cada pulsotipo se designa NAP y el número del tipo (por ejemplo, NAP1, NAP2, etc.)22,103.
Para citar un ejemplo de la aplicación de esta técnica, Gal et al. (2005), desarrollaron y aplicaron un protocolo de PFGE a 50 aislamientos del ribotipo 001 de C. difficile, donde la digestión con la enzima de restricción Sma-I le proporcionó bandas patrones diferentes y reproducibles102.
Análisis con endonucleasas de restricciónEl REA, al igual que en PFGE, utiliza enzimas específicas que cortan el ADN bacteriano en muchos más fragmentos que son separados por electroforesis en un gel de agarosa. La enzima más utilizada es la Hind-III, una enzima “cortadora” de 6 pb con numerosos sitios de restricción en el genoma. Este método arroja resultados bastante discriminatorios, pero el patrón de bandas obtenido es más difícil de interpretar y de reproducir comparado con la PFGE22,103. Los aislamientos que muestran 6 o menos diferencias visibles en las bandas de restricción son nombrados con el mismo grupo y designados por letras, mientras que los patrones idénticos de restricción son nombrados con números (por ejemplo, CF1, CF2)103.
En 1987, Kuijper et al., usando un análisis de digestión con enzimas de restricción, describieron en 2 pacientes con CSM adquirida en el ámbito hospitalario, patrones de restricción del ADN bacteriano muy similares a los hallados en otros aislamientos de la bacteria del mismo ambiente hospitalario, demostrando así la utilidad que tiene la técnica para estudiar la epidemiología de C. difficile113.
Clabots et al. (1993), con su grupo de investigación, desarrollaron un sistema de tipificación con REA HindIII para el ADN completo de 1.965 aislamientos de C. difficile. Los resultados obtenidos de esta colección de aislamientos permitieron organizarlos en 75 grupos, de los cuales 43 fueron citotoxina positiva, 28 citotoxina negativa y 4 grupos fueron incluidos en cepas toxigénicas y no toxigénicas. La tipificación con REA fue capaz de discriminar las diferencias obtenidas con cepas de referencia y con otros tipos de C. difficile identificados por otras técnicas, concluyendo que la técnica es sensible, discriminatoria, reproducible y rápida104. Este grupo de investigación mantiene una colección de aislamientos clínicos de C. difficile de varios sitios del mundo, obtenidos en un período de 20 años, aproximadamente. Esta colección fue importante en el momento de identificar la cepa epidémica BI/NAP1/027 y poder demostrar que ya se había aislado en el pasado, pero que otros factores favorecieron el incremento de su virulencia, como por ejemplo, la utilización de fluoroquinolonas103.
ToxinotipificaciónLa toxinotipificación es un método basado en PCR en el cual las cepas de C. difficile, de acuerdo con la longitud y patrones de restricción de 2 fragmentos (el B1 y A3) del PaLoc, son clasificadas en toxinotipos.
Rupnik et al. (1998), estudiaron en 219 aislamientos de 22 serogrupos de C. difficile los cambios en los genes que codifican para la producción de toxinas. Comparando los genes de las cepas problema con la cepa de referencia para C. difficile VPI 10463, y utilizando polimorfismos de longitud de fragmentos de restricción (RFLPs), se reconocieron 5 patrones diferentes en tcdB y 2 en tcdA. De acuerdo con los cambios y delecciones encontradas en los genes, las cepas fueron divididas en 10 grupos, del toxinotipo del I al X105. Actualmente, se conocen 27 toxinotipos diferentes (del I al XXVII)22.
Análisis de repeticiones en tándem de número variable en locus múltiplesEl análisis de repeticiones en tándem de MLVA es un método que cuenta el número de alelos repetidos en el genoma para una serie de loci conservados que son amplificados por PCR22.
Marsh et al. (2006), implementaron un MLVA para subtipificar cepas de C. difficile obtenidas de un brote en el 2001 en un hospital de cuidado terciario; los loci identificados fueron comparados con REA, encontrando el mismo tipo esperado. La técnica es un método que permite la detección de brotes de C. difficile y contribuye en el estudio de la epidemiología de la transmisión nosocomial114.
RibotipificaciónAsí como PFGE es la técnica más utilizada en Norteamérica, la ribotipificación es la más utilizada en Europa. Es el método de tipificación molecular universal de bacterias, utilizada para la discriminación de los diferentes serotipos bacterianos. El ribotipo se define como un grupo de cepas con idénticas bandas patrones, donde una diferencia en una sola banda representa un nuevo ribotipo. Esta técnica se basa en la amplificación, por medio de primers específicos, de la región espaciadora interna transcrita que se encuentra entre el rARN 16S y 23S. El resultado de estas amplificaciones son bandas entre 200 y 700 pb visualizadas, normalmente, sobre un gel de agarosa o también a través de un software46,103,108,115.
Muchas investigaciones han implementado la ribotipificación para el desarrollo de sus estudios, siendo las condiciones de Stubbs et al. (1999) o Bidet et al. (1999), las más utilizadas hasta el momento46,103,115. Stubbs et al. describieron por PCR una librería de 116 ribotipos de la bacteria, donde el tipo 001 fue el más común asociado a infecciones intrahospitalarias en el Reino Unido46.
Análisis de secuencias de locus múltiplesEl MLST es una técnica muy similar al MLVA, que permite el estudio de la epidemiología y filogenia de C. difficile. Esta técnica permite el estudio de la relación entre especies de bacterias, a través de la amplificación y secuenciación de aproximadamente 450–500 pb de fragmentos internos del genoma de, generalmente, 5 a 7 genes housekeeping. La diferencia encontrada en la secuencia de cada housekeeping se considera un alelo diferente, y cada alelo define un tipo de secuencia103.
MLST tiene la ventaja de que los tipos de secuencias arrojados no son ambiguos y fácilmente comparables a través de Internet con una base de datos de otros aislamientos103.
Tratamiento y susceptibilidad antimicrobianaEl tratamiento para el manejo de la CSM está basado en hidratación y la suspensión de los antibióticos causantes de la diarrea, esto con el fin de restaurarle al paciente la microbiota normal del TGI y su resistencia a la colonización por patógenos como C. difficile.
Desde comienzos de los años ochenta hasta la actualidad, el tratamiento con metronidazol y vancomicina ha demostrado ser efectivo contra la bacteria. Para complicaciones clínicas de la EACD, vancomicina provee una mayor respuesta a la enfermedad que el metronidazol43,116–119. El manejo de estas formas complicadas y recurrentes puede ir desde el suministro de fluidos intravenosos, vasopresores, vancomicina oral o intravenosa, metronidazol intravenoso, enemas, restablecimiento de la flora intestinal, hasta llegar, como única opción, a la colectomía22,120,121.
La susceptibilidad de la bacteria a ciertos antibióticos varía de un lugar a otro. Diversos estudios han reportado cifras diferentes de resistencia de la bacteria al metronidazol, además de cefotaxime, ciprofloxacina, moxifloxacina, levofloxacina, eritromicina, clindamicina, tetraciclina, rifampicina, imipenem y ácido fusídico122,123.
Impacto de las enfermedades asociadas a Clostridium difficile en la sociedad, salud y economíaHistóricamente, la mortalidad atribuible a infección por C. difficile ha sido baja, como un resultado directo o indirecto de infección, que ocurre aproximadamente en menos del 2% de casos. Sin embargo, los sobrecostos atribuibles a infección por C. difficile sugieren una carga sustancial sobre los sistemas de salud médica. De 1999 a 2003 en Massachusetts, se atribuyó a EACD un total de 55,380 días de hospitalización y 55,2 millones de dólares43. Se estima que en la actualidad, en EE. UU., hay 250.000 casos cada año, los cuales producen mayor estancia hospitalaria, con un costo adicional de 1,1 billones de dólares124.
Actualmente, la EACD constituye un problema creciente de salud pública en este entorno debido, principalmente, a los siguientes factores:
- a)
- b)
En la mayoría de casos, el tratamiento para la diarrea, independiente de la presencia o no de la CSM, es empírico, basado en hidratación y manejo con antimicrobianos como metronidazol y vancomicina principalmente43, lo que puede llevar a la no resolución de la enfermedad, estancias hospitalarias prolongadas y recurrencias.
- c)
La eliminación de la microbiota autóctona y el aumento de resistencia en C. difficile por el uso indiscriminado de antibióticos122,123, tanto en el ámbito hospitalario como de la comunidad.
La EACD se considera la principal enfermedad del TGI asociada al cuidado de la salud en pacientes hospitalizados > 65 años y que reciban tratamientos con antibióticos de amplio espectro. Debido al modo de transmisión de la bacteria, sus factores de virulencia, mecanismos de patogenicidad, y la aparición de posibles cepas epidémicas o resistentes a antibióticos, este tipo de enfermedad podría considerarse un problema de salud pública en las instituciones de salud.
Sin embargo, la ausencia de información e investigaciones en el medio no ha logrado evidenciar la verdadera prevalencia del microorganismo y de la EACD. Por tanto, esta revisión pretende retomar esta temática, contextualizar y dilucidar la importancia de realizar investigaciones aplicadas al futuro que permitan resolver esta problemática.
Conflicto de interesesLos autores manifiestan que no tienen conflictos de intereses para la publicación de este artículo.