Establecer la correlación entre la detección de superantígenos (MSSA) y la susceptibilidad a oxacilina de Staphylococcus aureus (MRSA) en aislamientos hospitalarios.
Materiales y métodosEn 81 aislamientos de Staphylococcus aureus de origen hospitalario, la susceptibilidad a oxacilina se estableció por un sistema automatizado y la detección de 22 genes de superantígenos fue realizada mediante PCR individual y múltiple.
ResultadosLa MRSA fue del 38,3%. Todos los aislamientos MRSA portaban uno o más genes de superantígenos; y el 92% de MSSA. El número de genotipos fue variable, pero un hallazgo relevante fue que el cluster egc solo fue detectado en MRSA (48,4%). Los genes no clásicos más detectados en MRSA fueron sem (53,1%) y seg (28,4%); y sem (37%) y seq (30,9%) para MSSA. El gen sec clásico (13,6%) fue más prevalente en MSSA, y en MRSA, los clásicos fueron de muy baja frecuencia. Para todos los genes, los genes seg, sej, sen, seo y seq mostraron diferencias estadísticamente significativas entre los aislamientos MRSA y MSSA.
ConclusiónEste estudio no permitió sacar conclusiones concluyentes para establecer la relación entre la detección de superantígenos y la susceptibilidad a oxacilina (MRSA vs. MSSA). Aunque, el número de genotipos fue variable, la presencia del cluster egc solamente en aislamientos MRSA es un hallazgo interesante, y en posteriores estudios se podría determinar la importancia del cluster egc.
To establish the relationship between the detection of superantigens (MSSA) and Staphylococcus aureus resistance to oxacillin in hospital isolates.
Material and methodsIn 81 isolates of Staphylococcus aureus of hospital origin, an oxacillin susceptibility test was performed by an automated system and 22 superantigenic genes were obtained using single and multiplex PCR.
ResultsThe MRSA was 38.3%. All MRSA isolates carried one or more genes for superantigens and 92.0% of MSSA. The numbers of genotypes for the 2 groups were variable, but the most important finding was that the egc cluster was detected only in MRSA (48.4%). The non-classic genes more often detected in MRSA were sem (53.1%) and seg (28.4%); in MSSA they were sem (37.0%) and seq (30.9%). The gen classic sec (13.6%) was more prevalent in MSSA and in MRSA; the classic genes were very low in frequency. For all genes, the genes: seg, sej, sen, seo and seq showed statistically significant differences between MRSA and MSSA isolates.
ConclusionThis study did not reveal a clear relationship between the detection of superantigens and oxacillin susceptibility (MRSA vs. MSSA). Although the number of genotypes varied, the presence of egc cluster only in the MRSA isolate was an important finding. Further studies are needed to establish the importance of the egc cluster.
Staphylococcus aureus (S. aureus) es uno de los patógenos humanos más importantes capaz de causar un amplio rango de infecciones, como infecciones de la piel y tejidos blandos, infecciones intrahospitalarias, intoxicación alimentaria, y también produce otras enfermedades más severas como síndrome de choque tóxico, endocarditis, osteomielitis, meningitis y neumonía1,2.
La patogenicidad de S. aureus es muy compleja, involucra numerosos productos bacterianos así como vías de regulación sofisticadas3. Han sido descritos muchos factores de virulencia, mecanismos de resistencia a los antibióticos, producción de exotoxinas y enzimas que contribuyen a la habilidad para colonizar y causar enfermedad1,2,4.
En Colombia, al igual que en otros países a nivel mundial, S. aureus es una causa importante de infecciones intrahospitalarias; a nivel local, el Hospital Universitario San Jorge de Pereira, Risaralda, reporta datos epidemiológicos de aislamientos de S. aureus del 9-12% entre los años 2010-2012, siendo la segunda causa de infecciones hospitalarias después de Escherichia coli.
Una de las propiedades de algunos factores de virulencia es la superantigenicidad, la cual se refiere a la capacidad de activación entre un 5-20% de linfocitos T con una producción masiva de citocinas proinflamatorias y quimiocinas que pueden producir fiebre, hipotensión y otros desórdenes que incluyen un choque potencialmente letal1,5,6, mientras que los antígenos convencionales solo activan un 0,01% de los linfocitos T. Los superantígenos incluyen enterotoxinas estafilocócicas clásicas y no clásicas (staphylococcal enterotoxins [SE]), la toxina del síndrome del choque tóxico y toxinas exfoliativas7. Varias enterotoxinas de S. aureus se incluyen en los serotipos clásicos SEA, SEB, SEC, SED y SEE. Sin embargo, técnicas moleculares han revelado enterotoxinas estrechamente relacionadas (no clásicas) donde se incluyen SEG, SEH, SEI, SEJ, SEK, SEL, SEM, SEN, SEO, SEP, SER y SEU1,4. Estos productos se han detectado en muestras de alimentos implicados en la intoxicación alimentaria y en muestras clínicas derivadas de pacientes infectados con S. aureus. Así, más de 20 genes de enterotoxinas han sido identificados junto a otras moléculas tipo enterotoxinas (enterotoxin–like molecules [SEI]) (SEA-SEIV) que conservan relaciones filogenéticas, estructura y secuencias2–4. Los superantígenos son codificados en plásmidos, bacteriófagos, islas de patogenicidad y en elementos genéticos móviles1–4. Los genes que codifican algunas enterotoxinas están físicamente agrupados; 6 genes (seg, sei, sem, sen, seo y seu) están localizados en egc (enterotoxin gene cluster)5.
La virulencia y la resistencia antimicrobiana en las cepas de S. aureus lo ha convertido en un problema de salud pública. La reconocida multirresistencia de S. aureus está generando un serio problema de salud pública a la hora de abordar el tratamiento y manejo de las infecciones estafilocócicas. Los primeros antimicrobianos utilizados en el tratamiento de las infecciones causadas por S. aureus fueron las penicilinas; la oxacilina y la meticilina fueron introducidas en la década de 1960, pero desafortunadamente a los pocos años las cepas de S. aureus se tornaron resistentes y fueron colectivamente denominados S. aureus meticilino resistentes (MRSA: methicillin-resistant S. aureus). Estas cepas portan el gen mecA, el cual se encuentra en un elemento genético móvil conocido como casete cromosómico estafilocócico mec (SCCmec: staphylococcal cassette chromosome mec).
El surgimiento de MRSA y otros antibióticos inicialmente ocurrió en los hospitales, y en tiempos más recientes han sido detectados circulando en la comunidad, aumentando la morbimortalidad en la población8,9. A nivel mundial se realiza un seguimiento epidemiológico de S. aureus resistente y lo mismo acontece en América Latina y Colombia donde existen muchos estudios relacionados con la resistencia a meticilina en S. aureus10–13.
Muchas de las cepas de S. aureus, especialmente MRSA, producen una o más exoproteínas estafilocócicas específicas, incluyendo superantígenos tipo enterotoxinas, toxina del síndrome del choque tóxico y toxinas exfoliativas7,8.
Existen diferentes métodos convencionales y moleculares para la detección de aislamientos MRSA. Así, uno de los primeros métodos de rutina para establecer la resistencia a meticilina fue la utilización de técnicas de dilución o difusión en disco para la oxacilina14. En muchos hospitales del mundo, en los paneles de los sistemas automatizados para identificación y determinación de la susceptibilidad antimicrobiana se incluye la susceptibilidad a oxacilina/cefoxitina14–16. En el caso del Hospital Universitario San Jorge de Pereira para la recolección de los aislamientos incluidos en el estudio se utilizó el sistema automatizado WalkAway/Microscan® (Dade Behring) incluyendo a la oxacilina.
La escasa información disponible en Colombia acerca del grado de variabilidad genética de los genes que codifican superantígenos con respecto a la susceptibilidad a oxacilina nos permitió plantear para nuestro medio una posible correlación entre el perfil genético de los superantígenos con la susceptibilidad a la oxacilina, considerando este método como patrón convencional de resistencia a meticilina (MRSA).
Materiales y métodosAislamientos bacterianosEn el período comprendido entre 2010 y 2012 se recolectó un total de 81 aislamientos de S. aureus del Hospital Universitario San Jorge de la ciudad de Pereira, Departamento de Risaralda, Colombia.
La identificación de los aislados clínicos y la susceptibilidad antibiótica fueron realizadas por el sistema automatizado WalkAway/Microscan® (Dade Behring) en el Hospital Universitario San Jorge, y confirmados por la PCR utilizando la pareja de iniciadores: Sa442-1 5’-AAT CTT TGT CGG TAC ACG ATA TTC TTC ACG -3’ y Sa442-2 5’-CGT AAT GAG ATT TCA GTA GAT AAT ACA ACA -3’, que amplificó una porción del gen específico de especie Sa442 de S. aureus17.
Clasificación de los aislamientos de Staphylococcus aureusLos 81 aislamientos se clasificaron de acuerdo al tipo de muestra clínica (sangre, secreciones y otros). Para determinar la presencia de aislamientos resistentes a meticilina (MRSA) y sensibles a meticilina (MSRSA: methicillin-sensitive Staphylococcus aureus) se utilizó la susceptibilidad a oxacilina, puesto que el sistema automatizado WalkAway/Microscan® no determinó la susceptibilidad a cefoxitina; y el origen hospitalario (HA: hospita-acquired) o de la comunidad (CA: community-acquired) fue establecido por el Hospital Universitario San Jorge.
Extracción de ADNLa extracción del ADN genómico se estandarizó por el método cetyltrimethylammonium bromide con lisostafina y la metodología descrita por Johnson et al.18. El ADN extraído se almacenó a −20°C en alícuotas de 20ng/μl, para su posterior amplificación por la PCR.
Identificación de los genes que codifican superantígenosLos 81 aislamientos se examinaron para detectar 22 genes de superantígenos, clasificados como genes de enterotoxinas clásicas: sea a see; gen de la toxina del síndrome de choque tóxico: tsst-1; genes de enterotoxinas no clásicas: seg a ser y seu; y los genes de las toxinas exfoliativas: eta, etb y etd, mediante el método de la PCR individual o múltiple. Se utilizaron 5 cepas de referencia como control positivo que contienen uno o más genes de los superantígenos: ATCC 700699, ATCC BAA-1707, ATCC 13565, ATCC 13566 y ATCC 19095 (FRI137). Los controles negativos fueron Streptococcus pyogenes ATCC 19615 y Pseudomonas aeruginosa ATCC 27853. Finalmente, se utilizó agua destilada estéril como control negativo de la reacción.
Las secuencias de nucleótidos para todos los iniciadores usados en este estudio y sus respectivos productos de amplificación han sido reportados previamente en la literatura19–22. Se realizaron 2 series de PCR múltiple para detectar los genes de las enterotoxinas clásicas (genes sea a see), la toxina del síndrome de choque tóxico (gen tsst-1) y las toxinas exfoliativas (genes eta y etb). La primera PCR múltiple detectó los genes de enterotoxinas sea a see y la segunda serie incluyó los genes de las toxinas exfoliativas eta y etb con la tsst-1 siguiendo la metodología descrita por Mehrotra, et al.19.
Para detectar los genes de las enterotoxinas no clásicas seg a ser y seu, y la toxina exfoliativa etd se realizaron PCR individuales; las parejas de iniciadores se describen en artículos previamente publicados19–22. Los controles positivos y negativos fueron los mismos descritos anteriormente. Los parámetros de amplificación de las PCR fueron previamente descritos por Omoe et al.20.
La detección y análisis de todos los productos amplificados se realizó por electroforesis en gel de agarosa al 2% teñido con bromuro de etidio.
Análisis estadísticoSe elaboraron tablas de contingencia y se analizaron diferentes correlaciones entre la presencia o ausencia de genes de los superantígenos frente a la susceptibilidad a la oxacilina, tipo de muestra clínica y el origen del aislamiento de S. aureus hospitalario o comunitario. Se aplicó la prueba exacta de Fisher y se consideraron estadísticamente significativos los valores de probabilidad (p≤0,05) con un intervalo de confianza del 95%.
ResultadosTodos los aislamientos de S. aureus identificados por el sistema automatizado WalkAway/Microscan® fueron confirmados por amplificación de 108pb del gen Sa442 (100%).
La distribución de los aislamientos según el género fue del 48,1% (39/81) para género masculino y del 51,9% (42/81) para género femenino.
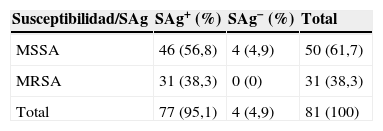
La prevalencia de los 22 genes detectados por la PCR en los 81 aislamientos de S. aureus fue del 95,1% (77/81) y en el 4,9% (4/81) no se detectó ningún gen.
La sensibilidad a oxacilina fue del 61,7% (50/81) para aislamientos MSSA, y la resistencia del 38,3% (31/81) para aislamientos MRSA. Todos los aislamientos MRSA (100%) portaban uno o más genes de superantígenos; y en el 92% de los MSSA. El número de genotipos para los 2 grupos fue variable, pero un hallazgo relevante fue que el cluster egc (genotipo gimnou) solo fue detectado en MRSA (48,4%). A nivel de detección de genes individuales, los más detectados en MRSA fueron sem (53,1%) y seg (28,4%); y para MSSA fueron sem (37%) y seq (30,9%). El gen sec (13,6%) fue más prevalente en MSSA, y en los MRSA se detectaron los clásicos con muy baja frecuencia.
En la tabla 1 se muestran los resultados de la correlación total de la detección o no de los superantígenos y la susceptibilidad a oxacilina; no hubo diferencia estadística significativa (p=0,1384).
Al comparar la susceptibilidad de la oxacilina frente al tipo de espécimen clínico, la resistencia en aislamientos de sangre fue del 6,2% (5/81), en secreciones del 19,8% (16/81) y en otros del 12,4% (10/81). Los aislamientos sensibles a oxacilina en sangre fueron del 22,2% (18/81), en secreciones del 28,4% (23/81) y en otros del 11,1% (9/81), con diferencia estadísticamente significativa (p=0,002).
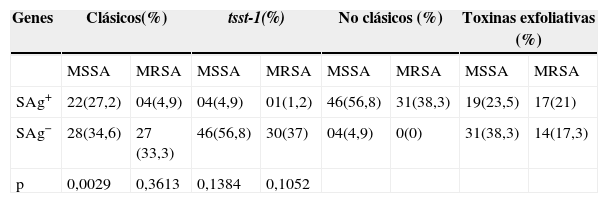
En la tabla 2 se muestran los resultados de las correlaciones entre la detección de superantígenos clásicos, toxina del síndrome del choque tóxico, superantígenos no clásicos y superantígenos de las toxinas exfoliativas frente a la susceptibilidad a oxacilina. El análisis estadístico mostró significación únicamente cuando se correlacionó con los superantígenos clásicos (p=0,0029); los demás análisis no mostraron diferencias significativas.
Correlación entre susceptibilidad a oxacilina frente a genes superantígenos: clásicos, tsst-1, no clásicos y toxinas exfoliativas
| Genes | Clásicos(%) | tsst-1(%) | No clásicos (%) | Toxinas exfoliativas (%) | ||||
|---|---|---|---|---|---|---|---|---|
| MSSA | MRSA | MSSA | MRSA | MSSA | MRSA | MSSA | MRSA | |
| SAg+ | 22(27,2) | 04(4,9) | 04(4,9) | 01(1,2) | 46(56,8) | 31(38,3) | 19(23,5) | 17(21) |
| SAg− | 28(34,6) | 27 (33,3) | 46(56,8) | 30(37) | 04(4,9) | 0(0) | 31(38,3) | 14(17,3) |
| p | 0,0029 | 0,3613 | 0,1384 | 0,1052 | ||||
El análisis estadístico de los resultados para los 22 genes analizados frente a la susceptibilidad a la oxacilina mostró que los genes seg, sej, sen, seo, seq y sec tuvieron diferencias estadísticas significativas entre los 2 grupos de aislamientos (MRSA vs. MSSA).
La distribución de la susceptibilidad a oxacilina en aislamientos HA y CA indicó que la resistencia fue mayor en los aislamientos HA (28,4%) que en los CA (9,8%) con diferencia estadísticamente significativa (p=0,0002).
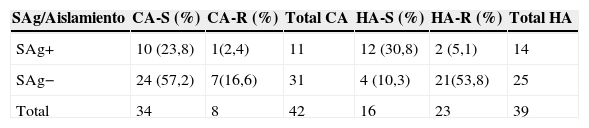
La comparación de la susceptibilidad a oxacilina y la detección de los superantígenos en los aislamientos de origen hospitalario y comunitario se muestran en la tabla 3. Los análisis estadísticos indicaron que solamente en los HA resistentes (HA-R) mostraron diferencia estadísticamente significativa (53,9% vs. 5,1%) con p=0,0001.
Correlación entre la susceptibilidad a oxacilina frente a genes de superantígenos clásicos en aislamientos de origen hospitalario o comunitario
| SAg/Aislamiento | CA-S (%) | CA-R (%) | Total CA | HA-S (%) | HA-R (%) | Total HA |
|---|---|---|---|---|---|---|
| SAg+ | 10 (23,8) | 1(2,4) | 11 | 12 (30,8) | 2 (5,1) | 14 |
| SAg− | 24 (57,2) | 7(16,6) | 31 | 4 (10,3) | 21(53,8) | 25 |
| Total | 34 | 8 | 42 | 16 | 23 | 39 |
CA-R: comunitario resistente; CA-S: comunitario sensible; HA-R: hospitalario resistente; HA-S: hospitalario sensible.
El S. aureus es un patógeno versátil capaz de causar muchos procesos infecciosos de alta morbimortalidad debido a la combinación de factores de virulencia mediada por toxinas, invasibilidad y resistencia antimicrobiana23,24.
La resistencia fenotípica a la meticilina se determina utilizando el antibiótico oxacilina y/o cefoxitina, por lo que se ha sugerido que estas cepas deberían llamarse S. aureus resistentes a oxacilina («oxacillin-resistant S. aureus», ORSA); sin embargo, es aceptado universalmente denominarlas cepas MRSA14–16. El estudio estableció que el 38,3% eran resistentes a oxacilina (MRSA) y el 61,7% sensibles a oxacilina (MSSA). La resistencia a nivel mundial es variable entre los países y aun a nivel regional o local; en Europa de acuerdo al reporte delEuropean Centre for Disease Prevention and Control, en el año 2011, la ocurrencia de MRSA fue estable o aun en decrecimiento en varios de los países europeos. El porcentaje de MRSA del 25% permaneció en 8 de los 28 que hicieron el reporte de aislamientos MRSA para el año 2011; el promedio fue de 38,3%, siendo el de menor prevalencia Noruega (0,3%) y el de mayor prevalencia Portugal (54,6%)25. En Colombia, para el 2006 fue reportada una frecuencia de MRSA del 47% con variaciones entre diferentes regiones9,26,27. La frecuencia de nuestro estudio es muy similar a la reportada en otras regiones de Colombia y Europa.
Adicionalmente a la resistencia de S. aureus a meticilina, varios reportes recientemente han analizado la prevalencia de los genes de superantígenos en aislamientos MRSA y MSSA. Sila et al.28 encontraron 7 genes más frecuentemente detectados en MRSA (sea, seb, sed, seg, sei, sej y eta), y para MSSA los genes pvl, tsst-1 y sec; el análisis estadístico de la frecuencia de comparación entre los 2 grupos (MRSA y MSSA) mostró diferencia significativa en la detección de los genes seg y sei. En nuestro estudio no fue posible ratificar este encuentro, puesto que los genes de enterotoxinas no clásicas más frecuentes fueron seg, sei, sen, seo y seu para MRSA, y para MSSA fueron seh, sej, sek, sel, sem y seq. Los genes clásicos fueron más frecuentes en MSSA que en MRSA, resultados comparables a los de Sila et al.28. Existen en la literatura diferentes combinaciones de genes, lo cual no permite sacar un patrón característico de superantígenos que caractericen a uno u otro grupo de aislamientos (MRSA vs. MSSA).
Los genes que codifican enterotoxinas no clásicas que mostraron diferencia significativa fueron: seg, sei, sen, seu y seo, pero para el total de genes no clásicos no hubo diferencia significativa. En cuanto a los genes de enterotoxinas clásicas, solo hubo diferencia significativa para el gen sec, y también existió diferencia significativa para el total de clásicas. Para los genes tsst-1 y las toxinas exfoliativas no hubo diferencia significativa. Lo anterior demuestra que, al igual que ha ocurrido con otros estudios, hasta ahora la correlación de la resistencia a oxacilina y la enterotoxigenicidad de S. aureus no ha sido claramente apoyada por los resultados publicados24. La única excepción a lo anterior es el papel de la resistencia a los antimicrobianos en la patogenicidad de S. aureus productor de enterotoxinas, que ha sido revelado bajo condiciones específicas como es el caso del desarrollo de diarrea asociada a antibióticos. La alteración de flora intestinal por la terapia antibiótica parece ser importante para la expresión de propiedades patogénicas de MRSA intestinal, capaz de sobrevivir en presencia de los antimicrobianos. Desde el punto de vista clínico, la severidad es mayor en los pacientes con diarrea asociada a antibióticos colonizados con MRSA enerotoxigénicos que en aquellos que sufren diarrea no asociada a antibióticos con MRSA o colonizados con MRSA no toxigénicos29.
En cuanto a la relación de resistencia a meticilina, la detección de los genes de superantígenos y el origen hospitalario o comunitario de los aislamientos de S. aureus, se evidenció que los genes seg, seu, sec y sea mostraron diferencia significativa dentro de los aislamientos HA, pero no hubo diferencias significativas en ninguno de los genes analizados para aislamientos CA. La anterior evidencia no establece si algunos de los genes detectados confieren especiales características de virulencia a los aislamientos HA-MRSA.
La detección del cluster egc únicamente en los aislamientos de MRSA en este estudio estaría en oposición a que la prevalencia del cluster egc en S. aureus es negativamente correlacionada con la severidad de la infección5,30. Lo anterior aumenta la controversia de la importancia del cluster egc en la virulencia del S. aureus, lo cual requiere hacer estudios comparativos entre aislamientos de diferentes orígenes (hospitalarios, comunitarios, portadores, etc.).
ConclusionesLa resistencia a meticilina (MRSA) por método fenotípico estableció que la prevalencia está acorde con la reportada en otros estudios. La presencia de superantígenos en MRSA no permite establecer un patrón claramente definido con respecto a MSSA. Sin embargo, aunque el número de genotipos para los 2 grupos de aislamientos fue variable, el hallazgo relevante del cluster egc (genotipo gimnou) en los aislamientos MRSA hace necesarios estudios adicionales de su verdadero impacto en la virulencia de S. aureus.
La relación entre resistencia a oxacilina, la detección de los genes de superantígenos y el origen hospitalario o comunitario de los aislamientos de S. aureus permitió establecer que algunos de los genes detectados mostraron diferencias significativas en los aislamientos MRSA pero no en MSSA.
Los resultados obtenidos en este estudio no permiten sacar conclusiones precisas para establecer la relación entre la detección de superantígenos y la susceptibilidad a oxacilina. Por ello se hace necesario continuar investigando este tipo de relaciones y poder finalmente establecer la verdadera asociación entre estos parámetros.
Responsabilidades éticasProtección de personas y animalesLos autores declaran que para esta investigación no se han realizado experimentos en seres humanos ni en animales.
Confidencialidad de los datosLos autores declaran que en este artículo no aparecen datos de pacientes.
Derecho a la privacidad y consentimiento informadoLos autores declaran que en este artículo no aparecen datos de pacientes.
FinanciaciónRecursos de la convocatoria interna de la Universidad Tecnológica de Pereira Risaralda Colombia
Conflicto de interesesLos autores declaran no tener conflictos de intereses.
El grupo investigador expresa los más sinceros agradecimientos a la Universidad Tecnológica de Pereira, por su generosa colaboración en el financiamiento y apoyo durante el desarrollo de la investigación y al grupo de bacteriólogas y directivas del Hospital Universitario San Jorge, E.S.E. Pereira por la donación de los aislamientos de S. aureus.