El tracto gastrointestinal abriga trillones de microorganismos, los cuales son indispensables para la salud. La microbiota gastrointestinal puede ser estudiada usando métodos de cultivo y moleculares. Las técnicas de secuenciación masiva tienen cada vez mayor aplicación debido a su alto rendimiento, costos cada vez más accesibles y la disponibilidad de software gratuito para el análisis de los datos generados. En este trabajo se revisan a detalle un gran número de artículos acerca de la microbiota gastrointestinal y su influencia en salud humana; en particular, se enfatiza la evidencia que sugiere una relación entre el ecosistema microbiano gastrointestinal y diversos procesos inmunes/inflamatorios y fisiológicos. Los trabajos analizados se discuten combinando un enfoque médico y conceptos actuales de ecología molecular microbiana. Es nuestro deseo que la presente revisión sea útil para los interesados en la microbiota gastrointestinal y su posible alteración para mantener, re-establecer y fortalecer la salud en el hospedero humano.
The gastrointestinal tract harbors trillions of microorganisms that are indispensable for health. The gastrointestinal microbiota can be studied using culture and molecular methods. The applications of massive sequencing are constantly increasing, due to their high yield, increasingly accessible costs, and the availability of free software for data analysis. The present article provides a detailed review of a large number of studies on the gastrointestinal microbiota and its influence on human health; particular emphasis is placed on the evidence suggesting a relationship between the gastrointestinal microbial ecosystem and diverse physiological and immune/inflammatory processes. Discussion of the articles analyzed combines a medical approach and current concepts of microbial molecular ecology. The present revision aims to be useful to those interested in the gastrointestinal microbiota and its possible alteration to maintain, re-establish and enhance health in the human host.
El tracto gastrointestinal (GI) de los seres humanos y otros mamíferos es un órgano complejo cuya función principal consiste en extraer y asimilar los nutrientes contenidos en los alimentos, así como proteger de amenazas biológicas y no biológicas provenientes del exterior. Desde la boca hasta el ano, más de 10trillones (>1013) de microorganismos habitan normalmente en el tracto GI de cada ser humano1. Estos microorganismos GI se diferencian evolutivamente en 3 dominios principales: Bacteria (>90%), Eukarya y Archaea. La microbiota GI bacteriana aporta indirectamente más de 9millones de genes al hospedero humano2, proporcionando así características funcionales únicas a nuestro sistema inmune y digestivo.
El tracto GI posee una microbiota autóctona que varía más entre individuos que dentro de un mismo sujeto a lo largo del tiempo3. Esta variación depende de la región anatómica específica y puede ser afectada por la edad4, la alimentación5 y el consumo de antibióticos6. A lo largo del tracto GI existe variación en la cantidad y diversidad de la microbiota. La cavidad oral de humanos contiene más de 600 especies de bacterias pertenecientes a más de 10 filos7, mientras que el estómago contiene al menos 5 diferentes filos, decenas de familias y cerca de 200 especies de bacterias8, incluyendo Helicobacter pylori, la cual es considerada como carcinógeno del grupoi y agente causal de cáncer, úlceras gástricas y duodenales9,10. El intestino delgado contiene una microbiota diversa pero poco abundante11, mientras que el intestino grueso contiene el mayor número y la mayor diversidad de grupos bacterianos de todo el tracto GI12. Desde un punto de vista fisiológico y evolutivo, es razonable encontrar una mayor diversidad y cantidad de especies bacterianas en un ambiente prácticamente estable de humedad, temperatura, y rico en nutrientes, tal y como el intestino grueso.
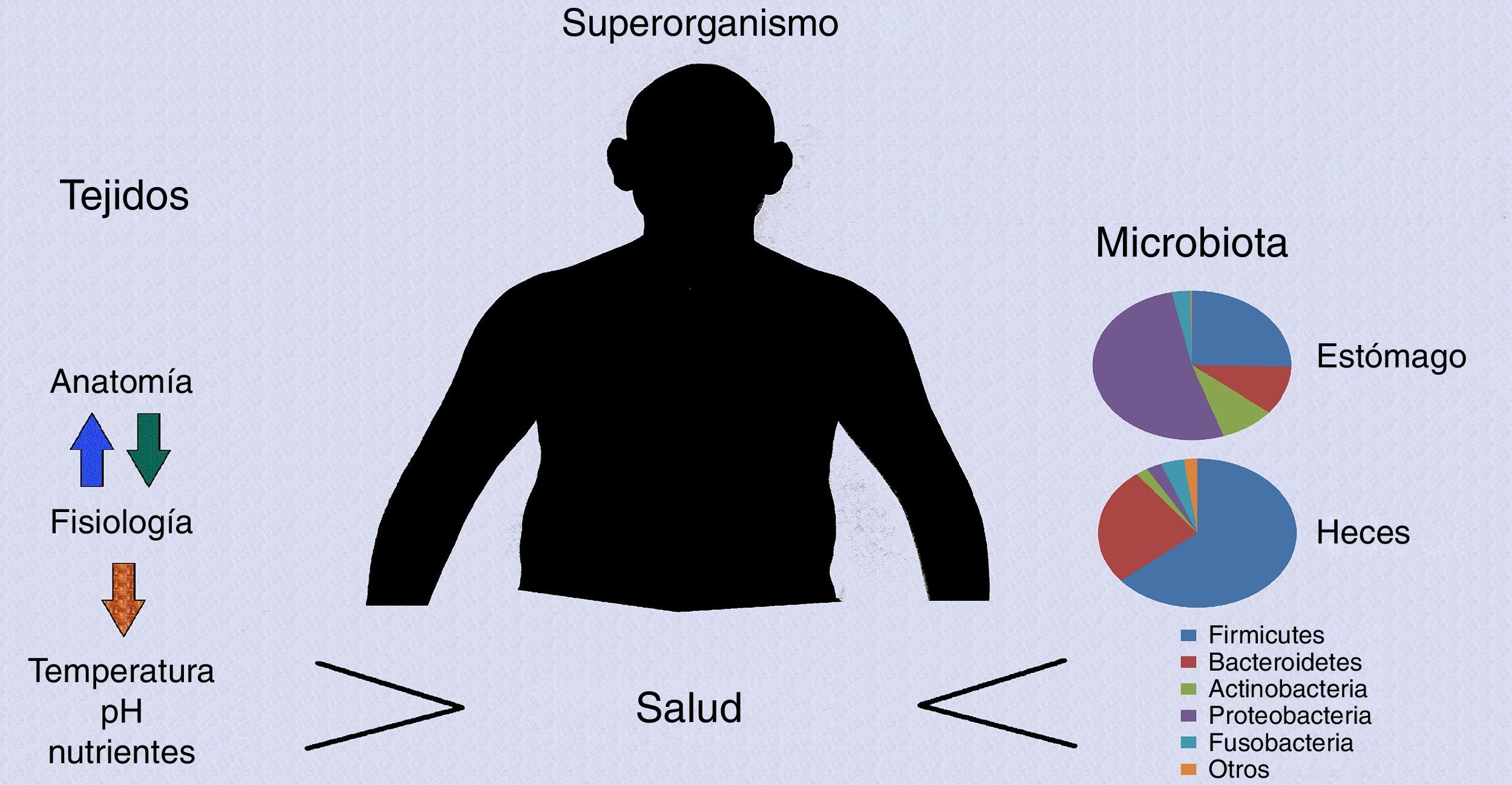
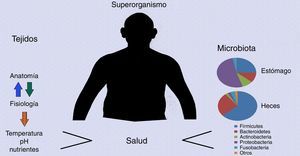
La presencia de formas de vida microscópicas en mucosas internas y externas se asocia a procesos de cooperación evolutiva: a lo largo del tiempo la asociación entre el hospedero humano y su microbiota asociada debió haber otorgado a ambos una ventaja en el ambiente, lo que permitió el éxito de su supervivencia como organismos simbiontes. Debido a que las bacterias aparecieron en la Tierra millones de años antes que cualquier célula animal o vegetal, se cree que el tracto GI de humanos y otros animales fue colonizado progresivamente por microorganismos ambientales13. Hoy en día, la microbiota GI se considera como otro sistema biológico dentro del cuerpo humano, impulsando a algunos investigadores a utilizar el término «superorganismo» (fig. 1) para definir la sólida asociación entre nuestros tejidos y los millones de virus, bacterias, archaeas y otros eucariotas microscópicos que cohabitan en ellos.
Superorganismo. El cuerpo humano (superorganismo, centro) está conformado por sus tejidos constituyentes (izquierda), cuyas características como temperatura, pH y disponibilidad de nutrientes están determinadas por la interrelación entre su anatomía y fisiología, y trillones de microorganismos (microbiota, derecha), cuya abundancia numérica supera por mucho la abundancia numérica de todas las células eucariotas que forman su hospedero. La estructura taxonómica y la actividad metabólica de la microbiota gastrointestinal varían grandemente dependiendo de la región anatómica donde habitan y de otros factores asociados con su hospedero. La salud del superorganismo depende de la homeostasis de sus tejidos y de su equilibrio con su microbiota. Las gráficas de pastel muestran la abundancia de filos bacterianos basados en Bik et al., 20068 (estómago) y Lozupone et al., 201214 (heces).
No existe duda de la gran influencia que ejerce la microbiota GI en la salud humana. Sin embargo, la información disponible acerca de este tema está dispersa en cientos de artículos científicos publicados en revistas con diversos criterios de publicación. Por otro lado, son pocos los artículos de revisión multidisciplinarios que contemplan aspectos tanto clínicos como microbiológicos de los microorganismos del tracto GI y su influencia en la salud. En vista de un creciente número de trastornos del tracto GI, hoy más que nunca la comunidad médica podría beneficiarse de una revisión de calidad con información concisa, actual y pertinente. Por lo tanto, el objetivo de este artículo de revisión fue revisar y discutir literatura pertinente acerca de la microbiota GI y su influencia en salud humana.
Caracterización de la microbiota gastrointestinalDesde hace décadas se sabe que solo una pequeña proporción de los microorganismos del tracto GI se pueden cultivar in vitro15. Esto se debe principalmente a la incapacidad de simular con precisión las condiciones de vida de los microorganismos en su hábitat natural. Una de las grandes desventajas de las técnicas de cultivo es que es imposible asegurar que el comportamiento fenotípico de un microorganismo en el laboratorio sea idéntico a su comportamiento in vivo, debido en parte a procesos de cooperación metabólica entre los microorganismos. Algunos investigadores han intentado cultivar a la mayoría de los microorganismos usando «culturómica», un término relativamente nuevo para describir la caracterización de microorganismos en gran escala usando métodos de cultivo16,17. Sin embargo, a pesar de los esfuerzos por estudiar a los microorganismos desde un punto de vista fenotípico, el uso de técnicas moleculares (ver abajo) permite caracterizar de manera más comprensiva a las comunidades microbianas en el tracto GI, así como en otros ambientes naturales18.
La principal diferencia entre los métodos tradicionales de cultivo y los métodos moleculares radica en la manera de caracterizar o identificar a los microorganismos. Mientras que los primeros intentan recrear las condiciones de vida naturales del microorganismo para que este pueda crecer in vitro y así poder estudiar su fenotipo, los métodos moleculares buscan la identificación de los organismos en base a similitudes genéticas, más comúnmente en el gen que codifica para la subunidad 16S del ARN ribosomal (gen 16S ARNr; ver Baker et al., 2003)19. Esto es posible debido a que este gen, de aproximadamente 1.542 nucleótidos de longitud19, ha cambiado relativamente poco a lo largo de la evolución, presumiblemente debido a su importancia para la supervivencia de las bacterias en relación con la producción de proteínas. A lo largo del tiempo, diferentes procesos evolutivos sobre la subunidad 16S del ARNr han originado la presencia de regiones conservadas (secuencias de nucleótidos idénticas en casi todos los tipos bacterianos) y regiones variables e hipervariables (secuencias de nucleótidos que varían entre diferentes grupos de bacterias), las cuales sirven para identificar y catalogar microorganismos en diferentes linajes filogenéticos, permitiendo así su estudio y análisis, por ejemplo, en procesos de salud y enfermedad. Otros genes que se han utilizado para catalogar a la microbiota GI incluyen genes que codifican para chaperoninas20 y la subunidad β de la ARN polimerasa21.
El desarrollo de técnicas de secuenciación masiva ha impulsado el descubrimiento de una diversidad de asociaciones entre microorganismos nunca antes imaginadas. Sin embargo, es importante recordar que la información evolutiva contenida en el gen 16S ARNr puede no ser representativa de la información evolutiva disponible en otras partes del genoma. En otras palabras, el patrón de sustitución de nucleótidos que ha sufrido el gen 16S ARNr puede ser diferente en otros genes (por ejemplo, chaperoninas) y por lo tanto su análisis puede arrojar resultados diferentes22. Aun así, una publicación reciente sugiere que los resultados obtenidos del gen 16S ARNr pueden extrapolarse al genoma completo y por lo tanto al perfil metabólico del microorganismo23. Independientemente del gen que se utilice para establecer relaciones evolutivas y taxonómicas entre las bacterias, las bases actuales de taxonomía microbiana dependen en gran parte de análisis filogenéticos moleculares, los cuales dependen de regiones conservadas de nucleótidos para lograr un alineamiento de secuencias no ambiguo. Esta es la razón principal de usar el gen 16S ARNr o cualquier otro gen conservado en el genoma.
Actualmente la caracterización de la microbiota GI se lleva a cabo en gran parte empleando técnicas moleculares. A continuación se describen brevemente las técnicas moleculares más utilizadas. Es importante notar que el término «especie» es controversial en la microbiología molecular moderna24, pero aun así es utilizado a lo largo de este trabajo para referirse a grupos relativamente emparentados de microorganismos. Por otro lado, el concepto de unidad taxonómica operacional (OTU, por sus siglas en inglés) se refiere a la asignación de secuencias (por ejemplo del 16S) a unidades taxonómicas específicas (por ejemplo familias, géneros y especies) basado en porcentajes de similitud entre secuencias de nucleótidos. El uso más común del concepto de OTU son aquellas secuencias del 16S que comparten el 97% o más de similitud con otra secuencia de referencia, las cuales son asignadas a la misma especie de microorganismos (OTU al 97%). Prácticamente todas las publicaciones donde se emplean secuencias de 16S de microorganismos utilizan el concepto de OTU, aunque su uso es controversial en taxonomía microbiana molecular.
Técnicas molecularesReacción en cadena de la polimerasaLa reacción en cadena de la polimerasa (PCR, por sus siglas en inglés) utiliza la enzima ADN polimerasa para añadir nucleótidos complementarios en hebras simples de ADN y por lo tanto replicar una secuencia específica de ADN. La acción de la polimerasa ocurre de manera sucesiva y exponencial, por lo que se tienen como resultado millones de copias del fragmento genético de interés (por ejemplo, una región semi-conservada del gen 16S). Los fragmentos amplificados de ADN pueden ser visualizados en geles de agarosa. Este tipo de PCR es conocido como PCR de punto final y su resultado es cualitativo, ya que indica la presencia o ausencia del fragmento de ADN. A pesar del gran número de microorganismos presentes en el tracto GI, la cantidad de ADN de cada grupo bacteriano es muy poca para su detección y análisis posterior, por lo que el PCR es una técnica conveniente para generar copias de ADN en cantidades suficientes que permitan el estudio de la microbiota GI.
PCR tiempo-realEl PCR tiempo-real es una técnica basada en el principio de amplificación exponencial como la PCR de punto final; sin embargo, la técnica de PCR tiempo-real permite cuantificar la presencia de fragmentos genéticos en las muestras. Esto se debe a la incorporación de moléculas fluorescentes en la reacción de PCR capaces de medir la amplificación de ADN en tiempo real25. Una desventaja de esta técnica es que el resultado que se obtiene son copias de ADN amplificadas, y se ha demostrado que las bacterias tienen diferente número de copias del gen 16S ARNr a nivel de cepa26. Además, estas copias del gen 16S dentro de un mismo genoma no necesariamente poseen la misma composición de nucleótidos27. Esto se traduce en la posibilidad de cuantificar incorrectamente el número de microorganismos usando PCR tiempo-real porque los resultados no pueden ser directamente extrapolados al número de organismos presentes en la muestra.
Hibridación fluorescente in situLa hibridación fluorescente in situ (FISH, por sus siglas en inglés) se utiliza para el estudio de la microbiota GI desde la década de 199028,29. A diferencia de la PCR tiempo-real, la técnica de FISH, aunque técnicamente más laboriosa, permite obtener una mejor estimación de los números reales de microorganismos y así ayudar a determinar con más precisión el papel que tienen los mismos en procesos de salud y enfermedad30.
Técnicas basadas en patrón de bandasEl resultado de utilizar la técnica de PCR usando una muestra de contenido intestinal son millones de copias de ADN provenientes de diferentes grupos microbianos. Estos productos de PCR (por ejemplo, del gen 16S ARNr) difieren entre ellos en su composición específica de nucleótidos. Estas diferencias en nucleótidos pueden ser distinguidas usando desnaturalizadores de ADN: a diferencia de las cadenas con un porcentaje mayor de adeninas y timinas, las cadenas con mayor porcentaje de guaninas y citocinas necesitan concentraciones más altas del agente desnaturalizador o más temperatura para desnaturalizarse. Dos técnicas basadas en este principio son la electroforesis en gel con gradiente de agente desnaturalizador y con gradiente de temperatura (DGGE y TGGE, por sus siglas en inglés, respectivamente). Aunque actualmente el uso de DGGE y/o TGGE no es recomendable debido a la incapacidad de estas técnicas para separar con precisión los productos de PCR31, diversos estudios han demostrado su utilidad en el área de probióticos32 y enfermedad inflamatoria intestinal33 (EII).
MicroarreglosLa secuenciación de genes provenientes de microorganismos del tracto GI ha originado una gran cantidad de secuencias genéticas disponibles para la comunidad científica. Esta información puede ser usada para diseñar oligonucleótidos capaces de hibridar al gen 16S ARNr, por ejemplo en la identificación de grupos microbianos específicos por medio de PCR. De manera similar, la información genética puede ser usada para diseñar oligonucleótidos capaces de ser hibridados por secuencias desconocidas presentes en una muestra biológica. Las placas de microarreglos consisten en un grupo de oligonucleótidos conocidos adheridos a una superficie sólida, los cuales son capaces de hibridar con secuencias en la muestra de interés. Esta técnica se ha usado en varias aplicaciones y su uso es común en el estudio de la ecología microbiana del tracto GI de humanos34,35. La principal desventaja de los microarreglos radica en el número y en los tipos de oligonucleótidos contenidos en la placa, ya que las secuencias que no se encuentren representadas en el microarreglo no podrán ser detectadas en la muestra. Esto es particularmente relevante para organismos con gran potencial para modular salud intestinal pero que históricamente han sido catalogados como desconocidos, tales como Christensenella minuta36,37. Sin embargo, algunos estudios han mostrado que los microarreglos y algunos tipos de secuenciación masiva ofrecen perfiles taxonómicos similares35.
Secuenciación clásicaLa fuente principal de información acerca de los microorganismos está resguardada en el orden de los nucleótidos de sus genes. Actualmente se conoce el orden de nucleótidos del gen del 16S ARNr en diferentes grupos bacterianos, los cuales se encuentran disponibles en bases de datos públicas, gratuitas y de gran calidad (por ejemplo, la Ribosomal Database Project, GreenGenes y Silva). La técnica de Sanger fue una de las primeras tecnologías usadas para descifrar el orden de nucleótidos en una secuencia genética38, y aún es utilizada en muchos laboratorios.
Biblioteca de clonasPara secuenciar por la técnica de Sanger, generalmente se crean bibliotecas o grupos de clones donde cada clon acarrea un tipo de secuencia proveniente de un tipo de microorganismo. La construcción de estas bibliotecas involucra la inserción de un plásmido conteniendo el fragmento genético de interés (por ejemplo, el gen 16S ARNr) en células de Escherichia coli de tal manera que cada célula bacteriana adquiera solamente un plásmido. Al crecer las E.coli en medio de cultivo semisintético, cada colonia poseerá una copia del gen en cuestión. En parte debido a las nuevas técnicas disponibles y al gran número de grupos microbianos, las bibliotecas de clonas se han utilizado menos frecuentemente en los últimos años. Sin embargo, esta técnica sigue siendo una buena alternativa para aquellos interesados en secuenciar los ∼1.500 nucleótidos del gen 16S ARN ribosomal.
Secuenciación masivaDurante los últimos años se han desarrollado varias tecnologías de secuenciación masiva para hacer más eficiente y menos costoso el uso de la secuenciación39. Entre las técnicas de secuenciación masiva más utilizadas y con mayor potencial de uso se encuentran Illumina40 e Ion Torrent41. Los datos generados, que pueden ser miles o millones de secuencias, se pueden analizar con paquetes computacionales gratuitos, como QIIME42 y Mothur43.
Otras tecnologíasOtras tecnologías moleculares disponibles para estudiar a la microbiota GI incluyen la metaproteómica44, el uso de isótopos estables para estudiar la utilización microbiana de sustratos específicos45, la citometría de flujo46 y el análisis de polimorfismos de longitud de fragmentos de restricción47. Es importante recordar que las conclusiones y la relevancia de los diferentes estudios acerca de la microbiota GI (ver abajo) están determinadas por la naturaleza de los métodos utilizados.
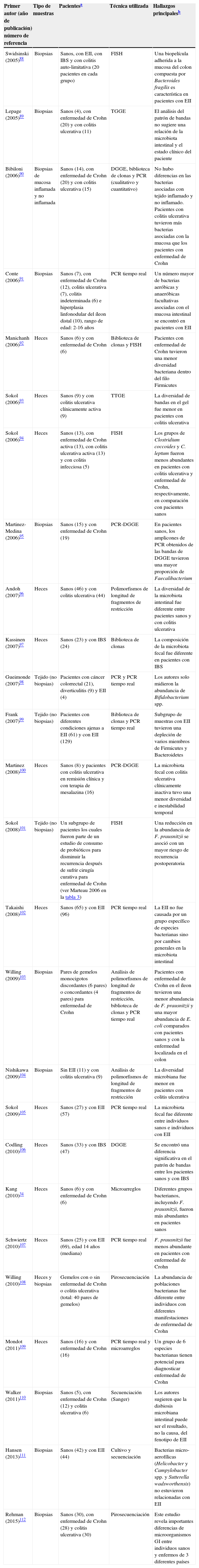
Microbiota gastrointestinal y su influencia en la salud y la enfermedad humanaLas siguientes tablas presentan un resumen de las publicaciones revisadas de mayor relevancia. La tabla 1 resume artículos de revisión principalmente acerca de la relación entre la microbiota intestinal y la salud del ser humano; la tabla 2 se enfoca en estudios clínicos de pacientes con EII o padecimientos similares, y la tabla 3 muestra otros aspectos importantes de interés clínico. A continuación se presenta un extracto de las ideas fundamentales mostradas en las tablas.
Resumen de artículos de revisión organizados por año de publicación
| Primer autor (año de publicación) número de referencia | Tema revisado/discutido | Comentarios |
|---|---|---|
| Hill (1975)48 | Ácidos biliares, bacterias intestinales y cáncer de colon | Uno de los primeros artículos que discuten la relación entre el metabolismo microbiano intestinal, los ácidos biliares y el cáncer de colon |
| Roediger (1980)49 | Bacterias anaeróbicas y colitis | Uno de los primeros artículos que discuten la posible relación entre las bacterias anaeróbicas, el colon y la colitis |
| Scheppach (1997)50 | Tratamiento de la mucosa inflamada del colon con ácidos grasos de cadena corta | Se discute la posibilidad de tratar pacientes con colitis usando enemas con ácidos grasos de cadena corta |
| Salminen (1998)51 | Inocuidad de los probióticos | Algunos autores del artículo eran empleados de compañías nutracéuticas |
| Cook (1998)52 | Ácidos grasos de cadena corta en salud y enfermedad | Los autores discuten varios trastornos intestinales, así como el metabolismo de los ácidos grasos de cadena corta |
| Apostolou (2001)53 | Propiedades de adhesión de probióticos y riesgo de bacteriemia | Los autores discuten la posibilidad de que una mejor adhesión al moco intestinal de algunos probióticos pueda representar un riesgo de bacteriemia |
| Salminen (2002)54 | Consumo de probióticos y bacteriemia | En Finlandia, un uso incrementado del probiótico L. rhamnosus cepa GG no ha originado un incremento en casos de bacteriemia causados por Lactobacillus |
| Vaarala (2003)55 | Efectos inmunológicos de probióticos, especialmente Lactobacillus spp. | El autor ofrece una perspectiva interesante desde un punto de vista pediátrico |
| Mai (2004)56 | Herramientas moleculares para estudiar a la microbiota intestinal | Los autores discuten diferentes técnicas y factores asociados con la microbiota desde un punto de vista molecular |
| Marteau (2004)57 | Microbiota intestinal y EII | Los autores incluyen una tabla útil con diferentes investigaciones acerca del uso de probióticos en EII |
| Sartor (2004)58 | Manipulación terapéutica de la microbiota intestinal en la EII | El autor incluye una tabla muy útil que resume varios estudios donde se han administrado antibióticos para tratar la EII |
| Tamboli (2004)59 | Disbiosis (disrupción en el balance de bacterias intestinales protectoras y dañinas) | El término disbiosis es ambiguo debido a que es muy difícil determinar qué bacterias son benéficas y cuáles son malignas |
| Thompson-Chagoyan (2005)60 | Relación entre la microbiota intestinal y el sistema inmune del intestino | Los autores ofrecen una buena perspectiva acerca de la posibilidad de una respuesta inmune anormal en contra de la microbiota intestinal |
| Egert (2006)61 | Función metabólica de la microbiota intestinal | La función metabólica de la microbiota intestinal es fundamental para entender su papel en salud o enfermedad. Este articulo ofrece una perspectiva interesante al respecto |
| O’Hara (2007)62 | Potencial terapéutico de cambios en la microbiota intestinal | El articulo hace énfasis en procesos como EII |
| Guarner (2007)63 | Prebióticos y EII | El autor ofrece una revisión concisa pero detallada del uso de prebióticos en EII |
| Goyette (2007)64 | Patogénesis molecular de la EII | Los autores implican a la EII con regiones genómicas y genes específicos |
| Xavier (2007)65 | Patogénesis de la EII | Los autores discuten 3 posibles tópicos responsables por la patogénesis de EII |
| Sheil (2007)66 | Efecto de probióticos en la EII | Síntesis de los efectos de probióticos (un tópico controversial, ver Bessenlink et al. En la tabla 3) en la EII |
| Eckburg (2007)67 | Microbiota intestinal y enfermedad de Crohn | Excelente discusión concisa acerca del posible papel de microorganismos en la enfermedad de Crohn |
| Peterson (2008)68 | Métodos moleculares para estudiar la microbiota intestinal y definir su relación con la salud EII | Excelente trabajo de revisión acerca del uso de tecnologías moleculares para estudiar la microbiota intestinal. Los autores también incluyen una lista interesante de áreas por estudiar |
| Issa (2008)69 | Relación entre Clostridium difficile e EII | Son pocos los artículos que hablan del tema de manera tan extensa |
| Sokol (2008)70 | Relación entre la microbiota intestinal y EII | Los autores han participado en diferentes investigaciones que han analizado comunidades microbianas en pacientes con EII |
| Khoshini (2008)71 | Sobrecrecimiento bacteriano en el intestino delgado (SIBO por sus siglas en inglés) | Los autores concluyen que no existen pruebas diagnósticas confiables para diagnosticar SIBO |
| Turroni (2008)72 | Diversidad de la microbiota intestinal y su contribución al funcionamiento del intestino | Se discuten aspectos de composición y funcionalidad de la microbiota intestinal con énfasis en Bifidobacterium spp. |
| Andoh (2009)73 | Tecnologías moleculares para estudiar la relación entre la microbiota intestinal y EII | Los autores discuten diferentes herramientas moleculares para estudiar microorganismos gastrointestinales en contexto de la EII |
| Dupont (2009)74 | Efectos antidiarreicos de arcillas minerales | Los autores describen la fisiopatología de la diarrea y los posibles mecanismos de acción de las arcillas minerales para contrarrestarla en niños con diarrea aguda |
| Round (2009)75 | Relación entre la microbiota intestinal y el sistema inmune intestinal | Se discute la posibilidad de que el sistema inmune del intestino es controlado por microorganismos, no para controlar a los microorganismos |
| Hill (2010)76 | La influencia de las señales de la microbiota intestinal en la homeostasis del sistema inmune | Uno de los mejores y más detallados trabajos de revisión acerca del tema |
| Luther (2010)77 | Relación entre la presencia de H. pylori en el estómago y la EII | Metaanálisis y revisión de literatura. Los resultados sugieren que la presencia de H. pylori puede tener un efecto benéfico en contra del desarrollo de la EII |
| Wlodarska (2010)78 | Respuesta inmune y alteración de la microbiota intestinal debida a antibióticos | Los autores discuten brevemente los cambios inmunológicos que se producen por la administración de antibióticos y su efecto en la microbiota intestinal |
| O’Toole (2010)4 | Cambios de la microbiota intestinal a lo largo de la vida de un individuo | Se discuten los cambios que sufre la microbiota intestinal desde la infancia hasta la tercera edad |
| Haller (2010)79 | Nutrigenómica | Se discute el impacto del estudio de la nutrición y su relación con la microbiota intestinal |
| Nell (2010)80 | Utilidad de modelos en ratones para estudiar procesos como la EII | Se discute el uso de ratones para estudiar la relación entre la microbiota intestinal y la EII |
| Hörmannsperger (2010)81 | Relevancia clínica de la comunicación entre bacterias probióticas y el sistema inmune intestinal | El artículo es discutido en el contexto de la EII |
| Chassaing (2011)82 | Relación entre microbiota residente y enteropatógenos en la patogénesis de EII | Los autores expanden las posibilidades de una relación entre microorganismos intestinales e EII argumentando la presencia de enteropatógenos |
| Fava (2011)83 | Relación entre la microbiota intestinal y la patogénesis de EII | Se discute la posible relación entre la microbiota intestinal y la aparición de la EII |
| Manichanh (2012)84 | Relación de la microbiota intestinal con EII | Se discute que los cambios en la microbiota intestinal pueden ser la causa y la consecuencia de la EII |
| Bushman (2013)85 | Relación entre la dieta, la microbiota intestinal y la EII | Se discute brevemente la relación potencial entre la dieta, la microbiota intestinal y procesos inflamatorios como la EII |
| Viladomiu (2013)86 | Mecanismos nutricionales con el potencial de proteger contra la inflamación intestinal | El articulo incluye el uso de probióticos y prebióticos para contrarrestar procesos inflamatorios (un tópico controversial, ver Bessenlink et al. en la tabla 3) |
| Sharon (2014)87 | Metabolitos producidos por la microbiota GI | Excelente referencia acerca de la síntesis, absorción y efectos fisiológicos de los miles de metabolitos producidos por los microorganismos GI |
EII: enfermedad inflamatoria intestinal
Resumen de las publicaciones revisadas con mayor relevancia clínica acerca de la relación entre la microbiota intestinal y diferentes manifestaciones de la enfermedad inflamatoria intestinal. Las publicaciones están organizadas por año de publicación
| Primer autor (año de publicación) número de referencia | Tipo de muestras | Pacientesa | Técnica utilizada | Hallazgos principalesb |
|---|---|---|---|---|
| Swidsinski (2005)88 | Biopsias | Sanos, con EII, con IBS y con colitis auto-limitativa (20 pacientes en cada grupo) | FISH | Una biopelícula adherida a la mucosa del colon compuesta por Bacteroides fragilis es característica en pacientes con EII |
| Lepage (2005)89 | Biopsias | Sanos (4), con enfermedad de Crohn (20) y con colitis ulcerativa (11) | TGGE | El análisis del patrón de bandas no sugiere una relación de la microbiota intestinal y el estado clínico del paciente |
| Bibiloni (2006)90 | Biopsias de mucosa inflamada y no inflamada | Sanos (14), con enfermedad de Crohn (20) y con colitis ulcerativa (15) | DGGE, biblioteca de clonas y PCR (cualitativo y cuantitativo) | No hubo diferencias en las bacterias asociadas con tejido inflamado y no inflamado. Pacientes con colitis ulcerativa tuvieron más bacterias asociadas con la mucosa que los pacientes con enfermedad de Crohn |
| Conte (2006)91 | Biopsias | Sanos (7), con enfermedad de Crohn (12), colitis ulcerativa (7), colitis indeterminada (6) e hiperplasia linfonodular del íleon distal (10), rango de edad: 2-16 años | PCR tiempo real | Un número mayor de bacterias aeróbicas y anaeróbicas facultativas asociadas con el mucosa intestinal se encontró en pacientes con EII |
| Manichanh (2006)92 | Heces | Sanos (6) y con enfermedad de Crohn (6) | Biblioteca de clonas y FISH | Pacientes con enfermedad de Crohn tuvieron una menor diversidad bacteriana dentro del filo Firmicutes |
| Sokol (2006)93 | Heces | Sanos (9) y con colitis ulcerativa clínicamente activa (9) | TTGE | La diversidad de bandas en el gel fue menor en pacientes con colitis ulcerativa |
| Sokol (2006)94 | Heces | Sanos (13), con enfermedad de Crohn activa (13), con colitis ulcerativa activa (13) y con colitis infecciosa (5) | FISH | Los grupos de Clostridium coccoides y C. leptum fueron menos abundantes en pacientes con colitis ulcerativa y enfermedad de Crohn, respectivamente, en comparación con pacientes sanos |
| Martinez-Medina (2006)95 | Biopsias | Sanos (15) y con enfermedad de Crohn (19) | PCR-DGGE | En pacientes sanos, los amplicones de PCR obtenidos de las bandas de DGGE tuvieron una mayor proporción de Faecalibacterium |
| Andoh (2007)96 | Heces | Sanos (46) y con colitis ulcerativa (44) | Polimorfismos de longitud de fragmentos de restricción | La diversidad de la microbiota intestinal fue diferente entre pacientes sanos y con colitis ulcerativa |
| Kassinen (2007)97 | Heces | Sanos (23) y con IBS (24) | Biblioteca de clonas | La composición de la microbiota fecal fue diferente en pacientes con IBS |
| Gueimonde (2007)98 | Tejido (no biopsias) | Pacientes con cáncer colorrectal (21), diverticulitis (9) y EII (4) | PCR y PCR tiempo real | Los autores solo midieron la abundancia de Bifidobacterium spp. |
| Frank (2007)99 | Tejido (no biopsias) | Pacientes con diferentes condiciones ajenas a EII (61) y con EII (129) | Biblioteca de clonas y PCR tiempo real | Subgrupo de muestras con EII tuvieron una depleción de varios miembros de Firmicutes y Bacteroidetes |
| Martinez (2008)100 | Heces | Sanos (8) y pacientes con colitis ulcerativa en remisión clínica y con terapia de mesalazina (16) | PCR-DGGE | La microbiota fecal con colitis ulcerativa clínicamente inactiva tuvo una menor diversidad e inestabilidad temporal |
| Sokol (2008)101 | Tejido (no biopsias) | Un subgrupo de pacientes los cuales fueron parte de un estudio de consumo de probióticos para disminuir la recurrencia después de sufrir cirugía curativa para enfermedad de Crohn (ver Marteau 2006 en la tabla 3) | FISH | Una reducción en la abundancia de F. prausnitzii se asoció con un mayor riesgo de recurrencia postoperatoria |
| Takaishi (2008)102 | Heces | Sanos (65) y con EII (96) | PCR tiempo real | La EII no fue causada por un grupo específico de especies bacterianas sino por cambios generales en la microbiota intestinal |
| Willing (2009)103 | Biopsias | Pares de gemelos monocigotos discordantes (6 pares) o concordantes (4 pares) para enfermedad de Crohn | Análisis de polimorfismos de longitud de fragmentos de restricción, biblioteca de clonas y PCR tiempo real | Pacientes con enfermedad de Crohn en el íleon tuvieron una menor abundancia de F. prausnitzii y una mayor abundancia de E. coli comparados con pacientes sanos y con la enfermedad localizada en el colon |
| Nishikawa (2009)104 | Biopsias | Sin EII (11) y con colitis ulcerativa (9) | Análisis de polimorfismos de longitud de fragmentos de restricción | La diversidad microbiana fue menor en pacientes con colitis ulcerativa |
| Sokol (2009)105 | Heces | Sanos (27) y con EII (57) | PCR tiempo real | La microbiota fecal fue diferente entre individuos sanos e individuos con EII |
| Codling (2010)106 | Heces | Sanos (33) y con IBS (47) | DGGE | Se encontró una diferencia significativa en el patrón de bandas entre los pacientes sanos y con IBS |
| Kang (2010)34 | Heces | Sanos (6) y con enfermedad de Crohn (6) | Microarreglos | Diferentes grupos bacterianos, incluyendo F. prausnitzii, fueron más abundantes en pacientes sanos |
| Schwiertz (2010)107 | Heces | Sanos (25) y con EII (69), edad 14 años (mediana) | PCR tiempo real | F. prausnitzii fue menos abundante en pacientes con enfermedad de Crohn |
| Willing (2010)108 | Heces y biopsias | Gemelos con o sin enfermedad de Crohn o colitis ulcerativa (total: 40 pares de gemelos) | Pirosecuenciación | La abundancia de poblaciones bacterianas fue diferente entre individuos con diferentes manifestaciones de enfermedad de Crohn |
| Mondot (2011)109 | Heces | Sanos (16) y con enfermedad de Crohn (16) | PCR tiempo real y microarreglos | Un grupo de 6 especies bacterianas tienen potencial para diagnosticar enfermedad de Crohn |
| Walker (2011)110 | Biopsias | Sanos (5), con enfermedad de Crohn (12) y colitis ulcerativa (6) | Secuenciación (Sanger) | Los autores sugieren que la disbiosis microbiana intestinal puede ser el resultado, no la causa, del fenotipo de EII |
| Hansen (2013)111 | Biopsias | Sanos (42) y con EII (44) | Cultivo y secuenciación | Bacterias micro-aerofílicas (Helicobacter y Campylobacter spp. y Sutterella wadsworthensis) no estuvieron relacionadas con EII |
| Rehman (2015)112 | Biopsias | Sanos (30), con enfermedad de Crohn (28) y colitis ulcerativa (30) | Pirosecuenciación | Este estudio revela importantes diferencias de microorganismos GI entre individuos sanos y enfermos de 3 diferentes países |
DGGE: gel electroforesis basado en un gradiente desnaturalizador de ADN; EII: enfermedad inflamatoria intestinal; FISH: hibridación fluorescente in situ; IBS: síndrome del intestino irritable; TGGE: gel electroforesis basado en un gradiente de temperatura; TTGE: gel electroforesis basado en un gradiente temporal de temperatura
Por favor, revise el texto principal para más información acerca de las ventajas y desventajas de cada una de estas técnicas moleculares para estudiar el ecosistema microbiano gastrointestinal.
Esta columna puede también contener especulaciones de los autores basadas en los hallazgos encontrados. Por ejemplo, analizar solamente un número limitado de grupos de microorganismos y referirse a toda la microbiota. Se recomienda revisar las referencias originales para detalles específicos acerca de los hallazgos principales u otra característica del estudio.
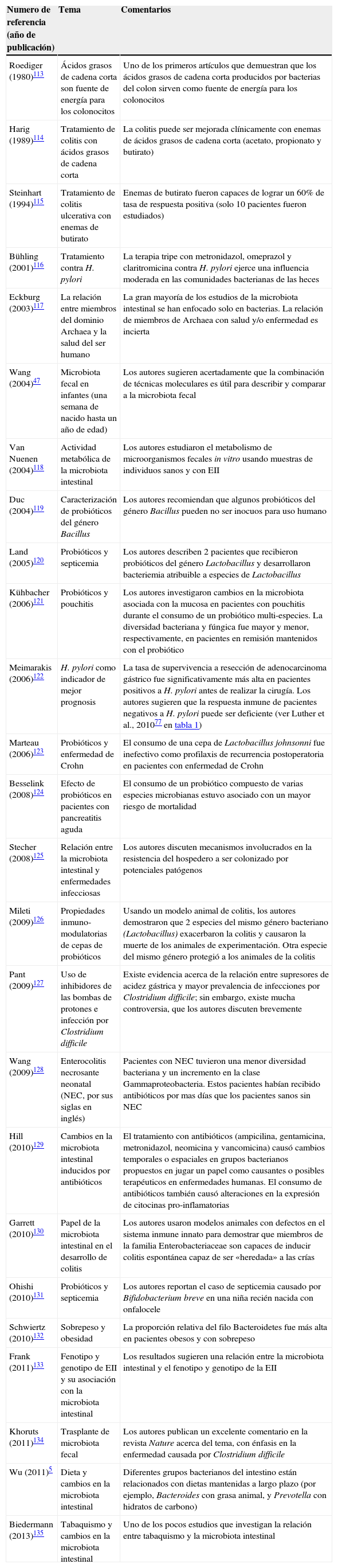
Resumen de otros artículos de interés para la comunidad gastroenteróloga organizados por año de publicación
| Numero de referencia (año de publicación) | Tema | Comentarios |
|---|---|---|
| Roediger (1980)113 | Ácidos grasos de cadena corta son fuente de energía para los colonocitos | Uno de los primeros artículos que demuestran que los ácidos grasos de cadena corta producidos por bacterias del colon sirven como fuente de energía para los colonocitos |
| Harig (1989)114 | Tratamiento de colitis con ácidos grasos de cadena corta | La colitis puede ser mejorada clínicamente con enemas de ácidos grasos de cadena corta (acetato, propionato y butirato) |
| Steinhart (1994)115 | Tratamiento de colitis ulcerativa con enemas de butirato | Enemas de butirato fueron capaces de lograr un 60% de tasa de respuesta positiva (solo 10 pacientes fueron estudiados) |
| Bühling (2001)116 | Tratamiento contra H. pylori | La terapia tripe con metronidazol, omeprazol y claritromicina contra H. pylori ejerce una influencia moderada en las comunidades bacterianas de las heces |
| Eckburg (2003)117 | La relación entre miembros del dominio Archaea y la salud del ser humano | La gran mayoría de los estudios de la microbiota intestinal se han enfocado solo en bacterias. La relación de miembros de Archaea con salud y/o enfermedad es incierta |
| Wang (2004)47 | Microbiota fecal en infantes (una semana de nacido hasta un año de edad) | Los autores sugieren acertadamente que la combinación de técnicas moleculares es útil para describir y comparar a la microbiota fecal |
| Van Nuenen (2004)118 | Actividad metabólica de la microbiota intestinal | Los autores estudiaron el metabolismo de microorganismos fecales in vitro usando muestras de individuos sanos y con EII |
| Duc (2004)119 | Caracterización de probióticos del género Bacillus | Los autores recomiendan que algunos probióticos del género Bacillus pueden no ser inocuos para uso humano |
| Land (2005)120 | Probióticos y septicemia | Los autores describen 2 pacientes que recibieron probióticos del género Lactobacillus y desarrollaron bacteriemia atribuible a especies de Lactobacillus |
| Kühbacher (2006)121 | Probióticos y pouchitis | Los autores investigaron cambios en la microbiota asociada con la mucosa en pacientes con pouchitis durante el consumo de un probiótico multi-especies. La diversidad bacteriana y fúngica fue mayor y menor, respectivamente, en pacientes en remisión mantenidos con el probiótico |
| Meimarakis (2006)122 | H. pylori como indicador de mejor prognosis | La tasa de supervivencia a resección de adenocarcinoma gástrico fue significativamente más alta en pacientes positivos a H. pylori antes de realizar la cirugía. Los autores sugieren que la respuesta inmune de pacientes negativos a H. pylori puede ser deficiente (ver Luther et al., 201077 en tabla 1) |
| Marteau (2006)123 | Probióticos y enfermedad de Crohn | El consumo de una cepa de Lactobacillus johnsonni fue inefectivo como profilaxis de recurrencia postoperatoria en pacientes con enfermedad de Crohn |
| Besselink (2008)124 | Efecto de probióticos en pacientes con pancreatitis aguda | El consumo de un probiótico compuesto de varias especies microbianas estuvo asociado con un mayor riesgo de mortalidad |
| Stecher (2008)125 | Relación entre la microbiota intestinal y enfermedades infecciosas | Los autores discuten mecanismos involucrados en la resistencia del hospedero a ser colonizado por potenciales patógenos |
| Mileti (2009)126 | Propiedades inmuno-modulatorias de cepas de probióticos | Usando un modelo animal de colitis, los autores demostraron que 2 especies del mismo género bacteriano (Lactobacillus) exacerbaron la colitis y causaron la muerte de los animales de experimentación. Otra especie del mismo género protegió a los animales de la colitis |
| Pant (2009)127 | Uso de inhibidores de las bombas de protones e infección por Clostridium difficile | Existe evidencia acerca de la relación entre supresores de acidez gástrica y mayor prevalencia de infecciones por Clostridium difficile; sin embargo, existe mucha controversia, que los autores discuten brevemente |
| Wang (2009)128 | Enterocolitis necrosante neonatal (NEC, por sus siglas en inglés) | Pacientes con NEC tuvieron una menor diversidad bacteriana y un incremento en la clase Gammaproteobacteria. Estos pacientes habían recibido antibióticos por mas días que los pacientes sanos sin NEC |
| Hill (2010)129 | Cambios en la microbiota intestinal inducidos por antibióticos | El tratamiento con antibióticos (ampicilina, gentamicina, metronidazol, neomicina y vancomicina) causó cambios temporales o espaciales en grupos bacterianos propuestos en jugar un papel como causantes o posibles terapéuticos en enfermedades humanas. El consumo de antibióticos también causó alteraciones en la expresión de citocinas pro-inflamatorias |
| Garrett (2010)130 | Papel de la microbiota intestinal en el desarrollo de colitis | Los autores usaron modelos animales con defectos en el sistema inmune innato para demostrar que miembros de la familia Enterobacteriaceae son capaces de inducir colitis espontánea capaz de ser «heredada» a las crías |
| Ohishi (2010)131 | Probióticos y septicemia | Los autores reportan el caso de septicemia causado por Bifidobacterium breve en una niña recién nacida con onfalocele |
| Schwiertz (2010)132 | Sobrepeso y obesidad | La proporción relativa del filo Bacteroidetes fue más alta en pacientes obesos y con sobrepeso |
| Frank (2011)133 | Fenotipo y genotipo de EII y su asociación con la microbiota intestinal | Los resultados sugieren una relación entre la microbiota intestinal y el fenotipo y genotipo de la EII |
| Khoruts (2011)134 | Trasplante de microbiota fecal | Los autores publican un excelente comentario en la revista Nature acerca del tema, con énfasis en la enfermedad causada por Clostridium difficile |
| Wu (2011)5 | Dieta y cambios en la microbiota intestinal | Diferentes grupos bacterianos del intestino están relacionados con dietas mantenidas a largo plazo (por ejemplo, Bacteroides con grasa animal, y Prevotella con hidratos de carbono) |
| Biedermann (2013)135 | Tabaquismo y cambios en la microbiota intestinal | Uno de los pocos estudios que investigan la relación entre tabaquismo y la microbiota intestinal |
Entre 1960 y 1980 se publicaron varios artículos importantes con respecto al ecosistema microbiano GI, en los cuales se discute la posible relación entre el metabolismo microbiano intestinal y la salud del ser humano15,136,137. Sin embargo, no fue sino hasta principios de la década de 1990 cuando algunos investigadores comenzaron a estudiar en detalle la relación entre los microorganismos y la inflamación en el tracto GI138. Los autores de este artículo discuten la relación entre los ácidos grasos volátiles, el mantenimiento de la integridad de la mucosa intestinal y la fermentación bacteriana en el colon. A partir de esta y otras contribuciones, una cantidad considerable de publicaciones han mostrado que la microbiota intestinal tiene un papel importante en la fisiopatología de la inflamación intestinal, en particular la EII, un proceso autoinflamatorio de alta frecuencia en países industrializados65, y síndrome de intestino irritable139 (tablas 1-3).
La relación entre la microbiota GI y los procesos inflamatorios del aparato GI es de gran importancia. No obstante, es importante mencionar que la mayoría de los estudios han utilizado solamente muestras fecales (tabla 2), y existe evidencia que sugiere que la microbiota puede ser diferente a lo largo del intestino grueso, así como entre la mucosa y la luz intestinal12,88,140-144. Aun a pesar de los inconvenientes de utilizar muestras fecales para demostrar una relación entre la microbiota intestinal y la salud del hospedero, cientos de estudios han mostrado su utilidad para estudiar el ecosistema microbiano en el intestino grueso en estados de salud y enfermedad (tabla 2).
La relación entre la microbiota intestinal y la inflamación en el tracto GI es compleja debido al gran número de moléculas involucradas145. Por un lado, las bacterias son capaces de sintetizar cientos de metabolitos146, cada uno con el potencial de modular la relación microorganismo-microorganismo y microorganismo-hospedero. Por otro lado, el hospedero humano sintetiza inmunoglobulinas, interleucinas, factores de necrosis tumoral, interferones, reguladores de transcripción de ADN y otras moléculas del sistema inmune, las cuales han sido implicadas en la aparición y/o desarrollo de la EII65,147. Una de las hipótesis para explicar el desarrollo de la EII es que el cuerpo reacciona inmunológicamente en contra de su propia microbiota148, una idea que ha generado un amplio número de investigaciones (tabla 1).
Uno de los objetivos fundamentales de los estudios de la relación de la EII y la microbiota GI es encontrar algún microorganismo o grupo de microorganismos que difieran notablemente en comparación con personas sin EII (tabla 2). La comprensión de estos cambios ayudaría a diseñar tratamientos enfocados a re-establecer el equilibrio perdido. Sin embargo, los artículos publicados han utilizado diferentes técnicas y se han enfocado en aspectos específicos de la EII y sus diferentes manifestaciones (tabla 2), por lo que ha sido difícil definir con claridad una microbiota específica de la EII. En un esfuerzo por re-establecer el balance en la microbiota intestinal, algunos probióticos y prebióticos se han utilizado como tratamiento de la EII (tablas 1 y 3); sin embargo, la mayoría de los clínicos continúan utilizando medicamentos para contener la inflamación en el hospedero149. Desafortunadamente poco se sabe acerca del efecto de estos medicamentos en la microbiota GI, pero artículos recientes han hecho progreso en el área150.
ObesidadEl sobrepeso y la obesidad son condiciones clínicas de gran importancia a nivel internacional. Desde un punto de vista microbiológico, es razonable pensar en que los diversos grupos bacterianos tienen capacidades diferentes para extraer y almacenar energía de los nutrientes151,152. Ley et al. (2005)153 fueron los primeros en describir esta relación mostrando una reducción del 50% en la abundancia relativa del filo Bacteroidetes en ratones obesos y un incremento proporcional en el filo Firmicutes. Estos resultados despertaron un gran interés por parte de la comunidad científica, en parte debido a conocimientos previos que asociaban a la microbiota intestinal con utilización y degradación de nutrientes154. Sin embargo, los resultados observados por Ley et al. fueron cambios al nivel de filo solamente; en este estudio no se observó ningún subgrupo especifico (familias, géneros o especies) de grupos bacterianos que disminuyeron o aumentaron de manera consistente dentro de los 2 grandes filos de Firmicutes y Bacteroidetes. La disminución en Bacteriodetes en pacientes obesos ha sido confirmada por algunos estudios, mientras que otras investigaciones han arrojado resultados sin diferencias o resultados que contradicen las observaciones originales de Ley et al.155. Estas discrepancias pueden deberse al modelo animal utilizado así como también a las condiciones específicas de cada estudio en particular, incluyendo la técnica utilizada para estimar la abundancia de los microorganismos. También es importante reconocer las grandes diferencias en las comunidades microbianas intestinales entre individuos3.
La relación entre la microbiota intestinal y el peso corporal parece obedecer a las características únicas de los individuos investigados. Un artículo publicado recientemente mostró resultados alentadores en el entendimiento de la relación entre la microbiota intestinal y la obesidad156. El estudio involucró el trasplante de microbiota de gemelos discordantes para obesidad (solo un gemelo sufría de obesidad) a ratones libres de microbiota. En este trabajo, los ratones inoculados con la microbiota del gemelo obeso desarrollaron mayores incrementos en masa corporal y adiposidad que los inoculados con la microbiota del gemelo no-obeso, lo cual sugiere fuertemente una asociación entre la microbiota y la condición corporal del hospedero. Estudios como este permiten estudiar más a fondo la contribución metabólica de la microbiota GI y su relación con el peso corporal del hospedero.
Con frecuencia los problemas de obesidad y sobrepeso son acompañados de varios trastornos fisiológicos como resistencia a insulina, hiperlipidemia y acumulación anormal de grasa en el hígado, conocido colectivamente como síndrome metabólico, el cual a su vez incrementa el riesgo de desarrollar enfermedades cardiovasculares. Un artículo demostró que los cambios metabólicos que acompañan a este síndrome se correlacionan con cambios en la composición de la microbiota GI en ratones genéticamente deficientes de receptores 5 tipo Toll, un componente del sistema inmune innato expresado en la mucosa intestinal157. Estos resultados soportan aún más la hipótesis de que la microbiota GI contribuye a la aparición, desarrollo y/o mantenimiento de la obesidad y síndrome metabólico.
Patologías asociadas a la infección por Helicobacter pyloriDe todo el aparato GI en el ser humano, el estómago es quizás el ambiente más hostil para la supervivencia microbiana, con valores de pH menores de 2 y una alta concentración de proteasas durante la digestión de los alimentos. Aun en estas condiciones inhóspitas y a diferencia de otros mamíferos como los perros158, el estómago humano abriga una gran diversidad microbiana de al menos 5 filos diferentes, incluyendo Proteobacteria y varias especies del género Helicobacter8. La abundancia de H.pylori en el estómago varia grandemente (1-99%) entre individuos, y su presencia parece no afectar la composición microbiana gástrica8. Esta bacteria es considerada como el agente causal de varios tipos de cáncer en el estómago, esófago, hígado y otros órganos, así como úlceras gástricas y duodenales9,10,159, pero su relación con la salud humana es controversial. Por ejemplo, un estudio sugiere que los pacientes negativos a H.pylori tienen menor probabilidad de sobrevivir a cáncer gástrico después de una resección de adenocarcinoma gástrico122, mientras que un metaanálisis reciente sugiere que esta bacteria puede tener un efecto protector en pacientes con EII77. La evidencia muestra que H.pylori tiene un papel importante en el desarrollo de diversas patologías gástricas y que hay participación importante de factores ambientales, tales como estatus socioeconómico o los hábitos alimentarios, además de factores bacterianos, del hospedero y de la microbiota gástrica acompañante160.
La presencia e importancia de otras especies de Helicobacter también es controversial, pero hay estudios que apuntan a una relación de las mismas con diferentes padecimientos como cáncer, litiasis biliar y enfermedades cardiacas161,162. Es interesante notar que H.pylori, y probablemente otras especies microbianas, tiene la capacidad de migrar a diferentes secciones dentro del estómago163, lo cual podría repercutir en los hallazgos y conclusiones reportadas entre los diferentes estudios.
Infestaciones parasitariasDecenas de diferentes tipos de parásitos pueden infestar el tracto GI de humanos. La relación entre la microbiota GI y los parásitos intestinales muy probablemente se originó hace millones de años, los primeros protegiendo al hospedero y los últimos intentando infestarlo. Un número cada vez mayor de publicaciones han investigado la relación de la microbiota GI con parásitos intestinales. Por ejemplo, un artículo reciente sugiere que la microbiota intestinal tiene un papel importante en el éxito de la eclosión de huevos del parásito Trichuris muris164. Por el otro lado, la microbiota autóctona puede también tener un papel protector como en la estimulación de células dendríticas para activar la respuesta inmune en contra de Toxoplasma gondii165. Sin embargo, otros han sugerido que algunos miembros de la microbiota podrían promover la infección por helmintos166. Sin duda, la microbiota intestinal juega un papel importante en la infección por estos y otros organismos (por ejemplo, Clostridium difficile, ver «Trasplante de microbiota fecal» abajo). Sería interesante considerar en el futuro estudios que analicen el efecto de diferentes antiparasitarios en la microbiota GI como mecanismo alterno de acción en contra de parásitos intestinales.
Otros trastornos y enfermedadesLa microbiota GI ha sido implicada en varias otras condiciones de interés clínico, como trastornos del espectro autista167,168, alergias75 y susceptibilidad al virus de influenza169. Sin embargo, son necesarias más investigaciones para esclarecer esta relación y sus implicaciones en el tratamiento terapéutico de los pacientes. También es importante recalcar que la causa de las enfermedades muchas veces es multifactorial y por lo tanto difícil de esclarecer completamente. Aun a pesar de las desventajas inherentes a la asociación de comunidades microbianas complejas con padecimientos clínicos específicos, los cuales también varían grandemente entre individuos, proyectos de secuenciación a gran escala como el Proyecto de Microbioma Humano170 o el American Gut Project jugarán un papel crucial en el entendimiento de la relación microorganismos-hospedero y su repercusión en salud y enfermedad.
Estrategias que contribuyen a mejorar la salud gastrointestinalProbióticos y prebióticosIliá Métchnikoff es famoso por su trabajo con macrófagos y otros componentes del sistema inmune, pero también fue uno de los primeros científicos que discutió la posible relación de ciertos microorganismos intestinales con la salud intestinal. En su libro clásico titulado La prolongación de la vida, Métchnikoff describe el uso de bacterias productoras de ácido láctico para contrarrestar la «putrefacción» intestinal171.
El término prebiótico fue usado por primera vez en 1995 por Gibson y Roberfroid para referirse a ingredientes no-digeribles en la dieta capaces de estimular el crecimiento y/o la actividad de uno o más grupos bacterianos en el intestino grueso, intentando mejorar la salud en el hospedero172. Los probióticos por el otro lado son definidos como organismos viables que cuando son administrados en cantidades adecuadas son capaces de proveer un beneficio al hospedero173. El uso y el significado de ambos términos han cambiado ligeramente debido a nuevos hallazgos clínicos, metabólicos y taxonómicos174,175.
Hoy en día, cepas específicas de grupos bacterianos como Bifidobacterium176, Enterococcus177, Lactobacillus178, Streptococcus179 y E.coli180 han sido utilizadas como probióticos. Levaduras como Saccharomyces boulardii también han sido utilizadas en múltiples estudios181. Además, se han propuesto algunas especies productoras de butirato como probióticos potenciales debido a su estrecha relación con la salud intestinal182. Una de las principales preocupaciones acerca del uso de los probióticos es que muchos productos comerciales no mencionan o no contienen los ingredientes descritos en las etiquetas183. Por otro lado, el uso de probióticos se ha asociado con el desarrollo de septicemia y mayor mortalidad en pacientes inmunosuprimidos o con enfermedad pancreática118,122,184, por lo que cualquier producto nutracéutico debe ser usado con cautela.
Diferentes autores han cuestionado: a)la eficacia de prebióticos, probióticos o su combinación (simbiótico); b)la eficacia de probióticos compuestos de una sola cepa, de una sola especie o de varias especies, y c)la eficacia de microorganismos aislados del mismo o de otro tipo de hospedero. En nuestro conocimiento; a)ningún artículo ha sido publicado comparando un simbiótico con sus componentes por separado (probióticos y prebióticos), aunque se espera que juntos funcionen mejor; b)2 artículos de revisión sugieren que la combinación de varios organismos en una misma formulación pueden ser más efectivos en promover la salud en el hospedero, pero ambos admiten desconocer los mecanismos185,186, y por último, c)la literatura indica que ciertas cepas o especies de microorganismos son capaces de mejorar la salud sin la necesidad de ser organismos autóctonos de la especie de hospedero187. De hecho, un probiótico no necesita provenir del aparato intestinal para ser efectivo clínicamente en casos de enfermedad GI181 y no necesariamente tienen que haber sido aislados del tracto GI de los seres humanos para tener el potencial de proporcionar beneficios en la salud de nuestra especie.
Trasplante de microbiota fecalLa trasplantación de microbiota fecal, terapia fecal o microbioterapia fecal consiste en la introducción de heces de un individuo sano en el tracto GI de otro individuo para curar una enfermedad específica, en particular la infección por Clostridium difficile188. Estudios recientes sugieren que el trasplante fecal es un procedimiento terapéutico eficiente para tratar infección recurrente por C.difficile189. Este procedimiento ha mostrado ser capaz de inducir la remisión en un gran porcentaje (>90%) de pacientes con enfermedad por C.difficile190. Existe también evidencia del potencial de la microbioterapia fecal para curar EII y otras enfermedades189,191,192.
Actualmente se desconoce el mecanismo exacto por el cual la microbioterapia fecal es efectiva, pero algunos sugieren que ayuda a restaurar las comunidades microbianas normales193, las cuales podrían no solo proteger contra la colonización de patógenos como C.difficile sino también suprimir su crecimiento194. Hamilton et al. (2013)194 utilizaron Illumina para estudiar la microbiota de 3 pacientes receptores de trasplante fecal a lo largo del tiempo y compararlo con la microbiota fecal del donador. A pesar de la gran similitud entre las muestras del donador y las muestras postrasplante de los receptores del trasplante, la abundancia relativa de miembros específicos de la microbiota varió considerablemente entre pacientes y entre muestras a diferentes tiempos postrasplante. Esta falta de consistencia en la abundancia de grupos microbianos específicos sugiere que el mecanismo de acción de los trasplantes fecales involucra otros aspectos de la microbiota (por ejemplo, actividad metabólica) además de un cambio en su composición taxonómica.
Arcillas mineralesLas arcillas minerales son materiales idealmente inertes cuyo consumo tiene el potencial de provocar cambios favorables en la salud GI. Aunque la mayoría de las investigaciones con estas arcillas se han centrado en sus efectos para prevenir la intoxicación por aflatoxinas195,196, estas arcillas han demostrado su utilidad en el tratamiento de signos clínicos GI. Por ejemplo, un estudio en Italia de más de 800 niños con diarrea aguda mostró que una arcilla de tipo esmectita redujo significativamente la duración de la diarrea197. Investigaciones más recientes han confirmado este efecto benéfico de arcillas esmectitas en controlar la diarrea en un gran número de pacientes pediátricos198. Los probables mecanismos de acción de la arcilla han sido discutidos e involucran protección de la capa de mucina, adsorción de compuestos tóxicos y modulación de la respuesta inflamatoria199–201. Esta modulación se puede dar debido a la adsorción de agentes inflamatorios intraluminales tales como interferones, interleucinas y otras citocinas. Otro posible mecanismo de acción de las arcillas en el tracto GI es modular la composición de la microbiota, pero desafortunadamente el efecto de estos compuestos en microorganismos intestinales ha sido estudiado casi exclusivamente usando métodos de cultivo200,202. Un artículo reciente utilizó Illumina (casi un millón de secuencias del gen 16S fueron generadas) para estudiar la microbiota fecal en un modelo animal de enfermedad de Crohn expuesto a ácido 2,4,6-trinitrobencenosulfónico (TNBS) con o sin arcilla esmectita201. Esfuerzos como este estudio de secuenciación masiva podrían ayudar a comprender mejor el efecto de arcillas minerales en la microbiota GI y su repercusión en la salud digestiva.
Marcadores microbianos de salud o enfermedad gastrointestinalLa medición de la abundancia de microorganismos GI no patogénicos para diagnosticar enfermedades aún no ha sido debidamente explorada, tal y como en el caso de las bacterias productoras de ácidos grasos de cadena corta o bacterias degradadoras de mucinas.
Cuantificación de baterías productoras de ácidos grasos de cadena cortaLos ácidos grasos de cadena corta sirven como fuente de energía para los colonocitos. Algunas bacterias intestinales son importantes productoras de estos compuestos203, por lo que varios estudios han intentado determinar su abundancia en pacientes con y sin EII (tabla 2). Por ejemplo, la bacteria Faecalibacterium prausnitzii fue inicialmente descubierta hace casi un siglo204, clasificada como Fusobacterium prausnitzii205, estudiada por estos y otros autores206, y finalmente reclasificada como Faecalibacterium prausnitzii207. F.prausnitzii es una bacteria que produce ácidos grasos de cadena corta en abundancia203, y varios estudios han mostrado una menor abundancia de esta bacteria en pacientes con EII (tabla 2). Sería interesante investigar en futuros estudios el potencial de la abundancia de esta u otras bacterias productoras de ácidos grasos de cadena corta en la detección y el diagnóstico de EII.
Cuantificación de bacterias degradadoras de mucinasOtro dato interesante del papel de la microbiota GI en la salud humana es la degradación de mucina por Akkermansia muciniphila. En 1990, investigadores ingleses mostraron una cantidad significativamente menor de mucina en biopsias de pacientes con colitis ulcerativa (10 pacientes) en comparación con biopsias de pacientes sanos (5 pacientes) o con enfermedad de Crohn (11 pacientes)208. Los autores de este artículo discuten que la destrucción de mucina tiene un gran valor diagnóstico, lo cual puede emplearse para el manejo terapéutico y definir el pronóstico del paciente. De manera interesante, no todos los miembros de la microbiota intestinal son capaces de degradar mucinas con la misma capacidad. En 2004 se logró aislar A.muciniphila a partir de materia fecal de humano. Esta cepa fue capaz de crecer en un medio con mucina como la única fuente de carbono y nitrógeno209. Poco después, se encontró que esta bacteria es un miembro abundante de la microbiota intestinal210. Estos resultados han sido confirmados en un estudio reciente, donde se demostró que A.muciniphila es uno de los principales degradadores de mucina en un modelo animal45. Si se toma en cuenta el potencial diagnóstico de la destrucción de mucinas, y soportado por estas investigaciones, el ejemplo de A.muciniphila es una clara demostración de cómo el estudio de la microbiota intestinal puede tener un valor diagnóstico en la práctica clínica. En nuestro conocimiento, sin embargo, ningún estudio ha sido publicado que investigue el valor diagnóstico de la abundancia de A.muciniphila en heces de pacientes.
ConclusionesEl hospedero humano y sus huéspedes microbianos han co-evolucionado por millones de años hasta convertirse en un solo superorganismo. Esta relación milenaria se ve reflejada en una convivencia frecuentemente armoniosa que observamos hoy en día en millones de personas que gozan de buena salud GI. Los resultados de las investigaciones acerca de la microbiota GI dependen de las diferentes técnicas de cultivo o moleculares utilizadas, por lo que es recomendable utilizar más de una técnica para corroborar los resultados.
En resumen, la microbiota GI juega un papel vital en la salud del ser humano. A pesar de los grandes avances en el entendimiento del ecosistema microbiano GI, más investigaciones son necesarias para definir las razones detrás de las variaciones entre individuos y su repercusión en el tratamiento de patologías digestivas. Un mejor conocimiento de la microbiota y su asociación con salud y enfermedad podría en el futuro permitir modificar este ecosistema exitosamente para mantener o mejorar el bienestar de los pacientes. El presente trabajo de revisión pretende despertar un mayor interés en la microbiota GI y su posible alteración en composición numérica, taxonómica y/o funcional para fortalecer la salud en el ser humano. Para esto, es necesario comenzar a visualizar a los pacientes como superorganismos cuyo bienestar no solo depende de la homeostasis de sus tejidos, sino también de su equilibrio con sus trillones de huéspedes microbianos.
FinanciaciónEl presente trabajo no recibió financiamiento por ninguna compañía o laboratorio.
Conflicto de interesesLos autores no tienen relación con ningún laboratorio, institución de educación u otra entidad lucrativa o no lucrativa en México u otros países que pudiera haber afectado el enfoque y revisión objetiva de los artículos científicos discutidos en este trabajo.
Los autores agradecen cordialmente al Consejo Nacional de Ciencia y Tecnología (CONACYT, México) por soporte financiero a través del programa SNI (Sistema Nacional de Investigadores).