Se desconocen los factores etiológicos exactos del cáncer colorrectal (CCR), aunque se ha intentado relacionar con factores genéticos y ambientales. La posible asociación con Fusobacterium nucleatum podría abrir posibilidades en el diagnóstico precoz.
ObjetivoRevisar los estudios que analizan la asociación entre F. nucleatum y el CCR.
MétodosSe utilizaron las publicaciones disponibles en la base de datos MEDLINE PubMed hasta el día 1 de enero de 2020, que incluían los términos «cáncer colorrectal» y «Fusobacterium nucleatum». Se realizó un metaanálisis con el software Stata.
ResultadosUn total de 57 artículos fueron incluidos en la revisión sistemática. El metaanálisis indicó una mayor presencia de F. nucleatum en muestras de tejido tumoral de CCR, con respecto a muestras control de tejido sano, con una odds ratio de 4,558 (IC 95%: 3,312-6,272), y cuando se utilizaron muestras control de adenomas colorrectales, con una odds ratio de 3,244 (IC 95%: 2,359-4,462).
ConclusiónHay una mayor presencia de F. nucleatum en el CCR. Sin embargo, se necesitan estudios que demuestren esta relación.
The etiological factors of colorectal cancer (CRC) are not precisely known, although genetic and environmental factors have been implicated. A possible association with Fusobacterium nucleatum may provide opportunities for an early diagnosis.
ObjectiveTo review studies that address the association between F. nucleatum and CRC.
MethodsThe MEDLINE PubMed database was searched using the terms «colorectal cancer» and «Fusobacterium nucleatum», retrieving publications published up to January 1 2020. Stata software was used for a meta-analysis.
ResultsThe systematic review included 57 articles. Meta-analysis results indicated a more frequent presence of F. nucleatum in CRC tumor tissue samples in comparison to control samples of healthy tissue, with an odds ratio of 4.558 (95% CI: 3.312-6.272), and in comparison, to control samples of colorectal adenomas, with an odds ratio of 3.244 (95% CI: 2.359-4.462).
ConclusionThere is a more frequent presence of F. nucleatum in the CRC. However, further studies are needed to verify this relationship.
La etiología del cáncer colorrectal (CCR) es multifactorial e incluyen alteraciones genéticas y epigenéticas1. También podrían intervenir factores extrínsecos como la disbiosis intestinal2, pero existe una variabilidad interindividual, debido a múltiples factores genéticos y ambientales3, por ello resultaría de especial interés investigar la existencia de un denominador microbiano común. Fusobacterium nucleatum (FN) ha sido una de las bacterias más estudiadas4–6. Es una bacteria anaerobia, gramnegativa, que reside en la cavidad oral como microbiota comensal, pero también es un patógeno oportunista en las enfermedades periodontales, principalmente en gingivitis y periodontitis7. Su poder patógeno reside en sus factores de virulencia, entre los que destacan: la presencia de fimbrias, lipopolisacáridos, factores inhibidores de la quimiotaxis de los leucocitos polimorfonucleares y la elaboración de metabolitos tóxicos tisulares8. Sin embargo, el más importante es la adhesina FadA, ya que se ha demostrado que es el mayor estimulante de la inflamación al crear un ambiente proinflamatorio crónico que activaría señales oncogénicas, estimulando las células epiteliales9,10. Estudios recientes han demostrado un aumento de FadA en el tejido neoplásico del CCR, junto con otros marcadores proinflamatorios como COX-2, IL-8, IL-6, IL1ß y TNF-α11.
La posibilidad de que FN pueda formar parte del proceso de carcinogénesis podría abrir posibilidades en el diagnóstico precoz. Por lo tanto, estaría justificado analizar exhaustivamente la evidencia actual sobre la relación entre FN y el CCR. El objetivo de este trabajo fue integrar la información disponible sobre la relación entre FN y el CCR, a través de un metaanálisis.
Material y métodosEl metaanálisis tuvo un componente cualitativo (revisión sistemática) y otro cuantitativo. La revisión sistemática fue una descripción de los trabajos publicados que considera los estudios individuales como «sujetos» de estudio. Así, se realizó una búsqueda sistemática de todos los artículos publicados, en inglés o español, en revistas indexadas en MEDLINE, ISI Web of Knowledge y Cochrane Library. Los términos de búsqueda utilizados fueron «colorectal cancer» y «Fusobacterium nucleatum». Los criterios de selección fueron el límite temporal, ya que la búsqueda se restringió a todos los artículos publicados, en formato paper y/o electrónico, antes de enero de 2020, el tipo de estudio, seleccionando los estudios de investigación, y el idioma, limitados a los redactados en inglés y en español. Se utilizó como criterios de exclusión los trabajos sobre la relación de FN con otras enfermedades diferentes al CCR, la relación del CCR con bacterias diferentes a FN y el aislamiento de FN en especies diferentes al ser humano; así como las revisiones. Se requirió que los pacientes con CCR estuvieran incluidos en los estudios, y que el objetivo del estudio fuera la búsqueda directa o indirecta de FN y que intentara establecer una posible asociación entre la infección y el CCR. Se utilizó un sistema de calidad interno, que verificó la fiabilidad del método anterior elegido, basado en la revisión detenida de cada publicación con rastreo de la bibliografía de las publicaciones para evitar las pérdidas.
El componente cuantitativo se refirió a la agrupación estadística de los resultados, actuando los estudios individuales como los sujetos de la investigación, obteniéndose, en cada publicación y en conjunto con las demás, la significación estadística del análisis, el intervalo de confianza al 95%, la odds ratio (OR) y los pesos de los trabajos. Para estimar la medida ponderada se utilizó el modelo de efectos aleatorios por el método de DerSimonian y Laird12, el cual se ve menos afectado ante la heterogeneidad entre los estudios. Para valorar la heterogeneidad se utilizó la prueba del inverso de la varianza. Además, se calculó el índice de inconsistencia I2 de Higgins, cuyo valor indica el porcentaje de variabilidad debida a la heterogeneidad entre estudios. Valores por encima del 75% indican un nivel de heterogeneidad fuerte, y sugeriría la necesidad de realizar estudios adicionales. Las pruebas de Begg13 y Egger14 se usaron para detectar sesgo de publicación. El análisis de los datos se realizó con el software Stata Release, versión 14.
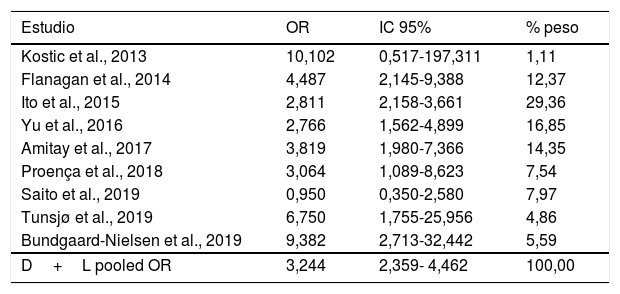
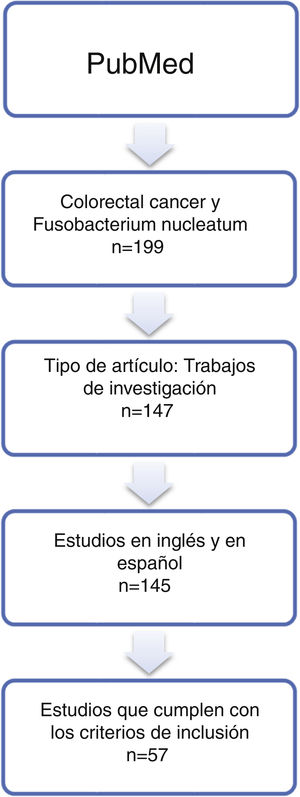
ResultadosRevisión sistemáticaLa búsqueda de los artículos recuperó 199 publicaciones, de los que se seleccionaron 57 trabajos (tabla 1 y fig. 1) que fueron publicados entre 2011 y 2019, al menos en su versión on-line. Se han denominado Controles-1 las muestras control de zona no tumoral (40 trabajos-70,2%: 15 tejidos adyacentes no tumorales; 4 muestras fecales; 2 muestras sanguíneas; una muestra salival; 18 muestras de tejidos sin tumor) y Controles-2 las obtenidas de adenomas (16-28,1%). La columna Prueba-1 indica la prueba descrita por los autores para detectar el microorganismo. Además, algunos de los trabajos incluyen el uso de pruebas de laboratorio adicionales, que se ha denominado Prueba-2. Para finalizar, se han recogido, también, la conclusión más importante de los artículos de investigación (columna «Comentarios artículo»).
Revisión sistemática de la bibliografía publicada en la base de datos MEDLINE PubMed hasta el día 1 de enero de 2020 sobre la relación entre Fusobacterium nucleatum y el cáncer colorrectal
| Autor | AñoPublicación física | Sexo total (m:h) | CasosCCR | SexoCasosCCR(m:h) | Control-1:No CCR (n) | SexoControl-1 | DescripciónGrupoControl-1 | Control-2:Adenomas (n) | SexoControl-2 | ¿Qué se analiza? | Prueba 1 | Prueba 2 | FN+/totalCasos | FN+/totalControl-1 | FN+/totalControl-2 | Comentarios artículos |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Castellarin et al.15 | 2012 | NC | 11 | NC | 99 | NC | Tejidos adyacentes no tumorales | NC | NC | RNA | PCR | NC | 11/11 | 9/11 | NC | Mayor porcentaje de FN en CCR que en tejido normal |
| Rubinstein et al.16 | 2013 | NC | 51 | NC | 14 | NC | Tejidos de sujetos sin tumor | NC | NC | RNAm | PCR | NC | NC | NC | NC | Mayor porcentaje de FadA en CCR que en tejido normal |
| Kostic et al.4 | 2013 | NC | 27 | NC | 31 | NC | Tejidos de sujetos sin tumor | 28 | NC | DNA y RNA | PCR | FISH | 27/27 | 15/31 | 24/28 | FN crea un estado proinflamatorio que favorece la progresión a CCR |
| Warren et al.17 | 2013 | NC | 65 | NC | 65 | NC | Tejidos adyacentes no tumorales | NC | NC | rRNA | Metatranscriptómica | NC | NC | NC | NC | Mayor porcentaje de FN en CCR que en tejido sano adyacente |
| Flanagan et al.18 | 2014 | NC | 122 | NC | 174 | NC | Tejidos adyacentes no tumorales | 52 | NC | DNA | PCR | NC | 70/122 | 33/174 | 12/52 | FN puede usarse como marcador para el CCR |
| Mira-Pascual et al.19 | 2014 | NC | 7 | NC | 5 | NC | Tejidos de sujetos sin tumor | NC | NC | DNA y RNA | PCR | Pirosecuenciación | 2/7 | 0/5 | NC | El aumento de niveles de las bacterias estudiadas se correlaciona con el CCR |
| Tahara et al.20 | 2014 | NC | 149 | NC | 89 | NC | Tejidos adyacentes no tumorales | NC | NC | DNA | PCR | NC | 78/149 | 27/89 | NC | Mayor porcentaje de FN en CCR que en controles |
| Ito et al.21 | 2015 | NC | 511 | 225:286 | 20 | NC | Tejidos de sujetos sin tumor | 456 | 39:59 | DNA | PCR | NC | 286/511 | 3/20 | 142/456 | Mayor porcentaje de FN en CCR que en controles |
| Mima et al.22 | 2015 | NC | 598 | NC | 558 | NC | Tejidos adyacentes no tumorales | NC | NC | DNA | PCR | NC | 76/598 | 19/558 | NC | FN crea un estado proinflamatorio que favorece la progresión a CCR |
| Yu et al.10 | 2015 | NC | 42 | NC | 52 | NC | Tejidos adyacentes no tumorales | 47 | NC | DNA | PCR | NC | NC | NC | NC | Mayor porcentaje de FN en CCR que en controles |
| Fukugaiti et al.23 | 2015 | 4:13 | 7 | 2:5 | 10 | 2:8 | Heces | NC | NC | DNA | PCR | FISH | 7/7 | 9/10 | NC | Mayor porcentaje de FN en CCR que en controles |
| Nosho et al.24 | 2016 | NC | 511 | 225:286 | NC | NC | No hay tejido control | NC | NC | DNA | PCR | NC | 44/511 | NC | NC | Menor porcentaje de positivos para FN en la cohorte japonesa |
| Repass et al.25 | 2016 | NC | 40 | NC | 40 | NC | Tejidos adyacentes no tumorales | NC | NC | DNA | PCR | NC | NC | NC | NC | Mayor porcentaje de FN en CCR que en tejido normal |
| Li et al.26 | 2016 | 46:55 | 101 | 46:55 | 101 | 46:55 | Tejidos adyacentes no tumorales | NC | NC | DNA | PCR | FISH | 88/101 | NC | NC | Mayor cantidad de FN en CCR se asocia con menor tiempo de supervivencia |
| Yu et al.27 | 2016 | NC | 93 | NC | 20 | NC | Tejidos de sujetos sin tumor | 112 | NC | DNA | PCR | NC | 62/93 | 4/20 | 47/112 | FN puede jugar un papel en la vía serrada de la carcinogénesis para CCR |
| Mima et al.28 | 2016 | NC | 1.102 | NC | NC | NC | No hay tejido control | NC | NC | DNA | PCR | NC | 138/1.102 | NC | NC | Aumenta la proporción de FN desde los CCR del recto a los cecales |
| Yu et al.5 | 2017 | NC | 74 | NC | 54 | NC | Tejidos de sujetos sin tumor | NC | NC | DNA | PCR | NC | 39/74 | 2/54 | NC | FN promueve la progresión de CCR a través de la modulación de las vías de la autofagia |
| Suehiro et al.29 | 2017 | NC | 158 | NC | 60 | NC | Tejidos de sujetos sin tumor | 19 | NC | DNA | PCR | NC | NC | NC | NC | Media de copias de FN: 17,5 en el grupo control, 122 adenoma, 317 CCR |
| Liang et al.30 | 2017 | NC | 203 | NC | 236 | NC | Tejidos de sujetos sin tumor | NC | NC | DNA | PCR | Secuenciación metagenómica | NC | NC | NC | Mayor porcentaje de FN en CCR que en controles |
| Wong et al.31 | 2017 | NC | 104 | NC | 102 | NC | Tejidos de sujetos sin tumor | 103 | NC | DNA | PCR | NC | NC | NC | NC | Mayor cantidad de FN en los adenomas con respecto a los controles p=0,022 |
| Mehta et al.32 | 2017 | NC | 1.019 | NC | NC | NC | No hay tejido control | NC | NC | DNA | PCR | NC | 125/1.019 | NC | NC | La asociación de la dieta con CCR es modificada por FN |
| Chen et al.33 | 2017 | NC | 98 | 40:58 | NC | NC | No hay tejido control | NC | NC | DNA | FISH | NC | 61/98 | NC | NC | FN invasivo activa la vía de señalización beta-catenina |
| Ye et al.34 | 2017 | NC | 25 | NC | 25 | NC | Tejidos adyacentes no tumorales | NC | NC | DNA | PCR | NC | 4/25 | 2/25 | NC | Mayor porcentaje de FN en CCR que en controles |
| Amitay et al.35 | 2017 | 220:280 | 46 | 15:31 | 231 | 121:110 | Tejidos de sujetos sin tumor | 223 | 84:139 | DNA | PCR | NC | 25/46 | 58/231 | 53/223 | Mayor porcentaje de FN en CCR que en controles |
| Park et al.36 | 2017 | 82:78 | 160 | 82:78 | NC | NC | No hay tejido control | NC | NC | DNA | PCR | NC | 107/160 | NC | NC | La mayor cantidad de FN en muestras tumorales se correlaciona con la presencia de macrófagos intratumorales |
| Eklöf et al.37 | 2017 | NC | 39 | 19:20 | 65 | 30:35 | Tejidos de sujetos sin tumor | NC | NC | DNA | PCR | NC | 27/39 | 15/65 | NC | Mayor cantidad de FN en CCR que en controles |
| Bullman et al.38 | 2017 | NC | 77 | NC | NC | NC | No hay tejido control | NC | NC | RNA | PCR | FISH | 45/77 | NC | NC | La reducción de FN con antibióticos produce una menor proliferación cancerígena |
| Drewes et al.39 | 2017 | NC | 58 | NC | 34 | NC | Tejidos de sujetos sin tumor | NC | NC | RNA | PCR | FISH | 17/58 | 1/34 | NC | Mayor cantidad de bacterias en CCR que en tejido sano |
| Yamaoka et al.40 | 2018 | NC | 100 | NC | 72 | NC | Tejidos adyacentes no tumorales | NC | NC | DNA | PCR | 75/100 | 46/72 | NC | Mayor cantidad de FN en CCR que en controles | |
| Guevara et al.6 | 2018 | NC | 37 | NC | 37 | NC | Tejidos de sujetos sin tumor | NC | NC | Anticuerpos anti-FN | ELISA | NC | 37/37 | 12/37 | NC | La proteína Fap2 de FN es antigénica |
| Dai et al.41 | 2018 | 206:320 | 255 | 92:163 | 271 | 114:157 | Heces | NC | NC | DNA | Metagenómica | NC | NC | NC | NC | Mayor porcentaje de FN en CCR que en controles |
| Liu et al.42 | 2018 | NC | 2.759 | NC | NC | NC | No hay tejido control | NC | NC | DNA | PCR | NC | 951/2.759 | NC | NC | Asociación positiva de dietas empíricas inflamatorias (EDIP) con CCR FN+ |
| Chen et al.43 | 2018 | NC | 25 | NC | 7 | NC | No hay tejido control | 8 | NC | DNA | FISH | Inmunofluorescencia | NC | NC | NC | Mayor expresión de TOX y CD4+ en tejidos FN− comparados con los FN+. Correlación negativa entre la abundancia de FN y la expresión de TOX |
| Guo et al.44 | 2018 | NC | 215 | NC | 156 | NC | Tejidos de sujetos sin tumor | NC | NC | DNA | PCR | NC | NC | NC | NC | La relación FN/Bifidobacterium tuvo una sensibilidad del 84,6% y una especificidad del 92,3% en detectar CCR |
| Komiya et al.45 | 2018 | NC | 14 | NC | NC | NC | No hay tejido control | NC | NC | DNA | PCR | NC | 8/14 | NC | NC | Muestras de FN en tejido tumoral y en saliva |
| Lee et al.46 | 2018 | NC | 246 | NC | NC | NC | No hay tejido control | NC | NC | DNA | PCR | NC | NC | NC | NC | Altos niveles de FN se asocian con una menor supervivencia en las metástasis de CCR |
| Rezasoltani et al.47 | 2018 | NC | NC | NC | 31 | 15:16 | Tejidos de sujetos sin tumor | 87 | 33:54 | DNA | PCR | NC | NC | NC | NC | Relación significativa entre el tamaño del pólipo y la cuantificación de FN |
| Hamada et al.48 | 2018 | 609:432 | 1.041 | 609:432 | NC | NC | No hay tejido control | NC | NC | DNA | PCR | NC | 135/1.041 | NC | NC | La asociación de FN con el CCR es diferente dependiendo de si el tumor tiene alterada la vía de los microsatélites |
| Oh et al.49 | 2018 | 231:362 | 593 | 231:362 | NC | NC | No hay tejido control | NC | NC | DNA | PCR | NC | 204/593 | NC | NC | El pronóstico del tumor en relación con FN depende de la localización del CCR |
| Proença et al.50 | 2018 | NC | 43 | NC | 70 | NC | Tejidos adyacentes no tumorales | 27 | NC | DNA | PCR | NC | 33/43 | 23/70 | 14/27 | FN es un factor de riesgo para CCR |
| Zhang et al.51 | 2019 | 41:53 | 94 | 41:53 | NC | NC | No hay tejido control | NC | NC | DNA | PCR | Microarray | 21/90 | NC | NC | FN induce la expresión del gen BIRC3 en CCR |
| Saito et al.52 | 2019 | 30:51 | 24 | 7:17 | 10 | 7:3 | Tejidos de sujetos sin tumor | 47 | 16:31 | rRNA | PCR | NC | 14/24 | NC | 28/47 | FN puede asociarse con CCR y adenomas, y es un potencial marcador diagnóstico para ambos |
| Guo et al.53 | 2019 | NC | 46 | NC | 36 | NC | Sangre | NC | NC | DNA | Secuenciación genómica | NC | NC | NC | NC | Mayor presencia de FN en los tejidos tumorales que en las muestras sanguíneas |
| Feng et al.54 | 2019 | 13:12 | 15 | 8:7 | 10 | 5:5 | Tejidos de sujetos sin tumor | NC | NC | DNA | PCR | Western blot | 10/15 | 5/10 | NC | La expresión de la proteína CREB se correlaciona con la metástasis de CCR ya sea en FN+ como FN− |
| Tunsjø et al.55 | 2019 | NC | 25 | NC | 22 | NC | Tejidos de sujetos sin tumor | 25 | NC | DNA | PCR | Secuenciación 16S rRNA | 15/25 | 5/25 | 4/22 | PCR para la detección de FN podría ser incluida como biomarcador de CCR |
| Bundgaard-Nielsen et al.56 | 2019 | 141:132 | 99 | 55:44 | 76 | 35:41 | Tejidos adyacentes no tumorales | 96 | 51:47 | DNA | PCR | Secuenciación 16S rRNA | 23/99 | NC | 3/96 | No hay evidencia de que FN intervenga en el desarrollo de adenomas, pero puede tener un papel en la transición de adenoma a CCR |
| Guven et al.57 | 2019 | NC | 71 | NC | 77 | NC | Saliva de controles no cancerosos | NC | NC | DNA | PCR | NC | NC | NC | NC | Mayor cantidad de FN en la saliva de los pacientes con CCR en comparación con los controles |
| Yachida et al.58 | 2019 | NC | 258 | NC | 291 | NC | Heces | 67 | NC | DNA | Electroforesis capilar | Estudio metagenómico y metabolómico | NC | NC | NC | La abundancia de FN se correlaciona con la progresión tumoral desde carcinoma intramucoso a estadios más avanzados |
| Leung et al.59 | 2019 | 10:9 | 19 | 10:9 | 19 | 10:9 | Tejidos adyacentes no tumorales | NC | NC | DNA | PCR | NC | NC | NC | NC | Mayor porcentaje de FN en CCR que en tejido normal |
| Kunzmann et al.60 | 2019 | 60:130 | 190 | 60:130 | 190 | 60:130 | Tejidos adyacentes no tumorales | NC | NC | DNA | PCR | NC | 61/190 | 129/190 | NC | FN es más abundante en el tejido tumoral que en tejido circundante |
| Butt et al.61 | 2019 | NC | 485 | NC | 485 | NC | Sangre | NC | NC | Anticuerpos anti-FN | Serología | NC | NC | NC | NC | Los anticuerpos dirigidos contra FN no tienen asociación con el riesgo de desarrollar CCR |
| De Carvalho et al.62 | 2019 | NC | 152 | NC | 57 | NC | Tejidos adyacentes no tumorales | NC | NC | DNA | PCR | NC | 35/152 | 6/57 | NC | La detección de DNA de FN se asocia con la localización proximal de los tumores, positividad para la mutación MSI, BRAF, pérdida de MLH1, MSH2 y PMS2. También con menor supervivencia |
| Chen et al.63 | 2019 | 36:55 | 91 | 36:55 | NC | NC | No hay tejido control | NC | NC | DNA | PCR | NC | 25/91 | NC | NC | Mayor cantidad de FN en el tejido del CCR se asocia con un menor tiempo de supervivencia |
| Mima et al.64 | 2019 | 208:304 | 256 | 208:304 | NC | NC | No hay tejido control | NC | NC | DNA | PCR | NC | 140/256 | NC | NC | Mayor cantidad de FN y otras bacterias asociadas al CCR aumentan el riesgo de rotura anastomótica tras la cirugía |
| Liang et al.65 | 2019 | NC | 274 | NC | 353 | NC | Heces | 353 | NC | DNA | PCR | Prueba inmunohistoquímica fecal | 71/274 | 97/385 | 145/353 | Identificación del marcador m3 para el diagnóstico de CCR |
| Haruki et al.66 | 2019 | 423:301 | 724 | 423:301 | NC | NC | No hay tejido control | NC | NC | DNA | PCR | NC | 99/724 | NC | NC | Asociación inversa entre la cantidad de FN y la expresión tumoral de BECN1 |
| Chen et al.67 | 2019 | NC | 148 | NC | NC | NC | No hay tejido control | NC | NC | DNA y RNA | PCR | FISH | 88/148 | NC | NC | FN se encuentra en mayor cantidad en los CCR con metástasis |
CCR: cáncer colorrectal; FN: Fusobacterium nucleatum; NC: no consta información.
Se comprobó un aumento del número de los trabajos publicados con el paso de los años, pero no fue hasta 2017 cuando aumentó significativamente esta investigación. El sexo de los casos se describió en 20 (35,1%) trabajos; en los Controles-1 en 11 (19,3%) estudios y en los Controles-2 en 5 (31,2%) trabajos. Hasta el año 2015 ningún trabajo describió el sexo de los participantes en el estudio. Los trabajos más recientes sí tienden a indicar el sexo de la población estudiada. Cuando se analiza la distribución de los estudios con datos cuantitativos, para los Casos, Controles-1 y Controles-2, estos aparecen indicados en 40 (70,2%), 22 (38,6%%) y 10 (25%) artículos, respectivamente.
Análisis cuantitativoEn cuanto al número de positivos en las muestras de CCR, los mayores porcentajes de positivos se obtuvieron en los estudios de Castellarin et al.15, Kostic et al.4, Fukugaiti et al.23 y Guevara et al.6, en los que la totalidad de las muestras de CCR analizadas obtuvieron un resultado positivo en las pruebas de laboratorio. Por el contrario, el menor porcentaje de resultados positivos se encontró en el estudio de Nosho et al.24, donde solo un 8,6% de las muestras arrojaron un resultado positivo. En cuanto al número de muestras de los Casos en cada trabajo, es muy variado. Así el menor número se encontró en los estudios de Mira-Pascual et al.19 y Fukugaiti et al.23, con un total de 7 casos analizados cada uno. El mayor tamaño muestral para los Casos se correspondió con el estudio de Liu et al.42, con un análisis de 2.759 muestras. Cabe destacar que solo hay 4 (7%) trabajos que superen los 1.000 individuos con CCR, y que fueron llevados a cabo entre los años 2017 y 2018.
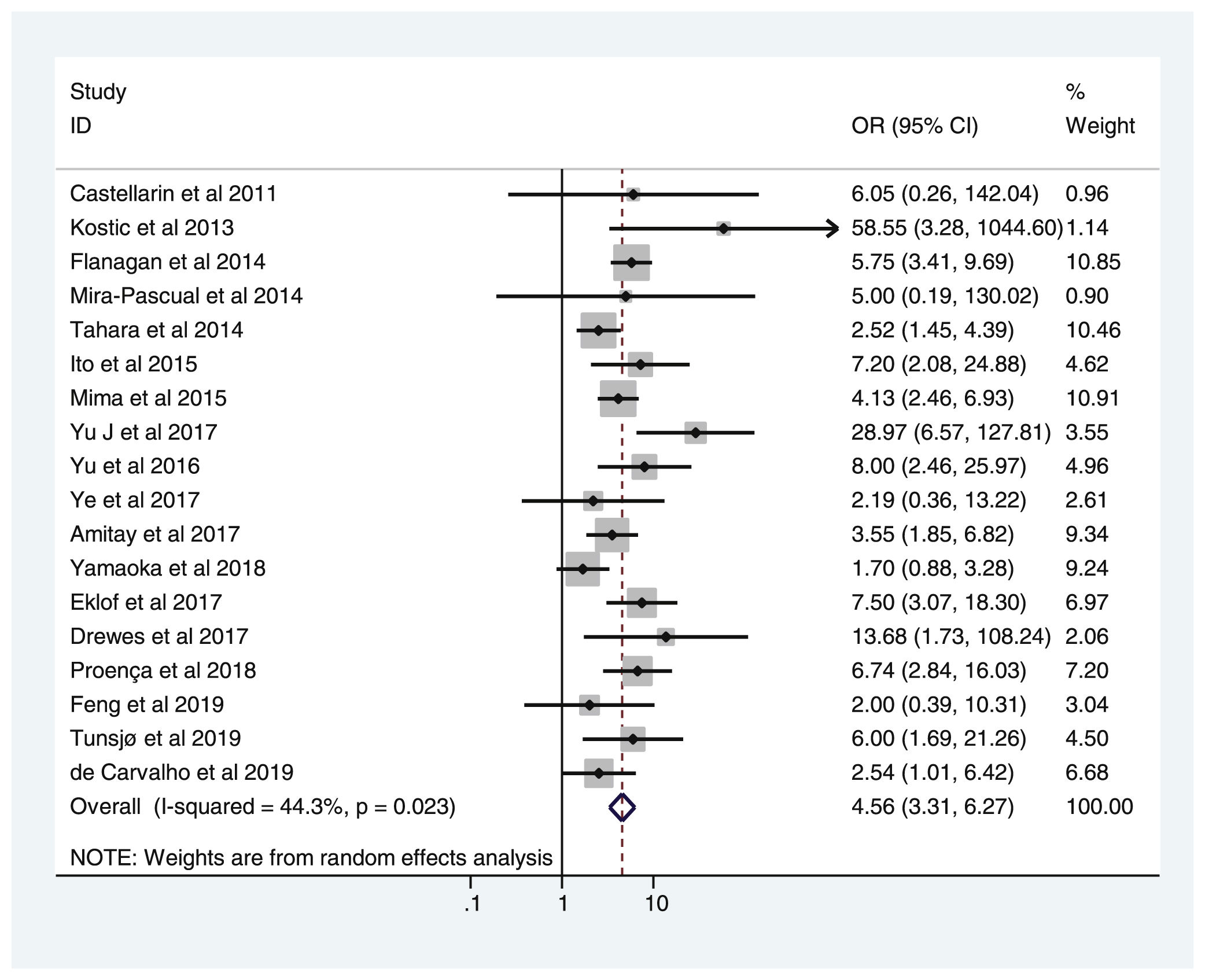
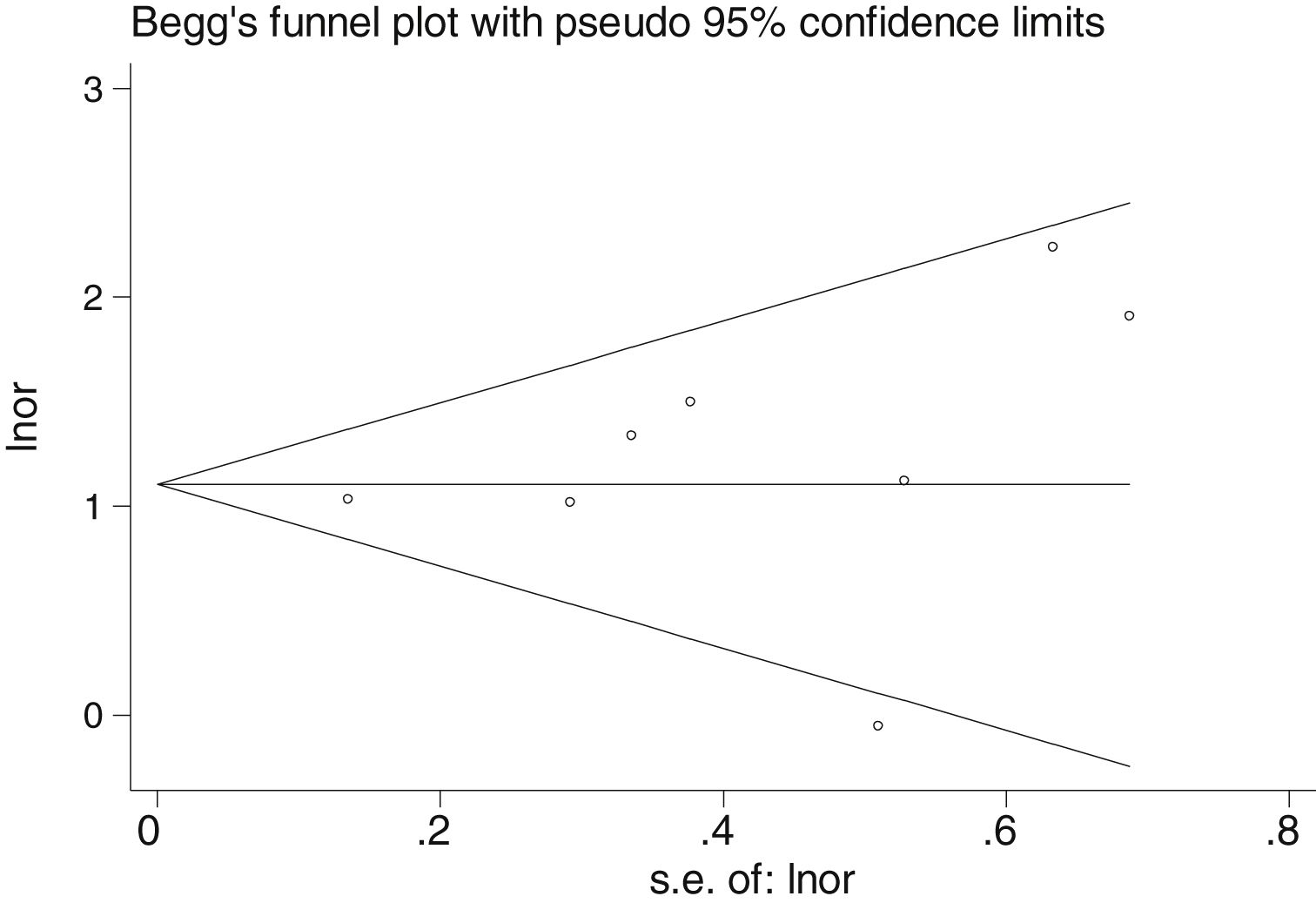
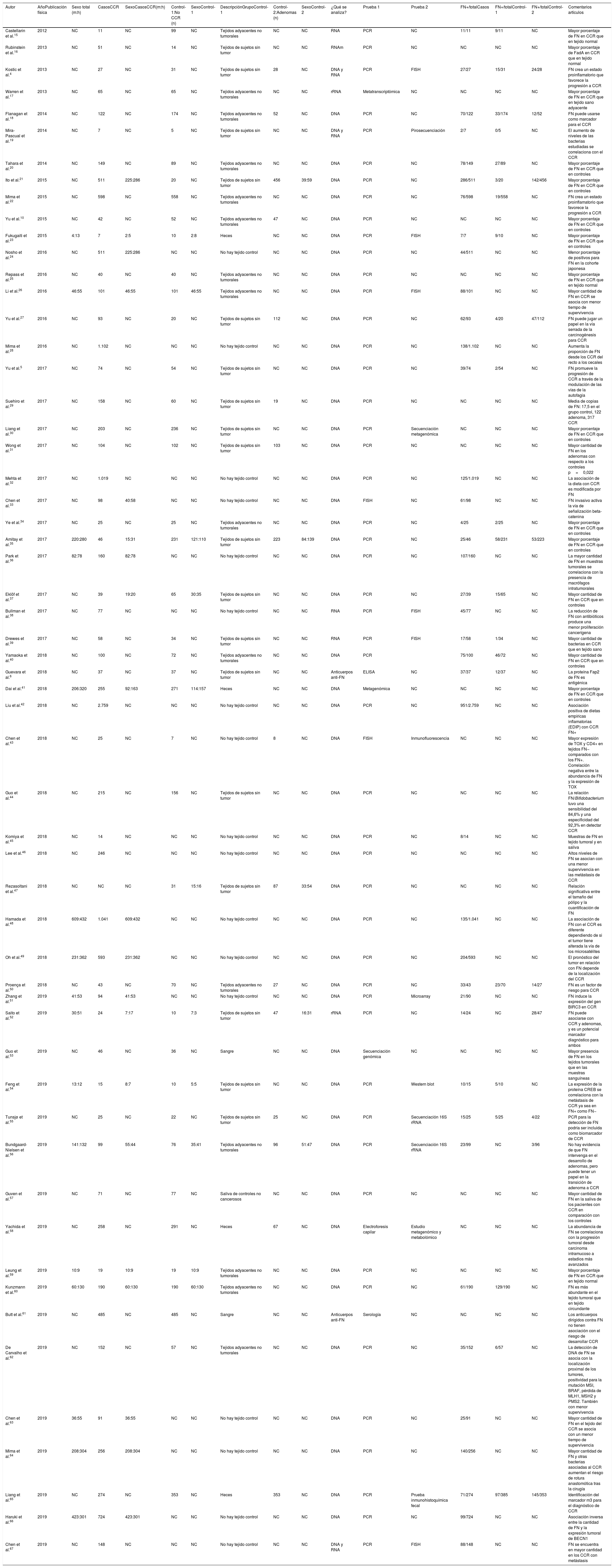
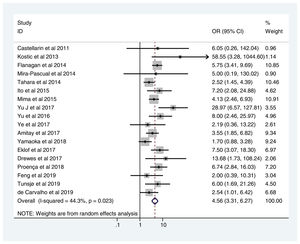
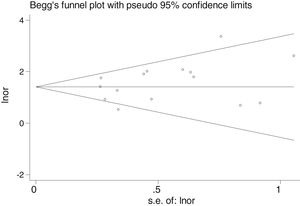
En la tabla 2 se reflejan los estudios (18 de los 40, 45%) que compararon, en Casos y Controles-1, la positividad de marcadores de infección por FN con datos cuantitativos de muestras de tejidos colónicos con pruebas de biología molecular. Por su peso, destacan los estudios Mima et al.28, Flanagan et al.18 y Tahara et al.20. Todos estos artículos tienen un peso superior al 10%. La estimación global de la OR de los resultados individuales obtenidos en estos trabajos dio un resultado de 4,558 (IC 95%: 3,312-6,272), que nos permite concluir que hay asociación significativa (p<0,001) entre FN y el CCR cuando se utilizaron los Controles-1. Para calcular la heterogeneidad se llevó a cabo la prueba del inverso de la varianza, con un valor de χ2exp.=30,54, con 17 grados de libertad y p=0,023, por lo que las diferencias encontradas entre los estudios no fueron debidas al azar. El estudio de heterogeneidad se corroboró utilizando la I2 de Higgins, con un resultado de I2=44,3% (tabla 2 y fig. 2). No hubo sesgo de publicación evidente, tal y como se indica en las pruebas de Begg y Egger que no fueron significativas (p=0,767 y p=0,210, respectivamente) (fig. 3).
Relación de los 18 estudios que compararon, en Casos y Controles-1, la positividad de marcadores de infección por Fusobacterium nucleatum en muestras de biopsia intestinal mediante biología molecular
| Estudio | OR | IC 95% | % peso |
|---|---|---|---|
| Castellarin et al., 2011 | 6,053 | 0,258-142,040 | 0,96 |
| Kostic et al., 2013 | 58,548 | 3,282-1.044,602 | 1,14 |
| Flanagan et al., 2014 | 5,752 | 3,413-9,693 | 10,85 |
| Mira-Pascual et al., 2014 | 5,000 | 0,192-130,024 | 0,90 |
| Tahara et al., 2014 | 2,523 | 1,449-4,393 | 10,46 |
| Ito et al., 2015 | 7,203 | 2,085-24,884 | 4,62 |
| Mima et al., 2015 | 4,130 | 2,463-6,926 | 10,91 |
| Yu et al., 2017 | 28,971 | 6,567-127,806 | 3,55 |
| Yu et al., 2016 | 8,000 | 2,465-25,968 | 4,96 |
| Ye et al., 2017 | 2,190 | 0,363-13,219 | 2,61 |
| Amitay et al., 2017 | 3,551 | 1,850-6,815 | 9,34 |
| Yamaoka et al., 2018 | 1,696 | 0,876-3,282 | 9,24 |
| Eklöf et al., 2017 | 7,500 | 3,074-18,297 | 6,97 |
| Drewes et al., 2017 | 13,683 | 1,730-108,242 | 2,06 |
| Proença et al., 2018 | 6,743 | 2,838-16,026 | 7,20 |
| Feng et al., 2019 | 2,000 | 0,388-10,309 | 3,04 |
| Tunsjø et al., 2019 | 6,000 | 1,693-21,262 | 4,50 |
| De Carvalho et al., 2019 | 2,543 | 1,007-6,421 | 6,68 |
| D+L pooled OR | 4,558 | 3,312-6,272 | 100,00 |
Heterogeneidad χ2=30,54 (g.l.=17) p=0,023.
I2 (variación en OR atribuible a la heterogeneidad)=44,3%.
Estimación de la varianza entre estudios Tau-cuadrado=0,1736.
Test de OR=1: z=9,31, p=0,000.
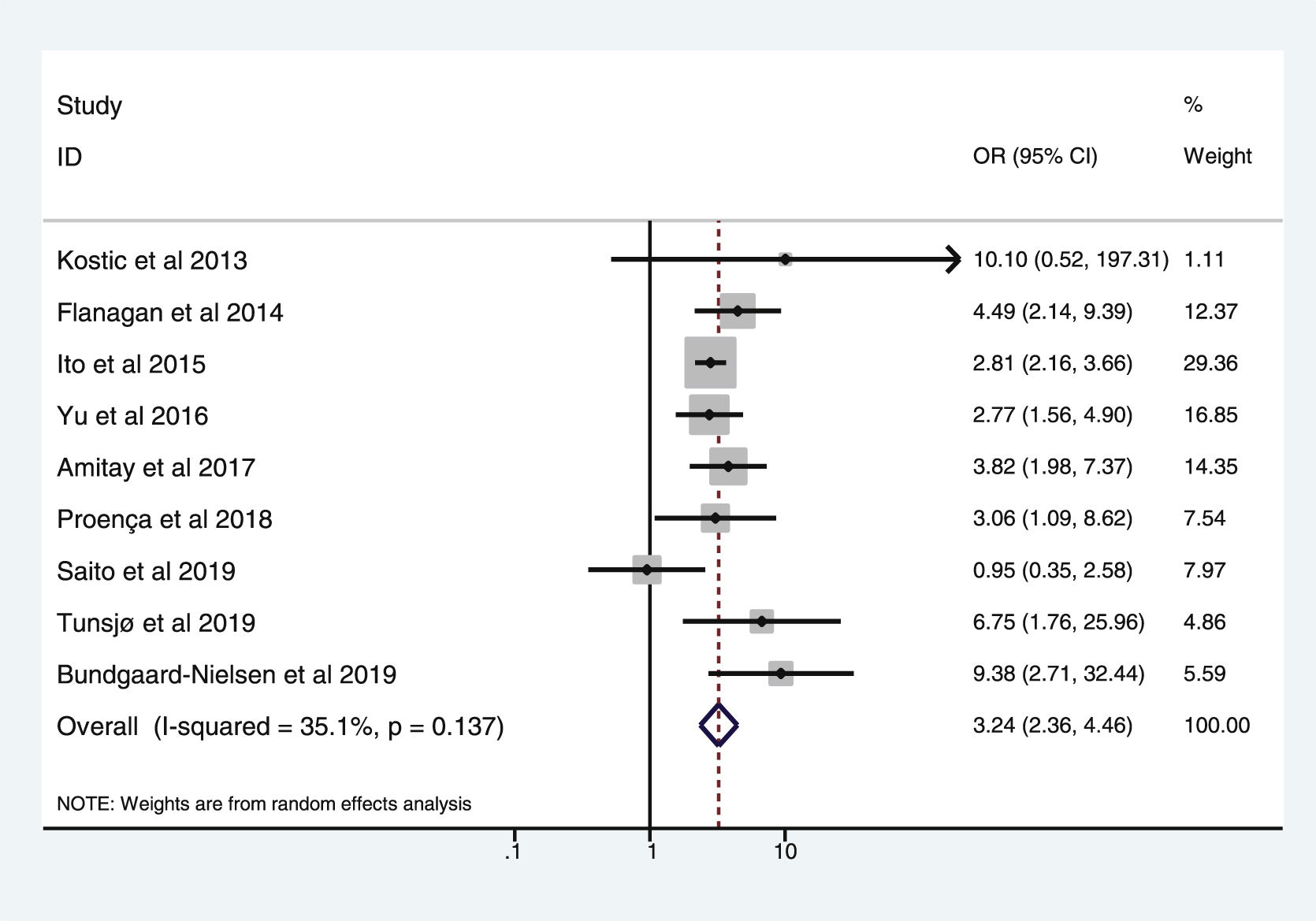
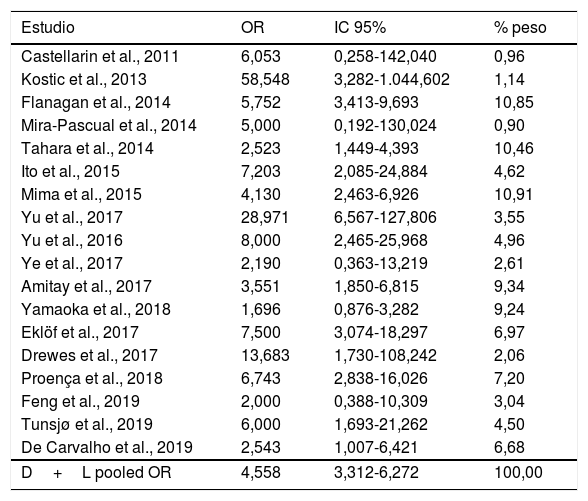
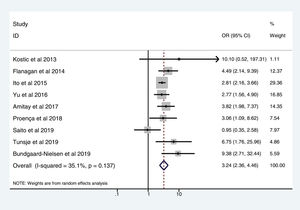
En la tabla 3 se reflejan los estudios (9 de los 16, 56,25%) que compararon, en Casos y Controles-2 (adenomas colorrectales), la positividad de marcadores genéticos de infección por FN con datos cuantitativos de muestras de tejidos colónicos con pruebas de biología molecular. Destacan por su peso los trabajos de Ito et al.21, Flanagan et al.18, Yu et al.27 y Amitay et al.35, con un peso superior al 12%. La estimación global de la OR de los resultados individuales obtenidos en estos trabajos dio un resultado de 3,244 (IC 95%: 2,359-4,462), lo cual muestra una asociación estadísticamente significativa entre la presencia de FN y el CCR (p<0,001). Para analizar la heterogeneidad se llevó a cabo la prueba del inverso de la varianza, con un valor de χ2exp.=12,33, con 8 grados de libertad y p=0,137, lo cual muestra que existe homogeneidad entre los estudios comparados. Además, la I2 de Higgins mostró un valor del 35,1%, indicando un bajo nivel de heterogeneidad (tabla 3 y fig. 4).
Relación de 9 estudios que compararon, en Casos y Controles-2, la positividad de los marcadores de infección por Fusobacterium nucleatum en muestras de biopsia intestinal mediante biología molecular
| Estudio | OR | IC 95% | % peso |
|---|---|---|---|
| Kostic et al., 2013 | 10,102 | 0,517-197,311 | 1,11 |
| Flanagan et al., 2014 | 4,487 | 2,145-9,388 | 12,37 |
| Ito et al., 2015 | 2,811 | 2,158-3,661 | 29,36 |
| Yu et al., 2016 | 2,766 | 1,562-4,899 | 16,85 |
| Amitay et al., 2017 | 3,819 | 1,980-7,366 | 14,35 |
| Proença et al., 2018 | 3,064 | 1,089-8,623 | 7,54 |
| Saito et al., 2019 | 0,950 | 0,350-2,580 | 7,97 |
| Tunsjø et al., 2019 | 6,750 | 1,755-25,956 | 4,86 |
| Bundgaard-Nielsen et al., 2019 | 9,382 | 2,713-32,442 | 5,59 |
| D+L pooled OR | 3,244 | 2,359- 4,462 | 100,00 |
Heterogeneidad χ2=12,33 (g.l.=8) p=0,137.
I2 (variación en OR atribuible a la heterogeneidad)=35,1%.
Estimación de la varianza entre estudios Tau-cuadrado=0,0719.
Test de OR=1: z=7,24 p=0,000.
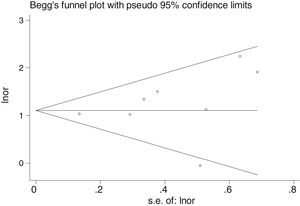
No hubo sesgo de publicación evidente, tal y como se indica en las pruebas de Begg y Egger que no fueron significativas (p=0,108 y p=0,441, respectivamente) (fig. 5).
DiscusiónEn este estudio se observa que la relación entre FN y el CCR es un tema que suscita interés, ya que hay un crecimiento exponencial de los trabajos que investigan esta relación. En cuanto al tamaño muestral de los estudios, 31 (54,4%) contaban con un número de muestras de casos inferior a 100, y solo 4 (7%) presentaban más de 1.000 muestras. Igualmente, en los trabajos publicados, solo 22 (55%) presentaban datos numéricos para los Controles-1 y 10 (17,5%) para los Controles-2. No obstante, en los últimos años se ha ido corrigiendo este hecho y en los estudios más actuales la tendencia es publicar los valores correspondientes. Ante todo, es necesario que los estudios que se lleven a cabo en el futuro presenten los datos numéricos cuantitativos para la positividad de FN en las muestras, ya que aporta una evidencia sólida y tratable estadísticamente sobre su asociación con el CCR. Otro hecho importante es la influencia del sexo en los resultados, que apenas ha sido estudiada en este campo, ya que en muchos estudios se prescinde de estos datos. El desglose por sexos de esta relación podría poner en evidencia si este podría considerarse un factor de riesgo en relación con la presencia de FN, ya que las enfermedades crónicas no transmisibles se relacionan con el estilo de vida de hombres y mujeres. En esta revisión sistemática solo se indica el sexo de los pacientes en 20 (35,1%) estudios del total de 57 artículos recopilados.
De forma semejante podrían estudiarse en futuros trabajos otras variables, como la dieta, ya que es un factor que algunos estudios, como el de Hussan et al.68, han recogido y evidenciado que una dieta rica en fibra se relaciona con un menor riesgo de desarrollar CCR a pesar de tener un mayor número de muestras positivas de FN en la mucosa colónica, lo que parecería contradictorio.
También cabe destacar que la mayoría de las muestras analizadas en los trabajos procedían de tejidos, es decir, las muestras de CCR procedían del tejido tumoral y las muestras de los controles procedían de tejido sano, ya que, en principio, su estudio debe aportar una información más completa. De lo publicado hasta la fecha, se ha deducido que existe una importante presencia de FN en el tejido del CCR en comparación con tejidos sanos adyacentes15,18,20,28,69, con un aumento gradual de FN desde la mucosa sana hasta el adenoma y de este al adenocarcinoma, lo que sugiere que FN puede jugar un papel importante desde las etapas más iniciales de la génesis tumoral colónica4,10,18,70; que hay una asociación entre esta presencia y la invasión de ganglios linfáticos por el CCR15,26,71 y que FN acompaña a las células tumorales en las metástasis38. De esta forma se ha explicado que la cantidad de FN en el CCR se relaciona con el estadio, resistencia a la quimioterapia, mayor recurrencia y menor supervivencia, siendo un factor predictivo independiente10,26,28,38,70,72,73.
Solo 4 (7%) trabajos llevaron a cabo los análisis en muestras fecales, uno (1,8%) en muestras sanguíneas y otro (1,8%) en muestras salivares, concluyendo que existe una sobreabundancia de FN en las heces de pacientes con CCR. FN es 132 veces más abundante en las heces de pacientes con CCR y su determinación se ha señalado como un marcador útil para detectar el CCR29–31, mostrando una sensibilidad y especificidad del 80,2% y 80,7%, respectivamente, que aumentan al 92-93% cuando se combina con la detección de sangre oculta en heces74. Sería de interés comprobar la asociación de FN y el CCR simultáneamente en muestras de tejido, fecales, salivares y sanguíneas, ya que en caso de aumentar los estudios y descubrir asociación significativa tendría un gran interés diagnóstico para el paciente puesto que son muestras relativamente fáciles de recoger y sin necesidad de llevar a cabo una colonoscopia para su obtención.
En base a todos los estudios realizados hasta la actualidad se podría aceptar la participación de FN en el mecanismo de carcinogénesis del CCR, ya que existe una asociación estadísticamente significativa entre la presencia de FN y el CCR; sin embargo, para determinar que esta bacteria es el verdadero origen del tumor deberían cumplirse los criterios de causalidad75, principalmente la temporalidad, dirección y asociación. En este caso no se puede demostrar el criterio de temporalidad, por el cual el efecto debe ser precedido por la causa. La mayoría de los estudios que investigan la asociación entre FN y el CCR son retrospectivos, por lo que no se puede saber si la bacteria estaba presente antes de desarrollar el tumor o fue adquirida después; entonces, se necesita más evidencia para confirmar esta causalidad.
Múltiples estudios, entre los que destacan por su peso en el metaanálisis los de Mima et al.28, Flanagan et al.18 y Tahara et al.20, confirman que existe una relación entre FN y el CCR cuando se compara tejido sano procedente de Controles-1 y los Casos de CCR, si bien es necesario clarificar esta asociación, ya que, entre otras cosas, actualmente no hay estudios prospectivos que demuestren que la presencia de la bacteria preceda al desarrollo del tumor. En la actualidad se conoce que el sustrato etiopatogénico se corresponde con la activación de un estado inflamatorio crónico derivado de la adhesión de la bacteria a través de un mecanismo de disbiosis intestinal. Todo este proceso lleva a un inicio de vías asociadas con la carcinogénesis colorrectal, destacando la vía Wnt/-beta-catenina cuya disregulación causa fallos en el crecimiento celular y progresión tumoral76. El estudio en profundidad de la activación de estas vías proinflamatorias y los mecanismos por los cuales se producen abrirá múltiples posibilidades diagnóstico-terapéuticas en uno de los tumores más incidentes de la actualidad, lo cual traería consigo un notable beneficio en el curso de esta enfermedad. Nuestro metaanálisis indica una mayor presencia de FN en muestras de tejido tumoral colorrectal con respecto a tejido sano con una OR de 4,558 y un intervalo de confianza al 95% de 3,312-6,272, lo que confirma la asociación significativa entre la presencia de FN en las muestras de tejido con el desarrollo de CCR; no obstante, es necesario clarificar el sentido de esta relación con estudios prospectivos que confirmen la temporalidad de la asociación, hecho que otros metaanálisis como el de Hussan et al.68 también mencionan en su trabajos.
Por otra parte, estudios recientes como el de Yu et al.70 describen el papel de FN en la quimiorresistencia en pacientes con CCR, indicando que un aumento de la cantidad de FN en el tejido tumoral se relaciona con una mayor tasa de quimiorresistencia. Por este motivo sería interesante la realización de más investigaciones sobre el tiempo de supervivencia de los pacientes en relación con la concentración de FN en el tejido tumoral, ya que este hecho podría significar la existencia de más dianas terapéuticas en esta enfermedad como el tratamiento erradicador de la bacteria.
En el caso de la comparación entre los Controles-2 (adenomas colorrectales) y los Casos de CCR, es importante destacar que no todos los artículos utilizaban muestras de los mismos tipos de adenomas colorrectales, estudiando una amplia variedad de tipos histológicos dentro de la denominación de adenoma colorrectal. Además, actualmente hay menos trabajos que relacionen la presencia de FN con los adenomas colorrectales, por tanto, la escasez de artículos unida a la falta de uniformidad de criterios para la selección de las muestras indica que en un futuro debería estudiarse esta asociación con más profundidad, debiéndose analizar también si FN contribuye más que a la formación del adenoma a la malignización de este.
Hay múltiples artículos, entre los que destacan Mima et al.28, Yu et al.5 y Lee et al.46, entre otros, que relacionan la supervivencia del CCR en función de la presencia o ausencia de FN, si bien no hay trabajos que demuestren que con la erradicación de la bacteria pudiera obtenerse un mejor pronóstico en el tratamiento del tumor o una mejora en el tratamiento quimioterápico. Tampoco hay estudios que relacionen el tener una buena higiene dental con la presencia de CCR, ya que al ser FN una bacteria colonizadora de la cavidad oral, podría estudiarse la relación entre su cantidad en dicha cavidad y en la mucosa colónica.
La principal limitación de esta revisión sistemática es la inclusión de trabajos publicados en MEDLINE PubMed y no en otras bases de datos ni las tesis doctorales sobre el tema. Por este motivo asumimos que, aunque escasa, se ha podido perder información publicada, que, por otro lado, presumiblemente podría ser poco relevante. En cuanto a la limitación del idioma, solo se han incluido trabajos publicados en inglés y en español, aunque la mayoría de las revistas indexadas en MEDLINE estaban publicadas en inglés, y solo se excluyeron por este motivo 2 trabajos. Incluso la asociación de mayor frecuencia de FN en las muestras de CCR no implica una causalidad que pueda permitir una estrategia preventiva. Podría ser una consecuencia del tejido tumoral que facilitara la infección posterior por FN.
ConclusionesEn base a los resultados de esta revisión sistemática y metaanálisis encontramos que existe asociación entre la presencia de FN y el CCR. Finalmente, para obtener una conclusión definitiva, es necesario que se desarrollen nuevos estudios comparativos, con un número suficiente de pacientes, que utilicen la combinación de varias técnicas microbiológicas para un mismo sujeto y muestra, y analicen simultáneamente tejidos neoplásico y sano, mediante técnicas estandarizadas y con una adecuada sensibilidad.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.