En Venezuela hay reportes de Klebsiella pneumoniae con carbapenemasa tipo KPC. Sin embargo, desde su primer reporte en el 2008, son muy escasos los estudios de epidemiología molecular que se han realizado en estos aislados.
MétodosLos objetivos de esta investigación fueron detectar la producción de betalactamasas de espectro extendido (BLEE) (blaTEM y grupo blaCTX-M-1) y determinar la relación genética de 30 aislados pertenecientes a brotes importantes de K. pneumoniae productores de carbapenemasa tipo KPC derivados por once centros sanitarios de diferentes estados de Venezuela entre enero de 2008 y diciembre de 2012 mediante electroforesis en campo pulsante (ECP).
ResultadosTodos los aislados fueron identificados como K. pneumoniae subsp. pneumoniae. Los aislados mostraron el mayor porcentaje de resistencia al ertapenem, un 97%. En todos los aislados se detectó el gen tipo KPC. El 73% presentó BLEE (en el 68% se detectó blaTEM y en el 27% blaTEM, CTX-M-1). En la ECP se detectaron 11 agrupaciones.
ConclusiónDurante los años 2008-2012 se demostró que existe una gran diversidad genética en los aislados en estudio. Se determinó que algunos aislados circularon en los 11 centros sanitarios. Los resultados de esta investigación plantean la necesidad de fortalecer la vigilancia epidemiológica y el desarrollo de actividades para prevenir y controlar este tipo de microorganismo.
In Venezuela, there have been some reports of carbapenemase KPC-producing Klebsiella pneumoniae. Nevertheless, since the first report in 2008, only a few studies have been done on their molecular epidemiology in this country.
MethodsThe aims of this study were to detect extended-spectrum betalactamase (ESBL)-producing (blaTEM and blaCTM-M-1) and to determine the genetic relationship between 30 isolates of carbapenemase KPC-producing K. pneumoniae taken from patients at eleven health centers in different states of Venezuela from January 2008 to December 2012, using pulsed-field gel electrophoresis (PFGE).
ResultsAll isolates were identified as K. pneumoniae subsp. pneumoniae. Isolates showed the highest resistance to the ertapenem, 97%. The KPC gene was detected in all studied strains. Seventy three percent showed ESBL, having the blaTEM in 68% and blaTEM, CTX-M-1 in 27% of the strains. Eleven groups were found using the field-pulsed gel electrophoresis.
ConclusionHigh genetic diversity was found during 2008-2012 in K. pneumoniae isolated at different states in Venezuela, some of them circulating at eleven health centers. Results showed the importance of performing epidemiologic studies and the need to develop some activities to control this type of microorganisms.
Klebsiella pneumoniae es considerada una de las especies de la familia Enterobacteriaceae que comúnmente puede adquirir genes de resistencia a carbapenémicos1. Entre 2001 y 2011, se reportó un incremento del 1,6 al 10,4% en el número de aislados de K. pneumoniae resistentes a carbapenémicos en Estados Unidos, debido a la producción de carbapenemasas tipo KPC. Se ha descrito que los pacientes infectados con K. pneumoniae productora de carbapenemasa tipo KPC tienen una mortalidad más alta que aquellos que se encuentran infectados con aislados productores de otros tipos de carbapenemasas (47,66 y 46,71%, respectivamente)2, considerándose como un problema de salud pública sin precedentes3.
El uso de técnicas moleculares como electroforesis en campo pulsante (ECP) ha permitido evaluar la diseminación de aislados productores de carbapenemasa tipo KPC4. Esta técnica posee un alto poder discriminatorio y buena reproducibilidad, siendo considerada la técnica estándar de referencia para la tipificación de bacterias5.
Desde el año 2008, se ha demostrado la presencia de K. pneumoniae productora de carbapenemasa tipo KPC en Venezuela, según los registros del Instituto Nacional de Higiene Rafael Rangel (datos no publicados). Posteriormente se han realizado otros reportes en Distrito Capital6, Carabobo y Zulia7 en coexistencia con otros determinantes de resistencia a betalactámicos, sin embargo, no hay reportes de la epidemiología molecular de estos primeros aislamientos en diferentes estados del país. El objetivo de la presente investigación fue detectar la producción de betalactamasas de espectro extendido (BLEE) (blaTEMyblaCTX-M-1) y determinar la relación genética mediante ECP en aislados de K. pneumoniae productores de carbapenemasa tipo KPC provenientes de diferentes centros sanitarios en diversos estados de Venezuela durante los años 2008 a 2012.
Materiales y métodosAislados estudiadosEn el Instituto Nacional de Higiene Rafael Rangel, mediante el algoritmo de la Red Latinoamericana de Vigilancia de la Resistencia a los Antimicrobianos8, los distintos centros sanitarios públicos y privados del país remiten cepas con fenotipos de resistencia inusuales para su confirmación y caracterización. Es así como se seleccionaron 30 aislados clínicos representativos de brotes hospitalarios importantes, no duplicados de diferentes centros sanitarios, de K. pneumoniae productores de carbapenemasa tipo KPC almacenados durante los años 2008-2012 en el cepario de la institución. La identificación de todos los aislados se realizó mediante pruebas bioquímicas convencionales.
La investigación cumple con los principios fundamentales de la bioética y los establecidos en la Declaración de Helsinki de la Asociación Médica Mundial9 y las pautas éticas internacionales para la investigación biomédica10.
Estudio de la sensibilidad antibióticaSe realizó por el método de Kirby-Bauer11 evaluando los discos de imipenem 10μg, meropenem 10μg y ertapenem 10μg (Difco & BBL, EE.UU.) según los puntos de corte establecidos en el documento M100-S25 del Clinical and Laboratory Standards Institute (CLSI)12. Las cepas Escherichia coli ATCC® 25922 y Pseudomonas aeruginosa ATCC® 27853 se utilizaron como controles de calidad de los discos de antibióticos.
Caracterización fenotípica de betalactamasasLa detección de carbapenemasas se realizó mediante el uso de inhibidores como el ácido 3-aminofenil-borónico 300μg/mL (Sigma-Aldrich, EE.UU.) para la búsqueda de serinoenzimas y etilendiaminotetraacético/mercaptoacetato de sodio 1.000mM para la búsqueda de metalobetalactamasas6. La detección de BLEE se realizó por el método confirmatorio de doble disco12.
Para controlar la actividad de los diferentes inhibidores se emplearon las cepas K. pneumoniae M1340313, K. pneumoniae M988514 y K. pneumoniae ATCC® 700603.
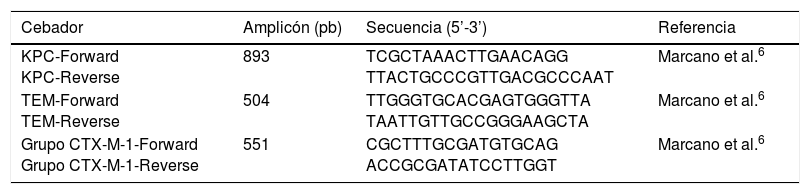
Caracterización genotípica de betalactamasasEn todos los aislados se amplificó el gen que codifica para la carbapenemasa tipo KPC. La detección de los genes que codifican para la betalactamasa tipo TEM y del grupo CTX-M-1 se realizó en aislados BLEE positivos. La detección se realizó mediante la técnica de reacción en cadena de la polimerasa (PCR) punto final. Los iniciadores empleados se muestran en la tabla 1.
Cebadores empleados para la detección de genes que codifican betalactamasas
| Cebador | Amplicón (pb) | Secuencia (5’-3’) | Referencia |
|---|---|---|---|
| KPC-Forward KPC-Reverse | 893 | TCGCTAAACTTGAACAGG TTACTGCCCGTTGACGCCCAAT | Marcano et al.6 |
| TEM-Forward TEM-Reverse | 504 | TTGGGTGCACGAGTGGGTTA TAATTGTTGCCGGGAAGCTA | Marcano et al.6 |
| Grupo CTX-M-1-Forward Grupo CTX-M-1-Reverse | 551 | CGCTTTGCGATGTGCAG ACCGCGATATCCTTGGT | Marcano et al.6 |
La extracción del ADN se realizó por el método de ebullición15 y las reacciones de amplificación y visualización de los productos se realizaron según protocolo empleado en otros estudios6.
Los controles positivos fueron: K. pneumoniae M1340313 y K. pneumoniae M988514.
Tipificación molecularLa relación genética entre los 30 aislados estudiados se determinó por ECP. Se empleó la enzima de restricción XbaI como indica el protocolo estandarizado de la red PulseNet para E. coli O157, Salmonella pps. y Shigella sonnei16. Se utilizó como estándar la Salmonella serovar Braenderup H9812 (ATCC® BAA-664).
Los resultados se analizaron con el método de UPGMA (unweighted pair group method using arithmetic average) y el coeficiente de Dice del programa Bionumeric, versión 4.0 (AppliedMaths, Sint-Martens-Latem, Bélgica). Los patrones de ECP se agruparon con una similitud ≥85%17. En el análisis de los geles de la ECP se emplearon los fragmentos de restricción con tamaños entre 1.135 a 54,7Kb. La interpretación de los patrones de restricción se basó en los criterios de Tenover et al.18. Cuando se encontraron diferencias mayores o iguales al 85% en el pulsotipo, los aislados fueron designados como tipos diferentes y se les asignaron letras (A, B, C, etc.). Cuando uno o más fragmentos fueron diferentes entre los aislados de un mismo tipo, se les asignó un subtipo, el cual fue designado con números arábigos.
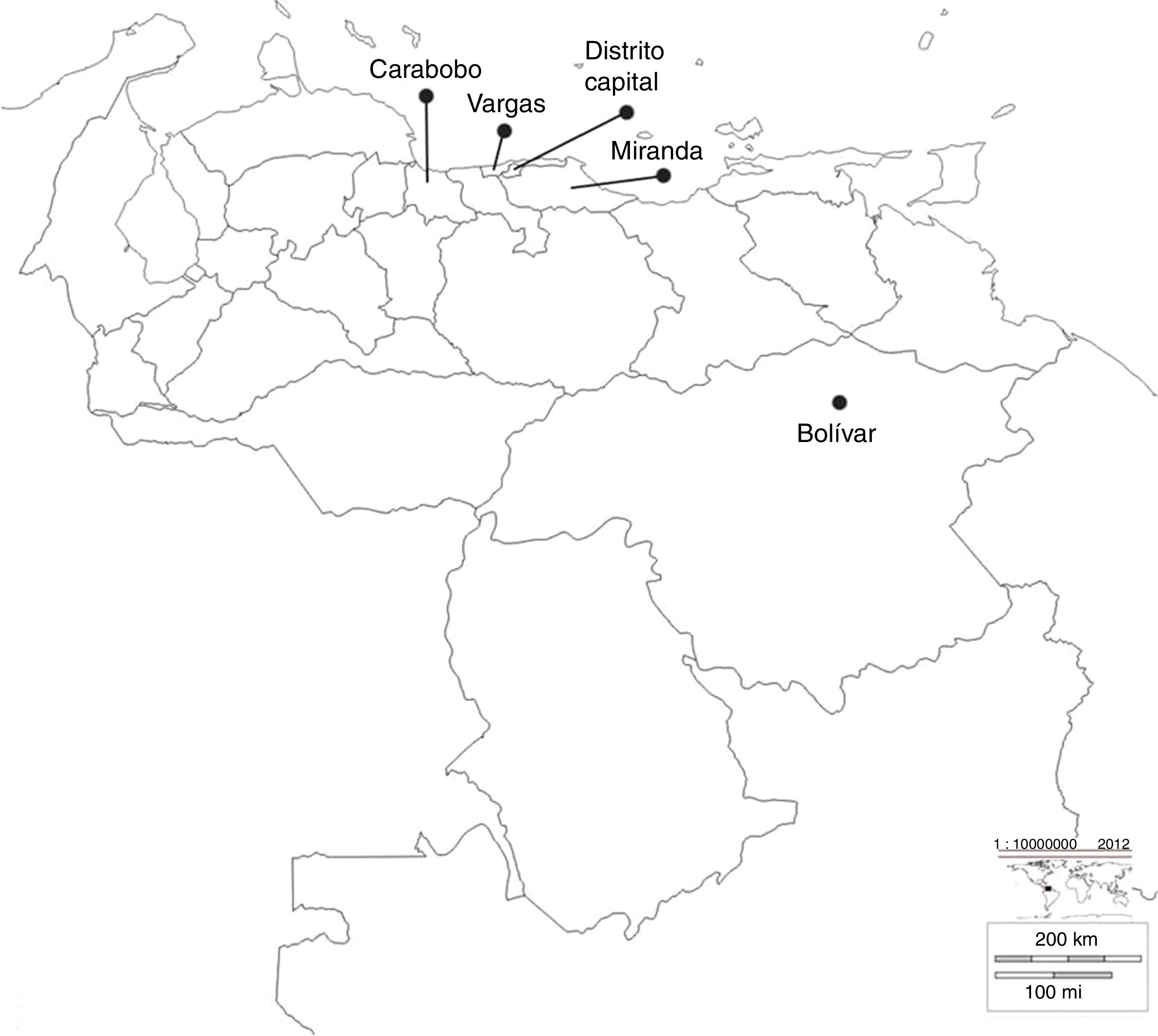
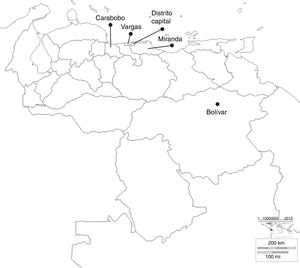
ResultadosAislados estudiadosSe analizaron 30 aislados de K. pneumoniae productores de carbapenemasa tipo KPC durante 2008-2012, provenientes de brotes importantes de diferentes centros sanitarios, ubicados en diversos estados de Venezuela, como se muestra en la figura 1.
Ubicación geográfica de los estados donde pertenecen los diferentes centros sanitarios incluidos en el estudio. Bolívar: Hospital Raúl Leoni del Instituto Venezolano de los Seguros Sociales (HRL IVSS); Carabobo: Ciudad Hospitalaria Dr. Enrique Tejera (CHET); Distrito Capital: Maternidad Santa Ana (MSA), Hospital Cardiológico Infantil Latinoamericano (HCIL), Hospital Dr. José María Vargas (HJMV), Hospital Dr. Miguel Pérez Carreño (HMP), Hospital de Niños J.M. de los Ríos (HNJMR), Clínica la Arboleda (CA) y Centro Médico Loira (CML); Miranda: Centro Médico Docente la Trinidad (CMDLT); Vargas: Hospital de Pariata (HP).
El 97, 93 y 90% de los aislados fueron categorizados como resistentes a ertapenem, meropenem e imipenem, respectivamente. El resto de los aislados fueron categorizados con susceptibilidad intermedia frente a los carbapenémicos.
Caracterización fenotípica de betalactamasasEn el 100% de los aislados se evidenció la sinergia entre el disco de ácido borónico y los carbapenémicos evaluados, demostrando la producción de serinoenzimas. Mientras que en la búsqueda de metaloenzimas no se observó sinergia con el EDTA/SMA. El 73% de los aislados fueron positivos en la detección de BLEE.
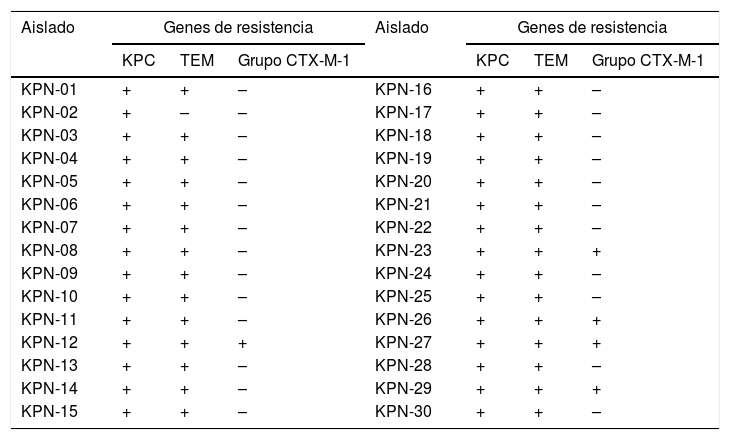
Caracterización genotípica de betalactamasasEn la tabla 2 se puede observar que en el 100% (n=30/30) de los aislados se detectó el gen que codifica para la carbapenemasa tipo KPC. En el 68% se detectó la coexistencia de blaTEM y blaKPC, mientras que en el 27% la coexistencia de blaKPC, blaTEM y blagrupoCTX-M-1.
Detección por PCR punto final de genes que codifican para betalactamasas en aislados de Klebsiella pneumoniae
| Aislado | Genes de resistencia | Aislado | Genes de resistencia | ||||
|---|---|---|---|---|---|---|---|
| KPC | TEM | Grupo CTX-M-1 | KPC | TEM | Grupo CTX-M-1 | ||
| KPN-01 | + | + | – | KPN-16 | + | + | – |
| KPN-02 | + | – | – | KPN-17 | + | + | – |
| KPN-03 | + | + | – | KPN-18 | + | + | – |
| KPN-04 | + | + | – | KPN-19 | + | + | – |
| KPN-05 | + | + | – | KPN-20 | + | + | – |
| KPN-06 | + | + | – | KPN-21 | + | + | – |
| KPN-07 | + | + | – | KPN-22 | + | + | – |
| KPN-08 | + | + | – | KPN-23 | + | + | + |
| KPN-09 | + | + | – | KPN-24 | + | + | – |
| KPN-10 | + | + | – | KPN-25 | + | + | – |
| KPN-11 | + | + | – | KPN-26 | + | + | + |
| KPN-12 | + | + | + | KPN-27 | + | + | + |
| KPN-13 | + | + | – | KPN-28 | + | + | – |
| KPN-14 | + | + | – | KPN-29 | + | + | + |
| KPN-15 | + | + | – | KPN-30 | + | + | – |
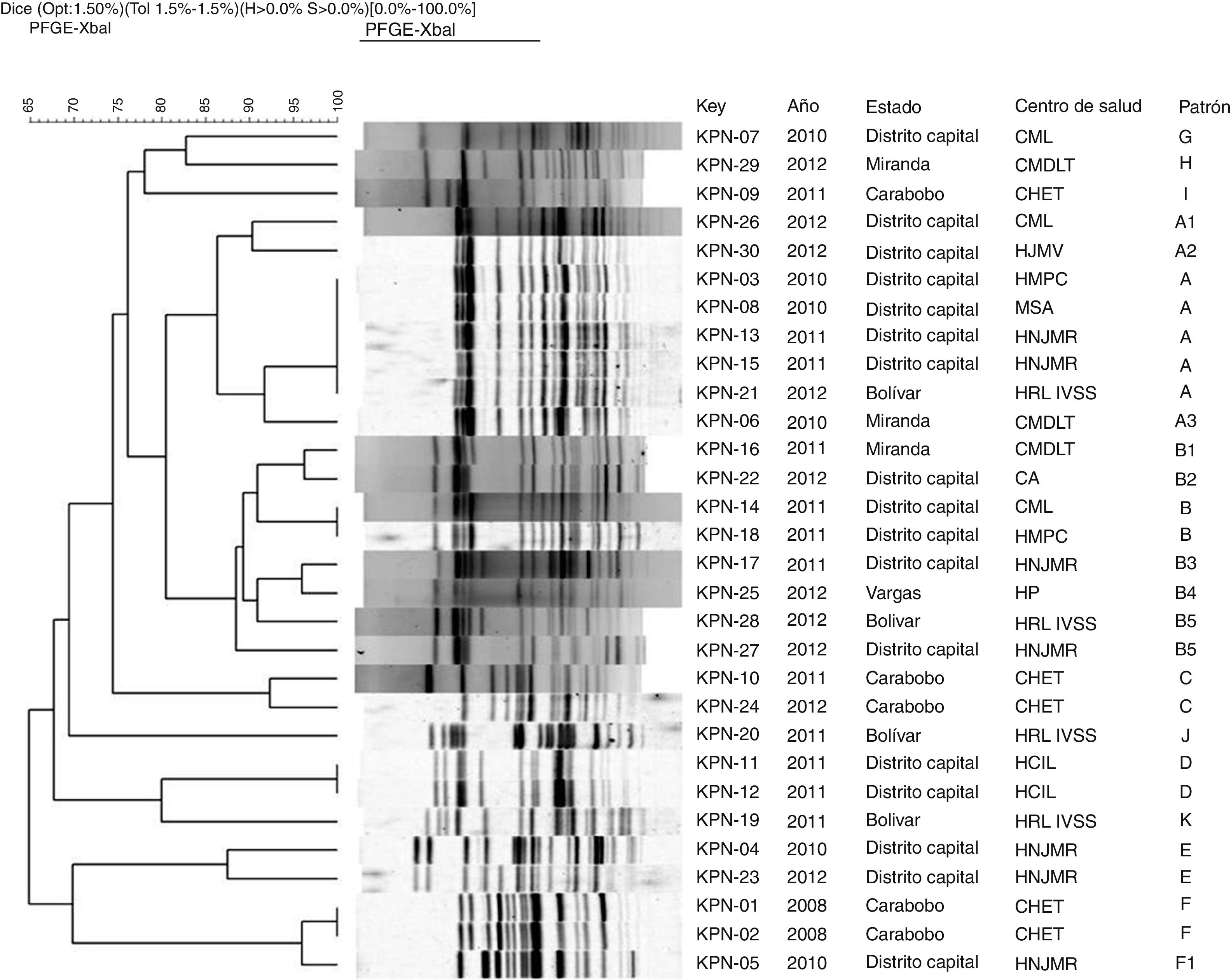
En todos los aislados se obtuvieron patrones de restricción con la enzima XbaI (Promega, EE.UU.). El número de fragmentos obtenidos en los patrones varió entre 13 y 19. En el análisis comparativo de los patrones de ADN genómico obtenidos por ECP se determinaron 11 agrupaciones de la letra A a la letra K (fig. 2), de las cuales 6 contenían al 83% de los aislamientos estudiados.
Dendrograma de ECP con enzima de restricción XbaI de 30 aislados de Klebsiella pneumoniae KPC de diferentes centros sanitarios de Venezuela durante 2008-2012. UPGMA, coeficiente de Dice tolerancia 1,5%. Optimización 1,5%.
CA: Clínica la Arboleda; CHET: Ciudad Hospitalaria Dr. Enrique Tejera; CMDLT: Centro Médico Docente la Trinidad; CML: Centro Médico Loira; ECP: electroforesis en campo pulsante; HCIL: Hospital Cardiológico Infantil Latinoamericano; HJMV: Hospital Dr. José María Vargas; HMPC: Hospital Dr. Miguel Pérez Carreño; HNJMR: Hospital de Niños J.M. de los Ríos; HP: Hospital de Pariata; HRL IVSS: Hospital Raúl Leoni del Instituto Venezolano de los Seguros Sociales; MSA: Maternidad Santa Ana; UPGMA: unweighted pair group method using arithmetic average.
En la agrupación A se detectaron 5 aislados con pulsotipos «indistinguibles» procedentes de Distrito Capital (KPN-03, KPN-08, KPC-13) y Bolívar (KPN-21), como se muestra en la figura 2. El resto de los aislados de este grupo fueron categorizados como «posiblemente relacionados» (subtipos A1-A3).
En la agrupación B, los aislados KPN-14 y KPN-18 provenientes de diferentes centros sanitarios del Distrito Capital fueron categorizados como «indistinguibles», mientras que 6 aislados de esta agrupación estuvieron «cercanamente relacionados». Estos pulsotipos corresponden a aislados derivados de centros sanitarios ubicados en diferentes estados.
Las agrupaciones C y E incluyeron, cada una, 2 aislados recuperados en años diferentes los cuales fueron categorizados como «cercanamente relacionados» y correspondieron a los centros sanitarios CHET y HNJMR, respectivamente. En las agrupaciones D y F se detectaron aislados «indistinguibles» en un mismo centro hospitalario.
Los aislados de los patrones G-K presentaron un porcentaje de similitud <85%, por lo que fueron categorizados como «aislados no relacionados», derivados entre 2010-2011 y correspondientes a 5 centros sanitarios ubicados en diferentes estados a excepción de 2 aislados recuperados en un mismo centro de salud, el Hospital Raúl Leoni del Instituto Venezolano de los Seguros Sociales (HRL IVSS), en el año 2011.
Al realizar el análisis por centro de salud, se observó que los aislados del HRL IVSS, CMDLT, HMPC y CML eran «aislados no relacionados». Sin embargo, en la CHET se detectaron clones intrahospitalarios. En el HNJMR, con un porcentaje de similitud ˃85%, se detectaron 3 agrupaciones de aislados cercanamente relacionados durante 2010-2012. Es importante resaltar que en las 3 agrupaciones se incluyeron aislados de diferentes años.
DiscusiónK. pneumoniae es un importante patógeno nosocomial, cuyos aislados productores de carbapenemasa tipo KPC han emergido como un problema significante debido a su amplia diseminación a nivel mundial mediante elementos móviles genéticos19.
En nuestro estudio, la mayoría de los aislados fueron resistentes a los carbapenémicos evaluados siendo el más afectado el disco de ertapenem, al igual que los resultados obtenidos en aislados productores de carbapenemasa tipo KPC de Noruega y Suecia20, donde el 100% de los aislados fueron resistentes a ertapenem, mientras que algunos fueron categorizados con susceptibilidad intermedia o resistentes a los otros carbapenémicos, por lo cual podría ser considerado como el disco que alertaría sobre la posible existencia de aislados de K. pneumoniae productores de carbapenemasa tipo KPC. Sin embargo, el uso exclusivo del ertapenem como antibiótico indicador de la sospecha de carbapenemasa tipo KPC presenta problemas de especificidad3.
La presencia de blaKPC en todos los aislados demostró la amplia distribución de este gen en diferentes aislados de Venezuela. Otros estudios realizados de manera aislada han reportado blaKPC en K. pneumoniae proveniente de los estados Zulia y Carabobo7. Cuzon et al. reconocen que, fuera de los Estados Unidos, K. pneumoniae productora de KPC es reportada con mayor frecuencia, encontrándose en países como Francia, Israel, Colombia, Brasil, Argentina, China, Grecia, Alemania, España, Reino Unido, Bélgica, Croacia, Noruega y Polonia, entre otros1,21.
El gen blaKPC ha sido reportado en plásmidos conjugativos en combinación con otros genes de resistencia que codifican para las betalactamasas del grupo CTX-M-1, TEM y SHV7. En los aislados en estudio, fue evidente la amplia distribución del gen que codifica para la betalactamasa tipo TEM, encontrándose en algunos casos en coproducción con las betalactamasas del grupo CTX-M-1. Es interesante destacar que, en un estudio realizado en América Latina, no se reporta el gen TEM en aislados de Venezuela, pero sí en alta incidencia en Guatemala, mientras que blaCTX-M fue detectado en aproximadamente el 50% de los aislados22. El estudio realizado por Cuzon et al., en diferentes países, demuestra la presencia de TEM-1 y SHV-11 en aislados de Estados Unidos, Grecia, Colombia, Brasil e Israel. Igualmente, CTX-M-2, CTX-M-12 y CTX-M-15 fueron reportadas en estos países a excepción de Estados Unidos, Grecia y Suiza, lo cual demuestra la amplia diseminación de este tipo de betalactamasa21.
Los resultados obtenidos por ECP demostraron una gran diversidad genética entre los aislados, a diferencia de lo reportado en Argentina por Gómez et al., quienes determinaron 5 agrupaciones en 69 aislados recolectados entre 2006-201023. Una de ellas incluía el 91% de los aislados, concluyendo que la diseminación en Argentina se debió a la existencia de un aislado epidémico, el cual posteriormente por multilocus sequence typing fue identificado como ST-258.
Sin embargo, los resultados de Cuzon et al. son acordes con los resultados obtenidos en nuestro estudio21. Ellos reportaron en 16 aislados de Colombia, Brasil, Israel, Estados Unidos, Suecia y Grecia heterogeneidad entre los aislados productores de carbapenemasa tipo KPC al determinar 9 pulsotipos. En China, Cao et al. demostraron que la diversidad genética en aislados de K. pneumoniae portadores de múltiples genes de resistencia, incluyendo blaKPC, permitía inferir sobre la fácil transmisión de los determinantes de resistencia entre especies bacterianas a través de elementos móviles genéticos24. Estos resultados fueron confirmados por la detección de una alta prevalencia de integrones y plásmidos en su estudio.
Además de observar una gran variedad de patrones, se determinó que algunos pulsotipos estuvieron circulando durante los años de estudio en los 11 centros sanitarios de diferentes estados de Venezuela, demostrando el éxito de este tipo de aislados para mantenerse en el tiempo y diseminarse, como se ha demostrado en otros estudios25.
La presencia de aislados similares de diferentes años y centros sanitarios podría estar relacionada con mecanismos tales como la diseminación inter e intrahospitalaria, colonización de pacientes, profesionales de la salud, etc.; equipo médico o fómites como vehículo de transmisión y el uso de antibióticos de amplio espectro26. Robledo et al. infieren que la combinación de múltiples factores contribuyen en la diseminación de aislados de K. pneumoniae productora de carbapenemasa tipo KPC, entre los cuales señalan: fallas en el equipo de salud en el seguimiento de las políticas establecidas para el control de infecciones; incremento del tránsito del personal y equipo médico de diferentes hospitales; la tardía detección de pacientes colonizados o infectados con microorganismos multidrogorresistentes adquiridos en otras instituciones; carencia de un sistema de alerta entre los laboratorios, y/o el uso inadecuado de antibióticos26. Estudios realizados en España y Grecia han inferido sobre la posible contaminación cruzada entre pacientes y el equipo médico, originando brotes importantes de K. pneumoniae productora de carbapenemasa tipo KPC. Sin embargo, en países como Israel, se ha determinado que los pacientes colonizados por este tipo de microorganismo juegan un rol importante en la diseminación de estos aislados27.
En los patrones obtenidos, se observaron aislados genéticamente relacionados en un mismo centro hospitalario en diferentes años, lo cual podría indicar la permanencia del microorganismo en el ambiente. Este factor se encuentra favorecido por las diferentes propiedades y características de esta bacteria. Algunos autores señalan que los seres humanos pueden ser portadores de K. pneumoniae durante muchos años, con el riesgo de adquirir infecciones causadas por este microorganismo y diseminarlo tanto en ambientes hospitalarios como en la comunidad o transmitirse entre personas, así como entre diferentes lugares de un mismo hospital y entre ciudades y países28.
En el análisis de los pulsotipos por años, se determinó que la diversidad genética de los aislados fue incrementándose en el transcurso del tiempo. Es posible que la existencia de subtipos esté relacionada con la sumatoria de eventos genéticos. La Sociedad Española de Infectología y Microbiología Clínica29 señala que este tipo de comportamiento es de esperar, pues al inicio o en la detección de los primeros aislados es probable que la diversidad genética sea baja, sin embargo, al irse acumulando cambios genéticos en las cepas epidémicas o endémicas posiblemente se incrementará la variedad de los patrones obtenidos por ECP.
De igual manera, Sabat et al. refieren que las inserciones o deleciones de elementos móviles genéticos, así como eventos de recombinación en el ADN genómico, pueden resultar en algunos casos como cambios en los patrones de ECP30.
En esta investigación, se evidenció la variabilidad de aislados productores de carbapenemasa de tipo KPC mediante ECP (policlonalidad) en Venezuela, describiendo aislados que permanecieron en el tiempo y la posible diseminación inter e intrahospitalaria en aislados que se encontraron genéticamente relacionados. En este sentido, es necesario fortalecer la vigilancia epidemiológica y el desarrollo de actividades para prevenir y controlar este tipo de aislados en los centros sanitarios de Venezuela.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Este estudio ha sido posible gracias al apoyo del Departamento de Bacteriología; Gerencia de Diagnóstico y Vigilancia Epidemiológica y la Gerencia de Docencia del Instituto Nacional de Higiene’“Rafael Rangel”. La colaboración del personal de las diferentes secciones de bacteriología de los centros sanitarios quienes derivaron las cepas estudiadas. La colaboración del personal del Laboratorio de Aislamiento e Identificación Bacteriana del Instituto Nacional de Higiene Rafael Rangel. La Dra. Omaira Da Mata y la Lcda. Cirana Rodríguez por la asesoría en la técnica de ECP.