Pseudomonas aeruginosa coloniza la mucosa respiratoria del paciente con fibrosis quística (FQ), seleccionándose en general una única cepa dominante en un proceso de variación genética.
MétodosSe obtuvieron secuencialmente 123 aislados de P. aeruginosa en 6 pacientes con FQ atendidos durante su seguimiento habitual o en las exacerbaciones a lo largo de 2-12 años en el Hospital Universitario Ramón y Cajal (Madrid). Otros 13 aislados se obtuvieron de un sólo paciente en un estudio a corto plazo (7 días). Se estudió su sensibilidad antimicrobiana por microdilución y caracterizaron mediante polimorfismo de fragmentos de restricción (RFLP) y secuenciación de los genes mucA y fpvA, que codifican respectivamente el regulador de la síntesis de alginato y un receptor de pioverdina.
ResultadosSe identificó en cada paciente una cepa dominante según el perfil de RFLP. Los polimorfismos de los genes mucA y fpvA se correlacionaron bien con dicho perfil, pero en dos pacientes hermanos se observó una relación entre cepas no evidente por RFLP. Las mutaciones sin sentido en mucA fueron exclusivas de la cepa dominante de cada paciente, reflejando el proceso adaptativo. La detección alternativa de los mismos polimorfismos en mucA o fpvA mostró la coexistencia de subpoblaciones en cada paciente. Esta hipótesis se confirmó en el estudio prospectivo de 7 días al aislar 6 variantes en un único paciente.
ConclusionesEn la colonización crónica por P. aeruginosa en pacientes con FQ coexisten variantes genotípicas no siempre detectadas por RFLP y con diferentes perfiles de sensibilidad.
Pseudomonas aeruginosa is able to colonize the lungs of cystic fibrosis patients (CF) in an adaptive process that results in the selection of a dominant strain through a process of genetic variation.
MethodsOne hundred and twenty tree isolates of P. aeruginosa were sequentially recovered from 6 CF patients during the routine follow-up or exacerbations over periods of 2 to 12 years in the Ramon y Cajal University Hospital (Madrid, Spain). Another 13 isolates were obtained from a single CF patient in a short-term study. They were analysed by restriction fragment length polymorphism (RFLP) and sequencing of mucA and fpvA genes, which code for the alginate biosynthesis regulator and a pyoverdin receptor, respectively, and their antibiotic susceptibility was studied by microdilution.
ResultsA dominant colonising strain was found in each patient based on the RFLP profile. The polymorphisms of mucA and fpvA genes correlated well with these profiles, but suggested a relationship between strains isolated from two brothers, not inferred by RFLP. Stop codon mutations in mucA were unique to each dominant strain, indicating the adaptive process suffered. The alternate detection of the same mucA and/or fpvA genotypic variants suggested the coexistence of several subpopulations. This hypothesis was confirmed in a prospective study in which 6 variants were isolated in 7 days from the same patient.
ConclusionsGenotypic variants of the P. aeruginosa dominant strains can coexist in the chronic colonization in CF patients. These variants can be undetected by RFLP and they might present variable antibiotic susceptibility.
La fibrosis quística (FQ) es una alteración hereditaria causada por mutaciones recesivas del gen que codifica el regulador de conductancia transmembrana de FQ (CFTR). La resultante pérdida de fluidez en las secreciones bronquiales facilita la colonización crónica por algunos patógenos oportunistas. Entre ellos, Pseudomonas aeruginosa es la bacteria mejor adaptada y contribuye significativamente a la morbilidad y mortalidad ligada a la FQ1–3. La colonización se produce por cepas ambientales, y suele hacerse permanente tras una conversión a fenotipo mucoide, haciéndose muy difícil la erradicación debido a la formación de biofilm4–6. El análisis genotípico en diferentes estudios ha mostrado que la colonización crónica se debe generalmente a una sola cepa de P. aeruginosa que se hace dominante, siendo infrecuente la adquisición de otras7–12. Recientemente se ha demostrado la persistencia de P. aeruginosa, a diferencia de Staphylococcus aureus o Haemophilus influenzae, en un modelo experimental en ratón de colonización crónica de la mucosa respiratoria13.
El proceso de colonización patogénica adaptativa de P. aeruginosa implica cambios genéticos, y los aislados en las secreciones respiratorias de los pacientes con FQ presentan una amplia variedad fenotípica. La más evidente es el morfotipo mucoide, debido a mutaciones en el gen mucA regulador del operón biosintético de alginato y en otros genes de ese operón2,14,15. En fases más tardías de colonización es frecuente la pérdida del fenotipo mucoide por reparación génica o, más comúnmente, por supresión extragénica16. Estos cambios se favorecen por procesos de hipermutación, frecuentes en estos aislados y que facilitan también la resistencia a antibióticos17.
Con el objetivo de conocer mejor la diversidad de las poblaciones de P. aeruginosa que colonizan la mucosa respiratoria del paciente de FQ, hemos analizado la secuencia de dos genes que experimentan gran variabilidad en esta bacteria: el propio mucA, y el que codifica el receptor del sideróforo pioverdina, fpvA. Aunque, como se ha citado, el fenotipo mucoide suele revertir, lo habitual es que las mutaciones sufridas en mucA se mantengan, quedando la huella del proceso adaptativo, por lo que se ha utilizado su polimorfismo para caracterizar aislados clínicos7,16,18. El receptor de pioverdina, FpvA, está codificado en una de las regiones más variables del genoma de P. aeruginosa15, y es posible que esté sujeto a selección positiva durante el proceso de colonización19. El uso de estos dos marcadores puede permitir conocer la presencia de variantes de la cepa colonizadora dominante que no se aprecien por polimorfismo de restricción (RFLP).
En el presente trabajo se ha realizado un análisis genotípico mediante RFLP y polimorfismo de un sólo nucleótido (SNP) de los aislados de 6 pacientes de FQ, obtenidos durante su seguimiento habitual o en las exacerbaciones del cuadro respiratorio en la unidad de FQ del Hospital Universitario Ramón y Cajal (Madrid). Posteriormente, se ha llevado a cabo un estudio prospectivo a corto plazo (una semana) en otro paciente para confirmar la coexistencia de variantes de una misma cepa colonizadora de P. aeruginosa.
MétodosPacientes, muestras, aislados bacterianos y sensibilidadLas muestras de esputo se obtuvieron durante exacerbaciones o en el seguimiento normal de 6 pacientes (denominados aleatoriamente FQ1 a FQ6) entre agosto de 1996 y mayo de 2007, así como durante un estudio prospectivo de siete días de un único paciente (FQ7). La cohorte estuvo compuesta por 4 hombres y 3 mujeres, con edades comprendidas entre 6 y 35 años, e incluyó dos hermanos (FQ2 y FQ3). Este estudio se realizó cumpliendo los requisitos éticos del Hospital Universitario Ramón y Cajal.
Las muestras de esputo se homogenizaron con N-acetil-cisteína y se procesaron mediante una técnica cuantitativa20. Los cultivos en agar Columbia con 5% de sangre y agar de MacConkey se incubaron aeróbicamente 24h a 37°C, seguidas de 24h a 25°C. Además, se inocularon placas de agar chocolate con bacitracina y se incubaron 48h at 37°C con 5% de CO2. Cada colonia fenotípicamente distinta de cada muestra de esputo se aisló e identificó mediante la reacción de la oxidasa y pruebas bioquímicas, utilizando paneles PASCO (Difco, Detroit, MI, EE. UU.) o WIDER (Fco. Soria Melguizo, Madrid, España). Estos mismos paneles se utilizaron para el estudio de sensibilidad a antibióticos interpretándose los resultados siguiendo los criterios del Clinical and Laboratory Standards Institute (CLSI). Ciento treinta y cinco aislados se identificaron como P. aeruginosa y fueron seleccionadas para este estudio.
Análisis del polimorfismo de restricción (RFLP)Los aislados de P. aeruginosa se cultivaron durante 18h a 37°C en agar Columbia con 5% de sangre y se resuspendieron (OD420=0,5) en tampón Tris 100mM, EDTA 100mM, pH 8,0. Se añadió proteinasa K a una concentración final de 30mgml−1 y la suspensión (400μl) se mezcló en proporción 1:1 con agarosa de bajo punto de fusión al 1% [p/v]. Las bacterias embebidas en el agar se trataron 1h a 54°C con proteinasa K (0,2mgml−1) en tampón de lisis (N-lauril-sarcosinato 1% [p/v] en Tris 50mM, EDTA 50mM, pH 8,0). El ADN librado se digirió con 40 U de endonucleasa SpeI durante una noche a 37°C en 100μl del tampón suministrado por el fabricante (Roche Diagnostics GmbH, Mannheim, Alemania). Los fragmentos de ADN se separaron por electroforesis en campo pulsado (PFGE) en un equipo CHEF-DR III (Bio-Rad, Hercules, CA, EEUU) utilizando geles de agarosa al 1,2% (p/v). Las condiciones de electroforesis fueron: una rampa lineal de 20 a 60 seg. durante 40h a 5Vcm−1, seguida de una rampa lineal de 5 a 10 seg. durante 20h a 6Vcm−1. Para la calibración y comparación de los geles se utilizó ADN de la cepa PAO-1, empleando el programa Diversity Database (Bio-Rad) y aplicando los criterios de similitud de Tenover et al21.
Análisis de los genes mucA y fpvAPara la amplificación por PCR de un fragmento interno del gen mucA (nt 42 a 544) se diseñaron los cebadores 5′-GGATAACGAAGCGGATGAAC-3′ y 5′-CGCTCTCTGTACCACTGACG-3′. Las condiciones empleadas para la PCR fueron: 1min a 95°C; 30 ciclos de 30 seg. a 95°C, 30 seg. a 54°C y 30s at 72°C; finalizando con un ciclo de 4min a 72°C. Para la amplificación de los alelos fpvA correspondientes a los tipos I, II y III se emplearon los cebadores FpvAI-1F/FpvAI-1R, FpvAII-2F/FpvAII-2R, y FpvAIII-3F/FpvAIII-3R descritos por Chial et al., obteniendo fragmentos de 326, 897, y 506 nt, respectivamente22. Los productos de PCR se purificaron mediante el GFX PCR DNA Purification Kit (GM Health Care, Buckinghamshire, UK) y se secuenciaron en doble cadena. Como cepa de referencia para detectar cambios en las secuencias de mucA y fpvA tipo I se utilizó P. aeruginosa PAO-1 (GeneBank AE004091). Las secuencias de los genes fpvA de tipo II se compararon con las de las cepas P. aeruginosa MSH (AY765263) y PA7 (CP000744), según la similitud detectada con el programa BLAST (www.ncbi.nlm.nih.gov/blast/Blast.cgi). Cada nuevo cambio detectado se comprobó mediante una nueva amplificación y secuenciación para evitar los errores de la ADN polimerasa.
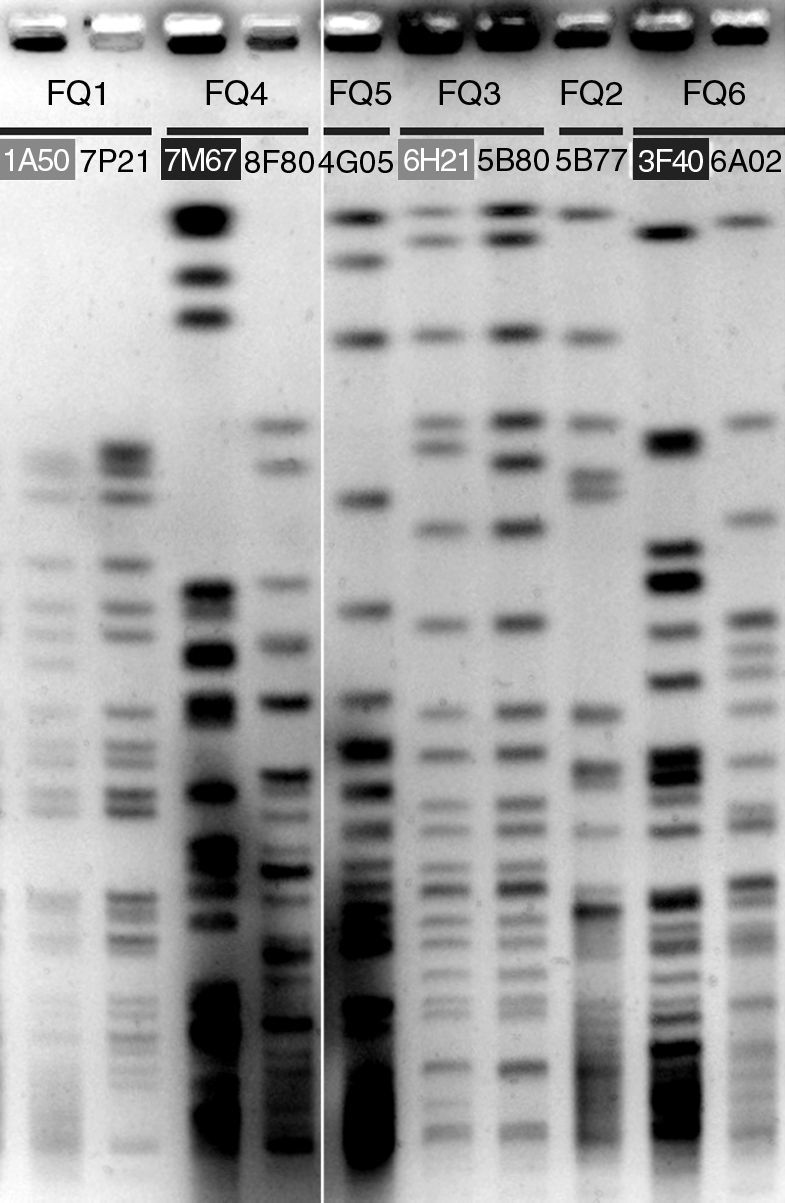
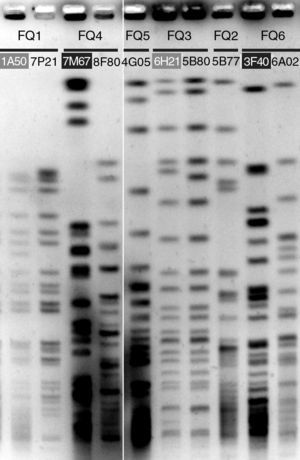
ResultadosRFLPLos perfiles de RFLP resultaron idénticos para todos los aislados de cada uno de los pacientes, indicando en cada caso la colonización por un único clon, con tan sólo cuatro excepciones. En dos aislados procedentes de los pacientes FQ1 y FQ3 (1A50 y 6H31 respectivamente; fig. 1) se observaron diferencias sólo en una o dos bandas con respecto al perfil del clon, mientras que en los pacientes FQ4 y FQ6 se detectó una cepa en cada uno de ellos (7M67 y 3F40 respectivamente; fig. 1) con un perfil totalmente distinto al del clon dominante, indicando una colonización temporal por una cepa distinta.
PFGE de las digestiones de ADN total con SpeI. Se eligió un aislado representativo del perfil RFLP dominante de cada paciente. Los restantes aislados que se muestran presentaban perfiles muy relacionados (rotulados con fondo gris) o totalmente diferentes (rotulados con fondo negro) del dominante.
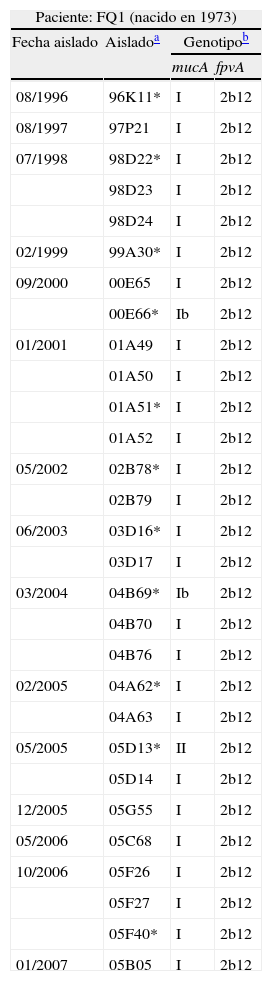
Al alinear las secuencias del gen mucA de los diferentes los aislados con la de la cepa PAO1 se detectaron 12 polimorfismos de un nucleótido (SNPs), cuya agrupación en los distintos aislados permitió definir tres genotipos: I (A342G) II (T126C, T198G, A243G, A342G, T381G) y III (T133C, C156T, A342G). La presencia de cambios adicionales con sustitución de aminoácidos (indicada entre corchetes) se utilizó para definir 5 variantes: Ia (I+C188G [A→G]), Ib (I+C394T [P→S]), IIa (II+C469T [R→C]), IIab (IIa+C467T [T→I]) y IIIa (III+C424A [Q→K]). Así mismo, se identificaron 8 mutaciones sin sentido que determinaban la aparición de un nuevo codón de terminación (en la posición indicada entre corchetes): Tres inserciones (G∴160 [nt 478], C∴171 [nt 478]; y GCCTGCT∴202 [nt 484]), cuatro deleciones (ΔT180 [nt 283], Δ187-197 [nt 466], ΔG430 [nt 439] y ΔC453 [nt 752]) y una transversión (C424T [nt 424]). El codón de terminación normal de mucA se sitúa en posición 584. La distribución por aislados y pacientes de muestra en la tabla 1.
Genotipado mucA/fpvA de los aislamientos de Pseudomonas aeruginosa
| Paciente: FQ1 (nacido en 1973) | |||
| Fecha aislado | Aisladoa | Genotipob | |
| mucA | fpvA | ||
| 08/1996 | 96K11* | I | 2b12 |
| 08/1997 | 97P21 | I | 2b12 |
| 07/1998 | 98D22* | I | 2b12 |
| 98D23 | I | 2b12 | |
| 98D24 | I | 2b12 | |
| 02/1999 | 99A30* | I | 2b12 |
| 09/2000 | 00E65 | I | 2b12 |
| 00E66* | Ib | 2b12 | |
| 01/2001 | 01A49 | I | 2b12 |
| 01A50 | I | 2b12 | |
| 01A51* | I | 2b12 | |
| 01A52 | I | 2b12 | |
| 05/2002 | 02B78* | I | 2b12 |
| 02B79 | I | 2b12 | |
| 06/2003 | 03D16* | I | 2b12 |
| 03D17 | I | 2b12 | |
| 03/2004 | 04B69* | Ib | 2b12 |
| 04B70 | I | 2b12 | |
| 04B76 | I | 2b12 | |
| 02/2005 | 04A62* | I | 2b12 |
| 04A63 | I | 2b12 | |
| 05/2005 | 05D13* | II | 2b12 |
| 05D14 | I | 2b12 | |
| 12/2005 | 05G55 | I | 2b12 |
| 05/2006 | 05C68 | I | 2b12 |
| 10/2006 | 05F26 | I | 2b12 |
| 05F27 | I | 2b12 | |
| 05F40* | I | 2b12 | |
| 01/2007 | 05B05 | I | 2b12 |
| Paciente: FQ2 (nacido en 1977) | |||
| Fecha aislado | Aisladoa | Genotipob | |
| mucA | fpvA | ||
| 09/2000 | 0E67* | III, ΔT180, Δ187-197 | 2b1 |
| 0E68* | III, Δ187-197 | 2b1 | |
| 11/2001 | 1F76* | III, Δ187-197 | 2b1 |
| 11/2002 | 2E43* | III, Δ187-197 | 2b1 |
| 2E44* | IIIa, Δ187-197 | 2b1 | |
| 03/2003 | 3B45* | III, Δ187-197 | 2b1 |
| 3B46 | IIIa | 2b1 | |
| 03/2004 | 4B48* | III, Δ187-197 | 2b1 |
| 02/2005 | 5B11* | III, Δ187-197 | 2b1 |
| 5B12 | IIIa, Δ187-197 | 2b1 | |
| 04/2005 | 5B76* | III, Δ187-197 | 2b1 |
| 5B77* | IIIa, Δ187-197 | 2b1 | |
| 06/2005 | 5D34* | III, Δ187-197 | 2b1 |
| 5D35 | IIIa, Δ187-197 | 2b1 | |
| 5D36 | IIIa, Δ187-197 | 2b1 | |
| 08/2005 | 5E41 | IIIa, Δ187-197 | 2b1 |
| 5E42* | III, Δ187-197 | 2b1 | |
| 09/2005 | 5F25* | III, Δ187-197 | 2b1 |
| 04/2006 | 6C26* | IIIa, Δ187-197 | 2b1 |
| 6C28* | III, Δ187-197 | 2b1 | |
| 08/2006 | 6E27 | IIIa, Δ187-197 | 2b1 |
| 12/2006 | 6H28* | III, Δ187-197 | 2b1 |
| 6H29 | IIIa, Δ187-197 | 2b1 | |
| 03/2007 | 7C14* | IIIa, Δ187-197 | 2b1 |
| Paciente: FQ3 (nacido en 1979) | |||
| Fecha aislado | Aisladoa | Genotipob | |
| mucA | fpvA | ||
| 04/2005 | 5B80 | III | 2b |
| 5C01* | III, G::160, ΔC453 | 2b | |
| 5C02 | III, ΔC453 | 2b | |
| 05/2005 | 5C51* | III, ΔC453 | 2b |
| 5C52 | III | 2b2 | |
| 10/2006 | 6F56 | III | 2b2 |
| 6F57 | III* | 2b | |
| 12/2006 | 6H21* | III, ΔC453, GCCTGCT::202 | 2b |
| 6H22 | III | 2b2 | |
| 6H23 | III | 2b2 | |
| Paciente: FQ4 (nacido en 1987) | |||
| Fecha aislado | Aislado | Genotipoa | |
| mucA | fpvA | ||
| 02/1997 | 7M67 | IIa, 171::C | 2c |
| 08/1998 | 8D51* | II, ΔG430 | 2c |
| 10/1998 | 8E62* | II, ΔG430 | 2c |
| 8E63 | II | 2c | |
| 12/1998 | 8F79* | II, ΔG430 | 2c |
| 8F80 | II | 2c | |
| 12/2004 | 4G62* | II, ΔG430 | 2c |
| 4G63 | II, ΔG430 | 2c | |
| 4G73* | II, ΔG430 | 2c | |
| 02/2007 | 7B73* | II, ΔG430 | 2c |
| 7B74 | II, ΔG430 | 2c | |
| 7B75* | II, ΔG430 | 2c | |
| 05/2007 | 7D62* | II, ΔG430 | 2c |
| Paciente: FQ5 (nacido en 1991) | |||
| Fecha aislado | Aislado | Genotipoa | |
| mucA | fpvA | ||
| 08/1997 | 7P23 | II | 2a1 |
| 08/1998 | 8D66 | II, C424T | 2a1 |
| 04/1999 | 9B44 | II | 2a1 |
| 9B45 | II | 2a1 | |
| 09/2000 | 0E05 | II | 2a1 |
| 0E06 | II | 2a1 | |
| 0E07 | II | 2a1 | |
| 10/2001 | 1E56 | II | 2a1 |
| 1E57 | II | 2a1 | |
| 10/2002 | 2D50 | II | 2a1 |
| 2D51 | II | 2a1 | |
| 09/2003 | 3E19 | I | 2a11 |
| 11/2004 | 4G03* | II, C424T | 2a1 |
| 4G04 | II, C424T | 2a1 | |
| 4G05* | II | 2a1 | |
| 4G06 | II | 2a1 | |
| 01/2005 | 5A17* | II, C424T | 2a1 |
| 5A19* | II, C424T | 2a1 | |
| 02/2005 | 5B16* | II | 2a1 |
| 5B17 | II, C424T | 2a1 | |
| 05/2005 | 5C38 | II, C424T | 2a1 |
| 5C39 | II, C424T | 2a1 | |
| 5C40 | II | 2a1 | |
| 10/2005 | 5F66* | II, C424T | 2a1 |
| 5F67 | II, C424T | 2a1 | |
| 02/2006 | 6B15* | II | 2a11 |
| 6B16 | II | 2a1 | |
| 06/2006 | 6D79 | II, C424T | 2a1 |
| 09/2006 | 6E55 | II, C424T | 2a1 |
| 6E56* | II, C424T | 2a11 | |
| 01/2007 | 7A22 | II, C424T | 2a11 |
| Paciente: FQ6 (nacido en 2002) | |||
| Fecha aislado | Aislado | Genotipoa | |
| mucA | fpvA | ||
| 10/2003 | 3F20 | Ia | 1 |
| 11/2003 | 3F40 | II | 1 |
| 09/2004 | 4E46 | Ia | 1 |
| 12/2004 | 4G77 | Ia | 1 |
| 4G78 | Ia | 1 | |
| 4G79 | Ia | 1 | |
| 05/2005 | 5C32 | I | 1 |
| 04/2005 | 5B69 | Ia | 1 |
| 06/2005 | 5D22 | Ia | 1 |
| 01/2006 | 6A02 | Ia | 1 |
| 02/2006 | 6B20 | Ia | 1 |
| 02/2007 | 7B45 | Ia | 1 |
| 7B46 | Ia | 1 | |
| 05/2007 | 7D43* | Ia | 1 |
Un asterisco indica aislado mucoide; se señalan con fondo gris aquellos con perfil PFGE distinto de la cepa dominante, y con fondo gris los que presentaron un perfil relacionado (fig. 1).
En la mayoría de los aislados se obtuvo amplificación positiva con los cebadores correspondientes al receptor de pioverdina tipo 2 (121 de 135; 89,6%), siendo los de restantes de tipo 1; no se detectó ninguno de tipo 3. La secuencias amplificadas del gen fpvA se alinearon con las representantes de los tipos descritos de receptor FpvA19,23: PAO1 para el tipo 1 y MSH, 2-146 y W15Dec11 para las diferentes subtipos del tipo 2. Dentro de los genes fpvA de tipo 2 se detectaron 20 SNPs, agrupados en 9 genotipos FpvA. Todos los aislados del tipo 2a presentaban el polimorfismo G960T, denominándolo subtipo 2a1. Algunos aislados presentaron, además de este cambio, transversiones que han permitido definir más variantes: 2a11 (2a1+G1315A [G→S]), 2a12 (2a1+G1283A [G→D]), 2a121 (2a12+C1258T [R→C]) y 2a1211 (2a121+C1466T [A→V]). Del mismo modo, dentro del tipo 2b se definieron las siguientes variantes: 2b1 (+ A699G), 2b11 (2b1+C1134T) y 2b2 (G1066A [V→M]). Finalmente, 13 aislados presentaron un alelo fpvA que no se correspondía con ninguna de las secuencias publicadas23, presentando los siguientes cambios con respecto a la de tipo 2b: A699G, G804A, C852T, G856A, C861T, C975T, A1044G, C1140T, G1152C, T1153C [F→L], T1161C, C1177T [L→F], G1242A y G1308A. Esta variante se denominó 2c. La distribución por aislados y pacientes de muestra en la tabla 1.
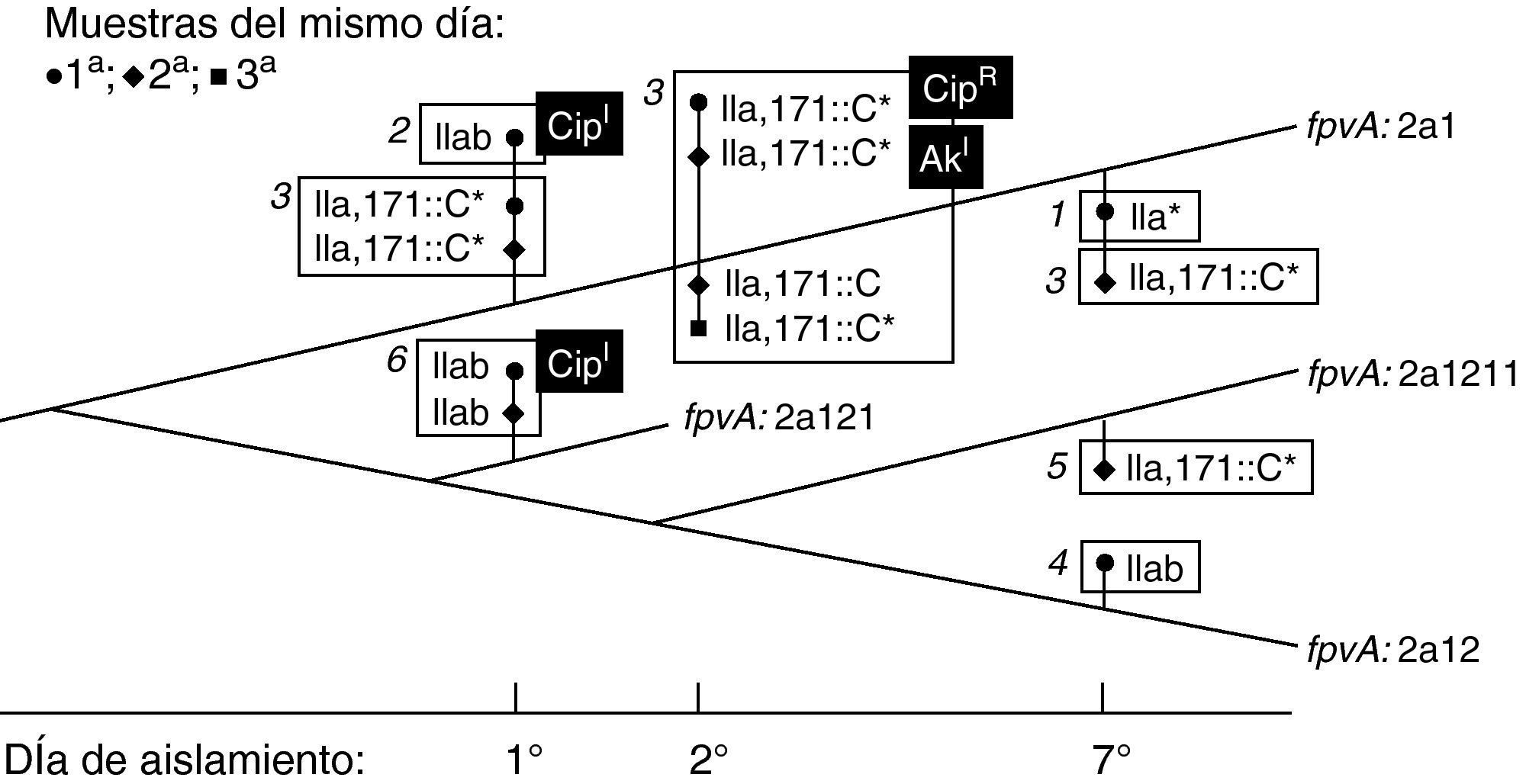
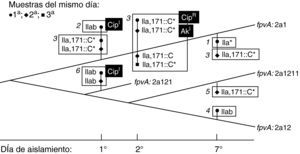
Coexistencia de variantes de la cepa dominantePara discriminar si las variantes detectadas por sus diferencias genotípicas en mucA y fpvA aparecen y desaparecen secuencialmente, o por el contrario pueden persistir en las secreciones respiratorias, se analizaron 7 muestras de esputo tomadas en tres días de una semana, procedentes de un paciente (FQ7) con colonización crónica establecida desde 1994. Se obtuvieron 13 aislamientos con morfotipos diferentes, con el mismo perfil PFGE (no mostrado), pero con diferencias en los genotipos mucA o fpvA. De este modo se pudieron definir 6 variantes genotípicas y proponer su aparición en base a la adquisición de las distintas mutaciones, como se esquematiza en la figura 2. Dos de los aislados mostraron sensibilidad intermedia a ciprofloxacino; un tercero era resistente a este antibacteriano, y un cuarto aislado presentó sensibilidad intermedia a amikacina, siendo los restantes 9 sensibles a ambos antibacterianos (fig. 2).
Representación de la posible evolución de la cepa de P. aeruginosa colonizadora del paciente FQ7, según el análisis de 13 aislados obtenidos de 7 muestras tomadas en tres días distintos. Las líneas gruesas indican el genotipo fpvA (cada nueva mutación marca una bifurcación, que se sitúa de forma arbitraria en el dibujo), mientras que el genotipo mucA de los aislados se indica junto al símbolo correspondiente a cada muestra tomada, en cada uno de los tres días. Las 6 variantes detectadas, según su genotipo fpvA-mucA, se indican en rectángulos numerados, teniendo en cuenta el número de mutaciones sufridas. Los aislamientos mucoides se señalan con un asterisco (*). Las desviaciones en sensibilidad a antibióticos de algunos aislados respecto al clon colonizador se indican en fondo negro: AkI, CMI amikacina=16μgml−1; CipI: CMI ciprofloxacino=2μgml−1; CipR: CMI ciprofloxacino=4μgml−1.
La colonización por P. aeruginosa en los pacientes con FQ se caracteriza por el establecimiento de una cepa clonal dominante, una vez que se ha alcanzado una fase de colonización estable. En los pacientes con FQ estudiados y según los perfiles de RFLP, sólo se ha detectado en dos de ellos, FQ4 y FQ6, la presencia de una cepa diferente a la que coloniza crónicamente. En ambos casos se aisló en las primeras muestras del estudio y no se detectó nuevamente, siendo posteriormente sustituida por la cepa dominante. Este hecho ha sido también observado en las fases de colonización intermitente posterior a la primocolonización24. Otros 2 aislados, de FQ1 y FQ3 mostraron diferencias en el perfil de RFLP insuficientes para adjudicarlas a un clon diferente según los criterios de Tenover et al21 (fig. 1 y tabla 1).
El desarrollo de un fenotipo mucoide, debido a la hiperproducción de alginato, es característico de las poblaciones de P. aeruginosa que colonizan las vías aéreas en casos de FQ2,14, facilitando la formación de biofilm y marcando generalmente la fase irreversible de colonización crónica. Este fenotipo se debe a mutaciones que afectan al operón de biosíntesis de alginato, especialmente al gen mucA (en un 85% de los casos, aproximadamente25), que codifica un regulador negativo de la síntesis. En nuestra cohorte, al aislamiento de cepas mucoides osciló entre el 25,9% (FQ5) y el 70,8% (FQ2), exceptuando FQ6, en quien sólo el último aislado (7%) fue mucoide, lo que se corresponde probablemente con su corta edad y reciente colonización; en el caso de FQ5 la detección de aislados mucoides se inicia sólo a partir de los 13 años (2004), mientras que en los restantes pacientes se detectan desde el comienzo del estudio (tabla 1).
Del total de 56 aislados mucoides, en el 70% (39) se detectaron mutaciones capaces de truncar la proteína MucA. Cada una de estas 8 mutaciones han sido exclusivas de un clon, y sólo dos de ellas se han descrito anteriormente: la más común es la deleción de una G en el grupo de 5 que ocupa la posición 426 a 430 (Δ430G), conocida como mucA227,11,18,26; otra es la mutación sin sentido C424T26. La distribución por pacientes sugiere que cada una es resultado de un suceso único en la cepa dominante (aunque se conserve también la cepa silvestre, como se discute más adelante): ΔGCGGCGGTCTC187 en FQ2, ΔC453 en FQ3, ΔG430 en FQ4 y C424T en FQ5 (tabla 1). Al igual que en otros estudios, todas las mutaciones afectan al dominio periplásmico de la proteína MucA (aminoácidos 113-170) implicado en la interacción con MucB. Se cree que la pérdida de interacción con MucA permite la degradación de MucA por proteasas periplásmicas27–29. En el 30% restante de los aislados mucoides no se detectaron mutaciones sin sentido o con desfase del marco de lectura. Se ha descrito que la frecuencia de mutaciones responsables de fenotipo mucoide en la zona no secuenciada de mucA es muy baja (<0,3%)25, por lo que la explicación más probable para estos aislados es la presencia de mutaciones en los genes mucB o mucD26,30,31.
Otros 17 aislados no fueron mucoides a pesar de poseer también alguna de esas mutaciones (tabla 1). Se ha descrito que la pérdida del carácter mucoso se debe normalmente a supresiones extragénicas16. Curiosamente, tres aislados presentaron una supresión intragénica del codon de terminación prematura sin perder el fenotipo mucoide: ΔT180+Δ187-197 (0E67 de FQ2), G∴160+ΔC453 (5C01 de FQ3) y GCCTGCT∴202+ΔC453 (6H21 de FQ3).
El genotipado del receptor de pioverdina mostró predominancia del tipo II, sujeto a una notable variabilidad, y ausencia del tipo III. Otros estudios han detectado una proporción similar de los tres tipos en pacientes de FQ, mientras que en los aislamientos ambientales de P. aeruginosa predomina el tipo I23,32. La distribución de tipos y variantes es coherente con la de los diferentes clones; la diferencia de subtipo en la cepa D24 de FQ1 va acompañada de un cambio en el perfil de PFGE y podría reflejar un fenómeno de recombinación19. No se detectó ninguna mutación sin sentido, lo que es consistente con la importancia de la captación de hierro para P. aeruginosa en la colonización pulmonar33.
Los genotipos mucA/fpvA dominantes en los aislados de cada paciente muestran una discriminación similar al RFLP: I/2b12 en FQ1, II/2c en FQ4, II/2a1 en FQ5 y Ia/1 en FQ6 (tabla 1). Sin embargo, los pacientes FQ2 y FQ3 mostraron mayor coincidencia: el mismo genotipo mucA III, y los genotipos fpvA 2b1 en FQ2 y 2b ó 2b2 en FQ3, que difieren sólo en un SNP. Esta similitud puede deberse a que ambos pacientes son hermanos y pueden haber sido colonizados inicialmente por una cepa común, pero la divergencia en los perfiles de PFGE y el período de estudio realizado no permite asegurarlo.
En todos los pacientes el genotipado mucA/fpvA reveló la presencia de variantes de la cepa dominante, por adquisición de SNPs o mutaciones diversas. La alternancia de tales variantes es especialmente evidente en FQ2 (mucA III ó IIIa), FQ3 (fpvA 2b ó 2b2) y FQ5 (mucA C424T o silvestre y fpvA 2a1 o 2a11), que se aíslan simultáneamente de las mismas muestras. La presencia de alelos silvestres de mucA intercalados con alelos delecionados, como ocurre en FQ2 (Δ187-189, ausente en el aislados B46) o FQ4 (ΔG430 ausente en E63 y F80) se explica más fácilmente por la persistencia de la cepa original que por retromutación. Para explorar este fenómeno se proyectó el estudio de muestras tomadas del mismo paciente (FQ7) en un intervalo corto de tiempo (para reducir la posibilidad de aparición de nuevas variantes) pero con mayor frecuencia (2-3 esputos diarios). Los resultados demuestran la coexistencia de hasta 6 variantes de la misma cepa, que no se detectaron en todas las muestras. Cuatro de los aislados, pertenecientes a dos variantes genéticas, habían desarrollado, bien resistencia a ciprofloxacino, o niveles de sensibilidad intermedia a este antibacteriano o a amikacina (fig. 2). Se ha demostrado que la hipermutación es un factor esencial en el desarrollo de resistencia durante la colonización en pacientes con fibrosis quística u otras enfermedades obstructivas pulmonares, y que está probablemente ligada a la propia adaptación a la colonización crónica34. Estos resultados indican que en el pulmón del enfermo de fibrosis quística pueden coexistir varias subpoblaciones de la cepa colonizadora, posiblemente ocupando diferentes nichos. La dificultad para diferenciarlas, especialmente si se parte de una única muestra, puede tener consecuencias a la hora de determinar su sensibilidad a antibacterianos.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Este trabajo ha sido financiado por el proyecto PI050569 del Fondo de Investigaciones Sanitarias (FIS). A.V. fue becaria del Ministerio de Medio Ambiente, y A.F.-O. contratada (CM08/00166) mediante una ayuda del Instituto de Salud “Carlos III”, Ministerio de Ciencia e Innovación. Agradecemos a P. Gómez su valiosa asistencia técnica y a la Unidad de Genómica, Parque Científico de Madrid/UCM, por el trabajo de secuenciación.