Hafnia alvei es un bacilo gramnegativo facultativamente anaeróbico que constituye parte de la flora intestinal humana. Hasta hace poco, las cepas de H. alvei, que se identificaban por métodos convencionales, por miniaturización o por sistemas automáticos, podían confundirse fácilmente con miembros de los géneros Serratia, Escherichia, Citrobacter, Yokenella, Obesumbacterium o Salmonella, por lo que era necesario realizar técnicas moleculares para su identificación definitiva en la práctica clínica. Además, recientemente se ha introducido una nueva especie de Hafnia,H. paralvei, que sin duda incluye a muchas de las cepas previamente identificadas en la literatura como H. alvei. Aunque el aislamiento de H. alvei de muestras clínicas humanas sigue siendo poco frecuente, han aumentado los artículos científicos que evidencian un aumento de la resistencia a los antibióticos en esta especie y es probable que este organismo gane cada vez más importancia en el futuro. Además, aunque H. alvei comparte algunos mecanismos de virulencia con otros enteropatógenos gramnegativos, se sabe poco sobre los factores que contribuyen a su patogénesis en humanos. El presente artículo revisa los métodos de identificación actuales, la resistencia a los antimicrobianos y los factores de virulencia de esta bacteria.
Hafnia alvei is a Gram-negative facultatively anaerobic bacillus that constitutes part of the human gut flora. Until recently, H. alvei strains could be mistakenly identified by conventional methods, miniaturisation or automatic systems as members of the Serratia, Escherichia, Citrobacter, Yokenella, Obesumbacterium or Salmonella genera. Consequently, molecular techniques were required for their definitive identification in the clinical laboratory. In addition, a new Hafnia species, H. paralvei, has recently appeared, which undoubtedly includes many of the strains reported in the literature as H. alvei. Alrhough H. alvei isolation from human clinical specimens remains uncommon, the development of drug resistance due to this species is emerging and it is likely that this organism will gain increasing importance in the future. Moreover, although H. alvei shares some virulence mechanisms with other Gram-negative enteropathogens, little is known about the factors that contribute to its pathogenesis in humans. The present article reviews the current identification methods, antimicrobial resistance and virulence factors of this bacterium.
Hafnia alvei es una bacteria bacilar Gram negativa anaeróbica facultativa. Este microorganismo se distribuye ampliamente en la naturaleza, y pertenece a la microbiota comensal intestinal de muchos animales, en algunos de ellos se considera como patógeno oportunista; especialmente en aves de corral y en peces, en los que se han descrito brotes que han afectado a un gran número de individuos1,2. H. alvei tiene también cierto interés para la industria alimentaria, donde se la asocia con el deterioro de algunos productos alimentarios, y frecuentemente se aísla de productos lácteos y carnes envasadas al vacío3,4. En el hombre, esta bacteria es un habitante normal del tracto intestinal, aunque algunos investigadores también la consideran un comensal del tracto respiratorio.
2Patología en humanosEn humanos, esta especie se ha asociado con un amplio abanico de infecciones que incluyen bacteremia5–8, septicemia9, neumonía10,11, endoftalmitis12, infecciones de articulaciones y heridas13, infecciones respiratorias14,15, síndrome urémico hemolítico16 y meningitis17. En patologías extraintestinales, como peritonitis o abscesos, también se ha reconocido a H. alvei como agente causal18–20. Su relación con casos de trastornos gastrointestinales ha recibido creciente atención desde hace varias décadas21, aunque la literatura revela que esta bacteria se ha asociado con casos de diarrea y gastroenteritis desde 195722–25. Por desgracia, la taxonomía de este género —y en general la mayoría de técnicas bacteriológicas y métodos de identificación— no estaba muy definida en aquella época, y los primeros artículos científicos sobre la relación entre Hafnia y enfermedades intestinales prácticamente no se han citado por investigaciones más recientes. La polémica respecto a si se puede considerar causante o no de patologías intestinales graves, como diarrea aguda, comenzó realmente a principios de la década de 1990, con los trabajos realizados por investigadores del International Center for Diarrheal Disease Research in Bangladesh y los Centers for Disease Control and Prevention (CDC) estadounidenses. En estos trabajos se aisló primeramente una cepa identificada como H. alvei en un niño de 9 meses que padecía una diarrea acuosa26. Esta cepa en cuestión y otras aisladas en la misma zona, no producían toxinas típicas implicadas en trastornos gastrointestinales, pero sí inducían diarrea en modelos animales de infección (conejo). Además, tras observar el íleon y el colon de los conejos infectados, se detectó, por microscopía electrónica, un tipo de lesiones parecidas a las que realizan algunas cepas de Escherichia coli enteropatogénicas, que suelen ser agentes causales de diarrea en humanos. A este trabajo siguieron otros in vitro e in vivo de estos mismos investigadores, con distintos colaboradores internacionales, que intentaron clarificar el posible papel de H. alvei como causante de diarrea en humanos27,28. En uno de esos estudios se aislaron nuevas cepas de esta bacteria de una docena de turistas finlandeses que habían experimentado diarrea durante un viaje a Marruecos29. Inmediatamente, otros investigadores comenzaron a publicar trabajos en los que se relacionaba H. alvei con casos de trastornos gastrointestinales en humanos30–33. A finales de esa década se comprobó que la mayoría de las cepas de H. alvei estudiadas eran en realidad biotipos “raros” de Escherichia y que, aunque presentaban positividad por reacción en cadena de la polimerasa (PCR) para el gen eaeA (del inglés attaching and effacing) —codifica para una intimina que interacciona con los enterocitos intestinales para producir las lesiones típicas de E. coli enteropatogénicas—, no presentaban otras toxinas. Posteriormente, los investigadores interesados en H. alvei comenzaron a realizar la PCR para este gen, y mayoritariamente verificaron que cepas aisladas de pacientes con diarrea y cuya identificación se acreditaba como H. alvei, no lo presentaban34. Así que, por ejemplo, muchas de las cepas causantes de diarrea en Bangladesh pasaron a denominarse E. albertii.
Sin embargo, hay una mayoría de estudios que describen la presencia mayoritaria —o incluso única— de esta bacteria en heces de niños con diarrea, con edades comprendidas entre 0 y 12 años5,35,36, por lo que esta sí que podría ser el agente etiológico de algunas diarreas infantiles.
3Taxonomía e identificaciónLa taxonomía de H. alvei ha experimentado notables cambios desde que en 1954 Møeller introdujera el género Hafnia, sobre la base de los patrones de decarboxilación de aminoácidos por especies de la familia Enterobacteriaceae. En años posteriores, algunos investigadores propusieron incluir a H.alvei dentro del género Enterobacter, pero mediante estudios de hibridación de ADN se comprobó que H. alvei no estaba tan cerca de Enterobacter como se pensaba37. En la década de 1990, estaba claro que las cepas que se identificaban primeramente como H. alvei presentaban una heterogenicidad fenotípica y genotípica evidente, por lo que se planteaba la necesidad de abordar definitivamente técnicas moleculares para dilucidar su intrincada variabilidad. Así, mediante estudios de hibridación de ADN, se comprobó que H. alvei contenía en realidad varias genomoespecies distintas, que unos años después se identificaron por el grupo de Janda et al38. Estos, apoyándose además en la cada vez más habitual secuenciación del gen 16S del ARN ribosómico bacteriano, consiguieron separar claramente 2 grupos distintos de cepas, a los que denominaron grupos de hibridación 1 y 2, cuyas cepas de referencia fueron la ATCC 29926 y ATCC 29927, respectivamente. Más adelante, Janda y su grupo se unieron al grupo de microbiología de la Ghent University, dirigido por G. Huys, que poseían gran experiencia en taxonomía bacteriana. Juntos consiguieron separar definitivamente los 2 grupos de bacterias del género Hafnia, y describieron al grupo de hibridación 2 como H. paralvei39. Así, H. alvei y H. paralvei son actualmente las especies de este género que se aíslan en los laboratorios de microbiología.
Sin embargo, la correcta adscripción de las cepas aisladas a cada uno de los 2 géneros era complicada, sobre todo para los laboratorios clínicos, que no podían abordar laboriosos protocolos de electroforesis de campo pulsado o hibridación ADN-ADN y solo disponían de los clásicos métodos bioquímicos o de las tiras API. Los métodos de identificación automática, como el VITEK o el MicroScan (en sus diferentes versiones), tampoco separaban estas 2 especies, por lo que en muchos casos se llegaban a confundir cepas de H. alvei o H. paralvei con especies de los géneros Serratia, Escherichia, Citrobacter, Yokenella y Obesumbacterium40,41.
Más adelante se desarrolló una serie de tests bioquímicos que parecían ayudar a la correcta identificación y clasificación de ambas especies. Para ello se emplearon los tests de la b-glucosidasa, de la utilización del malonato, de la fermentación de la D-arabinosa, de la fermentación de salicina y de la hidrólisis de la esculina39,42. Estos 5 tests eran en un principio asequibles a la mayoría de laboratorios, pues algunos de ellos se utilizaban clásicamente en la identificación con pruebas bioquímicas convencionales. Algunos investigadores incluso utilizaron fagos específicos de H. alvei para su correcta identificación43. Así, H. alvei sería positiva para la b-glucosidasa y para la utilización del malonato, negativa para la fermentación de la D-arabinosa y variable para la fermentación de la salicina y para la hidrólisis de la esculina, mientras que H. paralvei sería negativa para todas las pruebas, menos para la fermentación de la D-arabinosa, en el que el resultado es variable.
Teniendo en cuenta que la nueva especie H. paralvei se describió en 2010 y que las técnicas para la diferenciación de ambas especies se han comenzado a incorporar muy lentamente por los laboratorios, cabe la duda razonable de que muchas de las cepas aisladas con anterioridad e identificadas como H. alvei, en realidad sean H. paralvei. De hecho, algunos investigadores han publicado recientemente trabajos donde se rectifica la identificación inicial de algunas cepas que, debido a la taxonomía de la época, se habían clasificado como H. alvei44. Por lo tanto, se sabe muy poco sobre la frecuencia de aislamiento de H. paralvei, y tampoco se sabe mucho sobre su virulencia o acerca de su resistencia antimicrobiana. Asumiendo que se han tratado siempre ambas especies como si fueran una sola, sería interesante recuperar cepas de estudios más antiguos de H. alvei y reidentificarlas según esas características genéticas y fenotípicas actuales.
Actualmente, H. alvei es una bacteria que crece perfectamente en medios bacteriológicos generales para el aislamiento de enterobacterias. En algunos casos, la bacteria supera ampliamente el número de unidades formadoras de colonias respecto a otros patógenos buscados habitualmente en estos medios, como Enterobacter, Klebsiella o E. coli.
Los laboratorios que se benefician de la utilización de sistemas MALDI-TOF se encontrarán con que las bases de datos no están totalmente implementadas para diferenciar entre H. alvei y H. paralvei, por lo que sería interesante optimizar dichas bases de datos. También lo sería optimizar un método de PCR para diferenciar ambas especies en la práctica clínica, y poder conocer así la frecuencia con la que aparecen tanto en coprocultivos como en infecciones intra o extraintestinales.
Por otro lado, gracias al abaratamiento de los costes de la secuenciación de genomas, ahora es más fácil clasificar correctamente ambas especies utilizando todo el contenido genómico. Actualmente, solo hay 4 genomas completos de H. alvei en la base de datos GeneBank. Estos corresponden a una cepa aislada de carne de pescado elaborada (cepa FB1) y a 3 cepas de origen humano: HUMV5920, PCM-1220 y CBA7135. Otro genoma completo procedente de una cepa de H. alvei (denominada bta3_1) aislada del tubo digestivo de una abeja se ha depositado recientemente en esta base de datos45. Además, la cepa CBA7124 de H. paralvei, aislada de heces en un paciente que presentaba enteritis, también se ha secuenciado totalmente y se ha anotado su genoma. Estos genomas completos —y una veintena más de cada especie secuenciados parcialmente— han ofrecido una visión global de su genoma central y de sus genomas accesorios27.
Debido a la secuenciación masiva de genomas bacterianos realizada en los últimos años, las especies H. alvei, H. paralvei y la recientemente descubierta H. psychrotolerans, actualmente se sitúan en una familia denominada Hafniaceae, dentro del orden Enterobacterales46.
4Factores de virulenciaAl observar los datos recogidos de los pocos genomas completos que hay depositados en las bases de datos, se puede constatar que, al igual que otras enterobacterias, H. alvei presenta genes para la comunicación bacteriana (quorum-sensing) basados en acilhomoserina lactona y moléculas de tipo AI-2 como moléculas inductoras, y también genes para la movilidad, quimiotaxis, formación de apéndices superficiales (fimbrias) y para formación de biocapas (biofilms). En H. paralvei, este sistema de comunicación con homoserina lactona y AI-2 también está presente en al menos la cepa de referencia ATCC 29927. La expresión de todos estos genotipos se ha comprobado in vitro. La conjunción de estos atributos (quorum-sensing, movilidad, fimbrias y biofilms) podría jugar un papel importante en fenómenos que relacionen la densidad de población bacteriana con los efectos patológicos o incluso con la permanencia de esta bacteria en instalaciones sanitarias47. H. alvei también presenta mecanismos de captación de hierro (sideróforos) que podrían participar en su patogénesis. También, aproximadamente el 50% de las cepas de H. alvei que se han enfrentado a suero humano son resistentes a él. Por desgracia, no se han ensayado mutantes de estos factores de virulencia en modelos in vitro o in vivo.
Como en la mayoría de enterobacterias, la expresión de algunos factores de virulencia está modulada por la temperatura. Así, H. alvei produce una biocapa más robusta y expresa flagelos laterales cuando se cultiva a temperatura ambiente48,49, por lo que la expresión de factores que promueven la formación de biofilm podría estar asociada con la supervivencia de esta especie en el ambiente hospitalario. Además, al contrario que con otros patógenos prioritarios, como por ejemplo Acinetobacter, Klebsiella o Enterobacter, tampoco se ha estudiado la resistencia de H. alvei a factores ambientales como desecación, falta de nutrientes, etc.
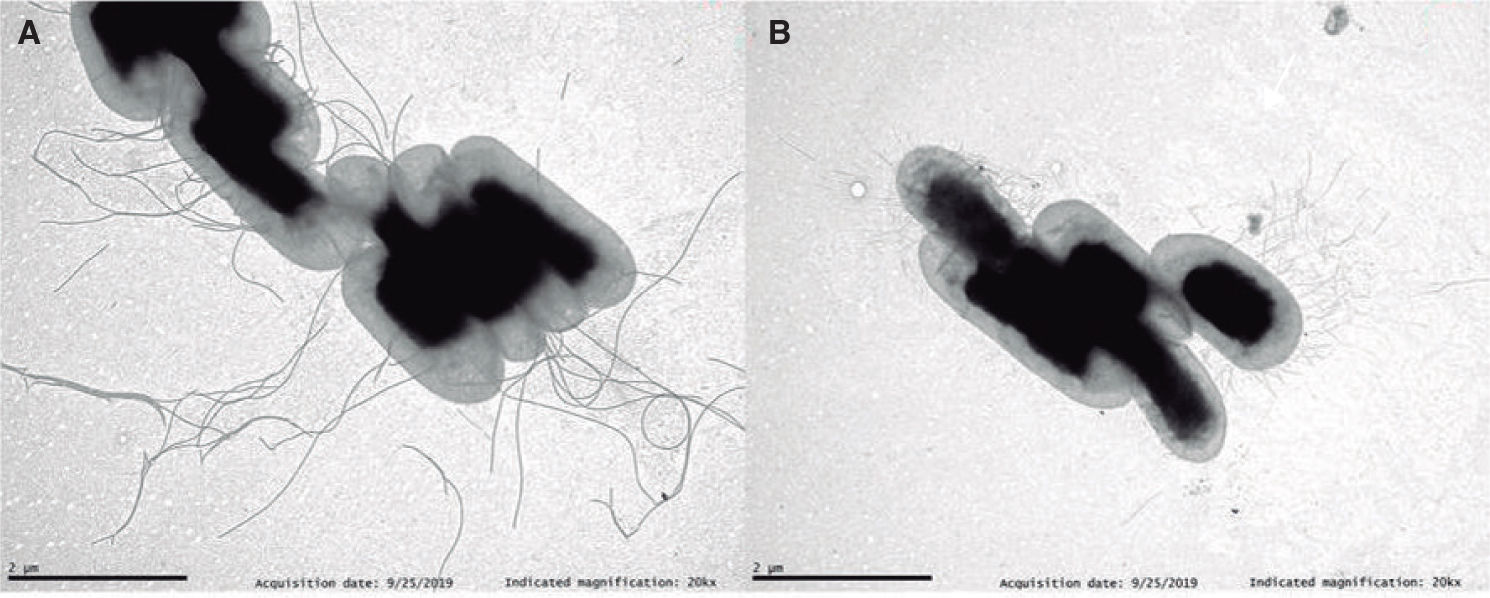
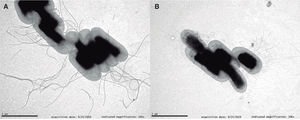
Siguiendo con el efecto de la temperatura sobre la expresión de factores de virulencia, las cepas de H. alvei cultivadas a 37°C no presentan flagelos, pero una parte de la población expresa fimbrias, que podrían estar implicadas en la patogénesis, mientras que otra parte no presenta apéndices superficiales. En la figura 1 se muestra una fotografía de microscopio electrónico de barrido de la morfología de una cepa de H. alvei cultivada a ambas temperaturas. La aparición en un mismo cultivo de células con o sin fimbrias podría deberse a la “variación de fase”, un sistema por el que la expresión de proteínas en las bacterias se activa (fase ON) o se inactiva (fase OFF) dependiendo de factores genéticos o epigenéticos. El fenómeno de variación de fase, también puede afectar en bacterias a la expresión de genes relacionados con el lipopolisacárido (LPS), la cápsula, la morfología y opacidad de las colonias y otras proteínas o genes reguladores. Sería interesante utilizar mutantes de H. alvei en fimbrias o flagelos para realizar ensayos de virulencia in vitro o in vivo.
Fotografía de microscopía electrónica de transmisión (TEM) de una cepa clínica de Hafnia alvei cultivada a 25°C (A) y a 37°C (B). La fotografía seha tomado en un microscopio de transmisión JEOL (JEM-1011) a 80kV, equipado con una cámara CCD Orius SC 1000 (GATAN), tras una tinción con ácido fosfotúngstico. Magnificación original, 320.000 aumentos.
La adherencia de H. alvei a células eucariotas in vitro no es muy elevada, y solo en ensayos largos de infección (más de 2h), algunas cepas tienen cierta capacidad de adherencia e invasión50. Además, los pocos estudios que se han realizado in vitro con células epiteliales humanas han utilizado un bajo número de aislados clínicos y se han realizado utilizando principalmente en células derivadas de tracto respiratorio.
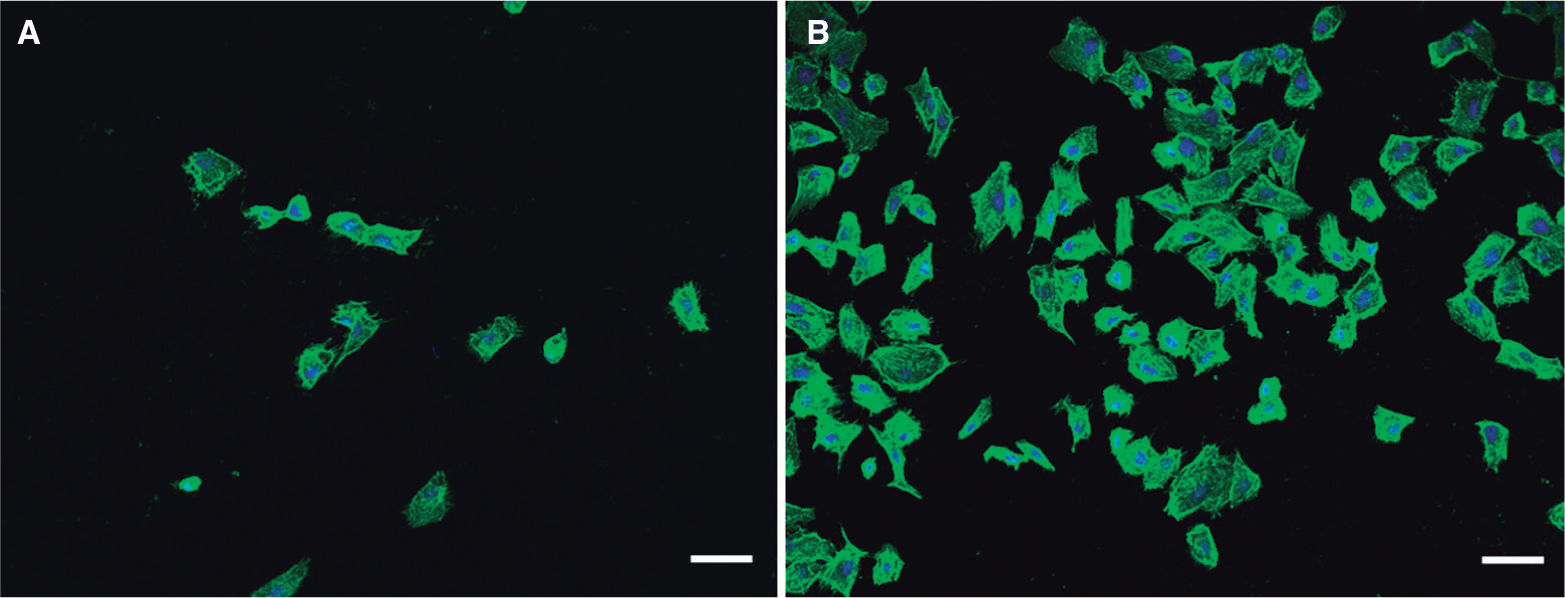
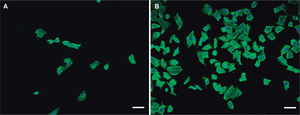
En el año 2006 se aisló una cepa de H. alvei de una niña de 11 años con diarrea y síndrome hemolítico urémico16; los productos extracelulares presentes en el sobrenadante de los cultivos de esta cepa presentaron efectos citopáticos cuando se añadieron a células Vero (células epiteliales del riñón de mono verde africano, Chlorocebus sp.), similares a los que producen las cepas de E. coli O157:H7. A partir de ese momento, el mismo grupo de investigación reunió una colección de cepas de H. alvei y H. paralvei para comprobar si este fenómeno era aislado o si, por el contrario, era común en esas especies. El resultado fue que un número mayor de cepas de H. alvei que de H. paralvei producían un efecto citopático42. Estos investigadores indicaron también que el número de cepas de H. alvei o H. paralvei con capacidad citotóxica en células Vero era del 70%. Sería interesante realizar estos ensayos utilizando células de colon humanas (hay incluso varias líneas celulares establecidas disponibles) para reproducir estos ensayos, ya que las células Vero (riñón de mono) no son representativas del epitelio intestinal humano. También se debe indicar que las enterobacterias producen grandes cantidades de LPS durante su crecimiento in vitro, y que la sensibilidad a toxinas de las células Vero es proporcional a la concentración y tipo de LPS y al tiempo de contacto con este. Un ejemplo del efecto citotóxico de cepas de H. alvei sobre células Vero puede verse en la figura 2. Nosotros no hemos encontrado esta toxicidad utilizando otras líneas celulares epiteliales humanas, ni tampoco utilizando células de sistema inmunitario primarias o establecidas50; pero el efecto citopático observado en células Vero indica que en los productos extracelulares de la bacteria podría haber una toxina lítica implicada en las patologías intestinales o extraintestinales achacadas a esta bacteria o que su LPS es especialmente tóxico en estas células.
Efecto citopático de una cepa clínica de Hafnia alvei sobre células Vero. Nótese la disrupción de la monocapa de células (A) comparada con un control de células sin infectar (B). El citoesqueleto celular está teñido (verde) con faloidina Atto-488 y los núcleos celulares (azul) con DAPI. Las preparaciones se han fotografiado con un microscopio confocal Nikon A1R y un objetivo 340 Plan-Fluor 1.3 NA. Barras de escala, 50μm.
Respecto a modelos animales de infección, hasta la fecha solo se ha utilizado el modelo Galleria mellonella en un ensayo con H. alvei51. En este trabajo se utilizó una cepa de H. alvei y otra de H. paralvei, y esta segunda presentó una dosis letal más baja (2,80×105 frente a 9,48×106). También se han utilizado peces (trucha y dorada), ratones y cobayas para los estudios de virulencia con Hafnia, pero en la mayoría de ensayos publicados no se contabilizó el inóculo infectivo de forma precisa, o las dosis letales fueron muy variables entre cepas21.
5Resistencia a los antibióticosNo hay muchos estudios en los que se haya analizado a la vez la sensibilidad a los antimicrobianos en un número grande de aislados clínicos. Cabe destacar el estudio de Laupland et al8, donde se analizaron cepas de H. alvei aisladas de 138 pacientes durante un período de 6 años, y el cultivo de orina fue la fuente principal de los aislamientos. Otros 2 estudios con gran número de cepas fueron los de Frutos et al52, con 111 aislados de coprocultivo en 2 años, Rodríguez-Guardado et al19, con 36 cepas, Fernández-Roblas et al53, con 32 cepas, y Lancelevee et al54, con 17 cepas. En la mitad de estudios publicados en los últimos 20 años, solo se realizó el análisis de la resistencia a antimicrobianos de una sola cepa de H. alvei, aislada en la gran mayoría de casos en monocultivo y a la que se consideró agente etiológico de la patología estudiada en esos trabajos. En muchos de los trabajos analizados se indica que H. alvei es resistente a ampicilina, cefalotina, cefoxitina, cefuroxima, penicilina, piperacilina y a las combinaciones amoxicilina-clavulánico y piperazilina-tazobactam, por lo que no se recomiendan estos agentes como de primera elección. En la gran mayoría de trabajos, H. alvei presentó sensibilidad a amikacina, cefoxatima, cefepime, ceftazidima, ceftriaxona, ciprofloxacino, gentamicina, imipenem, meropenem, quinolonas y a la combinación trimetoprim-sulfametoxazol. En otros estudios, las cepas de H. alvei analizadas presentaron variabilidad respecto a su sensibilidad a tetraciclina, cotrimoxazol, cefpodoxima, gentamicina y nitrofurantoina.
Es de destacar que la gran mayoría de cepas estudiadas en infecciones causadas por H. alvei procedían de la comunidad, y solo en contados casos se ha podido verificar una infección nosocomial por este patógeno. La revisión de los patrones de resistencia o susceptibilidad de cepas ambientales (p. ej., en cepas aisladas de animales o alimentos) queda fuera del objetivo de esta revisión.
Hasta 2017, los trabajos en los que se había determinado la sensibilidad de Hafnia a colistina eran escasos18,42,44,55. En el trabajo de Günthard y Pennekamp de 201618, 54 de las 72 cepas analizadas fueron resistentes a este antibiótico de último recurso. En 2017 se realizó un estudio con cepas aisladas de humanos y se estudió su sensibilidad a colistina mediante 3 métodos diferentes56. En conjunto, la tasa de resistencia a colistina fue del 96% (n=25 cepas). En otro estudio se aislaron 8 cepas de Hafnia a partir de individuos sanos y una a partir de un paciente recibido en atención primaria, que presentaron valores de MIC para colistina de 4 (n=5) u 8 (n=4)57. Estos datos sobre resistencia a colistina parecen colocar a este género cerca de otros géneros de enterobacterias que son resistentes a colistina de forma natural, como Proteus, Providencia, Morganella y Serratia.
H. alvei posee en su cromosoma una b-lactamasa inducible (clase C de Amber) con actividad cefalosporinasa, que generalmente confiere resistencia a aminopenicilinas y cefalosporinas de primera generación, pero no a cefalosporinas de tercera y cuarta generaciones. Sin embargo, en 2010 se aisló una cepa de Hafnia resistente a carbapenémicos (ertapenem, meropenem e imipenem)58, lo que es de especial importancia porque los carbapenémicos son prácticamente la última línea de defensa contra infecciones causadas por enterobacterias resistentes a cefalosporinas de tercera y cuarta generaciones. Además, se ha evidenciado una relación entre varias b-lactamasas AmpC cromosómicas de H. alvei y de otras enterobacterias, con cefalosporinasas plasmídicas presentes en especies homólogas o heterólogas, como K. pneumoniae, por lo que estas especies podrían ser finalmente reservorios de cefalosporinasas codificadas en plásmidos, con el consiguiente peligro de diseminación de sus genes de resistencia59,60.
6ConclusiónEsta revisión está basada en 3 aspectos de H. alvei, su taxonomía, su patogenicidad y su resistencia o sensibilidad a los antimicrobianos. En cuanto a la taxonomía, parece claro que algunas de las cepas que se venían identificando en los laboratorios de los hospitales como H. alvei eran en realidad H. paralvei. Todos los sistemas de identificación rápidos aún no están optimizados para separar estas 2 especies, aunque en la práctica clínica tampoco es que esto parezca muy urgente. Sería interesante separar H. alvei de H. paralvei (p. ej., con alguna prueba rápida como la PCR), para conocer un poco más sobre su virulencia y su patogenicidad. Respecto a su virulencia, aún se está lejos de comprender cómo este habitante de la microbiota normal humana puede llegar a causar patologías tan diferentes. Faltan ensayos con mutantes, buenos modelos celulares y también estandarizar algún modelo animal que pueda arrojar pistas concretas sobre su virulencia. Respecto a la resistencia a los antimicrobianos, H. alvei es en general un patógeno fácil de tratar con antibióticos, pero varias publicaciones nos han alertado ya sobre el aumento de las resistencias de los aislados clínicos y sobre la resistencia intrínseca de esta bacteria a colistina. La reciente aparición de algunas cepas resistentes a carbapenémicos debe mantenernos alerta sobre este patógeno oportunista.
Conflicto de interesesEl autor declara no tener ningún conflicto de intereses.