Un ejemplo de ingeniería evolutiva en bacterias patógenas es la emergencia en Salmonella enterica de plásmidos híbridos que han surgido por asociación de determinantes de resistencia (R) a antimicrobianos con plásmidos de virulencia (V) específicos de los serotipos typhimurium y choleraesuis. Los plásmidos VR poseen en común el operón spv (Salmonella plasmid virulence, que interviene en la infección sistémica) aunque difieren en la presencia de otros determinantes V y en el perfil de genes R. Los del serotipo typhimurium se han encontrado en Europa, y presentan regiones R con diferente grado de complejidad, pudiendo incluir integrones de clase 1 y distintos transposones. Los del serotipo choleraesuis proceden de cepas aisladas en Taiwán y sólo confieren resistencia a ampicilina y sulfonamidas. Ambos serotipos son zoonóticos y la formación de plásmidos VR puede aportar una ventaja selectiva en determinadas circunstancias, originando cepas más difíciles de tratar y con mayor potencial epidémico.
An example of evolutive engineering in bacterial pathogens is the emergence of hybrid virulence-resistance (VR) plasmids in Salmonella enterica, resulting from an association between antimicrobial resistance determinants and specific virulence plasmids of the S. typhimurium and S. choleraesuis serotypes. VR plasmids all possess the spv (Salmonella plasmid virulence) operon, which is involved in systemic infection; however, they differ in the presence of other virulence determinants and in the resistance gene profile. VR plasmids of S. typhimurium have been found in Europe, and show resistance regions with different levels of complexity that can include class 1 integrons and various transposons. VR plasmids of S. choleraesuis, detected in strains isolated in Taiwan, only confer resistance to ampicillin and sulfonamides. Both serotypes are zoonotic and the presence of hybrid VR plasmids may confer an adaptive advantage under certain conditions, resulting in bacterial strains that are more difficult to treat and have a higher epidemic potential.
La evolución de las bacterias patógenas en cuanto a la adquisición de determinantes de virulencia (V) y/o de resistencia (R) a agentes antimicrobianos se debe, fundamentalmente, a la incorporación, a menudo secuencial, de piezas de ADN, cuya combinación origina nuevos elementos genéticos, que quedan inmediatamente sometidos a la selección natural en un ambiente cambiante1. Las piezas que intervienen en este proceso de ingeniería evolutiva son de 3 tipos: operativas (entre las que se incluyen los genes que codifican y regulan las funciones V y R), translocativas (como secuencias de inserción, integrasas, transposasas, resolvasas, etc.) y dispersivas (entre las que se incluyen islas genómicas, bacteriófagos, plásmidos, transposones e integrones-casetes génicas)1,2. Al día de hoy, múltiples evidencias apoyan la idea de que una bacteria saprofita o comensal puede transformarse en patógena sólo por recibir, mediante transferencia horizontal, un determinado elemento genético2–4. Como ejemplo, se podría tomar una cepa ancestral de Escherichia coli, comensal habitual en el intestino de los animales homeotermos, que al adquirir la isla de patogenicidad SPI1 (Salmonella pathogenicity island) daría lugar a una cepa enteroinvasiva y, por tanto, patógena, conduciendo a la separación del género Salmonella. SPI1 contiene 2 grupos de genes (inv-spa y prg-org) que codifican un sistema se secreción de tipo III (SST3), mediante el cual la bacteria secreta e inyecta diversas proteínas efectoras (o de patogenicidad) en el citosol de la célula hospedadora. Parte de estas proteínas fuerzan la penetración de Salmonella en células no fagocíticas gracias a la modificación del citoesqueleto de actina. Un segundo grupo de proteínas efectoras será responsable de la enteropatogenicidad, causando inflamación del epitelio intestinal y síntomas diarreicos5. En otros momentos del largo proceso evolutivo hacia las actuales representantes de Salmonella, se fueron ganando nuevos determinantes V, cuyos productos permiten a la bacteria atravesar barreras y manipular las células del hospedador en sitios específicos durante el curso de la infección. Por ejemplo, SPI2 codifica un segundo SST3 requerido para la supervivencia de la bacteria, tanto en células epiteliales como en macrófagos4,6. Hace años se postuló que un 4% (unos 200 genes) del genoma de la cepa tipo de Salmonella (S. enterica serotipo typhimurium LT2) estaba involucrado en la patogenicidad para el ratón7, hecho confirmado por la posterior secuenciación del genoma de esta cepa8. En las salmonelas actuales, la mayor parte de los determinantes V están localizados en el cromosoma formando islas, islotes, operones o genes sueltos, pero también se conoce la presencia de éstos en plásmidos específicos de algunos serotipos no tifoideos de S. enterica8–11. Estos serotipos, transmitidos fundamentalmente por alimentos, son responsables de un gran número de enteritis (salmonelosis) en el ámbito mundial, y pueden originar infecciones sistémicas en personas inmunodeprimidas12–15.
Plásmidos de virulencia en SalmonellaLa existencia de plásmidos V en S. enterica (pS-V) se puso por primera vez de manifiesto en el serotipo typhimurium (pSLT) en el año 198216, y posteriormente se encontró en un número limitado de otros serotipos, todos pertenecientes a la subespecie 1. Algunos de ellos son particularmente frecuentes en infecciones humanas, como enteritidis (pSEV; 60kb), choleraesuis (pSCV; 50kb), dublin (pSDV; 80kb), además de typhimurium (pSLT, pSTV; 90kb)10,11. Los plásmidos V poseen una región de 7,8kb, altamente conservada, que contiene los genes spv (Salmonella plasmid virulence) necesarios durante la fase sistémica de la enfermedad en hospedadores específicos (experimentales/naturales: typhimurium en ratón/ganado vacuno, enteritidis en ratón/pollo, choleraesuis en cerdo y dublin en ganado vacuno)9,10,17. El locus spv está formado por 5 genes designados spvRABCD,8,9,11. El gen spvR codifica un regulador positivo esencial para la expresión de los otros genes spv. El producto de spvB es una ADP ribosil transferasa que actúa sobre la actina, bloqueando la conversión de G-actina en F-actina y provocando una desestabilización del citoesqueleto de células eucarióticas18. En el ratón, mutaciones en los genes spv no afectan a la colonización inicial del intestino, pero sí a la infección sistémica, ya que estos genes son necesarios para la supervivencia y multiplicación de la bacteria en el interior de los macrófagos17,18.
Dependiendo del serotipo, los plásmidos V pueden contener otros genes asociados a virulencia (fig. 1) como son: a) El operón pefBACDI (plasmid-encoded fimbriae), implicado en la síntesis de un tipo de fimbrias que intervienen en la adherencia de la bacteria a las células epiteliales del intestino delgado de ratón10,19. El operón pef está ausente en pSDV, el plásmido del serotipo dublin que, en cambio, codifica un tipo de fimbrias relacionadas con las K88 de E. coli10; b) los genes rsk y rck podrían estar implicados en la resistencia de Salmonella a la acción bacteriolítica del suero. El primero se encuentra en los plásmidos V de typhimurium, enteritidis y choleraesuis, y el segundo sólo en los de typhimurium y enteritidis. La resistencia al suero mediada por el producto del gen rck está asociada con la inhibición de la polimerización del componente C9 del sistema del complemento, impidiendo la formación de complejos de ataque de membrana y, por tanto, la disrupción de la bacteria10,20; c) el locus spf, presente en 3 plásmidos V (pSTV, pSEV y pSCV) está implicado en la supresión de la activación de la respuesta inmune del hospedador, y en el caso de pSTV está asociado con la producción de IL-12p40 por macrófagos del ratón11,21; d) otros genes V son mig-5, tlpA y srgA, que codifican, respectivamente, una anhidrasa carbónica inducible en macrófagos, una proteína que actúa como termosensor reprimiendo su propia expresión cuando hay un aumento de temperatura y una posible disulfuro oxidorreductasa, esencial para la biogénesis de fimbrias de codificación plasmídica10,22; e) otros loci codifican funciones de replicación y transferencia por conjugación. Sin embargo, mientras pSLT es un plásmido autotransferible, al menos bajo determinadas condiciones10,23, que posee el origen de transferencia del factor F (oriT) y una región tra compleja, los otros 3 plásmidos (pSEV, pSDV y pSCV) han perdido esta capacidad, por presentar diferentes deleciones en la región tra10,23; f) en los plásmidos V se han detectado, además, un número de loci de función desconocida8,9.
Representación esquemática de los mapas genéticos de 4 plásmidos V de Salmonella. Los plásmidos pSLT/pSTV, pSEV, pSCV y pSDV (específicos de serotipos typhimurium, enteritidis, choleraesuis y dublin, respectivamente) se presentan alineados respecto a pSLT. Deleción determinada: ----; deleción no determinada:
En cuanto al origen de los plásmidos V, se piensa que en algún momento del proceso evolutivo pudo tener lugar la integración del factor F (en el caso de pSTV, pSEV y pSCV) o de un plásmido relacionado (en el caso de pSDV, que pertenece a un linaje diferente), en el cromosoma de Salmonella, próximo al operón spv. Posteriormente, el factor F integrado se escindiría del cromosoma arrastrando al locus spv y generando los ancestros de los plásmidos V actualmente presentes en Salmonella11. Esta hipótesis viene apoyada por la presencia de dicho operón en el cromosoma de aislamientos pertenecientes a las subespecies II, IIIa, IV y VII de S. enterica. Sin embargo, la detección de genes relacionados con transposasas flaqueando la región spv, sugiere que esta región pudo haber sido adquirida mediante transposición11.
Plásmidos híbridos derivados de pSLT y portadores del integrón InH detectados en España en el serotipo typhimuriumUna nueva etapa en la ingeniería evolutiva de Salmonella ha sido la emergencia, aparentemente reciente, de plásmidos híbridos con determinantes V y R. Ya se ha indicado que en la mayor parte de los casos, la resistencia a antimicrobianos en las bacterias es consecuencia de la adquisición de nueva información genética portada por algún tipo de elemento móvil, y los transposones y las casetes génicas localizadas en integrones son entidades clave en la formación y evolución de los plásmidos1,2,10,24,25. Frecuentemente, los integrones forman parte de transposones y éstos, a su vez, suelen estar localizados en plásmidos, a menudo conjugativos. Estos plásmidos pueden ser transferidos a otras bacterias, y también insertarse en el cromosoma y/o en otros plásmidos presentes en el nuevo hospedador o en el inicial. Un ejemplo relevante lo constituyen los integrones portadores de la casete génica aadA1, sola o en combinación con otras casetes de adquisición posterior, que forman parte de transposones del tipo Tn21, que pueden estar localizados en diferentes plásmidos o en el cromosoma, unas veces solos y otras en combinación con transposones y/o integrones adicionales, formando agrupaciones con diferente grado de complejidad estructural24,25.
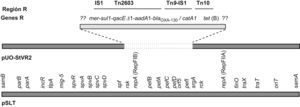
En Salmonella se han detectado plásmidos VR en los serotipos typhimurium26–30 y choleraesuis11,31. Uno de los plásmidos híbridos de typhimurium, designado pUO-StVR2, fue descubierto en nuestro laboratorio en cepas clínicas de este serotipo, aisladas en Asturias entre 1993 y 199928. Estas cepas compartían el fenotipo pentarresistente (ACSSuT, resistencia a ampicilina, cloranfenicol, estreptomicina/espectinomicina, sulfonamidas y tetraciclina) de un grupo pandémico de typhimurium, asociado preferentemente al fagotipo DT104, que ha sido objeto de numerosas revisiones bibliográficas32–34. Ahora bien, cabe destacar que en el tipo pandémico, los genes responsables de la resistencia son blaPSE−1, floR, aadA2, sul1 y tet(G), respectivamente, y que estos genes se localizan en una isla cromosómica de 43kb, denominada SGI1. Esta isla contiene un integrón de clase 1 complejo, con 2 sitios attI1, el primero ocupado por la casete aadA2 y el segundo por el gen blaPSE−134–38. Por el contrario, el fenotipo ACSSuT conferido por pUO-StVR2 se debe a los genes blaOXA−1 (designado también blaOXA−3039), catA1, aadA1, sul1 y tet(B)28,30,38. El plásmido pUO-StVR2 tiene un tamaño de aproximadamente 130kb (inicialmente estimado en 140kb), es conjugativo, pertenece al grupo de incompatibilidad IncFII y porta la mayor parte de los genes característicos de pSTV (locus spv, rck, traT, traX, oriT, samA, samB, repA [RepFIIA], parA, parB, srgA), siendo defectivo para la región que abarca el operón pef y en el gen rsk28,30,38. Se piensa que pUO-StVR2 podría proceder de pSTV tras haber ganado (en un solo paso o de forma secuencial) una región R compleja, de unas 45kb (fig. 2), que incluye un integrón de clase 1, designado InH38, con una región variable de unas 2.000 pb que contiene las casetes génicas blaOXA−1-aadA1, y una región 3’ constante con los genes qacEΔ1-sul1. En la región R de pUO-StVR2 se localizan, además, 3 transposones: a) Tn2603, relacionado con Tn21 (perteneciente a la familia Tn3) y portador del integrón InH y del operón mer que codifica resistencia a mercurio40,41; b) Tn9, que aporta el gen catA1 y se encuentra ligado a Tn2603, y c) Tn10 donde se localiza el gen tet(B)25. La secuencia de nucleótidos de la región R, que se está determinando actualmente, aportará información acerca de su origen y los mecanismos de ensamblaje de las distintas piezas que la constituyen (genes R, integrón, secuencias de inserción y transposones, así como de su sitio de inserción en el plásmido pSTV).
Mapas de los plásmidos pSLT y pUOStVR2. Aparentemente pUOStVR2 (ca. 125kb) es un derivado de pSLT (94kb), en el que se ha insertado una compleja región R de unas 40kb, provocando la deleción de una región que afecta al operón pef y al gen rsk. Datos tomados de las referencias 8, 9, 61 y de nuestra propia investigación.
Estudios en curso, realizados en colaboración con el Centro Nacional de Microbiología (CNM) han revelado la existencia de variantes de pUO-StVR2, designadas pUO-StVR4 a VR942, presentes en cepas multirresistentes de typhimurium recogidas en diferentes laboratorios españoles. Algunos de estos plásmidos confieren a sus hospedadores el mismo fenotipo R y genotipo VR que pUO-StVR2, pero se diferencian en cuanto a tamaño y/o número de sitios de corte con distintas endonucleasas (XbaI, SphI, HindIII, EcoRI, etc.). Éste es el caso de los plásmidos pUO-StVR4 y pUO-StVR5. Por otro lado, pUO-StVR7 porta un segundo integrón en cuya región variable (aproximadamente, 1.000pb) se identificó la casete génica aadA22, que difiere de aadA1 en 21 nucleótidos. La ganancia de dicho integrón afecta al genotipo R, pero no al fenotipo. Sin embargo, en el caso de otros plásmidos, la pérdida o adquisición de determinantes R se traduce en una alteración del fenotipo. Así, pUO-StVR6 y pUO-StVR7 no confieren resistencia a cloranfenicol y tetraciclina, debido probablemente a una deleción que afecta a los genes catA1 y tet(B) y, por tanto, a los transposones Tn9 y Tn10; mientras que pUO-StVR8 y pUO-StVR9 han ganado un determinante de resistencia a trimetroprim (aún no identificado), ampliando así el espectro de resistencia.
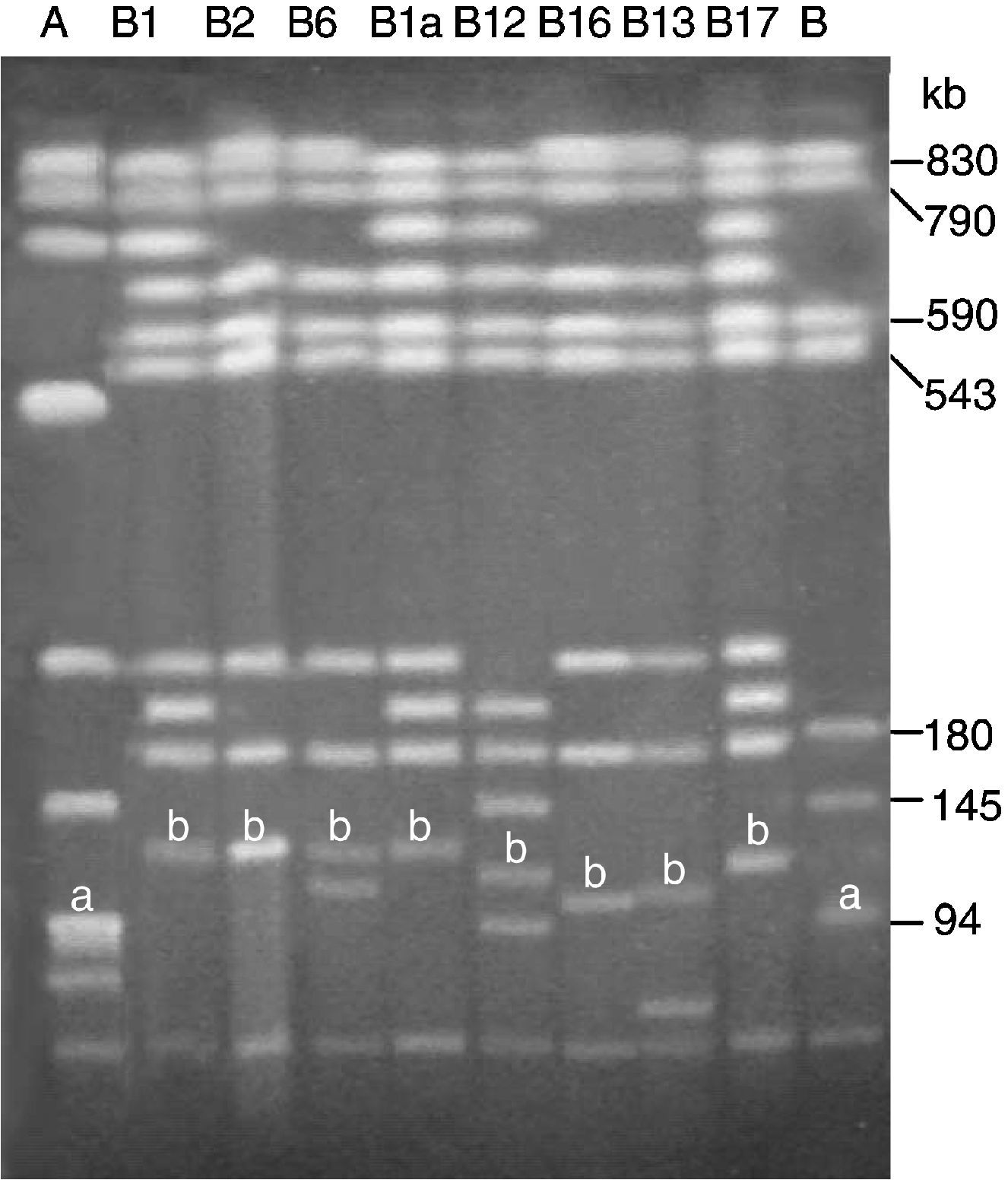
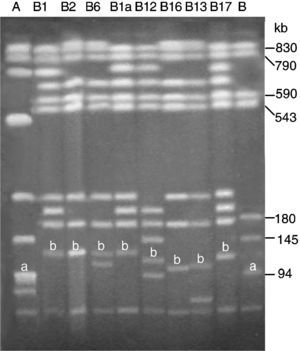
Las primeras cepas de typhimurium portadoras de pUO-StVR2 se asignaron a un mismo grupo genómico por presentar perfiles de genes R, genes V, integrones y macrorrestricción genómica (XbaI-PFGE) idénticos y claramente diferentes de los mostrados por otras cepas multirresistentes del mismo serotipo28. Sin embargo, la fagotipificación de estas cepas reveló una cierta heterogeneidad, ya que fueron no fagotipables, de patrón no reconocido, o pertenecían a fagotipos bien definidos: DT104 o DT120. En estudios con aislamientos posteriores recogidos en Asturias (2001–2002)30 y en otras regiones españolas (2002–2004)42 (datos no publicados) se han encontrado éstos y otros fagotipos (DT104b, DT208, DT41, DT27, DT193, DT160 y DTU302), y además se ha revelado que, aunque la mayoría de los aislamientos generaban perfiles de macrorrestricción genómica con XbaI y BlnI idénticos a los aislamientos más antiguos, algunos originaron perfiles diferentes (fig. 3). También es reseñable la detección, en baja frecuencia, de aislamientos portadores de pUO-StVR2 con fenotipo/genotipo R extendido, no atribuible a la presencia del plásmido híbrido. Cabe destacar la detección de genes de resistencia a trimetoprim (dfrA12, aportado por un plásmido de resistencia compatible con pUO-StVR2, y dfrA1, presente en el cromosoma y un gen sin identificar en el plásmido híbrido)30,42, gentamicina (gen de localización plasmídica, aún sin identificar)42, y ácido nalidíxico (debido a una mutación puntual que cambia el aminoácido Asp87 del gen gyrA por Asn [GAC a AAC])42 (datos no publicados). Además, se identificaron aislamientos con el mismo fenotipo R pero distinto genotipo R, que incluye los genes strA/B-sul2 (resistencia a estreptomicina-sulfadiacina) localizados en plásmidos de pequeño tamaño (<15kb). Estos datos apoyan el mantenimiento de los aislamientos característicos del grupo a lo largo de la última década en España, junto con la aparición de nuevas variantes, lo que supone una evolución intragrupo, además de la ya constatada evolución plasmídica30,42.
Análisis de macrorrestricción genómica mediante PFGE utilizando la endonucleasa BlnI de aislamientos de Salmonella typhimurium portadores de diferentes plásmidos del grupo pUO-StVR. A y B, perfiles mostrados por aislamientos de S. typhimurium fuera de grupo: A, LSP 14/92 (representativo del clon pentarresistente DT104) y B, cepa tipo LT2 (pSLT). B1, B2, B6, B1a, B12, B16, B13 y B17, perfiles generados por LSP 31/93 (pUO-StVR2/ca. 130kb), LSP 137/01 (pUO-StVR2/ca. 130kb), LSP 174/01 (pUO-StVR2/ca. 125kb+pUO-StR12/ca. 110kb), CNM 42/04 (pUO-StVR4/ca. 135kb), CNM 173/02 (pUO-StVR5/ca. 115kb), CNM 171/02 (pUO-StVR6/ca. 105kb), CNM 179/02 (pUO-StVR7/ca. 105kb), CNM 79/04 (pUO-StVR8/ca. 120kb), respectivamente. Este último perfil también fue generado por CNM 181/04 (pUO-StVR9/ca.120kb). a: plásmido pSLT de 94kb específico del serotipo typhimurium; b: fragmentos correspondientes a plásmidos pUO-StVR identificados mediante hibridación utilizando como sonda el plásmido pUO-StVR2.
En cuanto a la diseminación o dispersión geográfica, los plásmidos pUO-StVR se han encontrado hasta la fecha en aislamientos clínicos procedentes de 21 ciudades españolas, en algún caso asociados a brotes alimentarios de tipo familiar o comunitario30,42,43. Por ello, al menos desde 2002, el grupo typhimurium pUO-StVR debe considerarse endémico en España42. Por otro lado, tanto pUO-StVR2 como pUO-StVR9 se han encontrado en aislamientos procedentes de cerdos enfermos, y pUO-StVR2 en aislamientos de productos cárnicos destinados a consumo humano42,43 (datos no publicados), lo cual indica la existencia de un reservorio animal.
De momento, las cepas de Salmonella con plásmidos del tipo pUO StVR2 sólo se han detectado y caracterizado en España. Este hecho podría ser interpretado de 2 formas: que estas cepas estén limitadas a nuestra área geográfica o, más probable, que no se han buscado con la metodología apropiada en otros países. En cualquier caso, presuponemos que su diseminación será cuestión de tiempo y en ella jugarán (o están ya jugando) un papel importante tanto los movimientos poblacionales (incluido el turismo), como de mercancías. A favor de este supuesto, tenemos como antecedentes cercanos la rápida diseminación espacial de 2 tipos pandémicos y prevalentes: el ya comentado typhimurium DT104 y enteritidis (fagotipos PT4 y PT1) que tiene como principal reservorio las gallináceas y como vehículo de transmisión los alimentos derivados, incluidos los huevos44–48. Además, datos en relación con la epidemiología de integrones de clase 1 en Salmonella, revelaron la presencia de integrones con la misma región variable que InH en aislamientos de typhimurium analizados en otros laboratorios de España49–51 y en diferentes países del sur de Europa: de origen clínico en Albania52,53 y en Francia54, y tanto clínicos como de productos de cerdo en Portugal55. Al menos una parte de los aislamientos de España51, Francia54 y Portugal55 generaba perfiles XbaI muy similares a los encontrados por nosotros28,30,42. Integrones de tipo InH se han detectado también en Noruega, en 5 aislamientos clínicos del serotipo typhimurium fagotipo DT120, y es destacable que las encuestas epidemiológicas realizadas a los pacientes indicaron que 4 de ellos podrían haber sido adquiridos en España durante los años 1996–199956. Sin embargo, en ninguno de estos casos se investigó la posible relación del integrón con plásmidos de virulencia del tipo pUO-StVR. Para ello bastaría con visualizar el ADN plasmídico mediante un método apropiado (p. ej., lisis alcalina, S1-PFGE, o macrorrestricción-PFGE utilizando una endonucleasa con un único sitio de reconocimiento en el plásmido)28,30,42, e hibridarlo con sondas específicas para los genes bla0XA−1, utilizado como marcador de resistencia y del integrón InH, y spvC, marcador de virulencia (fig. 3).
Otros plásmidos híbridos de virulencia/resistencia en SalmonellaProcesos de transposición y/o recombinación entre pSTV y diferentes plásmidos R, han conllevado a la emergencia de otros plásmidos híbridos (portadores o no de integrones tipo InH) en la Europa mediterránea. En Italia, en 1997, se encontró una cepa clínica de typhimurium que contenía un plásmido VR de 110kb (denominado t-ST4), aparentemente no conjugativo pero transferible a E. coli por transformación, y perteneciente al grupo de incompatibilidad IncFII29. Este plásmido poseía genes característicos de pSLT (spvC, rck, pefA y traT), portaba un integrón con la misma región variable que InH (designado In-t8), junto a un segundo integrón (In-t7) con la casete aadB (resistencia a gentamicina y kanamicina), respectivamente asociados a transposones del tipo Tn21 y Tn1696, así como otros 3 genes R: catA1, dfrA23 y sul1, éste último en la región 3′ constante de los integrones. En cuanto a cepas de typhimurium aisladas en Francia, aunque se estudiaron en menor profundidad, se pudo constatar que expresaban el fenotipo ACSSuT, en el cual la resistencia a ampicilina se debía a los genes blaTEM−1 o blaPSE−1, localizados en plásmidos pertenecientes a los grupos de incompatibilidad IncFIC/P/Q, que hibridaban con una sonda spvCD26.
Sin embargo, el caso más preocupante y bajo vigilancia epidemiológica especial, fue descrito en 1997 por el CNM. Se trata de una variante monofásica del serotipo typhimurium, carente del gen fljB, con fórmula antigénica [4,5,12:i:-], multirresistente (fenotipo ACGSSuTp±T), y adscrita al fagotipo U30257,58. Esta variante, considerada por el CNM como serotipo para el seguimiento epidemiológico, había sido aislada esporádicamente entre 1993 y 1996, pasando posteriormente a ocupar la cuarta posición en frecuencia en nuestro país, por detrás de enteritidis, typhimurium y Hadar (http://cne.isciii.es/htdocs/bes/bes2005.htm). La comparación de esta variante con la cepa tipo typhimurium LT2, realizada mediante microarrays, demostró que la primera carecía del operón fljAB y de otras regiones del genoma, como parte del regulón responsable de la asimilación anaeróbica de la alantoína, varios profagos y el gen iroB59. Ante la alerta de la emergencia de este clon, con resistencia extendida a gentamicina y trimetoprim respecto al fenotipo ACSSuT prevalente, nuestro laboratorio optó por participar en su estudio encontrando que los aislamientos [4,5,12:i:-] recogidos en Asturias entre 1997 y 2000, analizados mediante macrorrestricción genómica con XbaI, generaban perfiles de bandas con una similitud >90% y, en su mayor parte, poseían un plásmido VR de aproximadamente 140kb (pUO-StVR3), no autotransferible a E. coli, perteneciente al grupo de incompatibilidad IncN, portador de una región R muy diferente de la encontrada en pUO-StVR2 (que incluye blaTEM−1-cmlA1-aac[3]-IV-aadA2-sul1-dfrA12±tet[A]; resistencia a ampicilina, cloranfenicol, gentamicina, estreptomicina, sulfonamidas, trimetoprim,±tetraciclina, respectivamente), y de un integrón de clase I (InI) con los genes qacEΔ1-sul1 en la región 3′ constante, y las casetes dfrA12-aadA2 en la región variable (de unas 1900pb). Además, contiene el locus spv pero no el operón pef ni los genes rck y rsk27,38 (datos no publicados). pUO-StVR3 deriva probablemente de un plásmido R que habría adquirido, al menos, la región spv de pSLT. El posible antecesor pudo haber sido pUO-StR4, que también se ha encontrado en aislamientos [4,5,12:i:-], que carece de genes de virulencia, pero comparte con pUO-StVR3 el integrón, el perfil de resistencia y el grupo de incompatibilidad. A día de hoy, el CNM ha identificado aislamientos [4,5,12:i:-] con diferentes perfiles R y plásmidos VR de tamaño entre 100–200kb (datos no publicados).
Es altamente interesante resaltar que en Taiwan, y presumiblemente en otras zonas asiáticas, ha tenido lugar un proceso paralelo de «ingeniería evolutiva» que ha conducido a la emergencia de plásmidos VR muy diferentes a los registrados en el serotipo typhimurium en países europeos. Esos plásmidos se han seleccionado en el serotipo choleraesuis, endémico en la zona, altamente invasivo, que generalmente requiere terapia antimicrobiana. Por ello, la resistencia a antimicrobianos puede constituir un serio problema11,31,60. Afortunadamente, la incidencia de choleraesuis en España y en otros países europeos es nula o muy baja (http://cne.isciii.es/htdocs/bes/bes2005.htm). Las cepas de choleraesuis analizadas31 habían sido recogidas de muestras clínicas en los años 1996 y 1997, albergaban plásmidos de gran tamaño (136 y 140kb) que portaban el gen spvC (utilizado como indicador del plásmidos de virulencia de Salmonella61) junto a los genes de resistencia blaTEM−1 y sul1. Como origen, los autores postulan la posible cointegración, mediante recombinación, de pSCV con plásmidos R. De hecho, en cepas clínicas correspondientes al mismo período, se identificó un plásmido de 90kb, portador de los genes sul1-blaTEM−1. Sin embargo, en ningún caso, la resistencia se asocia con la presencia de integrones o transposones31.
Importancia de la emergencia de plásmidos híbridos de virulencia/resistencia en bacteriasFinalmente, cabe destacar que la emergencia de plásmidos híbridos VR en salmonelas no tifoideas (bacterias zoonóticas, con amplio rango de hospedador y fuerte potencial epidémico) constituye un nuevo e interesante ejemplo de evolución bacteriana que puede conducir a la coselección de 2 grupos de determinantes (V y R) para una mejor adaptación de la bacteria en diferentes circunstancias: a) en situaciones no infectivas, pero en presencia de antimicrobianos, los determinantes R permiten la selección simultánea de los determinantes V, y b) en situaciones infectivas la presencia de los factores V garantiza el mantenimiento de los determinantes R, incluso en ausencia de la presión selectiva ejercida por el uso de antimicrobianos. Aunque la adquisición de nuevos plásmidos puede ocasionar un coste inicial a la bacteria receptora, se ha demostrado que éste puede ser compensado por mutaciones en el genoma del hospedador, que restauran, o incluso mejoran, su eficacia biológica en pocas generaciones2. Por tanto, los plásmidos VR aportan una gran ventaja adaptativa, dando lugar a la emergencia de «nuevas» cepas patógenas que podrían ser más difíciles de tratar. Esto constituye un grave problema sanitario, máxime si se tiene en cuenta la posible incorporación de determinantes R asociados a transposones en el cromosoma bacteriano, donde se estabilizan, así como la transferencia horizontal de los plásmidos VR, no sólo a otras cepas y serotipos de Salmonella sino también a diferentes géneros bacterianos. Este último hecho se ha constatado experimentalmente al transferirse distintos plásmidos híbridos a E. coli, vía conjugación o transformación28–30,42. La diseminación de estas «nuevas» cepas patógenas podría contribuir al incremento en la prevalencia de la salmonelosis, tanto en humanos como en animales.
Las autoras desean expresar su agradecimiento a todas las personas e instituciones que, de alguna manera, han contribuido a la investigación recogida en esta revisión, muy especialmente a las Dras. Beatriz Guerra (Federal Institute for Risk Assessment, Berlin, Germany), M. Ángeles González-Hevia (Laboratorio de Salud Pública, Principado de Asturias) y Aurora Echeita (Centro Nacional de Microbiología, Madrid).
Financiación
Nuestro trabajo acerca del tema se ha desarrollado en el marco de proyectos del Ministerio de Sanidad y Consumo, Fondo de Investigación Sanitaria (PI00-0184; PI02-0172 y PI05-2489) y Ministerio de Educación y Ciencia (SAF-2005-04212).