Globalamente la situación epidemiológica de la enfermedad por micobacterias no tuberculosas (MNT) es desconocida debido a que no es una enfermedad de notificación en la mayoría de los países, sin embargo los diversos reportes de infecciones y brotes por MNT publicados sugieren su creciente incremento en los últimos años. Tradicionalmente la identificación se realiza a través de metodologías bioquímicas, las cuales permiten la diferenciación inicial entre M. tuberculosis y MNT, y en algunos casos la identificación de la especie micobacteriana. Sin embargo, esta metodología tiene importantes limitaciones técnicas y la obtención de resultados requiere tiempos prolongados. Por otra parte, la introducción de metodologías basadas en biología molecular ha facilitado significativamente el diagnóstico de las MNT desde el laboratorio.
ObjetivoEstablecer la frecuencia de MNT en los cultivos positivos para bacilos ácido alcohol resistentes remitidos al Laboratorio de Salud pública de Bogotá en un período de 12 meses.
Materiales y métodosUn total de 100 cultivos provenientes de hospitales públicos y privados de Bogotá fueron identificados a través de pruebas bioquímicas y de las metodologías moleculares; PRA (PCR-análisis de restricción) y PCR múltiplex. Así mismo, la presencia de especies de baja prevalencia, así como el estudio de los casos no concluyentes se realizó través de análisis de secuenciación del blanco molecular 16SrDNA.
ResultadosEl estudio identificó MNT en el 11% de los cultivos a través de la metodología PRA. Adicionalmente, esta metodología molecular permitió detectar la ocurrencia de más de una especie micobacteriana en el 4% de los cultivos evaluados. Interesantemente un nuevo patrón de restricción-PRA para la especie M. kubicae es reportado en nuestro estudio.
ConclusiónEl uso de un algoritmo de identificación micobacteriana que incluya la metodología molecular PRA incrementa el poder diagnóstico de los métodos convencionales, y contribuye al mejor conocimiento de la epidemiología de las MNT y control de las micobacteriosis.
Global epidemiology of non-tuberculous mycobacteria (NTM) is unknown due to the fact that notification is not required in many countries, however the number of infection reports and outbreaks caused by NTM suggest a significant increase in the last years. Traditionally, mycobacteria identification is made through biochemical profiles which allow to differentiate M. tuberculosis from NTM, and in some cases the mycobacteria species. Nevertheless, these methods are technically cumbersome and time consuming. On the other hand, the introduction of methods based on molecular biology has improved the laboratory diagnosis of NTM.
ObjectiveTo establish the NTM frequency in positive cultures for acid-fast bacilli (AAFB) which were sent to Laboratorio de Salud Pública de Bogotá over a 12 month period.
Materials and methodsA total of 100 positive cultures for acid-fast bacilli from public and private hospitals from Bogotá were identified by both biochemical methods and the molecular methods PRA (PCR-restriction enzyme analysis) and multiplex-PCR. Furthermore, low prevalence mycobacteria species and non-interpretable results were confirmed by 16SrDNA sequentiation analysis.
ResultsIdentification using the PRA method showed NMT occurrence in 11% of cultures. In addition, this molecular methodology allowed to detect the occurrence of more than one mycobacteria in 4% of the cultures. Interestingly, a new M. kubicae pattern of PCR-restriction analysis is reported in our study.
ConclusionUsing a mycobacteria identification algorithm, which includes the molecular method PRA, improves the diagnostic power of conventional methods and could help to advance both NTM epidemiology knowledge and mycobacteriosis control.
Las micobacterias no tuberculosas (MNT) pueden ser saprofitas, patógenas u oportunistas y adicionalmente de rápido y de lento crecimiento1. En la actualidad se han descrito más de 150 especies2,3, y aunque estos microorganismos son agentes causales de enfermedad su patogenicidad varía entre especies4.
Por otra parte, hay limitada evidencia de transmisión de MNT persona-persona; se han descrito brotes causados por exposición al mismo reservorio5,6 y varios reservorios han sido claramente identificados para algunas de las especies micobacterianas; animales marinos para M. marinum, tuberías para M. avium complex y agua de grifo para M. gordonae, entre otros7. Así mismo las MNT han sido frecuentemente asociadas a diversas condiciones de salud, siendo agentes causales de enfermedad en poblaciones de pacientes con fibrosis quística, enfermedad pulmonar crónica, virus de la inmunodeficiencia humana y otras condiciones inmunosupresoras6–9.
A nivel global, la situación epidemiológica de la enfermedad por MNT es desconocida debido a que no es una enfermedad de notificación en la mayoría de los países; sin embargo los diversos reportes de infecciones y brotes por MNT publicados sugieren incrementos en la proporción de casos atribuidos a estos agentes infecciosos10. En Colombia se han descrito series de casos de infecciones oportunistas y hospitalarias causadas por MNT11,12, así como brotes en pacientes sometidos a mesoterapia13. Así mismo, un estudio realizado en pacientes infectados con el virus de la inmunodeficiencia humana aisló MNT en el 5,3% al 28% de los casos14. Sin embargo, en Colombia se desconoce la prevalencia de micobacteriosis debido a la falta de conocimiento de estas especies como causantes de enfermedad, y a limitaciones para su diagnóstico, el cual se realiza principalmente a través pruebas bioquímicas, a pesar del amplio conocimiento en la técnica molecular PRA (PCR-análisis de restricción)13,15-17.
Tradicionalmente, el diagnóstico de MNT se basa en pruebas bioquímicas, las cuales tardan varias semanas, son dispendiosas y algunas veces no logran diferenciar entre miembros de complejos18. Sin embargo, la descripción de la metodología de identificación molecular mediante reacción en cadena de la polimerasa PCR-análisis de restricción del gen hsp65 se ha convertido en una herramienta útil para la identificación rápida de micobacterias a un nivel inferior al de especie, y estudios globales han confirmado la reproducibilidad de la técnica17, así como su utilidad para la identificación directa de la especie desde la muestra clínica en corto tiempo y con alta especificidad19.
Actualmente otras metodologías basadas en métodos moleculares —PCR múltiplex, PCR-hibridación— han sido exitosamente evaluadas para la identificación de micobacterias tuberculosas y no tuberculosas20,21. Así mismo, los recientes avances en espectrometría de masas han hecho posible el uso de la técnica matrix-assisted laser desorption ionization- time-of-flight para identificar de forma reproducible las diferentes especies de micobacterias mediante la obtención de un espectro de masas único para cada una de estas22.
Materiales y métodosMuestras de cultivosUn total de 107 cultivos positivos para BAAR fueron remitidos al Laboratorio de Salud Pública de Bogotá durante 12 meses. Los cultivos se obtuvieron a partir de muestras de pacientes que acudieron a diferentes centros hospitalarios públicos y privados de la ciudad. En la mayoría de los casos no fue posible obtener información de la velocidad de crecimiento y pigmentación de los cultivos. Así mismo, un total de 20 cultivos de las cepas control M. fortuitum ATCC 6841, M. avium ATCC 15769 y M. tuberculosis ATCC 25177 se incluyeron como controles, para lo cual se analizaron en ciego a través de las metodologías moleculares.
La identificación bioquímica se llevó a cabo a través de las pruebas bioquímicas catalasa 37°C, catalasa 68°C, nitratos y niacina, o la metodología comercial TBc i test (Becton, Dickinson, EE. UU.) las cuales permiten únicamente la diferenciación entre M. tuberculosis y MNT.
Extracción del ADNEl ADN fue extraído y obtenido a partir de todas las suspensiones de los cultivos previamente identificados a través de las metodologías fenotípicas mediante el kit comercial QUIamp DNA mini and Blood Mini (Quiagen) con algunas modificaciones. Brevemente, la suspensión de colonias fue resuspendida en el buffer de extracción (20mM Tris-HCl pH 8; 2mM EDTA, 1,2% Triton; Lisozyma 20mg/ml), incubadas 30min/37°C, las proteínas fueron depletadas con proteinasa K. Seguidamente se adicionó el buffer AL y se incubó a 56°C/30min, se precipitó con etanol absoluto y se purifico a través de las columnas provistas por el kit, el ADN se resuspendió en buffer AE. El ADN extraído fue cuantificado a través de electroforesis en gel de agarosa al 0,8%, y fue almacenado a –20°C hasta su uso.
Metodología reacción en cadena de la polimerasa-análisis de restricciónPara la identificación molecular se siguió el procedimiento descrito por Telenti et al.23. Primero se amplificó un fragmento de 439bp a partir del ADN extraído de los cultivos de M. tuberculosis, empleando los oligonucleótidos TB11 (5’ ACCAACGATGGTGTGTCCAT) y TB12 (5’ CTTGTCGAACCGCATACCCT), a continuación el producto de la PCR se llevó a digestión con las endonucleasas HaeIII y BstEII. Finalmente, los productos digeridos fueron evaluados en un gel de agarosa al 3%, y la identificación de la especie micobacteriana se realizó siguiendo las recomendaciones establecidas en el sitio Web: http://app.chuv.ch/prasite, así como mediante comparación con otros algoritmos publicados24.
Reacción en cadena de la polimerasa múltiplexPara la identificación molecular se siguió el procedimiento descrito por Chia et al.20. Nueve pares de iniciadores fueron empleados para la identificación de 9 especies micobacterianas. Primero, para la detección de micobacterias de lento crecimiento se emplearon 7 pares de iniciadores, 6 de los cuales tienen como blanco molecular la región 16S–23S rRNA ITS-1 y el séptimo para la detección de un fragmento del gen que codifica para el antígeno ESAT-6 específico de M. tuberculosis complex (MTBC) TBF1a GTGGGGCGTAGGCCGTGA y TBR ACCTGGAACAAGTCCGAGTG, M. avium-intracellulare complex MACF CCCTGAGACAACACTCGGTC y MACR1a GAAATTACACATTTCGATGAAC, M. gordonae GORF CGACAACAAGCTAAGCCAGA GORR CAACGCATACATTTTGATGC, M. scrofulaceum SCOF TCGGCTCGTTCTGAGTGGTG y SCOR1a CGGCCACGCATCCGTTTA, M. kansasii MKF1a TCGGGCGCCCACCAGGAAT y MKR1a CGGCCCCGGACTTCTTTCGT, M. szulgai SZUF AACACTCAGGCTTGGCCAGA y SZUR GAGGGCAGCGCATCCAATTG, MTBC/M. kansasii/M. marinum ESAT-F GATCCCATGACAGAGCAGCAGT y ESAT-R TCTGCAGCGCGTTGTTCAGCT por otra parte, para la identificación de micobacterias rápido crecimiento; un par de iniciadores tiene como blanco la ITS-1, y el otro amplifica un fragmento del gen hsp65; M. fortuitum complex MFOR-F GCTGAGCTCGTCAAAGAGGTC y MFOR-R CACCTCCTTGGCGGACTTCAGG, M. chelonae-abscessus complex CHEF1a TGGTTACTCGCTTGGTGAATAT y CHER1a CCAGCGTTTCAATTCTATTGA. La mezcla de reacción de PCR se preparó con 1×PCR buffer, 0,5U of HotStarTaq DNA polimerasa (Invitrogen), 200μmol/l of each deoxinucleotido trifosfato, y 0,2μmol/l de cada iniciador. El ciclo de amplificación consistió de una denaturación inicial a 95°C/15min y luego 35 ciclos de denaturación a 95°C/1min, anillaje a 60°C (para lentas crecedoras) o 55°C (para rápidas crecedoras)/1min, y extensión a 72°C/1min. El tamaño de los productos de PCR fue evaluado mediante electroforesis en agarosa al 3%.
Análisis de secuenciación 16SrDNALas muestras con resultados inconcordantes se llevaron a amplificación para el blanco molecular 16SrDNA utilizando los iniciadores universales. Los productos se analizaron mediante secuenciación comercial. Los cromatogramas se analizaron en el programa BioEdit v7.2.5, y las secuencias nucleotídicas obtenidas fueron analizadas mediante el programa BLAST (www.ncbi.nlm.nhi.gov) y la base de datos de Ribosomal Differentiation of Medical Microorganisms (RIDOM) (http://www.ridom.de). Para los análisis de 16SrDNA la identificación de especie fue confirmada si la secuencia nucleotídica obtenida presentaba una similitud≥99%.
ResultadosUn total de 107 cultivos de micobacterias obtenidos de pacientes de Bogotá entre el 11 de enero de 2013 y el 01 de diciembre de 2013 fueron incluidos en nuestro estudio.
De los 107 cultivos 7 no pudieron incluirse dentro del análisis debido a que 5 estaban contaminados, y 2 correspondieron a cultivos duplicados, por lo cual un total de 100 muestras se analizaron de forma independiente a través de cada una de las metodologías seleccionadas (tabla 1).
Resultados de la identificación a través de las pruebas bioquímicas, PRA y PCR múltiplex
| MetodologíaResultado | P. bioquímicas | PRA | PCR múltiplex |
|---|---|---|---|
| M. tuberculosis | 66 | 83 | 89 |
| MNT | 26 | 11(2M.avium, 2M. gordonae, 3M. kubicae, M. brumae, M. intracelulare, M. fortuitum, M. abscessus) | 7(3M.avium, 2M. gordonae, M. fortuitum, M. chelonae) |
| Mixtaa | No procede | 4 | 0 |
| Inconcluyenteb | 8 | 2 | 4 |
| Total | 100 | 100 | 100 |
Cambios efectuados: n.° referencia 6, 19, 31.
Un total de 83 cultivos fueron identificados como M. tuberculosis a través de PRA, de los cuales 82 fueron concordantes con los resultados arrojados por la PCR múltiplex; y el restante cultivo no mostró amplificación para los blancos moleculares evaluados. Así mismo, las pruebas bioquímicas solo concordaron en identificar M. tuberculosis en 66 cultivos, mientras que en 13 casos sugirieron presencia de MNT y en 4 resultados no concluyentes.
En cuanto a la identificación de MNT, mientras PRA permitió identificar MNT en 11 cultivos, la PCR múltiplex identificó MNT en 7 cultivos; un cultivo identificado como M. intracellulare a través de PRA fue identificado como M. avium a través de esta metodología, un M. abscessus fue clasificado como M. chelonae y M. brumae como M. tubeculosis. En 2 cultivos no se obtuvo amplificación. En cuanto a los resultados de la identificación bioquímica se encontraron resultados concordantes para 10 de los cultivos identificados como MNT mediante PRA; y el cultivo identificado como M. brumae fue identificado como M. tubeculosis. Este cultivo fue posteriormente analizados a través de secuenciación 16SrDNA (tabla 2) que sugirió la presencia de M. brumae.
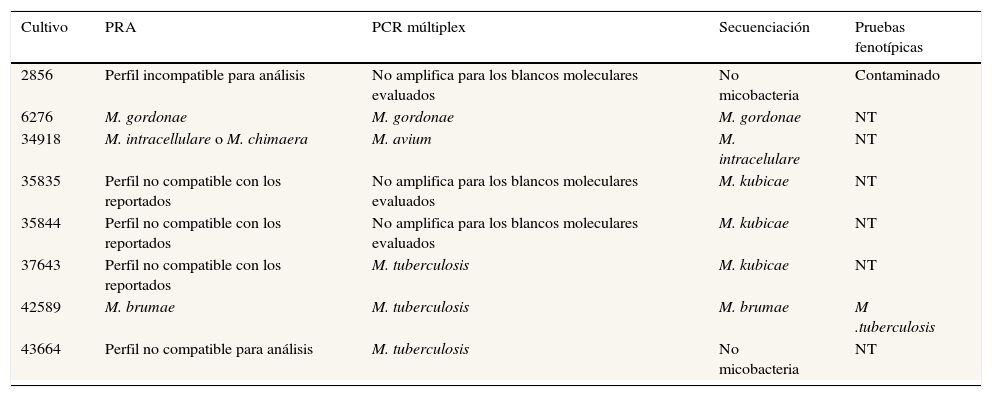
Resultados del análisis de secuenciación
| Cultivo | PRA | PCR múltiplex | Secuenciación | Pruebas fenotípicas |
|---|---|---|---|---|
| 2856 | Perfil incompatible para análisis | No amplifica para los blancos moleculares evaluados | No micobacteria | Contaminado |
| 6276 | M. gordonae | M. gordonae | M. gordonae | NT |
| 34918 | M. intracellulare o M. chimaera | M. avium | M. intracelulare | NT |
| 35835 | Perfil no compatible con los reportados | No amplifica para los blancos moleculares evaluados | M. kubicae | NT |
| 35844 | Perfil no compatible con los reportados | No amplifica para los blancos moleculares evaluados | M. kubicae | NT |
| 37643 | Perfil no compatible con los reportados | M. tuberculosis | M. kubicae | NT |
| 42589 | M. brumae | M. tuberculosis | M. brumae | M .tuberculosis |
| 43664 | Perfil no compatible para análisis | M. tuberculosis | No micobacteria | NT |
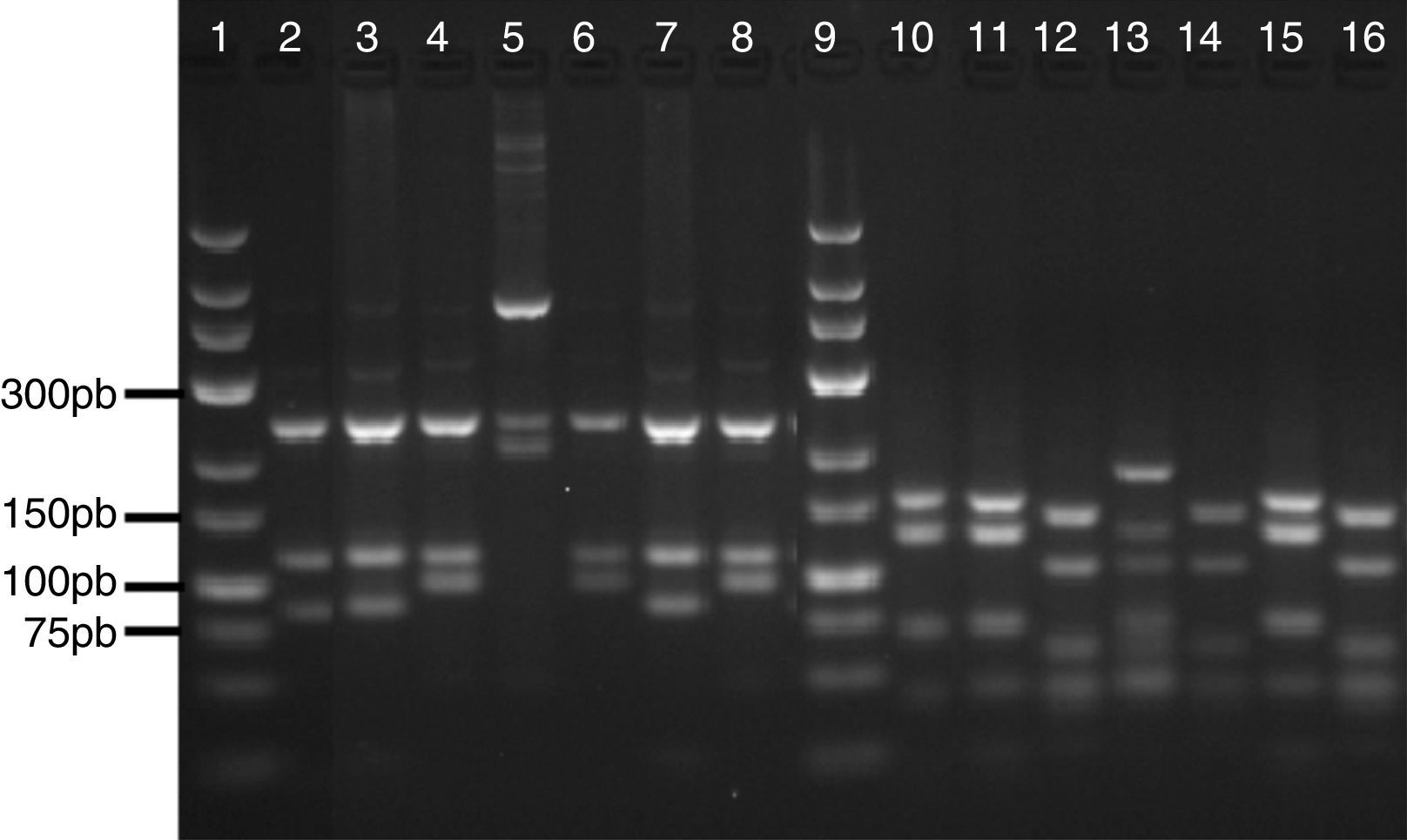
Es importante resaltar que en 3 de los cultivos identificados como MNT a través de PRA, los cuales mostraron idéntico patrón de restricción, los algoritmos disponibles no permitieron definir la especie micobacteriana, por lo cual las muestras fueron analizadas a través de secuenciación 16SrDNA, que permitió establecer la presencia de M. kubicae en todos los casos (tabla 2 y fig. 1).
Por otra parte, la metodología PRA permitió detectar la ocurrencia de infecciones mixtas, en nuestro caso M. tubeculosis y MNT simultáneamente en 4 cultivos. Sin embargo, estos cultivos fueron identificados como M. tubeculosis a través de la PCR múltiplex, y en 3 casos como MNT a través de las pruebas bioquímicas.
Extrañamente 2 cultivos no pudieron ser analizados a través de PRA, ya que se obtuvo resultados positivos a través de la amplificación, pero los patrones de restricción fueron totalmente incompatibles a los algoritmos disponibles. Uno de estos cultivos se identificó como M. tubeculosis a través de PCR múltiplex y el otro no amplificó, mientras que la identificación bioquímica identificó uno como MNT y otro mostró resultados no concluyentes. Estos 2 cultivos fueron analizados posteriormente mediante secuenciación 16SrDNA, la cual sugirió la presencia de otros géneros bacterianos (tabla 2).
En cuanto a los resultados de la identificación del total de los cultivos control mostraron 100% de concordancia en los resultados obtenidos a través de las 3 metodologías utilizadas.
DiscusiónLas metodologías moleculares han mostrado alto poder diagnóstico para la diferenciación de micobacterias25,26. Sin embargo, métodos fenotípicos basados en pruebas bioquímicas continúan siendo la metodología estandarizada para la identificación de las especies micobacterianas en un amplio número de laboratorios26,27.
En este estudio el 26% de los cultivos estudiados mostraron resultados compatibles para MNT a través de las pruebas bioquímicas, sin embargo a través de las metodologías moleculares PRA y PCR múltiplex se encontraron frecuencias de MNT del 11% y 7% respectivamente; esta notoria diferencia en los resultados obtenidos podría atribuirse a las conocidas limitaciones de las metodologías fenotípicas: a) baja reproducibilidad; b) los fenotipos esperados no son absolutos y pueden mostrar sustancial variabilidad; y c) la base de datos de las características fenotípicas está limitada a especies comunes28. Así mismo, las metodologías moleculares permiten obtener resultados satisfactorios con un bajo número de colonias, y aun en cultivos almacenados por largos periodos de tiempo; factores que contrariamente en las pruebas bioquímicas son indispensables para la obtención de resultados24.
Estos hallazgos sugieren la necesidad de complementar las metodologías fenotípicas con otros métodos de mayor poder diagnóstico29, y resaltan la importancia de confirmar la presencia de una MNT en por lo menos 2 muestras diferentes ante la sospecha de micobacteriosis4,15,30.
En el 82% (9 de 11) de los casos de MNT encontrados en este estudio a través de PRA fue posible establecer la especie micobacteriana a través de los patrones publicados en http://app.chuv.ch/prasite, resultados que fueron 100% concordantes con el análisis de secuenciación, hecho que confirma el alto poder diagnóstico de la metodología PRA para la identificación de micobacterias15,17,26,31. Sin embargo, para el 18% faltante (3 de 11) no fue posible establecer con certeza la especie debido a que los patrones de restricción reportados fueron incompatibles con nuestros hallazgos, situación que ha sido documentada en otros estudios32. Finalmente, la identificación de estos aislamientos con idéntico perfil de restricción fue posible con el análisis de secuenciación que sugirió para los 3 casos la presencia M. kubicae. Interesantemente, nuestros resultados evidencian un nuevo perfil de restricción (fig. 1) que podrá ser considerado ante la sospecha de M. kubicae; micobacteria de lento crecimiento, escotocromógena y hasta la fecha no reportada como agente causal de infección29. Interesantemente en este estudio la metodología molecular PRA permitió la identificación de esta especie en 3 cultivos obtenidos de 3 pacientes, en el mismo periodo de tiempo y en el mismo centro hospitalario; sin embargo, para ninguno de los casos fue posible reconfirmar la infección por esta MNT, ya que no fue posible la obtención de una segunda muestra para ningún caso, hecho que sugiere la ocurrencia de colonización por esta micobacteria o más posiblemente contaminación cruzada en el laboratorio.
En cuanto a los resultados obtenidos a través de la PCR múltiplex en el 6% de los cultivos no se obtuvo amplificación para los blancos evaluados, lo cual obedece a una limitación de la técnica, que únicamente detecta 7 de las especies micobacterianas más frecuentes20; sin embargo, el creciente número de MNT causantes de infección33 y el poco conocimiento que se tiene acerca de la epidemiología de estos agentes infecciosos en Colombia15 requiere de metodologías que permitan la identificación de un amplio rango de micobacterias4,25.
Entre los hallazgos interesantes, en un caso identificado como M. tuberculosis a través de las metodologías fenotípicas y PCR múltiplex la metodología PRA detectó la presencia de M. brumae, resultado que se confirmó a través de análisis de secuenciación 16SrDNA; estos resultados coinciden con hallazgos previamente reportados que sugieren baja concordancia entre las pruebas fenotípicas y esta metodología molecular, así como a la limitación de las pruebas fenotípicas y PCR múltiplex para identificar únicamente especies micobacterianas comunes31,28. Por otra parte, en 2 cultivos no pudo llevarse a cabo el análisis de restricción PRA a pesar de contar con resultados positivos para la PCR hsp65, hecho que puede explicarse por la presencia de este blanco molecular en otros géneros bacterianos34, y que pudo esclarecerse con los resultados del análisis de secuenciación que sugirieron ausencia de micobacterias.
Hwang et al.35 detectaron la ocurrencia en simultáneo de infecciones mixtas por micobacterias tuberculosas y no tuberculosas a través de la metodología molecular GenoBlot Assay. En nuestro estudio la metodología molecular PRA nos permitió identificar la coexistencia de más de una micobacteria en un cultivo, siendo esta una de las bondades de esta metodología molecular, y lo cual en la práctica clínica resulta de gran interés para el éxito terapéutico4.
Con la discrepancia observada entre los resultados arrojados por las diferentes metodologías evaluadas en este estudio, y la ocurrencia de patrones PRA no compatibles con lo reportado32, la utilización de herramientas robustas como la secuenciación36 nos permitió confirmar o excluir la presencia micobacteriana, así como identificar con certeza la especie, resultados que indican el valor diagnóstico de esta metodología como apoyo para el análisis e interpretación de casos especiales como los observados en nuestro estudio.
Estudios previos realizados en Colombia37 empleando metodologías de identificación fenotípica registraron un total de 19 casos por MNT para el año 2003, resultados que no muestran variaciones significativas frente a nuestros hallazgos; identificación fenotípica 25% (22 de 89), PRA 12% (11 de 89) y PCR múltiplex 11% (10 de 89), sin embargo la comparación de resultados de las pruebas fenotípicas estuvo limitada en este estudio al hecho de que la identificación fenotípica realizada correspondió únicamente a la diferenciación de micobacterias tuberculosas de MNT, sin llegar a definir la especie. Así mismo, la notoria diferencia en la frecuencia de MNT obtenida a través de las metodologías fenotípicas vs las pruebas moleculares empleadas en este estudio puede ser explicada por el alto poder diagnóstico de estas últimas17,23,31, las cuales discriminaron la presencia de más de una micobacteria en simultáneo; cultivos identificados como MNT por metodologías fenotípicas fueron identificados como cultivos mixtos de M. tuberculosis y MNT a través del análisis molecular. Interesantemente, un estudio multicéntrico que investigo 36.099 aislados de muestras clínicas de pacientes provenientes de 14 países identificó como especies más frecuentes Mycobacterium avium complex, M. gordonae, M. xenopi, M. kansasii y M. fortuitum38. Así mismo, Castro et al.15 en Colombia, utilizando la metodología PRA para la identificación de 70 cultivos de MNT recolectados durante 11 años de todo el país, reportaron como especies prevalentes: M. fortuitum, M. abscessus, M. gordonae, M. chelonae y M. intracelulare; a pesar de las notorias diferencias entre los estudios mencionados concernientes a tamaño y tipo de muestra nuestros resultados muestran concordancia en las especies de MNT detectadas, y nosotros encontramos en el 63% de las MNT aisladas M. avium, M. gordonae, M. abscessus y M. fortuitum.
ConclusiónEl uso de un algoritmo de identificación micobacteriana que incluya la metodología molecular PRA incrementa el poder diagnóstico de los métodos convencionales, y contribuye significativamente al mejor conocimiento de la epidemiología y control de la infección por MNT.
FinanciaciónEl presente trabajo fue desarrollado con fondos del Laboratorio de Salud Pública de Bogotá.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.