La espectrometría de masas Matrix-Assisted Laser Desorption-Ionisation Time-of-Flight (MALDI-TOF) es una técnica rápida y fiable de identificación bacteriana.
MétodosSe evalúa un método para identificación directa del hemocultivo fácil, rápido de realizar y de bajo coste.
ResultadosEl porcentaje de identificaciones con puntos de corte de ≥1,7 y ≥1,4 para aceptable identificación de especie y género fue del 77,5 y del 93,8%, respectivamente.
ConclusiónEl buen funcionamiento, la rapidez y el bajo coste hacen que esta técnica sea apropiada y fácil de implantar en los laboratorios de microbiología clínica.
Matrix-Assisted Laser Desorption-Ionisation Time-of-Flight (MALDI-TOF) Mass Spectrometry is rapid and accurate for the bacterial identification.
MethodsWe have evaluated a less laborious and less time consuming method for microorganism identification directly from positive blood cultures.
ResultsWhen we considered the scores ≥1.7 and ≥1.4 for acceptable identification of species and genus, the percentage of identification was 77.5% and 93.9%, respectively.
ConclusionsThis method is reliable, rapid and cost-effective for implementation in routine use in clinical microbiology laboratories.
La espectrometría de masas Matrix-Assisted Laser Desorption-Ionisation Time-of-Flight (MALDI-TOF) ha demostrado ser una técnica rápida y fiable para la identificación bacteriana en los laboratorios de microbiología clínica. Su realización directamente a partir de los hemocultivos positivos permite reducir considerablemente el tiempo de obtención del resultado respecto a los métodos convencionales, ofreciendo la posibilidad de implementar un tratamiento antimicrobiano precoz más dirigido en los pacientes con sepsis.
Hasta la fecha se han publicado varios protocolos, algunos basados en diferentes centrifugaciones y lavados para eliminar los eritrocitos1 y otros que utilizan tubos separadores de suero2, ambos seguidos de una extracción con ácido fórmico/acetonitrilo.
Bruker Daltonics (Bremen, Alemania) ha comercializado un método, Sepsityper®, con el cual se han obtenido buenos resultados de identificación3 pero que, al incluir también un paso de extracción, resulta laborioso cuando se utiliza en la rutina diaria del laboratorio de microbiología clínica.
En un estudio realizado con la base de datos de ANDROMAS4 se ha descrito un método que permite reducir el coste y el tiempo utilizando saponina como agente lisante de eritrocitos. Más recientemente, Martiny et al.5 han desarrollado un procedimiento parecido pero utilizando la base de datos de BRUKER (Biotyper), eliminando una de las 2 fases de lavado y sin ningún tipo de extracción, mostrando buenos resultados.
El objetivo de nuestro trabajo ha sido evaluar los resultados de las identificaciones bacterianas obtenidas con MALDI-TOF directamente del hemocultivo, utilizando una modificación de la técnica de Martiny et al. que podría mejorar la tasa de identificaciones sin aumentar el tiempo de realización.
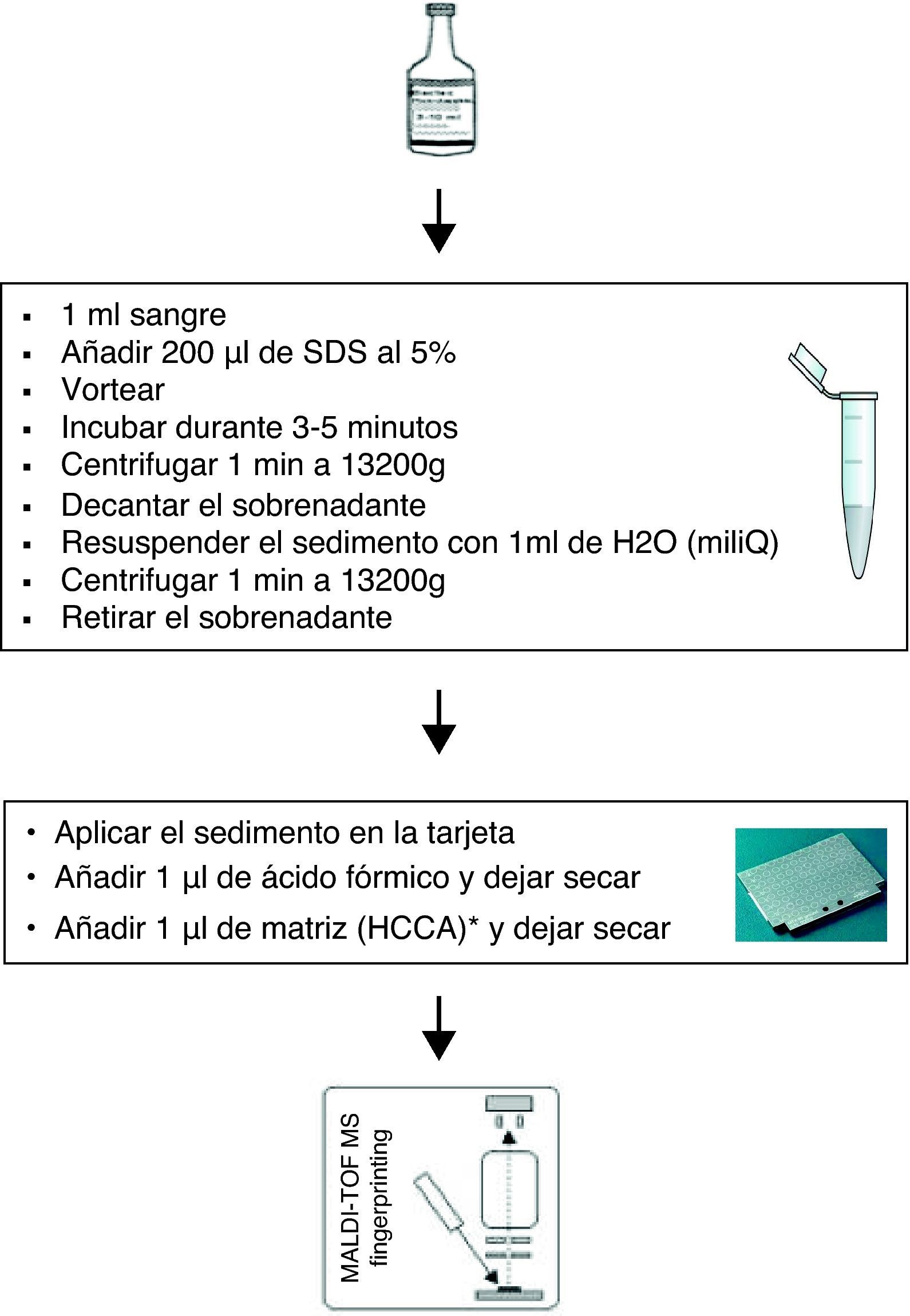
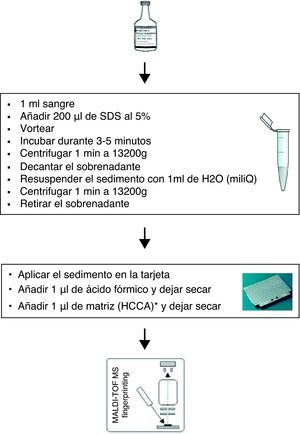
MétodosDurante el periodo comprendido entre febrero y abril 2012, 100 hemocultivos (BACTEC Plus Aerobic/Anaerobic, Becton Dickinson, Franklin Lakes, NJ, EE.UU.) positivos elegidos aleatoriamente se estudiaron con la técnica de MALDI-TOF y se compararon simultáneamente con los métodos convencionales utilizados en nuestro laboratorio: tinción de Gram, WIDER (Soria Melguizo, España), Vitek (BioMerieux, Marcy L’Etoile, Francia) y MALDI-TOF de las colonias. La técnica de MALDI-TOF directamente del hemocultivo se realizó como la descrita por Martiny pero se añadió un procedimiento de extracción rápida con ácido fórmico6 sobre la extensión de la muestra en la tarjeta y se utilizó como detergente dodecilsulfato sódico (SDS) en lugar de saponina (fig. 1).
Las muestras se analizaron en un microflex LT system, calibrado con Bruker Bacterial Test Standard. Para la identificación se sumaron hasta 240 disparos en modo automático, el espectro resultante se importó al software MALDI BioTyper 3.0 y se comparó con la base de datos existente. El análisis se realizó siempre por duplicado y se retuvo el valor más alto de las 2 determinaciones. Los resultados de las identificaciones se clasificaron en diferentes franjas de valores y se caracterizaron utilizando 2 tipos de puntos de corte. Por una parte, los recomendados por los fabricantes, en donde ≥2 identifica como especie, entre 1,9 y 1,7 identifica como género y <1,7 no identifica. Por otra, los basados en la experiencia de otros autores2,7 que consideran que puntos de corte más bajos (PCMB) como ≥1,7 y ≥1,4 son aceptables para especie y género, respectivamente, y <1,4, «no identifica».
ResultadosDe los 100 hemocultivos positivos, 2 fueron falsos positivos, los cuales, con valores <1,4, se clasificaron por la técnica de MALDI-TOF como «no identifica» con ambos puntos de corte.
Los 98 restantes presentaron la siguiente distribución: 25 bacilos gramnegativos (BGN), 71 grampositivos (GP) y 2 levaduras. No hubo ningún hemocultivo polimicrobiano.
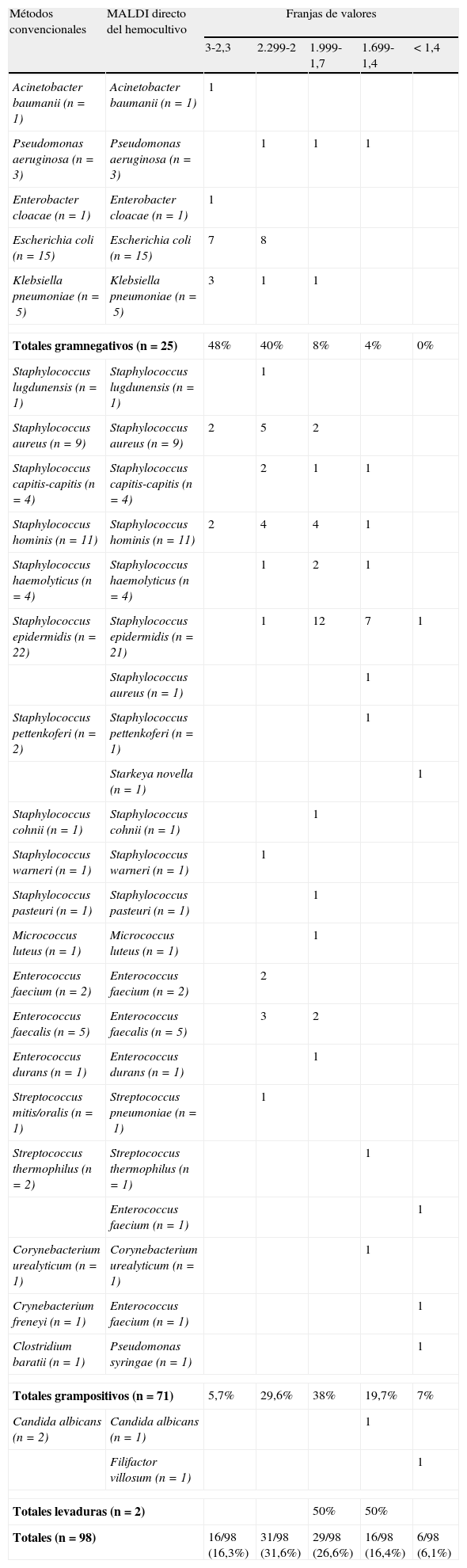
En la tabla 1 se reflejan los microorganismos aislados y la comparación de su identificación por los métodos convencionales usados en el laboratorio con la resultante del MALDI directo del hemocultivo con sus correspondientes valores.
Comparación de la identificación bacteriana con los métodos convencionales y la técnica rápida de MALDI-TOF directa del hemocultivo
| Métodos convencionales | MALDI directo del hemocultivo | Franjas de valores | ||||
| 3-2,3 | 2.299-2 | 1.999-1,7 | 1.699-1,4 | <1,4 | ||
| Acinetobacter baumanii (n=1) | Acinetobacter baumanii (n=1) | 1 | ||||
| Pseudomonas aeruginosa (n=3) | Pseudomonas aeruginosa (n=3) | 1 | 1 | 1 | ||
| Enterobacter cloacae (n=1) | Enterobacter cloacae (n=1) | 1 | ||||
| Escherichia coli (n=15) | Escherichia coli (n=15) | 7 | 8 | |||
| Klebsiella pneumoniae (n=5) | Klebsiella pneumoniae (n=5) | 3 | 1 | 1 | ||
| Totales gramnegativos (n=25) | 48% | 40% | 8% | 4% | 0% | |
| Staphylococcus lugdunensis (n=1) | Staphylococcus lugdunensis (n=1) | 1 | ||||
| Staphylococcus aureus (n=9) | Staphylococcus aureus (n=9) | 2 | 5 | 2 | ||
| Staphylococcus capitis-capitis (n=4) | Staphylococcus capitis-capitis (n=4) | 2 | 1 | 1 | ||
| Staphylococcus hominis (n=11) | Staphylococcus hominis (n=11) | 2 | 4 | 4 | 1 | |
| Staphylococcus haemolyticus (n=4) | Staphylococcus haemolyticus (n=4) | 1 | 2 | 1 | ||
| Staphylococcus epidermidis (n=22) | Staphylococcus epidermidis (n=21) | 1 | 12 | 7 | 1 | |
| Staphylococcus aureus (n=1) | 1 | |||||
| Staphylococcus pettenkoferi (n=2) | Staphylococcus pettenkoferi (n=1) | 1 | ||||
| Starkeya novella (n=1) | 1 | |||||
| Staphylococcus cohnii (n=1) | Staphylococcus cohnii (n=1) | 1 | ||||
| Staphylococcus warneri (n=1) | Staphylococcus warneri (n=1) | 1 | ||||
| Staphylococcus pasteuri (n=1) | Staphylococcus pasteuri (n=1) | 1 | ||||
| Micrococcus luteus (n=1) | Micrococcus luteus (n=1) | 1 | ||||
| Enterococcus faecium (n=2) | Enterococcus faecium (n=2) | 2 | ||||
| Enterococcus faecalis (n=5) | Enterococcus faecalis (n=5) | 3 | 2 | |||
| Enterococcus durans (n=1) | Enterococcus durans (n=1) | 1 | ||||
| Streptococcus mitis/oralis (n=1) | Streptococcus pneumoniae (n=1) | 1 | ||||
| Streptococcus thermophilus (n=2) | Streptococcus thermophilus (n=1) | 1 | ||||
| Enterococcus faecium (n=1) | 1 | |||||
| Corynebacterium urealyticum (n=1) | Corynebacterium urealyticum (n=1) | 1 | ||||
| Crynebacterium freneyi (n=1) | Enterococcus faecium (n=1) | 1 | ||||
| Clostridium baratii (n=1) | Pseudomonas syringae (n=1) | 1 | ||||
| Totales grampositivos (n=71) | 5,7% | 29,6% | 38% | 19,7% | 7% | |
| Candida albicans (n=2) | Candida albicans (n=1) | 1 | ||||
| Filifactor villosum (n=1) | 1 | |||||
| Totales levaduras (n=2) | 50% | 50% | ||||
| Totales (n=98) | 16/98 (16,3%) | 31/98 (31,6%) | 29/98 (26,6%) | 16/98 (16,4%) | 6/98 (6,1%) | |
A pesar de retener solo el microorganismo con el valor más alto de las 2 determinaciones, cabe destacar que coincidieron las identificaciones en todos los duplicados a excepción de 7 casos que correspondían a cepas con valor <1,4 y, por lo tanto, fueron considerados como «no identifica».
Utilizando los puntos de corte comerciales, se identificaron correctamente a nivel de género el 96% de los BGN y el 73,3% de los GP, a nivel de especie el 88% de los BGN y el 35,3% de los GP, y no se identificaron el 4% de los BGN y el 26,7% de los GP. Cuando se utilizaron los PCMB se obtuvo identificación correcta de género en el 100% de los BGN y en el 93% de los GP, identificación correcta de especie en el 96% de BGN y el 73,3% de GP, y el 7% de los GP no fueron identificados.
Dentro del grupo de los GP, la única identificación errónea fue un Streptococcus mitis, que se identificó como Streptococcus pneumoniae, un error bastante común debido a la gran semejanza de estas 2 especies8 y que se produjo con los 2 tipos de punto de corte. En las últimas actualizaciones del software ya se advierte que la técnica no es capaz de diferenciar entre estas 2 especies, limitando la identificación a S. pneumo/mitis, por lo que más que un error debe considerarse una limitación de la técnica MALDI-TOF.
El único anaerobio aislado, Clostridium baratii, no fue identificado. Respecto a las levaduras, solo una pudo identificarse a nivel de género y cuando se utilizaron los PCMB.
El porcentaje total de identificaciones correctas usando los puntos de corte comerciales fue de 77,5% a nivel de género, 47,9% a nivel de especie y 22,5% no fueron identificados. Al utilizar los PCMB, las cifras fueron del 93,9, del 77,5 y del 6,1%, respectivamente.
DiscusiónNuestro protocolo, como el descrito por Martiny, es perfectamente adaptable a la rutina de trabajo en un laboratorio de microbiología clínica y proporciona un mayor porcentaje de identificaciones correctas que puede deberse al uso del método de extracción rápido con ácido fórmico. Saffert et al.9, combinando SDS y una extracción larga, obtienen resultados muy similares a los nuestros con una serie muy semejante.
La gran diferencia entre el porcentaje de identificaciones correctas entre BGN y GP es un hecho ya constatado en otras series1,5. En nuestro estudio se ha aislado un mayor porcentaje de GP y observamos que la baja tasa de identificación correcta, sobre todo a nivel de especie y con los puntos de corte establecidos por los comerciantes (35,3%), se debe principalmente a especies de estafilococos coagulasa negativos, que en muchos casos son considerados contaminantes pero que son aislados frecuentemente en hemocultivos. La mejora que resulta cuando aplicamos los PCMB (73,3%) podría ser mayor con la ampliación del número de espectros de cada especie incluida en la base de datos10.
Pensamos que la fiabilidad de las identificaciones con PCMB, ya demostrada por varios artículos, sugiere un reajuste de los puntos de corte. Nosotros también observamos en este estudio la buena concordancia de las identificaciones de MALDI-TOF y de los métodos convencionales cuando se aplican PCMB. Algunos autores consideran adecuado aplicar en estos casos criterios adicionales, como la necesidad de que coincidan las 3 primeras identificaciones de MALDI-TOF o que exista una diferencia significativa (generalmente 0,3) entre las primeras identificaciones coincidentes y la primera discrepante. En nuestro estudio, aunque solo hemos aplicado el criterio de PCMB, hemos observado que además en todos los casos en que se consideró aceptable identificación de género o aceptable identificación de especie, las 3 primeras identificaciones de MALDI-TOF coincidían en el género y en la especie, respectivamente. En 15 de los 16 microorganismos con valores en el intervalo de 1,7 a 1,4, la identificación con ambos métodos coincidía incluso también a nivel de especie, aunque 5 coincidían solo en la primera identificación proporcionada por MALDI-TOF.
Como limitación de nuestro trabajo tendríamos que destacar la casi ausencia de anaerobios (solo un aislado) o de levaduras (solo 2), que no nos permite sacar conclusiones. En ambos tipos de microorganismos se ha descrito su dificultad para ser identificados por la técnica de MALDI-TOF y se ha sugerido la necesidad de ampliar la base de datos para ellos. Algunos autores mejoran sus resultados con las 2 modificaciones que aquí se realizan, la extracción rápida con ácido fórmico y PCMB similares a los aquí utilizados6.
Por último nos gustaría resaltar el bajo coste de este método, tanto en reactivos como en tiempo. La técnica para un hemocultivo se realiza en aproximadamente 20min, pero si consideramos que en la rutina diaria se suelen trabajar varios hemocultivos a la vez, el tiempo por hemocultivo se vería aún más reducido.
Concluimos que los buenos resultados, unidos a la rapidez y al bajo coste de esta técnica, hacen de ella una medida apropiada y fácil de implantar dentro de los laboratorios de microbiología clínica.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.