Introducción
En los últimos años, la contribución de la quimioterapia a la infección por el virus de la inmunodeficiencia humana (VIH) ha sido muy significativa en términos de supervivencia y mejora de la calidad de vida. Sin embargo, un tratamiento curativo para la infección sigue siendo un objetivo incierto, ya que no se consigue la eliminación completa de virus, que se mantiene latente y con capacidad de reactivación en un pequeño reservorio celular1,2. Todo esto hace que se haya estimulado la aplicación a este problema de otras alternativas terapéuticas como el trasplante de médula, la terapia celular, la inmunoestimulación, la utilización de vacunas terapéuticas e intervenciones genéticas dentro de lo que se ha venido en llamar terapia génica3.
Si se tiene en cuenta que el VIH se integra dentro del genoma de la célula del hospedador se puede considerar el sida como una enfermedad genética adquirida4. Aunque se ha intentado la curación genética del provirus mediante procedimientos de escisión5, la mayor parte de los diseños de terapia génica anti-VIH se han dirigido a la introducción de un gen antiviral dentro de la célula que prevenga la infección o inhiba la expresión del virus. Este concepto fue originalmente bautizado por David Baltimore como "inmunización intracelular"6 y constituye el objetivo último de la terapia génica. Esta disciplina, que nació fruto del extraordinario avance en genética y patología molecular, ha generado muchas expectativas desde sus inicios a principios de los noventa que no siempre se han visto acompañadas por resultados clínicos convincentes7. La realidad ha demostrado que, al igual que nuestra comprensión de la enfermedad producida por el VIH, el desarrollo de la terapia génica es mucho más complejo de lo que se preveía. La tarea de introducir y expresar genes en células somáticas necesita de un detallado conocimiento de las bases moleculares de la enfermedad y también de la mejora de nuestras herramientas para conseguir la transferencia de genes8. En relación con estos avances el campo de la terapia génica ha experimentado, como veremos a continuación, momentos de intensa euforia y decepción, a la vez que se han descrito formidables herramientas para el tratamiento molecular como el mecanismo de ARN de interferencia (ARNi).
Estrategias moleculares anti-VIH
El VIH es un retrovirus complejo que aparte de los productos de los genes gag, pol y env de los retrovirus simples utiliza seis proteínas accesorias cuya función es esencial para la replicación del virus y completar el ciclo infectivo. Algunos de estos productos, fundamentalmente tat y rev, junto a los receptores necesarios para la infección celular, han sido los candidatos más importantes contra los que se han dirigido la mayoría de las intervenciones moleculares.
Proteína tat
La proteína tat es un potente transactivador del promotor LTR del VIH-1 y es esencial para la replicación del virus9. Se han utilizando dos mecanismos para interferir con tat: en el primero se expresa un ARN antisentido correspondiente a la región TAR, de forma que su unión impida la localización de tat en esta estructura y por lo tanto su función4. Otra estrategia de bloqueo de tat consiste en la expresión de grandes cantidades de fragmentos de ARN correspondientes a TAR, utilizándolos como señuelo para disminuir el número de moléculas efectivas de tat que puedan unirse al TAR retroviral10.
Rev
La porción carboxiterminal de rev contiene una región rica en residuos de leucina que funciona como señal de exportación nuclear a través de la interacción con la maquinaria de transporte celular11. Se han construido diferentes transdominantes de Rev pero el más utilizado es el conocido como RevM10, que contiene una mutación precisamente en la región de interacción con proteínas celulares12. Este producto RevM10 se ha utilizando por varios grupos en ensayos clínicos en seres humanos13,14 con resultados muy limitados15,16. RevM10 tiene algunas características muy atractivas desde el punto de vista de su utilización como antiviral: carece de toxicidad celular, es muy poco inmunogénico y el desarrollo de variantes resistentes, aunque recientemente se han logrado inducir in vitro17, parece más difícil que cuando se utiliza una diana enzimática.
Receptores
Los aislamientos detectados en las primeras fases de la infección utilizan invariablemente la molécula CCR5 como correceptor18). La significación de este hecho cobró una mayor dimensión al descubrirse que algunos individuos homozigóticos para una deleción de 32 pb en el gen de CCR519,20, presente hasta en el 1% de la raza blanca21, eran resistentes a la infección por el VIH-1 y que incluso los individuos portadores de esta deleción de manera heterozigótica, si bien podían infectarse, presentaban una evolución clínica más lenta22,23. Parece claro que la interferencia con la expresión de CCR5 y CXCR4 podría tener importantes beneficios terapéuticos. Es importante puntualizar que si bien la completa anulación de CCR5 es el caso de los homozigóticos para D32CCR5, no parece asociarse con ninguna inmunodeficiencia, el bloqueo de CXCR4 podría ser más problemático, ya que, al menos en ratones, la anulación de CXCR4 tiene consecuencias graves sobre la hematopoyesis y el desarrollo cerebral, aunque su anulación en etapas posteriores al desarrollo podría ser mejor tolerada.
Una de las alternativas para bloquear la expresión de los correceptores en la membrana celular es la utilización de sus mismos ligandos naturales modificados de tal manera que puedan unirse intracelularmente a sus receptores y a la vez quedar bloqueados en el retículo endoplásmico (RE), limitando así la accesibilidad del virus para este correceptor. Estas moléculas que pueden inhibir el transporte de sus receptores hacia la membrana celular se conocen como "intracinas"24. Las intracinas, cuyo potencial antiviral se está evaluando, están basados en RANTES, MIP-1α y MIP-1β para CCR5 (MIP-1β es completamente específico para CCR5 mientras que RANTES y MIP-1α también pueden bloquear otros receptores) y SDF-1 para CXCR5.
Aparte de las intracinas se pueden utilizar otras estrategias para bloquear la expresión de estos correceptores a un nivel diferente, por ejemplo mediante la utilización de ribozimas. Los ribozimas catalizan el corte específico de secuencias de ARN y debido a su eficiencia y a la simplicidad de su diseño se han convertido en interesantes herramientas para la supresión selectiva de expresión de genes25. Recientemente26 se ha realizado el diseño y estudio funcional de una ribozima, hammerhead, RzR5-76, que hidroliza de manera específica el ARN mensajero (ARNm) de CCR5. Los resultados de este ribozima en un modelo de transfección in vitro indican que su actividad catalítica permite una importante reducción en la expresión del correceptor CCR5 en la superficie celular26. Frente al receptor CD4 se han utilizado igualmente diferentes estrategias, entre ellas la fabricación de quimeras de CD4 con señales de retención en RE27.
Herramientas para la transferencia genética
Uno de los factores limitantes para convertir la terapia génica en una alternativa terapéutica realista es sin duda el desarrollo de vehículos genéticos o vectores seguros y efectivos, con los que se pueda conseguir la restauración de un defecto genético o, como es el caso del sida, transferir resistencia antiviral a las poblaciones celulares susceptibles. Hay diferentes posibilidades de introducir ADN dentro de una célula (tabla 1), pero en la actualidad son los vectores basados en virus recombinantes, y sobre todo los retrovirus, los más utilizados.
Retrovirus
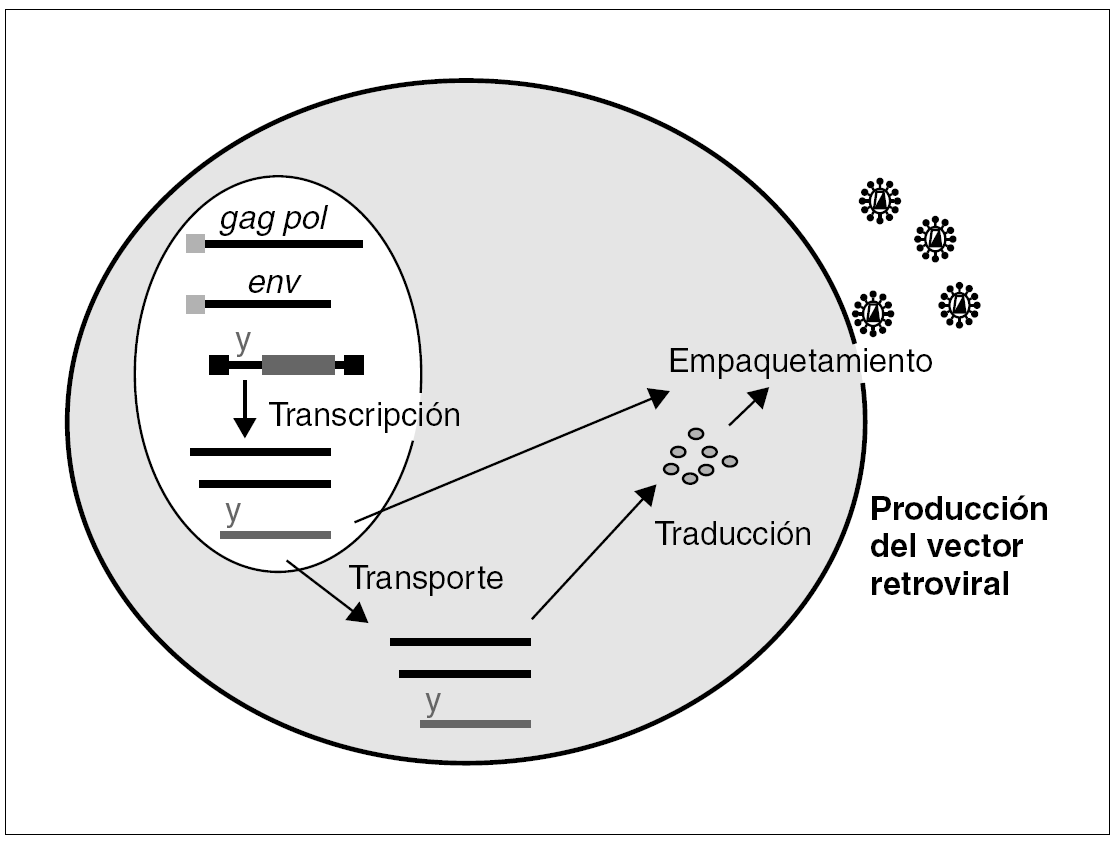
Las propiedades que les hacen tan peligrosos desde el punto de vista patogénico son precisamente las que les convierten en instrumentos muy atractivos como vectores en terapia génica: infectan las células muy eficazmente y una vez integrado su genoma, sobre todo si está desposeído de proteínas estructurales, su expresión es inmunológicamente muy silente. ¿Cómo se fabrica un retrovirus recombinante? El planteamiento inicial es sencillo, a partir de la estructura de un oncorretrovirus murino simple, el virus de la leucemia murina (MLV) (fig. 1), se eliminan la mayor parte de las secuencias gag, pol y env, que codifican sus productos estructurales, y se sustituyen por la secuencia del gen que queremos expresar. El resto de los componentes estructurales de la partícula viral se aportan en trans, dirigidos por un promotor heterólogo. De esta forma se pueden producir partículas retrovirales defectivas, con capacidad para infectar células e integrar su genoma, pero, al carecer este de los genes estructurales, sin la capacidad de producir un nuevo ciclo infectivo.
Figura 1. Célula empaquetadora para la producción de retrovirus recombinantes: los componentes estructurales, gag-pol y env, de la partícula retroviral así como el ARN genómico se introducen en la célula por medio de transfección de diferentes plásmidos. Todos estos componentes se empaquetan y generan una partícula infecciosa defectiva, carece de genes estructurales, que integrará el transgén en la célula diana.
Vectores lentivirales
A pesar de la experiencia acumulada con los vectores basados en oncorretrovirus murinos, existe una importante limitación para su utilización con fines terapéuticos derivada de su incapacidad para transducir células que no se encuentren activamente dividiéndose, como células musculares, nerviosas o hematopoyéticas.
Esta es la principal razón para utilizar vectores basados en lentivirus que, debido a las propiedades cariofílicas de sus complejos preintegración, consiguen infectar con eficacia células en reposo28,29. Los primeros vectores lentivirales fueron los derivados del mismo VIH-128,29 y posteriormente se han utilizado otros lentivirus: VIH-230, virus de la inmunodeficiencia del simio (SIV)31 y virus de la inmunodeficiencia en felinos (FIV)32. Evidentemente la utilización de vectores basados en agentes con capacidad patogénica debe realizarse bajo condicionantes de bioseguridad extrema que hagan imposible la recombinación y generación de variantes competentes33-35, por ejemplo el diseño de vectores lentivirales incluye en la actualidad un mínimo de secuencias originales, escasamente el 40%, e introduce mecanismos de seguridad como autoinactivación del promotor (LTR)33,36,37.
Una de las aplicaciones más atractivas de los vectores lentivirales es la transducción de precursores, células madre, con la mayor capacidad de autorrenovación y diferenciación posible. De esta manera se podría asegurar la expresión del transgén en células diferenciadas, por ejemplo linfocitos CD4+ resistentes al VIH en el caso del sida, que derivarían ilimitadamente de esta población de células madre. Los estudios en el modelo animal de reconstitución hematopoyética NOD/Scid, un ratón con inmunodeficiencia combinada grave y déficit de actividad natural killer (NK)38, son muy prometedores: se ha demostrado que la repoblación de todo el sistema hematopoyético humano a partir de precursores de sangre de cordón que han sido transducidos ex vivo con un marcador genético por medio de un vector lentiviral y que la expresión del marcador se mantiene durante meses39. Así mismo se ha podido demostrar transducción y expresión mantenida en otros tejidos con células quiescentes, cerebro28,29 y retina40 en otros modelos experimentales utilizando vectores lentivirales.
No obstante, la utilización de vectores basados en VIH-1 despierta una lógica prevención y es también comprensible que a pesar de los resultados conseguidos en los estudios preclínicos, las exigencias de bioseguridad retarden su aplicación en seres humanos. También es previsible que sean precisamente pacientes ya infectados con VIH-1 los primeros en los que se utilicen vectores lentivirales. Si bien el vector retroviral defectivo que expresa el marcador de resistencia puede de hecho ser rescatado por el virus natural, esto incluso podría tener el beneficio añadido de expandir el gen de resistencia33.
Situación de los estudios de terapia génica en infección por VIH hasta el año 2000
Se han autorizado hasta esa fecha un número considerable de estudios clínicos que utilizan terapia génica para el tratamiento de la infección por VIH-1. Estos estudios han consistido en fases I y II en las que el objetivo fundamental ha sido confirmar la seguridad de los procedimientos y la ausencia de toxicidad de las intervenciones genéticas sobre células humanas. Los resultados de eficacia clínica que utilizan estos vectores retrovirales de primera generación han sido muy limitados. El estudio más significativo con resultados publicados hasta la fecha16 ha permitido demostrar, en pacientes infectados con el VIH-1, una supervivencia más prolongada de los linfocitos CD4 que expresan el gen antiviral RevM10 en comparación con aquellos que expresan un control no funcional. Sin embargo, esta ventaja selectiva de la población celular protegida no consiguió una reducción significativa de la carga viral, probablemente debido a que la cantidad de células CD4 transducidas, en torno al 0,1% de las circulantes, es demasiado pequeña para modular la expresión de la enfermedad.
Problemas en la utilización de vectores retrovirales
En abril de 2000 se comunicó por el grupo de Alain Fischer y Marina Cavazzana-Calvo del Hospital Necker de París el primer estudio de terapia génica en el que se conseguía un éxito terapéutico inequívoco. El protocolo clínico se realizó sobre un grupo de 10 niños con una forma de inmunodeficiencia combinada grave ligada al cromosoma X (SCIDX1) por la ausencia de la subunidad g de los receptores de IL-2, 4, 7, 9, 15 y 21. El tratamiento consistía en la reposición, mediante un vector retroviral del receptor deficitario (IL2Rgc) en células hematopoyéticas CD34 autólogas. Después de la reinfusión de las células modificadas, 9 de los 10 niños presentaron una rápida repoblación cuantitativa y funcional41. La curación por medio de terapia génica se había conseguido. Sin embargo, todo el extraordinario optimismo generado por estos resultados se transformó en decepción al comprobar que dos de los niños estaban desarrollando leucemia42. El estudio de la población de células leucémicas confirmó que en los 2 casos el retrovirus que expresaba el gen IL2Rgc se había integrado en las proximidades del promotor del oncogén linfocitario LMO243. Estos graves efectos secundarios obligaron a una moratoria en la realización de ensayos con retrovirus en la mayor parte de los países. En el caso de la deficiencia de IL2Rg se puede haber dado una secuencia de desgraciadas circunstancias: la sobreexpresión de IL2Rg puede ser en sí misma oncogénica lo cual, unido al alto número de células modificadas, aumenta la probabilidad de inserción cerca de otro oncogén. Serían precisamente estas células las que tienen un mayor potencial de selección durante la repoblación y eventualmente podrían desarrollar leucemia44.
La integración de los retrovirus y el VIH en el genoma humano se consideraba aleatoria pero en la actualidad sabemos que se produce en zonas claramente preferentes (hotspots). La disponibilidad de la secuencia del genoma humano junto a una mayor capacidad de analizar regiones de inserción gracias a la amplificación y secuenciación robotizada ha permitido determinar que el VIH se inserta preferentemente en genes activos45. En el caso de los retrovirus murinos las zonas preferentes parecen ser regiones justamente en las proximidades de los promotores46, lo cual, indudablemente, introduce una mayor prevención en la utilización de estos vectores47.
ARN de interferencia
La reciente descripción del mecanismo conocido como ARN de interferencia (ARNi) ha desencadenado un extraordinario interés y se ha convertido en un corto espacio de tiempo en una de las áreas más activas de investigación en biología. El fenómeno que tiene como protagonista al ARNi consiste en el silenciamiento específico de la expresión de determinados genes por fragmentos cortos de ARN de doble cadena (ARNdc). En 1998 Fire et al, utilizando como modelo experimental el nematodo Caenorhabditis elegans, confirmaron que un fenómeno curioso de silenciamiento postranscripcional, observado inicialmente en plantas48, estaba mediada por fragmentos de ARNdc complementarios al ARNm de los genes silenciados, y comprobaron que pequeñas cantidades de ARNdc pueden tener efectos de inhibición muy significativos49. Tres años después, en 2001, Elbashir et al50,51 demuestran que el protagonista fundamental del fenómeno de interferencia es un pequeño fragmento de ARNdc de 21 nucleótidos y presentan datos que confirman que estos fragmentos pequeños de ARN inhibidores (ARN de pequeña interferencia o ARNpi) son activos no sólo en células de plantas e invertebrados, sino también de vertebrados superiores y humanos. Este descubrimiento abre un campo totalmente nuevo en la comprensión de la regulación génica y al mismo tiempo permite vislumbrar la disponibilidad de una herramienta con un extraordinario potencial.
A diferencia del interferón (IFN), el ARNi es un proceso exquisitamente específico que actúa selectivamente sobre el ARN para el que es exactamente complementario. Adicionalmente, los fragmentos de ARNdc de pequeño tamaño (< 30 nucleótidos) mediadores de la interferencia parecen no inducir la maquinaria de activación del IFN.
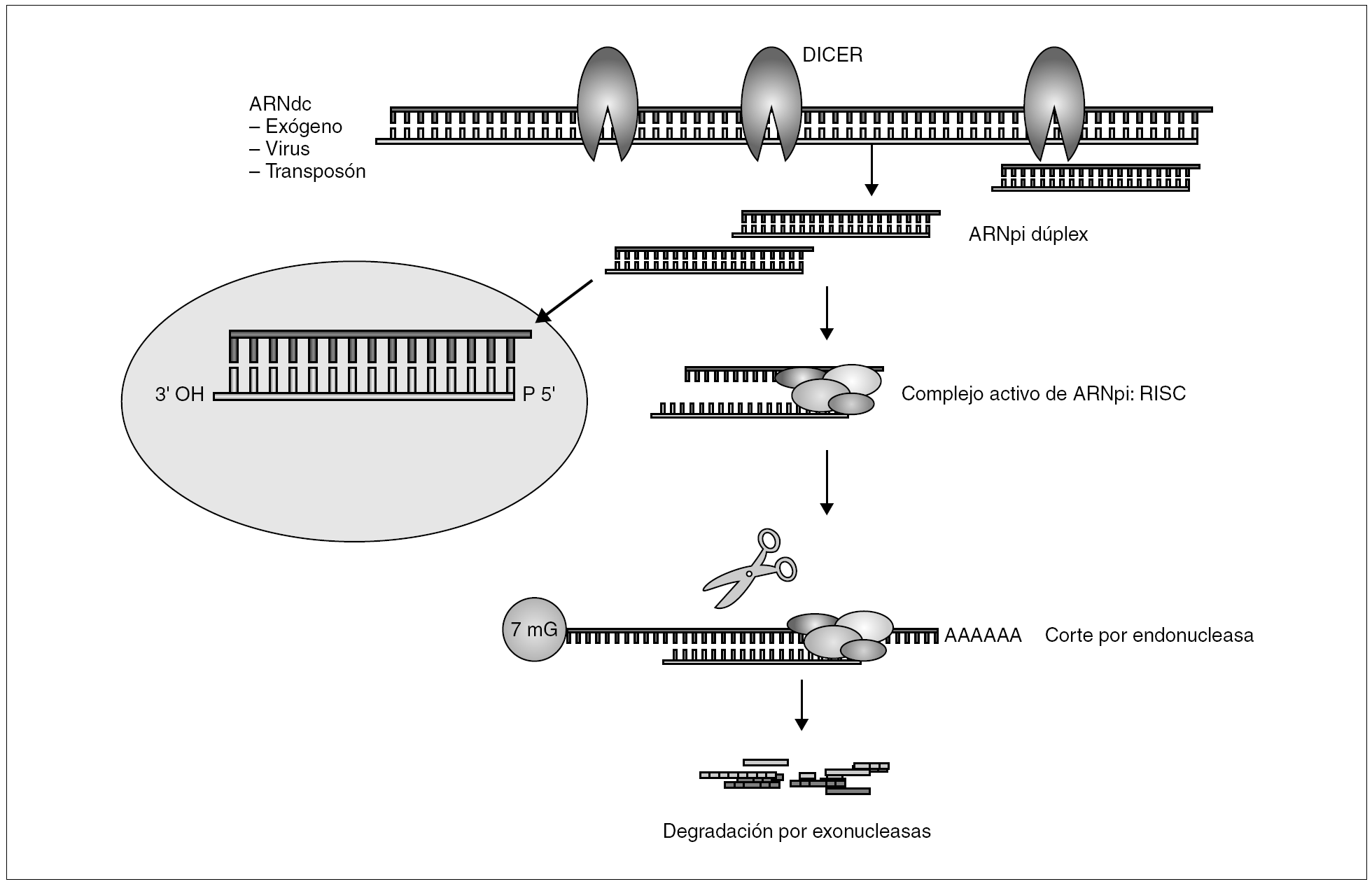
El mecanismo general de la producción de interferencia mediada por ARN se describe en la figura 2. La presencia de ARNdc intracelular activa una endonucleasa específica de ARN de doble cadena denominada DICER. Esta enzima corta el ARNdc en fragmentos de 21-23 nucleótidos con extremos 3´ libres que constituye el auténtico mediador del proceso de silenciamiento secuencia-específico52. A continuación los fragmentos de ARNpi forman un complejo activo con una serie de nucleasas y helicasas denominado RISC (RNA-induced silencing complex)53. Durante el ensamblaje de RISC, muy probablemente una sola de las hebras es capaz de desencadenar interferencia54. Utilizando esta hebra como guía, RISC se une al ARNm homólogo y cataliza el corte de la secuencia, por una ARNsa distinta de DICER, a una corta distancia del sitio de unión55. Los fragmentos de ARN son posteriormente degradados por exonucleasas celulares, completando así el proceso de silenciamiento. En algunos organismos, C. elegans y plantas, se ha propuesto la existencia de un ciclo de amplificación de silenciamiento, a partir de ARNpi secundario generado por DICER, sobre nuevo ARNdc sintetizado por una ARN polimerasa dependiente de ARN (RdRP). Esta RdRP utiliza como molde el ARNm y como primer una de las hebras de los fragmentos de ARNpi56. Este mecanismo de amplificación se ha relacionado con la respuesta sistémica que exhibe el ARNi en plantas y C. elegans donde el silenciamiento se extiende a zonas lejanas e incluso se puede transmitir a la progenie49,57. Este efecto sistémico no se ha confirmado en vertebrados, probablemente por la ausencia de RdRP. El fenómeno de interferencia mediado por ARN es un proceso común a la mayor parte de las células eucarióticas (no existe en bacterias) y parece un antiguo mecanismo defensivo que antecede a la separación evolutiva entre animales y plantas. Sin embargo, el mecanismo básico de interferencia ha podido ir desarrollándose en distintos sentidos. En plantas cumple una función fundamentalmente defensiva frente a virus y tiroides, en vertebrados superiores y seres humanos parece tener una función más bien reguladora de la expresión genética. Esto se puso en evidencia con el descubrimiento de algunos genes cuyo producto no era una proteína, sino fragmentos cortos de ARN con interesantes propiedades reguladoras llamados microARN (miARN)58.
Figura 2. Mecanismo general de funcionamiento de ARNi.
La maquinaria de silenciamiento parece estar conservada en mamíferos, como indican los experimentos de silenciamiento al introducir ARNpi sintético dentro de la célula; sin embargo, no tenemos pruebas que el fenómeno de interferencia tenga un importante papel antiviral en la infección natural, tal como sucede en plantas. Es posible que la inmunidad adaptativa basada en el reconocimiento de estructuras de proteínas haya podido suplantar al mecanismo de interferencia. Es así mismo llamativo que el ARNi funcione como sistema defensivo en plantas e invertebrados que precisamente carecen de inmunidad adaptativa.
Aplicaciones del ARNi: ¿cómo se utiliza?
La forma más sencilla de introducir ARNi dentro de la célula es mediante un procedimiento relativamente sencillo de transfección utilizando ARN sintético. La mayor parte de los experimentos realizados hasta el momento han utilizado esta estrategia con células en cultivo, obteniendo resultados consistentes y reproducibles. Es también posible introducir dúplex de ARN sintético directamente en algunos organismos vivos como sucede en C. elegans59,60, en el que el fenómeno de interferencia se consigue por "ingestión", simplemente añadiendo el ARNpi en el medio de cultivo49. En vertebrados parece algo más complicado: utilizando grandes cantidades de ARN sintético es posible introducir ARNpi en ratones por vía intravenosa a través de un sistema de inyección hidrodinámica61,62. El ARNpi aplicado por este mecanismo en el ratón se localiza rápidamente dentro de células hepáticas y parece tener una sorprendente estabilidad de al menos 7-10 días62. Los inconvenientes de esta estrategia de administración directa son el elevado coste del ARN sintético (a diferencia del ADN) y sobre todo la duración del silenciamiento que lógicamente es transitoria mientras se mantenga la presencia del dúplex de ARN intracelular. Una alternativa para conseguir más estabilidad es utilizar uno de los vectores genéticos, en forma de plásmidos, diseñados para expresar ARNdc. Por último, se ha demostrado que es posible generar vectores basados en retrovirus y lentivirus recombinantes que pueden integrar en la célula diana un casete que exprese ARNpi63-65. Este sistema, al integrar el provirus en el genoma celular, permite la expresión estable de ARNpi y sería por tanto uno de los más atractivos para aplicaciones terapéuticas.
Aplicaciones terapéuticas del RNAi. La prueba de concepto
Una de las aplicaciones más espectaculares con ARNi ha sido la realizada por el grupo de Judy Lieberman sobre un modelo de hepatitis fulminante en ratón inducida por Fas62. La administración de un anticuerpo agonista de Fas produce en ratón la muerte por apoptosis hepática masiva Fas-dependiente en 2-3 días. Utilizando ARNpi sintético y un sistema de inyección intravenosa hidrodinámico, que produce un incremento fugaz de la presión sanguínea66,67 consiguieron proteger a los ratones a los que se inyectó ARNpi específico del ARN mensajero de Fas. Los ratones a los que se inyectó ARNpi inespecífico o modificaciones del ARNpi de Fas no fueron protegidos, demostrando así la especificidad del proceso. La prueba de concepto de que el ARNi puede curar parece establecida, sin embargo existen todavía importantes reservas sobre las cantidades de ARN sintético que serían necesarias y la posible aplicación del sistema hidrodinámico en humanos que exigiría la infusión intravenosa de más de un litro de fluido en pocos segundos66,67.
El sistema inmunitario del genoma. ARNi como antiviral
Con relación al posible papel del ARNi como antiviral en seres humanos existen todavía algunos puntos que deben ser mejor comprendidos para valorar el futuro de esta herramienta. ¿Se produce ARNpi durante una infección natural? aunque como hemos visto el ARNi en plantas se ha desarrollado evolutivamente, con toda probabilidad, como mecanismo defensivo frente a virus y transposones, todavía está por demostrar que en vertebrados el ARNi forme parte de la respuesta natural frente a la infección por virus68. Perece que la expresión de DICER puede ser muy baja en células diferenciadas y sólo en algunas células embrionarias se detectan elevadas concentraciones de DICER y se conservaría, por lo tanto, la función natural antiviral del ARNi69. Por otra parte, tampoco se han podido identificar genes virales cuya función sea evitar la acción del ARNi, en contraste con los numerosos ejemplos de virus que dedican muchos de sus genes a dificultar una adecuada respuesta de IFN, presentación de antígenos o respuesta inmunitaria adaptativa70-73. La maquinaria de silenciamiento está sin embargo conservada y la introducción de ARNi por diferentes técnicas se ha demostrado muy efectiva en diferentes modelos in vitro de infección por virus, entre los que se encuentran los virus de la hepatitis C74-77 y B78, papiloma79, polio80, rotavirus81 y otros virus ARN82.
Aunque el entusiasmo por la utilización de ARNi como antiviral es grande, existen algunos problemas que deben tenerse en cuenta. La extraordinaria especificidad del ARNi, que exige una casi perfecta homología entre los 21-23 nucleótidos que establecen la guía del silenciamiento, también puede ser un aspecto particularmente vulnerable a la hora de aplicarlo como herramienta antiviral, existe ya la observación realizada por Gitlin et al80 trabajando con un modelo de ARNi para poliovirus en el que detectaron una variante que escapaba al mecanismo de interferencia. Esta variante presentaba una única sustitución en la zona central diana del ARNi.
El ARNi en la infección por VIH
La infección por VIH ha sido lógicamente uno de los primeros objetivos de aplicación de la tecnología de ARNi. No en vano es una de las infecciones en que se dispone de un conocimiento más preciso de los mecanismos moleculares y celulares implicados en el proceso. En el caso del VIH existen datos que indican que el ARN genómico puede ser inactivado por ARNi antes de la integración83 e incluso se han conseguido evidenciar reducciones significativas del ARN de VIH antes de la primera hora postinfección en células transfectadas con ARNpi sintético frente a regiones de vif y nef84. La posibilidad de impedir la entrada del genoma de VIH en el núcleo de la célula es extraordinariamente importante y este aspecto debe ser ampliamente estudiado en modelos de infección más estables y utilizando células y aislamientos primarios.
Teóricamente, las mismas construcciones de ARNi activas frente al ARN genómico serían activas frente a los correspondientes ARNm que codifican las proteínas estructurales y accesorias del VIH. Existen diferentes trabajos que confirman la utilidad del ARNi para bloquear el mensajero de proteínas accesorias: Rev85, Tat83, Vif84 y estructurales, fundamentalmente CA86,87. Si como parece el ARNi es capaz de unirse y destruir tanto el ARN genómico del VIH, como el ARNm producido por el provirus integrado, nos encontraríamos con un método único con actividad frente a fases tempranas y tardías de la infección84,88.
Por último, se ha demostrado que es posible aplicar el ARNi para impedir la expresión de las moléculas receptoras del virus en la membrana celular. Utilizando ARNpi sintético se ha demostrado un efecto significativo sobre la infección al bloquear la expresión de CD487, CXCR4 y CCR586. La utilización de ARNi frente a productos celulares podría tener ventajas como sugiere un interesante trabajo en el que se estudió la estabilidad de ARNpi sintético en macrófagos humanos. El ARNpi de p24 tuvo una vida media de menos de 7 días en células no infectadas, frente a los más de 15 días de persistencia del dirigido frente a CCR586, mientras que en células infectadas fue comparable, lo que sugiere que la presencia del ARN diana puede ser necesaria para la estabilidad del ARNpi.
La utilidad del ARNi parece confirmarse igualmente en modelos más fisiológicos utilizando células primarias86,89 y modelos estables para la expresión mantenida de ARNpi86. En este último trabajo se ha utilizado un sistema de transducción mediante un lentivirus recombinante que expresa ARNpi anti-CCR5 en linfocitos primarios en los que se consiguió reducir la expresión de CCR5 por un factor de 10. Esto resultó en una disminución por un factor de 3-7 de la infección por VIH90. Igualmente se han comunicado resultados alentadores inhibiendo de forma estable la producción de tat y la expresión de CCR5 en macrófagos primarios mediante un vector lentiviral91.
Se conoce bien la extraordinaria capacidad del VIH para generar variabilidad y escapar de la presión selectiva inducida por la respuesta inmunitaria92,93 y de los fármacos antirretrovirales94, incluyendo las diseñadas para antagonizar la función de los receptores95,96. No es sorprendente que se haya comunicado la aparición de mutantes de escape a la acción del ARNpi, en este caso por una variante de VIH que presentaba una mutación puntual en la zona diana del ARNi, 21 nucleótidos, correspondiente a la región de tat. Será por tanto necesario, para asegurar el éxito de la aplicación terapéutica del ARN de interferencia en la infección por VIH, utilizar la combinación de diferentes factores del virus y de la célula.
Nuestra capacidad para curar, o contribuir a la curación de una enfermedad como el sida con métodos moleculares depende por una parte de la disposición de una herramienta eficaz, y el ARNi parece serlo, pero también será necesario la puesta a punto de sistemas para hacer llegar el ARNi a las células susceptibles de forma eficaz y segura. Hoy somos más conscientes de las posibilidades y también de las limitaciones de un campo que debe avanzar apoyado en el conocimiento científico y en la consecuente búsqueda de soluciones para los problemas presentes.