Los objetivos de este trabajo fueron estudiar la presencia de β-lactamasas de espectro extendido (BLEE), investigar la ubicación de los genes que codifican para esas enzimas y determinar la relación clonal de cepas de Pseudomonas aeruginosa resistentes a la ceftazidima aisladas de pacientes mexicanos con fibrosis quística.
MetodologíaSe determinó el perfil de resistencia a 11 antibióticos (CLSI) y la detección fenotípica de las BLEE siguiendo un método de difusión en discos de papel filtro adaptado para P. aeruginosa. La caracterización de los genes de las BLEE y de integrones se realizó por PCR y secuenciación del ADN, mientras que el análisis de la relación clonal se realizó por PFGE.
ResultadosDe las 148 cepas en estudio, 22 resultaron resistentes a la ceftazidima y el análisis por PCR y secuenciación reveló la presencia del gen blaOXA-141 en 7 cepas, 6 resistentes y una sensible a la ceftazidima. Asimismo, blaGES se detectó en 11 cepas. Los genes intI2 e intI3 no se detectaron por PCR, pero en las 6 cepas resistentes a la ceftazidima se descubrió el gen blaOXA-141 en un integrón de la clase 1. El análisis de la relación clonal de los aislamientos mostró que la mayoría de los pacientes se infectaron a lo largo del periodo de estudio con cepas de P. aeruginosa que presentaron patrones diferentes, principalmente en los individuos que no tenían relación familiar.
ConclusionesEste trabajo demuestra la existencia del gen blaOXA-141 asociado a un integrón de clase 1 en varias cepas de P. aeruginosa, así como de genes blaGES cuya localización y variante están en estudio en el grupo de investigación. Lo anterior, aunado a la diversidad de cepas capaces de infectar a individuos sensibles, sugiere un riesgo de dispersión de las cepas de P. aeruginosa productoras de BLEE entre la población mexicana que padece fibrosis quística
The aims of this research were to study the presence of extended spectrum β-lactamases (ESBL) to investigate the location of the genes encoding these enzymes, and determine the clonal relationship of strains of ceftazidime-resistant Pseudomonas aeruginosa isolated from Mexican patients with cystic fibrosis.
MethodsWe determined the resistance profile to 11 antibiotics (CLSI) and phenotypic ESBL detection following a disk diffusion method adapted for P. aeruginosa. Characterization of ESBL genes and integrons was performed by polymerase chain reaction (PCR) and DNA sequencing, while analysis of the clonal relationship was performed by pulsed field gel electrophoresis (PFGE).
ResultsOf the 148 strains studied, 22 were resistant to ceftazidime, and analysis by PCR and sequencing revealed the presence of the gene blaOXA-141 in 7 strains, 6 of which were resistant and one, susceptible to ceftazidime. In addition, blaGES was detected in 11 strains. intI2 and intI3 genes were not detected by PCR, but in the 6 ceftazidime-resistant strains, the blaOXA-141 gene was determined in a class 1 integron. Analysis of the clonal relationship of isolates showed that the majority of patients were infected during the study period with P. aeruginosa strains that exhibit different patterns, especially in individuals without a familial relationship.
ConclusionsThis report demonstrates the existence of the blaOXA-141 gene associated with a class 1 integron in several strains of P. aeruginosa, as well as blaGES genes, and their location and variants are being studied by our research group. This, combined with the diversity of strains able to infect several susceptible individuals, suggests the risk of spread of P. aeruginosa-strain ESBL producers among Mexican populations with cystic fibrosis.
Pseudomonas aeruginosa es un patógeno oportunista causante de infecciones con altos índices de morbilidad y mortalidad en huéspedes inmunocomprometidos y pacientes con fibrosis quística. La prevalencia de la bacteria en el tracto respiratorio de los pacientes con fibrosis quística es elevada, debido a la rápida adaptación fisiológica y genética del microorganismo al medio ambiente del huésped, por lo que requieren una mayor atención médica, así como el uso obligado de antibióticos1. El tratamiento continuo con antibióticos β-lactámicos y aminoglucósidos es importante para evitar el deterioro pulmonar en estos pacientes, pero actúa como un mecanismo de selección de cepas capaces de resistir a la acción de los antimicrobianos2.
P. aeruginosa presenta diversos mecanismos de resistencia hacia los agentes terapéuticos, y los principales son la producción de β-lactamasas, la presencia de bombas de expresión activa y la pérdida o la disminución de la expresión de la porina OprD3, lo que en conjunto contribuye a la selección de cepas multirresistentes a los antibióticos4. Los genes que codifican para la síntesis de las β-lactamasas de espectro extendido (BLEE) pueden estar localizados a nivel cromosomal, o bien en algún elemento genético como plásmidos conjugativos, transposones, integrones e islas de patogenicidad, facilitando así su rápida diseminación. En este sentido, los integrones participan de manera determinante en la acumulación y la diseminación de los genes de resistencia5.
La adquisición de genes que codifican para la síntesis de BLEE permite a las bacterias contar con uno de los mecanismos principales de resistencia hacia las cefalosporinas de tercera y cuarta generación e incluso a monobactames. Estas enzimas son variadas y con muchos tipos, debido a mutaciones en respuesta a la alta presión del uso de antibióticos6. En las bacterias gramnegativas se han detectado múltiples tipos de BLEE con más de 800 variedades; en cepas clínicas de P. aeruginosa se han determinado los tipos TEM, SHV, CTX-M, GES, VEB, PER y OXA7, y actualmente suman unas 120 variedades8. Tanto OXA-2 (con 6 derivadas) como OXA-10 (con 10 derivadas) se consideran los prototipos con características de BLEE del tipo OXA que se han podido detectar en cepas clínicas9. Actualmente se continúan incorporando resultados que muestran nuevas variantes de las BLEE del tipo OXA en cepas de P. aeruginosa, como la OXA-161 referida por Juan et al. en 200910, la cual confiere resistencia a la ceftazidima. Sin embargo en otras enzimas, como la OXA-141, se puede consultar la secuencia nucleotídica que está depositada en la base de datos provista por Jacoby en la web de nomenclatura de β-lactamasas [http://www.lahey.org/studies/], pero no contamos con publicaciones que proporcionen información adicional. De igual forma, en México no contamos con estudios que detecten la presencia de BLEE en aislados de P. aeruginosa de origen clínico.
Los objetivos de este trabajo fueron estudiar la presencia de BLEE en cepas de P. aeruginosa resistentes a la ceftazidima, investigar la ubicación de los genes que codifican para esas enzimas y determinar la relación clonal entre las cepas aisladas de pacientes mexicanos con fibrosis quística.
MétodosMaterial biológicoSe estudiaron 148 cepas de P. aeruginosa aisladas en el Laboratorio de Bacteriología Experimental del Instituto Nacional de Pediatría del Distrito Federal, México, a partir de aspirados bronquiales de 76 pacientes que presentaban fibrosis quística, durante 2000 y 200111. La colección de cepas se ha mantenido en tubos con caldo soya tripticaseína y glicerol al 15% a –70°C.
La cepa de P. aeruginosa ATCC 27853 se utilizó para establecer el control de calidad de la prueba de sensibilidad con discos de papel filtro, además de no presentar alguna BLEE. Se empleó una cepa de Escherichia coli de origen clínico productora de SHV-5 como control positivo de producción de BLEE (donada por la Dra. Castro-Alarcón, de la Universidad Autónoma de Guerrero, México). La cepa de P. aeruginosa 208 de origen clínico fue usada como control positivo de integrón de clase 1 y de BLEE de la variedad OXA-2 (colección de cepas del laboratorio).
Pruebas de sensibilidadSe evaluó la sensibilidad a 11 antibióticos: aztreonam (ATM), cefepima (FEP), ceftazidima (CAZ), ceftizoxima (ZOX), ciprofloxacino (CIP), imipenem (IPM), meropenem (MEM), netilmicina (NET), norfloxacino (NOR), ofloxacino (OFX) y piperacilina (PIP) (adquiridos de Becton, Dickinson Company, Maryland, EE.UU.) mediante el método de difusión con disco de papel de filtro, siguiendo las recomendaciones del Clinical and Laboratory Standard Institute (CLSI)12.
La determinación de la concentración inhibitoria mínima (CIM) de la ceftazidima (Sigma-Aldrich, St. Louis, Missouri, EE.UU.), se realizó por el método de diluciones seriadas en agar, siguiendo las recomendaciones y criterios de interpretación de resultados del CLSI12.
Detección fenotípica de BLEESe estableció la presencia de BLEE empleando la prueba de sinergismo de doble disco, ceftazidima 30μg y amoxicilina-ácido clavulánico 20/10μg, dispuestos con una separación de 6mm13.
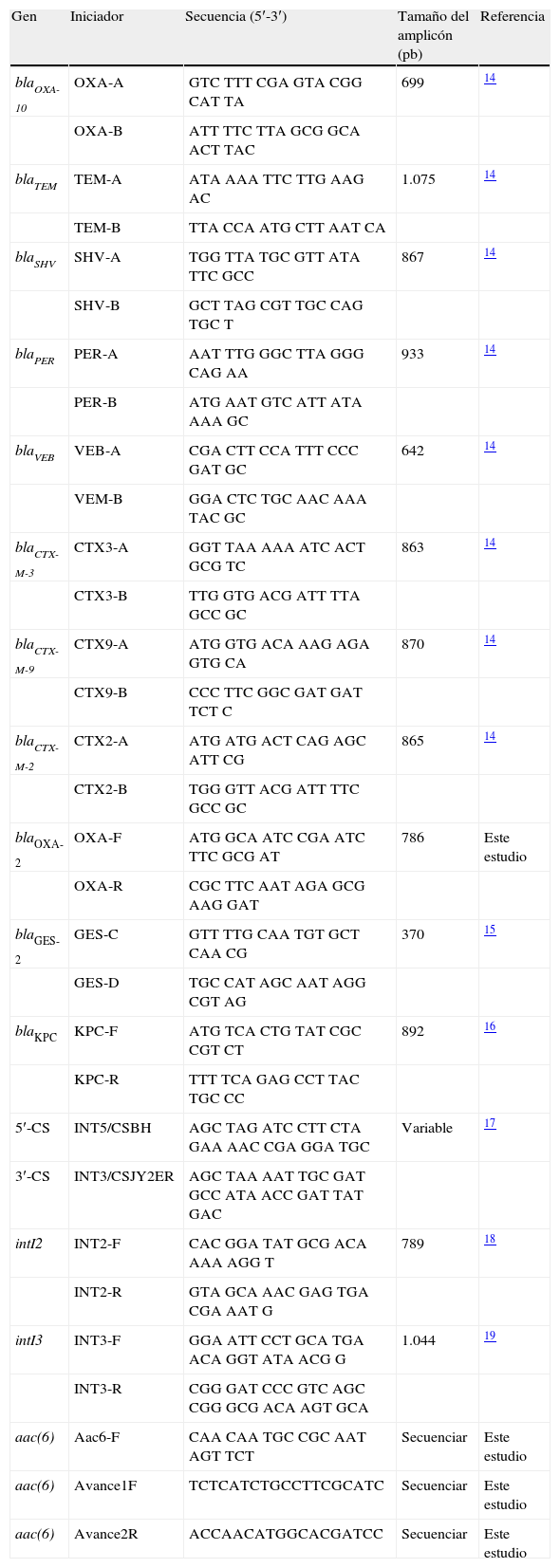
Detección de genes de BLEE mediante PCRPara realizar la caracterización genética por PCR se usó como molde el ADN genómico obtenido con el producto DNeasy Tissue kit (QIAGEN, Hilden, Germany), así como iniciadores específicos para detectar variantes de las BLEE, los cuales fueron previamente reportados por Jiang et al. (2006)14: TEM, SHV, CTX-M, PER, VEB y OXA; Bradford et al. (2004)15: KPC-2 (carbapenemasa que puede degradar ceftazidima), y Weldhagen y Prinsloo (2004)16: GES-2 (tabla 1). Las condiciones de reacción también fueron tomadas de los autores.
Iniciadores empleados para la detección de BLEE y la caracterización de integrones
| Gen | Iniciador | Secuencia (5′-3′) | Tamaño del amplicón (pb) | Referencia |
| blaOXA-10 | OXA-A | GTC TTT CGA GTA CGG CAT TA | 699 | 14 |
| OXA-B | ATT TTC TTA GCG GCA ACT TAC | |||
| blaTEM | TEM-A | ATA AAA TTC TTG AAG AC | 1.075 | 14 |
| TEM-B | TTA CCA ATG CTT AAT CA | |||
| blaSHV | SHV-A | TGG TTA TGC GTT ATA TTC GCC | 867 | 14 |
| SHV-B | GCT TAG CGT TGC CAG TGC T | |||
| blaPER | PER-A | AAT TTG GGC TTA GGG CAG AA | 933 | 14 |
| PER-B | ATG AAT GTC ATT ATA AAA GC | |||
| blaVEB | VEB-A | CGA CTT CCA TTT CCC GAT GC | 642 | 14 |
| VEM-B | GGA CTC TGC AAC AAA TAC GC | |||
| blaCTX-M-3 | CTX3-A | GGT TAA AAA ATC ACT GCG TC | 863 | 14 |
| CTX3-B | TTG GTG ACG ATT TTA GCC GC | |||
| blaCTX-M-9 | CTX9-A | ATG GTG ACA AAG AGA GTG CA | 870 | 14 |
| CTX9-B | CCC TTC GGC GAT GAT TCT C | |||
| blaCTX-M-2 | CTX2-A | ATG ATG ACT CAG AGC ATT CG | 865 | 14 |
| CTX2-B | TGG GTT ACG ATT TTC GCC GC | |||
| blaOXA-2 | OXA-F | ATG GCA ATC CGA ATC TTC GCG AT | 786 | Este estudio |
| OXA-R | CGC TTC AAT AGA GCG AAG GAT | |||
| blaGES-2 | GES-C | GTT TTG CAA TGT GCT CAA CG | 370 | 15 |
| GES-D | TGC CAT AGC AAT AGG CGT AG | |||
| blaKPC | KPC-F | ATG TCA CTG TAT CGC CGT CT | 892 | 16 |
| KPC-R | TTT TCA GAG CCT TAC TGC CC | |||
| 5′-CS | INT5/CSBH | AGC TAG ATC CTT CTA GAA AAC CGA GGA TGC | Variable | 17 |
| 3′-CS | INT3/CSJY2ER | AGC TAA AAT TGC GAT GCC ATA ACC GAT TAT GAC | ||
| intI2 | INT2-F | CAC GGA TAT GCG ACA AAA AGG T | 789 | 18 |
| INT2-R | GTA GCA AAC GAG TGA CGA AAT G | |||
| intI3 | INT3-F | GGA ATT CCT GCA TGA ACA GGT ATA ACG G | 1.044 | 19 |
| INT3-R | CGG GAT CCC GTC AGC CGG GCG ACA AGT GCA | |||
| aac(6) | Aac6-F | CAA CAA TGC CGC AAT AGT TCT | Secuenciar | Este estudio |
| aac(6) | Avance1F | TCTCATCTGCCTTCGCATC | Secuenciar | Este estudio |
| aac(6) | Avance2R | ACCAACATGGCACGATCC | Secuenciar | Este estudio |
Se realizó la detección de integrones en todas las cepas de estudio mediante la técnica de PCR empleando las condiciones de reacción recomendada por Yatsuyanagi et al. (2004)17 para la clase 1, por Ploy et al. (2000)18 para la clase 2 y por Collis et al. (2002)19 para la clase 3 (tabla 1). La caracterización de los integrones se realizó mediante PCR y secuenciación en avanzada, diseñándose iniciadores en cada avance, tomando como molde las secuencias parciales de los amplicones que se obtenían durante este proceso hasta completar la región interna del integrón. El diseño de los iniciadores se llevó a cabo empleando el programa Oligo-Perfect (Invitrogen) (tabla 1).
Purificación, secuenciación y análisis bioinformático de los ampliconesLa purificación de los productos de PCR se efectuó con el sistema comercial DNAclean & concentrator-5™ (Zymo Research, Orange, California, EE.UU.). La secuenciación de los amplicones obtenidos se realizó con el sistema de Big Dye Terminador versión 3.1 (Cycle Sequencing Kit, Applied Biosystems Foster City, California, EE.UU.) y un secuenciador automático ABIPRISM 3100 (Applied Biosystems). Cada uno de los amplificados fue sometido a un análisis con el programa BLASTn para corroborar la identidad, y posteriormente fueron comparados con el programa ClustalX 2.1.
Identificación de los patrones clonales de las cepas en estudio mediante electroforesis en campos pulsados (PFGE)Se siguió el procedimiento operativo estándar para PFGE de Salmonella, establecido por el CDC en la página del PulseNet (http://www.cdc.gov/pulsenet/protocols.htm), con ligeras modificaciones, utilizando la enzima BcuI (SpeI) (Fermentas, Lituania) y el equipo CHEF Mapper® XA (Bio-Rad). La interpretación de los patrones de restricción obtenidos se realizó con base en los criterios de Tenover et al20,21.
ResultadosSensibilidad antimicrobiana y fenotipo BLEEDe las 148 cepas de P. aeruginosa incluidas en este estudio, 64 (43,2%) fueron sensibles y 84 (56,8%) fueron resistentes al menos a uno y hasta a 8 de los 11 antibióticos probados, detectándose resistencia principalmente a los antibióticos ceftizoxima (52%), ceftazidima (14,9%), piperacilina (10,8%), imipenem (8,8%) y ofloxacino (8,8%). Se observó que las 22 cepas resistentes a la ceftazidima presentaron resistencia a uno y hasta a 7 antibióticos más (tabla 2).
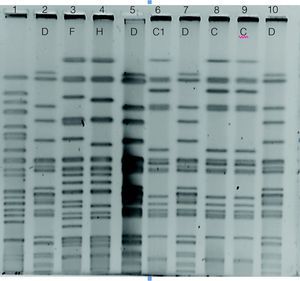
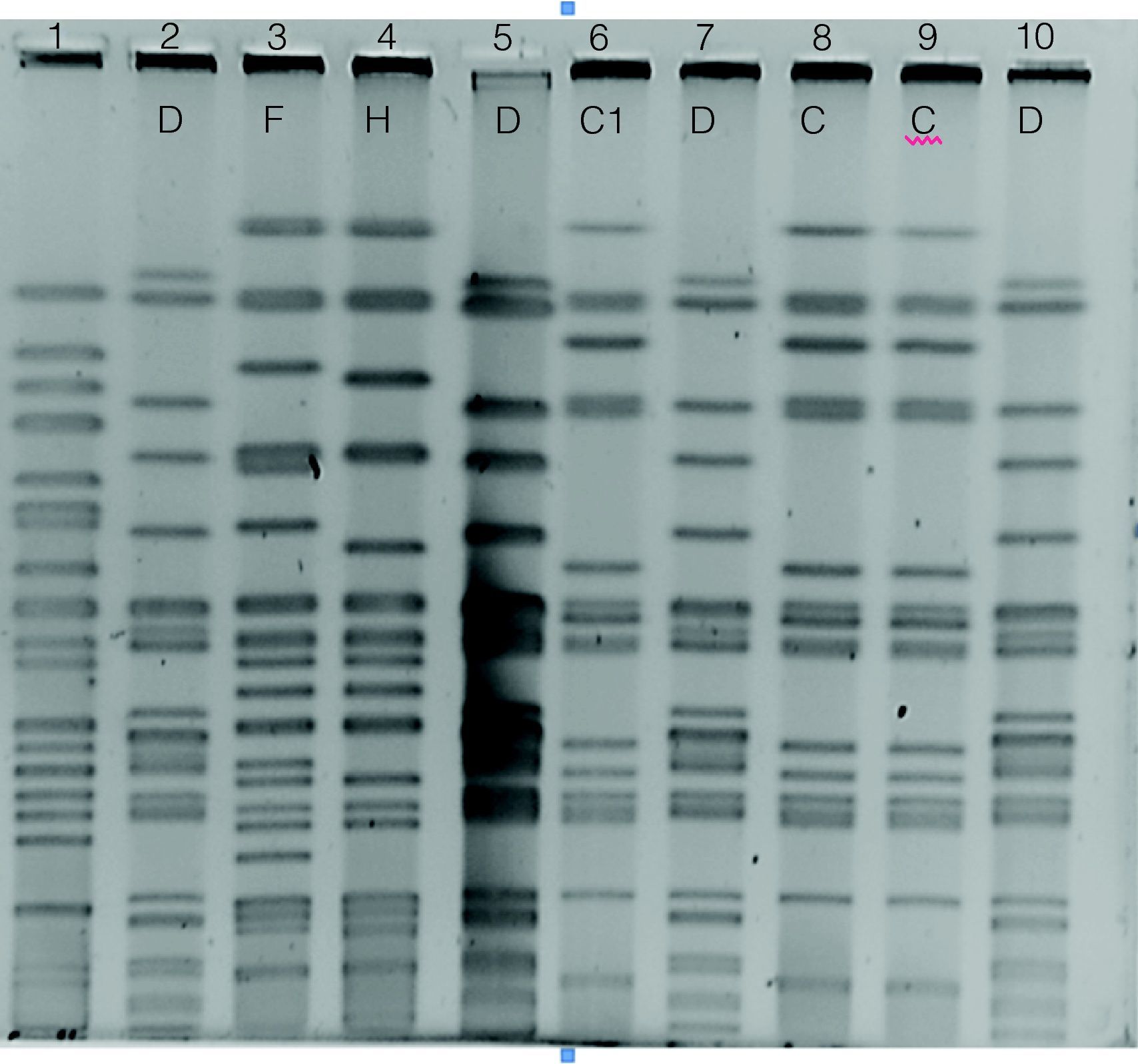
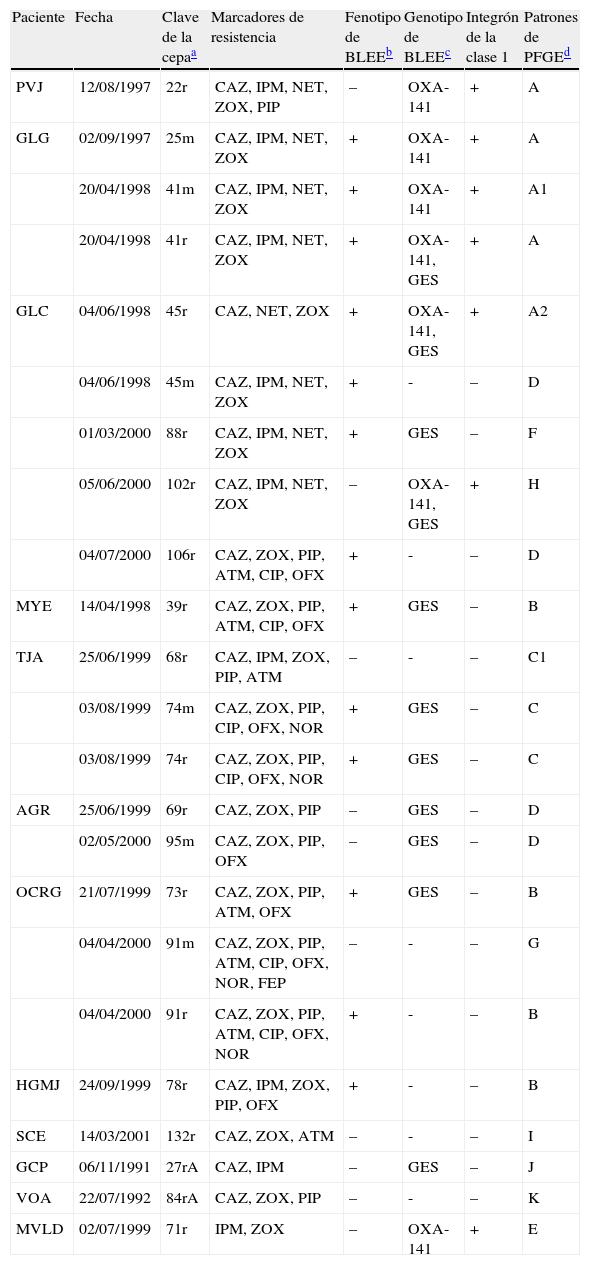
Características fenotípicas y genotípicas de las cepas de Pseudomonas aeruginosa aisladas de pacientes con fibrosis quística
| Paciente | Fecha | Clave de la cepaa | Marcadores de resistencia | Fenotipo de BLEEb | Genotipo de BLEEc | Integrón de la clase 1 | Patrones de PFGEd |
| PVJ | 12/08/1997 | 22r | CAZ, IPM, NET, ZOX, PIP | – | OXA-141 | + | A |
| GLG | 02/09/1997 | 25m | CAZ, IPM, NET, ZOX | + | OXA-141 | + | A |
| 20/04/1998 | 41m | CAZ, IPM, NET, ZOX | + | OXA-141 | + | A1 | |
| 20/04/1998 | 41r | CAZ, IPM, NET, ZOX | + | OXA-141, GES | + | A | |
| GLC | 04/06/1998 | 45r | CAZ, NET, ZOX | + | OXA-141, GES | + | A2 |
| 04/06/1998 | 45m | CAZ, IPM, NET, ZOX | + | - | – | D | |
| 01/03/2000 | 88r | CAZ, IPM, NET, ZOX | + | GES | – | F | |
| 05/06/2000 | 102r | CAZ, IPM, NET, ZOX | – | OXA-141, GES | + | H | |
| 04/07/2000 | 106r | CAZ, ZOX, PIP, ATM, CIP, OFX | + | - | – | D | |
| MYE | 14/04/1998 | 39r | CAZ, ZOX, PIP, ATM, CIP, OFX | + | GES | – | B |
| TJA | 25/06/1999 | 68r | CAZ, IPM, ZOX, PIP, ATM | – | - | – | C1 |
| 03/08/1999 | 74m | CAZ, ZOX, PIP, CIP, OFX, NOR | + | GES | – | C | |
| 03/08/1999 | 74r | CAZ, ZOX, PIP, CIP, OFX, NOR | + | GES | – | C | |
| AGR | 25/06/1999 | 69r | CAZ, ZOX, PIP | – | GES | – | D |
| 02/05/2000 | 95m | CAZ, ZOX, PIP, OFX | – | GES | – | D | |
| OCRG | 21/07/1999 | 73r | CAZ, ZOX, PIP, ATM, OFX | + | GES | – | B |
| 04/04/2000 | 91m | CAZ, ZOX, PIP, ATM, CIP, OFX, NOR, FEP | – | - | – | G | |
| 04/04/2000 | 91r | CAZ, ZOX, PIP, ATM, CIP, OFX, NOR | + | - | – | B | |
| HGMJ | 24/09/1999 | 78r | CAZ, IPM, ZOX, PIP, OFX | + | - | – | B |
| SCE | 14/03/2001 | 132r | CAZ, ZOX, ATM | – | - | – | I |
| GCP | 06/11/1991 | 27rA | CAZ, IPM | – | GES | – | J |
| VOA | 22/07/1992 | 84rA | CAZ, ZOX, PIP | – | - | – | K |
| MVLD | 02/07/1999 | 71r | IPM, ZOX | – | OXA-141 | + | E |
ATM: aztreonam; CAZ: ceftazidima; CIP: ciprofloxacino; FEP: cefepima; IPM: imipenem; NET: netilmicina; NOR: norfloxacino; OFX: ofloxacino; PIP: piperacilina; ZOX: ceftizoxima.
Posteriormente se realizó un análisis de las 22 cepas que resultaron resistentes a la ceftazidima, que fueron recuperadas de 11 pacientes seguidos a lo largo de su tratamiento en el INP. En el paciente 3 se aislaron cinco cepas durante el periodo de atención de su enfermedad. Los valores de la CIM para las cepas fueron de 32-256μg/ml. En 13/22 (59,1%) de las cepas resistentes a la ceftazidima se observó que la prueba de sinergismo de doble disco en la que se usaron discos de amoxicilina-ácido clavulánico y ceftazidima con una separación de 6mm, produjo una sinergia discreta (diferente al resultado franco que presenta la enterobacteria utilizada como testigo), lo que sugirió la presencia de BLEE (tabla 2).
Identificación genotípica de BLEEEn las 148 cepas en estudio se realizó la búsqueda de los genes más frecuentes que codifican para BLEE en P. aeruginosa, empleando los iniciadores indicados en la tabla 1. No se detectó la presencia de BLEE de los tipos TEM, SHV, PER, VEB, CTX-2, CTX-3, CTX-9 y OXA-10. Sin embargo, con los iniciadores dirigidos al gen blaOXA-2 se detectó la presencia de la BLEE OXA-2 en 7/148 cepas de estudio, las cuales, con excepción de una cepa, resultaron resistentes a la ceftazidima, además de provenientes de 4 pacientes. El análisis de cada una de las secuencias de los 7 amplificados reveló una identidad del 100% con OXA-141, del 99% con las variantes OXA-2, 15, 34, 143 y 144, y del 90% con la variante OXA-3. El alto porcentaje de identidad entre las variantes 15, 34, 141, 143 y 144 con la variante OXA-2 se debe a que son derivadas de esta última. Posteriormente, se corroboró la variante OXA presente en las cepas de estudio mediante alineamientos de las secuencias obtenidas para el gen blaOXA-2 en este estudio y las secuencias disponibles en el servidor NCBI para las variantes de blaOXA-(2, 3, 15, 34, 141, 144 y 161), códigos de acceso en GenBank: FJ855130, L07945, U63835, AF350424, EF552405, FJ617207, GQ202693, respectivamente. Empleando el programa Chromas Pro 1.5 (Technelysium Pty Ltd.) y ClustalX2.1, encontramos que las siete cepas de P. aeruginosa mostraron una BLEE del tipo y variante OXA-141, caracterizada por el cambio nucleotídico de G162S (posición tomada con base en la secuencia de la variante OXA-2) (tabla 2).
Para entender el resultado fenotípico con ausencia de un gen correspondiente en las cepas resistentes a la ceftazidima se realizó la búsqueda de genes menos frecuentes que codifican para β-lactamasas en P. aeruginosa, empleando los iniciadores indicados en la tabla 1. No se detectó la presencia de la β-lactamasa del tipo KPC; sin embargo, con los iniciadores dirigidos al gen blaGES se detectó un amplicón del tamaño esperado en 11 cepas, incluso en 3 que presentaron también el gen blaOXA-141 (tabla 2).
Caracterización de los integrones detectadosEn ninguna de las 148 cepas se obtuvo una PCR positiva para la detección de los genes intI2 e intI3. Únicamente se logró amplificar el gen de la integrasa de clase 1 (intI1) en las 7 cepas que presentan la BLEE del tipo OXA-141 (tabla 2).
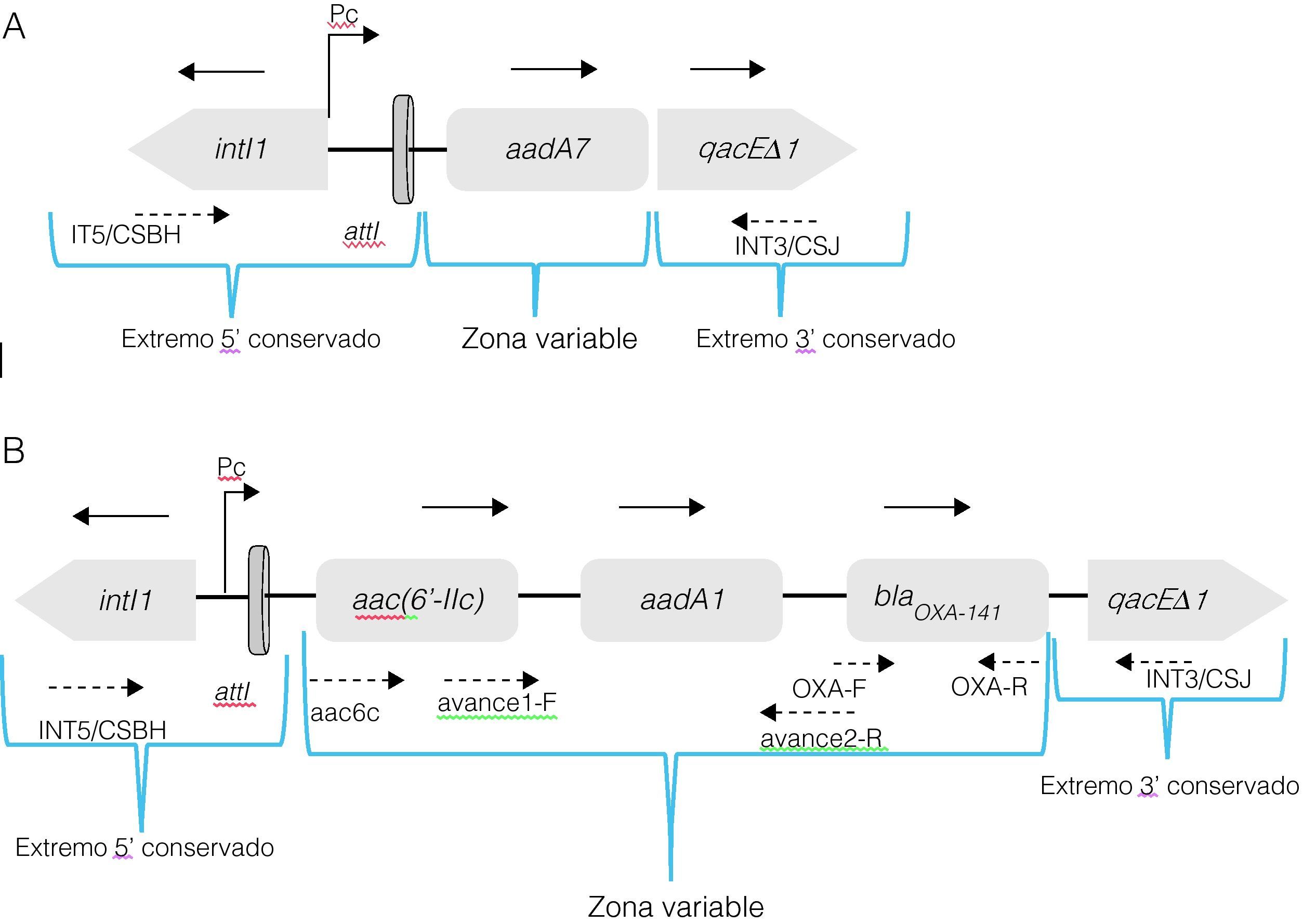
En las 6 cepas resistentes a la ceftazidima (22r, 25m, 41m, 41r, 45r y 102r) se obtuvieron amplificados del mismo tamaño de ∼3,5kb en la región que abarca de la región conservada de la IntI1 a la región variable 3′ que contiene a los casetes de resistencia, mientras que la cepa sensible a la ceftazidima (71r) generó un fragmento de ∼1,5kb (fig. 1). La estructura del integrón clase 1 de la cepa 71r mostró un solo casete, el aadA7, que codifica para una 6-adenil transferasa, pero no se ubicó en este el casete que codifica para blaOXA-141 y blaGES. Tomando como molde las secuencias parciales de los integrones detectados, se llevó a cabo el diseño de iniciadores para realizar PCR y secuenciaciones en avanzada, determinando de esta forma la zona variable de los integrones de clase 1 de mayor tamaño. Las regiones variables de todos estos integrones están constituidas por el gen aac(6′-IIc), que se relaciona con la resistencia a aminoglucósidos, seguido río abajo por aadA1, que también confiere resistencia a los aminoglucósidos y finalmente blaOXA-141, pero en ningún integrón se detectó la presencia del gen blaGES (fig. 2).
Estructura esquemática (no a escala) de los integrones de clase 1 caracterizados en cepas de Pseudomonas aeruginosa aisladas de pacientes con fibrosis quística. En A) se representa la organización del integrón de ∼1,5kpb de la cepa 71r de P. aeruginosa, que presenta identidad con intI1 [integrasa], aadA7 (GenBank número de acceso: DQ899757) [resistencia a aminoglucósidos] y qacΔE1 [gen que codifica la resistencia a las sales cuaternarias de amonio]. En B) se representa el integrón de ∼3,5kpb conservado en las 6 cepas de P. aeruginosa, que presentando identidad con intI1 [integrasa], aac(6′-IIc) (GenBank número de acceso: AF162771) [resistencia a aminoglucósidos], aadA1 (GenBank número de acceso: HQ454494) [resistencia a aminoglucósidos], blaOXA-141 (GenBank número de acceso: EF552405) [β-lactamasa de espectro extendido] y qacΔE1 [gen que codifica la resistencia a las sales cuaternarias de amonio]. Las flechas completas indican la orientación de la transcripción. Las flechas punteadas muestran los iniciadores empleados.
Para establecer la relación epidemiológica de las 22 cepas resistentes a la ceftazidima y con respecto a las 7 cepas que presentan el gen blaOXA-141, se realizó un análisis de PFGE. Se detectaron 11 patrones clonales designados de la A-K (fig. 2); además, el análisis de las bandas obtenidas en las cepas con el patrón A permitió detectar dos subtipos, A1 y A2. Encontramos que cepas aisladas de dos hermanos presentaron patrones clonales relacionados, independientemente del fenotipo bacteriano en primer aislamiento (mucoide o no mucoide) y de la fecha de aislamiento (cepas 25m, 41m y 41r del paciente GLG y cepa 45r del paciente GLC). También encontramos que una cepa aislada de otro paciente sin relación familiar con los anteriores (cepa 22r del paciente PVJ) mostró el mismo patrón de PFGE. En el caso del paciente GLC se pudo establecer la presencia de 5 clones de P. aeruginosa diferentes (A2, D, F, H y G) durante el tiempo de estudio; además de compartir el tipo clonal D con el paciente AGR, con quien no tiene relación familiar. De igual manera, se observó que algunos pacientes únicamente estuvieron infectados por un solo clon de P. aeruginosa, el cual no se presentó en algún otro paciente. La cepa tipo B fue identificada en 3 pacientes sin relación familiar (tabla 2).
DiscusiónLos pacientes con fibrosis quística presentan exacerbaciones respiratorias, lo que hace necesario un tratamiento con antibióticos para restaurar las funciones básicas de los pulmones y así evitar complicaciones respiratorias. El tratamiento convencional incluye una combinación de antibióticos antiseudomonas, empleando un antibiótico β-lactámico como piperacilina-tazobactam, ceftazidima, cefepima, meropenem, imipenem o aztreonam, así como un aminoglucósido, que puede ser amikacina, gentamicina o tobramicina22. Los pacientes de este estudio recibieron tratamientos a base de ceftazidima, tobramicina, amikacina o combinación de ceftazidima y alguno de los aminoglucósidos, lo que debió favorecer en ellos la selección de cepas resistentes a los β-lactámicos y a los aminoglucósidos. Algunas cepas demostraron resistencia al imipenem, lo que no se debe a una presión selectiva, ya que según los expedientes clínicos y los protocolos establecidos en el nosocomio, los pacientes no recibieron ese carbapeneme para su tratamiento. En P. aeruginosa un mecanismo común de resistencia a la ceftazidima corresponde a la producción de BLEE y carbapenemasas que actúan contra las cefalosporinas y/o contra los carbapenemes23, lo que ha quedado documentado en reportes de brotes causados por cepas de P. aeruginosa que producen BLEE del tipo OXA24.
La detección en las enterobacterias de BLEE por métodos fenotípicos es fácil, pero en el caso de P. aeruginosa esto no resulta así, ya que las cepas pueden presentar varios mecanismos alternos de resistencia, como son la sobreexpresión de la β-lactamasa AmpC y/o de las bombas de expresión activa, lo que puede enmascarar la inhibición realizada por el ácido clavulánico sobre la BLEE25. En este estudio eso pudo suceder, ya que en varias cepas que se demostró la resistencia a la ceftazidima no se obtuvo un resultado positivo en la detección fenotípica de BLEE, pero se logró detectar la presencia de los genes blaOXA-141 y blaGES.
La adquisición de los genes blaOXA se ha asociado con elementos genéticos como los integrones de clase 1, transposones y secuencias de inserción26. Este estudio es el primer informe en México de la identificación y caracterización de cepas clínicas de P. aeruginosa productoras de la BLEE OXA-141, y se observa una relación directa entre la presencia del gen OXA-141 e integrón de clase 1, hallazgo que concuerda con el de Laudy et al. en 2007 en Polonia [http://www.lahey.org/studies/]. Adicionalmente, la organización genética de los 6 integrones resultó ser la misma; este hecho es muy interesante, ya que de los 3 pacientes en los que se detectaron las cepas con integrones, solo 2 fueron convivientes, lo que sugiere que se pudo haber dado un cruce de cepas con el tercer individuo al asistir al hospital a recibir atención médica. Aún no hemos logrado esclarecer la localización del gen blaOXA-141 detectado en la cepa 71r de nuestro estudio, ya que no se encuentra dentro de la zona variable del integrón de clase 1 hallado, y esto es de gran interés debido a la ausencia de resistencia a β-lactámicos de espectro extendido y no así de carbapenemes. Todas las cepas blaOXA-141 presentaron resistencia a β-lactámicos y la presencia de los casetes aac(6′-II) y aadA1 y aadA7, responsables de la resistencia al aminoglucósido probado, lo que representa un problema terapéutico en nuestra institución, ya que el régimen antimicrobiano para estos pacientes es a base de cefalosporinas de tercera generación en combinación con aminoglucósidos.
De los 76 pacientes incluidos en este proyecto, 4 presentaron siete cepas de P. aeruginosa con los genes blaOXA-141 e integrones de la clase 1. La caracterización genética por PFGE reveló que 5 de las cepas de los pacientes PVJ, GLG y GLC tienen una relación clonal, mientras que el aislamiento del paciente MVLD no presentó relación epidemiológica con ninguna de las cepas en estudio. El paciente GLG presentó 3 aislamientos del patrón A, mientras que su familiar GLC tiene 5 aislamientos pero únicamente uno del tipo A, lo que habla del recambio de cepas que puede darse a lo largo del tiempo en los pacientes con fibrosis quística. En este último se aprecia que el paciente fue infectado con 2 cepas que presentan el mismo integrón de clase 1 y la OXA-141; con todo, el patrón de PFGE de ambas cepas resultó diferente, por lo cual se puede sospechar una transferencia horizontal de información genética intraespecie. En otros pacientes no se detectó la presencia de OXA-141, pero puede participar la β-lactamasa GES detectada, la cual se presentó en cepas de los patrones A, B, C, D, F y H; aunque también puede ser atribuido a la participación de otros mecanismos de resistencia que pudieran presentarse, como bombas de expresión activa e impermeabilidad de la membrana, ya que pueden participar de forma individual o en conjunto en la resistencia a los antibióticos de cepas de P. aeruginosa3. En las cepas en las que fue posible detectar la presencia del gen blaOXA encontramos que se trata de la misma variante, OXA-141, y este hallazgo concuerda con el de Laudy et al. en 2007 en Polonia en un aislado de P. aeruginosa [http://www.lahey.org/studies/other.]; hasta la fecha no se ha descrito dicha enzima en alguna otra parte del mundo (una oxacilinasa de espectro extendido, de resistencia adquirida, reportada en P. aeruginosa y asociada a un integrón de clase 1).
En conclusión, en 6 aislamientos de P. aeruginosa de niños con fibrosis quística se detectó la presencia del gen blaOXA-141 asociado a un integrón de clase 1, siendo el primer reporte de ello a nivel mundial. Así mismo se evidenció la presencia del gen blaGES en 11 de las cepas resistentes a la ceftazidima, siendo este estudio pionero en el reporte de este tipo de BLEE en pacientes con fibrosis quística, lo que alerta de la necesidad de establecer mecanismos permanentes de monitorización de los genes que confieren la resistencia a los antibióticos para frenar la selección y la dispersión de los genes que confieren resistencia.
FinanciaciónM.G.V. y J.E.D.R. recibieron beca y apoyo económico para realizar esta investigación por el CONACyT y el IPN, México. R.M.R.A. y G.A.O. son becarios de los programas COFAA-IPN y EDD-IPN. Esta investigación fue soportada por SIP-IPN-México; claves: 20091358 y 20101151. R.C.J. recibió apoyo del INP-SSA-México con clave 039/2008.
Conflicto de interesesLos autores declaran que no existe ningún conflicto de intereses.





![Estructura esquemática (no a escala) de los integrones de clase 1 caracterizados en cepas de Pseudomonas aeruginosa aisladas de pacientes con fibrosis quística. En A) se representa la organización del integrón de ∼1,5kpb de la cepa 71r de P. aeruginosa, que presenta identidad con intI1 [integrasa], aadA7 (GenBank número de acceso: DQ899757) [resistencia a aminoglucósidos] y qacΔE1 [gen que codifica la resistencia a las sales cuaternarias de amonio]. En B) se representa el integrón de ∼3,5kpb conservado en las 6 cepas de P. aeruginosa, que presentando identidad con intI1 [integrasa], aac(6′-IIc) (GenBank número de acceso: AF162771) [resistencia a aminoglucósidos], aadA1 (GenBank número de acceso: HQ454494) [resistencia a aminoglucósidos], blaOXA-141 (GenBank número de acceso: EF552405) [β-lactamasa de espectro extendido] y qacΔE1 [gen que codifica la resistencia a las sales cuaternarias de amonio]. Las flechas completas indican la orientación de la transcripción. Las flechas punteadas muestran los iniciadores empleados. Estructura esquemática (no a escala) de los integrones de clase 1 caracterizados en cepas de Pseudomonas aeruginosa aisladas de pacientes con fibrosis quística. En A) se representa la organización del integrón de ∼1,5kpb de la cepa 71r de P. aeruginosa, que presenta identidad con intI1 [integrasa], aadA7 (GenBank número de acceso: DQ899757) [resistencia a aminoglucósidos] y qacΔE1 [gen que codifica la resistencia a las sales cuaternarias de amonio]. En B) se representa el integrón de ∼3,5kpb conservado en las 6 cepas de P. aeruginosa, que presentando identidad con intI1 [integrasa], aac(6′-IIc) (GenBank número de acceso: AF162771) [resistencia a aminoglucósidos], aadA1 (GenBank número de acceso: HQ454494) [resistencia a aminoglucósidos], blaOXA-141 (GenBank número de acceso: EF552405) [β-lactamasa de espectro extendido] y qacΔE1 [gen que codifica la resistencia a las sales cuaternarias de amonio]. Las flechas completas indican la orientación de la transcripción. Las flechas punteadas muestran los iniciadores empleados.](https://static.elsevier.es/multimedia/0213005X/0000003000000009/v1_201305090349/S0213005X12000481/v1_201305090349/es/main.assets/thumbnail/gr1.jpeg?xkr=ue/ImdikoIMrsJoerZ+w96p5LBcBpyJTqfwgorxm+Ow=)