Las técnicas moleculares, como PCR múltiples, presentan la ventaja de poder detectar de forma simultánea un mayor número de virus respiratorios que el cultivo celular, y de forma más rápida, en un solo análisis1,2. Aunque el cultivo por shell vial también analiza varios virus de forma simultánea permitiendo detectar coinfecciones, lo hace con una sensibilidad mucho menor3. La RT-PCR permite ampliar las posibilidades diagnósticas para aquellos virus que, por sus características, no son detectables por otros métodos o son de difícil cultivo. En este sentido, la RT-PCR múltiple podría aportar al diagnóstico la detección de rinovirus, que cada día parece más justificado por su incidencia y relación con procesos respiratorios de gravedad más o menos severos en niños y pacientes con EPOC4,5. Además, también permite el diagnóstico de otros virus menos frecuentes, como coronavirus y virus de la gripe C. En el presente estudio se ha evaluado la eficacia diagnóstica de una técnica de PCR múltiple para virus respiratorios, en comparación con técnicas tradicionales de cultivo celular en ámbito hospitalario.
Se trabajaron muestras de frotis faríngeos (FF), lavados nasales (LN) y lavados broncoalveolares (BAL). Todas las muestras se analizaron mediante shell vial como método de referencia, en comparación con una RT-nested PCR múltiple. Mediante shell vial, se estudió la presencia de virus de la gripe A (GA), gripe B (GB), virus respiratorio sincitial (VRS), virus parainfluenza humano (VPIh) y adenovirus (ADV), permitiendo su crecimiento en 48h. Para ello, se utilizaron líneas celulares comerciales (Vircell®, España) y la identificación de cada virus aislado se realizó por tinción con anticuerpos monoclonales (IMAGEN Respiratory Screen, DAKO). La RT-nested PCR múltiple utilizada1,2 consistió en 2 PCR en las que cada una fue diseñada para la detección de un grupo de virus. La primera de ellas contenía los primers para la detección de GA, GB, gripe C (GC), VRS A, VRS B y ADV, y la otra, los de VPIh 1, VPIh 2, VPIh 3, VPIh 4, enterovirus, rinovirus, coronavirus 229E y coronavirus OC43. La extracción de ácidos nucleicos se llevó a cabo por medio del extractor automático EasyMag (Biomerieux, Francia).
Se analizaron 488 muestras respiratorias procedentes de igual número de pacientes, correspondientes a 255 LN, 210 FF y 23 BAL; 180/488 (36,7%) fueron positivas para algún virus respiratorio por cultivo celular y 293/488 (60%) lo fueron para algún virus respiratorio de los 14 detectables por PCR múltiple (p<0,001). Ello supone un aumento del 62,8% en la detección por PCR múltiple con respecto al cultivo celular. Si comparamos sólo los 5 virus detectables por ambas técnicas, 230/488 (47,1%) fueron positivas por PCR múltiple (p<0,001); en este caso, la PCR sólo supone un incremento del 27,8%.
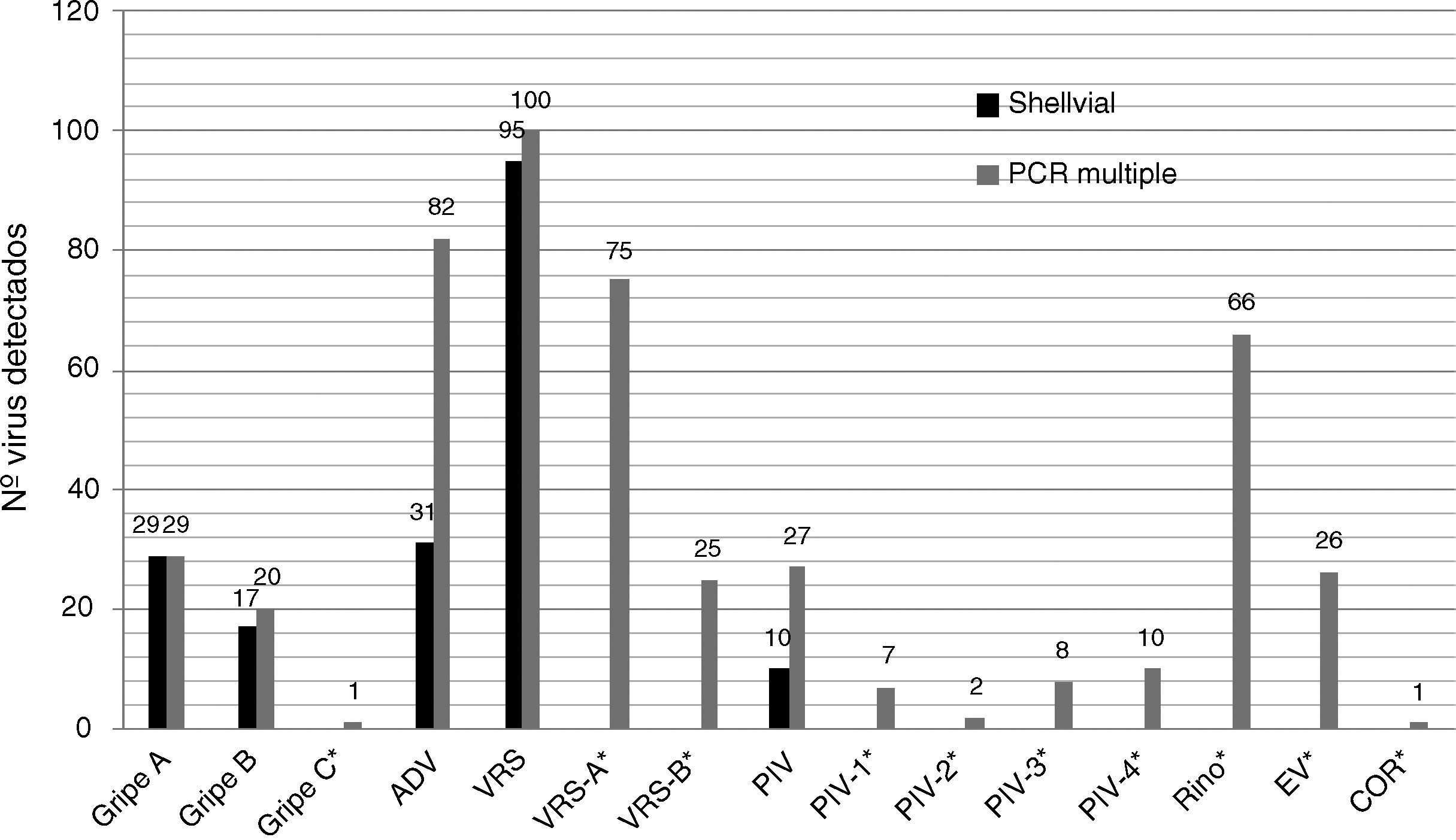
Se detectaron 2 coinfecciones dobles (1% sobre las muestras positivas) y ninguna triple por cultivo celular. Por el contrario, se detectaron 48 coinfecciones dobles (16,4% sobre las positivas) y 5 triples (1,7%) por PCR múltiple. El número total de virus respiratorios diagnosticados mediante aislamiento en cultivo celular fue de 182 y el número de virus respiratorios detectados por PCR fue de 352. Además de los virus diagnosticados por ambas técnicas, se detectaron otros que sólo pudieron ser investigados mediante PCR (fig. 1). En la tabla 1 se muestran los valores de validez de las pruebas diagnósticas considerando al cultivo celular como método de referencia. En este sentido, se aconseja el manejo cautelar de los datos de especificidad, ya que en dicha tabla se consideran falsos positivos a los detectados sólo por PCR, hecho que presuntivamente no se debe a la inespecificidad, sino más bien a la falta de sensibilidad del cultivo celular frente a la PCR.
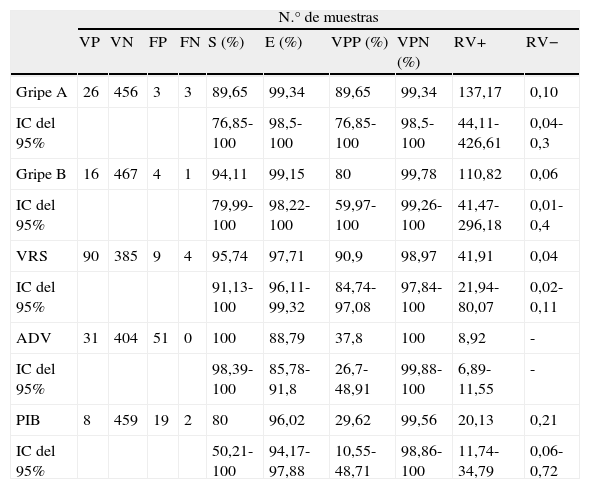
Sensibilidad, especificidad y valores predictivos positivo y negativo de la RT-PCR nested en la detección de los virus respiratorios estudiados en relación con el cultivo celular.
| N.° de muestras | ||||||||||
| VP | VN | FP | FN | S (%) | E (%) | VPP (%) | VPN (%) | RV+ | RV− | |
| Gripe A | 26 | 456 | 3 | 3 | 89,65 | 99,34 | 89,65 | 99,34 | 137,17 | 0,10 |
| IC del 95% | 76,85-100 | 98,5-100 | 76,85-100 | 98,5-100 | 44,11-426,61 | 0,04-0,3 | ||||
| Gripe B | 16 | 467 | 4 | 1 | 94,11 | 99,15 | 80 | 99,78 | 110,82 | 0,06 |
| IC del 95% | 79,99-100 | 98,22-100 | 59,97-100 | 99,26-100 | 41,47-296,18 | 0,01-0,4 | ||||
| VRS | 90 | 385 | 9 | 4 | 95,74 | 97,71 | 90,9 | 98,97 | 41,91 | 0,04 |
| IC del 95% | 91,13-100 | 96,11-99,32 | 84,74-97,08 | 97,84-100 | 21,94-80,07 | 0,02-0,11 | ||||
| ADV | 31 | 404 | 51 | 0 | 100 | 88,79 | 37,8 | 100 | 8,92 | - |
| IC del 95% | 98,39-100 | 85,78-91,8 | 26,7-48,91 | 99,88-100 | 6,89-11,55 | - | ||||
| PIB | 8 | 459 | 19 | 2 | 80 | 96,02 | 29,62 | 99,56 | 20,13 | 0,21 |
| IC del 95% | 50,21-100 | 94,17-97,88 | 10,55-48,71 | 98,86-100 | 11,74-34,79 | 0,06-0,72 | ||||
E: especificidad; FN: falso negativo; FP: falso positivo; IC: intervalo de confianza; RV +: razón de verosimilitud positiva; RV –: razón de verosimilitud negativa; S: sensibilidad; VN: verdadero negativo; VP: verdadero positivo; VPN: valor predictivo negativo; VPP: valor predictivo positivo.
La edad media±desviación estándar en años de los pacientes por tipo de virus detectado correspondió a 1±1,14 para VRS, 4,5±5,91 para gripe A, 6,5±3,42 para gripe B, 6,2±12,04 para ADV, 1,6±2,11 para PIVh, 1,5±3,37 para rinovirus y 3,4±3,44 para enterovirus. En relación con el sexo, en todos los tipos de muestras predominaron los varones sobre las mujeres, con una media global del 60% para varones y el 40% para mujeres (p=0,0046).
El rendimiento en las muestras de LN fue muy superior al resto, tanto en técnicas de shell vial (45,5%) como por PCR (70,6%). No obstante, las muestras de FF también muestran rendimientos aceptables tanto por shell vial (30,5%) como por PCR (53,8%), teniendo en cuenta que se trata de la toma más fácil y menos traumática para el paciente. Sin embargo, el BAL presentó rendimientos mínimos para shell vial (4%) y nulos para PCR, datos de valor limitado por la escasa representación en la serie estudiada (n=23).
En términos globales, casi el doble (36,7% vs 60%) de las muestras positivas se diagnosticaron por PCR en comparación al cultivo celular. Si bien hay que tener en cuenta que, mediante PCR, se aumentó el espectro diagnóstico a 14 virus respiratorios frente a sólo 5 virus investigados por shell vial. Si se comparan solo los 5 virus respiratorios detectables por ambas técnicas, la PCR presentó un 47,1% de muestras positivas, un 27,8% (50/180) más que por shell vial, que sumado a los no diagnosticables por cultivo celular, suponen un 62,8% (113/180) de incremento en detecciones por PCR. Otros estudios comparativos de cultivo por shell vial y técnicas de PCR similares a la ensayada, mostraron incrementos en la detección de virus respiratorios entre el 12 y el 25% mediante PCR6,7, por lo que los resultados obtenidos se encuentran entre los rangos más elevados de rendimiento diagnóstico.
La PCR múltiple detectó 94 virus adicionales no testados por shell vial (66 rinovirus, 26 enterovirus, 1 virus gripal C y 1 coronavirus). Así pues, de estos resultados se puede inferir la importancia clínica que los picornavirus tienen en la etiología de las infecciones respiratorias, con frecuencia infravalorados en la práctica clínica por falta de diagnóstico etiológico específico8, aunque no siempre la sola presencia de algunos de estos virus puede demostrar la infección. Por otra parte, el hallazgo de coinfecciones representó una proporción importante de las muestras, siendo las dobles y triples un 16 y un 2% por PCR, respectivamente, y sólo un 1% las coinfecciones dobles diagnosticas por shell vial. Las coinfecciones por PCR comunicadas por otras series oscilan entre el 2 y el 30%9,10; sin embargo, son escasas las coinfecciones comunicadas por cultivo celular11. En la presente serie, los virus más frecuentes en coinfecciones fueron los VRS, rinovirus y adenovirus; alguno de ellos está presente en más del 80% de las coinfecciones. Las combinaciones más frecuentes fueron VRS-rinovirus (10), VRS-adenovirus (9) y adenovirus-rinovirus (8).
En relación con el sexo, aparecieron mayores porcentajes de varones que de mujeres en cuanto a sintomatología de infección respiratoria vírica y existe una diferencia significativa asociada al sexo, al igual que en otra serie comunicada12. Sin embargo, en otros estudios no se ha podido demostrar dicha diferencia13,14. Cabe apuntar que, a pesar de los resultados, es muy posible que las infecciones respiratorias de origen viral afecten por igual a ambas poblaciones.
En conclusión, el diagnóstico molecular en las infecciones respiratorias virales constituye una herramienta diagnóstica que, además de presentar mayor rapidez, sensibilidad, especificidad y espectro diagnóstico que el cultivo celular, puede permitir el estudio en profundidad de coinfecciones, así como las asociaciones más frecuentes y su epidemiología.