Se presenta el análisis anual de los resultados remitidos durante el año 2017 por los participantes inscritos en el Programa de Control de Calidad de la Sociedad Española de Enfermedades Infecciosas y Microbiología Clínica (SEIMC), que incluye las áreas de bacteriología, serología, micología, parasitología, micobacterias, virología, microbiología molecular y de detección genotípica de mecanismos de resistencia bacteriana. Los resultados obtenidos por los centros participantes resaltan, de nuevo, la adecuada capacitación de la inmensa mayoría de los laboratorios españoles de microbiología clínica, como ya venía sucediendo en los últimos años. Sin embargo, el programa muestra de nuevo que es posible obtener un resultado erróneo, incluso en determinaciones de la mayor trascendencia y en cualquier laboratorio. Una vez más, se destaca la importancia de complementar el control interno que cada laboratorio lleve a cabo con estudios de intercomparación externos, como los que ofrece el Programa SEIMC.
This article provides an analysis of the results obtained in 2017 by the participants inscribed in the External Quality Control Programme of the Spanish Society of Infectious Diseases and Clinical Microbiology (SEIMC), which includes controls for bacteriology, serology, mycology, parasitology, mycobacteria, virology, molecular microbiology, and genotypic bacterial resistance. The results obtained in 2017 confirm the excellent skill and good technical standards found in previous editions. However, the programme again showed that erroneous results can be obtained in any laboratory and even in clinically relevant determinations. Once again, the results of this program highlight the need to implement both internal and external controls, as in the SEIMC programme.
Las distintas determinaciones que se realizan en los laboratorios de microbiología clínica tienen repercusión tanto en el diagnóstico clínico de los pacientes como en el ámbito de la salud pública. Estos laboratorios se enfrentan actualmente a desafíos como la rápida detección de la resistencia bacteriana, para así poder prevenir fallos en el tratamiento antimicrobiano, la identificación de patógenos emergentes y la implementación de nuevas técnicas moleculares, y, además, estos resultados deben ser precoces y con un coste-beneficio adecuado. Para evitar errores que puedan tener consecuencias en los pacientes, los profesionales de microbiología clínica muestran cada vez un creciente interés en asegurar la fiabilidad de los resultados emitidos1,2. Para ello, estos laboratorios deben disponer de controles de calidad, tanto internos como externos, que abarquen todas las fases del proceso analítico. Estos controles de calidad permiten detectar errores, tanto sistemáticos como aleatorios, con la consiguiente posibilidad de introducir, si procede, las medidas correctoras adecuadas3–6.
La participación en programas de intercomparación externa entre diferentes laboratorios permite la obtención de diversos beneficios derivados del análisis conjunto de datos aportados por los centros participantes, así como la detección de errores o inconsistencias atribuibles a algunas metodologías o sistemas, comerciales o no, que puedan ser el punto de partida de estudios más profundos y concluyentes2,6, tal como se observa a lo largo de este artículo. Además, estos programas pueden aprovecharse para instaurar actividades de formación continuada que ayuden en la introducción de medidas correctoras y que repercutan en la mejora continua de la calidad. Esta ha sido una característica definitoria del Programa del Control de Calidad de la Sociedad Española de Enfermedades Infecciosas y Microbiología Clínica (SEIMC) —CCS—7–18 y es coherente con lo indicado en la Norma UNE-EN ISO 1518919, que otorga a la formación una importancia de primer orden. En el presente número extraordinario de la revista de EnfermedadesInfecciosas y Microbiología Clínica (EIMC), además del análisis general de los resultados remitidos por los participantes a lo largo del año 2017 para las áreas de serología, bacteriología trimestral y mensual, micología, parasitología, micobacteriología, virología, microbiología molecular y de detección genotípica de mecanismos de resistencia bacteriana, con sus principales conclusiones y enseñanzas, se presenta una serie de revisiones de los distintos temas sobre los que versaban los controles remitidos en este año. Las áreas de control de calidad de la carga viral de los virus de la inmunodeficiencia humana tipo 1 (VIH-1), de la hepatitis C (VHC) y de la hepatitis B (VHB) se presentan en un documento aparte. Se puede obtener información más detallada en el sitio web del Programa de Control de Calidad SEIMC20.
2Análisis de datos de los controles de serologíaDurante el año 2017 se realizaron 8 envíos de serología (S-1A/17, S-1B/17, S-2A/17, S-2B/17, S-3A/17, S-3B/17, S-4A/17 y S-4B/17) a 183 centros inscritos en esta área. Previamente, para cada uno de los controles, se solicitó a 2 laboratorios con experiencia en serología la realización de estas determinaciones, que se utilizarían posteriormente como valor asignado de comparación y para la emisión de los certificados individuales a cada participante. Algunas de las características y resultados de dichos controles se resumen en la tabla 1.
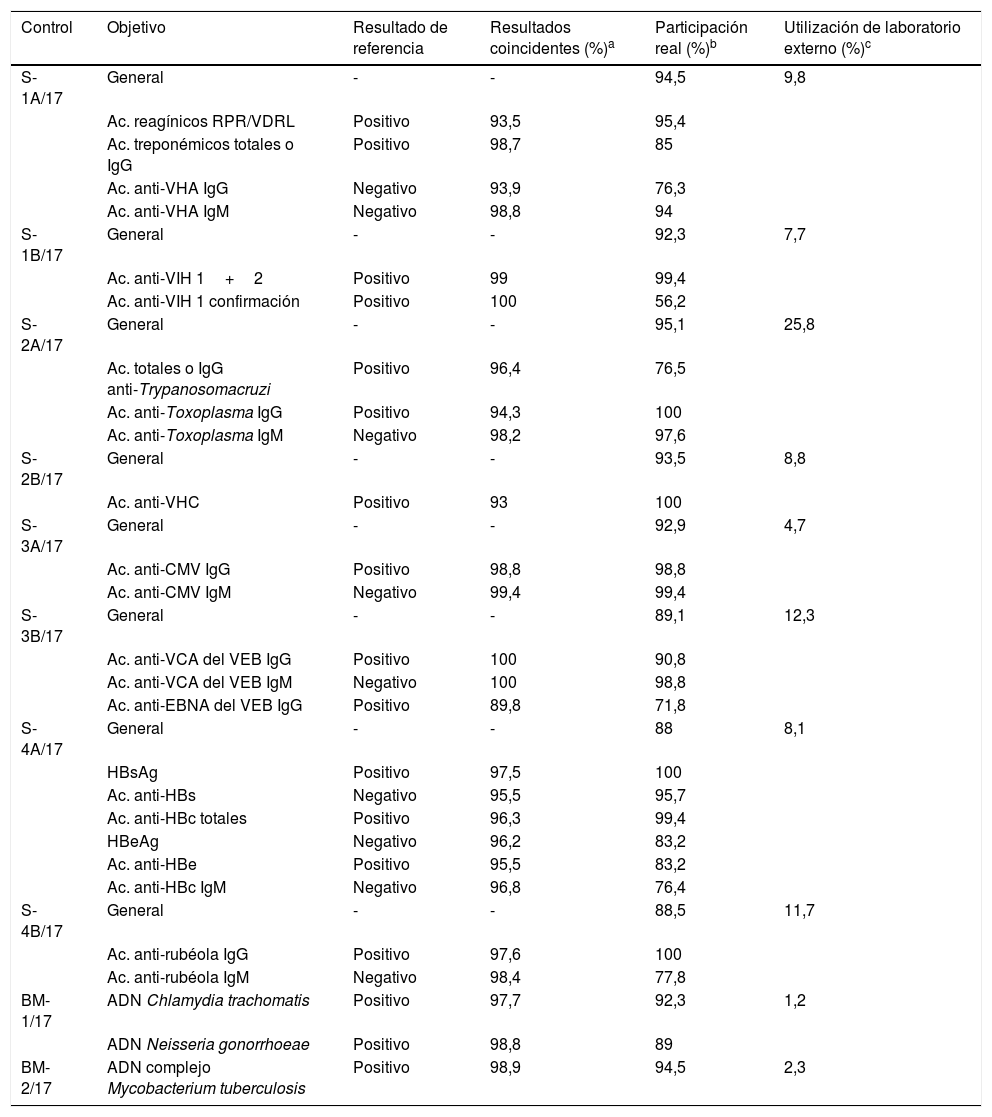
Resumen de los controles de serología y microbiología molecular del año 2017a
| Control | Objetivo | Resultado de referencia | Resultados coincidentes (%)a | Participación real (%)b | Utilización de laboratorio externo (%)c |
|---|---|---|---|---|---|
| S-1A/17 | General | - | - | 94,5 | 9,8 |
| Ac. reagínicos RPR/VDRL | Positivo | 93,5 | 95,4 | ||
| Ac. treponémicos totales o IgG | Positivo | 98,7 | 85 | ||
| Ac. anti-VHA IgG | Negativo | 93,9 | 76,3 | ||
| Ac. anti-VHA IgM | Negativo | 98,8 | 94 | ||
| S-1B/17 | General | - | - | 92,3 | 7,7 |
| Ac. anti-VIH 1+2 | Positivo | 99 | 99,4 | ||
| Ac. anti-VIH 1 confirmación | Positivo | 100 | 56,2 | ||
| S-2A/17 | General | - | - | 95,1 | 25,8 |
| Ac. totales o IgG anti-Trypanosomacruzi | Positivo | 96,4 | 76,5 | ||
| Ac. anti-Toxoplasma IgG | Positivo | 94,3 | 100 | ||
| Ac. anti-Toxoplasma IgM | Negativo | 98,2 | 97,6 | ||
| S-2B/17 | General | - | - | 93,5 | 8,8 |
| Ac. anti-VHC | Positivo | 93 | 100 | ||
| S-3A/17 | General | - | - | 92,9 | 4,7 |
| Ac. anti-CMV IgG | Positivo | 98,8 | 98,8 | ||
| Ac. anti-CMV IgM | Negativo | 99,4 | 99,4 | ||
| S-3B/17 | General | - | - | 89,1 | 12,3 |
| Ac. anti-VCA del VEB IgG | Positivo | 100 | 90,8 | ||
| Ac. anti-VCA del VEB IgM | Negativo | 100 | 98,8 | ||
| Ac. anti-EBNA del VEB IgG | Positivo | 89,8 | 71,8 | ||
| S-4A/17 | General | - | - | 88 | 8,1 |
| HBsAg | Positivo | 97,5 | 100 | ||
| Ac. anti-HBs | Negativo | 95,5 | 95,7 | ||
| Ac. anti-HBc totales | Positivo | 96,3 | 99,4 | ||
| HBeAg | Negativo | 96,2 | 83,2 | ||
| Ac. anti-HBe | Positivo | 95,5 | 83,2 | ||
| Ac. anti-HBc IgM | Negativo | 96,8 | 76,4 | ||
| S-4B/17 | General | - | - | 88,5 | 11,7 |
| Ac. anti-rubéola IgG | Positivo | 97,6 | 100 | ||
| Ac. anti-rubéola IgM | Negativo | 98,4 | 77,8 | ||
| BM-1/17 | ADN Chlamydia trachomatis | Positivo | 97,7 | 92,3 | 1,2 |
| ADN Neisseria gonorrhoeae | Positivo | 98,8 | 89 | ||
| BM-2/17 | ADN complejo Mycobacterium tuberculosis | Positivo | 98,9 | 94,5 | 2,3 |
Ac.: anticuerpos; Ag: antígeno; CMV: citomegalovirus; EBNA: antígeno nuclear del virus de Epstein-Barr; HBeAg: antígeno “e” del virus de la hepatitis B; HBsAg: antígeno de superficie del virus de la hepatitis B; IgG: inmunoglobulina G; IgM: inmunoglobulina M; RPR/VDRL: rapid plasma reagin/venereal disease research laboratory; VCA: antígeno de la cápside viral; VEB: virus de Epstein-Barr; VHA: virus de la hepatitis A; VHC: virus de la hepatitis C; VIH: virus de la inmunodeficiencia humana.
En el control S-1A/17 se solicitó la detección de diversos marcadores serológicos de sífilis y los anticuerpos de tipo inmunoglobulina (Ig)G e IgM frente al virus de la hepatitis A (VHA). Los 2 laboratorios que actuaron de referencia confirmaron la existencia de anticuerpos reagínicos y treponémicos en la muestra control, y resultaron positivas las pruebas de RPR (rapid plasma reagin, con un título de 1/4) y los anticuerpos totales frente a Treponema pallidum. Por el contrario, los anticuerpos anti-VHA de las clases IgG e IgM fueron negativos. En cuanto a los resultados de los participantes, hubo coincidencia general con los de referencia para la detección de estos diversos marcadores, con escasas discrepancias.
En el control S-1B/17 se solicitó la detección de los anticuerpos frente al VIH de los tipos 1 y 2. De acuerdo con el valor asignado, se confirmó que la muestra era positiva para VIH-1. De nuevo, hubo concordancia entre los resultados de los participantes con el valor asignado, con algunas discrepancias ocasionales. Aun así, hay que señalar por su importancia clínica a los 2 centros (1,0%) que informaron un resultado falsamente negativo para el VIH. Por otra parte, cabe destacar que tan solo el 56,2% de los participantes confirmaron el resultado positivo obtenido debido, en parte, a que en bastantes ocasiones realizaban la confirmación en una segunda muestra de suero y así lo hicieron constar en sus comentarios.
El control S-2A/17 versaba acerca de la detección de los anticuerpos frente a Trypanosoma cruzi, así como de los anticuerpos de tipo IgG e IgM frente a Toxoplasma gondii. Los 2 laboratorios de referencia detectaron la presencia de anticuerpos IgG frente a T. cruzi y frente a Toxoplasma, mientras que los anticuerpos anti-Toxoplasma IgM fueron negativos. De nuevo, hubo concordancia entre los resultados de los participantes, con algunas discrepancias ocasionales.
En el control S-2B/17 se requirió a los participantes la detección de los anticuerpos frente al VHC. De acuerdo con el valor asignado, los anticuerpos anti-VHC fueron positivos. Hay que resaltar que, de nuevo, hubo concordancia entre los resultados de los participantes con el valor asignado, si bien con algunas discrepancias ocasionales. Sin embargo, hay que destacar, por su importancia clínica, los 5 centros que obtuvieron un resultado negativo para los anti-VHC y los 8 centros que informaron un resultado indeterminado. Asimismo, tan solo el 40,9% de los participantes confirmaron el resultado positivo obtenido del VHC debido a que, en parte, en bastantes ocasiones los participantes realizaban esta última prueba en una segunda muestra de suero.
El control S-3A/17 versaba acerca de la detección de los anticuerpos de tipo IgG e IgM frente al citomegalovirus (CMV). Sobre la base del valor asignado, los anticuerpos IgG frente al CMV fueron positivos en la muestra control y los de la clase IgM negativos. De nuevo, hubo concordancia entre los resultados de los participantes con el valor asignado, con apenas resultados discrepantes.
En el control S-3B/17 se solicitó la determinación de los anticuerpos de tipo IgG e IgM frente al antígeno de la cápside viral (VCA) y de los anticuerpos de tipo IgG frente al antígeno nuclear del virus de Epstein Barr —VEB— (EBNA). Respecto al valor asignado, los anticuerpos anti-VCA y anti-EBNA de la clase IgG fueron positivos, y los anti-VCA IgM fueron negativos. En cuanto a los resultados de los participantes, hubo coincidencia general con el valor asignado, aunque con algunas discrepancias ocasionales en los anti-EBNA IgG, con relación a un determinado método.
En el control S-4A/17 se requirió la detección de los antígenos de superficie del VHB (HBsAg) y del antígeno “e” del VHB (HBeAg), y de los anticuerpos frente al antígeno de superficie del VHB (anti-HBs), frente al antígeno “e” del VHB (anti-HBe) y frente al antígeno del core del VHB (anti-HBc) del VHB. Los marcadores del VHB de referencia sugerían un patrón de hepatitis crónica en fase no replicativa (o con actividad replicativa mínima), ya que el HBsAg, los anti-HBc y los anti-HBe fueron los 3 positivos, mientras que el resto de los marcadores solicitados fueron negativos. En cuanto a los resultados de los participantes, hubo coincidencia general con los de referencia en todas las determinaciones solicitadas, con algunas discrepancias ocasionales. Como dato negativo, por su relevancia clínica, hubo 3 centros que informaron erróneamente un resultado falsamente negativo para el HBsAg.
Y, por último, en el control S-4B/17 se solicitó la detección de los anticuerpos de las clases IgG e IgM frente al virus de la rubéola. De nuevo hubo concordancia entre los resultados de los participantes con los de referencia, con alguna discrepancia esporádica.
La participación real fue ≥ 88% en los 8 controles remitidos, mientras que el uso de soporte externo osciló entre un 2,4 y un 25,8%. Los menores porcentajes de utilización de un centro externo se produjeron para la serología de lúes, VHB, VHC, VIH y CMV, determinaciones al alcance de la gran mayoría de los laboratorios de microbiología clínica. Por el contrario, los mayores porcentajes fueron para la serología de T. cruzi (que fue requerido por el 25,8% de los centros participantes).
En resumen, el nivel de capacitación en serología de los laboratorios españoles se puede considerar como satisfactorio. Señalar que, incluso en las mejores condiciones (como el procesamiento de un control de calidad), se obtienen resultados erróneos, incluyendo determinaciones críticas como el HBsAg y los anti-VHC y anti-VIH 1+2. Por ello, los centros deben establecer un alto nivel de control mediante la validación clínica de los resultados.
3Análisis de datos de los controles de bacteriologíaEn el año 2017 hubo 232 inscritos en el área de bacteriología (tabla 2). En el control B-1/17 se remitió una cepa de Streptococcus gallolyticus. Esta bacteria se había aislado en hemocultivos de un paciente de 78 años con una probable neoplasia de colon. La participación real (92,3%) fue buena, similar a la de otros controles. En cuanto a la identificación, si bien únicamente el 79,0% de los participantes respondieron S. gallolyticus, al agrupar esta especie con Streptococcus bovis, estrechamente relacionadas, el porcentaje de identificaciones aceptables alcanzó el 95,8%. En el estudio de sensibilidad, hubo concordancia con el del valor asignado para la mayoría de los antibióticos, con algunos errores ocasionales.
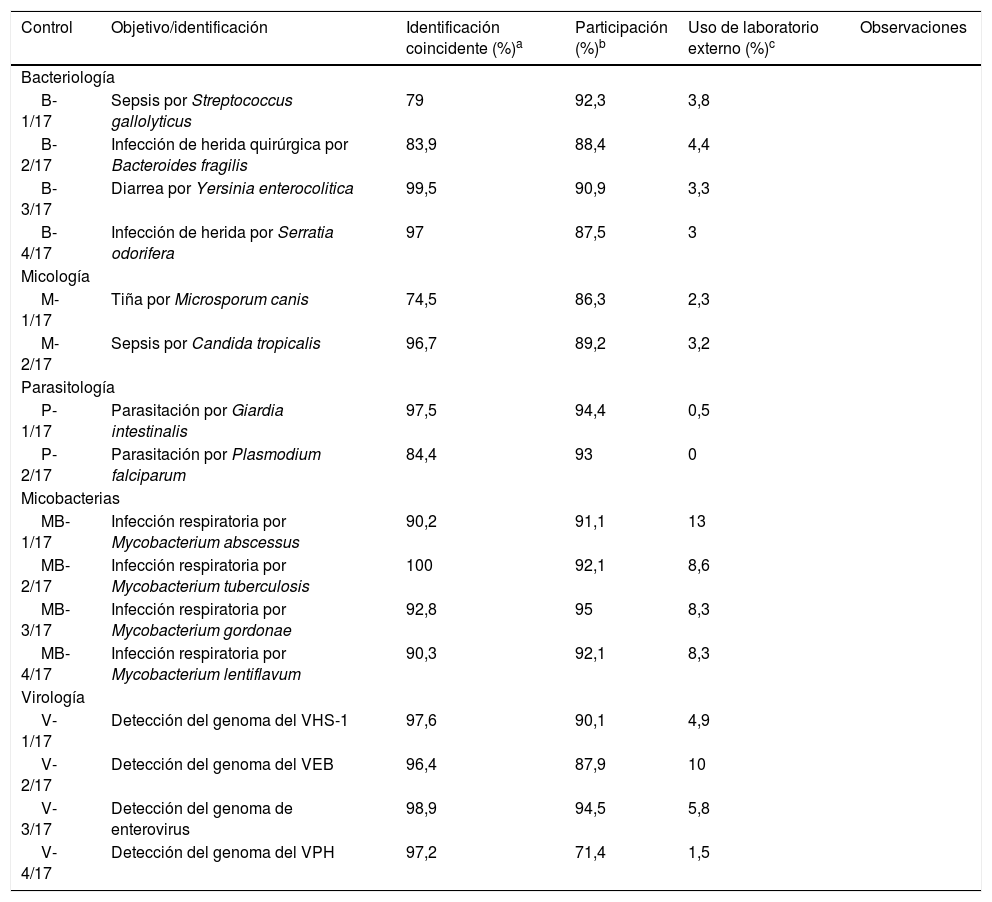
Resumen de los resultados obtenidos en otros controles del año 2017
| Control | Objetivo/identificación | Identificación coincidente (%)a | Participación (%)b | Uso de laboratorio externo (%)c | Observaciones |
|---|---|---|---|---|---|
| Bacteriología | |||||
| B-1/17 | Sepsis por Streptococcus gallolyticus | 79 | 92,3 | 3,8 | |
| B-2/17 | Infección de herida quirúrgica por Bacteroides fragilis | 83,9 | 88,4 | 4,4 | |
| B-3/17 | Diarrea por Yersinia enterocolitica | 99,5 | 90,9 | 3,3 | |
| B-4/17 | Infección de herida por Serratia odorifera | 97 | 87,5 | 3 | |
| Micología | |||||
| M-1/17 | Tiña por Microsporum canis | 74,5 | 86,3 | 2,3 | |
| M-2/17 | Sepsis por Candida tropicalis | 96,7 | 89,2 | 3,2 | |
| Parasitología | |||||
| P-1/17 | Parasitación por Giardia intestinalis | 97,5 | 94,4 | 0,5 | |
| P-2/17 | Parasitación por Plasmodium falciparum | 84,4 | 93 | 0 | |
| Micobacterias | |||||
| MB-1/17 | Infección respiratoria por Mycobacterium abscessus | 90,2 | 91,1 | 13 | |
| MB-2/17 | Infección respiratoria por Mycobacterium tuberculosis | 100 | 92,1 | 8,6 | |
| MB-3/17 | Infección respiratoria por Mycobacterium gordonae | 92,8 | 95 | 8,3 | |
| MB-4/17 | Infección respiratoria por Mycobacterium lentiflavum | 90,3 | 92,1 | 8,3 | |
| Virología | |||||
| V-1/17 | Detección del genoma del VHS-1 | 97,6 | 90,1 | 4,9 | |
| V-2/17 | Detección del genoma del VEB | 96,4 | 87,9 | 10 | |
| V-3/17 | Detección del genoma de enterovirus | 98,9 | 94,5 | 5,8 | |
| V-4/17 | Detección del genoma del VPH | 97,2 | 71,4 | 1,5 | |
VEB: virus de Epstein-Barr; VHS: virus del herpes simple; VPH: virus del papiloma humano.
El control B-2/17 se refería a un cuadro de infección de herida quirúrgica por Bacteroides fragilis. El porcentaje de participación real (88,4%) fue ligeramente inferior a la de los otros controles, lo que podría deberse, al tratarse de una bacteria anaerobia, a una mayor dificultad de crecimiento e identificación. En cuanto a la necesidad de un laboratorio externo, fue requerido por el 4,4% de los centros. Respecto a la identificación de la cepa remitida, el 83,9% de los participantes identificaron correctamente dicha especie. En el estudio de sensibilidad, los laboratorios mostraron unos resultados concordantes con el valor asignado, con la excepción de la cefoxitina, con bastante discrepancia entre los diferentes centros debido a diferencias en la interpretación en una dilución.
En el control B-3/17 se envió una cepa de Yersinia enterocolitica aislada en coprocultivos de una niña de 4 años con un cuadro de diarrea y fiebre. De nuevo, la identificación constituyó el objetivo fundamental del control, con un porcentaje de respuestas acertadas muy alto (99,5%). Asimismo, el porcentaje de participación fue del 90,9%, similar al de otros controles, mientras que solo un 3,3% de los centros hicieron uso de un laboratorio externo. Respecto al estudio de sensibilidad, con algunos errores anecdóticos, hubo una amplia concordancia para todos los antibióticos.
Finalmente, el control B-4/17 contenía una cepa de Serratia odorifera. Había sido aislada a partir de un cultivo de un exudado y de una biopsia de herida en un paciente de 71 años que había sufrido un traumatismo con un objeto punzante. Los porcentajes de participación (87,5%) y de utilización de laboratorio externo (3,0%) fueron similares a los de otros controles, mientras que la gran mayoría de los participantes (97,0%) llegaron a la identificación de especie. Respecto al estudio de sensibilidad, existió concordancia general con el antibiograma de referencia, con algunas discrepancias que afectaron especialmente a la ampicilina y a la amoxicilina-clavulánico, debido a la existencia de una b-lactamasa cromosómica inducible de clase C en la especie Serratia marcescens, que afectaría a estos 2 antibióticos, pero no en S. odorífera.
En resumen, los participantes mostraron un buen nivel de capacitación y competencia, incluso para controles, con un mayor nivel de dificultad diagnóstica a priori. Asimismo, la participación en los 4 controles fue bastante alta, superior al 87% en todos ellos.
4Análisis de datos de los controles de micologíaDurante el año 2017 se realizaron 2 envíos a los 204 centros inscritos en esta área (tabla 2). En el primero de ellos (M-1/17), se remitió un hongo filamentoso identificado como Microsporum canis. Este hongo había sido aislado a partir de una muestra de escamas de la cabeza de un paciente de 16 años, que vivía en un medio rural rodeado de animales domésticos. El índice de participación fue del 86,3%, similar al de otros controles de micología. Respecto a la identificación, el porcentaje de centros que informaron esta especie no fue muy alto (74,5%), si bien, en conjunto, un 85,2% de los centros encuadraron correctamente el hongo dentro del género Microsporum (porcentaje de respuestas aceptables por el Programa). Sus características macroscópicas, junto con el estudio microscópico con azul de lactofenol, fueron los métodos más usados para su identificación por la práctica totalidad de los participantes.
El segundo envío (M-2/17) contenía un liófilo con Candida tropicalis. Esta levadura se había aislado en hemocultivos y en un urocultivo de un paciente de 39 años ingresado en UCI. El índice de participación fue alto (89,2%), similar al de otros controles. Asimismo, la gran mayoría de los participantes (el 96,7%) identificaron correctamente esta levadura, lo que demostró el buen rendimiento de los métodos comerciales de identificación que se utilizaron, mayoritariamente la espectrometría de masas, las galerías de pruebas bioquímicas y el cultivo en medio de agar cromogénico. El antifungigrama lo realizó el 78,6% de los participantes que respondieron C. tropicalis, porcentaje similar a otros controles de levaduras recientes, y mostró unos resultados concordantes con el valor asignado para la gran mayoría de los antifúngicos.
A modo de conclusión, estos resultados muestran la buena capacitación, de la mayoría de los laboratorios participantes, para la identificación de las levaduras y de los hongos filamentosos más frecuentes.
5Análisis de datos de los controles de parasitologíaDurante 2017 se realizaron 2 envíos a los 213 laboratorios inscritos en esta área (tabla 2). En el primero de ellos (P-1/17) se remitió un concentrado de heces en el que los 2 laboratorios que actuaron de referencia detectaron, mediante un examen microscópico de las heces tras concentración, la presencia de quistes de Giardia intestinalis (Giardia lamblia o Giardia duodenalis). El índice de participación fue del 94,4%, similar al de otros controles de parasitología, mientras que el porcentaje de los laboratorios que necesitaron soporte externo fue tan solo del 0,5%. Como cabría esperar, el parásito más frecuentemente informado fue G. intestinalis (el 97,5% de los participantes).
En el segundo control (P-2/17), se remitió a los participantes un portaobjetos con una extensión sanguínea teñida con Giemsa. La muestra pertenecía a un varón de 36 años procedente de Guinea Ecuatorial, con un cuadro de escalofríos, fiebre alta, artralgias y cefalea. Los 2 laboratorios que actuaron de referencia visualizaron, mediante examen microscópico de la extensión, anillos intraeritrocitarios que se correspondían a una parasitación por Plasmodium falciparum. El índice de participación real fue del 93,0%, similar al de otros controles, mientras que ningún centro hizo uso de un laboratorio externo. A efectos de comparación, el Programa de Control de Calidad aceptó como respuesta válida la identificación de P. falciparum, bien sea como única especie observada (respuesta óptima), o bien, junto con otra especie de Plasmodium (respuesta aceptable). Así, el 76,8% de los centros observaron únicamente P. falciparum, mientras que otro 7,6% de estos informaron P. falciparum acompañado de otra especie de Plasmodium; por lo que, el porcentaje de acierto total fue del 84,4%.
En general, se puede concluir que, al igual que viene sucediendo en los últimos años, los participantes del CCS presentan una alta capacitación respecto a la detección parasitológica, ya que se constatan unos altos porcentajes de identificaciones correctas con una ausencia de utilización de un laboratorio externo.
6Análisis de datos de los controles de micobacteriasDurante el año 2017 se remitieron 4 controles a los 101 laboratorios inscritos en el área de micobacteriología (tabla 2). El primero de ellos (MB-1/17) contenía una cepa identificada como Mycobacterium abscessus. Se había aislado a partir de muestras de esputo de una mujer de 64 años con febrícula vespertina, sensación disneica y tos persistente. El porcentaje de participación fue del 91,1%, mientras que la necesidad de recurrir a un laboratorio externo fue del 13,0%, porcentajes similares a los de otros controles con micobacterias no tuberculosas. El Programa de CCS aceptó como óptima la identificación de especie M. abscessus, y como aceptables las respuestas Mycobacterium chelonae/abscessus,Mycobacterium abscessus/immunogenum y M. chelonae grupo III, dada la elevada similitud genética que presentan estas especies. Así, el 90,2% de los laboratorios contestaron correctamente M. abscessus, mientras que el porcentaje de las respuestas aceptables alcanzó el 97,8%. Respecto a los métodos usados para la identificación, todos los centros que informaron M. abscessus emplearon la espectrometría de masas, o bien algún método molecular (principalmente la hibridación inversa). El estudio de sensibilidad fue realizado por el 56,7% de los participantes que identificaron M. abscessus, y las técnicas mayoritarias fueron la microdilución seguida de las tiras de concentración. Se observó coincidencia entre los laboratorios participantes con el antibiograma del valor asignado en todos los antimicrobianos ensayados con la única excepción de la claritromicina, con una moderada discrepancia entre los centros. Ello se debía a que la cepa, aunque era sensible a los 3 días de incubación, poseía una mutación en el gen erm(41), que le confería resistencia inducible a los macrólidos.
En el control MB-2/17, se remitió una cepa identificada como Mycobacterium tuberculosis. Procedía de varias muestras de esputo de una paciente de 67 años que presentaba desde hacía tres meses un cuadro de astenia, febrícula de predominio vespertino, tos escasamente productiva y ligera disnea. El porcentaje de participación (92,1%) fue similar al de otros controles, mientras que la necesidad de recurrir a un laboratorio externo fue menor (8,6%). Desde el Programa, se consideró óptima la identificación de especie M. tuberculosis y como aceptables las respuestas complejo M. tuberculosis y M. tuberculosis/Mycobacterium canetti. Así, el 55,9% de los participantes informaron complejo M. tuberculosis, otro 38,7% respondió M. tuberculosis y un 5,4% contestó M. tuberculosis/M. canetti, por lo que el porcentaje de acierto total alcanzó el 100,0% de los participantes. Las técnicas mayoritarias para la identificación fueron la hibridación inversa, seguida de la PCR a tiempo real y de la espectrometría de masas. En cuanto al estudio de sensibilidad, se realizó por el 78,5% de los centros, predominando la dilución en medio líquido, con una excelente concordancia entre las respuestas de los participantes y el valor asignado.
Respecto al control MB-3/17, se trataba de una cepa de Mycobacterium gordonae. Se había aislado en una muestra de esputo de un paciente de 76 años con una tos escasamente productiva y aumento de su disnea habitual. El porcentaje de participación fue del 95,0%, el más alto de los 4 controles; mientras que la necesidad de recurrir a un laboratorio externo fue del 8,3%. Asimismo, el porcentaje de acierto en la identificación fue bastante bueno, ya que el 90,7% de los participantes identificaron la cepa como M. gordonae y otro 2,1% respondieron Mycobacterium paragordonae, con lo que el porcentaje de respuestas aceptables alcanzó el 92,8%. Para la identificación se empleó principalmente la hibridación inversa, de forma única o junto a otros métodos moleculares, y la espectrometría de masas. El estudio de sensibilidad lo realizó únicamente el 6,7% de los centros, ya que la mayoría consideraron que M. gordonae no era patógeno.
Por último, en el control MB-4/17 se remitió una cepa identificada como Mycobacterium lentiflavum. Procedía de una muestra de esputo de un paciente de 79 años con una tos escasamente productiva y aumento de su disnea habitual. Los porcentajes de participación (92,1%) y de utilización de laboratorio externo (8,3%) fueron similares a los de los últimos controles de micobacterias. El Programa CCS aceptó únicamente como válida la identificación M. lentiflavum, especie informada por la mayoría de los participantes (90,3%). Como en el resto de los últimos controles de micobacterias, los métodos mayoritariamente empleados para la identificación fueron las pruebas de hibridación inversa y la espectrometría de masas. En cuanto al estudio de sensibilidad, se realizó tan solo por el 13,1% de los participantes, ya que muchos de ellos consideraron esta especie como no patógena o de significado clínico dudoso.
7Análisis de datos del control de microbiología molecularEn el año 2017 se realizaron 2 envíos de microbiología molecular a los participantes (tabla 1). En el primer control (BM-1/17) se remitió una alícuota de exudado uretral de un varón de 25 años con un cuadro de micción dolorosa y exudado uretral amarillento, que relataba haber mantenido relaciones sexuales con diferentes parejas. Se solicitó a los participantes la detección del genoma de Chlamydia trachomatis y Neisseria gonorrhoeae. El valor asignado de referencia fue el de detección positiva del genoma de ambos microorganismos. Para este primer control se enviaron un total de 91 muestras, y aportaron resultados valorables un 92,3% de ellos, mientras que la utilización de un laboratorio externo fue solo del 1,2%. Del conjunto de las determinaciones efectuadas, un 97,7% fueron positivas a C. trachomatis y un 98,8% lo fueron a N. gonorrhoeae, con un claro predominio de la PCR a tiempo real.
En el segundo control (BM-2/17) se remitió una alícuota procedente de un paciente de 36 años que presentaba un cuadro de malestar general y pérdida de peso de mes y medio de evolución. Se solicitó a los participantes la detección del genoma del complejo M. tuberculosis. El porcentaje de participación real en este control fue del 94,5%, con una utilización de un laboratorio externo del 2,3%. Todas las determinaciones realizadas excepto 1 (el 98,9%) fueron positivas, coincidiendo con el valor asignado. El método mayoritariamente empleado para la detección de ADN de este complejo fue la PCR a tiempo real, destacando especialmente el reactivo Xpert® MTB/RIF de Cepheid.
8Análisis de datos del control de virologíaEn 2017 se realizaron 4 envíos de virología (tabla 2). El primer control (V-1/17) consistía en una alícuota de líquido cefalorraquídeo (LCR) de un paciente de 25 años con meningitis. Se solicitó a los participantes la detección del genoma del virus del herpes simple (VHS) 1+2. El valor asignado fue el de detección positiva del genoma de VHS-1, con una detección negativa del genoma del VHS-2. La muestra de LCR fue remitida a los 91 centros inscritos en esta área, de los que un 90,1% emitieron hoja de respuesta con datos evaluables. Todas las determinaciones efectuadas excepto 2 (el 97,6%) fueron positivas para la detección de genoma del VHS-1. Respecto a los métodos empleados, hubo un claro predominio de la PCR a tiempo real, especialmente de los reactivos de RealCycler® de Progenie.
En el segundo control (V-2/17) se remitió una alícuota de exudado faringoamigdalar que procedía de una paciente de 13 años con un cuadro de mononucleosis infecciosa. Se solicitó a los participantes la detección del genoma del VEB. El porcentaje de participación fue del 87,9%, similar a otros controles. Todas las determinaciones efectuadas excepto 3 (el 96,4%) fueron positivas para VEB, coincidiendo con el valor asignado. En cuanto a los métodos utilizados, casi todos los participantes emplearon la PCR a tiempo real, con un predominio de los reactivos RealStar® de Altona y RealCycler® de Progenie.
En el tercer control (V-3/17) se remitió otra alícuota de LCR para la detección de enterovirus. La muestra procedía de un niño de 3 años con una meningitis. El porcentaje de participación real fue del 94,5%, el más alto de los 4 controles de virología. Todos los participantes excepto 1 (el 98,9%) detectaron el enterovirus en la muestra de LCR, resultado coincidente con el valor asignado. La mayoría de las determinaciones fueron por PCR a tiempo real, con un predominio del reactivo Xpert® de Cepheid.
En el último control de esta área (V-4/17) se envió una alícuota de exudado endocervical para la detección del virus del papiloma humano (VPH). La historia clínica correspondía a una paciente de 31 años que presentaba pequeñas lesiones sobreelevadas en el cérvix uterino. La participación fue solo del 71,4%, la más baja de los 4 controles de virología. Todos los participantes excepto 2 (el 97,2%) detectaron el VPH en la muestra remitida, coincidiendo con el valor asignado. El método más empleado fue de nuevo la PCR a tiempo real, con un predominio de los reactivos cobas® de Roche y Anyplex™ II de Seegene.
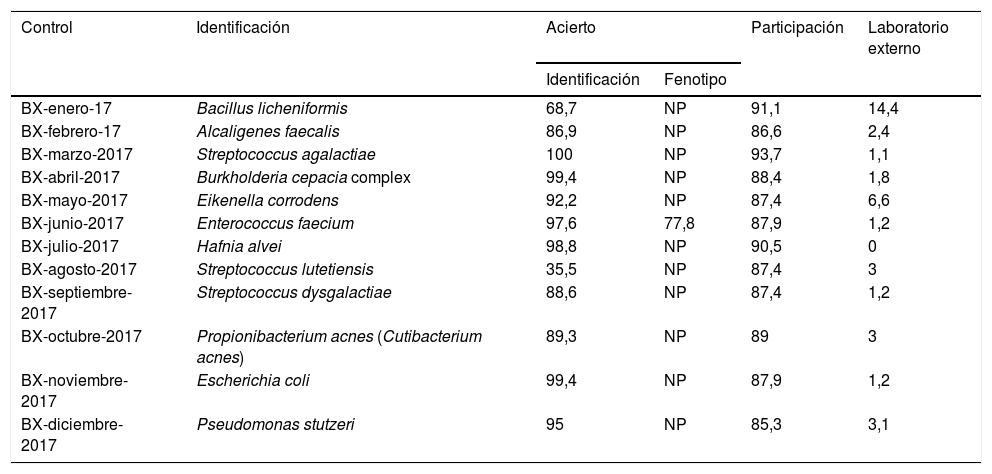
9Análisis de datos de los controles de bacteriología mensualA lo largo del año 2017 se enviaron 12 controles mensuales de bacteriología a 190 centros inscritos. La participación media fue del 88,6%, con escasas oscilaciones (85,3-93,7%).
La utilización de laboratorio externo fue baja en 10 de los 12 controles, oscilando entre el 0,0 y el 3,1% (tabla 3). Los porcentajes más altos de uso de un centro externo ocurrieron en los controles de enero y mayo, en los que se remitieron, respectivamente, una cepa de Bacillus licheniformis (el 14,4% de soporte externo) y otra cepa de Eikenella corrodens (6,6%). Ello se debe, como cabe suponer, a una mayor dificultad en la identificación de estas cepas.
Características y porcentajes de participación, acierto y uso de laboratorio externo en los controles de bacteriología mensual del año 2017
| Control | Identificación | Acierto | Participación | Laboratorio externo | |
|---|---|---|---|---|---|
| Identificación | Fenotipo | ||||
| BX-enero-17 | Bacillus licheniformis | 68,7 | NP | 91,1 | 14,4 |
| BX-febrero-17 | Alcaligenes faecalis | 86,9 | NP | 86,6 | 2,4 |
| BX-marzo-2017 | Streptococcus agalactiae | 100 | NP | 93,7 | 1,1 |
| BX-abril-2017 | Burkholderia cepacia complex | 99,4 | NP | 88,4 | 1,8 |
| BX-mayo-2017 | Eikenella corrodens | 92,2 | NP | 87,4 | 6,6 |
| BX-junio-2017 | Enterococcus faecium | 97,6 | 77,8 | 87,9 | 1,2 |
| BX-julio-2017 | Hafnia alvei | 98,8 | NP | 90,5 | 0 |
| BX-agosto-2017 | Streptococcus lutetiensis | 35,5 | NP | 87,4 | 3 |
| BX-septiembre-2017 | Streptococcus dysgalactiae | 88,6 | NP | 87,4 | 1,2 |
| BX-octubre-2017 | Propionibacterium acnes (Cutibacterium acnes) | 89,3 | NP | 89 | 3 |
| BX-noviembre-2017 | Escherichia coli | 99,4 | NP | 87,9 | 1,2 |
| BX-diciembre-2017 | Pseudomonas stutzeri | 95 | NP | 85,3 | 3,1 |
NP: no procede.
Los porcentajes de identificaciones correctas conseguidos por los participantes fueron elevados en 10 de los controles, y se alcanzó un máximo en los controles a priori más sencillos (Streptococcus agalactiae, Escherichia coli) y también en el complejo Burkholderia cepacia; con un porcentaje de acierto superior al 99,0% en todos ellos. Por el contrario, los menores índices de identificaciones correctas se obtuvieron con la cepa de B. licheniformis mencionada anteriormente (el 68,7% de aciertos) y con una cepa de Streptococcus lutetiensis (el 35,5% de aciertos). Respecto al control con B. licheniformis, si bien el 68,7% de los participantes respondieron esta especie, al agruparse las otras identificaciones aceptadas como válidas por parte del CCS (género Bacillus o cualquier especie de este), dicho porcentaje aumentó hasta un 91,9%. En cuanto al control con S. lutetiensis, al agrupar las respuestas de las especies que forman parte del complejo S. bovis (que incluye S. lutetiensis), el porcentaje de identificaciones aceptadas aumentó hasta un 96,4%.
En una ocasión, la cepa enviada presentaba una característica fenotípica especial que constituía el verdadero objetivo perseguido por el control. Así, en el control de junio, el 77,8% de los centros informaron explícitamente que la cepa de Enterococcus faecium remitida era resistente a la vancomicina y teicoplanina (fenotipo VanA), por lo que los resultados se pueden considerar como óptimos, aunque algo mejorables.
En resumen, los porcentajes de participación y acierto son altos para casi todos los controles y se confirma de nuevo que los laboratorios de nuestro país están bien capacitados para los análisis bacteriológicos.
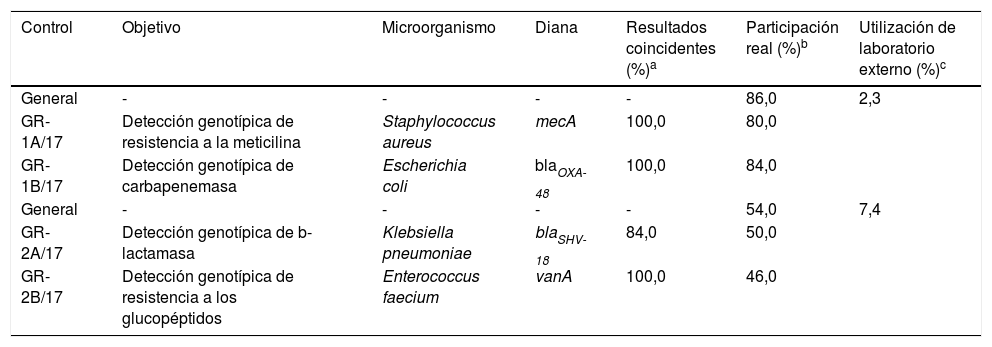
10Análisis de datos del control de genotipos de resistenciaDurante el año 2017 se remitieron 4 controles, separados en 2 envíos, a los 50 laboratorios inscritos en el área de detección genotípica de mecanismos de resistencia bacteriana.
El primer envío costaba de una cepa de Staphylococcus aureus, en la que se solicitaba la detección molecular del gen de resistencia a la meticilina (control GR-1A/17), y de una cepa de E. coli en la que se requería la detección genotípica de carbapenemasa (control GR-1B/17). El porcentaje de participación real en alguno de estos 2 controles fue del 86,0%, superior al de 2016 (que fue del 71,4%), mientras que tan solo un 2,3% de los centros hicieron uso de un centro externo (tabla 4). En cuanto a los resultados obtenidos, todos los participantes (el 100,0%) detectaron tanto el gen mecA en la cepa de S. aureus como el gen productor de OXA-48 en la cepa de E. coli.
Resumen de los controles de genotipos de resistencia del año 2017
| Control | Objetivo | Microorganismo | Diana | Resultados coincidentes (%)a | Participación real (%)b | Utilización de laboratorio externo (%)c |
|---|---|---|---|---|---|---|
| General | - | - | - | - | 86,0 | 2,3 |
| GR-1A/17 | Detección genotípica de resistencia a la meticilina | Staphylococcus aureus | mecA | 100,0 | 80,0 | |
| GR-1B/17 | Detección genotípica de carbapenemasa | Escherichia coli | blaOXA-48 | 100,0 | 84,0 | |
| General | - | - | - | - | 54,0 | 7,4 |
| GR-2A/17 | Detección genotípica de b-lactamasa | Klebsiella pneumoniae | blaSHV-18 | 84,0 | 50,0 | |
| GR-2B/17 | Detección genotípica de resistencia a los glucopéptidos | Enterococcus faecium | vanA | 100,0 | 46,0 |
En el segundo envío se remitieron una cepa de Klebsiella pneumoniae, para la detección genotípica de b-lactamasa (control GR-2A/17), y una cepa de E. faecium, para la detección del gen de resistencia a los glucopéptidos (control GR-2B/17). El porcentaje de participación real en alguno de estos 2 controles fue del 54,0%, inferior a otros controles, con una utilización de laboratorio externo del 7,4%. Respecto a los resultados, si bien todos los participantes (el 100,0%) detectaron el gen vanA en la cepa de E. faecium, únicamente el 59,3% de estos detectaron el gen productor de SHV en la cepa de K. pneumoniae.
11ConclusiónLos resultados obtenidos a lo largo del período analizado confirman, una vez más, la buena capacitación general de los laboratorios de microbiología, sin duda atribuible, entre otros, a la incorporación de profesionales bien entrenados y con conocimientos sólidos. Aun así, como en cualquier programa de control externo, se pone de manifiesto que la obtención de algún resultado erróneo, incluso en las determinaciones de mayor trascendencia, es un riesgo que puede presentarse en cualquier laboratorio. Todo ello no hace más que resaltar la importancia de complementar el control de calidad interno, que cada laboratorio lleva a cabo, con los ejercicios de intercomparación externos7–18, como los que ofrece el CCS.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.