La suplementación de la dieta con un 10% (p/v) de fructosa en el agua de bebida durante 14 días en ratas produce hipertrigliceridemia y esteatosis hepática como consecuencia de una reducción en la expresión y en la actividad transcripcional de PPARα en el hígado. En el presente trabajo hemos estudiado en un modelo in vitro, la línea celular FaO, el posible mecanismo por el cual la fructosa reduce la expresión de PPARα.
Material y métodosLas células FaO se incubaron en ausencia o presencia de fructosa 25mM. Se obtuvieron extractos totales y nucleares para la determinación de los niveles relativos de ARNm por RT-PCR, y de proteínas por Western Blot, de aquellas enzimas y factores de transcripción implicados en las alteraciones producidas por la fructosa. Asimismo, se valoró la actividad PP2A.
ResultadosLa incubación con fructosa redujo la expresión de PPARα y sus genes diana ACO y CYP4A1, e incrementó los niveles de proteína ChREBP y ARNm de su gen diana L-PK. El inhibidor de PP2A ácido okadaico anuló el incremento de la actividad PP2A mediado por la fructosa y evitó parcialmente la inducción de L-PK, pero no modificó la reducción de la expresión de PPARα, ni tampoco la de ACO y CYP4A1.
ConclusionesEl aumento de los niveles de proteína ChREBP y de ARNm de L-PK, así como la represión de PPARα mediados por la fructosa, se reproducen en la línea celular FaO tratada con fructosa 25mM. Los experimentos realizados en presencia de ácido okadaico sugieren que la activación de PP2A por metabolitos de la fructosa no juega un papel relevante en la reducción de la expresión de PPARα producida por la fructosa.
The addition of fructose in drinking water (10% w/v) for two weeks to rats induces hypertriglyceridemia and fatty liver by reducing the expression and transcriptional activity of PPARα in liver. Using an in vitro model, hepatoma FaO cells, we have studied the possible mechanism underlying the fructose-mediated reduction of PPARα expression.
Material and methodsFaO cells were incubated with or without 25mM fructose. Total and nuclear extracts were obtained and used to assess the relative levels of specific mRNAs by RT-PCR, and proteins by Western Blot, of those enzymes and transcription factors involved in fructose-induced alterations. PP2A activity was also determined.
ResultsFructose treatment reduced PPARα mRNA levels. Consequently, the expression of ACO and CYP4A1, two PPARα target genes, also decreased. Incubation of FaO cells with fructose increased both protein levels of ChREBP and mRNA levels of L-PK. Okadaic acid, a PP2A inhibitor, blunted the fructose-induced increase in PP2A activity, and partially prevented the fructose-mediated induction of L-PK expression, but it did not modify the effect of fructose on PPARα and its target genes.
ConclusionsBoth the increase in ChREBP and L-PK expression, and the decrease in PPARα mRNA levels were reproduced in FaO cells incubated with 25mM fructose. Experiments performed in the presence of okadaic acid suggest that activation of PP2A by intermediary metabolites of fructose is not involved in fructose-mediated decrease of PPARα expression.
En las últimas décadas, la prevalencia de obesidad y síndrome metabólico ha alcanzado proporciones prácticamente epidémicas en los países occidentales1. Este hecho se ha relacionado, entre otros, con factores ambientales como la reducción de la actividad física o el cambio en los hábitos dietéticos de la población2. Además del aumento de la ingesta calórica total, se ha observado un cambio en el tipo de nutrientes que constituyen la dieta, cobrando especial importancia el aumento del consumo de fructosa que ha tenido lugar en los últimos 30 años, principalmente como edulcorante de bebidas refrescantes3,4. En este sentido, varios estudios relacionan el consumo de fructosa con la aparición de obesidad5 y alteraciones metabólicas como la hipertrigliceridemia6,7.
En trabajos previos observamos que la suplementación de la dieta con un 10% de fructosa (p/v) en el agua de bebida durante 14 días produce hipertrigliceridemia y esteatosis hepática en ratas Sprague Dawley macho8,9. Para ello, es necesaria la conjunción de dos factores: en primer lugar, la activación de la síntesis de los ácidos grasos por un aumento de la expresión y actividad del factor de transcripción ChREBP (carbohydrate response element-binding protein), mediado a su vez por un aumento de la expresión de la proteína fosfatasa 2 A (PP2A), y en segundo lugar, la reducción de la expresión y de la actividad transcripcional de PPARα (peroxisome proliferator-activated receptor α), que conduce a la disminución del catabolismo de los ácidos grasos.
En el presente estudio se ha utilizado una línea celular de hepatoma de rata, las células FaO, con el fin de profundizar en los mecanismos moleculares responsables de la reducción de la expresión de PPARα mediada por la fructosa. Los resultados muestran que este efecto es independiente de la activación de la fosfatasa PP2A.
Material y métodosCultivo celularSe utilizó la línea celular de hepatoma de rata FaO, adquirida en la European Collection of Cell Cultures (ECACC). Las células se cultivaron en medio DMEM low glucose (Gibco, Invitrogen) suplementado con 10% de FBS Gold (PAA) y antibiótico (100U/ml de penicilina y 100μg/ml de estreptomicina, Gibco, Invitrogen). Para el tratamiento de las células, la proporción de suero se redujo al 1% y las células se incubaron en ausencia (medio control) o presencia de fructosa (Sigma) a una concentración 25mM (medio fructosa) durante 24h.
El inhibidor ácido okadaico (Sigma) fue utilizado a una concentración 20nM, realizando un pre-tratamiento de 30min previo a la adición de fructosa en el medio.
Análisis de ARNmPara el aislamiento del ARN total de las células se utilizó el reactivo de Trizol (Invitrogen) siguiendo las instrucciones del fabricante. La cuantificación del ARN se realizó mediante la lectura de absorbancia a 260nm en un espectrofotómetro (PerkinElmer, Lambda 45). Se verificó la integridad del ARN sometiéndolo a una electroforesis en condiciones desnaturalizantes en un gel de agarosa al 1% con bromuro de etidio para la visualización de las bandas de ARN ribosomal 18S y 28S.
Los niveles relativos de ARNm fueron determinados mediante la reacción de la transcriptasa inversa acoplada a la reacción en cadena de la polimerasa (RT-PCR). Se sintetizó ADN complementario (ADNc) a partir de 0.5μg de ARN total utilizando 125ng de oligonucleótidos inespecíficos (random hexamers, Roche Diagnostics), 500μM de cada dNTP (Sigma), 200U de la enzima transcriptasa inversa (M-MVL-RT) y 20U de RNasa Out (Invitrogen), en un tampón que contenía 50mM Tris-HCl (pH8,3), 75mMKCl, 3mMMgCl2, 10mMDTT, todo ello en un volumen final de 20μl. La reacción de la transcriptasa inversa se realizó sometiendo las muestras a las siguientes etapas: 5min a 65°C, 5min a 4°C, 2min a 37°C, 10min a 25°C, 50min a 37°C y 15min a 70°C.
La reacción de PCR se llevó a cabo usando 5μl del producto de la reacción de RT, 0,5μg de cada oligonucleótido específico (sentido y antisentido), 200μM de cada dNTP, 1U de Taq ADN polimerasa (Invitrogen) y 0,25μCi de α-[32P]dATP (PerkinElmer) en tampón Tris-HCl 20mM pH8,5 y 2,5mMMgCl2, en un volumen final de reacción de 50μl. Para evitar hibridaciones inespecíficas, el ADNc y la enzima fueron separados de los oligonucleótidos específicos y los dNTP mediante una capa de parafina que funde cuando la reacción alcanza los 55-60°C, permitiendo entonces que todos los reactivos entren en contacto.
La reacción de PCR se realizó en un Termociclador MJ Research (Ecogen) y consta de las siguientes fases: 1) desnaturalización de los oligonucleótidos y ADNc a 94°C durante 1min; 2) n ciclos de amplificación. Cada ciclo consiste en una desnaturalización a 92°C, 1min, seguida de una hibridación con los oligonucleótidos específicos, a 60°C 1min y 15s y una etapa de síntesis a 72°C 1min 50s, 3) etapa de síntesis final, a 72°C 5min. Se diseñaron estudios previos con el fin de establecer las condiciones óptimas de amplificación de los diferentes genes a estudiar, determinando el número de ciclos y la cantidad de ARN necesaria para que la relación ARN/producto final fuera lineal y trabajáramos siempre por debajo de las condiciones de saturación. Para normalizar los resultados se usó el gen control APRT. El número de ciclos de amplificación utilizados en cada caso, así como las secuencias de los oligonucleótidos específicos para cada gen y el tamaño del producto de PCR, se describen en la tabla 1. Cinco microlitros de la reacción de PCR se sometieron a electroforesis en gel de poliacrilamida al 5%, los productos de amplificación fueron escaneados (Personal Molecular Imager, Bio Rad) y se cuantificaron las bandas radiactivas con el software Quantity One (BioRad). Todos los resultados se expresan como los valores de ARNm de cada uno de los genes estudiados normalizados por los valores de ARNm del gen control, APRT.
Oligonucleótidos específicos utilizados para la reacción de PCR
| Genes | Acceso GenBank | Secuencias sentido y antisentido | Producto PCR (bp) | Ciclos amplificación |
| ACO | NM017340 | 5’-ACTATATTTGGCCAATTTTGTG-3’ 5’-TGTGGCAGTGGTTTCCAAGCC-3’ | 195 | 24 |
| APRT | L04970 | 5’-AGCTTCCCGGACTTCCCCATC-3’ 5’-GACCACTTTCTGCCCCGGTTC-3’ | 329 | 21-24 |
| CYP4A1 | NM175837 | 5’-CTGGCTTCCTCCAAGTGGCCT-3’ 5’-TTGCTTCCCCAGAACCATCGA-3’ | 509 | 25 |
| L-PK | M11709 | 5’-TATGGCGGACACCTTCCTGGA-3’ 5’-GCTGAGTGGGGAGGTTGCAAA-3’ | 250 | 21 |
| PPARα | M88592 | 5’-GGCTCGGAGGGCTCTGTCATC-3’ 5’-ACATGCACTGGCAGCAGTGGA-3’ | 654 | 25 |
Los extractos nucleares de células FaO se prepararon siguiendo el protocolo descrito por Andrews y Faller10, y la concentración proteica se determinó siguiendo el método de Bradford11.
Así, 30μg de extractos nucleares se sometieron a electroforesis en geles de poliacrilamida-SDS al 10%. Posteriormente, las proteínas se transfirieron a membranas Immobilon polyvinylidene diflouride (PVDF) (Millipore) y se bloquearon durante 1h a temperatura ambiente en solución de TBS-0,1% Tween-20 con un 5% de leche en polvo desnatada. Posteriormente, las membranas se incubaron con anticuerpo primario anti-ChREBP (Santa Cruz Biotechnology) a una dilución 1:500 en la misma solución de bloqueo durante unas 16h a 4°C. Después de 4 lavados de 5min cada uno, la membrana se puso a contactar con anticuerpo secundario anti-goat (Santa Cruz Biotechnology) a una dilución 1:3.000 durante 1h a temperatura ambiente. La detección se realizó utilizando el reactivo ECL chemiluminescence kit HRP (Amersham Biosciences) y las bandas de proteína fueron cuantificadas mediante el programa Quantity One (Bio Rad). Para el control de carga las membranas se incubaron con β-actina (Sigma-Aldrich). El tamaño de las proteínas se comprobó utilizando marcadores de peso molecular (Invitrogen).
Actividad PP2APara la realización del ensayo de actividad PP2A se aisló dicha proteína mediante la técnica de inmunoprecipitación, y posteriormente se evaluó su actividad fosfatasa mediante el kit colorimétrico Sensolyte pNPP Protein Phosphatase Assay Kit (Anaspec).
Los extractos celulares se obtuvieron según las recomendaciones del fabricante del kit, y la concentración proteica se analizó por el método de Bradford11. Para la inmunoprecipitación, se partió de 100μg de proteína y se añadieron 4μg de anticuerpo anti-PP2A (Millipore). La mezcla se llevó a un volumen final de 500μl con tampón TBS que contenía un 2% (p/v) de BSA, y se dejó en rotación durante 2h a 4°C. Pasado este tiempo, se añadieron 50μl de proteína A/G agarosa (Santa Cruz) y la nueva mezcla se incubó durante unas 16h a 4°C, en rotación constante. Posteriormente se realizaron 3 lavados del complejo proteína PP2A-anticuerpo-proteína A/G agarosa con 700μl de tampón TBS, centrifugando a 1.000g durante 5min y a 4°C para descartar el sobrenadante. Tras el último lavado se añadieron 60μl de Assay Buffer y 60μl de la mezcla de reacción pNPP (preparados según instrucciones del fabricante) a dicho complejo, incubando las muestras durante 45min a temperatura ambiente y en rotación constante para permitir la defosforilación del sustrato pNPP. Por último, se realizó una centrifugación de la mezcla de reacción a 1.000g durante 5min y a temperatura ambiente, y 80μl de los sobrenadantes se transfirieron a una placa de 96 pocillos. El grado de defosforilación del sustrato en las distintas muestras se determinó por lectura de su absorbancia a 405nm en un espectrofotómetro de microplaca (Benchmark Plus, Bio Rad).
EstadísticaLos resultados se expresan como la media±desviación estándar. Las diferencias significativas se establecieron mediante la prueba de Student's o bien mediante una ANOVA con contraste a posteriori con la prueba de comparación múltiple de Tukey. El nivel de significación estadística se estableció en p≤0,05.
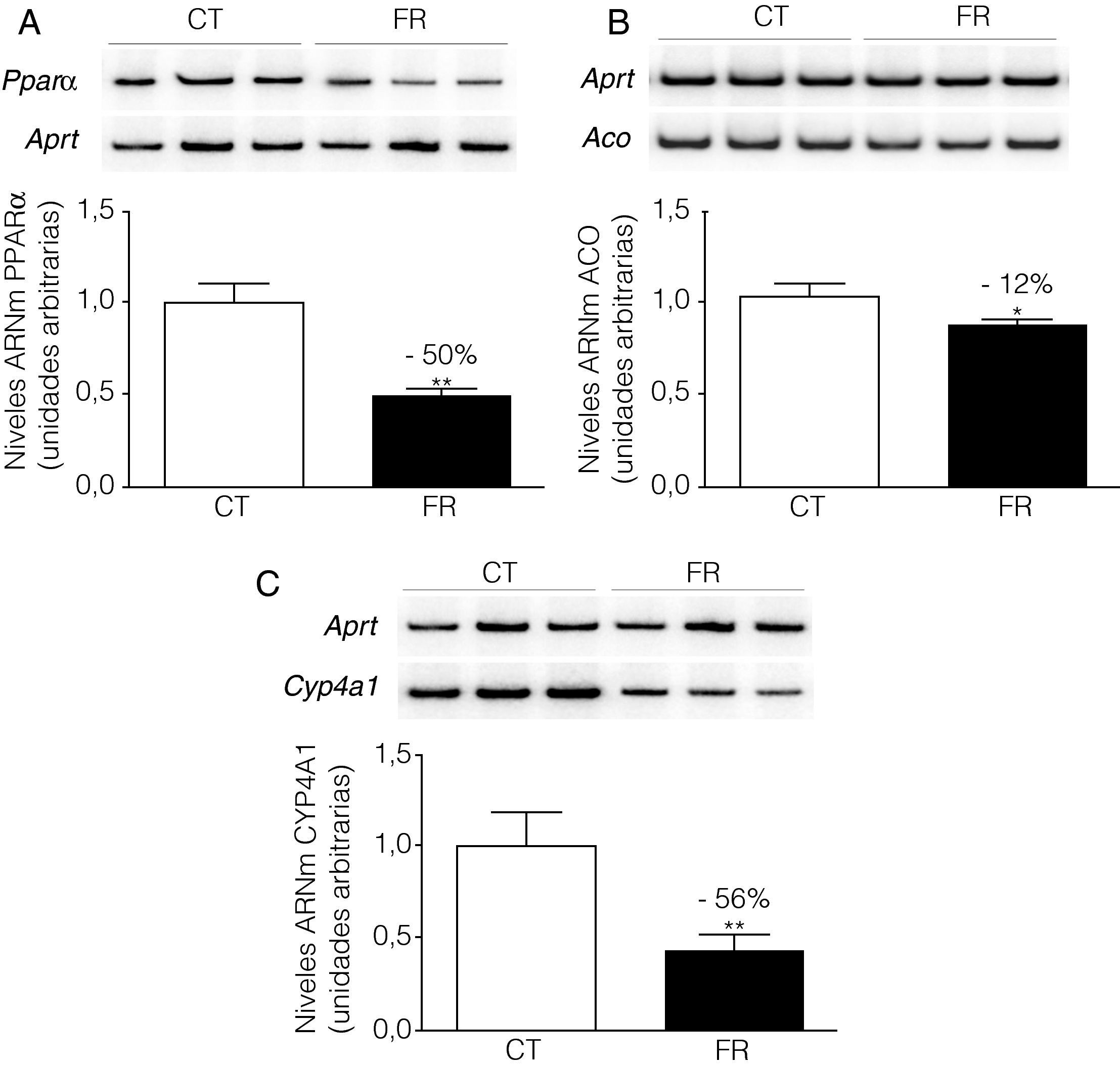
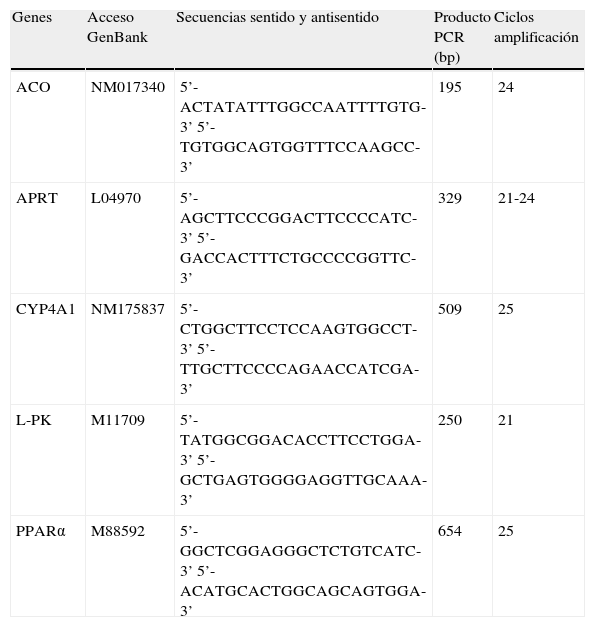
ResultadosLa fructosa reduce la expresión de PPARα y de sus genes diana en la línea celular FaOLa incubación de las células FaO con fructosa a una concentración 25mM durante 24h redujo un 50% los niveles de ARNm correspondientes a PPARα (fig. 1A). Del mismo modo, la expresión de las enzimas ACO (Acil-CoA oxidasa) y CYP4A1 (citocromo P450 4A1), ambos genes diana de PPARα, se vio reducida en un 12 y un 56%, respectivamente (figs. 1B y C).
La fructosa reduce la expresión de PPARα y sus genes diana en células FaO. Ensayo de RT-PCR para los genes PPARα (A), ACO (B) y CYP4A1 (C) en células FaO incubadas en ausencia (condición control, CT) o presencia de fructosa 25mM (condición fructosa, FR) durante 24h. Las gráficas muestran los niveles relativos de ARNm correspondientes a cada gen, normalizados por los niveles de ARNm del gen control, APRT, y expresados como la media±desviación estándar para n=4 muestras por grupo de tratamiento (*p<0,05 y **p<0,01 respecto a la condición control).
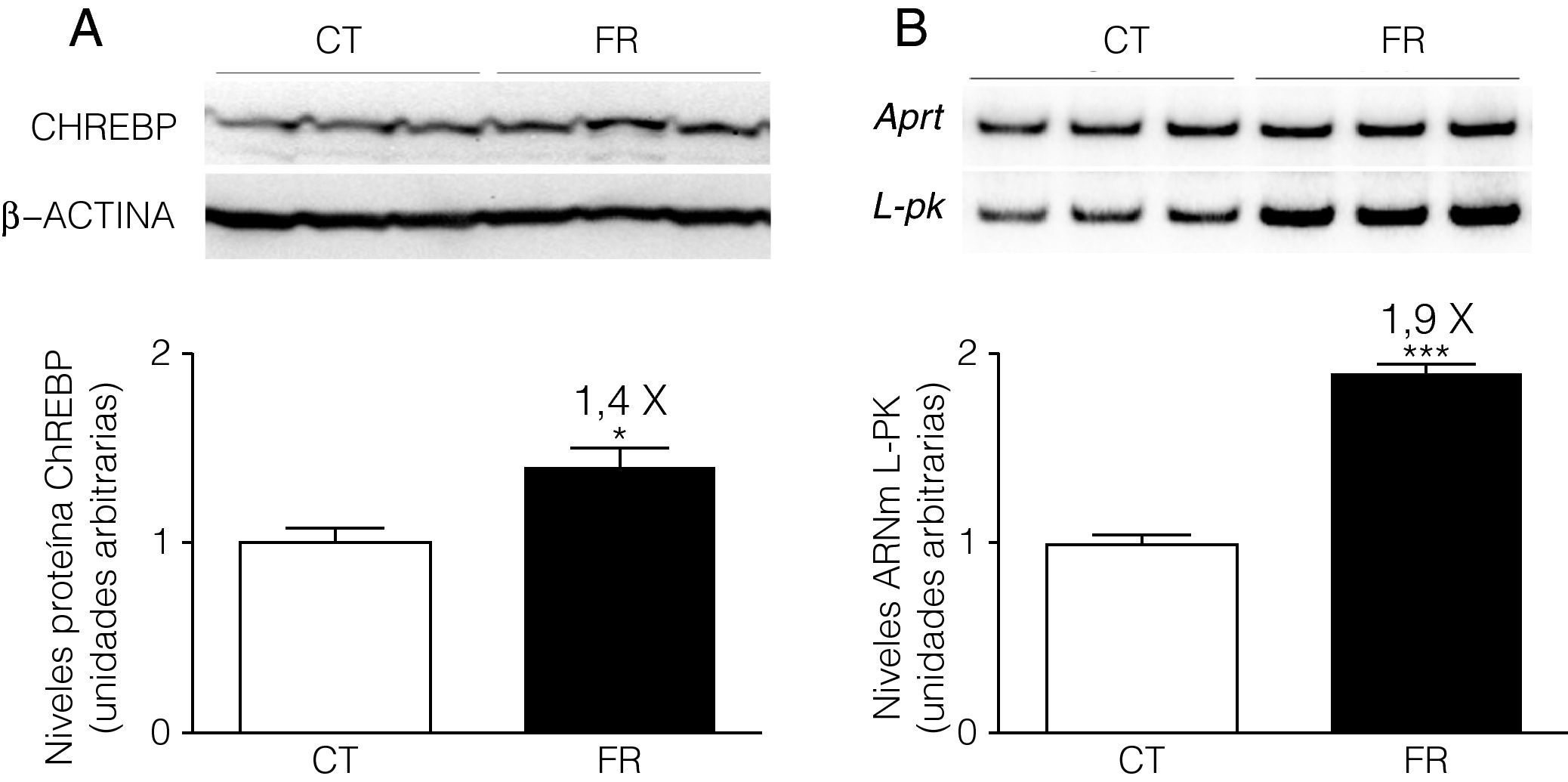
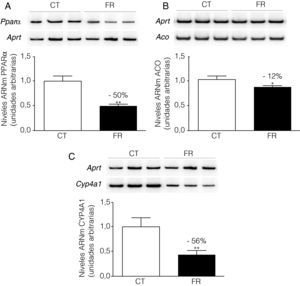
Al igual que sucede in vivo8, en la línea celular FaO la fructosa aumenta los niveles de proteína ChREBP en el núcleo (1,4X) (fig. 2A). Asimismo, la expresión del gen diana de ChREBP, piruvato cinasa hepática (liver pyruvate kinase, L-PK), se vio incrementada 1,9 veces por la adición de fructosa al medio (fig. 2B).
La fructosa aumenta la expresión de ChREBP y L-PK en células FaO. A) Gráfica de los niveles proteicos de ChREBP en extractos nucleares de células FaO incubadas en ausencia (condición control, CT) o presencia de fructosa 25mM (condición fructosa, FR) durante 24h. Cada barra representa la media±desviación estándar para n=4 muestras por grupo de tratamiento. En la parte superior de la gráfica se puede observar la autorradiografía representativa del ensayo de Western Blot. La cantidad de proteína cargada se confirmó por el método de Bradford, y la uniformidad de carga en cada línea mediante la determinación de la señal de la proteína β-actina (*p<0,05 respecto a la condición control). B) Niveles relativos de ARNm de L-PK en muestras de células FaO control (CT) y fructosa (FR), determinados por la técnica de RT-PCR y normalizados por la expresión del gen APRT. Cada valor representa la media±desviación estándar de n=4 muestras por grupo, expresado en unidades arbitrarias (***p<0,001 respecto a la condición control).
Está ampliamente descrito que los hidratos de carbono como la glucosa o la fructosa activan, a través de su metabolito intermediario xilulosa-5-fosfato, la proteína PP2A. Una vez activa, PP2A defosforila a ChREBP, promoviendo su localización nuclear y su unión al ADN, pudiendo activar así la transcripción de sus genes diana, como L-PK12,13. Con el fin de averiguar si existe una relación entre el aumento de la actividad PP2A y el descenso de la expresión de PPARα se utilizó un inhibidor de la proteína PP2A, el ácido okadaico, a una concentración 20nM14.
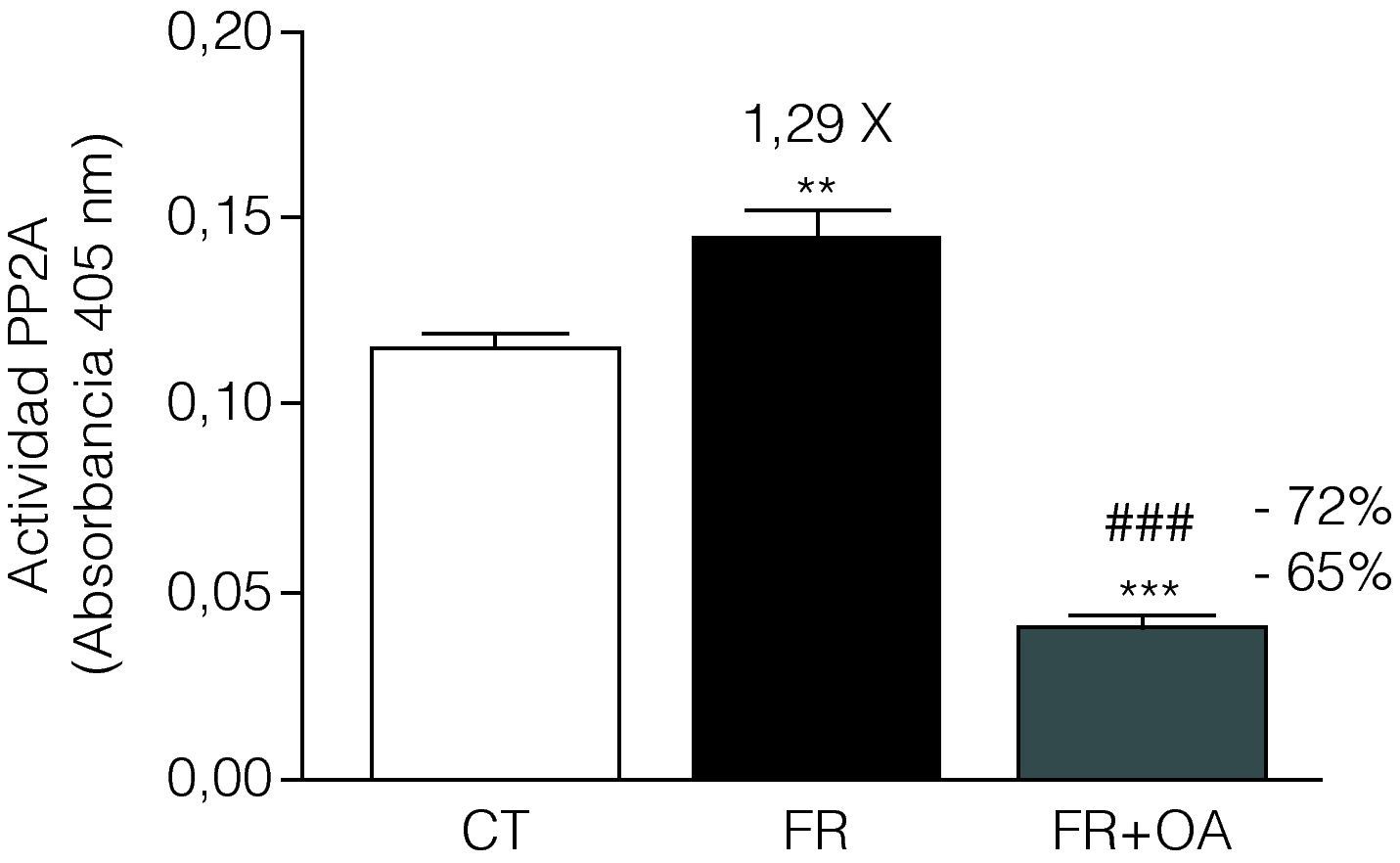
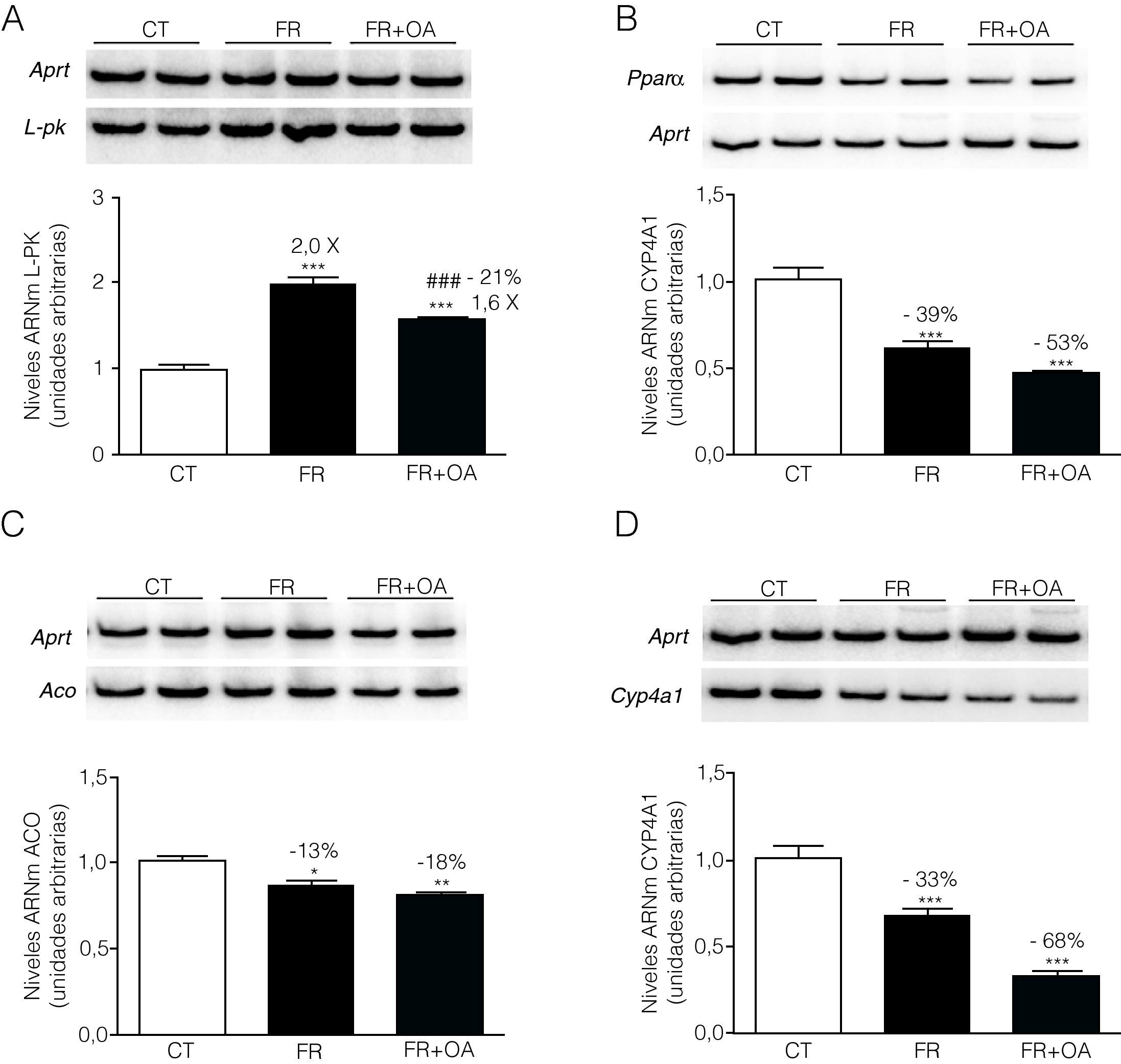
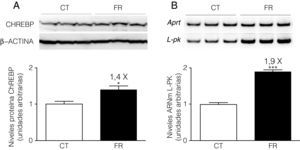
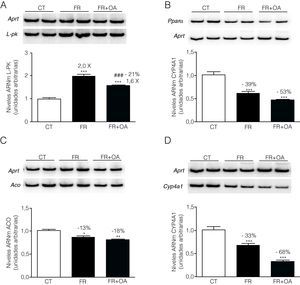
El tratamiento de las células FaO con fructosa aumentó un 29% la actividad de la proteína fosfatasa PP2A, mientras que la adición de ácido okadaico al medio redujo dicha actividad hasta niveles inferiores incluso a los que presentaban las células control, anulando completamente la inducción producida en presencia de fructosa (fig. 3). Por otra parte, la incubación con ácido okadaico produjo una anulación parcial (reducción del 21%) de la inducción de L-PK mediada por la fructosa (fig. 4A), pero no modificó la reducción de la expresión de PPARα (fig. 4B). Del mismo modo, el ácido okadaico tampoco anuló el efecto de la fructosa sobre la expresión de los genes diana de PPARα, ACO y CYP4A1 (figs. 4C y D).
La actividad de la fosfatasa PP2A en células FaO aumenta por el tratamiento con fructosa, efecto que se anula por la adición de ácido okadaico en el medio de cultivo. Ensayo de actividad PP2A en células FaO incubadas en ausencia (condición control, CT), presencia de fructosa 25mM (condición fructosa, FR) o bien en presencia de fructosa 25mM y ácido okadaico 20nM (condición fructosa+ácido okadaico, FR+OA) durante 24h. La gráfica muestra la absorbancia a 405nm del sustrato defosforilado por PP2A, expresada como la media±desviación estándar para n=4 muestras por grupo de tratamiento (**p<0,01 y ***p<0,001 respecto a la condición control; ###p<0,01 respecto a la condición fructosa).
El ácido okadaico anula de forma parcial el efecto de la fructosa sobre la expresión de L-PK, pero no modifica la represión de PPARα y sus genes diana. Ensayo de RT-PCR para los genes L-PK (A), PPARα (B) ACO (C) y CYP4A1 (D) en células FaO incubadas en ausencia (condición control, CT), presencia de fructosa 25mM (condición fructosa, FR), o bien en presencia de fructosa 25mM y ácido okadaico 20nM (condición fructosa+ácido okadaico, FR+OA) durante 24h. Las gráficas muestran los niveles relativos de ARNm correspondientes a cada gen, normalizados por los niveles de ARNm del gen control, APRT, expresados como la media±desviación estándar para n=4 muestras por grupo de tratamiento (*p<0,05, **p<0,01 y ***p<0,001 respecto a la condición control; ###p<0,01 respecto a la condición fructosa).
En trabajos previos8,9, nuestro grupo de investigación demostró que la administración de fructosa al 10% en el agua de bebida a ratas provoca hipertrigliceridemia y esteatosis hepática. Estas manifestaciones tenían lugar como consecuencia de la reducción del catabolismo de los ácidos grasos, mediada por una reducción de la actividad transcripcional de PPARα, junto con un incremento de la actividad transcripcional de ChREBP, asociado a un aumento de los niveles de proteína PP2A en estos animales.
Las dietas ricas en hidratos de carbono, como la glucosa y la fructosa, aumentan los niveles del metabolito xilulosa-5-fosfato. La xilulosa-5-fosfato activa la proteína fosfatasa PP2A, que a su vez defosforila a ChREBP, promoviendo su translocación al núcleo, donde activa la transcripción de sus genes diana12,13.
Dado que las líneas celulares son herramientas útiles en el estudio de los mecanismos moleculares por los cuales se produce una determinada alteración, se ha utilizado la línea celular de hepatoma de rata FaO para estudiar la posible implicación de la activación de PP2A en la reducción de la expresión de PPARα y sus genes diana.
Los resultados del presente trabajo muestran que en esta línea celular se reproducen tanto el incremento de los niveles de proteína ChREBP y ARNm de L-PK, como el descenso de la expresión de PPARα y sus genes diana —efectos característicos producidos en el hígado de ratas alimentadas con fructosa en el agua de bebida8— cuando estas células se incuban en presencia de fructosa 25mM, constituyendo un buen modelo para el estudio de las alteraciones metabólicas producidas por la fructosa.
Por otra parte, los resultados obtenidos sugieren que el aumento de la expresión de ChREBP y L-PK observados por el tratamiento con fructosa es parcialmente independiente del incremento en la actividad de la proteína PP2A. Confirmando esta teoría, la incubación de las células FaO con fructosa aumenta la actividad de dicha fosfatasa en un 29%, y la adición de ácido okadaico —conocido inhibidor de PP2A— al medio de cultivo, aunque anula la inducción de la actividad PP2A, solo atenúa parcialmente la inducción de L-PK mediada por la fructosa. Aun en presencia de ácido okadaico, la fructosa continúa aumentando la expresión de L-PK, lo que sugiere la existencia de un mecanismo adicional e independiente de PP2A por el cual la fructosa incrementa la expresión de dicha enzima. En este sentido, la fosfatasa PP2A regula la actividad transcripcional de ChREBP a través de su defosforilación, pero existen otras modificaciones postraduccionales de ChREBP, tales como la acetilación o la acetil-glucosaminación, que aumentan su capacidad de unión al ADN y su actividad transcripcional15,16. De este modo, cabe la posibilidad de que la fructosa promueva dichas modificaciones postraduccionales, contribuyendo a la inducción de L-PK.
Ravnskjaer et al.17 describieron que la glucosa disminuía la expresión de PPARα en células β pancreáticas a través de la activación de PP2A. Sin embargo, con el presente trabajo se demuestra que este no es el mecanismo por el cual la fructosa reduce la expresión de PPARα en células hepáticas, ya que la expresión de dicho factor de transcripción y de sus genes diana no se ve afectada por la adición de ácido okadaico al medio de tratamiento. Recientemente, Boergesen et al.18 han descrito que el aumento de la actividad del factor de transcripción ChREBP conduce a la represión de PPARα en células β pancreáticas. Siguiendo con la hipótesis planteada anteriormente, la fructosa, a través de modificaciones postraduccionales independientes de la activación de PP2A, podría estar aumentando la actividad transcripcional de ChREBP, conduciendo a una menor expresión de PPARα y sus genes diana. No obstante, se requieren más estudios para confirmar que este mecanismo molecular sea el responsable de la represión de PPARα mediada por la fructosa en el hígado.
FinanciaciónEste trabajo ha sido financiado por el MICINN, proyecto SAF2010-15664.
AutoríaM. Alegret y J.C. Laguna contribuyeron al diseño del presente estudio. La obtención de los resultados experimentales fue llevada a cabo por A. Rebollo y N. Roglans. Todos los autores contribuyeron a la interpretación de los resultados obtenidos. La redacción y la revisión del manuscrito fueron realizadas por A. Rebollo, M. Alegret y J.C. Laguna.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Una comunicación referente a esta línea de trabajo, titulada «Papel de PP2A en el control de la expresión de PPARα por fructosa en células FaO de hepatoma de rata», fue presentada en el XXIII Congreso de la SEA (Córdoba 2010) y galardonada con una Mención Especial.